Генетика, 2019, T. 55, № 1, стр. 94-99
Филогенетический анализ для кыргызской породы лошадей по 17 микросателлитным маркерам
Ж. Т. Исакова 1, *, Б. И. Токтосунов 2, В. Н. Кипень 3, **, Л. В. Калинкова 4, Э. Т. Талайбекова 1, Н. М. Алдашева 1, А. Х. Абдурасулов 2
1 Институт молекулярной биологии и медицины
720040 Бишкек, Киргизия
2 Институт биотехнологии национальной академии наук
720071 Бишкек, Киргизия
3 ГНУ "Институт генетики и цитологии" НАН Беларуси
220072 Минск, Республика Беларусь
4 Всероссийский научно-исследовательский институт коневодства
391105 Рязанская область, пос. Дивово, Россия
* E-mail: jainagul@mail.ru
** E-mail: slavakipen@rambler.ru
Поступила в редакцию 08.02.2018
После доработки 03.05.2018
Принята к публикации 18.04.2018
Аннотация
Результаты данного исследования являются первыми в оценке субпопуляционной подразделенности кыргызской породы лошадей. Генотипирование лошадей проводилось по 17 микросателлитным локусам, рекомендованным Международным обществом генетики животных (ISAG). На основании результатов генотипирования по методу AMOVA рассчитаны парные генетические дистанции для лошадей кыргызской породы по отношению к 31 другой породе. Показано, что лошади кыргызской породы генетически наиболее близки к Уэльскому пони и Полукровной лошади.
Изучение генетической структуры популяций аборигенных пород лошадей имеет важное значение для поддержания разнообразия, улучшения селекционной работы и определения места их геногеографического происхождения [1]. Недостаточная изученность генофонда аборигенных пород лошадей Центральной и Восточной Азии не позволяет однозначно дифференцировать географическое место начала процессов доместикации [1, 2]. К настоящему времени численность конного поголовья на территории стран СНГ сократилась более чем в 10 раз по сравнению с началом XX в. [3]. Негативные последствия данного процесса выражены, в первую очередь, в снижении общего уровня генетического разнообразия. В связи с этим на современном этапе в коневодстве актуально проведение исследований генетического потенциала аборигенных пород лошадей для оценки и поддержания высокого уровня их генетического разнообразия с целью недопущения негативных последствий инбридинга и эффекта “бутылочного горлышка” [4]. Сохранение фенотипических породных характеристик, связанных с экстерьерными особенностями, продуктивностью и жизнеспособностью лошадей, возможно лишь при сохранении “специфических генных комплексов и сбалансированной системы генов и аллелей” [5].
Предполагается, что дикий предок современной лошади (Equus caballus) вымер. В качестве предполагаемых диких предков лошади рассматриваются два вида: тарпан (E. ferus) и лошадь Пржевальского (E. przewalskii). Лошадь была одомашнена предположительно около 3700–3100 г. до н. э. на севере современного Казахстана (Ботайская культура) [6]. Однако по результатам молекулярно-генетических исследований, проведенных Vila et al. [7] и Jansen et al. [8], современное представление о доместикации и последующей интрогрессии лошади таково, что, вероятнее всего, имели место многократные независимые случаи одомашнивания.
Экстерьерные характеристики и хозяйственное назначение лошадей кыргызской породы представлены в исследованиях Рипар с соавт. [9]. Начало же исследованиям генетического разнообразия кыргызской породы лошадей с использованием микросателлитных локусов было положено в работе Исаковой и др. [10].
Цель настоящего исследования – провести филогенетический анализ для кыргызской породы лошадей по 17 микросателлитным маркерам относительно 31 другой породы.
Материалом для генетического исследования по 17 STR-локусам (AHT4, AHT5, ASB2, ASB17, ASB23, CA425, HMS1, HMS2, HMS3, HMS6, HMS7, HTG4, HTG6, HTG7, HTG10, LEX3, VHL20) послужили образцы волос с луковицами, взятые у взрослого поголовья кыргызских лошадей, находящихся в высокогорных экспериментальных зонах Джети-Огузского и Тонского районов Иссык-Кульской области (19 животных) и в Кара-Кульджинском районе Ошской области (16 животных) Республики Кыргызстан. Результаты молекулярно-генетического исследования представлены в нашей предыдущей публикации [10]. Статистическую обработку данных проводили с использованием программ GenAIEx v. 6.5 [11], STRUCTURE v. 2.3.4 [12], PAST v. 3.17 [13] и POPHELPER v. 1.0.10 [14]. С использованием GenAIEx v. 6.5 были рассчитаны следующие показатели: среднее число аллелей на локус (Na), число эффективных аллелей (Ne), уровни ожидаемой (He) и наблюдаемой (Ho) гетерозиготности, значение информационного индекса Шеннона (I), коэффициенты FIS и FST, расчет генетических дистанций по методу AMOVA (Analysis of molecular variance) [15]. В программе STRUCTURE v. 2.3.4 по методу Pritchard с соавт. был рассчитан критерий Q, который характеризует принадлежность каждого отдельного животного к соответствующей породе [12]. С использованием веб-приложения POPHELPER v. 1.0.10 произведена графическая интерпретация результатов, полученных в STRUCTURE v. 2.3.4, а также рассчитан параметр ΔK. В программе PAST v. 3.17 выполнено построение дендрограммы на основе расчета генетических дистанций по методу AMOVA.
Современная порода кыргызских лошадей обнаруживает высокий уровень внутрипородной генетической вариабельности [10]. В 17 исследованных микросателлитных локусах было идентифицировано 135 аллелей, в том числе 38 редких аллелей (с частотой встречаемости менее 5.0%). Число аллелей в каждом локусе варьировало от 4 до 13, при среднем значении 7.941 ± 0.525. Для представителей кыргызской породы лошадей показано наличие редких аллелей – D/F/M/O/S для STR-локуса ABS17, G/Q – для ASB23, F/G/I/P – для CA425, I – для HMS1, O/P – для HMS2, Q – для HMS7, N – для HTG и Q – для HTG10. В дополнение к стандартизованной номенклатуре [16] у кыргызской лошади был выявлен не характерный для изученных заводских пород аллель 20 (код в данном исследовании О) для STR-локуса HMS1.
В качестве контрастных популяций для исследования межпопуляционной дифференциации – расчета коэффициента инбридинга субпопуляций относительно всей популяции FST – были использованы 35 групп лошадей, представленных следующими породами: Андалузская (AND), Аппалуза (APP), Арабская чистокровная (ARA), Камполина (CAM), Коннемара пони (CON), Дартмурский пони (DAR), Голландская тяжеловозная (DUT), Фалабелла (FAL), Фелл пони (FEL), Фиордская (FJO), Фризская (FRI), Гронингенская (GRO), Хакнэ (HAC), Хафлингер (HAF), Исландская (ICE), Ирландский коб (IRI), Каспийская (KAS), Польский коник (KON), Липпиццанская (LIP), Лузитано (LUS), Арьежский пони (MER), Мини-лошадь (MIN), Нью-Форест-пони (NEW), Шетлендский пони (SHE), Шайр (SHI), Американская стандартбредная (STA), Теннессийская прогулочная (TEN), Чистокровная верховая (THO), Тинкер (TIN), Полукровная (WAR), Уэльский пони (WEL). Были использованы данные о генотипах 8676 животных, представленных в открытом доступе [16]. Дополнительно был проведен анализ с использованием STRUCTURE v. 2.3.4, направленный на формирование наиболее однородных групп обозначенных выше пород лошадей [16], который позволил нивелировать разницу в объемах выборки для каждой группы лошадей, минимизировав риск потерять потенциально важную генетическую информацию (т.е. объемы выборок были сокращены до 50 голов для выборок ARA, DUT, FAL, FEL, FJO, FRI, HAC, HAF, ICE, IRI, MIN, HE2, STA, WAR1, WAR2, WAR3, WEL1 и WEL2). Результаты проведенного анализа представлены в табл. 1.
Таблица 1. Генетическая характеристика 35 популяций лошадей различных пород по результатам генотипирования 17 STR-локусов (название популяции соответствует принятой классификации в исследовании [16])
| Популяция | N | Na | Ne | I | Ho# | He# | FIS# |
|---|---|---|---|---|---|---|---|
| AND | 45 | 6.529 | 3.372 | 1.375 | 0.668 | 0.659 | –0.017 |
| APP | 54 | 6.882 | 3.859 | 1.491 | 0.713 | 0.713 | 0.007 |
| ARA | 50 | 6.000 | 3.142 | 1.300 | 0.621 | 0.638 | 0.032 |
| CAM | 61 | 6.765 | 3.604 | 1.417 | 0.667 | 0.681 | 0.022 |
| CON | 40 | 6.824 | 4.144 | 1.542 | 0.726 | 0.736 | 0.024 |
| DAR | 22 | 5.353 | 3.227 | 1.312 | 0.631 | 0.657 | 0.027 |
| DUT | 50 | 5.529 | 3.191 | 1.272 | 0.663 | 0.630 | –0.045 |
| FAL | 50 | 6.647 | 3.479 | 1.399 | 0.693 | 0.679 | –0.017 |
| FEL | 50 | 5.882 | 3.796 | 1.421 | 0.715 | 0.692 | –0.033 |
| FJO | 50 | 6.471 | 3.864 | 1.479 | 0.705 | 0.708 | 0.001 |
| FRI | 50 | 4.000 | 2.106 | 0.834 | 0.452 | 0.450 | –0.024 |
| GRO | 27 | 6.059 | 3.884 | 1.483 | 0.752 | 0.727 | –0.032 |
| HAC | 50 | 5.765 | 3.220 | 1.294 | 0.645 | 0.647 | 0.009 |
| HAF | 50 | 5.235 | 3.355 | 1.289 | 0.642 | 0.646 | 0.006 |
| ICE | 50 | 6.529 | 3.867 | 1.457 | 0.692 | 0.688 | –0.013 |
| IRI | 50 | 7.000 | 3.732 | 1.487 | 0.715 | 0.710 | –0.006 |
| KAS | 17 | 5.941 | 4.140 | 1.511 | 0.816 | 0.731 | –0.125 |
| KIR | 35 | 7.941 | 5.080 | 1.751 | 0.761 | 0.779 | 0.022 |
| KON | 51 | 6.235 | 3.705 | 1.422 | 0.708 | 0.690 | –0.030 |
| LIP | 45 | 6.235 | 3.461 | 1.382 | 0.729 | 0.694 | –0.051 |
| LUS | 43 | 6.471 | 4.071 | 1.490 | 0.725 | 0.706 | –0.019 |
| MER | 23 | 5.529 | 3.326 | 1.339 | 0.698 | 0.671 | –0.035 |
| MIN | 50 | 7.059 | 4.062 | 1.535 | 0.718 | 0.714 | –0.005 |
| NEW | 54 | 7.941 | 4.405 | 1.632 | 0.760 | 0.733 | –0.039 |
| SHE1 | 57 | 6.706 | 3.554 | 1.409 | 0.677 | 0.677 | –0.005 |
| SHE2 | 50 | 5.941 | 3.487 | 1.371 | 0.653 | 0.681 | 0.037 |
| SHI | 29 | 5.235 | 3.015 | 1.268 | 0.649 | 0.641 | –0.017 |
| STA | 50 | 6.235 | 3.661 | 1.451 | 0.715 | 0.702 | –0.018 |
| TEN | 23 | 5.176 | 3.204 | 1.295 | 0.679 | 0.653 | –0.039 |
| THO | 55 | 5.118 | 3.542 | 1.336 | 0.688 | 0.685 | –0.008 |
| TIN | 28 | 6.412 | 3.741 | 1.483 | 0.714 | 0.713 | 0.001 |
| WAR1 | 50 | 6.882 | 4.248 | 1.574 | 0.749 | 0.748 | 0.001 |
| WAR2 | 50 | 7.647 | 4.463 | 1.636 | 0.740 | 0.751 | 0.012 |
| WAR3 | 50 | 6.706 | 4.097 | 1.531 | 0.743 | 0.730 | –0.015 |
| WEL1 | 50 | 7.118 | 4.225 | 1.582 | 0.712 | 0.738 | 0.038 |
| WEL2 | 50 | 7.529 | 4.218 | 1.621 | 0.718 | 0.746 | 0.039 |
Примечание. N – число животных в группе, Na – среднее число выявленных аллелей на локус (No of Different Alleles per Locus), Ne – число эффективных аллелей (No of Effective Alleles), I – индекс разнообразия Шеннона (Shannon’s Information Index), Ho – наблюдаемая гетерозиготность (Observed Heterozygosity), He – ожидаемая гетерозиготность (Expected Heterozygosity), FIS – индивидуальный индекс фиксации (Fixation Index); # среднее для 16 STR-локусов (кроме LEX3).
Таким образом, нами были получены генотипы по 17 STR-локусам для 35 лошадей кыргызской породы. Дополнительно сформированы 35 групп для 31 породы лошадей общим объемом 1574 животных (26 758 генотипов).
При расчете значения показателя FST (по алгоритму AMOVA) для двух географически разобщенных групп кыргызских лошадей – группа № 1 из Джети-Огузского и Тонского районов Иссык-Кульской области (19 животных), группа № 2 из Кара-Кульджинского района Ошской области (16 животных) – не было найдено статистически значимых различий. Дальнейшие расчеты при оценке показателей Na, Ne, He, Ho, I и FST для сравнения группы лошадей кыргызской породы с 35 группами (31 порода лошадей), представленными в исследовании Van de Goor et al. [16], проводились, исходя из данного положения.
Из данных табл. 1. видно, что для кыргызской породы лошадей cреднее число выявленных аллелей на локус для 17 микросателлитов является одним из наибольших среди исследованных групп и составляет 7.941 ± 0.525. Аналогичное значение данного показателя отмечено лишь для одной группы из оставшихся 35, а именно – для породы Нью-Форест-пони (NEW) и составляет 7.941 ± ± 0.621 аллелей. Для показателя Ne, отражающего число эффективных аллелей, расчетное значение для группы KIR составило 5.080 ± 0.364 и оказалось также выше, чем для оставшихся групп, включая группу NEW (4.405 ± 0.380 аллеля). Таким образом, для исследуемой выборки лошадей кыргызской породы показаны высокие значения показателей Na и Ne, это может свидетельствовать о значительном генетическом разнообразии данной породы в сравнении с другими, анализируемыми в рамках настоящего исследования, породами.
Также показано, что для кыргызской породы лошадей характерны максимальные, усредненные по 16 STR-локусам, значения ожидаемой гетерозиготности (He), в сравнении с оставшимися 35 группами: показатель ожидаемой гетерозиготности составил 0.779 ± 0.017, наблюдаемой гетерозиготности (Ho) – 0.761 ± 0.029.
Среднее арифметическое значение индекса Шеннона, отражающего сложность структуры сообщества, для группы KIR также оказалось наиболее высоким среди исследованных групп и составило 1.751 ± 0.066. Данный факт свидетельствует о средней сложности структуры популяций исследованных кыргызских лошадей.
Таким образом, по результатам оценки среднего числа аллелей на локус (Na), числа эффективных аллелей (Ne), уровней ожидаемой (He) и наблюдаемой (Ho) гетерозиготности, а также значения информационного индекса Шеннона (I) можно констатировать о высоких значениях внутрипопуляционного генетического разнообразия кыргызской породы лошадей и значительном генетическом потенциале данной породы.
Анализ субпопуляционной структуры исследуемых 36 групп лошадей, включая группу лошадей кыргызской породы, был проведен с использованием программы STRUCTURE v. 2.3.4 [12] и веб-приложения POPHELPER v. 1.0.10 [14].
Для некоторых групп лошадей, например Коннемара пони (CON), Гронингенской лошади (GRO), Каспийской лошади (KAS), Арьежского пони (MER), Нью-Форест-пони (NEW), Теннессийской прогулочной лошади (TEN), Полукровной лошади (WAR), Уэльского пони (WEL), а также для Кыргызской лошади (KIR) характерна весьма гетерогенная структура исследуемой выборки, вплоть до значения K = 10. Отношение ΔK/K соответствовало значениям 4.595/4 и 4.640/8 для CON, 1.107/6 – GRO, 1.694/4 и 2.074/10 – KAS, 1.797/4 – MER, 3.191/3 и 2.814/8 – NEW, 4.925/3 – TEN, 1.540/4 – WAR1, 2.065/6 и 2.601/8 – WAR2, 2.126/3 – WAR3, 12.835/5 и 8.642/8 – WEL1, 2.941/5 и 3.356/9 – WEL2, 6.228/8 – KIR. Данный факт может свидетельствовать, в первую очередь, о сложной и разнообразной схеме скрещивания при породообразовании обозначенных групп, а также о наличии значительного генетического разнообразия, которое, в свою очередь, в эволюционном контексте ассоциировано со способностью адаптироваться к условиям окружающей среды, а также с продолжающимися процессами эволюции пород.
На основе рассчитанных в GenAIEx v. 6.5 парных генетических дистанций FST (по методу AMOVA) была построена дендрограмма в программе PAST v. 3.17 (рис. 1). Оценка надежности ветвей филогенетического дерева проведена с использованием бутстрэп-анализа [17] с использованием 1000 случайных выборок.
Рис. 1.
Дендрограмма на основе матрицы попарных генетических дистанций FST между изучаемыми группами лошадей (по методу AMOVA).
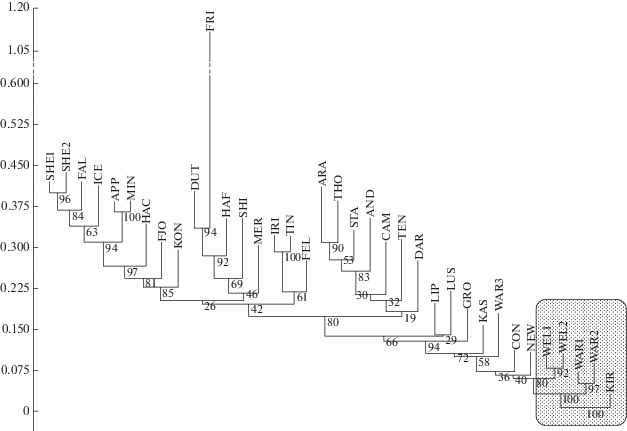
При кластеризации изучаемых групп лошадей, основанной на рассчитанных значениях генетических дистанций методом AMOVA (рис. 1), наблюдаются некоторые закономерности. Из дендрограммы видно, что группа KIR генетически наиболее близка к группам WEL (Уэльский пони) и WAR (Полукровная лошадь) – генетическое расстояние (отн. ед.) равно 0.028 (WEL2/KIR), 0.0286 (WEL1/KIR), 0.0202 (WAR1/KIR) и 0.0205 (WAR2/KIR). В целом группа KIR располагается в субкластере с группами WAR и WEL, для которых ранее показаны высокие значения среднего числа аллелей на локус (Na), числа эффективных аллелей (Ne), уровней ожидаемой (He) и наблюдаемой (Ho) гетерозиготности (табл. 1).
Таким образом, результаты проведенного нами анализа, направленного на оценку аллелофонда и генетической характеристики кыргызской породы лошадей по микросателлитным маркерам, свидетельствуют о значительном генетическом потенциале и высоком генетическом разнообразии аборигенной для Кыргызской Республики породы лошадей. Однако сохранение данного разнообразия в отдаленной перспективе может быть достигнуто только путем использования в селекционной схеме принципов рационального и эффективного природопользования.
Список литературы
Воронкова В.Н. Оценка генетического разнообразия лошадей Саяно-Алтайского региона с использованием ядерных и митохондриальных ДНК маркеров: Автореф. дис. … канд. биол. наук. М.: ИОГен, 2012. 46 с.
Warmuth V., Eriksson A., Bower M.A. et al. European domestic horses originated in two holocene refugia // PLoS One. 2011. V. 30. P. 1–7 (e18194). doi 10.1371/ journal.pone.0018194
Ливанова Т.К., Ливанова М.А. Все о лошади. М.: АСТ-ПРЕСС СКД, 2002. 384 с.
Molecular genetic characterization of animal genetic resources // FAO Animal Production and Health Guidelines. № 9. FAO. Publ. in 2011. 85 p.
Столповский Ю.А. Концепция и принципы генетического мониторинга для сохранения in situ пород доместицированных животных // С.-х. биология. 2010. № 6. С. 3–8.
Olsen S.L. Early horse domestication on the Eurasian steppe // Documenting Domestication: New Genetics and Archaeological Paradigms / Eds Zeder M.A., Emshwiller E., Smith B.D., Bradley D.G. California, USA: Univ. California Press, 2006. P. 245–269.
Vilà C., Leonard J.A., Götherström S. et al. Widespread origins of domestic horse lineages // Science. 2001. V. 291(5503). P. 474–477. doi 10.1126/science.291.5503.474
Jansen T., Foster P., Levine M.A. et al. Mitochondrial DNA and the origins of the domestic horse // Proc. Natl Acad. Sci. USA. 2002. V. 99(16). P. 10905–10910. doi 10.1073/pnas.152330099
Рипар Ж., Уатто С., Перез С. Кыргызская лошадь, сохранение и использование скачки на выносливость и экотуризм. Практическое указание / Перевод А. Илиязова. Бишкек, 2007. 32 с.
Исакова Ж.Т., Токтосунов Б.И., Кипень В.Н. и др. Генетический портрет кыргызской лошади // Коневодство и конный спорт. 2018. № 1. С. 21–23.
Peakall R., Peter E. Smouse GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update // Bioinformatics. 2012. V. 28. P. 2537–2539. doi 10.1093/bioinformatics/bts460
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155(2). P. 945–959.
Hammer Q., Harper A.T., Ryan P.D. PAST: Paleontological statistics software package for education and data analysis // Palaeontol. Electronica. 2001. V. 4. P. 1–9.
Francis R.M. POPHELPER: an R package and web app to analyse and visualize population structure // Mol. Ecol. Res. 2017. V. 17. P. 27–32. doi 10.1111/1755-0998.12509
Excoffier L. Analysis of molecular variance inferred from metric distances among dna haplotypes: Application to human mitochondrial dna restriction data / Eds Excoffier L., Smouse P.E., Quattro J.M. // Genetics. 1992. V. 131. P. 479–491.
Van De Goor L.H.P., Panneman H., Van Haeringen W.A. A proposal for standardization in forensic equine DNA typing: allele nomenclature for 17 equine-specific STR loci // Anim. Genet. 2010. V. 41(2). P. 122–127. doi 10.1111/j.1365-2052.2009.01975.x
Zharkikh A. Statistical properties of bootstrap estimation of phylogenetic variability from nucleotide sequences. I. Four taxa with a molecular clock // Mol. Biol. Evol. 1992. V. 9(6). P. 1119–1147. doi 10.1093/oxfordjournals.molbev.a040782
Дополнительные материалы отсутствуют.


