Генетика, 2019, T. 55, № 1, стр. 28-39
Мобильные элементы в эволюции регуляторных сетей
С. А. Пирогов 1, *, О. Г. Максименко 1, П. Г. Георгиев 1
1 Институт биологии гена Российской академии наук
119334 Москва, Россия
* E-mail: biologsp@gmail.com
Поступила в редакцию 16.01.2018
После доработки 26.03.2018
Принята к публикации 05.03.2018
Аннотация
В последнее десятилетие идет активное исследование взаимоотношений популяции мобильных элементов (МЭ) и остального генома. Выявляются случаи использования регуляторных последовательностей в составе МЭ для регуляции различных генов и организации генома. Такими регуляторными последовательностями могут быть сайты связывания транскрипционных факторов, энхансеры и инсуляторы. МЭ могут содержать промоторы, которые могут быть одомашнены при замещении ими исходных промоторов генов или при формировании de novo длинных некодирующих ДНК. Кроме того, известны многие примеры одомашнивания белков, кодируемых МЭ, как-то транспозаз, протеаз и белков Gag. Данный обзор освещает роль МЭ в эволюции регуляторных сетей и определяющие эту роль принципы эволюции МЭ.
Одним из фундаментальных вопросов биологии является соотношение между организменной и геномной сложностью живых организмов. Геном прокариот обладает высокой генной плотностью, в отличие от генома большинства эукариот, которые, однако, сложнее организованы [1–3]. В биологическом контексте эта тенденция находит выражение в накоплении в геномах эукариот условно нефункциональных элементов: повторов и мобильных элементов. После расшифровки достаточного числа геномов были обнаружены два парадоксальных факта: организменная сложность не соотносится ни с величиной генома (C-парадокс [4]), ни с числом генов (N-парадокс [5]). Это стало причиной двух неоднозначных следствий. Во-первых, несоответствие сложности и размера генома позволило предположить, что значительная часть генома никак не влияет на развитие и жизнедеятельность организмов. Во-вторых, набор генов также не объясняет сложность организмов. Это привело к поиску других определяющих факторов.
У всех эукариот и многих прокариот в геноме обнаружены эгоистичные элементы, способные к изменению своей локализации и потому получившие название мобильных, а их совокупность была названа мобиломом. По мере исследования функциональных элементов генома выяснялось, что теми или иными функциями обладает большая часть генома, в том числе и многие мобильные элементы [6]. В то же время обнаружение огромного числа регуляторных элементов генома и транскрипционных факторов, а также открытие гистонового кода, факторов ремоделирования хроматина и транскрипции, охватывающей подавляющую часть генома, серьезно расширили наши представления о сетях регуляции генов, что позволило говорить о них как об источниках организменной сложности [7]. Открытие обширной вовлеченности мобилома в регуляторные сети стало поводом обратить внимание на значительную часть генома, которую раньше называли “мусорной” [8–10]. Возможно, мобилом является важным, хотя и не единственным источником организменной сложности. Однако число мобильных элементов (МЭ) и их относительная доля в геноме не имеют прямого соотношения со сложностью. Можно предположить, что мобильные элементы являются не только непосредственными участниками регуляторных сетей, но и модуляторами эволюционного развития этих сетей.
Настоящий обзор посвящен рассмотрению роли мобилома в формировании регуляторных сетей и обеспечении таких фундаментальных свойств биологических систем, как сложность [11], эволюционируемость [12], устойчивость [13, 14] и неадаптивность [15–17], в свете эволюционных законов и принципов, закладываемых экологией популяций.
МЭ И ГЕНОМНОЕ ОКРУЖЕНИЕ
Мобильные элементы эукариот разделяют на два основных класса: ретротранспозоны и ДНК-транспозоны. Ретротранспозоны распространяются путем “копирования и вставки” в ходе транскрипции с последующим использованием обратной транскриптазы, а ДНК-транспозоны – путем “вырезания и вставки” при помощи транспозаз. Поэтому число ретротранспозонов растет при каждом акте транспозиции, в отличие от ДНК-транспозонов, копийность которых изменяется только при репликации генома. К ретротранспозонам относятся: LTR-транспозоны (например, эндоретровирусы), имеющие промотор PolII и кодирующие 5–6 белков; LINE, имеющие промотор PolII и две рамки считывания; SINE, имеющие промотор PolIII и не кодирующие белки. К ДНК-транспозонам относятся классические TIR-транспозоны, а также Helitron и Maverick [18]. Часто транспозоны теряют кодирующую часть и получаются укороченные формы. Для LTR-транспозонов они получили название solo-LTR [19], а для TIR-транспозонов – MITE [20].
Согласно общепринятой оценке количество транспозонов в геноме человека составляет около 46%, а для некоторых растительных геномов – почти 90% [21, 22]. Биоинформатические подходы позволили выявить значительное количество новых повторяющихся элементов и участков ДНК, произошедших от них. Это дало возможность говорить о том, что более двух третей генома человека произошли из транспозонов [23]. Однако за длительный период эволюции подобные участки могли утратить признаки происхождения из МЭ, поэтому верхняя граница возраста ретротранспозона, выявляемого биоинформатичскими методами, составляет около 200 млн лет [24].
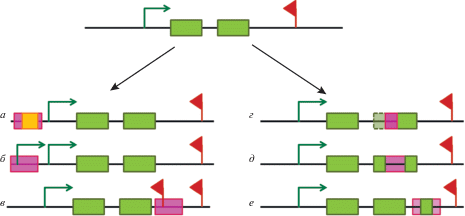
МЭ играют определенную роль в формировании и эволюции генов и их участков: промоторов, экзонов, интронов, сайтов полиаденилирования и сайтов сплайсинга (рис. 1). Примерно 90% генов человека содержат МЭ по меньшей мере в одном из интронов [25]. Существует теория, согласно которой интроны эукариот произошли из самосплайсирующихся интронов II группы [26], которые также можно отнести к МЭ [27]. Формирование интронов в ряде эукариотических ветвей продолжается и по сей день, в том числе за счет транспозонов (например, формирование интронных элементов у зеленых водорослей) [28] (рис. 1д). Процесс формирования новых экзонов (экзонизация) в результате встраивания МЭ в кодирующие участки гена хорошо изучен у млекопитающих (например, произошедший из MIR экзон в гене ZF639 [29]) [30] (рис. 1е). Кроме этого, в процессе внедрения МЭ в кодирующую часть гена часто формируются новые сайты сплайсинга и сайты полиаденилирования, позволяющие транскрибировать альтернативные сплайс-формы (одним из наиболее изученных примеров образования и новых сайтов сплайсинга, и альтернативной терминирующей области является ген ATRN [31]) [32, 33] (рис. 1в, 1г). Однако функциональная значимость сплайс-вариантов гена, индуцированных внедрением МЭ, остается под вопросом. Многие промоторные [34] и цис-регуляторные участки [35] также произошли из МЭ (классическим примером является альтернативный промотор в гене Agouty у мышей, отвечающем за окраску шерсти [36]) (рис. 1в, 1б). Так, существуют оценки, согласно которым более 30% сайтов старта транскрипции человека лежат в пределах МЭ [37]. Особо следует выделить формирование тканеспецифичных альтернативных промоторов из транспозонов, в частности в ооцитах, плаценте, плюрипотентных и эритроидных клетках (см. в обзоре [38]).
Рис. 1.
Роль МЭ (фиолетовый прямоугольник) в эволюции и формировании генов. а – образование цис-регуляторного участка (энхансера, сайленсера или инсулятора, желтый квадрат); б – образование альтернативного промотора, в частности тканеспецифичного (обозначен зеленой стрелкой); в – образование альтернативного сайта полиаденилирования и терминирующей области (обозначен красным флажком); г – образование альтернативного сайта сплайсинга; д – образование нового интрона; е – образование нового экзона (экзонизация). Экзоны показаны зелеными прямоугольниками. Цветной рисунок см. в электронной версии.
ЭВОЛЮЦИЯ, ОТБОР И ПОПУЛЯЦИОННАЯ ГЕНЕТИКА МЭ
Происхождение МЭ в эукариотическом геноме до сих пор остается неясным. Предположительно многие группы МЭ были унаследованы от архейного предка и эндосимбионтов; например, ретротранспозоны LINE произошли от интронов II группы [39]. В то же время процесс приобретения ретротранспозонов из ретровирусов происходил на протяжении всей эволюции эукариот [40], а SINE-элементы произошли de novo из 7SL РНК или тРНК [41].
МЭ получили возможность интенсивной экспансии у эукариот, в особенности многоклеточных, благодаря малому размеру их популяций по сравнению с прокариотами и, следовательно, слабому отбору против МЭ. В подтверждение этого популяционные исследования показывают, что в малочисленных популяциях количество транспозонов в геномах достоверно выше по сравнению с большими популяциями [42]. Слабый очищающий отбор и интенсивный дрейф в малых популяциях практически снял ограничения на геном по размеру [43]. С другой стороны, приобретение митохондрий эукариотами позволило энергетически поддерживать распространяющуюся ДНК [44]. В результате в геноме эукариот сформировались две популяции генетических элементов: оригинальные гены с регуляторными элементами и мобильные элементы. Также любопытной особенностью транспозонов является периодичность активности: после длительных периодов низкой активности наступают периоды “вспышек” транспозиций [45, 46]. Подобные пики активностей транспозонов сопряжены с резким увеличением числа мутаций, что описано для ряда сельскохозяйственных растений [47].
Популяции МЭ в геноме существенно неоднородны как по составу, так и по активности. Вместе с тем принципы отбора МЭ имеют некоторое сходство с общими принципами популяционной генетики: так, под более сильным отбором находятся транспозоны, представленные в большем числе [48]. Неактивные МЭ, неспособные к увеличению численности и перемещению, находятся под действием отбора и дрейфа только на уровне популяции организмов. Активные МЭ представляют собой популяцию, в отношении которой действует внешний отбор, направленный на подавление активности элементов. При наличии такого отбора в популяции D. melanogaster преобладает делеция МЭ над их инсерцией, в то время как в случае отсутствия внешнего отбора частота инсерций значительно превосходит частоту делеций [48, 49]. При этом внутри самой популяции МЭ действует внутренний отбор, который направлен против недостаточно приспособленных элементов [46]. Приспособленностью обычно считают способность элемента сохранять активность в любых участках генома. МЭ могут избирательно встраиваться вблизи активных генов (например, R1- и R2-транспозоны интегрируются в локус 28S рДНК [50]) или внутри неактивных областей генома (например, Ty5 ретротранспозон S. cerevisiae [51]) [52, 53]. Сейчас стали известны многие механизмы выбора сайта интеграции (например, место интеграции ретротранспозона Ty3 определяется через взаимодействие с TFIIIB [54]) [55]. Внутри МЭ могут сохраняться и формироваться новые участки связывания белков – как подавляющих, так и активирующих их транскрипцию. Впоследствии такие сайты связывания могут быть использованы геномом и могут участвовать в регуляции активности близлежащих генов.
Параллельно внешний отбор со стороны генома “хозяина” вырабатывает способы подавления излишней экспансии мобилома за счет инактивации. Одно из ярких проявлений внешнего отбора – накопление специфических механизмов, подавляющих активность МЭ в герминальных и эмбриональных клетках: система piRNA-сайленсинга [56], система эпигенетического маркирования [57], метилтрансферазы [58] и KRAB-ZFP белки. В генеративных и эмбриональных тканях активные МЭ эволюционировали в направлении наиболее эффективной транспозиции [59]. Вместе с тем МЭ в соматических тканях преимущественно неактивны [53].
Любопытно, что транспозиционная активность МЭ может быть полезна для генома: в эмбриональных клетках активные ретротранспозоны приводят к выработке интерферонных белков, что увеличивает шансы успешно противостоять потенциальной вирусной инфекции [60]. Регуляция плюрипотентного статуса клетки за счет регуляторных элементов и промоторов, произошедших из транспозонов, – другой пример одомашнивания МЭ [61]. Проявлением прямой взаимосвязи эволюции МЭ и остального генома является “гонка вооружений” между появлением новых семейств МЭ и эволюцией белков репрессии. Так, вспышки активности транспозонов совпадают с активной дупликацией и дивергенцией KRAB-ZFP белков [62]. У семейства мышиных интенсивная транспозиция привела к появлению новой метилтрансферазы, активной только в герминальных клетках [58].
Отдельного упоминания заслуживают эволюционные и экологические взаимосвязи между автономными мобильными элементами и неавтономными, амплифицирующимися за счет автономных. Показано, что характер этих отношений может описываться подобно модели “хищник–жертва” Вольтерра–Лотки [46]. Неоднозначны и представления о взаимоотношениях ДНК-транспозонов и ретротранспозонов. Например, гомологи белка CENP-B у дрожжей, произошедшие из транспозаз ДНК-транспозонов, участвуют в репрессии ретротранспозонов [63].
Стоит отметить, что при воздействии внешних стрессовых факторов часто заметно увеличивается активность МЭ [64]. Активность транспозонов проявляется при воздействии высоких и низких температур, засолении растений, появлении инфекционного агента, повреждении ДНК, оксидативном стрессе и ингибировании трансляции [47, 65]. Кроме того, показано, что происходит активация транспозиции при стрессе в генеративных клетках растений. Остается неясным, стоит ли считать такой процесс исключительно эгоистичной попыткой транспозона воспользоваться отключением защищающих геном механизмов или такая реакция закреплена и внешним отбором как индукция ненаправленного поиска адаптации.
МОБИЛОМ И РЕГУЛЯТОРНЫЕ СЕТИ ГЕНОМА
Ретротранспозоны содержат сайты связывания многих клеточных транскрипционных факторов: c-Myc, p53, Oct4, Nanog и другие [66–68]. По последним данным, от 20 до 40% всех сайтов связывания транскрипционных факторов расположены в МЭ, причем две трети сайтов являются тканеспецифичными и большинство – видоспецифичными [69]. Накопление таких сайтов может быть как результатом внутреннего отбора, так и неадаптивным нейтральным процессом. В ряде случаев положительное влияние представляется очевидным: например, Oct4 и Nanog позволяют транспозону оставаться активным в эмбриональных стволовых клетках [70]. На данный момент с помощью биоинформатических и экспериментальных методов выявлены энхансеры, PRE-элементы и инсуляторы, произошедшие из МЭ или находящиеся в их составе [66, 71–74]. Недавно был проведен широкомасштабный биоинформатический поиск инсуляторов в МЭ на CD4+ T-клетках, показавший их большое количество и, неожиданно, почти полное отсутствие перекрываний с сайтами связывания инсуляторного белка CTCF [75]. Также в ходе инактивации транспозонов часто затрагиваются соседние гены, что позволяет регулировать их активность [76, 77].
Существуют два пути формирования регуляторных областей из МЭ: de novo формирование и использование уже существующего регуляторного участка [78]. De novo формирование промотора или энхансера в МЭ может происходить в результате процесса, названного “epistatic capture”, когда в ходе эволюции последовательно формируются и закрепляются все более аффинные сайты связывания транскрипционных факторов (ТФ). Готовые участки связывания ТФ, необходимые для транскрипции собственных ретровирусных РНК, также включаются в регуляторные сети генома [79]. При этом в процессе распространения транспозона совокупность сформировавшихся в нем регуляторных элементов функционирует единообразно для всех генов, которые оказались под их влиянием [8]. Кроме того, выявлена закономерность, согласно которой на более древних транспозонах располагается больше энхансеров и сайтов связывания ТФ по сравнению с молодыми [80].
Необходимо признать, что в процессе эволюции генерация регуляторных элементов в основном происходит неадаптивно: формирование значительного числа сайтов связывания ТФ может быть объяснено дезаминированием метилированных участков CpG, “проскальзыванием” динуклеотидных повторов ДНК-полимеразой или результатом работы репарационных систем [81–83]. Некодирующая ДНК, произошедшая из мобилома, большую часть времени лишена эволюционных ограничений и подчинена принципам нейтральной эволюции, поэтому она играет принципиально важную роль в появлении в геноме новых особенностей [84, 85].
В эволюции регуляторных сетей важную роль играет способность мобильных элементов активироваться в стрессовых условиях. Случайное повышение активности МЭ может приводить к заметным изменениям в геноме: от полногеномных перестроек до локальных мутаций [46]. Неблагоприятные эффекты возросшей активности МЭ имеют для популяции меньшее значение по сравнению с возможностью создать геномный ландшафт, соответствующий адаптированному к новым условиям среды фенотипу [84]. Возможность стресс-опосредованной и быстрой перестройки регуляторной сети с помощью МЭ играет важную роль в эволюции растений [47, 86, 87]. Среди механизмов сайленсинга выделяется группа чрезвычайно разнообразных и быстро эволюционирующих белков KRAB-ZFP, специфично связывающихся с МЭ [88, 89]. Данная система может иметь два различных следствия, интересных в контексте регуляторного эффекта: экзаптация сайленсинга или избегание сайленсинга. Последнее достигается за счет перехода МЭ в форму solo-LTR, не распознаваемую белками KRAB-ZFP, что позволяет одомашнивать позитивные регуляторные элементы в составе МЭ. Экзаптация сайленсинга подразумевает возможность выключения генов на различных этапах онтогенеза и в различных тканях за счет инактивации близлежащего эндоретровируса [90, 91]. В таком случае ретротранспозон может стать элементом регуляторной сети, способным как связывать регуляторные белки, так и быть во включенном/выключенном состоянии благодаря функционированию KRAB-ZFP белков. В общем случае вырисовывается эволюционная модель, обладающая элементами “гонки вооружений” между KRAB-белками и мобиломом, внутренней конкуренцией транспозонов, положительным отбором одомашненных элементов и деградацией неприспособившихся элементов [92, 93].
Нередко сами транспозоны становятся непосредственными участниками репрессии других МЭ – таким способом популяция МЭ сама обеспечивает геном механизмами подавления их вредной повышенной активности. Так, в ходе эндогенизации ретровирусов часто наступает стадия, когда одна из копий становится источником микроРНК, подавляющей активность других копий, что ведет к их постепенной деградации [94]. Кроме того, недавно у растений была обнаружена особая группа регуляторных РНК – эпигенетически активируемые малые интерферирующие РНК (easiRNA), образующиеся также из активных транспозонов в пыльце, которые подавляют активность МЭ своего же класса [95]. Это одновременно демонстрирует и эффект участия транспозонов в собственной репрессии, и действие систем репрессии только в генеративных тканях. В свою очередь, малые некодирующие РНК, в частности piRNA и endo-siRNA, происходящие из МЭ и подавляющие их экспрессию, могут влиять на экспрессию обычных генов (в том числе на широко известные гены Nanos и Oscar) [96].
МЭ И ЭВОЛЮЦИЯ СЛОЖНОСТИ
На данный момент существует значительное число работ, посвященных выявленным функциям МЭ в регуляции генов, вовлеченных в эмбриональное развитие, плюрипотентность, иммунитет и т.д. [38, 97–101]. Необходимо отметить, что элементы, сохранившие мобильность и содержащие ТФ-связывающие сайты, напрямую связаны с важнейшей особенностью многоклеточных организмов: формированием многообразия в ходе раннего онтогенеза. Особо интересным примером является ремоделирование генома за счет перемещения и распространения многочисленных сайтов связывания белка CTCF, играющего важную роль в регуляции экспрессии генов в ходе эмбриогенеза и участвующего в организации архитектуры генома [72].
МЭ и вызванные ими перестройки регуляторных сетей играют важную роль в эволюции сложности, причем данное явление прослеживается на разных уровнях. На уровне крупнейших эволюционных переходов они, вероятно, важны в эукариогенезе: бактериальные интроны II группы стимулировали образование сплайсосомы, ядерной оболочки, а также появление линейных хромосом и альтернативного сплайсинга [102]. Более того, возможно, они участвовали в эволюции многоклеточности и появлении социальных организмов [103]. На уровне эволюции млекопитающих важно отметить крупномасштабные перестройки регуляторных сетей, совпавшие с отделением млекопитающих от рептилий, возникновением эутериев и появлением приматов [104]. Постепенно начинает проясняться роль коэволюции МЭ и KRAB-ZFP белков в формировании регуляторных хабов у рептилий и млекопитающих, а, следовательно, и их роль в эволюции регуляторных сетей [105].
Более частным примером участия МЭ в перестройке регуляторных сетей является цефализация у головоногих: внезапная экспансия SINE-элементов привела к изменению паттернов взаимодействующих генов, что позволило сформировать высокоразвитую нервную систему [106]. В мозге человека остаются активными ретротранспозоны семейства L1, что необычно для соматических тканей [107]. Также известно, что некоторые piRNA экспрессируются в нейронах Aplysia, грибовидных телах Drosophila и мозге мышей. Нарушение биогенеза этих РНК приводит к нарушениям в запоминании у Aplysia и Drosophila и к гиперактивности мышей [108–110]. Отдельной областью крупномасштабной регуляции экспрессии генов является дозовая компенсация у животных, когда целая половая хромосома либо инактивируется у гомогаметного пола (как у млекопитающих), либо активируется у гетерогаметного пола (как у плодовой мушки). Несмотря на принципиально разные молекулярные механизмы дозовой компенсации, для них характерна связь с МЭ. В случае млекопитающих было показано происхождение из эндогенного ретровируса А-повторов длинной нкРНК Xist, играющих принципиальную роль в ее репрессивной активности за счет взаимодействия с PRC2-комплексом [111, 112]. В случае с дозовой компенсацией у Drosophila miranda ситуация еще более любопытная – значительная часть сайтов связывания комплекса дозовой компенсации на новой Х-хромосоме возникла в ходе небольшой делеции в МЭ [113].
ДЛИННЫЕ НЕКОДИРУЮЩИЕ РНК И МЭ
Множество длинных некодирующих РНК (lncRNA) перекрываются с эндоретровирусами или повторяющимися элементами (LINE и SINE): до 80% всех lncRNA человека содержат последовательности МЭ, из них более 10% имеют промоторы, произошедшие из МЭ. Это позволяет предположить появление lncRNA в результате формирования промоторов de novo или с использованием существующих промоторов МЭ [92, 114]. С другой стороны, увеличение длины транскрипта также может происходить за счет близлежащих МЭ. Так, около 40% всех последовательностей lncRNA происходят из МЭ [115]. Показано, что промоторы lncRNA намного более эволюционно пластичны, чем промоторы белок-кодирующих генов [114]. Кроме этого, в большинстве lncRNA МЭ формируют экзоны – в среднем 20% всех экзонов возникли из МЭ [114], которые могут участвовать во взаимодействии с определенными белками хроматина или в регуляции экспрессии РНК (например, за счет специфичной РНК-интерференции). Особенно интересен вариант, когда lncRNA содержит участок, связывающий определенные микроРНК, что позволяет контролировать их влияние на некоторый ген [116]. Другим вариантом является генерация микроРНК против МЭ, находящихся в том числе внутри генов, что позволяет регулировать экспрессию этих генов [117]. Эта концепция получила название RIDL-гипотезы (Repeat Insertion Domains of LncRNAs), которая предполагает множество возможных функций, определяемых МЭ в составе lncRNA; примеры таких функций описаны для многих lncRNA (в том числе Xist, ANRIL, FENDRR) [118]. Наконец, показано участие повторяющихся элементов (особенно Alu) и некоторых других транспозонов в формировании кольцевых РНК (circRNA), чья роль в различных клеточных процессах сейчас активно исследуется [119, 120].
Посредством lncRNA мобильные элементы могут оказывать влияние на широкий круг генов в тех случаях, когда требуется очень тонкая настройка уровня экспрессии (эмбриогенез и поддержание плюрипотентности) [121]. Примером такой РНК может служить linc-RoR, играющая важную роль в репрограммировании фибробластов в плюрипотентные клетки и состоящая из различных ретротранспозонов [122]. Эта РНК также имеет промотор, произошедший из семейства МЭ HERVH, что характерно для многих lncRNA, экспрессирующихся в плюрипотентных клетках [123]. Однако некоторые функционально важные lncRNA, например HOTAIR, не содержат МЭ в своей последовательности или промоторе. Особое значение имеет исследование связи рака и экспрессии определенных lncRNA, произошедших из МЭ, что подробно описано в обзоре [98].
Эволюционная пластичность регуляторных сетей серьезно ограничена тем фактом, что экспрессия многих генов находится под сильным отбором. Изменение уровня или профиля экспрессии такого гена в процессе перестройки регуляторной сети (например, с участием МЭ) приведет с большой вероятностью к снижению приспособленности и, как следствие, к снижению выживаемости организма. Таким образом, вовлечение МЭ в непосредственную эволюцию регуляторных сетей требует их высокой устойчивости, при которой большинство радикальных изменений обладают относительно незначительным эффектом на экспрессию жизненно важных генов. Тем не менее возможность радикальной перестройки с помощью МЭ также играет важную роль в решении проблемы градуализма, когда постепенным движением по ландшафту приспособленности нельзя перейти с одного пика приспособленности на другой [65].
Проблему негативного влияния радикальных перестроек регуляторных сетей может нивелировать появление промежуточного уровня регуляции. То есть промежуточный регулятор повышает устойчивость регуляторной сети. Таким промежуточным элементом – буфером регуляторных действий – могут являться длинные некодирующие РНК, представленные в большом количестве в геноме эукариот и обладающие широчайшим спектром функциональности [124]. Другим примером буферов регуляторных сетей могут быть скрытые энхансеры [125].
Существует много противоречивых данных относительно очищающего отбора lncRNA; показано, что у человека для подавляющего большинства lncRNA очищающий отбор отсутствует, в отличие от D. melanogaster [126]. В любом случае обычно гены, кодирующие lncRNA, находятся под более слабым отбором, чем гены, кодирующие белки, но под большим, чем нейтральные области [115].
Таким образом, формирование регуляторных участков на транспозонах оказывает влияние на экспрессию lncRNA, которые, в свою очередь, оказывают весьма слабое опосредованное и неадаптивное влияние на экспрессию генов, что в итоге приводит к повышению устойчивости регуляторной сети. Это объясняет, с одной стороны, огромное число lncRNA в геномах и, с другой стороны, сложности в выявлении их функций.
ЭКЗАПТАЦИЯ ПРОТЕОМА МЭ
Кроме экзаптации регуляторных последовательностей из МЭ, необходимо сказать о процессе одомашнивания закодированных в транспозонах белков. Одомашниванию подверглись представители различных семейств транспозонов: так, например, из pogo-семейства произошли белки CENP-B и bab1, из Ac-семейства – BEAF-32, из Hsmar1-семейства – SETMAR, из MuDR – FHY3. При этом почти все одомашненные белки содержат в своем составе каталитический кор и ДНК-связывающие домены, произошедшие из транспозаз. Более того, белок BEAF-32 также содержит hATC-димеризационный домен транспозазного происхождения [78]. Классическим примером экзаптированных белков считаются Rag1/2 белки V(D)J-рекомбинации [127]. Особо широко представлена экзаптация белков ДНК-транспозонов у растений, причем одни и те же белки могли одомашниваться неоднократно [47, 128].
На сегодняшний день выявлено множество белков, произошедших из транспозаз, а также были обнаружены случаи одомашнивания других белков из МЭ: интеграз (произошедших, скорее всего, из особой группы МЭ Maverick), белка Gag (из которого произошли несколько семейств белков, задействованных в широком спектре клеточных функций), протеаз (например, белок SASPase) [129, 130]. Также из ретровирусов произошли такие белки, как теломеразы и белки группы syncytin (экспрессирующиеся в синцитиотрофобласте плаценты), произошедшие из гена Env. Недавно было показано, что два белка Arabidopsis, участвующих в сайленсинге перицентромерной области, также имеют домен, родственный аминокислотной последовательности eORF ретротранспозона gypsy [131].
Подводя итог, стоит отметить, что гены транспозаз являются наиболее распространенными и вездесущими генами в живой природе. Поэтому возможности экзаптации транспозаз в качестве ДНК-связывающих и олигомеризующихся белков в составе регуляторных сетей являются обширными [132].
ЗАКЛЮЧЕНИЕ
В последнее десятилетие стало очевидно, что мобилом – полноценный участник геномных регуляторных сетей за счет формирования регуляторных участков, образования некодирующих транскриптов и одомашнивания белков, кодируемых МЭ. Вместе с тем эволюция мобилома обладает специфичными чертами и происходит отчасти независимо от остального генома. Более того, между мобиломом и остальным геномом существуют взаимоотношения, подобные модели “паразит–хозяин”. В совокупности особые свойства мобилома позволяют считать его одним из основных источников эволюционной пластичности у эукариот: стабильное состояние генома перемежается короткими интервалами динамичных перестроек, часто связанными с активацией МЭ [65]. Важно отметить, что роль крупных делеций и дупликаций в эволюции генома, опосредованных пластичностью мобилома, и их потенциальное адаптивное значение только предстоит оценить [133]. В частности, растительные геномы демонстрируют огромный вклад МЭ в разнообразие размеров геномов, в которых видны следы многочисленных циклов амплификаций МЭ, полиплоидизации с последующим уменьшением дуплицированных хромосом и их объединением [134, 135].
МЭ являются основными источниками геномной пластичности, позволяющими многообразно и эффективно перестраивать геном посредством транспозиции и негомологичной рекомбинации [136]. МЭ представляются идеальными кандидатами на роль регуляторов эволюционируемости, так как их активность определяется одновременно и внешними, и внутренними популяционными факторами, а также многочисленными системами защиты генома.
Транспозоны обеспечивают эволюционируемость, в то время как эволюция мобилома представляет собой эволюцию эволюционируемости. Фундаментальной чертой эволюции МЭ в геноме является противопоставление “эгоистичных ценностей” транспозонов и “общего блага” всего генома, что также является одним из основных двигателей эволюции мобилома. Другое фундаментальное противопоставление в эволюции мобилома – отбор на разных уровнях и неадаптивное блуждание. По всей видимости, включение МЭ в регуляторные сети в значительной степени происходит случайно и отбор, чье значение может быть весьма важным, присутствует лишь незначительную часть эволюционного времени. В 1999 г. была предложена концепция конструктивной нейтральной эволюции, в основе которой лежит принцип неадаптивного функционального храповика; эта концепция может служить объяснением эволюции клеточной сложности [137, 138]. По всей видимости, эволюция регуляторных сетей под действием мобилома в значительной мере соответствует принципам конструктивной нейтральной эволюции. В конечном счете это дает основание предполагать, что биологическая сложность эукариот обеспечивается МЭ куда в большей степени, чем предполагалось ранее.
Работа выполнена при финансовой поддержке Российского научного фонда (проект № 17-74-20155).
Список литературы
Koonin E.V. The Logic of Chance: The Nature and Origin of Biological Evolution. FT Press, 2011. P. 227.
McShea D.W. Functional complexity in organisms: parts as proxies // Biol. Philos. 2000. V. 15. № 5. P. 641–668. doi 10.1023/A:1006695908715
Adami C. What is complexity? // BioEssays. 2002. V. 24. № 12. P. 1085–1094. doi 10.1002/bies.10192
Elliott T.A., Gregory T.R. What’s in a genome? The C‑value enigma and the evolution of eukaryotic genome content // Philos. Trans. R. Soc. B. Biol. Sci. 2015. V. 370. № 1678. P. 20140331. doi 10.1098/rstb.2014.0331
Claverie J.M. Gene number. What if there are only 30,000 human genes? // Science. 2001. V. 291. № 5507. P. 1255–1257. doi 10.1126/SCIENCE.1058969
Dunham I., Kundaje A., Aldred S.F. et al. An integrated encyclopedia of DNA elements in the human genome // Nature. 2012. V. 489. № 7414. P. 57–74. doi 10.1038/ nature11247
Carroll S.B. Evo-devo and an expanding evolutionary synthesis: a genetic theory of morphological evolution // Cell. 2008. V. 134. № 1. P. 25–36. doi 10.1016/ j.cell.2008.06.030
Lynch V.J. A copy-and-paste gene regulatory network // Science. 2016. V. 351. № 6277. P. 1029–1030. doi 10.1126/science.aaf2977
Sundaram V., Cheng Y., Ma Z. et al. Widespread contribution of transposable elements to the innovation of gene regulatory networks // Genome Res. 2014. V. 24. № 12. P. 1963–1976. doi 10.1101/gr.168872.113
Doolittle W.F. Is junk DNA bunk? A critique of ENCODE // Proc. Natl Acad. Sci. USA. 2013. V. 110. № 14. P. 5294–5300. doi 10.1073/pnas.1221376110
McShea D.W., Brandon R.N. Biology’s First Law: The Tendency for Diversity and Complexity to Increase in Evolutionary Systems. Univ. Chicago Press, 2010. 170 p.
Pigliucci M. Is evolvability evolvable? // Nat. Rev. Genet. 2008. V. 9. № 1. P. 75–82. doi 10.1038/nrg2278
Masel J., Trotter M.V. Robustness and evolvability // Trends Genet. 2010. V. 26. № 9. P. 406–414. doi 10.1016/j.tig.2010.06.002
Wagner A. Robustness and Evolvability in Living Systems. Princeton Univ. Press, 2007. 384 p.
Koonin E.V. Splendor and misery of adaptation, or the importance of neutral null for understanding evolution // BMC Biol. 2016. V. 14. P. 114. doi 10.1186/s12915-016-0338-2
Fernández A., Lynch M. Non-adaptive origins of interactome complexity // Nature. 2011. V. 474. № 7352. P. 502–505. doi 10.1038/nature09992
Lynch M. The frailty of adaptive hypotheses for the origins of organismal complexity // Proc. Natl Acad. Sci. USA. 2007. V. 104. Suppl. 1. P. 8597–8604. doi 10.1073/pnas.0702207104
Wicker T., Sabot F., Hua-Van A. et al. A unified classification system for eukaryotic transposable elements // Nat. Rev. Genet. 2007. V. 8. № 12. P. 973–982. doi 10.1038/nrg2165
Belshaw R., Watson J., Katzourakis A. et al. Rate of recombinational deletion among human endogenous retroviruses // J. Virol. 2007. V. 81. № 17. P. 9437–9442. doi 10.1128/JVI.02216-06
Feschotte C., Mouchès C. Evidence that a family of miniature inverted-repeat transposable elements (MITEs) from the Arabidopsis thaliana genome has arisen from a pogo-like DNA transposon // Mol. Biol. Evol. 2000. V. 17. № 5. P. 730–737. doi 10.1093/oxfordjournals.molbev.a026351
Schnable P.S., Ware D., Fulton R.S. et al. The B73 maize genome: complexity, diversity, and dynamics // Science. 2009. V. 326. № 5956. P. 1112–1115. doi 10.1126/science.1178534
Lander E.S., Linton L.M., Birren B. et al. Initial sequencing and analysis of the human genome // Nature. 2001. V. 409. № 6822. P. 860–921. doi 10.1038/35057062
de Koning A.P.J., Gu W., Castoe T. et al. Repetitive elements may comprise over two-thirds of the human genome // PLoS Genet. 2011. V. 7. № 12. P. e1002384. doi 10.1371/journal.pgen.1002384
Brosius J. The contribution of RNAs and retroposition to evolutionary novelties // Genetica. 2003. V. 118. № 2–3. P. 99–115. doi 10.1023/A:1024141306559
Zhang Y., Romanish M.T., Mager D.L. Distributions of transposable elements reveal hazardous zones in mammalian introns // PLoS Comput. Biol. 2011. V. 7. № 5. P. e1002046. doi 10.1371/journal.pcbi.1002046
Rogozin I.B., Carmel L., Csuros M., Koonin E.V. Origin and evolution of spliceosomal introns // Biol. Direct. 2012. V. 7. № 1. P. 11. doi 10.1186/1745-6150-7-11
Piégu B., Bire S., Arensburger P., Bigot Y. A survey of transposable element classification systems – A call for a fundamental update to meet the challenge of their diversity and complexity // Mol. Phylogenet. Evol. 2015. V. 86. P. 90–109. doi 10.1016/j.ympev.2015.03.009
Huff J.T., Zilberman D., Roy S.W. Mechanism for DNA transposons to generate introns on genomic scales // Nature. 2016. V. 538. № 7626. P. 533–536. doi 10.1038/nature20110
Krull M., Petrusma M., Makalowski W. et al. Functional persistence of exonized mammalian-wide interspersed repeat elements (MIRs) // Genome Res. 2007. V. 17. № 8. P. 1139–1145. doi 10.1101/gr.6320607
Schmitz J., Brosius J. Exonization of transposed elements: A challenge and opportunity for evolution // Biochimie. 2011. V. 93. № 11. P. 1928–1934. doi 10.1016/j.biochi.2011.07.014
Tang W., Gunn T.M., McLaughlin D.F. et al. Secreted and membrane attractin result from alternative splicing of the human ATRN gene // Proc. Natl Acad. Sci. USA. 2000. V. 97. № 11. P. 6025–6030. doi 10.1073/ pnas.110139897
Lee J.Y., Ji Z., Tian B. Phylogenetic analysis of mRNA polyadenylation sites reveals a role of transposable elements in evolution of the 3'-end of genes // Nucl. Acids Res. 2008. V. 36. № 17. P. 5581–5590. doi 10.1093/nar/gkn540
Cowley M., Oakey R.J., Venables P. et al. Transposable elements re-wire and fine-tune the transcriptome // PLoS Genet. 2013. V. 9. № 1. P. e1003234. doi 10.1371/journal.pgen.1003234
Huda A., Bowen N.J., Conley A.B., Jordan I.K. Epigenetic regulation of transposable element derived human gene promoters // Gene. 2011. V. 475. P. 39–48. doi 10.1016/j.gene.2010.12.010
Jordan I.K., Rogozin I.B., Glazko G.V., Koonin E.V. Origin of a substantial fraction of human regulatory sequences from transposable elements // Trends Genet. 2003. V. 19. № 2. P. 68–72. doi 10.1016/S0168-9525(02)00006-9
Morgan H.D., Sutherland H.G., Martin D.I., Whitelaw E. Epigenetic inheritance at the agouti locus in the mouse // Nat. Genet. 1999. V. 23. № 3. P. 314–318. doi 10.1038/15490
Faulkner G.J., Kimura Y., Daub C.O. et al. The regulated retrotransposon transcriptome of mammalian cells // Nat. Genet. 2009. V. 41. № 5. P. 563–571. doi 10.1038/ng.368
Thompson P.J., Macfarlan T.S., Lorincz M.C. Long Terminal Repeats: from parasitic elements to building blocks of the transcriptional regulatory repertoire // Mol. Cell. 2016. V. 62. № 5. P. 766–776. doi 10.1016/ j.molcel.2016.03.029
Eickbush T.H., Malik H.S. Origins and evolution of retrotransposons // Mob. DNA II. Am. Soc. Microbiol. 2002. P. 1111–1144. doi 10.1128/9781555817954.ch49
Katzourakis A., Gifford R.J. Endogenous viral elements in animal genomes // PLoS Genet. 2010. V. 6. № 11. P. e1001191. doi 10.1371/journal.pgen.1001191
Kramerov D.A., Vassetzky N.S. Origin and evolution of SINEs in eukaryotic genomes // Heredity. 2011. V. 107. № 6. P. 487–495. doi 10.1038/hdy.2011.43
Tollis M., Boissinot S. The evolutionary dynamics of transposable elements in eukaryote genomes // Genome Dyn. 2012. V. 7. P. 68–91. doi 10.1159/000337126
Lynch M. The Origins of Genome Architecture. Sin. Ass, 2007. 494 p.
Lane N., Martin W. The energetics of genome complexity // Nature. 2010. V. 467. № 7318. P. 929–934. doi 10.1038/nature09486
Le Rouzic A., Boutin T.S., Capy P. Long-term evolution of transposable elements // Proc. Natl Acad. Sci. USA. 2007. V. 104. № 49. P. 19375–19380. doi 10.1073/pnas.0705238104
Hua-Van A., Le Rouzic A., Boutin T.S. et al. The struggle for life of the genome’s selfish architects // Biol. Direct. 2011. V. 6. № 1. P. 19. doi 10.1186/1745-6150-6-19
Lisch D. How important are transposons for plant evolution? // Nat. Rev. Genet. 2012. V. 14. № 1. P. 49–61. doi 10.1038/nrg3374
Petrov D.A., Fiston-Lavier A.-S., Gonzalez J. et al. Population genomics of transposable elements in Drosophila melanogaster // Annu. Rev. Genet. 2014. V. 48. № 5. P. 561–581. doi 10.1093/molbev/msq337
Adrion J.R., Song M.J., Schrider D.R. et al. Genome-wide estimates of transposable element insertion and deletion rates in Drosophila melanogaster // Genome Biol. Evol. 2017. V. 9. № 5. P. 1329–1340. doi 10.1093/gbe/evx050
Luan D.D., Korman M.H., Jakubczak J.L., Eickbush T.H. Reverse transcription of R2Bm RNA is primed by a nick at the chromosomal target site: a mechanism for non-LTR retrotransposition // Cell. 1993. V. 72. № 4. P. 595–605.
Zou S., Voytas D.F. Silent chromatin determines target preference of the Saccharomyces retrotransposon Ty5 // Proc. Natl Acad. Sci. USA. 1997. V. 94. P. 7412–7416.
Levin H.L., Moran J.V. Dynamic interactions between transposable elements and their hosts // Nat. Rev. Genet. 2011. V. 12. № 9. P. 615–627. doi 10.1038/ nrg3030
Chuong E.B., Elde N.C., Feschotte C. Regulatory activities of transposable elements: from conflicts to benefits // Nat. Rev. Genet. 2016. V. 18. № 2. P. 71–86. doi 10.1038/nrg.2016.139
Qi X., Sandmeyer S. In vitro targeting of strand transfer by the Ty3 retroelement integrase // J. Biol. Chem. 2012. V. 287. № 22. P. 18589–18595. doi 10.1074/ jbc.M111.326025
Sultana T., Zamborlini A., Cristofari G., Lesage P. Integration site selection by retroviruses and transposable elements in eukaryotes // Nat. Rev. Genet. 2017. V. 18. № 5. P. 292–308. doi 10.1038/nrg.2017.7
Aravin A.A., Sachidanandam R., Bourc’his D. et al. A piRNA pathway primed by individual transposons is linked to de novo DNA methylation in mice // Mol. Cell. 2008. V. 31. № 6. P. 785–799. doi 10.1016/j.molcel.2008.09.003
Elsässer S.J., Noh K.-M., Diaz N. et al. Histone H3.3 is required for endogenous retroviral element silencing in embryonic stem cells // Nature. 2015. V. 522. № 7555. P. 240–244. doi 10.1038/nature14345
Barau J., Teissandier A., Zamudio N. et al. The DNA methyltransferase DNMT3C protects male germ cells from transposon activity // Science. 2016. V. 354. № 6314. P. 909–912.
Haig D. Transposable elements: Self-seekers of the germline, team-players of the soma // BioEssays. 2016. V. 38. № 11. P. 1158–1166. doi 10.1002/ bies.201600125
Grow E.J., Flynn R.A., Chavez S.L. et al. Intrinsic retroviral reactivation in human preimplantation embryos and pluripotent cells // Nature. 2015. V. 522(7555). P. 221–225. doi 10.1038/nature14308
Izsvák Z., Wang J., Singh M. et al. Pluripotency and the endogenous retrovirus HERVH: Conflict or serendipity? // BioEssays. 2016. V. 38. № 1. P. 109–117. doi 10.1002/bies.201500096
Jacobs F.M.J., Greenberg D., Nguyen N. et al. An evolutionary arms race between KRAB zinc-finger genes ZNF91/93 and SVA/L1 retrotransposons // Nature. 2014. V. 516. № 7530. P. 242–245. doi 10.1038/nature13760
Cam H.P., Noma K., Ebina H. et al., Host genome surveillance for retrotransposons by transposon-derived proteins // Nature. 2008. V. 451. № 7177. P. 431–436. doi 10.1038/nature06499
Piacentini L., Fanti L., Specchia V. et al. Transposons, environmental changes, and heritable induced phenotypic variability // Chromosoma. 2014. V. 123. № 4. P. 345–354. doi 10.1007/s00412-014-0464-y
Oliver K.R., Greene W.K. Transposable elements: powerful facilitators of evolution // BioEssays. 2009. V. 31. № 7. P. 703–714. doi 10.1002/bies.200800219
Kunarso G., Chia N.-Y., Jeyakani J. et al. Transposable elements have rewired the core regulatory network of human embryonic stem cells // Nat. Genet. 2010. V. 42. № 7. P. 631–634. doi 10.1038/ng.600
Bourque G., Leong B., Vega V.B. et al. Evolution of the mammalian transcription factor binding repertoire via transposable elements // Genome Res. 2008. V. 18. № 11. P. 1752–1762. doi 10.1101/gr.080663.108
Thornburg B.G., Gotea V., Makałowski W. Transposable elements as a significant source of transcription regulating signals // Gene. 2006. V. 365. P. 104–110. doi 10.1016/j.gene.2005.09.036
Sundaram V., Cheng Y., Ma Z. et al. Widespread contribution of transposable elements to the innovation of gene regulatory networks // Genome Res. 2014. V. 24. № 12. P. 1963–1976. doi 10.1101/gr.168872.113
Wang T., Zeng J., Lowe C.B. et al. Species-specific endogenous retroviruses shape the transcriptional network of the human tumor suppressor protein p53 // Proc. Natl Acad. Sci. USA. 2007. V. 104. № 47. P. 18613–18618. doi 10.1073/pnas.0703637104
de Souza F.S.J., Franchini L.F., Rubinstein M. Exaptation of transposable elements into novel cis-regulatory elements: is the evidence always strong? // Mol. Biol. Evol. 2013. V. 30. № 6. P. 1239–1251. doi 10.1093/ molbev/mst045
Schmidt D., Schwalie P.C., Wilson M.D. et al. Waves of retrotransposon expansion remodel genome organization and CTCF binding in multiple mammalian lineages // Cell. 2012. V. 148. № 1–2. P. 335–348. doi 10.1016/j.cell.2011.11.058
Lunyak V.V., Prefontaine G.G., Nunez E. et al. Developmentally regulated activation of a SINE B2 repeat as a domain boundary in organogenesis // Science. 2007. V. 317. № 5835. P. 248–251. doi 10.1126/science.1140871
Bire S., Casteret S., Piégu B. et al. Mariner transposons contain a silencer: possible role of the polycomb repressive complex 2 // PLoS Genet. 2016. V. 12. № 3. P. e1005902. doi 10.1371/journal.pgen.1005902
Wang J., Vicente-García C., Seruggia D. et al. MIR retrotransposon sequences provide insulators to the human genome // Proc. Natl Acad. Sci. USA. 2015. V. 112. № 32. P. E4428–E4437. doi 10.1073/pnas.1507253112
Hollister J.D., Gaut B.S. Epigenetic silencing of transposable elements: a trade-off between reduced transposition and deleterious effects on neighboring gene expression // Genome Res. 2009. V. 19. № 8. P. 1419–1428. doi 10.1101/gr.091678.109
Lisch D., Bennetzen J.L. Transposable element origins of epigenetic gene regulation // Curr. Opin. Plant. Biol. 2011. V. 14. № 2. P. 156–161. doi 10.1016/ j.pbi.2011.01.003
Feschotte C. Transposable elements and the evolution of regulatory networks // Nat. Rev. Genet. 2008. V. 9. № 5. P. 397–405. doi 10.1038/nrg2337
Cohen C.J., Lock W.M., Mager D.L. Endogenous retroviral LTRs as promoters for human genes: A critical assessment // Gene. 2009. V. 448. № 2. P. 105–114. doi 10.1016/j.gene.2009.06.020
Simonti C.N., Pavličev M., Capra J.A. Transposable element exaptation into regulatory regions is rare, influenced by evolutionary age, and subject to pleiotropic constraints // Mol. Biol. Evol. 2017. V. 34. № 11. P. 2856–2869. doi 10.1093/molbev/msx219
Baptiste B.A., Ananda G., Strubczewski N. et al. Mature microsatellites: mechanisms underlying dinucleotide microsatellite mutational biases in human cells // G3 (Bethesda). 2013. V. 3. № 3. P. 451–463. doi 10.1534/ g3.112.005173
Zemojtel T., Kielbasa S.M., Arndt P.F. et al. CpG deamination creates transcription factor-binding sites with high efficiency // Genome Biol. Evol. 2011. V. 3. P. 1304–1311. doi 10.1093/gbe/evr107
Kuzu G., Kaye E.G., Chery J. et al. Expansion of GA dinucleotide repeats increases the density of CLAMP binding sites on the X-chromosome to promote Drosophila dosage compensation // PLoS Genet. 2016. V. 12. № 7. P. e1006120. doi 10.1371/journal.pgen.1006120
Rebollo R., Romanish M.T., Mager D.L. Transposable elements: an abundant and natural source of regulatory sequences for host genes // Annu. Rev. Genet. 2011. V. 46. № 1. P. 21–42. doi 10.1146/annurev-genet-110711-155621
Koonin E.V., Wolf Y.I. Constraints and plasticity in genome and molecular-phenome evolution // Nat. Rev. Genet. 2010. V. 11. № 7. P. 487–498. doi 10.1038/nrg2810
Negi P., Rai A.N., Suprasanna P. Moving through the stressed genome: emerging regulatory roles for transposons in plant stress response // Frontiers Plant Sci. 2016. V. 7. № 1448. doi 10.3389/FPLS.2016.01448
Dubin M.J., Mittelsten Scheid O., Becker C. Transposons: a blessing curse // Curr. Opin. in Plant Biology. 2018. V. 42. P. 23–29. doi 10.1016/ J.PBI.2018.01.003
Nowick K., Hamilton A.T., Zhang H., Stubbs L. Rapid sequence and expression divergence suggest selection for novel function in primate-specific KRAB-ZNF genes // Mol. Biol. Evol. 2010. V. 27. № 11. P. 2606–2617. doi 10.1093/molbev/msq157
Yang P., Wang Y., Macfarlan T.S. The role of KRAB-ZFPs in transposable element repression and mammalian evolution // Trends Genet. 2017. V. 33. № 11. P. 871–881. doi 10.1016/j.tig.2017.08.006
Ecco G., Cassano M., Kauzlaric A. et al. Transposable elements and their KRAB-ZFP controllers regulate gene expression in adult tissues // Dev. Cell. 2016. V. 36. № 6. P. 611–23. doi 10.1016/j.devcel.2016.02.024
Gifford W.D., Pfaff S.L., Macfarlan T.S. Transposable elements as genetic regulatory substrates in early development // Trends Cell Biol. 2013. V. 23. № 5. P. 218–226. doi 10.1016/j.tcb.2013.01.001
Friedli M., Trono D. The developmental control of transposable elements and the evolution of higher species // Annu. Rev. Cell Dev. Biol. 2015. V. 31. № 1. P. 429–451. doi 10.1146/annurev-cellbio-100814-125514
Thomas J.H., Schneider S. Coevolution of retroelements and tandem zinc finger genes // Genome Res. 2011. V. 21. № 11. P. 1800–1812. doi 10.1101/gr.121749.111
Blumenstiel J.P. Evolutionary dynamics of transposable elements in a small RNA world // Trends Genet. 2011. V. 27. № 1. P. 23–31. doi 10.1016/j.tig.2010.10.003
Creasey K.M., Zhai J., Borges F. et al. miRNAs trigger widespread epigenetically activated siRNAs from transposons in Arabidopsis // Nature. 2014. V. 508. № 7496. P. 411–415. doi 10.1038/nature13069
McCue A.D., Slotkin R.K. Transposable element small RNAs as regulators of gene expression // Trends Genet. 2012. V. 28. № 12. P. 616–623. doi 10.1016/j.tig.2012.09.001
Chuong E.B., Elde N.C., Feschotte C. Regulatory activities of transposable elements: from conflicts to benefits // Nat. Rev. Genet. 2016. V. 18. № 2. P. 71–86. doi 10.1038/nrg.2016.139
Göke J., Ng H.H. CTRL+INSERT: retrotransposons and their contribution to regulation and innovation of the transcriptome // EMBO Rep. 2016. V. 17. № 8. P. e201642743. doi 10.15252/embr.201642743
Chuong E.B., Elde N.C., Feschotte C. Regulatory evolution of innate immunity through co-option of endogenous retroviruses // Science. 2016. V. 351. № 6277. P. 1083–1087. doi 10.1126/science.aad5497
Feschotte C., Gilbert C. Endogenous viruses: insights into viral evolution and impact on host biology // Nat. Rev. Genet. 2012. V. 13. № 4. P. 283–296. doi 10.1038/nrg3199
Ge S.X. Exploratory bioinformatics investigation reveals importance of “junk” DNA in early embryo development // BMC Genomics. 2017. V. 18. № 1. P. 200. doi 10.1186/s12864-017-3566-0
Koonin E.V. The origin of introns and their role in eukaryogenesis: a compromise solution to the introns-early versus introns-late debate? // Biol. Direct. 2006. V. 1. № 1. P. 22. doi 10.1186/1745-6150-1-22
Koonin E.V. Viruses and mobile elements as drivers of evolutionary transitions // Philos. Trans. R. Soc. London B Biol. Sci. 2016. V. 371. № 1701. P. 20150442. doi 10.1098/rstb.2015.0442
Jurka J., Bao W., Kojima K.K. et al. Distinct groups of repetitive families preserved in mammals correspond to different periods of regulatory innovations in vertebrates // Biol. Direct. 2012. V. 7. P. 36. doi 10.1186/ 1745-6150-7-36
Imbeault M., Helleboid P.-Y., Trono D. KRAB zinc-finger proteins contribute to the evolution of gene regulatory networks // Nature. 2017. V. 543. № 7646. P. 550–554. doi 10.1038/nature21683
Albertin C.B., Simakov O., Mitros T. et al. The octopus genome and the evolution of cephalopod neural and morphological novelties // Nature. 2015. V. 524. № 7564. P. 220–224. doi 10.1038/nature14668
Muotri A.R., Chu V.T., Marchetto M.C.N. et al. Somatic mosaicism in neuronal precursor cells mediated by L1 retrotransposition // Nature. 2005. V. 435. № 7044. P. 903–910. doi 10.1038/nature03663
Perrat P.N., DasGupta S., Wang J. et al. Transposition-driven genomic heterogeneity in the Drosophila brain // Science. 2013. V. 340. № 6128. P. 91–95. doi 10.1126/ science.1231965
Rajasethupathy P., Antonov I., Sheridan R. et al. A role for neuronal piRNAs in the epigenetic control of memory-related synaptic plasticity // Cell. 2012. V. 149. № 3. P. 693–707. doi 10.1016/j.cell.2012.02.057
Nandi S., Chandramohan D., Fioriti L. et al. Roles for small noncoding RNAs in silencing of retrotransposons in the mammalian brain // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 45. P. 12697–12702. doi 10.1073/pnas.1609287113
Zhao J., Sun B.K., Erwin J.A. et al. Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome // Science. 2008. V. 322. № 5902. P. 750–756. doi 10.1126/science.1163045
Elisaphenko E.A., Kolesnikov N.N., Shevchenko A.I. et al. A dual origin of the Xist gene from a protein-coding gene and a set of transposable elements // PLoS One. 2008. V. 3. № 6. P. e2521. doi 10.1371/journal.pone.0002521
Ellison C.E., Bachtrog D. Dosage compensation via transposable element mediated rewiring of a regulatory network // Science. 2013. V. 342. № 6160. P. 846–850. doi 10.1126/science.1239552
Kannan S., Chernikova D., Rogozin I.B. et al. Transposable element insertions in long intergenic non-coding RNA genes // Front. Bioeng. Biotechnol. 2015. V. 3. № 71. doi 10.3389/fbioe.2015.00071
Kelley D., Rinn J. Transposable elements reveal a stem cell-specific class of long noncoding RNAs // Genome Biol. 2012. V. 13. № 11. P. R107. doi 10.1186/ gb-2012-13-11-r107
Durruthy-Durruthy J., Sebastiano V., Wossidlo M. et al. The primate-specific noncoding RNA HPAT5 regulates pluripotency during human preimplantation development and nuclear reprogramming // Nat. Genet. 2015. V. 48. № 1. P. 44–52. doi 10.1038/ng.3449
Piriyapongsa J., Jordan I.K. A family of human microRNA genes from miniature inverted-repeat transposable elements // PLoS One. 2007. V. 2. № 2. P. e203. doi 10.1371/journal.pone.0000203
Johnson R., Guigo R. The RIDL hypothesis: transposable elements as functional domains of long noncoding RNAs // RNA. 2014. V. 20. № 7. P. 959–976. doi 10.1261/rna.044560.114
Liang D., Wilusz J.E. Short intronic repeat sequences facilitate circular RNA production // Genes Dev. 2014. V. 28. № 20. P. 2233–2247. doi 10.1101/ gad.251926.114
Qu S., Zhong Y., Shang R. et al. The emerging landscape of circular RNA in life processes // RNA Biol. 2016. V. 14. № 8. P. 992–999. doi 10.1080/ 15476286.2016.1220473
Göke J., Lu X., Chan Y.-S. et al. Dynamic transcription of distinct classes of endogenous retroviral elements marks specific populations of early human embryonic cells // Cell Stem Cell. 2015. V. 16. № 2. P. 135–141. doi 10.1016/j.stem.2015.01.005
Loewer S., Cabili M.N., Guttman M. et al. Large intergenic non-coding RNA-RoR modulates reprogramming of human induced pluripotent stem cells // Nat. Genet. 2010. V. 42. № 12. P. 1113–1117. doi 10.1038/ng.710
Lu X., Sachs F., Ramsay L. et al. The retrovirus HERVH is a long noncoding RNA required for human embryonic stem cell identity // Nat. Struct. Mol. Biol. 2014. V. 21. № 4. P. 423–425. doi 10.1038/nsmb.2799
Rinn J.L., Chang H.Y. Genome regulation by long noncoding RNAs // Annu. Rev. Biochem. 2012. V. 81. P. 145–166. doi 10.1146/annurev-biochem-051410-092902
Cannavò E., Khoueiry P., Garfield D.A. et al. Shadow enhancers are pervasive features of developmental regulatory networks // Curr. Biol. 2016. V. 26. № 1. P. 38–51. doi 10.1016/j.cub.2015.11.034
Haerty W., Ponting C.P., Meader S. et al. Mutations within lncRNAs are effectively selected against in fruit fly but not in human // Genome Biol. 2013. V. 14. № 5. P. R49. doi 10.1186/gb-2013-14-5-r49
Roth D.B., Craig N.L. VDJ recombination: a transposase goes to work // Cell. 1998. V. 94. № 4. P. 411–414.
Joly-Lopez Z., Hoen D.R., Blanchette M. et al. Phylogenetic and genomic analyses resolve the origin of important plant genes derived from transposable elements // Mol. Biol. Evol. 2016. V. 33. № 8. P. 1937–1956. doi 10.1093/molbev/msw067
Volff J.-N. Turning junk into gold: domestication of transposable elements and the creation of new genes in eukaryotes // BioEssays. 2006. V. 28. № 9. P. 913–922. doi 10.1002/bies.20452
Naville M., Warren I.A., Haftek-Terreau Z. et al. Not so bad after all: retroviruses and long terminal repeat retrotransposons as a source of new genes in vertebrates // Clin. Microbiol. Infect. 2016. V. 22. № 4. P. 312–323. doi 10.1016/j.cmi.2016.02.001
Ikeda Y., Pélissier T., Bourguet P. et al. Arabidopsis proteins with a transposon-related domain act in gene silencing // Nat. Commun. 2017. V. 8. P. 15122. doi 10.1038/ncomms15122
Aziz R.K., Breitbart M., Edwards R.A. Transposases are the most abundant, most ubiquitous genes in nature // Nucl. Acids Res. 2010. V. 38. № 13. P. 4207–4217. doi 10.1093/nar/gkq140
Kapusta A., Suh A., Feschotte C. Dynamics of genome size evolution in birds and mammals // Proc. Natl Acad. Sci. USA. 2017. V. 114. № 8. P. E1460–E1469. doi 10.1073/pnas.1616702114
Wendel J.F., Jackson S.A., Meyers B.C., Wing R.A. Evolution of plant genome architecture // Genome Biol. 2016. V. 17. № 1. P. 37. doi 10.1186/s13059-016-0908-1
Vicient C.M., Casacuberta J.M. Impact of transposable elements on polyploid plant genomes // Ann. Bot. 2017. V. 120. № 2. P. 195–207. doi 10.1093/aob/ mcx078
Wagner G.P., Altenberg L. Perspective: complex adaptations and the evolution of evolvability // Evolution (N.Y.). 1996. V. 50. № 3. P. 967. doi 10.2307/2410639
Stoltzfus A. On the possibility of constructive neutral evolution // J. Mol. Evol. 1999. V. 49. № 2. P. 169–181. doi 10.1007/PL00006540
Speijer D. Does constructive neutral evolution play an important role in the origin of cellular complexity? // BioEssays. 2011. V. 33. № 5. P. 344–349. doi 10.1002/bies.201100010
Дополнительные материалы отсутствуют.


