Генетика, 2019, T. 55, № 1, стр. 3-11
Гены HD-Zip и их роль в адаптации растений к факторам внешней среды
А. Б. Щербань *
Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
* E-mail: atos@bionet.nsc.ru
Поступила в редакцию 09.01.2018
После доработки 21.02.2018
Принята к публикации 07.02.2018
Аннотация
В данном обзоре представлена характеристика уникального для растений HD-Zip-семейства гомео-бокс-содержащих генов и рассматривается их участие в молекулярных механизмах устойчивости к отдельным неблагоприятным факторам внешней среды, таким как засуха, недостаток света, патогены. Продемонстрировано важное значение генов HD-Zip в модуляции и объединении сигналов от различных гормон-зависимых генетических каскадов, контролирующих адаптацию растений к тем или иным внешним факторам.
Гомеобокс-содержащие гены были впервые обнаружены у дрозофилы [1] в результате анализа гомеозисных мутаций, приводящих к трансформации одних частей тела в другие. Данные гены кодируют транскрипционные факторы (ТФ), управляющие процессами роста и дифференцировки в организме. Отличительной особенностью этих факторов является наличие гомеодомена HD – последовательности из 60 аминокислот, которая формирует трехспиральную третичную структуру, обеспечивающую специфическое взаимодействие с промоторными районами генов-мишеней [2]. Первым HD-содержащим геном, изолированным из геномов растений, был ген Knotted1 кукурузы Zea mays, влияющий на дифференцировку листовых тканей и получивший свое название благодаря характерному фенотипу растений, несущих этот ген (наличие узелков возле срединной и боковых жилок листа) [3]. С момента открытия этого гена в 1991 г. было установлено большое число других сходных генов растений, различающихся по структуре и размеру гомеодомена, а также наличию дополнительных доменов [4]. Однако, несмотря на то что эти генетические факторы участвуют в процессе формирования и дифференцировки клеток, ни один из них не проявлял гомеозисных эффектов, характерных для животных.
Среди гомеобокс-содержащих генов растений выделяется особое семейство HD-Zip, для которого характерна комбинация участка, кодирующего гомеодомен HD, с участком так называемой “лейциновой застежки” (leucinezipper, LZ). Последняя обеспечивает образование белковых димерных молекул и только в такой форме ТФ HD-Zip способен связывать ДНК-мишень [5]. По отдельности каждый из двух указанных доменов встречается у всех эукариот, однако их сочетание в одном белке характерно только для растений, включая как мхи и папоротники, так и покрытосеменные растения [6]. На основании структурных особенностей гены HD-Zip подразделяются на классы I–IV, из которых наиболее изучены в функциональном плане классы III и IV. Последние участвуют в процессах дифференцировки различных тканей, включая апикальную меристему, сосудистые пучки, ткани эпидермиса различных органов (корней, стебля) [7–14]. HD-Zip-гены, относящиеся к классам I–II, рано отделились от группы генов III–IV, судя по особенностям их структуры [15], тем не менее и для них было показано участие в ряде формообразовательных процессов, таких как развитие семядоли и листа [16, 17], определение срока колошения у озимой пшеницы [18]. Отличительная черта всех классов HD-Zip-генов заключается в изменчивости их паттернов экспрессии в зависимости от внешних факторов (свет, температура, увлажненность и др.), а также от действия фитогормонов, что предполагает участие этих генов в различных генетических каскадах, контролирующих адаптивные реакции растений к меняющимся, в том числе стрессовым, факторам внешней среды [19]. Цель настоящей статьи – обзор накопленных данных, посвященных этому аспекту функционирования HD-Zip-семейства регуляторных генов.
СТРУКТУРНАЯ ХАРАКТЕРИСТИКА ОСНОВНЫХ КЛАССОВ ГЕНОВ HD-Zip
HD-Zip-гены каждого класса представлены в отдельных геномах многочисленными паралогами, которые могут различаться как по своей структуре, так и по фенотипическому проявлению, связанному с уровнем экспрессии в различных тканях. Тем не менее гены одного класса, как правило, характеризуются общим механизмом своей регуляции, а также специфическим набором мишеней в составе тех генетических каскадов, в которых они принимают участие [20].
Классы I–II
HD-Zip-гены, относящиеся к классам I и II, кодируют подобные по структуре ТФ, имеющие небольшой размер (170–250 а. о.) относительно белков двух других классов (см. ниже) и состоящие из высококонсервативного HD, менее консервативного домена LZ и вариабельных N- и C-концевых районов (рис. 1). Основные функциональные домены HD и LZ содержат характерные наборы аминокислот, которые внутри отдельных классов не различаются даже у отдаленных видов растений [21, 22]. Однако, несмотря на большое структурное сходство генов обоих классов, между ними имеются следующие характерные различия:
Рис. 1.
Схема основных классов белков HD-Zip (модифицирована из [20]). HD – гомеодомен; LZ – лейциновый зиппер; START – домен (см. расшифровку в тексте); SAD – домен, прилегающий к START-домену; MEKHLA – домен, содержащий высококонсервативные аминокислотные остатки: Met, Glu, Lys, His, Leu, Ala.
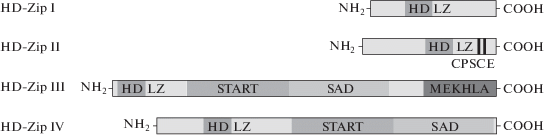
1. Гены класса HD-Zip II содержат три экзона и два интрона, расположенных в участках HD, кодирующих α-спирали 2 и 3. У класса HD-Zip I имеются гены с одним интроном: либо в составе домена LZ, либо в составе α-спирали 1, а также гены с комбинацией того и другого интрона [21].
2. Гены HD-Zip II кодируют дополнительный CPSCE-мотив (получивший название от последовательности аминокислот Cys, Pro, Ser, Cys, Glu в буквенном коде) в составе С-концевого района. Этот мотив отвечает за образование мультимерных белковых молекул за счет межмолекулярных Cys–Cys-связей. Такие молекулы не способны проходить через ядерную оболочку, поэтому активность ТФ может регулироваться скоростью образования мультимерных форм в зависимости от окислительно-восстановительного (редокс) потенциала цитоплазмы клетки [23].
3. Продукты генов HD-Zip I и HD-Zip II имеют различную специфичность сайтов-мишеней, в качестве которых выступают псевдопалиндромные последовательности CAATNATTG с различающимся центральным нуклеотидом: А/Т – в случае HD-Zip I и C/G – в случае HD-Zip II. Эта специфичность, по-видимому, обусловлена различными аминокислотами в позициях 46 и 56 α-спирали 3: Ala и Trp у HD-Zip I; Glu и Thr у HD-Zip II, наряду с различной пространственной ориентацией у обоих белков аминокислотного остатка Arg55, который выполняет центральную роль во взаимодействии с молекулой ДНК [24].
HD-Zip-белки обоих классов способны взаимодействовать с ДНК только в форме димеров, при этом большое значение для детерминации силы взаимодействия (но не специфичности) имеет структура N-концевого района и петли между первой и второй α-спиралями [25, 26].
Классы III–IV
Продукты генов HD-Zip III–IV относятся к одним из наиболее крупных ТФ растений и содержат дополнительные структурные домены, отсутствующие у предыдущей группы генов: START (Steroidogenic Acute Regulatory protein-related lipid Transfer) и консервативный SAD (START-associated domain). HD-Zip III-факторы, кроме указанных двух доменов, имеют еще один дополнительный С-концевой домен MEKHLA [20] (рис. 1).
Функция START-домена (~200 а. о.) до сих пор точно не установлена. У животных данный домен был обнаружен в различных белках, связанных с транспортом липидов или стеролов, но ни в одном из случаев он не был ассоциирован с ДНК-связывающим доменом [27]. Предполагается, что у растений аффинность белков, содержащих этот домен, к специфическим ДНК-сайтам может регулироваться взаимодействиями белок-липид/cтерол как прямыми, так и через другой белок-посредник [28]. Данная гипотеза подтверждается работой [29], в которой было показано участие одного из ТФ HD-Zip IV в фосфолипидной сигнальной системе клеток корня у арабидопсиса. По аналогии с механизмом транспорта рецепторов глюкокортикоидов у млекопитающих [30] предполагается также, что взаимодействие липидов со START-доменом необходимо для транспорта ТФ HD-Zip III–IV в ядро клетки. Функция SAD-домена пока неизвестна, но в одном случае установлено, что присутствие этого домена необходимо для осуществления регуляторной функции белка HD-Zip IV у хлопчатника [31].
MEKHLA-домен ТФ HD-Zip III имеет высокую степень гомологии к PAS-домену, присутствующему во многих белках всех видов организмов [32]. Последний является внутриклеточным сенсором, способным воспринимать такие сигналы как свет, концентрация кислорода, редокс-потенциал цитоплазмы. Факт присутствия сходного домена у белков HD-Zip III указывает на их вовлеченность в новую потенциальную сигнальную систему, которую еще предстоит изучить.
Взаимодействие ТФ HD-Zip III–IV с ДНК менее изучено, чем в случае двух предыдущих классов, поскольку сайты-мишени для этих белков более полиморфны. Общая особенность этих сайтов – наличие последовательности TAAA [20].
Далее будет рассмотрено участие генов HD-Zip различных классов в формировании устойчивости к некоторым неблагоприятным факторам внешней среды.
УСТОЙЧИВОСТЬ К ЗАСУХЕ
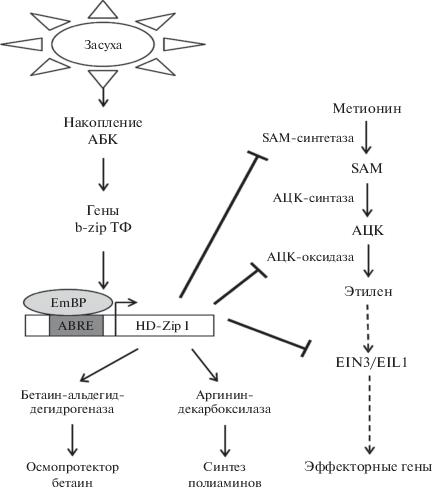
Ограниченный доступ воды для растений приводит к изменению ряда морфофизиологических признаков, а именно к уменьшению скорости испарения, замедлению роста и развития растения, раннему цветению и др. [33]. Эти процессы контролируются рядом растительных гормонов, включая абсцизовую кислоту (АБК), этилен, ауксин, а также генетической системой, опосредующей действие указанных гормонов на метаболические процессы, связанные с пролиферацией и дифференциацией клеток различных тканей [34]. Особую роль в этой системе выполняют гены HD-Zip-семейства, в особенности относящиеся к классу HD-Zip I. На различных растительных объектах, таких как рис, арабидопсис, подсолнечник, мягкая пшеница, был установлен всплеск транскрипционной активности генов этого класса в условиях засухи [21, 22, 35, 36]. Аналогичный эффект происходит под действием экзогенного гормона АБК, что свидетельствует об участии генов HD-Zip I в АБК-опосредованном ответе на данный вид стресса. Конститутивная экспрессия этих генов в трансгенных растениях сопровождается замедлением процессов, связанных со старением, что способствует повышению устойчивости к засухе [37]. Это позволило предположить, что фактор HD-Zip I участвует в регуляции сигнальной системы “гормона старения” – этилена. В дальнейшем это предположение подтвердилось при изучении ТФ Hahb4 подсолнечника [35]. В целом роль гена HD-Zip I в интегрированной системе, определяющей устойчивость к дефициту воды, можно описать в рамках схемы на рис. 2. Под действием засухи в различных тканях растения происходит накопление “гормона стресса” АБК, который через гены-посредники вызывает индукцию гена HD-Zip I в результате связывания белкового фактора EmBP с сайтами АBRE (ABscisic acid Responsive Elements) в составе промотора данного гена. Активация HD-Zip I приводит к репрессии этиленового пути в результате подавления активности основных генов, контролирующих биосинтез этилена: SAM (S-аденозилметионинсинтетазы) и АЦК-оксидазы. Последний фермент катализирует реакцию окисления 1-аминоциклопропан-1-карбоновой кислоты (АЦК) с образованием этилена [38]. Дополнительно этиленовый путь негативно регулируется в результате супрессии EIN3 и EIL1 факторов, служащих трансдукторами этиленового сигнала [39]. Кроме репрессии этиленового пути, активация HD-Zip I сопровождается активацией предполагаемых генов-мишеней, не связанных непосредственно с этим каскадом, в том числе генов, кодирующих бетаин-альдегиддегидрогеназу и аргининдекарбоксилазу [35]. Первый фермент контролирует биосинтез известного осмопротектора бетаина [40], второй вовлечен в биосинтез полиаминов – регуляторов ответа на осмотический стресс [41]. Таким образом, устойчивость к недостатку воды обеспечивается ингибированием этилен-индуцируемого процесса старения, что приводит, в частности, к поддержанию активного фотосинтеза в течение более длительного периода, позволяя растению синтезировать различные метаболиты для защиты от стресса, в том числе осмопротекторы. Ген HD-Zip I при этом выполняет роль посредника между сигнальными системами, контролируемыми гормонами АБК и этиленом.
Рис. 2.
Участие ТФ HD-Zip I в регуляции ответа на стресс засухи. Объяснение в тексте. Непрерывные линии со стрелками и ограничителями на концах обозначают индуцирующие и супрессирующие влияния, соответственно. Прерывистые линии и стрелки обозначают пути передачи сигнала, представленные упрощенно. SAM – S-аденозилметионин; AЦК – 1-амино-циклопропан-1-карбоновая кислота; ABRE – элементы промотора, контролирующие реакцию на АБК. Г-образная линия со стрелкой обозначает старт трансляции.
Интересно, что помимо генов HD-Zip I в детерминации устойчивости к недостатку воды могут принимать участие гены, относящиеся к классу HD-Zip IV. Было показано, что у мутантов арабидопсиса с повышенной экспрессией гена HDG11 данного класса возрастала устойчивость к засухе [42]. Мутантный фенотип характеризовался более развитой корневой системой и редуцированным числом устьиц. В биохимическом плане наблюдался повышенный уровень АБК, а также некоторых ферментов, отвечающих за детоксикацию активных форм кислорода и поддержание осмотического давления. На основании повышения уровня АБК авторы предполагали в данном случае наличие механизма устойчивости, аналогичного вышеописанному для генов HD-Zip I. Однако, поскольку нет данных, подтверждающих участие HD-Zip IV в регуляции этиленового каскада, этот механизм может быть иным. Нельзя также полностью исключить наличие различных механизмов регуляции и в случае генов, относящихся к одному классу. Так, под влиянием водного дефицита у части генов HD-Zip I арабидопсиса уровень экспрессии повышается, тогда как у другой части, наоборот, понижается [21].
СИНДРОМ “ИЗБЕГАНИЯ ТЕНИ”
Синдром “избегания тени” (SAS, Shade Avoidance Syndrome) широко распространен среди покрытосеменных растений. Понижение соотношения красного света к дальнему красному (R/FR) является сигналом затененности растения, возникающей из-за высокой плотности посевов. Данный сигнал воспринимается системой белков-сенсоров, в качестве которых выступают фитохромы А и В [43]. В ответ на этот сигнал происходят быстрые морфологические изменения, связанные с удлинением стебля за счет торможения роста листовых пластинок. Эти изменения полностью обратимы как только растение перерастает своих соседей и увеличивается доля красного света, необходимого для фотосинтеза. Соответствующие изменения экспрессии генов, сопровождающие данный морфологический синдром, также характеризуются быстротой и обратимостью [44]. К числу генов, реагирующих на изменение соотношения R/FR подобным образом, а именно резким увеличением уровня экспрессии при низком соотношении и столь же быстрым снижением этого уровня при высоком соотношении, относятся гены арабидопсиса ATHB2 (класс HD-Zip II) и PIL1, который принадлежит к классу ТФ с основным доменом типа спираль–петля–спираль (Basic–helix–loop–helix, bHLH) [44, 45]. Помимо указанных генов в регуляции ответа на затененность принимает участие ген HFR1, выступающий в качестве негативного регулятора [46]. В целом эти данные наряду с другими раскрывают генетический механизм ответа, базирующийся на динамическом балансе позитивных и негативных регуляторов (рис. 3).
Рис. 3.
Генетическая регуляция ответа на затененность. Характеристика генов (белковых факторов) дана в тексте. Ген, относящийся к классу HD-Zip II, выделен рамкой. Отношения супрессии и индукции, а также до конца не исследованные пути регуляции обозначены линиями как на рис. 2.
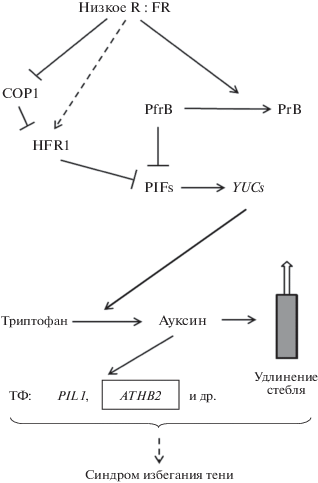
Одним из основных сенсоров в этом ответе является фитохром В [47]. В результате снижения соотношения R/FR до критического уровня данный белок меняет свою конформацию, переходя из формы, воспринимающей дальний красный свет (PfrB), в форму, воспринимающую красный свет (PrB). Это приводит к снижению ингибиторного эффекта PfrB на активность белков PIF (Phytochrome Interacting Factors), которые по своей структуре относятся к bHLH-типу ТФ. В свою очередь, PIF-белки индуцируют гены YUCs, контролирующие биосинтез гормона ауксина из предшественника триптофана (Trp) в листьях [47]. Этот гормон затем транспортируется в клетки стебля, где стимулирует процесс элонгации. Ауксин модулирует экспрессию огромного количества ТФ, в числе которых вышеуказанные гены ATHB2 и PIL1 [48]. Последние могут участвовать в объединении сигнальных каскадов, контролируемых различными гормонами, и формировании комплексного адаптивного ответа на стресс, однако детали этого процесса пока не изучены. Одновременно с активацией генов – позитивных регуляторов ответа происходит индукция негативного регулятора HFR1, продукт которого связывается с белками PIF, инактивируя их [49]. В условиях освещенности этот продукт подвергается деградации в результате убиквитин-лигазной активности белка, кодируемого геном COP1 (Constitutive Photomorphogenic 1) [50]. В условиях тени, под действием дальнего красного света, указанная активность COP1 уменьшается, что и приводит к увеличению активных форм HFR1 [51] (рис. 3). Постепенное их накопление обеспечивает переход из стадии позитивной регуляции SAS в стадию негативной регуляции в том случае, когда этот синдром не привел к положительному результату.
Таким образом, как в случае ответа на засуху, так и в реакции на недостаток света продемонстрирована важная роль генов HD-Zip I–II в качестве потенциальных модуляторов адаптивных реакций растений на эти виды стресса. Наряду с представленными работами была показана корреляция уровней экспрессии указанных генов с другими видами абиотических стрессовых воздействий, такими как засоленность, низкая температура [21, 52, 53], однако в этих случаях механизмы регуляции пока не ясны. Пока неизвестны примеры участия генов HD-Zip I–II в реакции на биотические факторы. Все известные примеры такого участия ограничиваются классом генов HD-Zip IV (см. далее).
ЗАЩИТА РАСТЕНИЙ ОТ ПАТОГЕНОВ
Как известно, кутикула растений, или неклеточный слой, покрывающий поверхностный эпителий и состоящий из воскоподобного вещества кутина, выполняет важную защитную функцию не только от абиотических воздействий, но и от патогенов [54]. Регуляция активности генов, вовлеченных в биосинтез кутикулы, пока недостаточно изучена. У арабидопсиса было обнаружено несколько путей биосинтеза, один из которых включает ген CFL1 (Curly Flag Leaf1) [55]. При этом было показано, что белок HDG1, относящийся к классу HD-Zip IV, взаимодействует с этим геном, негативно влияя на его активность. Кроме этого, был исследован ген ZmOCL1 того же класса у кукурузы и показано его участие в регуляции ряда генов, которые отвечают за транспорт и метаболизм липидов и могут участвовать в формировании кутикулы и определении ее структурных и физических свойств [56]. В числе этих генов-мишеней белка ZmOCL1: nsLTPII (белок переноса липидов типа 2), AtCXE-18 (карбоксилэстераза), WBC11/ABCG11 (переносчики АВС-типа), SEC14 (фосфатидил-инозитол-транспортный белок). Белок LTPII, кодируемый первым геном, играет существенную роль в транспорте липидов кутикулы через клеточную стенку [57]. Было установлено, что некоторые LTP-белки принимают участие в защите от грибных инфекций и насекомых [58, 59]. Кроме того, было показано, что сверхэкспрессия гена HvLTP2 у ячменя повышает устойчивость к бактерии Pseudomonas syringae [60], тогда как мутация в гене LPTG1 арабидопсиса приводит к чувствительности к грибным инфекциям [61]. К субсемейству LTP белков с выраженным антигрибным и антибактериальным действием относятся дефензины растений (γ-тионины) [62]. У пшеницы группа этих генов специфически экспрессируется во внешнем клеточном слое развивающегося зерна [63]. Как было показано в трансгенных экспериментах, ген HD-Zip IV, индуцируемый в ответ на поранение, может активировать промоторы нескольких генов, кодирующих дефензины.
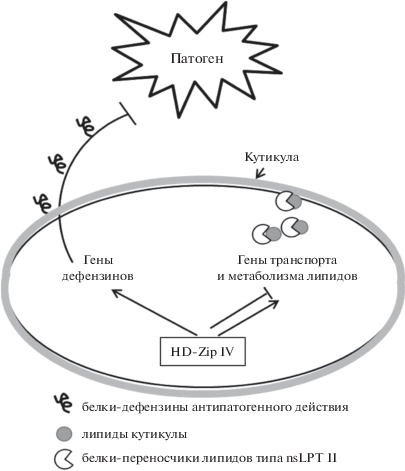
Таким образом, ТФ HD-Zip IV могут быть задействованы в системах защиты от патогенов либо прямо, активируя белки, имеющие антипатогенное действие, либо непрямым способом – через регуляцию белков, участвующих в формировании защитного слоя растений (рис. 4).
Рис. 4.
Участие ТФ HD-Zip IV в биотическом стрессе. Прямое антипатогенное действие дефензинов обозначено дугой с ограничителем на конце. Две параллельные линии со стрелкой и ограничителем указывают на разное влияние (как супрессирующее, так и индуцирующее) белковых факторов HD-Zip IV на гены, связанные с транспортом и метаболизмом липидов.
ЗАКЛЮЧЕНИЕ
Участие HD-Zip-генов в адаптивных реакциях растений реализуется через разнообразные механизмы, связанные со специфическими для каждого внешнего фактора сигнальными генетическими системами. При этом данные гены являются промежуточным звеном, объединяющим различные каскады и направляющим сигналы внешней среды на гены-эффекторы устойчивости. Это и определяет важное значение генов HD-Zip в селекции в качестве модуляторов целого комплекса адаптивных реакций растений к отдельным внешним, в том числе неблагоприятным, факторам.
Работа выполнена при финансовой поддержке Российского научного фонда (проект № 14-14-00161).
Список литературы
Garber R.L., Kuroiwa A., Gehring W.J. et al. Genomic and cDNA clones of the homeotic locus Antennapedia in Drosophila // EMBO J. 1983. V. 2. P. 2027–2036. doi 10.1002/j.1460-2075.1983.tb01696.x
Moens C.B., Selleri L. Hox cofactors in vertebrate development // Dev. Biol. 2006. V. 291. P. 193–206. doi 10.1016/j.ydbio.2005.10.032
Vollbrecht E., Veit B., Sinha N., Hake S. The developmental gene Knotted-1 is a member of a maize homeobox gene family // Nature. 1991. V. 350. P. 241–243. doi 10.1038/350241a0
Bharathan G., Janssen B.-J., Kellogg E.A., Sinha N. Did homeodomain proteins duplicate beforethe origin of angiosperms, fungi, and metazoa? // Proc. Natl Acad. Sci. USA. 1997. V. 94. P. 13749–13753.
Sessa G., Morelli G., Ruberti I. The Athb-1 and-2 HD-Zip domainshomodimerize forming complexes of different DNA binding specificities // EMBO J. 1993. V. 12. P. 3507–3517.
Schena M., Davis R.W. HD-Zip protein members of Arabidopsis homeodomain protein superfamily // Proc. Natl Acad. Sci. USA. 1992. V. 89. P. 3894–3898.
Zhong R., Ye Z-H. IFL1, a gene regulating interfascicular fiberdifferentiation in Arabidopsis, encodes a homeodomain-leucine zipperprotein // Plant Cell. 1999. V. 11. P. 2139–2152. doi 10.1105/tpc.11.11.2139
McConnell J.R., Emery J., Eshed Y. et al. Role of PHABULOSA and PHAVOLUTA in determining radial patterningin shoots // Nature. 2001. V. 411. P. 709–713. doi 10.1038/35079635
Ohashi-Ito K., Fukuda H. HD-Zip III homeobox genes that includea novel member, ZeHB-13 (Zinnia)/ATHB-15 (Arabidopsis), are involvedin procambium and xylem cell differentiation // Plant Cell Physiol. 2003. V. 44. P. 1350–1358. doi 10.1093/ pcp/pcg164
Rerie W.G., Feldmann K.A., Marks M.D. The glabra2 gene encodesa homeodomain protein required for normal trichome development in Arabidopsis // Genes Dev. 1994. V. 8. P. 1388–1399. doi 10.1101/gad.8.12.1388
Di Cristina M., Sessa G., Dolan L. et al. The Arabidopsis Athb-10 (GLABRA2) is an HD-Zip protein requiredfor regulation of root hair development // Plant J. 1996. V. 10. P. 393–402. doi 10.1046/j.1365-313X.1996.10030393.x
Masucci J., Rerie W., Foreman D. et al. The homeobox gene GLABRA2 is required forposition-dependent cell differentiation in the root epidermis of Arabidopsis thaliana // Development. 1996. V. 122. P. 1253–1260.
Ohashi Y., Oka A., Ruberti I. et al. Ectopically additive expression of GLABRA2 alters the frequency and spacing of trichome initiation // Plant J. 2002. V. 29. P. 359–369. doi 10.1046/j.0960-7412.2001.01214.x
Abe M., Katsumata H., Komeda Y., Takahashi T. Regulation of shoot epidermal cell differentiation by a pair of homeodomain proteins in Arabidopsis // Development. 2003. V. 130. P. 635–643. doi 10.1242/dev.00292
Chan R.L., Gago G.M., Palena C.M., Gonzalez D.H. Homeoboxes inplant development // Biochim. Biophys. Acta. 1998. V. 1442. P. 1–19. doi 10.1016/ S0167-4781(98)00119-5
Aoyama T., Dong C., Wu Y. et al. Ectopic expression of the Arabidopsis transcriptional activator Athb-1 alters leaf cell fate in tobacco // Plant Cell. 1995. V. 7. P. 1773–1785. doi 10.1105/tpc.7.11.1773
Hanson J., Johannesson H., Engstrom P. Sugar-dependent alterations in cotyledon and leaf development in transgenic plants expressing the HDZip gene ATHB13 // Plant Mol. Biol. 2001. V. 45. P. 247–262.
Li G., Yu M., Fang T. et al. Vernalization requirement duration in winter wheat is controlled by TaVRN-A1 at the protein level // Plant J. 2013. V. 76. P. 742–753. doi 10.1111/tpj.12326
Brandt R., Cabedo M., Xie Y., Wenkel S. Homeodomain leucine-zipper proteins and their role in synchronizing growth and development with the environment // J. Integr. Plant Biol. 2014.V. 56. P. 518–526. doi 10.1111/ jipb.12185
Ariel F.D., Manavella P.A., Dezar C.A., Chan R.L. The true story of the HD-Zip family // Trends Plant Sci. 2007. V. 12. № 9. P. 419–426. doi 10.1016/ j.tplants.2007.08.003
Henriksson E., Olsson A.S.B., Johannesson H. et al. Homeodomain leucine zipper class I genes in Arabidopsis. Expression patterns and phylogenetic relationships // Plant Physiology. 2005. V. 139. P. 509–518. doi 10.1104/pp.105.063461
Agalou A., Purwantomo S., Overnaes E. et al. Agenome-wide survey of HD-Zip genes in rice and analysis of drought responsive family members // Plant Mol. Biol. 2008. V. 66. P. 87–103. doi 10.1007/s11103-007-9255-7
Tron A.E., Bertoncini C.W., Chan R.L., Gonzalez D.H. Redox regulation of plant homeodomain transcription factors // J. Biol. Chem. 2002. V. 277. P. 34800–34807. doi 10.1074/jbc.M203297200
Tron A.E., Comelli R.N., Gonzalez D.H. Structure of homeodomain-leucine zipper/DNA complexes studied using hydroxyl radical cleavage of DNA and methylation interference // Biochemistry. 2005. V. 44. P. 16796–16803. doi 10.1021/bi0513150
Palena C.M., Tron A.E., Bertoncinim C.W. et al. Positively charged residues at the N-terminalarm of the homeodomain are required for efficient DNA binding byhomeodomain-leucine zipper proteins // J. Mol. Biol. 2001. V. 308. P. 39–47. doi 10.1006/jmbi.2001.4563
Tron A.E., Welchen E., Gonzalez D.H. Engineering the loop region of a homeodomain-leucinezipper protein promotes efficient binding to a monomeric DNA binding site // Biochemistry. 2004. V. 43. P. 15845–15851. doi 10.1021/bi048254a
Ponting C.P., Aravind L. START: A lipid-binding domain in StAR, HD-ZIP and signaling proteins // Trends Biochem. Sci. 1999. V. 24. P. 130–132. doi 10.1016/S0968-0004(99)01362-6
Schrick K., Nguyen D., Karlowski W.M., Mayer K.F. START lipid/sterol-binding domainsare amplified in plants and are predominantly associated with homeodomain transcription factors // Genome Biol. 2004. V. 5(6). R41. doi 10.1186/gb-2004-5-6-r41
Ohashi Y., Oka A., Rodrigues-Pousada R. et al. Modulation of phospholipid signaling by GLABRA2 in root-hair pattern formation // Science. 2003. V. 300. P. 1427–1430. doi 10.1126/science.1083695
Kumar R., Thompson E.B. Gene regulation by the glucocorticoid receptor: Structure: Function relationship // J. Steroid Biochem. Mol. Biol. 2005. V. 94. P. 383–394. doi 10.1016/j.jsbmb.2004.12.046
Zhang F., Zuo K., Zhang J. et al. An L1 box binding protein, GbML1, interacts with GbMYB25 to control cotton fibre development // J. Exp. Bot. 2010. V. 61. P. 3599–3613. doi 10.1093/jxb/erq173
Mukherjee K., Bürglin T.R. MEKHLA, a novel domain with similarity to PAS domains, isfused to plant homeodomain-leucine zipper III proteins // Plant Physiol. 2006. V. 140. P. 1142–1150. doi 10.1104/pp.105.073833
Skirycz A., Inzé D. More from less: plant growth under limited water // Curr. opinion Biotechnol. 2010. V. 21. P. 197–203. doi 10.1016/j.copbio.2010.03.002
Shinozaki K., Yamaguchi-Shinozaki K. Gene networks involved in drought stress response and tolerance // J. Exp. Bot. 2007. V. 58. P. 221–227. doi 10.1093/ jxb/erl164
Manavella P.A., Arce A.L., Dezar C.A. et al. Cross-talk between ethylene and drought signalling pathways is mediated by the sunflower Hahb-4 transcription factor // Plant J. 2006. V. 48(1). P. 125–137. doi 10.1111/j.1365-313X.2006.02865.x
Harris J.C., Sornaraj P., Taylor M. et al. Molecular interactions of the γ-clade homeodomain-leucine zipper class I transcription factors during the wheat response to water deficit // Plant Mol. Biol. 2016. V. 90. P. 435–452. doi 10.1007/s11103-015-0427-6
Dezar C.A., Gago G.M., Gonzalez D.H., Chan R.L. Hahb-4, a sunflower homeobox-leucine zipper gene, is a developmental regulator and confers drought tolerance to Arabidopsis thaliana plants // Transgenic Res. 2005. V. 14. P. 429–440.
Yang S.F., Hoffman N.E. Ethylene biosynthesis and its regulation inhigher-plants // Annu. Rev. Plant Physiol. Mol. Biol. 1984. V. 35. P. 155–189.
Chao Q., Rothenberg M., Solano R. et al. Activation of the ethylene gas response pathway in Arabidopsis by the nuclear protein ETHYLENE-INSENSITIVE3 and related proteins // Cell. 1997. V. 89. P. 1133–1144. doi org/10.1016/S0092-8674(00)80300-1
Sakamoto A., Murata N. Genetic engineering of glycinebetaine synthesis in plants: current status and implications forenhancement of stress tolerance // J. Exp. Bot. 2000. V. 51. P. 81–88.
Capell T., Bassie L., Christou P. Modulation of the polyamine biosynthetic pathway in transgenic rice confers tolerance to drought stress // Proc. Natl Acad. Sci. USA. 2004. V. 101. P. 9909–9914. doi 10.1073/ pnas.0306974101
Yu H., Chen X., Hong Y.-Y. et al. Activated expression of an Arabidopsis HD-START protein confers drought tolerance with improved root system and reduced stomatal density // Plant Cell. 2008. V. 20. P. 1134–1151. doi 10.1105/tpc.108.058263
Chen M., Chory J., Fankhauser C. Light signal transduction in higher plants // Annu. Rev. Genet. 2004. V. 38. P. 87–117.
Salter M.G., Franklin K.A., Whitelam G.C. Gating of the rapid shade avoidance response by the circadian clock in plants // Nature. 2003. V. 426. P. 680–683. doi 10.1038/nature02174
Carabelli M., Morelli G., Whitelam G., Ruberti I. Twilight zone and canopy shade induction of the ATHB-2 homeobox gene in green plants // Proc. Natl Acad. Sci. USA. 1996. V. 93. P. 3530–3535.
Sessa G., Carabelli M., Sassi M. et al. A dynamic balance between gene activation and repression regulates the shade avoidance response in Arabidopsis // Genes & Development. 2005. V. 19. P. 2811–2815. doi 10.1101/ gad.364005
Iglesias M.J., Sellaro R., Zurbriggen M.D. et al. Multiple links between shade avoidance and auxin networks // J. Exp. Bot. 2017. V. 69. P. 213–218. doi 10.1093/ jxb/erx295
Chapman E.J., Estelle M. Mechanism of auxin-regulated gene expression in plants // Annu. Rev. Genet. 2009. V. 43. P. 265–285. doi 10.1146/annurev-genet-102108-134148
Hornitschek P., Lorrain S., Zoete V. et al. Inhibition of the shade avoidance response by formation of non-DNA binding bHLH heterodimers // EMBO J. 2009. V. 28. P. 3893–3902. doi 10.1038/emboj.2009.306
Lau O.S., Deng X.W. The photomorphogenic repressors COP1 and DET1: 20 years later // Trends in Plant Science. 2012.V. 17. P. 584–593. doi 10.1016/j.tplants.2012.05.004
Pacín M., Semmoloni M., Legris M. et al. Convergence of CONSTITUTIVE PHOTOMORPHOGENESIS 1 and PHYTOCHROME INTERACTING FACTOR signalling during shade avoidance // New Phytol. 2016. V. 211. P. 967–979. doi 10.1111/nph.13965
Ariel F., Diet A., Verdenaud M. et al. Environmental regulation of lateral root emergence in Medicago truncatula requires the HD-Zip I transcription factor HB1 // Plant Cell. 2010. V. 22. P. 2171–2183. doi 10.1105/tpc.110.074823
Chen X., Chen Z., Zhao H. et al. Genome-wide analysis of soybean HD-Zip gene family and expression profiling under salinity and drought treatments // PLoS One. 2014. V. 9. e87156. doi 10.1371/journal.pone.0087156
Jeffree C.E. The fine structure of the plant cuticle // Annual Plant Reviews. V. 23: Biology of the Plant Cuticle. Oxford, UK: Blackwell Publ. Ltd., 2007. P. 11–125.
Wu R., Li S., He S. et al. CFL1, aWW domain protein, regulates cuticle development by modulating the function of HDG1, a class IV homeodomain transcription factor, in rice and Arabidopsis // Plant Cell. 2011. V. 23. P. 3392–3411. doi 10.1105/tpc.111.088625
Javelle M., Vernoud V., Depege-Fargeix N. et al. Overexpression of the epidermis-specific homeodomain-leucine zipper IV transcription factor Outer Cell Layer1 in maize identifies target genes involved in lipid metabolism and cuticle biosynthesis // Plant Physiol. 2010. V. 154. P. 273–286. doi 10.1104/pp.109
DeBono A., Yeats T.H., Rose J.K.C. et al. Arabidopsis LTPG is a glycosyl phosphatidylinositol-anchored lipid transfer protein required for export oflipids to the plant surface // Plant Cell. 2009. V. 21. P. 1230–1238. doi 10.1105/tpc.108.064451
Zottich U., da Cunha M., Carvalho A.O. et al. Purification, biochemical characterization and antifungal activity of a new lipid transfer protein (LTP) from Coffea canephora seeds with alpha-amylase inhibitor properties // Biochim. Biophys. Acta. 2011. V. 4. P. 375–383. doi 10.1016/j.bbagen.2010.12.002
Boutrot F., Chantret N., Gautier M.-F. Genome-wide analysis of the rice and Arabidopsis non-specific lipid transfer protein (nsLtp) gene families and identification of wheat nsLtp genesby EST data mining // BMC Genomics. 2008. V. 9(86). P. 1–19. doi 10.1186/1471-2164-9-86
Molina A., Garcia-Olmedo F. Enhanced tolerance to bacterial pathogens caused by the transgenic expression of barley lipid transfer protein LTP2 // Plant J. 1997. V. 12. P. 669–675. doi 10.1046/j.1365-313X.1997.00605.x
Lee S.B., Go Y.S., Bae H.J. et al. Disruption of glycosylphosphatidylinositol-anchored lipid transfer protein gene altered cuticular lipid composition, increased plastoglobules, and enhanced susceptibility to infection by the fungal pathogen Alternaria brassicicola // Plant Physiol. 2009. V. 150. P. 42–54. doi 10.1104/ pp.109.137745
Thomma B.P., Cammue B.P., Thevissen K. Plant defensins // Planta. 2002. V. 216(2). P. 193–202. doi 10.1007/s00425-002-0902-6
Kovalchuk N., Li M., Wittek F. et al. Defensin promoters as potential tools for engineering disease resistance in cereal grains // Plant Biotech. J. 2010. V. 8. P. 47–64. doi 10.1111/j.1467-7652.2009.00465.x
Дополнительные материалы отсутствуют.


