Генетика, 2019, T. 55, № 10, стр. 1198-1207
Сигналы направленного отбора в популяциях народов Сибири и европейской части России
В. А. Степанов 1, *, В. Н. Харьков 1, К. В. Вагайцева 1, И. Ю. Хитринская 1, А. В. Бочарова 1, Н. А. Колесников 1, А. А. Зарубин 1, А. А. Попович 1, А. В. Марусин 1, М. Г. Сваровская 1, П. Триска 2, Т. В. Татаринова 3
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
2 Второй медицинский факультет, Карлов университет
11100 Прага, Чехия
3 Факультет биологии, Университет Ла Верн
CA91750 Ла Верн, Калифорния, США
* E-mail: vadim.stepanov@medgenetics.ru
Поступила в редакцию 07.02.2019
После доработки 16.05.2019
Принята к публикации 21.05.2019
Аннотация
Адаптация популяций человека к экстремальными климато-географическим условиям среды обитания, опосредованная естественным отбором, может являться одним из основных факторов формирования генетической структуры популяций Северной Евразии. Используя данные полногеномного набора однонуклеотидных полиморфных маркеров (SNP), мы провели поиск сигналов направленного отбора в 5 популяциях Сибири и европейской части России. Для каждой из популяций обнаружено от 113 до 185 регионов генома, формирующих протяженные гомозиготные блоки гаплотипов, содержащие в совокупности 771 ген. Кросс-популяционный поиск мишеней отбора выявил еще около 150 участков генома, в том числе – 57 пересекающихся с результатами гаплотипического анализа в отдельных популяциях. Среди локусов генома, несущих наиболее выраженные сигналы направленного отбора в северных популяциях, можно выделить области генов SLC30A9, CACNA1C, KCNQ5, ABCA1, ALDH1A2, CSMD1, RBFOX1, WWOX и ряд других. Биоинформационный анализ показал, что основными биологическими процессами, в которые вовлечены мишени отбора в северных популяциях, являются процессы ответа на внешние стимулы, включая белки, нутриенты и глюкозу, и защитные реакции организма, включая воспалительный ответ. Сеть белок-белковых взаимодействий продуктов генов, находящихся в регионах направленного отбора в геноме, формирует выраженные кластеры, соотносящиеся с некоторыми из указанных биологических процессов. Результаты работы свидетельствуют, что механизмы не-нейтральной микроэволюции могут играть существенную роль в формировании генетической структуры популяций человека в ходе долговременной адаптации к неблагоприятным условиям среды обитания.
Генетическая структура популяций человека формировалась в ходе расселения современного человека из Африки и в период последующих миграций, процессов метисации, изоляции расстоянием и обмена генами. Среди факторов популяционной динамики, сыгравших роль в формировании наблюдаемых в популяциях частот генов и генотипов, несомненно, присутствует и естественный отбор. Однако роль отбора относительно других факторов динамики в глобальном масштабе относительно невелика, и выявление сигналов отбора на фоне доминирующих эффектов потока генов и генетического дрейфа представляет собой нетривиальную задачу.
Поиск сигналов естественного отбора в популяциях человека на полногеномном уровне является одним из актуальных направлений современной популяционной геномики человека. Использование больших геномных данных позволяет хотя бы частично преодолеть проблему относительной слабости сигналов отбора и выявить их на протяженных участках генома, даже если на уровне отдельных генетических маркеров эти сигналы стерты действием более мощных факторов – миграций и генетического дрейфа. Разработан ряд современных тестов, применяемых для поиска сигналов естественного отбора на геномных гаплотипических данных и данных по частотам аллелей – тест на “длинные гаплотипы” (LRH, long range haplotypes), протяженные гомозиготные гаплотипы (EHH, extended haplotype homozygosity), интегрированный гаплотипический балл iHS (integrated haplotype score), кросс-популяционный тест протяженных гомозиготных гаплотипов XP-EHH (cross-population extended haplotype homozygosity), тест PBS (population branch statistic) и др. [1–4].
Одной из частей ареала современного человека, где направленный отбор мог играть существенную роль в формировании генетической структуры популяции, адаптированной к средовым условиям, является Северная Евразия. Этот регион, расположенный в зонах резко континентального, субарктического и арктического климата, характеризуется наиболее суровыми условиями среды обитания. Ряд недавних исследований показывает, что генетические системы, связанные с жировым метаболизмом, устойчивостью к температурным шокам, тонусом сосудистого русла и др., могут играть существенную роль в адаптации коренных популяций к неблагоприятным или экстремальным климато-географическим условиям, локальным условиям среды обитания, способу хозяйствования [5–9]. В наших предыдущих работах нам удалось выявить связь генетического разнообразия определенных генов и их генетических маркеров в популяциях Северной Евразии с климато-географическими условиями среды обитания и обнаружить сигналы действия естественного отбора в ряде генов, кандидатных для адаптации к холодному климату [10–14].
Задачей настоящего исследования является поиск сигналов действия естественного отбора в популяциях северной Евразии в рамках полногеномного позиционного подхода с помощью анализа гомозиготных гаплотипических блоков.
МАТЕРИАЛЫ И МЕТОДЫ
Популяционные выборки и генотипирование. В работе были использованы данные широкогеномного генотипирования пяти популяций населения Сибири, европейской части России на высокоплотных матрицах “Illumina”, опубликованные нами ранее [15]. В анализ были включены выборки из популяций коренных народов Сибири – хантов (N = 29) и кето (N = 31); и народов европейской части России: коми (N = 32), карелов (N = 35) и удмуртов (N = 30). Кроме того в сравнительном кросс-популяционном анализе были использованы данные по популяциям украинцев (N = 36) из той же работы и японцев (N = 120) из проекта “1000 геномов” [16]. Выборки кетов, коми, карелов, удмуртов и украинцев были генотипированы на микроматрицах “Illumina Infinium 610-Quad” (610 тыс. SNP), выборка хантов – на “Illumina Infinium 370-Duo” (370 тыс. SNP).
Тесты на естественный отбор. Поиск сигналов направленного отбора в популяциях Сибири, Северной Азии и севера европейской части России на основе анализа данных по полногеномному набору SNP проводили в пакете SelScan с помощью геномных тестов на протяженную гомозиготность гаплотипов (iHS и XP-EHH) [17]. Оба теста основаны на том, что направленный отбор по какому-либо маркеру приводит к гомозиготизации участка генома вокруг мишени отбора за счет того, что отбирается весь гаплотип, сцепленный с мишенью. Тесты iHS/nSL и XP-EHH взаимно дополняют друг друга, поскольку первый эффективен при относительно невысоких частотах отбираемого аллеля, а второй – при высоких и близких к фиксации частотах. Расчеты для обоих тестов проводили вдоль всего генома в пределах рамки в 10 тпн при числе SNP в рамке не менее 20.
Тест iHS (интегрированный гаплотипический балл, integrated haplotype score) оценивает степень гомозиготности гаплотипа (EHH, extended haplotype homozygosity) вокруг тестируемого SNP при сравнении гаплотипов для предкового и производного аллелей [3]. В пакете SelScan реализован усовершенствованный вариант теста iHS – статистика nSL (number of Segregating sites by Length) [18]. Ключевое отличие теста nSL от классического iHS заключается в том, что nSL сравнивает длину гомозиготных фрагментов на двух гаплотипах (для предкового и производного аллелей) в терминах числа мутаций (полиморфных сайтов), а не пар нуклеотидов. Тем самым nSL не зависит от привязки к генетической карте и является более устойчивым к эффектам вариабельности в частоте рекомбинации или темпе мутирования. Кроме того, это свойство nSL делает его более удобным для анализа данных с относительно низкой плотностью маркерного покрытия, как, например, в случае наших данных для полногеномных чипов емкостью 610 тыс. SNP.
Для теста nSL в качестве сигнала направленного отбора выбирали участки генома, для которых абсолютное эмпирическое значение стандартизованной статистики |nSL| превышало границу 98% квантиля. Иначе говоря, выявляли гаплотипы-аутлайеры, имеющие необычно высокую степень гомозиготизации вокруг предкового или производного аллеля, попадающую в 2 крайних процента распределения эмпирических значений. В качестве дополнительных показателей, оценивающих степень гомозиготизации блока в области сигнала отбора, оценивали число SNP в блоке с экстремальными значениями статистики iHS (|iHS| > 2), общее число SNP в блоке и долю SNP с экстремальными значениями iHS.
Кросс-популяционный тест на протяженную гомозиготность гаплотипа направлен на выявление SNP, находящихся под действием отбора лишь в одной из двух сравниваемых популяций. XP-EHH (cross-population extended haplotype homozygosity) оценивает гомозиготизацию блока вокруг тестируемого SNP при сравнении гаплотипов в двух разных популяциях – т.е. тестирует различия в уровне гомозиготизации участков генома в изучаемой популяции по сравнению с контрольной. Для XP-EHH в качестве достоверного сигнала отбора использовали значения Z-балла (‒log(p-value)) больше 2 для каждого из SNP в рамке. Кросс-популяционный тест был проведен для всех популяций, кроме хантов, из-за более низкой плотности SNP в генотипических данных у последних по сравнению с контрольными популяциями.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Выявление сигналов направленного отбора
Результаты поиска гомозиготных гаплотипов, сформированных под действием направленного отбора в изучаемых популяциях, с помощью теста nSL приведены в табл. 1. Для каждой из популяций обнаружено от 113 до 185 регионов генома, формирующих протяженные гомозиготные блоки, с числом SNP в блоке от 20 до 122. Примечательно, что популяции европейской части России имеют большее число сигналов отбора (блоков протяженной гомозиготности), чем исследованные популяции Сибири. В общей сложности по всем популяциям в область блоков протяженной гомозиготности гаплотипов попадает 771 ген. Для 191 из них сигналы направленного отбора повторяются более чем в одной популяции, а остальные являются уникальными для отдельных популяций.
Таблица 1.
Результаты поиска сигналов направленного отбора с помощью теста nSL
| Популяция | Число регионов генома с |nSL| > 98% квантиля | Среднее число SNP в гомозиготном блоке (от–до) | Гены в десяти наиболее значимых гомозиготных регионах |
|---|---|---|---|
| Ханты | 113 | 27 (20–66) | KCNMA1 CRYBB1 CRYBA4 MIAT SLCO1B3 CFTR TIMP3 SYN3 SLC16A7 UCK1 UQCR10 |
| Кеты | 169 | 29 (20–81) | SLC30A9 EXOC6 CYP26C1 CYP26A1 ARHGEF7 C13orf16 GPR125 HDAC4 EYS FAM110A ANGPT4 ALDH1A2 |
| Коми | 183 | 29 (20–122) | SLC30A9 CACNA1C RHOJ INHBB PRKCH SOBP TBC1D15 MRS2P2 |
| Карелы | 185 | 31 (20–121) | SLC30A9 CDH10 TRIM39 HCG18 SETX C9orf171 TTF1 ELMO1 PPP2R2B COL6A5 ZNF323 ZKSCAN4 ZNF187 PGBD1 ZNF193 NKAPL |
| Удмурты | 182 | 28 (20–111) | SLC30A9 ACPL2 MAP4K5 CDKL1 SOX17 NRXN3 DNHD1 ARFIP2 FXC1 CACHD1 |
Тест XP-EHH, основанный на сравнении гомозиготных блоков в тестируемой и контрастной популяции сравнения оказался менее информативным для поиска сигналов отбора в наших данных. Для сравнения популяций холодного климата (Северная Европа и Сибирь), принадлежащих к монголоидному и европеоидному антропологическим типам, мы выбрали евразийские популяции, проживающие в умеренном климате – европеоидов (украинцы) и монголоидов (японцы). Популяции сравнения были выбраны таким образом, чтобы оценить межпопуляционные различия, связанные с климатической адаптацией в Евразии, а не в более глобальном масштабе. В случае более контрастных по происхождению популяций (например, при сравнении сибиряков с африканцами) мы, вероятно, тестировали бы отличия от африканцев, общие для евразийских популяций, а не внутриконтинентальную дифференциацию.
Кросс-популяционный тест выявил в исследованных популяциях от 22 до 28 регионов генома с экстремально повышенной гомозиготностью по сравнению с японцами и от 14 до 25 регионов при сравнении с украинцами. Обобщенные результаты поиска сигналов отбора с помощью кросс-популяционного теста на протяженную гомозиготность гаплотипов приведены в табл. 2. Всего кросс-популяционный тест обнаруживает участки генома, представленные 145 генами, 37 из которых повторяются в двух и более межпопуляционных сравнениях. 57 регионов повышенной гомозиготности, выявленные при межпопуляционных сравнениях, совпадают с участками экстремально высокой гомозиготности гаплотипов по данным теста nSL. В частности, среди совпадающих участков генома, демонстрирующих значимые сигналы направленного отбора, по результатам обоих тестов находятся локусы генов KCNQ5, ALDH1, SLC9A9, ABCA1, LRRN1, SLC16A10 и др. Всего же по результатам тестов nSL и XP-EHH выявляется 835 генов, расположенных в участках генома, несущих сигналы направленного отбора.
Таблица 2.
Результаты поиска сигналов направленного отбора с помощью теста XP-EHH
| Популяции сравнения | Число регионов генома со значимыми сигналами отбора | Среднее число SNP в гомозиготном блоке (от–до) | Гены в десяти наиболее значимых гомозиготных регионах |
|---|---|---|---|
| Кеты–японцы | 28 | 20–61 | KCNQ5 ALDH1 FAM110A ANGPT4 GUCY2E LRRC32 |
| LRRK1 ALDH1A3 PRKCH TMEM30B | |||
| AIFM2 H2AFY2 TYSND1 NCAM1 | |||
| Кеты–украинцы | 14 | 20–58 | OSBPL6 PRKG1 DYNC2LI1 ABCG8 ABCG5 SMC6 GEN1 |
| MSGN1 PGR INSC DAB1 | |||
| Карелы–японцы | 21 | 20–83 | LGALS3BP CANT1 TIMP2 NIPSNAP3B ABCA1 NIPSNAP3A |
| SETX C9orf171 TTF1 SOBP LRRN1 NTNG2 SETX PTGFR | |||
| ABCA1 | |||
| Карелы–украинцы | 21 | 20–41 | PTGFR TLR10 KLF3 TLR1 COL13A1 COL25A1 GPC5 |
| REG1B REG1A REG1P REG3A NBAS REG3G | |||
| Коми–японцы | 22 | 20–64 | KCNQ5 BNC2 SOBP NIPSNAP3B NIPSNAP3A INHBB |
| ABCA1 SLC12A1 | |||
| Коми–украинцы | 25 | 20–53 | ADAMTS8 INSR OR10G3 SALL2 COL13A1 IMMP2L NLGN1 |
| FMN2 GREM2 GNG12 GADD45A | |||
| Удмурты–японцы | 23 | 20–53 | SOX17 KCNQ5 EGFLAM SLC12A1 B3GALT1 ELMO1 AIFM2 |
| H2AFY2 TYSND1 PRUNE2 LRRN1 | |||
| Удмурты–украинцы | 24 | 20–57 | IMMP2L SLC16A10 KIAA1919 ERBB4 RMD3 PTGFR |
В табл. 3 приведены 19 генов, сигналы направленного отбора для которых найдены более чем в трех популяциях по результатам теста nSL или в трех и более межпопуляционных сравнениях по тесту XP-EHH. Среди наиболее ярких мишеней отбора следует отметить гаплотипы в локусе гена SLC30A9, который показывает наиболее экстремальные значения теста nSL в четырех популяциях – у кетов, карелов, коми и удмуртов (табл. 1). Ген SLC30A9 относится к семейству генов, кодирующих белки – переносчики растворимых веществ. Продукт гена является переносчиком ионов цинка и кроме трансмембранного переноса катионов задействован в процессах регуляции транскрипции. Недавно Zhang et al. [23] обнаружили сигналы дифференцирующего отбора в генах-транспортерах цинка, включая SLC30A9, в африканских и не-африканских популяциях. Гаплотипы гена SLC30A9 у африканцев йоруба в этой работе демонстрировали значимые сигналы направленного отбора гаплотипов по предковым аллелям, тогда как в популяциях Восточной Азии (у китайцев) отбор шел в пользу производных аллелей. В наших данных все четыре популяции характеризуются высокими положительными значениями nSL для этого участка генома, характеризующими однонаправленное действие отбора во всех популяциях в пользу производных (не-африканских) аллелей.
Таблица 3.
Гены под направленным отбором, представленные в более чем 3 популяциях (тест nSL) или в трех и более межпопуляционных сравнениях (XP-EHH)
| Ген | Функция продукта |
|---|---|
| MACROD2 | Деацетилаза О-ацетил-АДФ-рибозы. Сигнальная молекула, деацитилирующая лизиновые остатки в гистонах и других белках |
| SETX | РНК/ДНК хеликаза. Участвует в регуляции транскрипции, модулируя связывание РНК-полимеразы II с хроматином |
| ALDH1A2 | Ретинальдегидрогеназа 2, член генного семейства альдегид-дегидрогеназ |
| SLC30A9 | Трансмембранный переносчик катионов. Участвует в транспорте молекул цинка, задействован в процессах регуляции транскрипции |
| HDAC4 | Деацетилаза гистонов |
| CACNA1C | Вольтаж-зависимая субъединица кальциевого канала. Опосредует в транспорте ионов кальция в клетки и участвует во множестве кальций-зависимых процессов, включая сокращение мышц, вывод гормонов и нейротрансмиттеров и др. |
| PLEKHH2 | Член семейства белков, гомологичных плекстрину. Участвует в стабилизации F-актина, препятствуя его деполимеризации |
| STK32B | Серин/треонин-киназа |
| TYSND1 | Пероксисомальная протеаза. Участвует в окислении жирных кислот |
| RBFOX1 | РНК-связывающий белок, регулирующий альтернативный сплайсинг тканеспецифичных экзонов |
| LRRN1 | Нейронный белок, обогащенный повторами лейцина |
| PRKCH | Кальций-зависимая протеинкиназа, участвующая в дифференцировке кератиноцитов и рецепторов пре-бета-клеток. Блокирует рецептор эпидермального фактора роста |
| ERBB4 | Тирозиновая протеинкиназа, играющая роль поверхностного рецептора для нейрорегуляторов и эпидермального фактора роста |
| KCNQ5 | Калиевый канал, участвующий в активации и деактивации электропроводимости нейронов |
| EGFLAM | Белок сетчатки, пикачурин |
| PTGFR | Рецептор простагландина F2-альфа. Участвует в передаче сигналов в различных рецепторных комплексах |
| H2AFY2 | Вариант гистона H2A, репрессирующий транскрипцию |
| ABCA1 | Белок АТФ-связывающей кассеты. Рецептор анионов, участвующий во внутриклеточном транспорте холестерина |
| PRRC2A | Белок, принимающий участие в сплайсинге пре-мРНК |
| LRP1B | Рецептор липопротеинов низкой плотности |
| PPP2R2B | Серин/треонин фосфатаза, модулирующая субстратную селективность и каталитическую активность ряда ферментов. Участвует с процессах апоптоза и жизнеспособности нейронов |
| DDX39B | РНК-хеликаза сплайсосом. Участвует в транспорте сплайсированных и несплайсированных мРНК из ядра в цитоплазму |
| SFMBT2 | Репрессор транскрипции гена гомеобоксов HOXB13 |
| GPR98 | Белок комплекса G-рецептора, участвующий в регуляции развития ЦНС |
| PHLDB2 | Член семейства белков, гомологичных плекстрину. Участвует в формировании постсинаптического аппарата |
| CSMD1 | Субъединица фосфатазы 1, потенциальный супрессор плоскоклеточной карциномы. Варианты этого гена по нашим данным ассоциированы с неврологическим и психиатрическими заболеваниям и когнитивными функциями [19–22] |
| NRXN3 | Нейрексин 3. Поверхностный белок нейронов, участвующий в распознавании клеток и клеточной адгезии |
| WWOX | Оксидоредуктаза. Участвует в процессах супрессии опухолей и в апоптозе |
| AIFM2 | Апоптоз-индуцирующий фактор 2, оксидоредуктаза |
Среди других повторяющихся в исследованных популяциях регионов генома, показывающих значимые сигналы направленного отбора, можно выделить и другие гены, вовлеченные в трансмембранный транспорт ионов (CACNA1C, KCNQ5, ABCA1), а также гены, участвующие в межклеточной передаче сигналов (MACROD2, STK32B, PRKCH, ERBB4, PTGFR), апоптозе (WWOX, AIFM2), регуляции транскрипции (SETX, HDAC4, H2AFY2, SFMBT2), сплайсинге и транспорте мРНК (RBFOX1, PRRC2A, DDX39B), метаболизме липидов и жирных кислот (TYSND1, LRP1B, ABCA1), регуляции функций нервной системы и процессах распознавания внешних сигналов (LRRN1, EGFLAM, PPP2R2B, GPR98, PHLDB2, CSMD1, NRXN3).
Биоинформационный анализ биологических процессов и белок-белковых взаимодействий продуктов генов, находящихся под действием направленного отбора
Приведенные выше данные свидетельствуют, вероятно, о неслучайности кластеризации мишеней направленного отбора по функциям или биологическим процессам, в которые вовлечены находящиеся под отбором гены. Для проверки функциональных взаимосвязей выявленных генов и обогащенности ими биологических процессов и молекулярных функций мы провели биоинформационый анализ с помощью онлайн ресурсов DAVID [24, 25] и STRING [26, 27].
Обогащенность биологических процессов генами под отбором была оценена с помощью ресурса DAVID (The Database for Annotation, Visualization and Integrated Discovery). Из полного списка 853 генов, которые находятся в регионах генома, выявленных как мишени для направленного отбора по результатам тестов nSL и XP-EHH, 602 по данным DAVID оказались аннотированными, т.е. “привязанными” к определенным биологическим процессам или молекулярным функциям. 14 биологических процессов были статистически значимо, с учетом поправки Бенджамини–Хохберга, обогащены генами, находящимися под отбором. Это означает, что представленность генов в этих биологических процессах оказалась значимо выше по сравнению со случайными ожиданиями. Список этих биологических процессов представлен в табл. 4. Основными биологическими процессами, потребовавшими “настройки” генетической структуры популяций севера Евразии по механизму направленного отбора, явились процессы ответа на внешние стимулы, включая белки, нутриенты и глюкозу; защитные реакции организма, включая воспалительный ответ; сигнальные взаимодействия и презентация клетками рецепторов.
Таблица 4.
Биологические процессы, обогащенные генами, находящимися под действием направленного отбора в исследованных популяциях
| Биологический процесс (генная онтология) | Число генов | p, Бенжамини–Хохберг |
|---|---|---|
| GO:0006986 ответ на развернутый белок | 17 | 0.00017 |
| GO:0051789 ответ на белковые стимулы | 20 | 0.00029 |
| GO:0006952 защитный ответ | 55 | 0.00110 |
| GO:0010033 ответ на органические субстраты | 60 | 0.00257 |
| GO:0007584 ответ на нутриенты | 20 | 0.00746 |
| GO:0046325 отрицательная регуляция импорта глюкозы | 6 | 0.00845 |
| GO:0010829 отрицательная регуляция транспорта глюкозы | 6 | 0.01392 |
| GO:0006954 воспалительный ответ | 32 | 0.01765 |
| GO:0046324 регуляция импорта глюкозы | 9 | 0.02270 |
| GO:0007242 внутриклеточный сигнальный каскад | 85 | 0.02250 |
| GO:0010827 регуляция транспорта глюкозы | 9 | 0.02335 |
| GO:0042573 метаболизм ретиноевой кислоты | 6 | 0.02358 |
| GO:0043062 организация внеклеточных структур | 20 | 0.02387 |
| GO:0030030 организация проекции клеток | 33 | 0.04335 |
При этом процессы, связанные с регуляцией транскрипции, функционированием нервной системы и метаболизма липидов, при анализе полного списка генов не достигают статистически достоверного уровня обогащенности. Однако если ограничиться рассмотрением только высокоповторяющихся генов, представленных в табл. 3, то среди значимых процессов появляются синаптическая передача, развитие нервной системы и организация синапса. Процессы метаболизма холестерина и транспорта липидов выявляются как статистически значимо обогащенные генами под отбором при анализе пересекающихся по результатам тестов nSL и XP-EHH генов.
Белок-белковые взаимодействия продуктов генов под отбором были визуализированы и проанализированы с помощью ресурса STRING (Search Tool for the Retrieval of Interacting Genes/Proteins). Анализ был проведен с учетом доказательств функциональных связей между белками, полученных экспериментально, на основе их ко-экспрессии и совместной представленности в биологических процессах. Соупоминание в текстах публикаций и базах данных было исключено из критериев взаимодействия. При максимальном уровне доказательности связей (highest confidence, 0.9) из 815 белков, аннотированных в STRING, 616 оказались вовлечены в общие сети белок-белковых взаимодействий (уровень значимости обогащенности сети p = 0.00123). Полученная сеть представлена на рис. 1.
Рис. 1.
Белок-белковые взаимодействия продуктов генов, находящихся в выявленных регионах протяженной гомозиготности.
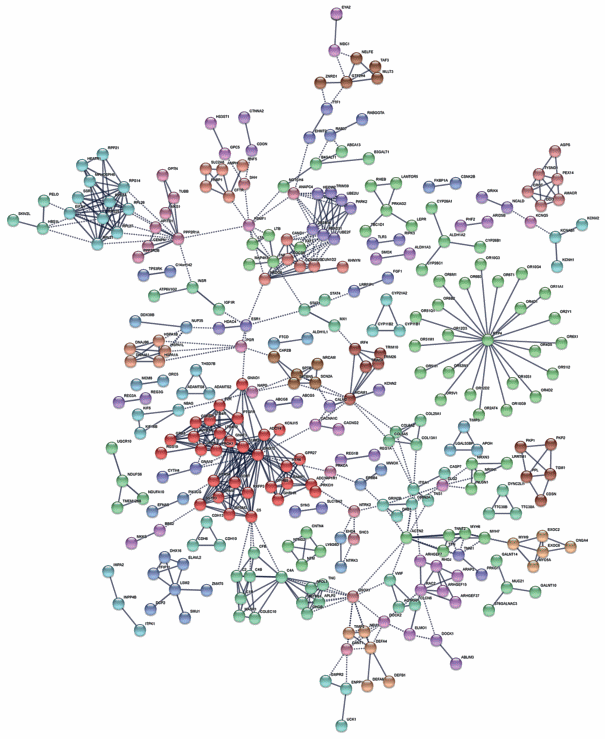
Кластеризация сети по алгоритму Маркова выявила 10 основных значимых кластеров, каждый из которых включает от 6 до 30 взаимосвязанных белков. Еще 62 кластера включали от 2 до 5 взаимосвязанных генных продуктов. Характеристика основных кластеров сети представлена в табл. 5. Главные кластеры сети родственны биологическим процессам, выявленным при анализе обогащенности, и включают белки, участвующие в сигнальных и рецепторных взаимодействиях, передаче сигналов, транспорте ионов, процессинге мРНК, гуморальной защите с помощью системы комплемента и др. Некоторым сюрпризом анализа сетевых взаимодействий оказался кластер обонятельных рецепторов, включающий 24 гена. Обогащенность сигналов отбора генами хеморецепторов не была выявлена другими подходами.
Таблица 5.
Основные кластеры сети белок-белковых взаимодействий продуктов генов, находящихся в регионах направленного отбора в геноме
| Номер кластера | Число белков | Характеристика кластера |
|---|---|---|
| 1 | 30 | Сигнальные и рецепторные взаимодействия, передача сигналов, транспорт ионов |
| 2 | 24 | Распознавание запахов. Кластер включает белок RTP 4 (транспортер рецепторов) в качестве центрального звена и 23 различных хеморецептора |
| 3 | 12 | В кластер входят компоненты комплемента и некоторые секреторные белки |
| 4 | 11 | Рибосомные белки и рибонуклеазы |
| 5 | 7 | Процессинг пре-мРНК |
| 6 | 7 | Комплекс убиквитин-лигаз и родственных им белков |
| 7 | 7 | Белки с ГТФазной активностью и факторы обмена гуанина |
| 8 | 7 | Серин/треониновые протеазы, центромерные и тубулиновые белки |
| 9 | 6 | Пероксисомальные белки |
| 10 | 6 | Куллин-связанные белки и белки убиквитин-лигазного комплекса |
Изученные в работе популяции большей частью являются достаточно изолированными. Их генетическая структура формировалась, вероятно, под существенным влиянием генетического дрейфа, который, так же как и направленный отбор, приводит к повышению уровня гомозиготности популяции и увеличению размеров гомозиготных блоков. Вследствие этого мы не можем исключить, что выявленная в изученных популяциях картина формирования протяженных гомозиготных блоков сложилась не только под действием направленного отбора, а является комплексным результатом эффектов основателя и отбора. В то же время следует заметить, что эффект дрейфа случаен в отношении регионов генома в разных популяциях, аллеля (предкового или произволдного) и функции гена. Полученные нами данные показывают довольно высокую степень повторяемости протяженных гомозиготных блоков в нескольких различных по происхождению популяциях, неслучайную направленность на производные аллели и функциональную взаимосвязь генов, несущих протяженные гомозиготные блоки. Кроме того, гомозиготные участки, сформировавшиеся под действием эффекта основателя, “размываются” со временем, укорачиваясь в каждом последующем поколении в условиях демографической экспансии и свободного скрещивания, тогда как блоки, сформированные отбором, стабилизируются при сохранении из поколения в поколение давления направленного отбора. Эти аргументы, а также применение тестов, специально разработанных для выявления сигналов отбора на фоне действия других факторов популяционной динамики и концентрация на наболее протяженных блоках, выпадающих из общей картины распределения гомозиготности по геному, позволяют нам рассматривать направленный отбор как ведущий фактор формирования повышенной гомозиготности в обсуждаемых регионах генома.
Таким образом, в проведенном исследовании с помощью тестов на поиск гомозиготных участков генома на полногеномном уровне в популяциях севера Евразии было выявлено существенное число сигналов направленного естественного отбора, вероятно, повлиявших на генетическую структуру популяции в процессе долговременной адаптации к климатогеографическим условиям обитания в резко континентальном или арктическом климате. Биоинформационный анализ показал, что основными биологическими процессами, в которые вовлечены мишени отбора в северных популяциях, являются процессы ответа на внешние стимулы, включая белки, нутриенты и глюкозу, и защитные реакции организма, включая воспалительный ответ. Большая часть генов, несущих сигналы направленного отбора, формирует сети взаимодействующих продуктов, вовлеченных в ключевые биологические процессы. В целом можно заключить, что механизмы не-нейтральной эволюции могут играть существенную роль в формировании генетической структуры популяций человека в ходе долговременной адаптации к неблагоприятным условиям среды обитания.
Работа поддержана грантом РФФИ № 18-29-13045 “Популяционная геномика и транскриптомика человека: поиск сигналов не-нейтральной эволюции”.
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики. От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Sabeti P.C., Reich D.E., Higgins J.M. et al. Detecting recent positive selection in the human genome from haplotype structure // Nature. 2002. V. 419. № 6909. P. 832–837. https://doi.org/10.1038/nature01140
Sabeti P.C., Varilly P., Fry B. et al. Genome-wide detection and characterization of positive selection in human populations // Nature. 2007. V. 449. № 7164. P. 913–918. https://doi.org/10.1038/nature06250
Voight B.F., Kudaravalli S., Wen X., Pritchard J.K. A map of recent positive selection in the human genome // PLoS Biol. 2006. V. 4. № 3. e72. https://doi.org/10.1371/journal.pbio.0040072
Hancock A.M., Witonsky D.B., Gordon A.S. et al. Adaptations to climate in candidate genes for common metabolic disorders // PLoS Genet. 2008. V. 4. № 2. e32. https://doi.org/10.1371/journal.pgen.0040032
Hancock A.M., Witonsky D.B., Alkorta-Aranburu G. et al. Adaptations to climate-mediated selective pressures in humans // PLoS Genet. 2011. V. 7. № 4. e1001375. https://doi.org/10.1371/journal.pgen.1001375
Lappalainen T., Salmela E., Andersen P.M. et al. Genomic landscape of positive natural selection in Northern European populations // Eur. J. Hum. Genet. 2010. V. 18. P. 471–478. https://doi.org/10.1038/ejhg.2009.184
Rasmussen M., Li Y., Lindgreen S. et al. Ancient human genome sequence of an extinct Palaeo-Eskimo // Nature. 2010. V. 463. P. 757–762. https://doi.org/10.1038/nature08835
Grossman S.R., Andersen K.G., Shlyakhter I. et al. Identifying recent adaptations in large-scale genomic data // Cell. 2013. V. 152. № 4. P. 703–713. https://doi.org/10.1016/j.cell.2013.01.035
Cardona A., Pagani L., Antao T. et al. Genome-wide analysis of cold adaptation in indigenous Siberian populations // PLoS One. 2014. V. 9. № 5. e98076. https://doi.org/10.1371/journal.pone.0098076
Степанов В.А., Канделария П., Кхо С. и др. Деканализация иммунного ответа при расселении современного человека: связь генетического разнообразия в генах иммунной системы с климато-геграфическими факторами // Мед. генетика. 2013. Т. 12. № 4. С. 8–18.
Чередниченко А.А., Трифонова Е.А., Вагайцева К.В. и др. Связь полиморфизма генов цитокинов и их рецепторов в популяциях человека с климатическими и географическими факторами // Генетика. 2014. Т. 50. № 10. С. 1254–1258. https://doi.org/10.7868/S0016675814100038
Степанов В.А. Эволюция генетического разнообразия и болезни человека // Генетика. 2016. Т. 52. № 7. С. 852–864. https://doi.org/10.7868/S0016675816070109
Stepanov V.A., Vagajceva K.V., Bocharova A.V., Khar’kov V.N. Development of multiplex genotyping method of polymorphic markers for genes involved in human adaptation to cold climate // Sci. Evol. 2016. V. 1. № 2. P. 92–101.
Cтепанов В.А., Харьков В.Н., Вагайцева К.В. и др. Поиск генетических маркеров адаптации к климату у населения Северной Евразии // Генетика. 2017. Т. 53. № 11. С. 1254–1266. https://doi.org/10.7868/S0016675817110121
Triska P., Chekanov N., Stepanov V. et al. Between Lake Baikal and the Baltic Sea: genomic history of the gateway to Europe // BMC Genet. 2017. V. 18. Suppl. 1. P. 110. https://doi.org/10.1186/s12863-017-0578-3
1000 Genomes Project Consortium, Auton A., Brooks L.D. et al. A global reference for human genetic variation // Nature. V. 526. № 7571. P. 68–74. https://doi.org/10.1038/nature15393
Szpiech Z.A., Hernandez R.D. selscan: an efficient multithreaded program to perform EHH-based scans for positive selection // Mol. Biol. Evol. 2014. V. 31. № 10. P. 2824–2827. https://doi.org/10.1093/molbev/msu211
Ferrer-Admetlla A., Liang M., Korneliussen T., Nielsen R. On detecting incomplete soft or hard selective sweeps using haplotype structure // Mol. Biol. Evol. 2014. V. 31. № 5. P. 1275–1291. https://doi.org/10.1093/molbev/msu077
Степанов В.А., Бочарова А.В., Марусин А.В. и др. Репликативный анализ ассоциаций генетических маркеров когнитивных признаков с болезнью Альцгеймера в российской популяции // Мол. биол. 2014. Т. 48. № 6. С. 952–962. https://doi.org/10.7868/S0026898414060160
Степанов В.А., Бочарова А.В., Садуакасова К.З. и др. Репликативное исследование подверженности шизофрении с ранним началом у казахов // Генетика. 2015. Т. 51. № 2. С. 227–235. https://doi.org/10.7868/S0016675815020149
Бочарова А.В., Степанов В.А., Марусин А.В., Харьков В.Н. и др. Анализ ассоциаций генетических маркеров шизофрении и ее когнитивных эндофенотипов // Генетика. 2017. Т. 53. № 1. С. 100–108. https://doi.org/10.7868/S0016675817010039
Stepanov V.A., Marusin A., Vagaytseva K. et al. Genetic variants in CSMD1 gene are associated with cognitive performance in normal elderly population // Genet. Res. Int. 2017. V. 2017. https://doi.org/10.1155/2017/6293826
Zhang C., Li J., Tian L. et al. Differential natural selection of human zinc transporter genes between african and Non-African populations // Sci. Rep. 2015. V. 5. P. 9658. https://doi.org/10.1038/srep09658
Huang D.W., Sherman B.T., Lempicki R.A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources // Nature Protoc. 2009. V. 4. № 1. P. 44–57. https://doi.org/10.1038/nprot.2008.211
Huang D.W., Sherman B.T., Lempicki R.A. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists // Nucl. Acids Res. 2009. V. 37. № 1. P. 1–13. https://doi.org/10.1093/nar/gkn923
Snel B., Lehmann G., Bork P., Huynen M.A. STRING: a web-server to retrieve and display the repeatedly occurring neighbourhood of a gene // Nucl. Acids Res. 2000. V. 28. № 18. P. 3442–3444.
Szklarczyk D., Morris J.H., Cook H. et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible // Nucl. Acids Res. 2017. V. 45. D1. D 362–368. https://doi.org/10.1093/nar/gkw937
Дополнительные материалы отсутствуют.


