Генетика, 2019, T. 55, № 10, стр. 1218-1230
Метод мультиплексного генотипирования полиморфных вариантов генов, ассоциированных с ожирением и индексом массы тела
Е. А. Трифонова 1, *, А. А. Попович 1, К. В. Вагайцева 1, А. В. Бочарова 1, М. М. Гавриленко 1, В. В. Иванов 1, В. А. Степанов 1
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
* E-mail: ekaterina.trifonova@medgenetics.ru
Поступила в редакцию 21.02.2019
После доработки 04.04.2019
Принята к публикации 14.05.2019
Аннотация
Ожирение является одной из глобальных проблем здравоохранения, приводящих к существенному экономическому и социальному ущербу как в развитых, так и в развивающихся странах. Избыточный вес и ожирение – ключевые факторы риска сахарного диабета, сердечно-сосудистых и онкологических заболеваний, обусловливающих высокую заболеваемость и смертность. В представленной статье разработан метод мультиплексного генотипирования полиморфных вариантов генов, ассоциированных с ожирением и вариабельностью показателя индекса массы тела (ИМТ), основанный на многолокусной ПЦР и MALDI-TOF-масс-спектрометрии молекул ДНК. Охарактеризованы частоты 51 однонуклеотидного полиморфизма генов-кандидатов ожирения в популяционной выборке русских г. Кемерово. Полученные результаты сопоставлены с данными для популяций из проекта “1000 геномов”. Показана ассоциация маркеров rs12446632 локуса LOC105371116 и rs16851483 гена RASA2 с вариабельностью ИМТ у русских г. Кемерово.
Ожирение представляет собой глобальную проблему здравоохранения, имеющую характер пандемии и ассоциированную со значительно возрастающим риском заболеваемости и смертности как в развитых, так и в развивающихся странах. По данным Всемирной организации здравоохранения, опубликованным в 2016 г., в мире более 1.9 млрд взрослых в возрасте старше 18 лет имели избыточный вес, из них свыше 650 млн страдали ожирением [1]. Распространенность избыточной массы тела и ожирения в Российской Федерации составляет 59.2 и 24.1% соответственно [2]. Результаты многочисленных эпидемиологических исследований выявили четкую связь между увеличением распространенности ожирения и ростом заболеваемости сахарным диабетом типа 2 (СД2), частоты сердечно-сосудистых (ССЗ), цереброваскулярных и онкологических заболеваний, а также репродуктивных нарушений, что обусловливает особую значимость данной проблемы.
Известно, что к развитию ожирения приводит дисбаланс между потребляемой и расходуемой энергией, в основе которого лежат нарушения метаболизма, генетическая предрасположенность, нарушение поведенческих реакций и влияние внешних факторов [3]. В настоящее время в зависимости от типа наследования выделяют несколько форм ожирения: моногенное ожирение, генетические синдромы, ассоциированные с ожирением, и ожирение, характеризующееся полигенным наследованием [4]. Генетическая компонента полигенной формы ожирения как самостоятельного фенотипа, так и как клинического проявления ряда метаболических и сердечно-сосудистых заболеваний, исследована достаточно подробно – описано более 1500 генетических маркеров, ассоциированных с ожирением или индексом массы тела (ИМТ), в рамках различных подходов (ассоциативных исследований типа “случай–контроль” для отдельных генетических маркеров, в полногеномном анализе ассоциаций (GWAS), метаанализах и др.) [5–8]. Однако, несмотря на огромное число работ по этой тематике, выполненных в ведущих лабораториях по всему миру, данные об ассоциации тех или иных полиморфных маркеров генов с развитием ожирения и вариабельностью показателя ИМТ в большинстве случаев носят противоречивый характер и требуют репликации в различных популяционных когортах. В связи с высоким уровнем наследуемости (по данным близнецовых исследований коэффициент наследуемости ИМТ может варьировать от 40 до 70%) и высокой медико-социальной значимостью данной проблемы изучение генетических основ ожирения и вариабельности ИМТ является чрезвычайно актуальной задачей и требует широких исследований в различных популяциях.
Цель данной работы заключается в разработке метода мультиплексного генотипирования полиморфных вариантов генов, ассоциированных с ожирением и вариабельностью показателя индекса массы тела согласно данным полногеномных ассоциативных исследований (GWAS). Возможное применение полученных мультиплексов связано с изучением молекулярных механизмов регуляции энергетического баланса, генетической архитектуры ожирения и наследственных факторов вариабельности ИМТ.
МАТЕРИАЛЫ И МЕТОДЫ
Формирование панели однонуклеотидных полиморфных маркеров
На основе анализа данных 92 полногеномных ассоциативных исследований, представленных в регулярно обновляющемся каталоге “GWAS Cataloge” национального института изучения генома человека США (National Human Genome Research Institute, NHGRS) [5], был сформирован список из 150 однонуклеотидных полиморфных маркеров (SNPs), ассоциированных с такими фенотипическими признаками как индекс массы тела и ожирение по результатам двух и более работ. Необходимо отметить, что большая часть данных SNPs (97%) была локализована в некодирующих участках генома (3'- и 5'-нетранслируемые регионы, интроны, межгенные регионы, ncRNA). 96 полиморфных маркеров из представленного списка были локализованы в 82 генах (список SNPs и перечень генов доступны по запросу у авторов), для 47 из которых были обнаружены специфичные и биологически значимые функциональные связи с помощью веб-ресурса “STRING” (рис. 1). Необходимо отметить, что только для девяти из этих генов (BDNF, FOXO3, FTO, GIPR, LOC105378797, LRRC53, MAP2K5, RARB и TFAP2B) была показана ассоциация с изученными фенотипами для нескольких SNPs.
Рис. 1.
Графическое отображение белок-белковых взаимодействий продуктов генов, ассоциированных по результатам GWAS с ожирением и показателем индекса массы тела. Толщина линии между белками отражает доверительный уровень (interaction score) связи.
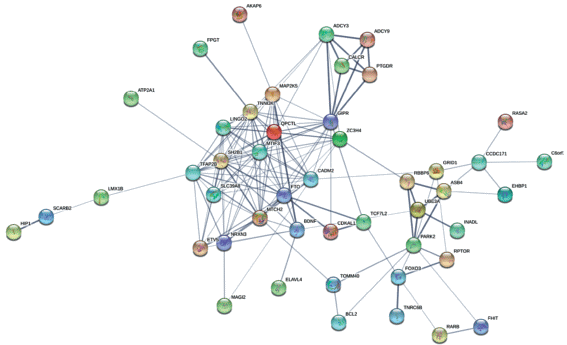
Анализ белок-белковых взаимодействий продуктов данных генов в базе данных “STRING” выявил несколько функционально значимых кластеров, включающих протеины, кодируемые следующими генами: ADCY3, ADCY9, AKAP6, AS, ASB4, ATP2A1, BCL2, BDNF, CALCR, CDKAL1, FHIT, FOXO3, FTO, GIPR, GNAT2, LRRC53, MAP2K5, MTCH2, PRKN, RARB, RBBP6, RPTOR, SH2B1, TCF7L2, TFAP2B, TNNI3K, TNRC6B, TOMM40, ZC3H4, которые вовлечены в такие GO-категории как клеточный гомеостаз, внутриклеточная трансдукция сигнала, положительная регуляция роста клеток, реакции клетки на внешние стимулы, межклеточные коммуникации (приводятся основные категории, для которых FDR < 0.0001).
Примечательно, что только 29 полиморфных маркеров (rs10182181, rs10938397, rs10968576, rs1121980, rs12446632, rs12463617, rs13130484, rs1516725, rs1558902, rs17381664, rs17782313, rs1800437, rs2030323, rs2112347, rs2206277, rs2207139, rs2531995, rs2568958, rs3101336, rs633715, rs7138803, rs7141420, rs7195386, rs7498665, rs7531118, rs887912, rs9568867, rs9816226, rs9941349) из 150 выявленных были ассоциированы с обоими из рассматриваемых фенотипов.
Дизайн мультиплекса, генотипирование образцов
В качестве технологии для генотипирования выбрали метод MALDI-TOF-масс-спектрометрии молекул ДНК в формате iPLEX Assay, реализованный на платформе “Sequenom MassARRAY4” (Agena Bioscience, США), включающей в себя систему для полимеразной цепной реакции (ПЦР) и масс-спектрометрическую установку для разделения амплифицированных продуктов по их массе и заряду. Система iPLEX Gold является точной и быстрой технологией для генотипирования однонуклеотидных полиморфных маркеров генов (SNP), позволяющей проводить высокопроизводительный (до 40 SNP в одной реакции) анализ ампликонов значительной протяженности, что подтверждается результатами многочисленных исследований [9, 10]. Подбор последовательностей праймеров для мультиплексного генотипирования методом MALDI-TOF-масс-спектрометрии проводили с помощью онлайн ресурса “Assay Design Suite v20” [11]. При подборе праймеров для мультиплексирования SNPs, показавшие ассоциацию в трех и более полногеномных исследованиях и/или локализованные в генах, продукты которых включены в кластеры белок-белковых взаимодействий, либо ассоциированные как с ожирением, так и с показателем ИМТ, отметили как приоритетные. В результате были сформированы два мультиплекса, содержащих 58 SNP (37 SNP, локализованных в 30 генах, и 21 SNP, расположенный в межгенных регионах), краткая характеристика которых представлена в табл. 1.
Таблица 1.
Характеристика маркеров, включенных в мультиплексы
| № п/п | ID SNP | Ген | Продукт гена | Локализация | Хромосомная локализация | Аллели | Предковый аллель | Ссылка |
|---|---|---|---|---|---|---|---|---|
| Мультиплекс 1 | ||||||||
| 1 | rs10938397 | – | – | Межгенная | chr4:45180510 | A/G | A | [24–32] |
| 2 | rs10968576 | LINGO2 | Белок 2, взаимодействующий с рецептором Nogo, содержащий богатые лейцином повторы и иммуноглобулиноподобный домен | Интрон | chr9:28414341 | A/G | A | [25–27, 29] |
| 3 | rs1121980 | FTO | Альфа-кетоглутарат-зависимая диоксигеназа | Интрон | chr16:53775335 | C/G/T | T | [33, 34] |
| 4 | rs11847697 | – | – | Межгенная | chr14:30045906 | C/T | C | [25, 27, 35] |
| 5 | rs12446632 | LOC105371116 | ncRNA | Интрон | chr16:19924067 | A/G | G | [26–29, 31] |
| 6 | rs12463617 | – | – | Межгенная | chr2:629244 | A/C/T | A | [35–37] |
| 7 | rs13130484 | – | – | Межгенная | chr4:45173674 | A/C/T | C | [26, 28, 31, 35] |
| 8 | rs1421085 | FTO | Альфа-кетоглутарат-зависимая диоксигеназа | Интрон | chr16:53767042 | C/T | T | [26, 28, 36, 38] |
| 9 | rs1558902 | FTO | Альфа-кетоглутарат-зависимая диоксигеназа | Интрон | chr16:53769662 | A/T | T | [25–30, 37, 39–41] |
| 10 | rs17381664 | ZZZ3 | Белок 3, содержащий цинковый палец типа ZZ | Интрон | chr1:77582646 | C/T | T | [26, 31] |
| 11 | rs17782313 | – | – | Межгенная | chr18:60183864 | C/T | T | [24, 31, 34, 38] |
| 12 | rs1800437 | GIPR | Рецептор, связанный с G-белком, для глюкозозависимого инсулинотропного пептида (ГИП) | Экзон | chr19:45678134 | C/G | G | [26, 31] |
| 13 | rs2030323 | BDNF | Нейротрофический фактор мозга | Интрон | chr11:27706992 | G/T | T | [26, 31, 42] |
| 14 | rs2033529 | – | – | Межгенная | chr6:40380914 | C/G/T | T | [27, 30, 31] |
| 15 | rs2112347 | LOC441087 | ncRNA | Интрон | chr5:75719417 | G/T | G | [25–27, 29–31] |
| 16 | rs2206277 | TFAP2B | Транскрипционный фактор AP-2-бета | Интрон | chr6:50830813 | A/G | G | [26, 30] |
| 17 | rs2207139 | – | – | Межгенная | chr6:50877777 | A/G | A | [26–29] |
| 18 | rs2531995 | ADCY9 | Аденилатциклаза 9-го типа | 3' UTR | chr16:3963466 | A/G | G | [26, 31, 37] |
| 19 | rs2568958 | LOC105378797 | ncRNA | Интрон | chr1:72299433 | A/C/G | G | [26, 43] |
| 20 | rs3101336 | LOC105378797 | ncRNA | Интрон | chr1:72285502 | A/G | A | [26–30, 36] |
| 21 | rs7141420 | NRXN3 | Нейрексин-3 | Интрон | chr14:79433111 | C/T | T | [26, 27, 29–31] |
| 22 | rs7195386 | RBBP6 | Белок 6, связывающий белок ретинобластомы | Интрон | chr16:24567137 | A/C/T | C | [30, 44] |
| 23 | rs7498665 | SH2B1 | Белок – член семейства медиаторов SH2-домена | Экзон | chr16:28871920 | A/G/T | G | [24, 26, 43] |
| 24 | rs7531118 | LOC105378797 | ncRNA | Интрон | chr1:72371556 | C/T | T | [26, 31] |
| 25 | rs7647305 | – | – | Межгенная | chr3:186116501 | C/T | T | [31, 32, 43] |
| 26 | rs887912 | – | – | Межгенная | chr2:59075742 | A/C/G | G | [25, 26] |
| 27 | rs9540493 | – | – | Межгенная | chr13:65631572 | A/G | A | [27, 29, 30] |
| 28 | rs9568867 | – | – | Межгенная | chr13:53533217 | G/A | G | [26, 30, 37] |
| 29 | rs9941349 | FTO | Альфа-кетоглутарат-зависимая диоксигеназа | Интрон | chr16:53791576 | C/T | C | [31, 45] |
| Мультиплекс 2 | ||||||||
| 30 | rs10182181 | – | – | Межгенная | chr2:24927427 | A/G | G | [26–30] |
| 31 | rs11671664 | GIPR | Рецептор, связанный с G-белком, для глюкозозависимого инсулинотропного пептида (ГИП) | Интрон | chr19:45669020 | A/G | G | [30, 37, 42, 46] |
| 32 | rs1167827 | HIP1 | Белок 1, взаимодействующий с протеином Хантингтона | 3' UTR | chr7:75533848 | A/G | G | [27, 29–31] |
| 33 | rs12429545 | – | – | Межгенная | chr13:53528071 | A/G | G | [27–31, 41] |
| 34 | rs12566985 | LRRC53, FPGT-TNNI3K, TNNI3K | Белок 53, содержащий богатый лейциновый повтор; транскрипция из соседних генов фукозо-1-фосфатгуанилилтрансферазы (FPGT) и TNNI3 взаимодействующей киназы (TNNI3K) | Интрон | chr1:74536509 | A/G | G | [27–30] |
| 35 | rs12940622 | RPTOR | Субъединица мишени рапамицина комплекса 1 у млекопитающих (mTORC1) | Интрон | chr17:80641771 | A/G | A | [27, 29, 30] |
| 36 | rs13021737 | – | – | Межгенная | chr2:632348 | A/G | A | [27–30] |
| 37 | rs13107325 | SLC39A8 | Член семейства генов-носителей SLC39, которые показывают структурные характеристики переносчиков цинка | Экзон | chr4:102267552 | A/C/T | C | [25, 27–29, 31] |
| 38 | rs13191362 | PRKN | Белок – компонент комплекса убиквитинлигазы E3 (паркин) | Интрон | chr6:162612318 | A/G | A | [27, 29, 31] |
| 39 | rs1514175 | LRRC53, FPGT-TNNI3K, TNNI3K | Белок 53, содержащий богатый лейциновый повтор; транскрипция из соседних генов фукозо-1-фосфатгуанилилтрансферазы (FPGT) и TNNI3 взаимодействующей киназы (TNNI3K) | Интрон | chr1:74525960 | C/T | T | [25, 35] |
| 40 | rs1516725 | ETV5 | ETS вариант 5 | Интрон | chr3:186106215 | C/T | C | [26, 27, 29, 30] |
| 41 | rs16851483 | RASA2 | Член GAP1-семейства ГТФаза-активирующих белков | Интрон | chr3:141556594 | G/T | G | [27, 29, 30] |
| 42 | rs17094222 | – | – | Межгенная | chr10:100635683 | C/T | T | [27, 29, 30] |
| 43 | rs2287019 | QPCTL | Глутаминил-пептид-циклотрансферазоподобный белок | Интрон | chr19:45698914 | C/T | C | [25, 27, 29] |
| 44 | rs3810291 | ZC3H4 | Белок 4, содержащий домен CCCH цинкового пальца | 3' UTR | chr19:47065746 | A/G | G | [25, 27, 29, 31, 37] |
| 45 | rs3817334 | MTCH2 | Белок – член семейства ядерно-кодированных транспортеров SLC25 | Интрон | chr11:47629441 | C/T | C | [25, 27, 29–31] |
| 46 | rs571312 | – | – | Межгенная | chr18:60172536 | G/T | G | [25, 31, 41] |
| 47 | rs633715 | – | – | Межгенная | chr1:177883445 | C/T | T | [26, 30, 31] |
| 48 | rs6567160 | – | – | Межгенная | chr18:60161902 | C/T | T | [27–30, 32, 47] |
| 49 | rs657452 | AGBL4 | АТФ/ГТФ-подобный связывающий белок 4 | Интрон | chr1:49124175 | A/G | A | [27, 29, 30] |
| 50 | rs6804842 | RARB | Бета-рецептор ретиноевой кислоты | Интрон | chr3:25064946 | A/G | A | [27, 29, 31] |
| 51 | rs6864049 | LOC105379158 | ncRNA | Интрон | chr5:124994829 | A/C/G/T | G | [27, 30, 31] |
| 52 | rs7138803 | – | – | Межгенная | chr12:49853685 | A/G/T | G | [25–31, 43] |
| 53 | rs7164727 | – | – | Межгенная | chr15:72801650 | C/G/T | C | [27, 29, 31] |
| 54 | rs7903146 | TCF7L2 | Транскрипционный фактор, содержащий бокс группы с высокой подвижностью (HMG) | Интрон | chr10:112998590 | C/G/T | T | [27, 29–31] |
| 55 | rs8050136 | FTO | Альфа-кетоглутарат-зависимая диоксигеназа | Интрон | chr16:53782363 | A/C | A | [31, 43, 48, 49] |
| 56 | rs9641123 | CALCR | Рецептор, связанный с G-белком, для кальцитонина | Интрон | chr7:93568420 | C/G | G | [27, 29, 31] |
| 57 | rs9816226 | – | – | Межгенная | chr3:186116710 | A/T | T | [25, 26] |
| 58 | rs987237 | TFAP2B | Транскрипционный фактор AP-2-бета | Интрон | chr6:50835337 | A/G | A | [25, 26, 28, 31] |
Материалом для генотипирования послужила выборка 95 индивидов, не состоящих в родстве и представляющих популяцию русских г. Кемерово. Средний возраст испытуемых составил 46.0 ± ± 10.8 года. Показатель ИМТ в данной популяционной выборке варьировал от 15.9 до 39.7 (среднее значение 26.9 ± 5.0).
Выделение ДНК проводили из венозной крови методом фенол-хлороформной экстракции. ПЦР, дефосфорилирование щелочной фосфатазой неинкорпорированных дезоксинуклеотидтрифосфатов, реакцию удлинения пролонгирующих праймеров путем включения дидезокситрифосфата (ddNTPs) с модифицированной массой (IPLEX реакция), очистку ПЦР-продуктов, перенос продуктов на спектрочип и ионизацию проводили согласно протоколу компании “Agena Bioscience” (CША). Праймеры были синтезированы компанией “Евроген” (Россия). Структура праймеров доступна по запросу у авторов. Более подробно процедура проведения генотипирования методом MALDI-TOF-масс-спектрометрии описана ранее [10]. Визуализацию результатов проводили посредством пакета программ Mass Array Typer Analyzer soft ware v. 4.0 (Agena Bioscience).
Статистическую обработку результатов исследования проводили с помощью пакетов программ Statistica V. 7.0 и ARLEQUIN. Эмпирически наблюдаемое распределение частот генотипов проверяли на соответствие теоретически ожидаемому при равновесии Харди–Вайнберга (РХВ) с помощью точного теста Фишера [12]. Для определения характера распределения полученных данных использовали критерий Шапиро–Уилка. Для анализа количественных признаков при сравнении независимых выборок с нормальным распределением использовали дисперсионный анализ [13]. Статистически значимыми считали различия для p < 0.05.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Оценка эффективности генотипирования
В рамках настоящей работы нами проведено два эксперимента по анализу эффективности метода – генотипирование двух разработанных мультиплексов (по 29 SNP в каждом) в планшете, включавшем 95 образцов из популяционной выборки русских и один контрольный образец без ДНК. Общая эффективность генотипирования оказалась достаточно высокой. Доля определенных генотипов из всех возможных (“callrate”) составила 86.5% для мультиплекса 1 и 92.7% для мультиплекса 2 (без учета контрольного образца). Низкая эффективность генотипирования была зафиксирована для rs10968576, rs11671664, rs2287019 и rs657452 (средняя доля определенных генотипов для всей выборки составила 16.67, 60.21, 65.62 и 69.79% соответственно). Генотипы трех из 58 анализируемых аллельных вариантов – rs13130484, rs17381664 и rs12566985 не удалось определить ни в одном образце, в связи с чем семь данных маркеров мы не рассматривали в последующем анализе. Без них эффективность генотипирования мультиплексов 1 и 2 составила 95.8 и 97.0% соответственно.
Популяционно-генетический анализ частот аллелей и генотипов
Частоты аллелей 51 SNP в популяции русских и европеоидов из международного проекта “1000 геномов”, генетическое разнообразие и значения отклонения от равновесия Харди–Вайнберга представлены в табл. 2. Распределение частот генотипов можно получить по запросу у авторов. В проанализированном массиве данных отклонение от равновесия Харди–Вайнберга среди 510 распределений частот генотипов было обнаружено в 26 случаях, что не превышает ожидаемого числа случайных отклонений. Не наблюдалось накопления отклонений от РХВ по отдельным маркерам или популяциям. Общий уровень генетического разнообразия популяций по исследованным маркерам оказался достаточно близок: средняя ожидаемая гетерозиготность варьирует от 0.361 у бенгальцев до 0.390 у британцев. Cредний уровень ожидаемой гетерозиготности в популяции русских составил 0.382. Наименьший уровень генетического разнообразия (показатель ожидаемой гетерозиготности составил менее 0.15) среди изученных популяций показан для локусов rs13107325, rs16851483 и rs11847697. Полокусные значения коэффициента генетической дифференциации для большинства маркеров (33 из 51) не превышают 2% (табл. 2). Лишь по пяти SNP (rs13107325, rs3810291, rs2531995, rs7195386, rs7531118) изученные популяции дифференцированы более чем на 5%. Средний же уровень дифференциации по 51 маркеру составляет 1.9%, что соответствует уровню генетических различий между исследованными популяциями по различным аутосомным маркерным системам, включая широкогеномные наборы SNP [14].
Таблица 2.
Частоты аллелей исследованных маркеров и генетическое разнообразие в популяции русских и в европеоидных популяциях проекта “1000 геномов”
| № п/п | Локус | Предковый аллель | Русские | Тосканцы | Иберийцы | Британцы | Финны | Пенджабцы | Индийцы из Гуджарата | Индийцы телугу | Ланкийские тамилы | Бенгальцы | Ожидаемая гетерозиготность | Fst | p-уровень |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | rs10182181 | G | 0.467 | 0.421 | 0.509 | 0.505 | 0.399 | 0.495 | 0.413 | 0.520 | 0.520 | 0.424 | 0.494 | 0.005 | 0.038 |
| 2 | rs10938397 | A | 0.542 | 0.603 | 0.626 | 0.549 | 0.535 | 0.656 | 0.612 | 0.603 | 0.652 | 0.640 | 0.476 | 0.003 | 0.101 |
| 3 | rs1121980 | A | 0.489 | 0.500 | 0.411 | 0.412 | 0.414 | 0.370 | 0.340 | 0.397 | 0.436 | 0.390 | 0.481 | 0.005 | 0.026 |
| 4 | rs1167827 | G | 0.423 | 0.584 | 0.556 | 0.549 | 0.414 | 0.469 | 0.568 | 0.471 | 0.510 | 0.424 | 0.492 | 0.012 | 0.000 |
| 5 | rs11847697 | C | 0.984 | 0.916 | 0.925 | 0.967 | 0.965 | 0.875 | 0.883 | 0.882 | 0.877 | 0.919 | 0.145 | 0.017 | 0.000 |
| 6 | rs12429545 | G | 0.909 | 0.883 | 0.930 | 0.885 | 0.864 | 0.802 | 0.864 | 0.824 | 0.828 | 0.808 | 0.238 | 0.010 | 0.002 |
| 7 | rs12446632 | G | 0.830 | 0.879 | 0.869 | 0.846 | 0.909 | 0.969 | 0.956 | 0.951 | 0.956 | 0.942 | 0.158 | 0.027 | 0.000 |
| 8 | rs12463617 | A | 0.186 | 0.206 | 0.154 | 0.165 | 0.177 | 0.125 | 0.170 | 0.127 | 0.172 | 0.145 | 0.271 | 0.000 | 0.480 |
| 9 | rs12940622 | A | 0.438 | 0.411 | 0.463 | 0.456 | 0.384 | 0.318 | 0.267 | 0.319 | 0.294 | 0.279 | 0.452 | 0.020 | 0.000 |
| 10 | rs13021737 | A | 0.110 | 0.206 | 0.150 | 0.165 | 0.182 | 0.109 | 0.141 | 0.118 | 0.152 | 0.134 | 0.248 | 0.003 | 0.122 |
| 11 | rs13107325 | C | 0.946 | 0.893 | 0.916 | 0.901 | 0.990 | 1.000 | 1.000 | 1.000 | 1.000 | 1.000 | 0.129 | 0.056 | 0.000 |
| 12 | rs13191362 | A | 0.914 | 0.855 | 0.822 | 0.846 | 0.960 | 0.943 | 0.932 | 0.922 | 0.907 | 0.936 | 0.170 | 0.020 | 0.000 |
| 13 | rs1421085 | T | 0.528 | 0.509 | 0.607 | 0.593 | 0.586 | 0.708 | 0.733 | 0.681 | 0.647 | 0.698 | 0.456 | 0.020 | 0.000 |
| 14 | rs1514175 | A | 0.424 | 0.425 | 0.383 | 0.396 | 0.470 | 0.531 | 0.408 | 0.544 | 0.569 | 0.541 | 0.489 | 0.015 | 0.000 |
| 15 | rs1516725 | C | 0.908 | 0.860 | 0.827 | 0.857 | 0.894 | 0.870 | 0.854 | 0.868 | 0.917 | 0.866 | 0.222 | 0.002 | 0.233 |
| 16 | rs1558902 | T | 0.522 | 0.509 | 0.607 | 0.593 | 0.586 | 0.708 | 0.733 | 0.681 | 0.647 | 0.698 | 0.456 | 0.021 | 0.000 |
| 17 | rs16851483 | G | 0.918 | 0.949 | 0.939 | 0.934 | 0.949 | 0.911 | 0.898 | 0.922 | 0.936 | 0.913 | 0.135 | 0.001 | 0.543 |
| 18 | rs17094222 | T | 0.815 | 0.846 | 0.771 | 0.775 | 0.798 | 0.849 | 0.898 | 0.902 | 0.843 | 0.884 | 0.267 | 0.012 | 0.000 |
| 19 | rs17782313 | T | 0.750 | 0.720 | 0.743 | 0.764 | 0.838 | 0.635 | 0.718 | 0.632 | 0.681 | 0.738 | 0.395 | 0.014 | 0.000 |
| 20 | rs1800437 | G | 0.790 | 0.776 | 0.808 | 0.780 | 0.742 | 0.896 | 0.816 | 0.907 | 0.848 | 0.860 | 0.287 | 0.015 | 0.000 |
| 21 | rs2030323 | A | 0.170 | 0.276 | 0.266 | 0.192 | 0.182 | 0.292 | 0.252 | 0.294 | 0.314 | 0.331 | 0.376 | 0.011 | 0.001 |
| 22 | rs2033529 | A | 0.700 | 0.790 | 0.710 | 0.747 | 0.652 | 0.880 | 0.859 | 0.853 | 0.873 | 0.855 | 0.317 | 0.037 | 0.000 |
| 23 | rs2112347 | G | 0.372 | 0.416 | 0.322 | 0.396 | 0.374 | 0.568 | 0.607 | 0.559 | 0.549 | 0.599 | 0.477 | 0.043 | 0.000 |
| 24 | rs2206277 | C | 0.678 | 0.818 | 0.850 | 0.797 | 0.788 | 0.833 | 0.714 | 0.696 | 0.760 | 0.814 | 0.342 | 0.016 | 0.000 |
| 25 | rs2207139 | A | 0.704 | 0.869 | 0.869 | 0.808 | 0.793 | 0.844 | 0.699 | 0.696 | 0.750 | 0.814 | 0.330 | 0.023 | 0.000 |
| 26 | rs2531995 | C | 0.410 | 0.332 | 0.439 | 0.407 | 0.419 | 0.641 | 0.621 | 0.647 | 0.676 | 0.657 | 0.467 | 0.067 | 0.000 |
| 27 | rs2568958 | G | 0.351 | 0.350 | 0.341 | 0.407 | 0.333 | 0.365 | 0.403 | 0.373 | 0.461 | 0.285 | 0.460 | 0.005 | 0.044 |
| 28 | rs3101336 | T | 0.348 | 0.355 | 0.341 | 0.407 | 0.333 | 0.359 | 0.403 | 0.377 | 0.461 | 0.285 | 0.460 | 0.005 | 0.036 |
| 29 | rs3810291 | G | 0.454 | 0.318 | 0.322 | 0.357 | 0.374 | 0.578 | 0.573 | 0.598 | 0.662 | 0.564 | 0.469 | 0.064 | 0.000 |
| 30 | rs3817334 | C | 0.625 | 0.589 | 0.561 | 0.544 | 0.601 | 0.672 | 0.684 | 0.632 | 0.618 | 0.663 | 0.468 | 0.004 | 0.059 |
| 31 | rs571312 | C | 0.758 | 0.720 | 0.743 | 0.764 | 0.838 | 0.641 | 0.718 | 0.637 | 0.681 | 0.738 | 0.393 | 0.013 | 0.000 |
| 32 | rs633715 | T | 0.857 | 0.804 | 0.846 | 0.775 | 0.823 | 0.844 | 0.801 | 0.882 | 0.828 | 0.907 | 0.270 | 0.006 | 0.022 |
| 33 | rs6567160 | T | 0.761 | 0.720 | 0.743 | 0.764 | 0.838 | 0.635 | 0.718 | 0.637 | 0.686 | 0.738 | 0.393 | 0.014 | 0.001 |
| 34 | rs6804842 | A | 0.375 | 0.407 | 0.453 | 0.407 | 0.520 | 0.615 | 0.621 | 0.588 | 0.618 | 0.622 | 0.480 | 0.037 | 0.000 |
| 35 | rs6864049 | G | 0.565 | 0.561 | 0.533 | 0.484 | 0.556 | 0.656 | 0.675 | 0.593 | 0.672 | 0.605 | 0.477 | 0.011 | 0.001 |
| 36 | rs7138803 | G | 0.581 | 0.626 | 0.659 | 0.681 | 0.672 | 0.667 | 0.597 | 0.632 | 0.701 | 0.680 | 0.452 | 0.002 | 0.234 |
| 37 | rs7141420 | T | 0.632 | 0.505 | 0.537 | 0.511 | 0.566 | 0.526 | 0.578 | 0.618 | 0.603 | 0.610 | 0.487 | 0.004 | 0.075 |
| 38 | rs7164727 | C | 0.355 | 0.336 | 0.252 | 0.346 | 0.268 | 0.432 | 0.413 | 0.373 | 0.456 | 0.442 | 0.456 | 0.017 | 0.000 |
| 39 | rs7195386 | C | 0.511 | 0.570 | 0.477 | 0.599 | 0.495 | 0.766 | 0.723 | 0.794 | 0.814 | 0.709 | 0.427 | 0.070 | 0.000 |
| 40 | rs7498665 | G | 0.410 | 0.285 | 0.294 | 0.346 | 0.364 | 0.193 | 0.296 | 0.255 | 0.157 | 0.180 | 0.389 | 0.028 | 0.000 |
| 41 | rs7531118 | T | 0.410 | 0.467 | 0.463 | 0.484 | 0.394 | 0.677 | 0.665 | 0.740 | 0.760 | 0.727 | 0.449 | 0.083 | 0.000 |
| 42 | rs7647305 | T | 0.139 | 0.168 | 0.252 | 0.231 | 0.192 | 0.224 | 0.301 | 0.191 | 0.186 | 0.238 | 0.331 | 0.008 | 0.012 |
| 43 | rs7903146 | T | 0.220 | 0.374 | 0.397 | 0.258 | 0.227 | 0.250 | 0.282 | 0.338 | 0.338 | 0.279 | 0.410 | 0.014 | 0.000 |
| 44 | rs8050136 | A | 0.462 | 0.463 | 0.374 | 0.396 | 0.389 | 0.276 | 0.243 | 0.304 | 0.338 | 0.285 | 0.446 | 0.021 | 0.000 |
| 45 | rs887912 | C | 0.753 | 0.692 | 0.706 | 0.714 | 0.768 | 0.818 | 0.796 | 0.848 | 0.907 | 0.913 | 0.319 | 0.033 | 0.000 |
| 46 | rs9540493 | A | 0.368 | 0.379 | 0.463 | 0.495 | 0.515 | 0.589 | 0.558 | 0.554 | 0.549 | 0.599 | 0.488 | 0.021 | 0.000 |
| 47 | rs9568867 | G | 0.908 | 0.883 | 0.930 | 0.885 | 0.864 | 0.802 | 0.864 | 0.819 | 0.828 | 0.808 | 0.239 | 0.011 | 0.001 |
| 48 | rs9641123 | G | 0.552 | 0.607 | 0.565 | 0.582 | 0.616 | 0.641 | 0.578 | 0.686 | 0.578 | 0.640 | 0.475 | 0.002 | 0.186 |
| 49 | rs9816226 | T | 0.844 | 0.841 | 0.766 | 0.797 | 0.833 | 0.833 | 0.728 | 0.838 | 0.863 | 0.820 | 0.297 | 0.007 | 0.014 |
| 50 | rs987237 | A | 0.694 | 0.818 | 0.855 | 0.797 | 0.788 | 0.833 | 0.714 | 0.701 | 0.760 | 0.831 | 0.338 | 0.015 | 0.000 |
| 51 | rs9941349 | C | 0.500 | 0.528 | 0.607 | 0.604 | 0.606 | 0.651 | 0.684 | 0.618 | 0.578 | 0.634 | 0.474 | 0.007 | 0.007 |
| Ожидаемая гетерозиготность | 0.382 | 0.387 | 0.383 | 0.390 | 0.370 | 0.368 | 0.381 | 0.377 | 0.373 | 0.361 | |||||
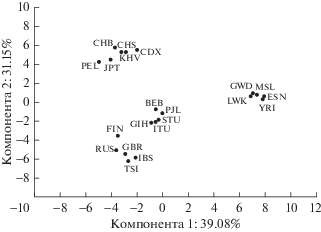
Взаимоотношения мировых популяций по частотам аллелей исследованных маркеров генов, ассоциированных с ожирением, полученные методом главных компонент по данным настоящей работы и проекта “1000 геномов”, представлены на рис. 2. Две первые главные компоненты (ГК) объясняют 70.23% вариабельности аллельных частот в анализируемых популяциях. Положение исследованных популяций в пространстве ГК1–ГК2 соответствует их географической принадлежности, что ожидалось, исходя из данных, полученных в предыдущих исследованиях аутосомных маркеров [15, 16]. Первая ГК, на долю которой приходится 39.08%, выделила кластер африканских популяций. В него вошли популяции йоруба, лухья, ишан, менде, жители Гамбии. Вторая главная компонента (31.15%), вероятно, разделяет выборки по восточно-евразийской генетической составляющей. Выборка русских локализована в кластере европейских популяций, при этом наибольшая генетическая близость из представленных популяций показана для русских, англичан, иберийцев и итальянцев.
Рис. 2.
Положение популяций в пространстве двух первых главных компонент частот аллелей 51 маркера. RUS – русские, GBR – англичане, FIN – финны, TSI – итальянцы, IBS – иберийцы, PEL – перуанцы, GIH – индийцы из Гуджарата, ITU – индийцы телугу, BEB – бенгальцы, CDX – даи, Китай, CHB, CHS – китайцы, JPT – японцы, KHV – вьетнамцы, STU – тамилы, PJL – пенджабцы, MSL – менде, GWD – жители Гамбии, ESN – ишаны, LWK – лухья, YRI – йоруба.
Оценка ассоциации изученных генетических маркеров с показателем ИМТ
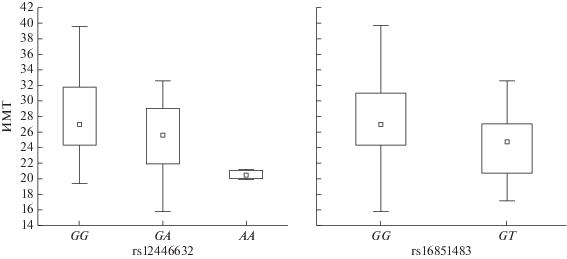
В представленной работе был проведен также анализ ассоциации SNPs с вариабельностью показателя ИМТ. Обнаружена ассоциация двух полиморфных маркеров из 51 изученного с показателем ИМТ: так, статистически значимо более высокие значения показателя ИМТ наблюдались у носителей генотипов GG и GA маркера rs12446632 локуса LOC105371116 (F = 4.90; p = 0.01), в котором расположен ген некодирующей РНК, а также у носителей генотипа GG маркера rs16851483 белок-кодирующего гена RASA2 (F = 5.56; p = 0.02) по сравнению с индивидами, обладающими генотипами АА и GT (рис. 3) соответственно. Функциональная значимость полиморфизма rs16851483 может быть обусловлена ключевой регуляторной функцией RAS-p21 ГТФаза-активирующего белка 2 в передаче сигнала по Ras/MAPK-пути и процессах дифференцировки, пролиферации и миграции клеток [17, 18]. Показано, что мутации гена RASA2 связаны с развитием меланомы у человека, а также синдромом Нунана [18, 19]. Нами не обнаружено данных, позволяющих охарактеризовать возможную значимость продукта гена LOC105371116 в формировании изученных фенотипов. Однако функциональная аннотация ассоциированного маркера локуса rs12446632 в онлайн-ресурсе HaploReg v4.1 выявила тесное сцепление данного варианта с рядом SNPs генов IQCK и GPRC5B [20]. Известно, что ген GPRC5B кодирует рецептор, сопряженный с G-белками, относящимися к ГТФазам и функционирующими в качестве вторичных посредников во внутриклеточных сигнальных каскадах, в том числе при передаче сигнала по MAPK-пути, в TGF-β и IFNγ-каскадах [17, 21]. Показано, что фосфорилирование этого рецептора тирозинкиназой Fyn и последующее его взаимодействие с Fyn через домен SH2 играют ключевую роль в инициации и прогрессии воспалительных процессов в жировой ткани, а также развитии инсулинорезистентности [22, 23].
Рис. 3.
Результаты анализа ассоциации изученных аллельных вариантов с показателем ИМТ у русских. Показаны минимальное и максимальное значение, медиана и квартильный размах.
Таким образом, в настоящем исследовании разработан высокопроизводительный метод мультиплексного генотипирования полиморфных маркеров генов, ассоциированных с ожирением и вариабельностью показателя индекса массы тела, по данным полногеномного анализа ассоциаций. Показана ассоциация маркеров rs12446632 локуса LOC105371116 и rs16851483 гена RASA2 с вариабельностью ИМТ у русских г. Кемерово, свидетельствующая о вовлеченности данных маркеров в формирование изменчивости изученного фенотипа и применимости разработанной панели в области исследований генетики ожирения и молекулярных механизмов регуляции энергетического баланса. Проведена оценка уровня генетического разнообразия изученной выборки и популяций из международного проекта “1000 геномов” по исследованным полиморфным вариантам. Предложенная в настоящем исследовании панель генетических маркеров отражает структуру генофонда населения мира и может быть использована для дальнейших популяционно-генетических, репликативных и ассоциативных исследований.
Исследование выполнено за счет гранта Российского фонда фундаментальных исследований (проект № 18-04-00758).
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Дедов И.И., Мельниченко Г.А., Романцова Т.И. Патогенетические аспекты ожирения // Ожирение и метаболизм. 2004. № 1. С. 3–9.
Дедов И.И., Мельниченко Г.А., Шестакова М.В. и др. Национальные клинические рекомендации по лечению морбидного ожирения у взрослых. 3-й пересмотр (лечение морбидного ожирения у взрослых) // Ожирение и метаболизм. 2018. Т. 15. № 1. С. 53–70. https://doi.org/10.14341/OMET2018153-70
Romieu I., Dossus L., Barquera S. et al. Energy balance and obesity: what are the main drivers? // Cancer Causes Control. 2017. V. 28. № 3. P. 247–258. https://doi.org/10.1007/s10552-017-0869-z
Huvenne H., Dubern B., Clément K., Poitou C. Rare genetic forms of jbesity: Clinical approach and current treatments in 2016 // Obes. Facts. 2016. V. 9. № 3. P. 158–173. https://doi.org/10.1159/000445061
GWAS Catalog [Electronic resource]. URL: https://www.ebi.ac.uk/gwas/. Accessed 12.2018.
Rohde K., Keller M., la Cour Poulsen L. et al. Genetics and epigenetics in obesity // Metabolism. 2019. V. 92. P. 37–50. https://doi.org/10.1016/j.metabol.2018.10.007
Srivastava A., Srivastava N., Mittal B. Genetics of Obesity // Indian J. Clin. Biochem. 2016. V. 31. № 4. P. 361–371. https://doi.org/10.1007/s12291-015-0541-x
Day F.R., Loos R.J. Developments in obesity genetics in the era of genome-wide association studies // J. Nutrigenet. Nutrigenomics. 2011. V. 4. № 4. P. 222–238. https://doi.org/10.1159/000332158
Gabriel S., Ziaugra L., Tabbaa D. SNP genotyping using the sequenom Mass ARRAY iPLEX platform // Curr. Protoc. Hum. Genet. 2009. V. 9. № 60. P. 2.12.1–2.12.18. https://doi.org/10.1002/0471142905.hg0212s60
Степанов В.А., Трифонова Е.А. Мультиплексное генотипирование однонуклеотидных полиморфных маркеров методом MALDI-TOF-масс-спектрометрии: частоты 56 SNP в генах иммунного ответа в популяциях человека // Мол. биология. 2013. Т. 47. № 6. С. 976–986. https://doi.org/10.7868/S0026898413060153
AgenaCx customer portal [Electronic resource]. URL: https://www.agenacx.com. Accessed 1.2019.
Вейр Б. Анализ генетических данных. М.: Мир, 1995. 400 с.
Гланц С. Медико-биологическая статистика. М.: Практика, 1999. 459 с.
Triska P., Chekanov N., Stepanov V. et al. Between Lake Baikal and the Baltic Sea: genomic history of the gateway to Europe // BMC Genet. 2017. V. 18. Suppl. 1. https://doi.org/10.1186/s12863-017-0578-3
Степанов В.А. Эволюция генетического разнообразия и болезни человека // Генетика. 2016. Т. 52. № 7. С. 852–864. https://doi.org/10.7868/S0016675816070109
Степанов В.А. Этногеномика населения Сибири и Средней Азии. Томск: Печатная мануфактура, 2002. 242 с.
The Human Gene Database GeneCards [Electronic resource]. URL: https://www.genecards.org/. Accessed. 12.2018.
Arafeh R., Qutob N., Emmanuel R. et al. Recurrent inactivating RASA2 mutations in melanoma // Nat. Genet. 2015. V. 47. № 12. P. 1408–1410. https://doi.org/10.1038/ng.3427
Tidyman W.E., Rauen K.A. Expansion of the RASopathies // Curr. Genet. Med. Rep. 2016. V. 4. № 3. P. 57–64.
HaploReg v4.1 [Electronic resource]. URL: https:// pubs.broadinstitute.org/mammals/haploreg/haploreg. php. Accessed. 12.2018.
Atanes P., Ruz-Maldonado I., Hawkes R. et al. Identifying signalling pathways regulated by GPRC5B in β‑Cells by CRISPR-Cas9-Mediated genome editing // Cell Physiol. Biochem. 2018. V. 45. № 2. P. 656–666. https://doi.org/10.1159/000487159
Kim Y.J., Greimel P., Hirabayashi Y. GPRC5B-Mediated sphingomyelin synthase 2 phosphorylation plays a critical role in insulin resistance // Science. 2018. V. 8. P. 250–266. https://doi.org/10.1016/j.isci.2018.10.001
Kim Y.J., Sano T., Nabetani T. et al. GPRC5B activates obesity-associated inflammatory signaling in adipocytes // Sci. Signal. 2012. V. 5. № 251. https://doi.org/10.1126/scisignal.2003149
Willer C.J., Speliotes E.K., Loos R.J. et al. Six new loci associated with body mass index highlight a neuronal influence on body weight regulation // Nat. Genet. 2009. V. 41. № 1. P. 25–34. https://doi.org/10.1038/ng.287
Speliotes E.K., Willer C.J., Berndt S.I. et al. Association analyses of 249 796 individuals reveal 18 new loci associated with body mass index // Nat. Genet. 2010. V. 42. № 11. P. 937–948. https://doi.org/10.1038/ng.686
Berndt S.I., Gustafsson S., Mägi R. et al. Genome-wide meta-analysis identifies 11 new loci for anthropometric traits and provides insights into genetic architecture // Nat. Genet. 2013. V. 45. № 5. P. 501–512. https://doi.org/10.1038/ng.2606
Locke A.E., Kahali B., Berndt S.I. et al. Genetic studies of body mass index yield new insights for obesity biology // Nature. 2015. V. 518. № 7538. P. 197–206. https://doi.org/10.1038/nature14177
Felix J.F., Bradfield J.P., Monnereau C. et al. Genome-wide association analysis identifies three new susceptibility loci for childhood body mass index // Hum. Mol. Genet. 2016. V. 25. № 2. P. 389–403. https://doi.org/10.1093/hmg/ddv472
Justice A.E., Winkler T.W., Feitosa M.F. et al. Genome-wide meta-analysis of 241 258 adults accounting for smoking behaviour identifies novel loci for obesity traits // Nat. Commun. 2017. V. 8. https://doi.org/10.1038/ncomms14977
Akiyama M., Okada Y., Kanai M. et al. Genome-wide association study identifies 112 new loci for body mass index in the Japanese population // Nat. Genet. 2017. V. 49. № 10. P. 1458–1467. https://doi.org/10.1038/ng.3951
Graff M., Scott R.A., Justice A.E. et al. Genome-wide physical activity interactions in adiposity – A meta-analysis of 200 452 adults // PLoS Genet. 2017. V. 13. № 4. e1006528. https://doi.org/10.1371/journal.pgen.1006528
Ng M.C.Y., Graff M., Lu Y. et al. Discovery and fine-mapping of adiposity loci using high density imputation of genome-wide association studies in individuals of African ancestry: African Ancestry Anthropometry Genetics Consortium // PLoS Genet. 2017. V. 13. № 4. e1006719. https://doi.org/10.1371/journal.pgen.1006719
Hinney A., Nguyen T.T., Scherag A. et al. Genome wide association (GWA) study for early onset extreme obesity supports the role of fat mass and obesity associated gene (FTO) variants // PLoS One. 2007. V. 2. № 12. e1361. https://doi.org/10.1371/journal.pone.0001361
Loos R.J., Lindgren C.M., Li S. et al. Common variants near MC4R are associated with fat mass, weight and risk of obesity // Nat. Genet. 2008. V. 40. № 6. P. 768–775. https://doi.org/10.1038/ng.140
Graff M., Ngwa J.S., Workalemahu T. et al. Genome-wide analysis of BMI in adolescents and young adults reveals additional insight into the effects of genetic loci over the life course // Hum. Mol. Genet. 2013. V. 22. № 17. P. 3597–3607. https://doi.org/10.1093/hmg/ddt205
Wheeler E., Huang N., Bochukova E.G. et al. Genome-wide SNP and CNV analysis identifies common and low-frequency variants associated with severe early-onset obesity // Nat. Genet. 2013. V. 45. № 5. P. 513–517. https://doi.org/10.1038/ng.2607
Wen W., Zheng W., Okada Y. et al. Meta-analysis of genome-wide association studies in East Asian-ancestry populations identifies four new loci for body mass index // Hum. Mol. Genet. 2014. V. 23. № 20. P. 5492–5504. https://doi.org/10.1093/hmg/ddu248
Meyre D., Delplanque J., Chèvre J.C. et al. Genome-wide association study for early-onset and morbid adult obesity identifies three new risk loci in European populations // Nat. Genet. 2009. V. 41. № 2. P. 157–159. https://doi.org/10.1038/ng.301
Scherag A., Dina C., Hinney A. et al. Two new loci for body-weight regulation identified in a joint analysis of genome-wide association studies for early-onset extreme obesity in French and german study groups // PLoS Genet. 2010. V. 6. № 4. e1000916. https://doi.org/10.1371/journal.pgen.1000916
Dorajoo R., Blakemore A.I., Sim X. et al. Replication of 13 obesity loci among Singaporean Chinese, Malay and Asian-Indian populations // Int. J. Obes. (Lond). 2012. V. 36. № 1. P. 159–163. https://doi.org/10.1038/ijo.2011.86
Warrington N.M., Howe L.D., Paternoster L. et al. A genome-wide association study of body mass index across early life and childhood // Int. J. Epidemiol. 2015. V. 44. № 2. P. 700–712. https://doi.org/10.1093/ije/dyv077
Okada Y., Kubo M., Ohmiya H. et al. Common variants at CDKAL1 and KLF9 are associated with body mass index in east Asian populations // Nat. Genet. 2012. V. 44. № 3. P. 302–306. https://doi.org/10.1038/ng.1086
Thorleifsson G., Walters G.B., Gudbjartsson D.F. et al. Genome-wide association yields new sequence variants at seven loci that associate with measures of obesity // Nat. Genet. 2009 V. 41. № 1. P. 18–24. https://doi.org/10.1038/ng.274
Comuzzie A.G., Cole S.A., Laston S.L. et al. Novel genetic loci identified for the pathophysiology of childhood obesity in the Hispanic population // PLoS One. 2012. V. 7. № 12. e51954. https://doi.org/10.1371/journal.pone.0051954
Cotsapas C., Speliotes E.K., Hatoum I.J. et al. Common body mass index-associated variants confer risk of extreme obesity // Hum. Mol. Genet. 2009. V. 18. № 18. P. 3502–3507. https://doi.org/10.1093/hmg/ddp292
Wen W., Cho Y.S., Zheng W. et al. Meta-analysis identifies common variants associated with body mass index in east Asians // Nat. Genet. 2012. V. 44. № 3. P. 307–311. https://doi.org/10.1038/ng.1087
Pei Y.F., Zhang L., Liu Y. et al. Meta-analysis of genome-wide association data identifies novel susceptibility loci for obesity // Hum. Mol. Genet. 2014. V. 23. № 3. P. 820–830. https://doi.org/10.1093/hmg/ddt464
Wan E.S., Cho M.H., Boutaoui N. et al. Genome-wide association analysis of body mass in chronic obstructive pulmonary disease // Am. J. Respir. Cell Mol. Biol. 2011. V. 45. № 2. P. 304–310. https://doi.org/10.1165/rcmb.2010-0294OC
Namjou B., Keddache M., Marsolo K. et al. EMR-linked GWAS study: investigation of variation landscape of loci for body mass index in children // Front Genet. 2013. V. 4. https://doi.org/10.3389/fgene.2013.00268
Дополнительные материалы отсутствуют.


