Генетика, 2019, T. 55, № 11, стр. 1263-1278
Генетическая гетерогенность диплоидного злака Aegilops tauschii, выявленная методами хромосомного анализа и электрофореза запасных белков зерна (глиадинов)
Е. Д. Бадаева 1, 2, *, А. В. Фисенко 3, С. А. Суржиков 2, А. А. Янковская 1, Н. Н. Чикида 4, С. А. Зощук 2, М. Х. Белоусова 4, А. Ю. Драгович 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
3 Главный ботанический сад им. Н.В. Цицина Российской академии наук
127276 Москва, Россия
4 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190121 Санкт-Петербург, Россия
* E-mail: katerinabadaeva@gmail.com
Поступила в редакцию 25.03.2019
После доработки 25.05.2019
Принята к публикации 30.05.2019
Аннотация
Генетическое разнообразие диплоидного злака Ae. tauschii Coss (2n = 2x = 14, DD), предка D-генома мягкой пшеницы, исследовано методами флуоресцентной гибридизации in situ (FISH) c 11 ДНК-зондами, представляющими сателлитные и микросателлитные последовательности и семейства 45S и 5S рРНК генов, и электрофоретического (ЭФ) анализа запасных белков зерна – глиадинов. Выявлена четкая генетическая дифференциация образцов на группы strangulata (Str) и tauschii (Tau), отличающиеся друга от друга наличием микросателлитных повторов GAAn и ACTn и распределением семейств сателлитных ДНК, особенно pAs1 последовательности. Установлено, что представители группы Str наиболее близки D-геному мягкой пшеницы по рисунку распределения изученных проб. Сравнение ЭФ-спектров глиадинов показало наибольшее сходство образцов Ae. tauschii из Армении и Азербайджана с мягкой пшеницей, что указывает на участие закавказской группы Str в формировании современного генофонда мягких пшениц.
Aegilops tauschii Coss (syn. Ae. squarrosa L.) – это диплоидный злак (2n = 14, DD), один из родителей мягкой пшеницы [1–6]. Он также участвовал в образовании ряда полиплоидных видов Aegilops, являясь при этом донором их цитоплазматического генома [7–9]. Ae. tauschii занимает обширный ареал в Восточной Азии, расположенный восточнее 40° в.д. Считается, что центр образования и генетического разнообразия Ae. tauschii находится в Иране – на южном побережье Каспийского моря и прилегающих районах Азербайджана [10, 11]. Оттуда он распространился на восток в виде “широкой полосы”, простирающейся от гор Копетдага (Туркменистан) до Алматы (Казахстан) на северо-востоке и ограниченной с юга горным массивом Тянь-Шаня и с севера степями Казахстана, Туркменистана и Узбекистана [11]. Далее вид продвинулся на юго-восток в горные районы Афганистана и Пакистана, дойдя до предгорья Гималаев в Индии (штат Кашмир) и Китая, который считается восточной границей ареала Ae. tauschii. В Китае вид обнаружен сравнительно недавно – в середине 50-х гг. ХХ в. Две популяции Ae. tauschii, значительно отличающиеся по морфолого-ботаническим признакам, найдены в долине р. Или Синьцзян-Уйгурского автономного района и в провинциях Шэньси и Хэнань – основных районах возделывания мягкой пшеницы, расположенных в средней части бассейна р. Хуанхэ [12]. Продвижение Ae. tauschii из центра образования в западном направлении шло в основном в Закавказье и Восточную Турцию, в меньшей степени – северную и восточную части Дуги плодородия (Сирия и Ирак). Окрестности г. Ракка на севере Сирии считаются западной границей ареала Ae. tauschii [13].
Вид адаптирован к разнообразным почвенно-климатическим условиям. В основном он обитает в глинистых полупустынях, на равнинах, в предгорьях и низкогорьях в районах с сухим климатом, однако встречается и во влажных широколиственных Гирканских лесах прикаспийского Ирана, и в субтропиках Абхазии [10, 11, 14–16]. В горных районах Ae. tauschii может подниматься до высоты 2700 м над у.м. [10, 11]. Нередко он занимает вторичные места обитания – заброшенные земли, пастбища, обочины дорог и окраины полей [15]. Во многих регионах Ae. tauschii, в смеси с другими видами эгилопсов, растет как сорняк на полях пшеницы, в садах и виноградниках. Предполагают, что эта особенность способствовала широкому распространению вида, которое шло параллельно расширению районов возделывания пшеницы. В качестве заносного вида Ae. tauschii зарегистрирован в США и некоторых странах Европы – Хорватии, Франции, Германии, Греции, Италии и Нидерландах [11].
Ae. tauschii характеризуется высокой изменчивостью морфологических, генетических и физиологических признаков растений [15–18]. Исходя из строения колоса, Eig [17] разделил Ae. squarrosa (=Ae. tauschii) на два подвида – ssp. eusquarrosa Eig (=ssp. tauschii) и ssp. strangulata (Eig) Tzvel. Представители ssp. tauschii (eusquarrosa) имеют удлиненный цилиндрический, иногда слабоверетеновидный колос, тогда как у ssp. strangulata колос четковидный, колоски квадратные. Образцы, относящиеся к подвиду strangulata (Str), в основном встречаются во влажных и сравнительно теплых горных районах: Гирканских лесах прикаспийского Ирана и в Закавказье [19]. Они характеризуются сравнительно невысокой изменчивостью морфолого-ботанических признаков растений, но обладают широким генетическим полиморфизмом. Представители подвида tauschii (Tau) намного более разнообразны по морфологии растений, но уровень генетического полиморфизма сравнительно невысок [20]. В пределах подвида tauschii выделяют три разновидности: var. typica, var. anathera и var. meyeri, различающиеся между собой строением колоса, наличием остей, габитусом и высотой растений, а также географическим распространением [15, 17, 19]. Наиболее широкие ареалы у разновидностей typica и anathera, тогда как var. meyri, считающийся промежуточным вариантом между ними, обнаружен в основном на западном побережье Каспийского моря [15].
В последние годы были накоплены данные, что, несмотря на морфологическое сходство, по генетическим характеристикам Ae. tauschii var. meyeri более близок подвиду strangulata, чем tauschii [2, 6, 21]. Несмотря на то что подвиды Ae. tauschii нередко произрастают совместно, гибриды между ними встречаются крайне редко [13, 18]. Считается, что Ae. tauschii ssp. strangulata был прародителем D-генома мягкой пшеницы [1, 13, 18, 22], тогда как ssp. tauschii участвовал в образовании полиплоидных видов Aegilops [23].
Ae. tauschii является ценным источником полезных признаков в селекции мягкой пшеницы [6, 24–26]. Многие образцы этого вида обладают высокой устойчивостью к болезням и абиотическим стрессам, а ряд генов, контролирующих устойчивость к листовой ржавчине, мучнистой росе, гессенской мухе и другим вредителям, был успешно интродуцирован в геном мягкой пшеницы от Ae. tauschii [27–30].
Близкое филогенетическое родство Ae. tauschii и мягкой пшеницы, а также его потенциал как донора ценных генов давно привлекали исследователей к изучению этого таксона. Внутривидовое разнообразие Ae. tauschii изучали с использованием изоферментных [16, 31–33], биохимических [18, 34–37], молекулярных [1, 2, 6, 12, 13, 38] и цитогенетических [21, 23, 39–41] маркеров. В результате проведенных исследований была разработана генетическая классификация С-окрашенных хромосом Ae. tauschii [39], построены генетические карты хромосом [42–44] и созданы SNP-панели для изучения генетического разнообразия этого вида и поиска целевых генов [12, 13, 22, 24, 45, 46]. Независимо от использованных подходов во всех работах выделяют две четко обособленные эволюционные линии Ae. tauschii [6, 13, 18, 20, 22, 47, 48], причем образцы, относящиеся к разным линиям, различались как по рисункам гибридизации с D-геномспецифичными зондами [21, 49], так и по спектрам запасных белков зерна – глиадинов [18, 35, 37].
В начале нового тысячелетия геном Ae. tauschii (4.3 млн пн) был секвенирован, а в качестве референсного был взят образец AL8/78, относящийся к подвиду strangulata как наиболее близкий D-геному мягкой пшеницы [50–52]. Результаты этих работ выявили сложную организацию генома Ae. tauschii, основную часть которого (85.9%) составляют мобильные элементы, преимущественно LTR-ретротранспозоны семейств Gypsy, 38.3%, CACTA, 16.8% и Copia, 16.5% [44]. Помимо этого, в D-геноме обнаружено избыточное количество сходных повторяющихся последовательностей, и он содержал больше дуплицированных диспергированных генов, чем другие виды злаков, геномы которых были секвенированы [51, 53]. Авторы предположили, что избыточное содержание сходных повторов часто приводит к ошибкам рекомбинации, в результате которых происходят дупликации генов и структурные перестройки хромосом, определяющие быструю эволюцию D-генома [51].
Крупный размер генома и большое количество повторяющихся последовательностей делают Ae. tauschii привлекательным объектом для цитогенетических исследований. Основными способами изучения кариотипов злаков на протяжении длительного времени были методы дифференциального окрашивания хромосом [54] и гибридизации in situ, однако число ДНК-зондов, пригодных для характеристики хромосом, было ограничено [23, 39, 41, 55–58]. Сравнительно недавно из генома мягкой пшеницы были изолированы несколько новых семейств повторов, некоторые из которых оказались специфичными для D-генома [59] и были использованы для анализа хромосом Ae. tauschii [21, 40], а также D-геномов пшениц и эгилопсов [59–62]. Совершенствование технологии FISH и разработка флуоресцентно-меченных олигонуклеотидных зондов, заменяющих стандартные плазмидные ДНК-пробы [63], существенно упростили цитогенетические исследования пшениц и их сородичей, что позволило проводить более масштабные анализы с привлечением большого числа образцов [62].
Широкие перспективы для изучения генетического разнообразия диких и культурных злаков, в частности пшеницы и ее сородичей, открывает изучение полиморфизма запасных белков зерна – глиадинов. Глиадины (Gli) относятся к классу проламинов, характеризующихся высоким содержанием глутамина и пролина и имеющих специфическую молекулярную структуру (размер, доменный состав, биохимические свойства) [64]. Глиадины оказывают существенное влияние на хлебопекарные качества пшеницы. Они кодируются двумя локусами – Gli-D1 и Gli-D2, расположенными на коротких плечах хромосом 1-й и 6-й гомеологических групп [65]. Показано, что Gli-локусы пшеницы и Ae. tauschii имеют сходную структурную организацию [66, 67]. Каждый локус представляет собой кластер тесносцепленных генов, контролирующий блоки полипептидов (компонентов), которые можно выявить электрофоретически. Для локусов характерен множественный аллелизм [36], позволяющий дифференцировать линии, образцы, сорта и разновидности, исследовать структуру популяций и определять филогенетическое родство между ними.
Настоящая работа посвящена оценке генетического разнообразия Ae. tauschii. Для оценки полиморфизма и сравнения двух подвидов были использованы методы электрофореза запасных белков зерна (глиадинов) и гибридизации in situ с 11 ДНК-зондами, представляющими сателлитные ДНК, семейства 5S (pTa794) и 45S (pTa71) рРНК генов и микросателлитные последовательности.
МАТЕРИАЛЫ И МЕТОДЫ
В работе были исследованы 44 образца Ae. tauschii, относящихся к подвидам strangulata (Str) и tauschii (Tau), из разных областей ареала вида (табл. 1). Методом электрофореза глиадинов (ЭФ) был исследован 41 образец, а цитогенетический анализ (FISH и C-banding соответственно) проведен на 13 образцах.
Таблица 1.
Список исследованных образцов и их происхождение
| № п/п | Коллекционный номер* | ssp. | var. | Хромосомная группа | Страна происхождения | Метод исследования** |
|---|---|---|---|---|---|---|
| 1 | К-79 | tauschii | typica | – | Дагестан, Деpбентский pайон, 8 км от аула Рукель (near from v. Rukel’) | ЭФ |
| 2 | K-249 | strangulata | strangulata | – | Туркменистан, Балканский велаят, Кара-Калинский район, Сюнт-Хасардагский хребет, горы Мезетли | ЭФ |
| 3 | K-258 | tauschii | typica | – | Таджикистан, Хатлонская, Khatlon Кумсаpгинский pайон, к-з “Победа” | ЭФ |
| 4 | К-285 | tauschii | typica | – | Азербайджан, Ахсуинский район, р. Пирсучай, Гидреманчайский мост | ЭФ |
| 5 | K-296 | strangulata | strangulata | – | Азербайджан, Варташенский район, дер. Якубли | ЭФ |
| 6 | К-329 | tauschii | typica | – | Азербайджан, Шемахинский район, с. Матраха | ЭФ |
| 7 | К-427 | tauschii | typica | – | Туркменистан, Кизил-Артек, Мешхед-Миссаринское плато | ЭФ |
| 8 | К-428 | tauschii | typica | – | Туркменистан, Кара-Кала, вдоль дороги от Яри-Калы к Шермопу | ЭФ |
| 9 | К-608 | tauschii | meyeri | – | Грузия, дер. Марнеули, по дороге Марнеула–Тбилиси, h = 530 м | ЭФ |
| 10 | К-624 | tauschii | typica | – | Азербайджан, Ахсуинский район, р. Пирсучай, Гидреманчайский мост | ЭФ |
| 11 | К-729 | strangulata | strangulata | – | Азербайджан, Шекинский район | ЭФ |
| 12 | K-865 | tauschii | typica | Tau | Неизв. | FISH, ЭФ |
| 13 | К-964 | tauschii | meyeri | – | Афганистан, Кабул, дорога Кабул–Кундуз, 3 км до перевала Саланг (Южный склон Гиндукуша) | ЭФ |
| 14 | К-966 | tauschii | meyeri | Tau | Афганистан, дорога Кабул–Кундуз, 56 км к югу от Пули-Хумри | FISH, ЭФ |
| 15 | К-974 | tauschii | meyeri | – | Афганистан, 18 км западнее Кундуза | ЭФ |
| 16 | К-985 | tauschii | typica | – | Афганистан, Джаузджан, 55 км по дороге Шиберган–Дарзаб | ЭФ |
| 17 | К-990 | tauschii | typica | – | Афганистан, Багдиз, 98 км по дороге Кайсар–Калай Нау | ЭФ |
| 18 | К-997 | tauschii | meyeri | – | Афганистан, Герат, 19 км по дороге Герат–Шиндад | ЭФ |
| 19 | К-999 | tauschii | typica | – | Афганистан, Нангархар, дорога Джалалабад–граница, 56 км | ЭФ |
| 20 | К-1098 | tauschii | meyeri | – | Азербайджан, Лерикский район, с. Талыш | ЭФ |
| 21 | К-1124 | strangulata | strangulata | – | Азербайджан, Нагорный Карабах, Степанокеpтский pайон, дорога Шушикенд–Сарушен | ЭФ |
| 22 | К-1155 | tauschii | solinii | Tau | Азербайджан, Лачинский район, г. Лачин, окрестности р. Акера | FISH, ЭФ |
| 23 | К-1202 | strangulata | strangulata | – | Армения, Ехегнадзорский район, к северу от с. Гетап | ЭФ |
| 24 | К-1659 | strangulata | strangulata | – | Афганистан | ЭФ |
| 25 | К-1804 | tauschii | meyeri | – | Узбекистан, Кашкадаpьинская обл., Гузаpский pайон, г. Дехканабад | ЭФ |
| 26 | К-1954 | strangulata | iranica | – | Иран, 26 км от г. Шираз, h = 1300 м | ЭФ |
| 27 | К-1958 | strangulata | iranica | Str | Иран, Гилян, г. Решт | C-b, ЭФ |
| 28 | К-1959 | strangulata | iranica | Str | Прикаспийский Иран, Сари-Мазандаран | FISH, ЭФ |
| 29 | К-1960 | strangulata | iranica | Str | Иран, дер. Байджан (Baijan), h = 1100 м | ЭФ |
| 30 | К-1661 | strangulata | strangulata | – | Иран, Керманшах, дер. Таги-Бостан (Taq-e Bostan) | ЭФ |
| 31 | К-1662 | strangulata | strangulata | Str | Иран, Фарс, дер. Доста-Арзан (Dasht-e Arzhan) | FISH, ЭФ |
| 32 | K-3187 | tauschii | typica | – | Армения, Мегринский район, с. Нювади | ЭФ |
| 33 | К-3298 | strangulata | strangulata | – | Иран, Западный Азербайджан, с. Хвой | ЭФ |
| 34 | К-3299 | strangulata | strangulata | – | Иран, Западный Азербайджан, 47.5 км на юго-восток от Маку к Хвой | ЭФ |
| 35 | К-4692 | tauschii | solinii | Tau | Казахстан, Южно-Казахстанская обл., Казгутский район, 5 км за поселком Рабат | C-b |
| 36 | К-4383 | tauschii | meyeri | Tau | Китай, Хэнань | FISH, ЭФ |
| 37 | К-4390 | tauschii | meyeri | Str | Китай, Синьцзян-Уйгурский АО, долина р. Или | FISH, ЭФ |
| 38 | K-4620 | tauschii | typica | – | Казахстан, Алмаатинская обл., Карасайский район, п. Жандосов. Остепненные склоны вдоль дороги | ЭФ |
| 39 | АЕ 149 | strangulata | typica | Str | Бывший СССР | FISH |
| 40 | АЕ 277 | tauschii | meyeri | Tau | Афганистан | FISH, ЭФ |
| 41 | AE 433 | tauschii | meyeri | – | Неизв., получен из США | ЭФ |
| 42 | АЕ 998 | tauschii | Tau | Болгария | FISH | |
| 43 | PI 560536 | tauschii | Турция, вдоль берега оз. Ван, 2 км СВ от Яйлияка (Yayliyaka) | ЭФ | ||
| 44 | TQ27 | strangulata | Str | Неизв. | C-b, FISH |
Примечание. ssp. – подвид; var. – разновидность; h – высота над ур. моря, м. * К – образцы из генбанка ВИР; АЕ – образцы из генбанка IPK; PI – образцы из генбанка USDA-ARS; TQ – образцы из генбанка Вейцмановского института науки. ** ЭФ – образец исследован методом электрофореза; C-b – образец исследован методом С-бэндинга; FISH – образец исследован методом флуоресцентной гибридизации in situ.
Цитогенетический анализ
Фиксацию материала, приготовление хромосомных препаратов и процедуру гибридизации in situ проводили в соответствии с протоколом [68].
В качестве ДНК-зондов были использованы следующие последовательности:
Плазмидные клоны
pTa71 – последовательность ДНК пшеницы длиной 9 тпн, кодирующая 18S, 5.8S и 26S рРНК гены, и спейсеры [69];
pTa794 – 420 пн-последовательность, включающая ген 5S рРНК пшеницы и межгенный спейсер [70];
pAs1 – фрагмент геномной ДНК Ae. tauschii длиной 1 тпн [56], относящийся к семейству Afa [71];
pSc119.2 – последовательность ДНК ржи длиной 120 пн [72];
pTa-s53 – фрагмент ДНК мягкой пшеницы длиной 587 пн [59];
pAesp_SAT86 – сателлитная ДНК с длиной мономера 86 пн, изолированная из генома Ae. speltoides [73].
ДНК метили биотином или флуоресцеином методом nick-трансляции с помощью Nick Translation Mix (Roche, Германия) в соответствии с рекомендациями фирмы-производителя.
Олигонуклеотидные зонды
pTa535-1 представлял собой олиго-пробу (5'‑AAA AAC TTG ACG CAC GTC ACG TAC AAA TTG GAC AAA CTC TTT CGG AGT ATC AGG GTT TC-3'), меченную по 5'-концу Су-3 или 6‑карбоксиметилродамином (TAMRA) [59, 63];
олиго-(CTT)10, меченный по 3'-концу Су-3 или 12-dUTP-флуоресцеином, синтезировали в лаборатории биологических микрочипов Института молекулярной биологии им. В.А. Энгельгардта РАН;
олиго-(ACT)10, меченный по 3'-концу Су-3, синтезировали в лаборатории биологических микрочипов Института молекулярной биологии им. В.А. Энгельгардта РАН;
олиго-(GTT)10-пробу, меченную по 3'-концу флуоресцеином-12, синтезировали в лаборатории биологических микрочипов Института молекулярной биологии им. В.А. Энгельгардта РАН.
Меченный флуоресцеином FAT-повтор амплифицировали методом ПЦР из геномной ДНК мягкой пшеницы, как описано в работе [74].
Препараты анализировали на микроскопе Zeiss-Imager D1 и отобранные метафазные пластинки фотографировали на цифровую видеокамеру AxioCam HRm с помощью пакета программ AxioImager, версия 4.6. Хромосомы классифицировали в соответствии с генетической номенклатурой [39]. Распределение разных типов последовательностей на хромосомах Ae. tauschii, выявленных методом FISH, сравнивали с рисунками С-окрашивания двух типичных образцов, представляющих подвиды strangulata (K-1958) и tauschii (К‑4692).
Электрофоретический анализ глиадинов
Электрофорез глиадинов проводили в полиакриламидном (ПААГ) геле в соответствии с опубликованной методикой [75]. Для каждого образца исследовали от трех до пяти зерен.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Сравнение С-окрашенных кариотипов двух образцов Ae. tauschii показало, что в целом они сходны по структуре кариотипов, общему содержанию гетерохроматина (ГХ) и рисункам С-бэндинга, но в то же время различались по распределению С-блоков на хромосомах 1D, 2D, 5D и 6D (рис. 1,l, m отмечены красными стрелками). Выявленные особенности были отмечены и у других изученных ранее образцов [23, 39], т.е. рисунки С-бэндинга специфичны для Ae. tauschii Str и Tau. Подвиды также различались по числу, размерам и распределению сигналов большинства исследованных зондов, выявленных методом FISH (рис. 1).
Рис. 1.
FISH. Распределение семейств повторяющихся ДНК pAs1 (a, b, d, e, g, h, j, n, o, зеленый псевдоцвет), pTa-535 (b, c, d, красный псевдоцвет), pTa-s53 (е, красный псевдоцвет), pAesp_SA86 (a, красный псевдоцвет), GAA (с, зеленый псевдоцвет; i, j, k, красный псевдоцвет), GTT (i, k, зеленый псевдоцвет), ACT (h, n, o, красный псевдоцвет), pSc119.2 (g, j, красный псевдоцвет) и cемейств 45S (красный) и 5S (зеленый псевдоцвет) рРНК генов (f) на хромосомах Ae. tauschii ssp. strangulata (b, k – K-1959; c, g, h – AE 149; k – K-4390), Ae. tauschii ssp. tauschii (a, d, i, n – K-966; f – TA 1588; e, j, o – K-1155). l, m – C-окрашенные метафазные пластинки образцов К-1958 (ssp. strangulata) и K-4692 (ssp. tauschii) соответственно. Хромосомы классифицированы по генетической номенклатуре. Масштабная линейка соответствует 10 мкм.
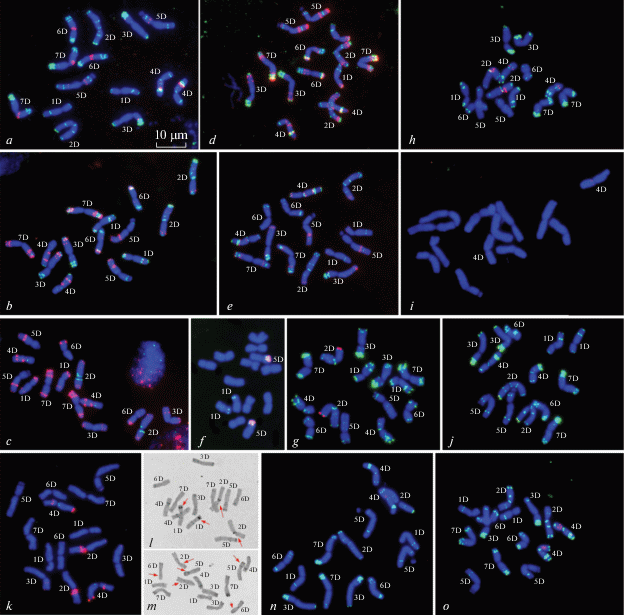
Последовательность (GTT)n у Ae. tauschii практически отсутствовала (рис. 1,i), и лишь у К-4390 (Str) в прицентромерных районах хромосом 3D и 4D детектировались точечные сигналы (рис. 1,k; 2). Микросателлит (GAA)n был обнаружен только у Str: крупные сигналы присутствовали на хромосомах 2DS и 4DS, более мелкие – на 1DS и 5DS, что соответствует данным Zhao et al. [21]. У образца К-966 из Афганистана очень крупные GAAn-сигналы наблюдались в субтеломерном участке 4DL (рис. 1,k; 2), но ни в иранском образце, ни в изученном ранее материале [21] такой локус не обнаружен. Zhao et al. [21] отмечали, что повтор (GAA)n у подвида tauschii практически отсутствует, что подтвердилось и в нашей работе (рис. 1,i). Другой исследовательской группе [40] не удалось выявить сигналов (GAA)n повтора ни в одном из исследованных ими образцов, независимо от таксономической принадлежности.
Микросателлитную последовательность ACTn на хромосомах Ae. tauschii ранее не картировали. В соответствии с полученными нами результатами она присутствует у всех образцов вида, однако число и хромосомная локализация сигналов варьировали между образцами. У Tau на 4D-хромосоме выявлено от одного до трех (ACT)10 сигналов (рис. 1,о, n). У образца К-1155 сигналы крупные и находились как в коротком (два), так и длинном плечах 4D (рис. 1,о), тогда как у К-966 слабый сигнал располагался в проксимальной части короткого плеча (рис. 1,n). В образцах Str (ACT)10 кластеры обнаружены на хромосомах 1DS, 2DS, 3DS и 4DS; их локализация перекрывалась с положением (GAA)n локусов.
Ранее было показано, что у мягкой пшеницы и некоторых других злаков кластеры микросателлитных последовательностей (GAA)n, (GTT)n и (ACT)n располагаются в районах конститутивного ГХ, выявляемого N-дифференциальным окрашиванием [76–79]. В D-геноме мягкой пшеницы четкие N-блоки видны на хромосомах 1D, 2D и 7D [80], две из которых – 1D и 2D – содержали сигналы (GAA)n и (ACT)n последовательностей в сходных позициях, однако на 7D ни одного из этих повторов обнаружено не было. Расхождение между данными N-бэндинга и FISH могут быть связаны с тем, что сравнение проводилось между D-геномом пшеницы и D-геномом его диплоидного предка, которые могли дивергировать друг от друга с момента полиплоидизации.
Гибридизация с пробами рРНК генов на хромосомах Ae. tauschii выявила два неравных локуса 5S рДНК, расположенных дистально на 1DS (крупный) и в середине плеча 5DS (мелкий), что соответствует опубликованным ранее данным [23, 57]. Единственный мажорный локус 45S рДНК находился на коротком плече хромосомы 5D (рис. 1,f), как сообщалось ранее [21, 23, 40, 41, 57]. Минорный сайт 45S рДНК на 7DL обнаружен лишь у некоторых образцов Ae. tauschii (рис. 2); ранее аналогичный локус был картирован на 7D-хромосоме мягкой пшеницы [81, 82].
Рис. 2.
Распределение разных типов ДНК-последовательностей на хромосомах Ae. tauschii, группа Tau. Номера образцов приведены сверху, комбинации зондов – внизу рисунка, цвет зонда соответствует цвету соответствующего сигнала. Розовые и зеленые стрелки указывают на позиции полиморфных сайтов. 1–7 – гомеологические группы.
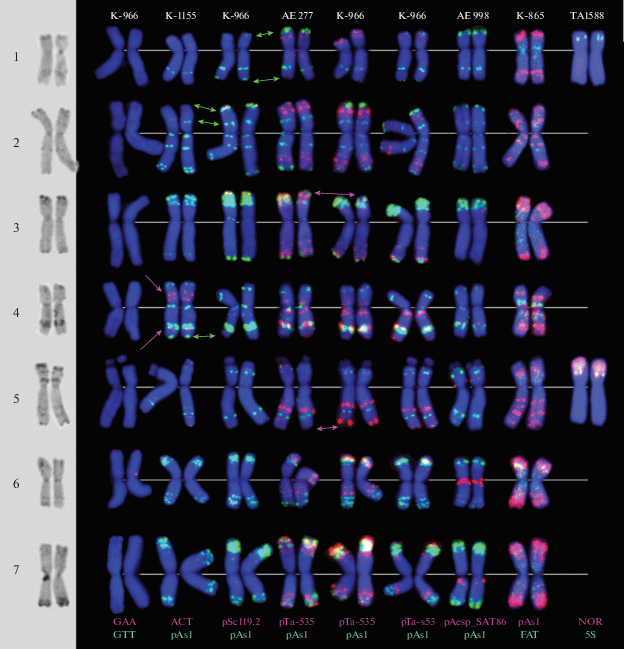
Сигналы pSc119.2-пробы у проанализированных нами (рис. 1,g, j; 2, 3), а также другими авторами [21, 23, 40, 41, 56] образцов Ae. tauschii были локализованы в субтеломерных участках трех или четырех пар хромосом: 2DS, 3DS, 4DS и реже 5DS. Размеры сигналов варьировали независимо от таксономической принадлежности образцов.
Подвиды Str и Tau не различались по рисункам гибридизации хромосом с pAesp_SAT86 повтором, однако мы наблюдали вариацию по размеру сигнала на 4DL. Кластеры этой последовательности выявлены также на хромосомах 3DS, 5DS (интерстициальные районы), 6D и 7D (прицентромерные районы; рис. 1,а; 2,3), наиболее крупные – на 4D, 6D и 7D. Полученная картина совпадает с локализацией пшеничного повтора рТа-713 [21, 40], являющегося гомологом pAesp_SAT86 [73], на хромосомах Ae. tauschii.
Рисунки гибридизации хромосом с рТа-535-зондом (рис. 1,b–d; 2, 3), полученные в настоящей работе, соответствовали приведенным другими авторами для Ae. tauschii [40], а также для D-генома мягкой пшеницы [59, 62, 63] и Ae. cylindrica [60]. В то же время они отличались от распределения зонда, описанного под названием pTa-535 в работе Zhao et al. [21]. Несмотря на выявленный между образцами полиморфизм, локализация сигналов рТа-535 была специфичной для индивидуальных хромосом Ae. tauschii, но несколько отличалась у представителей двух подвидов. Различия по хромосомам 2D, 3D, 6D и 7D были выражены более отчетливо (рис. 2, 3).
Рис. 3.
Распределение разных типов ДНК-последовательностей на хромосомах Ae. tauschii, группа Str. Номера образцов приведены сверху, комбинации зондов – внизу рисунка, цвет зонда соответствует цвету соответствующего сигнала. Розовые и зеленые стрелки указывают на позиции полиморфных сайтов. 1–7 – гомеологические группы.
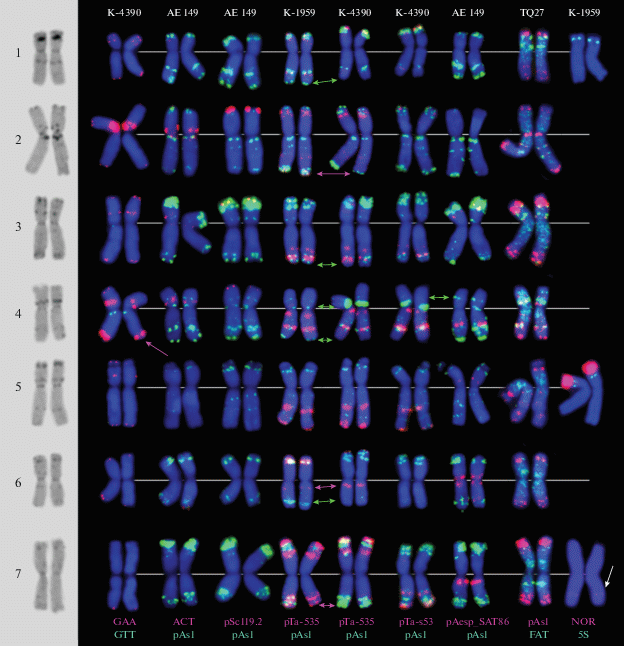
Ранее было показано, что D-геномы пшениц и эгилопсов обогащены FAT-повтором, который диффузно метит все хромосомы по длине и на этом фоне формирует несколько интенсивно флуоресцирующих кластеров [74]. Подвиды Tau и Str различались по числу и локализации таких кластеров, которые выявлялись только на двух парах хромосом – 4DS и 6DS образца Tau (рис. 2), но на четырех парах хромосом – 1DS, 3DS, 4D, 7DL – Str (рис. 3).
Рисунки мечения хромосом pAs1 зондом, полученные в настоящем исследовании (рис. 1–3), соответствовали таковым, приведенным другими авторами [39, 56, 58], и были идентичными распределению ДНК-зонда, описанного как рТа-535 в работе Zhao et al. [21]. pAs1-последовательность, относящаяся к семейству Afa-повторов [71], оказалась наиболее информативной не только для идентификации хромосом Ae. tauschii, но и как маркер для определения подвидов. Четкие различия между подвидами Str и Tau наблюдались по распределению зонда на хромосомах 2D, 3D, 4D, 5D и 6D, тогда как различия по хромосомам 1D и 7D не превышали пределов изменчивости, выявленной внутри каждого из них (рис. 2, 3).
Образцы Ae. tauschii, отнесенные к группе Str по результатам FISH или С-бэндинга [23], ведут происхождение из Ирана (К-1958, К-1959, К‑1960, KU-2160; TA 2529), Закавказья (К-588, K‑2194, G-3, AE 149), Афганистана (К-1740) и Китая (К-4390, Синьцзян-Уйгурский АО). Следует отметить, что связь популяции Ae. tauschii из Синьцзян-Уйгурского района Китая с иранскими формами была ранее установлена Wei et al. [12] методом ДНК-фингерпринтинга. В группу Tau вошли образцы более разнообразного географического происхождения: из Афганистана (АЕ 277 и К-966) и Пакистана (ТА 2381), Закавказья (G-2, К-1155, К-1656, К-1782) и Турции (ТА 1588), Туркменистана (G-1) и Казахстана (К-1611), Китая (К-4383 из Хэйнаня), и неизвестного происхождения (К-865, табл. 1).
Таким образом, исследование генетического разнообразия Ae. tauschii методом FISH подтвердило принадлежность всех генотипов к одному биологическому виду, но при этом выявило четкую внутривидовую дифференциацию, соответствующую таксономической классификации. Все образцы Ae. tauschii имели сходное содержание гетерохроматина, выявляемого С-окрашиванием по Гимза [23, 39], и идентичное расположение локусов 5S и 45S рДНК. Они были сходными по числу и распределению сайтов pSc119.2 и pAesp_SAT86 повторов, которые отличались от других диплоидных видов пшениц и Aegilops [56, 57], являясь, таким образом, видоспецифическим признаком.
Геномы всех образцов Ae. tauschii характеризовались избыточным содержанием повторов pAs1, pTa-535, pTa-s53, формирующих крупные, нередко перекрывающиеся кластеры в субтеломерных и интерстициальных районах хромосом, однако распределение сигналов на хромосомах подвидов Str и Tau несколько отличалось. Четкие различия между Str и Tau наблюдались и по наличию и распределению (GAA)n и (ACT)n кластеров, содержание которых у Str-образцов значительно превышало таковое у Tau.
В отличие от большинства других видов Aegilops, расположение сигналов (GAA)10- и (ACT)10-олиго-проб лишь частично совпадало с локализацией ГХ бэндов, выявляемых методом N-бэндинга на хромосомах D-генома пшеницы [83]. C другой стороны, рисунки С-бэндинга хромосом Ae. tauschii оказались весьма сходными с распределением pAs1-пробы. Эти два факта свидетельствуют в пользу того, что ГХ-блоки, выявляемые на хромосомах D-генома С-окрашиванием по Гимза, имеют другой нуклеотидный состав, чем ГХ-блоки хромосом А и В геномов полиплоидных пшениц или S, T, M, U, N и C геномов Aegilops, образованных преимущественно (GAA)n-микросателлитом [76, 84–87]. Str-образцы более близки к D-геному мягкой пшеницы в содержании и хромосомной локализации GAAn и ACTn последовательностей, распределении pAs1-повтора. Это еще раз подтверждает участие подвида strangulata в происхождении мягкой пшеницы [1, 20, 42, 51].
Для получения более полного представления о внутривидовой изменчивости Ae. tauschii и его роли в происхождении T. aestivum мы провели анализ электрофоретических спектров глиадина у 41 линии Ae. tauschii разного географического происхождения и таксономической принадлежности. Исследование продемонстрировало высокое генетическое разнообразие этого вида (рис. 4,а), что соответствует данным других авторов [18, 35, 36]. По числу ЭФ-компонентов глиадинов (бэндов на ЭФ-спектре), контролируемых хромосомами D-генома, Ae. tauschii значительно превышает мягкую пшеницу. Так, суммарное число компонентов глиадина, контролируемых D-геномом, у мягкой пшеницы составляет 6–11, а у Ae. tauschii – 14–30 (рис. 4,а). Снижение разнообразия глиадинов у мягкой пшеницы было описано ранее [36] и может быть обусловлено элиминацией части генетического материала родительских геномов при образовании мягкой пшеницы [3], а также эффектом “бутылочного горлышка” при формировании полиплоидного вида.
Рис. 4.
Разнообразие ЭФ-спектров глиадинов у Ae. tauschii. а – сравнение образцов разного географического происхождения (номера образцов приведены вверху рисунка). М.808 – стандартный сорт мягкой пшеницы Мироновская 808; Без. 1 – Безостая-1; Gli-D1 и Gli-D2 – зоны ЭФ-спектра, компоненты которых контролируются соответствующими глиадиновыми локусами. Кружками отмечены компоненты глиадина мягкой пшеницы, контролируемые локусом Gli-D1, ромбиками – локусом Gli-D2; б – ЭФ-спектры Ae. tauschii К-1098 (а) и К-1202 (b) в сравнении с сортом мягкой пшеницы Мироновская 808 (с). Слева дано схематическое изображение блоков, контролируемых локусами Gli-D1g, Gli-D2e; в – ЭФ-спектры Ae. tauschii К-1098 (а), К-1124 (b), К-79 (c), К-3299 (d) в сравнении со стандартным сортом мягкой пшеницы Мироновская 808 (е, с блоком Gli-D2e).
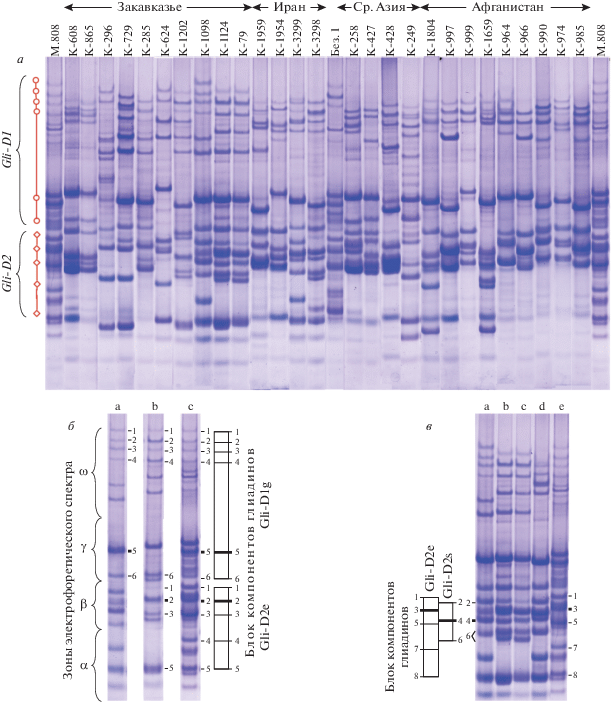
В исследованной нами выборке три пары образцов: К-1659 (Афганистан)–К-1662 (Иран), К‑865 (неизв.)–К-608 (Грузия), а также К-1958 (Иран)–К-1938 (Ирак) имели идентичные спектры глиадинов. При таком большом генетическом разнообразии, которое мы наблюдаем у Ae. tauschii (только по локусу Gli-D1 у исследованных линий обнаружено более 20 аллелей), можно с большой вероятностью предположить, что образцы с идентичными спектрами являются дубликатами. Очень близкми по ЭФ-спектру оказались образцы К-4562 и К-966, которые различались только по минорным компонентам из γ-зоны (рис. 4,а).
Следует отметить, что группы компонентов глиадинов, выявленные у некоторых образцов, были идентичными или очень близкими по подвижности блокам компонентов мягкой пшеницы, контролируемым локусом Gli-D1 (1-я гомеологическая группа хромосом). Так, у К-3187, K‑1098 и K-1202 из Закавказья (Армения и Азербайджан) идентифицированы блоки компонентов, идентичные блоку Gli-D1g (рис. 4,б), который достаточно широко распространен у сортов мягкой пшеницы [75]. Образец К-1202 из Армении имел идентичный с мягкой пшеницей набор компонентов, контролируемый не только 1D, но 6D-хромосомой – блок Gli-D2е, как у сорта мягкой пшеницы Мироновская 808 (рис. 4,б). То же самое характерно и для образца К-1098 из Азербайджана, только у него набор компонентов, контролируемых локусом Gli-D2, больше соответствовал блоку Gli-D2s (рис. 4,в).
Несмотря на то что в исследованном материале не было найдено вариантов, идентичных блокам Gli-D2 мягкой пшеницы (6-я гомеологическая группа), мы выявили несколько образцов Ae. tauschii со сходными по расположению и подвижности компонентов спектрами. Это – К-79 из Дагестана, К-964, К-974 и К-990 из Афганистана; К-1954, К-1959 и К-3299 из Ирана; К-428 из Туркменистана, К-329, К-624, К-1098 и К-1124 из Азербайджана. Варианты спектра, выявленные у этих образцов, были сходны с блоком Gli-D2s мягкой пшеницы (рис. 4,в). Следует отметить, что по сравнению с аллелями Gli-D1g и Gli-D2e, часто встречающимися у пшеницы, Gli-D2s является редким и поэтому вероятность того, что формы Ae. tauschii со спектром, похожим на Gli-D2s, являются донорами генома D мягкой пшеницы, невысока.
Полученные в настоящей работе данные подтверждают результаты других исследователей о наличии двух генетически четко обособленных групп Ae. tauschii, соответствующих подвидам strangulata (Str) и tauschii (Tau). Образцы Str характеризуются более сложной организацией генома, которая выражается, в частности, в наличии кластеров микросателлитных повторов, которые практически отсутствуют у представителей подвида Tau, а также более сложными спектрами запасных белков зерна – глиадинов. Анализ изменчивости некодирующих последовательностей хлоропластной ДНК, а также ряда генов ядерного генома показал, что разделение Ae. tauschii на два подвида произошло на самых ранних этапах эволюции вида [20, 88]. По общему содержанию и распределению семейств повторяющихся ДНК и семейств рРНК генов D-геном Ae. tauschii более сходен с А-геномами диплоидных видов пшеницы [73], чем с любым из существующих видов Aegilops. Исходя из гипотезы происхождения геномов диплоидных Aegilops в результате гомоплоидной гибридизации древних прародителей Ae. speltoides (2n = 2x = 14, геном SS) и диплоидных пшениц (2n = 2x = 14, AA) [89], D-геном современного Ae. tauschii можно считать одним из наиболее “древних” и филогенетически близких именно А-геному, тогда как вклад S-генома в его эволюцию, по-видимому, невелик. Это хорошо согласуется с опубликованными недавно данными, согласно которым хлоропластный геном Ae. tauschii ведет происхождение от A-геномных предков, при этом в процессе стабилизации гомоплоидного гибрида сохранялись преимущественно гены ядерно-цитоплазматического комплекса материнского родителя [90].
Данные цитогенетического исследования и ЭФ-анализа глиадинов подтверждают, что D-геном мягкой пшеницы был унаследован от образца Ae. tauschii, относящегося к подвиду strangulata. Хотя большинство данных указывают на прикаспийские районы Ирана как центр образования мягкой пшеницы [1, 20], наиболее близкие по составу глиадинов к этой культуре образцы обнаружены в Армении и Азербайджане. Это дает основание предположить, что кавказские формы Ae. tauschii внесли существенный вклад в формирование современного генофонда мягкой пшеницы.
Образцы были получены из коллекций генетических ресурсов растений ВИР им. Н.И. Вавилова, Института культурных растений (IPK), Гатерслебен (Германия), USDA-ARS, Абердин, Айдахо, США и Вейцмановского института науки, Реховот (Израиль).
Работа выполнена в рамках бюджетных проектов № 0112-2019-0002 ИОГен РАН и № 0662-2019-0006 ВИР при частичной поддержке гранта РФФИ 17-04-00087.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Dvořák J., Luo M.C., Yang Z.L., Zhang H.B. The structure of the Aegilops tauschii genepool and the evolution of hexaploid wheat // Theor. Appl. Genet. 1998. V. 97(4). P. 657–670. https://doi.org/10.1007/s001220050942
Lelley T., Stachel M., Grausgruber H., Vollmann J. Analysis of relationships between Aegilops tauschii and the D genome of wheat utilizing microsatellites // Genome. 2000. V. 43(4). P. 661–668. http://dx.doi.org/doi:10.1139/gen-43-4-661
Feldman M. Origin of cultivated wheat // The World Wheat Book. A History of Wheat Breeding / Eds. Bonjean A.P., Angus W.J. Ltd.: Londres; Paris; N.Y.: Tec & Doc/Intersept, 2001. P. 3–56.
McFadden E.S., Sears E.R. The origin of Triticum spelta and its free-threshing hexaploid relatives // J. Hered. 1946. V. 37(3). P. 81–107. https://doi.org/10.1093/oxfordjournals.jhered.a105590
Kihara H. Die entdeckung des DD-analysators beim Weizen // Agric. and Hortic. Tokio. 1944. V. 19. P. 889–890.
Lubbers E.L., Gill K.S., Cox T.S., Gill B.S. Variation of molecular markers among gehically diverse accessions of Triticum tauschii // Genome. 1991. V. 34(3). P. 354–361. https://doi.org/10.1139/g91-057
Feldman M., Levy A.A. Origin and evolution of wheat and related Triticeae species // Alien Introgression in Wheat: Cytogenetics, Molecular Biology, and Genomics / Eds. Molnár-Láng M., Ceoloni C., Doležel J. Intern. Publ.: Springer, 2015. P. 21–76.
Kihara H. Considerations on the evolution and distribution of Aegilops species based on the analyser-method // Cytologia. 1954. V. 19(4). P. 336–357. https://doi.org/10.1508/cytologia.19.336
Tsunewaki K. Plasmon analysis as the counterpart of genome analysis // Methods of Genome Analysis in Plant: Their Merrits and Piffals / Ed. Jauhar P.P. CRC Press: Boca Ration, 1996. P. 271–299.
Kilian B., Mammen K., Millet E. et al. Aegilops // Wild crop relatives: Genomics and breeding resources // Cereals / Ed. Kole C. Berlin; Heidelberg: Springer, 2011. P. 1–76.
Van Slageren M.W. Wild Wheats: A Monograph of Aegilops L. and Amblyopyrum (Jaub. et Spach) Eig (Poaceae). Wageningen: Wageningen Agricultural Univ., Wageningen and ICARDA, Aleppo, Syria, 1994. 514 p.
Wei H., Li J., Peng Z. et al. Relationships of Aegilops tauschii revealed by DNA fingerprints: The evidence for agriculture exchange between China and the West // Progress Natural Sci. 2008. V. 18(12). P. 1525–1531. https://doi.org/10.1016/j.pnsc.2008.05.022
Singh N., Wu S., Tiwari V. et al. Genomic analysis confirms population structure and identifies inter-lineage hybrids in Aegilops tauschii // Front. Plant Sci. 2019. V. 10(9). https://doi.org/10.3389/fpls.2019.00009
Жуковский П.М. Критико-систематический обзор видов рода Aegilops L. // Тр. прикл. бот., генет., селек. 1928. T. 28(1). С. 417–609.
Kihara H., Tanaka M. Morphological and physiological variation among Aegilops squarrosa strains collected in Pakistan, Afghanistan and Iran // Preslia. 1958. V. 30. P. 241–251.
Dudnikov A.J. Aegilops tauschii Coss.: allelic variation of enzyme-encoding genes and ecological differentiation of the species // Gen. Res. Crop Evol. 2014. V. 61(7). P. 1329–1344. https://doi.org/10.1007/s10722-014-0115-4
Eig A. Monographisch-kritische Ubersicht der Gattung Aegilops // Repertorium specierum novarum regni vegetabilis. Berlin: Beihefte, 1929. V. 55. 228 p.
Dudnikov A.J. Polymorphism of gliadins in Aegilops tauschii Coss. local populations in two primary habitats in Dagestan // Gen. Res. Crop Evol. 2018. V. 65(3). P. 845–854. https://doi.org/10.1007/s10722-017-0575-4
Dudnikov A.J. Searching for an effective conservation strategy of Aegilops tauschii genetic variatio // Cereal Res. Com. 2009. V. 37(1). P. 31–36. https://doi.org/10.1556/CRC.37.2009.1.4
Dudnikov A.J. Polymorphism of Got2 DNA sequences sheds light on Aegilops tauschii Coss. intraspecies divergence and origin of Triticum aestivum L. // Gen. Res. Crop Evol. 2017. V. 64(7). P. 1623–1640. https://doi.org/10.1007/s10722-016-0461-5
Zhao L., Ning S., Yi Y. et al. Fluorescence in situ hybridization karyotyping reveals the presence of two distinct genomes in the taxon Aegilops tauschii // BMC Genomics. 2018. V. 19(1). P. 3. https://doi.org/10.1186/s12864-017-4384-0
Wang J., Luo M.C., Chen Z. et al. Aegilops tauschii single nucleotide polymorphisms shed light on the origins of wheat D-genome genetic diversity and pinpoint the geographic origin of hexaploid wheat // New Phytol. 2013. V. 198(3). P. 925–937. https://doi.org/10.1111/nph.12164
Badaeva E.D., Amosova A.V., Muravenko O.V. et al. Genome differentiation in Aegilops. 3. Evolution of the D-genome cluster // Plant Syst. Evol. 2002. V. 231(1–4). P. 163–190. https://doi.org/10.1007/s006060200018
Rawat N., Schoen A., Singh L. et al. TILL-D: An Aegilops tauschii TILLING resource for wheat improvement // Front. Plant Sci. 2018. V. 9(1665). https://doi.org/10.3389/fpls.2018.01665
Rehman A., Evans N., Gianibelli M.C., Rose R.J. Allelic variations in high and low molecular weight glutenins at the Glu-D(t) locus of Aegilops tauschii as a potential source for improving bread wheat quality // Austr. Jour. Agric. Res. 2008. V. 59(5). P. 399–405. https://doi.org/10.1071/ar07229
Suneja Y., Gupta A.K., Bains N.S. Stress adaptive plasticity: Aegilops tauschii and Triticum dicoccoides as potential donors of drought associated morpho-physiological traits in wheat // Front. Plant Sci. 2019. V. 10(211). https://doi.org/10.3389/fpls.2019.00211
Schneider A., Molnár I., Molnár-Láng M. Utilisation of Aegilops (goatgrass) species to widen the genetic diversity of cultivated wheat // Euphytica. 2008. V. 163(1). P. 1–19. https://doi.org/10.1007/s10681-007-9624-y
Gill B.S., Raupp W.J., Sharma H.C. et al. Resistance in Aegilops squarrosa to wheat leaf rust, wheat powdery mildew, greenbug, and Hessian fly // Plant Dis. 1986. V. 70. P. 553–556. https://doi.org/10.1094/PD-70-553
McIntosh R.A., Yamazaki Y., Dubkovsky G. et al. Catalogue of Gene Symbols for Wheat // The 12th Intern. Wheat Genetics Symp., 8–13 Sept. 2013. Yokohama, Japan, 2013. P. 395.
Saluja M., Kaur S., Bansal U. et al. Molecular mapping of linked leaf rust resistance and non-glaucousness gene introgressed from Aegilops tauschii Coss. in hexaploid wheat Triticum aestivum L. // Plant Gen. Res.: Characterization and Utilization. 2017. V. 16(1). P. 82–88. https://doi.org/10.1017/S1479262116000460
Jaaska V. Aspartate aminotransferase and alcohol dehydrogenase isoenzymes: Intraspecific differentiation in Aegilops tauschii and the origin of the D genome polyploids in the wheat group // Plant Syst. Evol. 1981. V. 137(4). P. 259–273. https://doi.org/10.1007/BF00982790
Nishikawa K., Furuta Y., Goshima H. Genetic studies of α-amylase isozymes in wheat. II. Reconstituted AABB tetraploid, Aegilops squarrosa and their synthetic AABBDD hexaploid // Jpn. J. Genet. 1975. V. 50(5). P. 409–416. https://doi.org/10.1266/jjg.50.409
Nishikawa K., Furuta Y., Wada T. Genetic studies on α‑amylase in wheat. III. Intraspecific variation in Aegilops squarrosa and the birthplace of hexaploid wheat // Jpn. J. Genet. 1980. 55(5). P. 325–336. https://doi.org/10.1266/jjg.55.325
Lafiandra D., Masci S., D’Ovidio R. et al. Relationship between the D genome of hexaploid wheats (AABBDD) and Ae. squarrosa as deduced by seed storage proteins and molecular marker analyses // Hereditas. 1992. V. 116(3). P. 233–238. https://doi.org/10.1111/j.1601-5223.1992.tb00147.x
Конарев В.Г. Белки пшеницы. М.: Колос, 1980. 351 с.
Yan Y.M., Hsam S.L.K., Yu J.Z. et al. Genetic polymorphisms at Gli-D-t gliadin loci in Aegilops tauschii as revealed by acid polyacrylamide gel and capillary electrophoresis // Plant Breed. 2003. V. 122(2). P. 120–124. https://doi.org/10.1046/j.1439-0523.2003.00824.x
Masci S., D’Ovidio R., Lafiandra D. et al. Electrophoretic and molecular analysis of alpha-gliadins in Aegilops species (Poaceae) belonging to the D-genome cluster and in their putative progenitors // Plant Syst. Evol. 1992. V. 179(1–2). P. 115–128. https://doi.org/10.1007/bf00938024
Saeidi H., Rahiminejad M.R., Heslop-Harrison J.S. Retroelement insertional polymorphisms, diversity and phylogeography within diploid, D-genome Aegilops tauschii (Triticeae, Poaceae) Sub-taxa in Iran // Ann. Bot. 2008. V. 101(6). P. 855–861. https://doi.org/10.1093/aob/mcn042
Friebe B., Mukai Y., Gill B.S. C-banding polymorphism in several accessions of Triticum tauschii (Aegilops squarrosa) // Genome. 1992. V. 35(2). P. 192–199. https://doi.org/10.1139/g92-030
Majka M., Kwiatek M.T., Majka J., Wisniewska H. Aegilops tauschii accessions with geographically diverse origin show differences in chromosome organization and polymorphism of molecular markers linked to leaf rust and powdery mildew resistance genes // Front. Plant Sci. 2017. V. 8. P. 1149. https://doi.org/10.3389/fpls.2017.01149
Molnár I., Kubaláková M., Šimková H. et al. Flow cytometric chromosome sorting from diploid progenitors of bread wheat, T. urartu, Ae. speltoides and Ae. tauschii // Theor. Appl. Genet. 2014. V. 127(5) P. 1091–1104. https://doi.org/10.1007/s00122-014-2282-2
Gill K.S., Lubbers E.L., Gill B.S. et al. A genetic linkage map of Triticum tauschii (DD) and its relationshiop to the D genome of bread wheat (AABBDD) // Genome. 1991. V. 34(3). P. 362–374. https://doi.org/10.1139/g91-058
Boyko E.V., Gill K.S., Mickelson-Yang L. et al. A high-density genetic linkage map of Aegilops tauschii, the D-genome progenitor of bread wheat // Theor. Appl. Genet. 1999. V. 99(1–2). P. 16–26.https://doi.org/10.1007/s001220051204
Zhao G., Zou C., Li K. et al. The Aegilops tauschii genome reveals multiple impacts of transposons // Nature Plants. 2017. V. 3(12). P. 946–955. https://doi.org/10.1038/s41477-017-0067-8
You F.M., Huo N., Deal K.R. et al. Annotation-based genome-wide SNP discovery in the large and complex Aegilops tauschii genome using next-generation sequencing without a reference genome sequence // BMC Genomics. 2011. V. 59. http://www.biomedcentral.com/1471-2164/12/59
Mizuno N., Yamasaki M., Matsuoka Y. et al. Population structure of wild wheat D-genome progenitor Aegilops tauschii Coss.: implications for intraspecific lineage diversification and evolution of common wheat // Mol. Ecol. 2010. V. 19(5). P. 999–1013. https://doi.org/10.1111/j.1365-294X.2010.04537.x
Dudnikov A.J. Spatial patterns of adenylate kinase, catalase, endopeptidase and fructose-1,6-diphosphatase encoding genes allelic variation in Aegilops tauschii Coss. // Gen. Res. Crop Evol. 2012. V. 59(1). P. 1–8. https://doi.org/10.1007/s10722-011-9659-8
Gogniashvili M., Jinjikhadze T., Maisaia I. et al. Complete chloroplast genomes of Aegilops tauschii Coss. and Ae. cylindrica Host sheds light on plasmon D evolution // Current Genet. 2016. V. 62(4). P. 791–798. https://doi.org/10.1007/s00294-016-0583-5
Бадаева Е.Д. Хромосомный анализ при исследовании происхождения B-(G-) геномов полиплоидных пшениц // Биол. мембраны. 2001. Т. 18. № 3. С. 216–229.
Akhunov E., Akhunova A., Dvořák J. BAC libraries of Triticum urartu, Aegilops speltoides and Ae. tauschii, the diploid ancestors of polyploid wheat // Theor. Appl. Genet. 2005. V. 111(8). P. 1617–1622. https://doi.org/10.1007/s00122-005-0093-1
Luo M.-C., Gu Y.Q., Puiu D. et al. Genome sequence of the progenitor of the wheat D genome Aegilops tauschii // Nature. 2017. V. 551. P. 498. https://doi.org/10.1038/nature24486
Jia J., Zhao S., Kong X. et al. Aegilops tauschii draft genome sequence reveals a gene repertoire for wheat adaptation // Nature. 2013. V. 496(7443). P. 91–95. https://doi.org/10.1038/nature12028
Huo N.X., Dong L.L., Zhang S.L. et al. New insights into structural organization and gene duplication in a 1.75-Mb genomic region harboring the α-gliadin gene family in Aegilops tauschii, the source of wheat D genome // Plant J. 2017. V. 92(4). P. 571–583. https://doi.org/10.1111/tpj.13675
Friebe B., Gill B.S. Chromosome banding and genome analysis in diploid and cultivated polyploid wheats // Methods in Genome Analysis in Plants: Their Merits and Pitfals / Ed. Jauhar P.P., New York; London; Tokyo. CRC Press: Boca Ration, 1996. P. 39–60.
Rayburn A.L., Gill B.S. Isolation of a D-genome specific repeated DNA sequence from Aegilops squarrosa // Plant Mol. Biol. Rep. 1986. V. 4(2). P. 102–109. https://doi.org/10.1007/BF02732107
Badaeva E.D., Friebe B., Gill B.S. Genome differentiation in Aegilops. 1. Distribution of highly repetitive DNA sequences on chromosomes of diploid species // Genome. 1996. V. 39(2). P. 293–306. https://doi.org/10.1139/g96-040
Badaeva E.D., Friebe B., Gill B.S. Genome differentiation in Aegilops. 2. Physical mapping of 5S and 18S-26S ribosomal RNA gene families in diploid species // Genome. 1996. V. 39(6). P. 1150–1158. https://doi.org/10.1139/g96-145
Rayburn A.L., Gill B.S. Molecular analysis of the D-genome of the Triticeae // Theor. Appl. Genet. 1987. V. 73(3). P. 385–388. https://doi.org/10.1007/BF00262505
Komuro S., Endo R., Shitaka K., Kato A. Genomic and chromosomal distribution patterns of various repeated DNA sequences in wheat revealed by a fluorescence in situ hybridization procedure // Genome. 2013. V. 56(3). P. 131–137. https://doi.org/10.1139/gen-2013-0003
Mirzaghaderi G., Houben A., Badaeva E. Molecular-cytogenetic analysis of Aegilops triuncialis and identification of its chromosomes in the background of wheat // Mol. Cytog. 2014. V. 7(1). P. 91. https://doi.org/10.1186/s13039-014-0091-6
Tang S., Tang Z., Qui L. et al. Developing new oligo probes to distinguish specific chromosomal segments and the A, B, D genomes of wheat (Triticum aestivum L.) using ND-FISH // Front. Plant Sci. 2018. V. 9(1104). https://doi.org/10.3389/fpls.2018.01104
Jiang M., Xiao Z.Q., Fu S.L., Tang Z.X. FISH karyotype of 85 common wheat cultivars/lines displayed by ND-FISH using oligonucleotide probes // Cer. Res. Commun. 2017. V. 45(4). P. 549–563. https://doi.org/10.1556/0806.45.2017.049
Tang Z., Yang Z., Fu S. Oligonucleotides replacing the roles of repetitive sequences pAs1, pSc119.2, pTa-535, pTa71, CCS1, and pAWRC.1 for FISH analysis // J. App. Genet. 2014. V. 55(3). P. 313–318. https://doi.org/10.1007/s13353-014-0215-z
Shewry P.R., Halford N.G. Cereal seed storage proteins: structures, properties and role in grain utilization // J. Exp. Bot. 2002. V. 53(370). P. 947–958. https://doi.org/10.1093/jexbot/53.370.947
Kasarda D.D., Bernardin J.E., Qualset C.O. Relationship of gliadin protein components to chromosomes in hexaploid wheats (Triticum aestivum L.) // Proc. Natl Acad. Sci. USA. 1976. V. 73(10). P. 3646–3650. http://www.pnas.org/cgi/content/abstract/73/10/3646
Dong L., Huo N., Wang Y. et al. Rapid evolutionary dynamics in a 2.8-Mb chromosomal region containing multiple prolamin and resistance gene families in Aegilops tauschii // Plant J. 2016. V. 87(5). P. 495–506. https://doi.org/10.1111/tpj.13214
Huo N., Zhang S., Zhu T. et al. Gene duplication and evolution dynamics in the homeologous regions harboring multiple prolamin and resistance gene families in hexaploid wheat // Front. Plant Sci. 2018. V. 9(673). https://doi.org/10.3389/fpls.2018.00673
Badaeva E.D., Ruban A.S., Aliyeva-Schnorr L. et al. In situ hybridization to plant chromosomes // Fluorescence In Situ Hybridization (FISH). Application Guide / Ed. Liehr T. Berlin: Springer, 2017. P. 477–494.
Gerlach W.L., Bedbrook J.R. Cloning and characterization of ribosomal RNA genes from wheat and barley // Nucleic Acids Res. 1979. V. 7(7). P. 1869–1885. https://doi.org/10.1093/nar/7.7.1869
Gerlach W.L., Dyer T.A. Sequence organization of the repeated units in the nucleus of wheat which contains 5S-rRNA genes // Nucl. Acids Res. 1980. V. 8(21). P. 4851–4865. https://doi.org/10.1007/BF00327389
Nagaki K., Tsujimoto H., Isono K., Sasakuma T. Molecular characterization of a tandem repeat, Afa family, and its distribution among Triticeae // Genome. 1995. V. 38(3). P. 479–486. https://doi.org/10.1139/g95-063
Bedbrook R.J., Jones J., O’Dell M. et al. A molecular description of telomeric heterochromatin in Secale species // Cell. 1980. V. 19(2). P. 545–560. https://doi.org/10.1016/0092-8674(80)90529-2
Badaeva E.D., Amosova A.V., Goncharov N.P. et al. A set of cytogenetic markers allows the precise identification of all A-genome chromosomes in diploid and polyploid wheat // Cytogenet. Genome Res. 2015. V. 146(1). P. 71–79. https://doi.org/10.1159/000433458
Badaeva E., Zoshchuk S.A., Paux E. et al. Fat element – a new marker for chromosome and genome analysis in the Triticeae // Chrom. Res. 2010. V. 18(6). P. 697–709. https://doi.org/10.1007/s10577-010-9151-x
Упелниек В.П., Новосельская-Драгович А.Ю., Шишкина А.А и др. Лабораторный анализ белков семян пшеницы. Технологическая инструкция “Диагностика сортового соответствия и чистоты семян пшеницы”. Методическое пособие. М.: ИОГен РАН, 2013. 173 с.
Pedersen C., Langridge P. Identification of the entire chromosome complement of bread wheat by two-colour FISH // Genome. 1997. V. 40(5). P. 589–593. https://doi.org/10.1139/gen-40-5-589
Cuadrado A., Schwarzacher T., Jouve N. Identification of different chromatin classes in wheat using in situ hybridization with simple sequence repeat oligonucleotides // Theor. Appl. Genet. 2000. V. 101(5). P. 711–717. https://doi.org/10.1007/s001220051535
Cuadrado A., Jouve N. The nonrandom distribution of long clusters of all possible classes of trinucleotide repeats in barley chromosomes // Chrom. Res. 2007. V. 15(6). P. 711–720. https://doi.org/10.1007/s10577-007-1156-8
Mukai Y., Friebe B., Gill B.S. Comparison of C-banding patterns and in situ hybridization sites using highly repetitive and total genomic rye DNA probes of ‘Imperial’ rye chromosomes added to ‘Chinese Spring’ wheat // Jpn. J. Genet. 1992. V. 67(2). P. 71–83. https://doi.org/10.1266/jjg.67.71
Endo T.R., Gill B.S. Somatic karyotype, heterochromatin distribution, and nature of chromosome differentiation in common wheat, Triticum aestivum L. em Thell. // Chromosoma. 1984. V. 89(5). P. 361–369. https://doi.org/10.1007/BF00331253
Mukai Y., Endo T.R., Gill B.S. Physical mapping of the 18S.26S rRNA multigene family in common wheat: Identification of a new locusc // Chromosoma. 1991. V. 100(2). P. 71–78. https://doi.org/10.1007/BF00418239
Jiang J., Gill B.S. New 18S-26S ribosomal RNA gene loci: chromosomal landmarks for the evolution of polyploid wheats // Chromosoma. 1994. V. 103(3). P. 179–185. https://doi.org/10.1007/BF00368010
Endo T.R., Gill B.S. Identification of wheat chromosomes by N-banding // Proc. 6th Intern. Wheat Genetics Symp., Nov. 28–Dec. 3 1983. Kyoto, Japan: Plant Germ-Plasm Inst., Kyoto Univ., 1984. P. 355–362.
Vrána J., Kubaláková M., Simková H. et al. Flow sorting of mitotic chromosomes in common wheat (Triticum aestivum L.) // Genetics. 2000. V. 156(4). P. 2033–2041. http://www.genetics.org/cgi/content/abstract/156/4/2033.
Adonina I.G., Goncharov N.P., Badaeva E.D. et al. (GAA)n microsatellite as an indicator of the A genome reorganization during wheat evolution and domestication // Comp. Cytogenet. 2015. V. 9. P. 533–547. https://doi.org/10.3897/CompCytogen.v9i4.5120
Molnár I., Vrána J., Burešová V. et al. Dissecting the U, M, S and C genomes of wild relatives of bread wheat (Aegilops spp.) into chromosomes and exploring their synteny with wheat // Plant J. 2016. V. 88(3). P. 452–467. https://doi.org/10.1111/tpj.13266
Cuadrado A., Cardoso M., Jouve N. Increasing the physical markers of wheat chromosomes using SSRs as FISH probes // Genome. 2008. V. 51. P. 809–815. https://doi.org/10.1139/G08-065
Dudnikov A.J. Chloroplast DNA non-coding sequences variation in Aegilops tauschii Coss.: evolutionary history of the species // Gen. Res. Crop Evol. 2012. V. 59(5). P. 683–699. https://doi.org/10.1007/s10722-011-9711-8
Marcussen T., Sandve S., Heier L. et al. Ancient hybridizations among the ancestral genomes of bread wheat // Science. 2014. V. 345(6194). https://doi.org/10.1126/science.1250092
Liu B., Li C., Wang J. et al. Cytonuclear coevolution following homoploid hybrid speciation in Aegilops tauschii // Mol. Biol. Evol. 2018. V. 36(2). P. 341–349. https://doi.org/10.1093/molbev/msy215
Дополнительные материалы отсутствуют.


