Генетика, 2019, T. 55, № 11, стр. 1369-1376
Аллельный полиморфизм гена INVINH1 у образцов кукурузы из коллекции ВИР
М. А. Филюшин 1, *, Е. А. Дьяченко 1, Э. Б. Хатефов 2, А. В. Щенникова 1, Е. З. Кочиева 1, 3, К. Г. Скрябин 1, 3
1 Федеральный исследовательский центр “Фундаментальные основы биотехнологии”
Российской академии наук
119071 Москва, Россия
2 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
3 Московский государственный университет им. М.В. Ломоносова,
кафедра биотехнологии
119991 Москва, Россия
* E-mail: michel7753@mail.ru
Поступила в редакцию 15.03.2019
После доработки 09.04.2019
Принята к публикации 29.04.2019
Аннотация
Определены последовательности гена ингибитора апопластных инвертаз INVINH1 у 71 образца кукурузы из коллекции Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (Россия). Идентифицировано 35 аллельных вариантов данного гена. У 21 образца кукурузы в положении –95 от старт-кодона была обнаружена протяженная вставка (143, 190, 192 и 210 пн). В белках INVINH1 выявлено 21 замещение аминокислотных остатков, шесть из которых потенциально радикальные (A49V, A49G, A65P, S97I, C99R, D108Y) и локализованы в функциональном домене PMEI.
Кукуруза (Zea mays L.) является основной зерновой культурой в мире, ежегодное производство которой составляет более 1.1 млрд т (FAOSTAT, 2017). В сухом зерне кукурузы содержится более 70% крахмала и около 10% белков, поэтому кукуруза является важным источником пищи для человека и животных, а также сырьем для получения этанола и биотоплива [1]. Также кукуруза является модельным организмом для молекулярно-генетических исследований, в том числе связанных с углеводным метаболизмом [2, 3].
К настоящему времени у кукурузы идентифицированы и описаны гены метаболизма крахмала и сахарозы [2–5], а также различные транскрипционные факторы, регулирующие данные процессы [6–8]. Одними из важнейших ферментов углеводного метаболизма растений являются инвертазы (INV, EC 3.2.1.26), которые катализируют необратимый гидролиз сахарозы в глюкозу и фруктозу [5]. Сахароза по флоэме транспортируется в апопласт, цитоплазму и вакуоль, в каждом из этих компартментов функционируют определенные инвертазы [9]. В геноме кукурузы идентифицирован 21 ген, кодирующий инвертазы различной клеточной локализации [5], которые играют важную роль во время раннего развития зерна кукурузы, превращая сахарозу, транспортируемую из листьев, в гексозы для поддержки клеточного деления в эндосперме и эмбрионе [10].
Активность инвертаз регулируется посттрансляционно ингибиторами инвертаз за счет формирования этими белками нековалентного комплекса (1 : 1), в результате чего контролируется гидролиз сахарозы [11]. Белки INVINH являются членами суперсемейства ингибиторов пектинметилэстераз (PMEI; EC 3.1.1.11) и содержат сигнальный пептид и домен PMEI [10]. Роль ингибиторов в регуляции активности инвертаз была показана с помощью оверэкспрессии или замалчивания экспрессии кодирующих их генов в модельных растениях [11]. Кроме того было показано влияние транскрипционной активности ингибиторов инвертаз на некоторые физиологические процессы, такие как развитие семян и плодов, старение листьев и ответной реакции на биотические и абиотические стрессы [12–14].
У кукурузы идентифицирован и охарактеризован только один ген ингибитора инвертаз – INVINH1, для которого показана специфичная экспрессия на ранних стадиях развития зерна и предположено его участие в регуляции синтеза и накопления крахмала в клетках эндосперма путем ингибирования апопластных инвертаз [10]. Ген ZmINVINH1 локализован на хромосоме 2 и не содержит интронов. У сахарного тростника, который, как и кукуруза, относится к трибе Andropogoneae, идентифицировано два гена ингибиторов инвертаз, один из которых, ShINH1, гомологичен INVINH1 Z. mays [11]. Несмотря на то, что к настоящему времени определены полные геномы девяти генотипов кукурузы Z. mays subsp. mays и подвида Z. mays subsp. mexicana (который считается одним из предков современной кукурузы), данные о полиморфизме гена INVINH1 отсутствуют.
Целью данного исследования стала идентификация и анализ последовательностей гена INVINH1 у сортов и линий кукурузы из коллекции Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (ВИР, г. Санкт-Петербург, Россия).
Для работы были использованы 71 образец кукурузы из коллекции ВИР, включая тетраплоидные линии и образцы дикорастущих популяций из Перу, а также один образец подвида Z. mays subsp. parviglumis (табл. 1). ДНК выделяли из четырехдневных проростков согласно [15]. На основе доступных в NCBI геномных последовательностей гена INVINH1 девяти известных генотипов Z. mays subsp. mays (B73, CML247, DK105, EP1, F7, Mo17, PE0075, PH207, W22) и подвида Z. mays subsp. mexicana были разработаны праймеры INH1-F (5'-ACCAGCCACAATCCATTTATTAG-3') и INH1-R (5'-CTTAATAAGCCCGAGATCATCA-3') для амплификации данного гена у анализируемых образцов кукурузы. Амплификацию последовательностей проводили с использованием реактивов для ПЦР (ООО “Диалат Лтд”) в следующих условиях: исходная денатурация (5 мин, 95°C); 35 циклов денатурации (30 с, 95°C), отжига (30 с, 56°C) и синтеза (1 мин 30 с, 72°C); завершающий синтез (3 мин, 72°C). Полученные ампликоны секвенировали с помощью праймеров INH1-F и INH1-R с использованием системы BigDye и Applied Biosystems 3730 DNA Analyzer (ЦКП Биоинженерия, ФИЦ Биотехнологии РАН). Анализ и выравнивание нуклеотидных и аминокислотных последовательностей проводили с помощью программы MEGA 7.0 (https://www.megasoftware.net/). Присутствие сигнального пептида и PMEI-домена в последовательности белков определяли с помощью программ SignalP4.0 (http://www.cbs.dtu.dk/ services/SignalP-4.0/) и NCBI-CDD (http:// www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) соответственно. Радикальные и нейтральные аминокислотные замещения были предсказаны в программе PROVEAN (http://provean.jcvi.org/seq_submit.php). Аллельный полиморфизм анализируемых последовательностей генов ингибиторов инвертаз определяли путем их сравнения с последовательностью INVINH1 генотипа инбредной линии B73, который используется в геномных исследованиях Z. mays subsp. mays в качестве референса [16].
Таблица 1.
Список анализируемых образцов кукурузы и выявленные для них аллельные варианты гена INVINH1 и варианты белка
| Линия/сорт/, подвид Z. mays | Номер по каталогу ВИР | Аллельный вариант гена INVINH1 | Вариант белка | Наличие (+)/отсутствие (–) вставки в положении –95 от старт-кодона, ее размер, пн |
|---|---|---|---|---|
| Тео-4 | С-1760 | A1 | P1 | +(192) |
| Тео 5465-1-1 | С-1761 | A5 | P5 | +(143) |
| МП4а-1 | С-1762 | A16 | P13 | – |
| МП4а-3 | С-1763 | A5 | P5 | +(143) |
| МП4а-4 | С-1764 | A17 | P14 | +(192) |
| МП4в | С-1765 | A18 | P15 | +(143) |
| 1/130-1 | С-1766 | A6 | P6 | +(210) |
| 1/99-3-3-1 | С-1767 | A1 | P1 | +(192) |
| 1/99-3-3-4 | С-1768 | A19 | P4 | – |
| 1/99-3-3-5 | С-1769 | A1 | P1 | +(192) |
| 1/99-3-3-7 | С-1770 | A20 | P16 | +(192) |
| A21 | P17 | – | ||
| КБЗ | К-23994 | A22 | P18 | – |
| С-42 | С-42 | A1 | P1 | +(190) |
| С-154 | С-154 | A23 | P19 | – |
| С-163 | С-163 | A2 | P2 | – |
| С-224 | С-224 | A4 | P4 | +(192) |
| С-237 | С-237 | A3 | P3 | – |
| С-282 | С-282 | A24 | P20 | +(192) |
| С-498 | С-498 | A8 | P8 | – |
| С-551 | С-551 | A1 | P1 | +(143) |
| С-561 | С-561 | A4 | P4 | +(192) |
| С-562 | С-562 | A25 | P10 | – |
| С-680 | С-680 | A8 | P8 | – |
| С-740 | С-740 | A2 | P2 | – |
| С-751 | С-751 | A12 | P10 | +(190) |
| A26 | P21 | – | ||
| С-764 | С-764 | A2 | P2 | – |
| С-765 | С-765 | A2 | P2 | – |
| С-782 | С-782 | A27 | P12 | – |
| С-787 | С-787 | A3 | P3 | – |
| С-794 | С-794 | A1 | P1 | – |
| С-802 | С-802 | A2 | P2 | – |
| С-803 | С-803 | A8 | P8 | – |
| С-814 | С-814 | A28 | P22 | +(143) |
| С-817 | С-817 | A1 | P1 | +(192) |
| С-818 | С-818 | A6 | P6 | +(210) |
| Куско-1 | К-23977 | A11 | P9 | – |
| Куско-3 | К-23976 | A11 | P9 | – |
| Куско-4 | К-23980 | A10 | P4 | – |
| Куско-5 | К-23979 | A29 | P1 | +(143) |
| Ранняя Лакомка | К-23993 | A1 | P1 | – |
| С-450 | С-450 | A13 | P11 | – |
| Тетрасил | К- 23427 | A15 | P3 | – |
| С-430 | С-430 | A12 | P10 | +(190) |
| A30 | P23 | – | ||
| 1170-1 | С-1244 | A14 | P12 | – |
| 1139-4 | С-1771 | A1 | P1 | – |
| 1139 -1 | С-1005 | A1 | P1 | – |
| 1137-3 | С-987 | A14 | P12 | – |
| 1176-10 | С-1286 | A1 | P1 | – |
| 1194-7 | С-1444 | A2 | P2 | – |
| 1142 -2 | С-1034 | A7 | P7 | – |
| 1169-10 | С-1233 | A3 | P3 | – |
| 1189-6 | С-1392 | A31 | P2 | – |
| 1189-9 | С-1396 | A2 | P2 | – |
| 1192-5 | С-1172 | A9 | P1 | – |
| 1160-9 | С-1190 | A3 | P3 | – |
| 1167-4 | С-1229 | A3 | P3 | – |
| 1178-8 | С-1773 | A2 | P2 | – |
| 1191-15 | С-1229 | A9 | P1 | – |
| 1188-1 | С-1774 | A2 | P2 | – |
| 1170-2 | С-1245 | A32 | P24 | – |
| 1158-1 | С-1168 | A7 | P7 | – |
| 1184-9 | С-1370 | A3 | P3 | – |
| 1177-5 | С-1304 | A7 | P7 | +(210) |
| 1150-9 | С-1116 | A6 | P6 | – |
| 1150-7 | С-1114 | A3 | P3 | – |
| 1184-4 | С-1364 | A2 | P2 | – |
| 1173-1 | С-1273 | A15 | P3 | – |
| 1205-2 | С-1522 | A33 | P25 | – |
| 1173-2 | С-1274 | A1 | P1 | – |
| 1149-1 | С-1100 | A4 | P4 | – |
| parviglumis | 984 | A13 | P11 | – |
| mexicana* | A4 | P4 | – | |
| B73* | A1 | P1 | +(190) | |
| DK105* | A1 | P1 | +(192) | |
| F7* | A1 | P1 | – | |
| PH207* | A1 | P1 | – | |
| CML247* | A10 | P4 | – | |
| EP1* | A4 | P4 | +(192) | |
| Mo17* | A35 | P27 | – | |
| W22* | A34 | P26 | – | |
| PE0075* | A5 | P5 | +(143) |
С помощью разработанных праймеров у 71 образца кукурузы из коллекции ВИР были амплифицированы и секвенированы последовательности гена INVINH1. Размер кодирующей части гена INVINH1 у всех анализируемых образцов был одинаков и составил 531 пн. При сравнении с референсной последовательностью INVINH1 образца B73 в исследуемых генах было найдено 37 нуклеотидных замен (6.97% от общей длины). Анализ распределения этих замен у анализируемых образцов кукурузы и генотипов из NCBI позволил выявить 35 аллельных вариантов гена INVINH1 (табл. 2). Аллельные варианты INVINH1 отличались от последовательности INVINH1 B73 SNPs. В подавляющем большинстве они представляли собой различные комбинации определенного набора SNPs (табл. 2), чем и объясняется большое количество выявленных аллельных вариантов. Пять аллельных вариантов были представлены единичными заменами.
Таблица 2.
Аллельные варианты гена INVINH1 и частота их встречаемости у анализируемых образцов кукурузы из коллекции ВИР
| Аллель | Нуклеотидные замены | Число образцов | Частота встречаемости, % |
|---|---|---|---|
| A1 | – | 12 | 16.2 |
| A2 | C93T; C146T | 10 | 13.5 |
| A3 | C93T; G121T; T389C; G451T; G481C; A531G | 7 | 9.5 |
| A4 | G214T; T234C | 3 | 4.1 |
| A5 | C186G; C195T; G214T; T389C; G481C; A531G | 2 | 2.7 |
| A6 | G12A; C24T; G121T; G214T; T389C; G451T | 3 | 4.1 |
| A7 | C93T; G121T; T389C | 3 | 4.1 |
| A8 | C93T; G121T; G214T; T389C; G451T; G481C; A531G | 3 | 4.1 |
| A9 | C348A | 2 | 2.7 |
| A10 | G214T | 1 | 1.4 |
| A11 | C146T; G214T | 2 | 2.7 |
| A12 | C19G; G385A; T389C; G481C; A531G | 2 | 2.7 |
| A13 | C19G; T389C; A531G | 2 | 2.7 |
| A14 | C146T; G385A; T389C; G481C; A531G | 2 | 2.7 |
| A15 | C93T; G121T | 2 | 2.7 |
| A16 | C93T; G121T; G290A; T389C; G451T; G481C; A531G | 1 | 1.4 |
| A17 | G76A; G214T; T234C | 1 | 1.4 |
| A18 | C186G; C195T; G214T; T389C; G398C | 1 | 1.4 |
| A19 | G214T | 1 | 1.4 |
| A20 | T295C; G315C; T389C; G481C; A531G | 1 | 1.4 |
| A21 | C146T; G214T; T234C; G385A; G481C | 1 | 1.4 |
| A22 | C93T; G121T; G290T; C341G; T389C; G451T; G471A; G481C; G498T; A531G | 1 | 1.4 |
| A23 | C19G; T389C; G481C; A531G | 1 | 1.4 |
| A24 | G102A; G273C; G290A; G358A | 1 | 1.4 |
| A25 | C19G; G385A; T389C; G481C; A531G | 1 | 1.4 |
| A26 | G76A; G102A; G290T; G312T; G315C; G322T; G363T; G429A; C454T | 1 | 1.4 |
| A27 | C146T; G385A; T389C; G481C; A531G | 1 | 1.4 |
| A28 | G398C | 1 | 1.4 |
| A29 | A531G | 1 | 1.4 |
| A30 | G76A; G322T; G337T; G429A | 1 | 1.4 |
| A31 | C93T; C146T; G471A | 1 | 1.4 |
| A32 | C93T; G121T; C146G; T389C; G481C | 1 | 1.4 |
| A33 | G193C; G207A; C348A | 1 | 1.4 |
| A34* | C93T; G121T; G214T; T389C; G481C; A531G | – | – |
| A35* | C93T; G121T; T389C; G481C; A531G | – | – |
Наиболее частым вариантом оказалась последовательность гена референсного образца B73 (аллель А1), выявленным у 12 (16.2%) исследуемых образцов кукурузы коллекции ВИР, а также у генотипов DK105, F7, PH207 из базы данных NCBI. Также были выявлены редкие аллели А16–A35, которые встречались только у одного из анализируемых образцов (табл. 2).
Прародителем современной кукурузы считается подвид Z. mays subsp. parviglumis [17]. Анализ полногеномных последовательностей современных линий кукурузы и гибридов с теосинте (Z. mays subsp. mexicana) показал, что около 10% генома кукурузы происходит от subsp. mexicana [16]. Последовательность INVINH1 у анализируемых образцов Z. mays subsp. mexicana и Z. mays subsp. parviglumis отличается от референса B73 двумя (аллель A4) и тремя (аллель А13) нуклеотидными заменами соответственно. Аллельный вариант А4 (G214T; T234C), характерный для subsp. mexicana, был выявлен у трех образцов (С-224, С-561 и 1149-1), а аллельный вариант А13 (C19G; T389C; A531G) subsp. parviglumis – у образца С-450. Интересно, что у исследуемого 71-го образца кукурузы замены G214T (mexicana), T389C и A531G (parviglumis) были одновременно выявлены в составе нескольких аллелей, в то время как замены T234С (mexicana) и C19G (parviglumis) оказались редкими и не встречались вместе, а также в комбинации с заменами T389C и A531G (parviglumis) и G214T (mexicana) соответственно.
Полученные ампликоны содержали, помимо кодирующей части, 5'-нетранслируемый участок, размер которого варьировал (400–600 пн). В положении –95 от старт-кодона у 21-го образца кукурузы была выявлена вставка: 143 пн (шесть образцов), 190 пн (три образца), 192 пн (девять образцов) и 210 пн (три образца) (табл. 1). Подобная вставка присутствовала также у четырех генотипов кукурузы (B73 (190 пн), DK105 (192 пн), EP1 (192 пн), PE0075 (143 пн)) из базы NCBI. Три анализируемые образца кукурузы (1/99-3-3-7, С-751 и С-430) были гетерозиготны по наличию/отсутствию описанной выше вставки (табл. 1).
Полученные кодирующие последовательности INVINH1 71-го образца кукурузы были транслированы. Мы показали, что 35 выявленных аллелей соответствовали 27 вариантам аминокислотной последовательности INVINH1 (рис. 1).
Рис. 1.
Варианты последовательности белка INVINH1, выявленные у анализируемых образцов кукурузы. Сигнальный пептид и PMEI-домен подчеркнуты сверху. Сайты замещения аминокислот показаны желтым фоном, радикальные выделены красным шрифтом со звездочкой сверху. Рамкой отмечены консервативные остатки цистеина (Cys28, Cys43, Cys95 и Cys142).
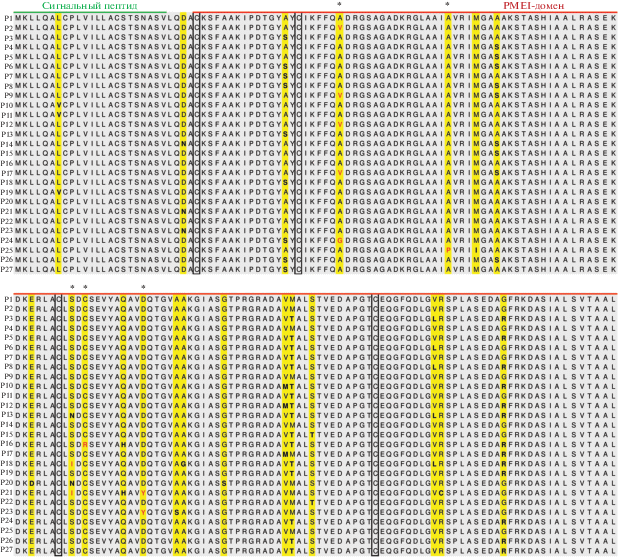
В аминокислотных последовательностях INVINH1 у всех анализируемых образцов был идентифицирован сигнальный пептид (1–23 а.о.) и домен PMEI (28–175 а.о.). В последовательности домена PMEI были выявлены четыре консервативных остатка цистеина (Cys28, Cys43, Cys95 и Cys142), которые, как было показано ранее, значимы для функции белка, поскольку образуют две дисульфидные связи и дополнительно фиксируют четырех-шпилечную структуру домена [18].
Всего в последовательностях белков INVINH1 было выявлено 21 замещение аминокислотных остатков, шесть из которых, согласно предсказанию программы PROVEAN, являются потенциально радикальными. Одно нейтральное замещение (L7V) находится в сигнальном пептиде, ответственном за транспортировку ингибитора в апопласт [10]. Остальные замещения локализованы в домене PMEI, в том числе шесть радикальных (A49V, A49G, A65P, S97I, C99R, D108Y) (рис. 1). Наличие радикальных замен в функциональном домене может влиять на пространственную структуру белка и, как следствие, его способность связываться с инвертазами, либо возможность принимать конформацию, необходимую для инактивации инвертаз [18].
Таким образом, была идентифицирована и проанализирована последовательность гена ингибитора апопластных инвертаз INVINH1 у 71-го образца кукурузы из коллекции ВИР. Сравнительный анализ нуклеотидных последовательностей INVINH1 анализируемых образцов кукурузы с референсной последовательностью Z. mays subsp. mays B73 позволил идентифицировать 35 аллельных вариантов, что подтверждает высокий уровень внутривидового геномного полиморфизма современной кукурузы, как было показано ранее [1, 16]. Более глубокое изучение функций ингибитора апопластных инвертаз INVINH1 и его связей с углеводным метаболизмом кукурузы может быть использовано для получения линий с измененным содержанием крахмала и сахаров в зерне.
Работа выполнена с использованием экспериментальной установки искусственного климата – ЭУИК (Институт биоинженерии ФИЦ Биотехнологии РАН), при поддержке РФФИ (№ 18-29-07007) и Министерства науки и высшего образования Российской Федерации.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объектов животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объектов людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Sun S., Zhou Y., Chen J. et al. Extensive intraspecific gene order and gene structural variations between Mo17 and other maize genomes // Nat. Genet. 2018. V. 50. № 9. P. 1289–1295. https://doi.org/10.1038/s41588-018-0182-0
Zhang X., Colleoni C., Ratushna V. et al. Molecular characterization demonstrates that the Zea mays gene sugary2 codes for the starch synthase isoform SSIIa // Plant Mol. Biol. 2004. V. 54. P. 865–879.
Yan H.B., Pan X.X., Jiang H.W., Wu G.J. Comparison of the starch synthesis genes between maize and rice: copies, chromosome location and expression divergence // Theor. Appl. Genet. 2009. V. 119. № 5. P. 815–825. https://doi.org/10.1007/s00122-009-1091-5
Chourey P.S., Jain M., Li Q.B., Carlson S.J. Genetic control of cell wall invertases in developing endosperm of maize // Planta. 2006. V. 223. № 2. P. 159–167.
Wang L., Zheng Y., Ding S. et al. Molecular cloning, structure, phylogeny and expression analysis of the invertase gene family in sugarcane // BMC Plant Biol. 2017. V. 17. № 1. Article 109. https://doi.org/10.1186/s12870-017-1052-0
Zhang Z., Zheng X., Yang J. et al. Maize endosperm-specific transcription factors O2 and PBF network the regulation of protein and starch synthesis // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 39. P. 10842–10847. https://doi.org/10.1073/pnas.1613721113
Cai H., Chen Y., Zhang M. et al. A novel GRAS transcription factor, ZmGRAS20, regulates starch biosynthesis in rice endosperm // Physiol. Mol. Biol. Plants. 2017. V. 23. № 1. P. 143–154. https://doi.org/10.1007/s12298-016-0404-9
Qi X., Li S., Zhu Y. et al. ZmDof3, a maize endosperm-specific Dof protein gene, regulates starch accumulation and aleurone development in maize endosperm // Plant Mol. Biol. 2017. V. 93. № 1–2. P. 7–20. https://doi.org/10.1007/s11103-016-0543-y
Ma H., Albert H.H., Paull R., Moore P.H. Metabolic engineering of invertase activities in different subcellular compartments affects sucrose accumulation in sugarcane cells // Functional Plant Biology. 2000. V. 27. P. 1021–1030. https://doi.org/10.1071/PP00029
Bate N.J., Niu X., Wang Y. et al. An invertase inhibitor from maize localizes to the embryo surrounding region during early kernel development // Plant Physiol. 2004. V. 134. № 1. P. 246–254.
Shivalingamurthy S.G., Anangi R., Kalaipandian S. et al. Identification and functional characterization of sugarcane invertase inhibitor (ShINH1): a potential candidate for reducing pre- and post-harvest loss of sucrose in sugarcane //Front. Plant Sci. 2018. V. 9. Article 598. https://doi.org/10.3389/fpls.2018.0059
Jin Y., Ni D.A., Ruan Y.L. Posttranslational elevation of cell wall invertase activity by silencing its inhibitor in tomato delays leaf senescence and increases seed weight and fruit hexose level // Plant Cell. 2009. V. 21. P. 2072–2089. https://doi.org/10.1105/tpc.108.063719
Xu X., Hu Q., Yang W.N., Jin Y. The roles of call wall invertase inhibitor in regulating chilling tolerance in tomato // BMC Plant Biol. 2017. V. 17. Article 195. https://doi.org/10.1186/s12870-017-1145-9
Zuma B., Dana M.B., Dongfang W.D. Prolonged expression of a putative invertase inhibitor in micropylar endosperm suppressed embryo growth in Arabidopsis // Front. Plant Sci. 2018. V. 9. Article 61. https://doi.org/10.3389/fpls.2018.00061
Филюшин М.А., Решетникова Н.М., Кочиева Е.З., Скрябин К.Г. Внутривидовая вариабельность ITS-последовательностей у паразитического растения Monotropa hypopitys L. из популяций европейской части России // Генетика. 2015. Т. 51. № 11. С. 1330–1334.
Yang N., Xu X.W., Wang R.R. et al. Contributions of Zea mays subspecies mexicana haplotypes to modern maize // Nat. Commun. 2017. V. 8. Article 1874. https://doi.org/10.1038/s41467-017-02063-5
van Heerwaarden J., Doebley J., Briggs W.H. et al. Genetic signals of origin, spread, and introgression in a large sample of maize landraces // Proc. Natl Acad. Sci. USA. 2011. V. 108. P. 1088–1092. https://doi.org/10.1073/pnas.1013011108
Brummell D.A., Chen R.K., Harris J.C. et al. Induction of vacuolar invertase inhibitor mRNA in potato tubers contributes to cold-induced sweetening resistance and includes spliced hybrid mRNA variants // J. Exp. Bot. 2011. V. 62. № 10. P. 3519–3534. https://doi.org/10.1093/jxb/err043
Дополнительные материалы отсутствуют.


