Генетика, 2019, T. 55, № 12, стр. 1461-1465
Оценка эффективности использования митохондриального маркера 12S для реконструкции филогении акантобделлид
А. В. Болбат 1, *, Н. В. Сороковикова 1, И. А. Кайгородова 1, 2
1 Лимнологический институт Сибирского отделения Российской академии наук
664003 Иркутск, Россия
2 Иркутский государственный университет, кафедра физико-химической биологии
664003 Иркутск, Россия
* E-mail: bolbatav@lin.irk.ru
Поступила в редакцию 17.01.2019
После доработки 13.03.2019
Принята к публикации 26.03.2019
Аннотация
На протяжении многих лет к решению спорных вопросов филогении применяются методы, основанные на молекулярном анализе консервативных участков генетического материала для расчета дивергенции организмов. В настоящей работе использован маркерный фрагмент гена 12S рРНК для уточнения филогенетического положения малоизученной фаунистической группы – древних пиявкоподобных паразитов рода Acanthobdella. Мозаичное сочетание признаков малощетинковых червей (Oligochaeta) и современных пиявок (Hirudinea) указывает на промежуточное эволюционное положение акантобделл. Полученные результаты позволяют говорить о хорошей применимости маркерного фрагмента 12S для филогенетического анализа на уровне родов и видов. Реконструированная нами филогенетическая история акантобделлид не согласуется с результатами ранних работ. Низкие статистические поддержки позволяют сделать заключение о непригодности маркера 12S для анализа филогении на надродовом уровне.
Митохондриальный геном животных является хорошим объектом для филогенетических исследований ввиду его гаплоидного наследования, отсутствия интронов и ограниченной рекомбинации [1]. Митохондриальный ген 12S рРНК часто используется для реконструкции молекулярной филогении, видовой идентификации и филогеографии (например, [2–4]). Длина его маркерного фрагмента варьирует в пределах 350–450 пар нуклеотидов ввиду большого количества делеций и инсерций, и он применялся для изучения событий дивергенции средней и большой давности [5]. В то же время скорость эволюции гена 12S оценивается как высокая, что может привести к насыщению в третьей позиции и искажению результатов исследования филогении таксонов высшего ранга [1]. Данный маркер уже был успешно использован для видового разграничения при исследовании европейских плоских пиявок [6].
На сегодняшний день систематика кольчатых червей остается актуальной и претерпевает множество изменений. Положение различных таксонов данного типа не определено однозначно и оценивается рядом авторов по-разному [7–9]. Acanthobdella peledina, обитающая в северной части Евразии [10] и, по некоторым данным, в Северной Америке [11, 12], является редким реликтовым пиявкоподобным паразитом, мозаично сочетающим в себе примитивные черты предков из подкласса малощетинковых червей с признаками, присущими современным пиявкам [10, 13]. Согласно современным представлениям о филогении и классификации, A. peledina вместе с сестринским видом A. livanowi занимает промежуточное эволюционное положение между подклассами Oligochaeta и Hirudinea. Впервые вывод об уникальном филогенетическом положении акантобделл был сделан Н.А. Ливановым в 1905 г. на основании исключительно морфологических характеристик [13]. Однако существовала и альтернативная точка зрения о принадлежности акантобделл к подклассу Oligochaeta и независимой конвергенции “пиявочных” черт [14]. Ввиду этого классификация пиявок и пиявкоподобных паразитов многократно менялась [10, 15, 16].
Первые работы по молекулярной филогении акантобделл начали проводиться на рубеже XX и XXI вв. на основе анализа ядерного гена 18S рРНК и митохондриального гена малой единицы цитохромоксидазы COI [7, 17]. В данных работах последовательности генов A. peledina уверенно кластеризовались в отдельные независимые ветви, занимающие промежуточное положение между подклассами Oligochaeta и Hirudinea, что согласуется с результатами морфологического анализа, проведенного Ливановым и подтвержденного современными морфологическими методами [18]. Однако несмотря на то что для A. peledina была получена последовательность гена 12S рРНК, она использовалась в филогенетическом анализе только как группа сравнения для укоренения дерева Hirudinea в целом [19]. Работ по установлению филогенетического положения представителей рода Acanthobdella по маркеру 12S проведено не было.
Для настоящей работы мы получили нуклеотидные последовательности образцов, собранных с хариуса и ленка на р. Верхняя Рассоха (приток р. Чечуй, бассейн р. Лена) в Киренском районе Иркутской области. Фрагмент гена 12S рРНК был амплифицирован с использованием пары универсальных праймеров [20]. В анализ была включена последовательность 12S рРНК A. peledina, полученная в 1999 г. [19]. В качестве группы сравнения для филогенетического анализа из международной базы генетических данных GenBank были загружены последовательности 55 представителей Polychaeta, Oligochaeta и Hirudinea. Реконструкцию деревьев по методу максимального правдоподобия (ML) проводили c использованием модели GTR+G+I, рекомендованной ModelTest, с помощью пакета программ MEGA7 [21]. Для байесовского анализа филогении (BI) мы использовали пакет программного обеспечения BEAST v. 2.5.1 [22] с моделью замен GTR+G и начальными параметрами, рекомендованными jModelTest v. 0.1 [23]. Сходимость ESS статистики байесовского анализа проверяли по программе Tracer v. 1.7.1.
При применении двух методов филогенетического анализа кластеризация различных последовательностей как на уровне видов, так и на уровне высших таксонов в целом совпала. При анализе таксонов высокого ранга выявлено отличие двух филогенетических схем, выраженное в разделении последовательностей на ML-дереве (рис. 1,а) на три массивные клады с четко выраженным расхождением олигохет и пиявок с многощетинковыми червями, выбранными нами в качестве внешней группы, тогда как при BI-анализе (рис. 1,б) последовательности разделились на две крупные клады, объединяющие представителей Oligochaeta и Hirudinea. При этом в ходе BI-анализа последовательности Polychaeta были отнесены в одну кладу с представителями Oligochaeta, что противоречит современной классификации [24].
Рис. 1.
ML-дерево (Log L= –4876.76) со значениями бутстреп-поддержек на 1000 репликах (а); BI-дерево со значениями байесовских апостериорных поддержек (б). Шкалы снизу соответствуют генетическим дистанциям.
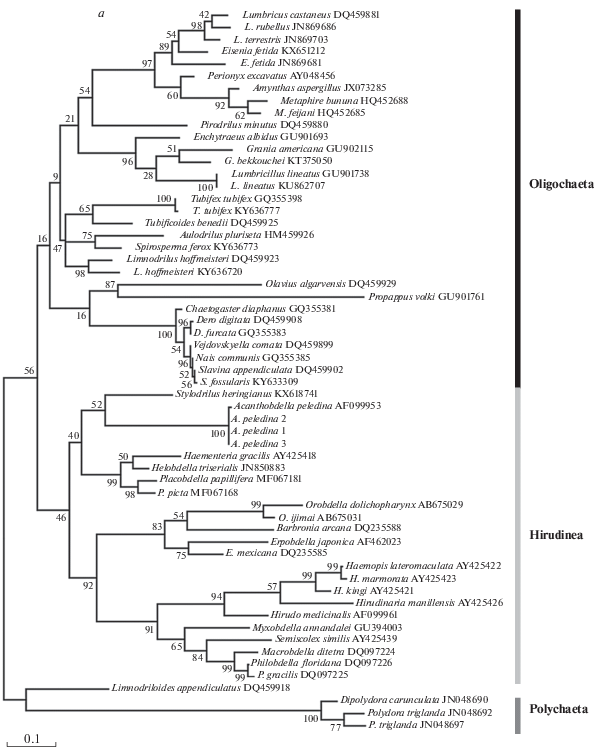
Рис. 1.
Окончание.
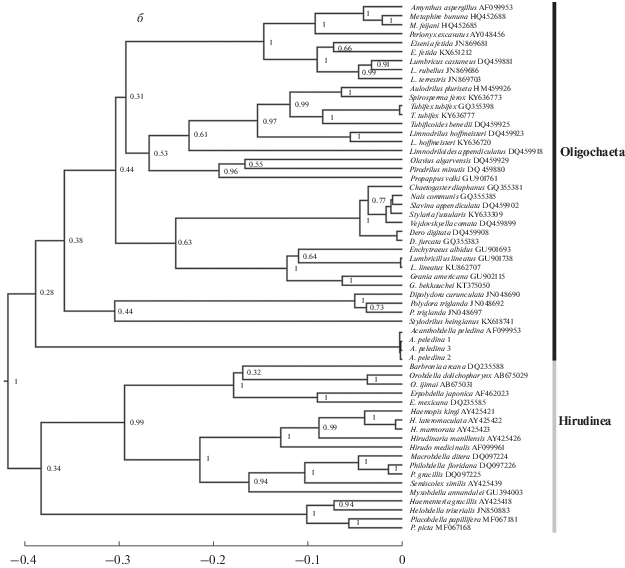
Последовательности вида A. peledina заняли разное положение при реконструкции филогении этими двумя методами. На ML-дереве последовательности акантобделлид расположились вместе с представителями подкласса Hirudinea. По характеру ветвления данного дерева можно сделать вывод, что род Acanthobdella является типичным представителем современных пиявок, что противоречит результатам морфологического анализа и ранних работ по молекулярной филогении. Байесовская реконструкция сгруппировала последовательности акантобделл в отдельную ветвь, более близкую к подклассу Oligochaeta.
Расположение филогенетической линии олигохеты Limnodriloides appendiculatus нестабильно и зависит от метода реконструкции.
Высокие статистические поддержки на уровне родов и видов подтверждают пригодность 12S в качестве маркерного фрагмента для анализа филогении на этом уровне, а также для видовой идентификации. В обоих деревьях наблюдается снижение значений статистической достоверности узлов по мере удаления от листьев к корню, что свидетельствует о большом количестве равновероятных сценариев эволюции и неспособности данного маркерного участка давать разрешенные узлы на высоких таксономических уровнях.
Известно, что бутстреп-поддержки оказываются зачастую ниже, чем байесовские апостериорные поддержки, поскольку при построении бутстреп-реплик каждый раз используется частичный набор данных из матрицы [25]. Несколько низких поддержек на уровне видов, наблюдаемых в байесовском дереве, вероятно, связаны с недостаточным таксономическим разнообразием в наборе данных.
Низкие статистические поддержки узлов у корневой части деревьев свидетельствуют о недостатке филогенетической информации в маркерном участке гена 12S рРНК для реконструкции древней эволюционной истории. Однако применение разных алгоритмов филогенетической реконструкции позволило успешно объединить последовательности в группы видового и родового уровней и одновременно выявило невозможность составить однозначную картину более ранней эволюции. Таким образом, несмотря на непригодность маркерного участка гена 12S рРНК для разрешения филогенетических отношений таксонов высокого порядка (семейств, отрядов и классов), этот фрагмент может быть с успехом использован для анализа филогении на видовом и родовом уровнях вследствие его высокой специфичности.
Авторы выражают благодарность Виктору Тараканову за помощь в сборе образцов.
Сбор образцов проводили в рамках Государственного задания 0345-2019-0002 (АААА-А16-116122110066-1) “Молекулярная экология и эволюция живых систем Центральной Азии в условиях глобальных экологических изменений”. Анализ образцов проводили в рамках гранта РФФИ № 17-29-05097.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Saccone C., De Giorgi C., Gissi C. et al. Evolutionary genomics in the Metazoa: the mitochondrial DNA as a model system // Gene. 1999. V. 238. P. 195–210.
Norris D.E., Klompen J.S.H., Black W.C. Comparison of the mitochondrial 12S and 16S ribosomal DNA genes in resolving phylogenetic relationships among hard ticks (Acari: Ixodidae) // Annals Entomol. Soc. America. 1999. V. 92(1). P. 117–129. https://doi.org/10.1093/aesa/92.1.117
Kaewkrajang N., Grootaert P., Boonrotpong S. Genetic variation of the long-legged flies phacaspis mitis complex (Diptera: Dolichopodidae) in peninsular Thailand inferred from three mitochondrial genes // J. Insect. Sci. 2018. V. 18(6): 6. https://doi.org/10.1093/jisesa/iey024
Mirzaei M., Ghahvei Y., Lefoulon E. et al. Morphological and molecular characterization of Onchocerca fasciata (Nematoda, Onchocercidae) from dromedary camels (Camelus dromedarius) in Iran // Parasite. 2018. V. 25: 50. https://doi.org/10.1051/parasite/2018045
Patwardhan A., Ray S., Roy A. Molecular markers in phylogenetic studies – a review // J. Phylogen. Evol. Biol. 2014. V. 2(2): 131. https://doi.org/10.4172/2329-9002.1000131
Trontelj P. Glossiphonia slovaca (Košel, 1973) (Hirudinea: Glossiphoniidae) iz Save pri Čatežu: nova vrsta pijavke za Slovenijo in vprašanje njene taksonomske pripadnosti // Natura Sloveniae. 2000. V. 2(2). P. 21–27.
Martin P. On the origin of the Hirudinea and the demise of the Oligochaeta // Proc. R. Soc. Lond. B. 2001. V. 268. P. 1089–1098. https://doi.org/10.1098/rspb.2001.1616
Erseus C., Kallersjo M. 18S rDNA phylogeny of Clitellata (Annelida) // Zool. Scripta. 2004. V. 33. P. 187–196. https://doi.org/10.1111/j.1463-6409.2004.00146.x
Minelli A., Sket B., Jong Y. Fauna Europaea: Annelida – Hirudinea, incl. Acanthobdellea and Branchiobdellea // Biodiversity Data J. 2014. V. 2. e4015. https://doi.org/10.3897/BDJ.2.e4015
Лукин Е.И. Пиявки пресных и солоноватых водоемов // Фауна СССР. Пиявки. Л.: Наука, 1976. С. 1–484.
Hauk A.K., Fallon M.J., Burger C.V. New host and geographical records for the leech Acanthobdella peledina Grube, 1851 (Hirudinea, Acanthobdellidae) // J. Parasitology. 1979. V. 65. № 6. P. 989. https://doi.org/10.2307/3280268
Holmquist C. A fish leech of the genus Acanthobdella found in North America // Hydrobiologia. 1974. V. 44(2–3). P. 241–245. https://doi.org/10.1007/BF00187273
Livanow N.A. Acanthobdella peledina Grube, 1851 // Morphol. Study. 1905. V. 72(5–8). P. 1–266.
Michaelsen W. Uber die Beziehungen der Hirudineen zu den Oligochaeten // Mitt. Zool. Mus. Hamburg. 1919. V. 36. P. 131–153.
Sawyer R.T. Leech Biology and Behaviour. Oxford: Clarendon Press, 1986. V. 2. 230 p.
Эпштейн В.М. Пиявки: Определитель паразитов пресноводных рыб фауны СССР / Под ред. Бауэр О. Л.: Наука, 1987. С. 340–372.
Siddal M.E., Apakupakul K., Burreson E.M. et al. Validating Livanow: molecular data agree that leeches, branchiobdellidans, and Acanthobdella peledina form a monophyletic group of oligochaetes // Mol. Phylogenet. Evol. 2001. V. 21(3). P. 346–351. https://doi.org/10.1006/mpev.2001.1021
Purschke G., Westheide W., Rohde D., Brinkhurst R.O. Morphological reinvestigation and phylogenetic relationship of Acanthobdella peledina (Annelida, Clitellata) // Zoomorphology. 1993. V. 113. P. 91–101. https://doi.org/10.1007/BF00403087
Trontelj P., Sket B., Steinbruk G. Molecular phylogeny of leeches: Congruence of nuclear and mitochondrial rDNA data sets and the origin of bloodsucking // J. Zool. Syst. Evol. Research. 1999. V. 37. P. 141–147. https://doi.org/10.1111/j.1439-0469.1999.00114.x
Hickson R.E., Simon C., Cooper A. et al. Conserved sequence motifs, alignment, and secondary structure for the third domain of animal 12S rRNA // Mol. Biol. Evol. 1996. V. 13(1). P. 150–169. https://doi.org/10.1093/oxfordjournals.molbev.a025552
Kumar S., Stecher G., Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33(7). P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Bouckaert R., Heled J., Kühnert D. et al. BEAST 2: a software platform for bayesian evolutionary analysis // PLoS Comput. Biol. 2014. V. 10(4): e1003537. https://doi.org/10.1371/journal.pcbi.1003537
Posada D. jModelTest: phylogenetic model averaging // Mol. Biol. Evol. 2008. V. 25(7). P. 1253–1256. https://doi.org/10.1093/molbev/msn083
Kojima S. Paraphyletic status of Polychaeta suggested by phylogenetic analysis based on the amino acid sequences of elongation factor-1α // Mol. Phylogenet. Evol. 1998. V. 9(2). P. 255–261. https://doi.org/10.1006/mpev.1997.0472
García-Sandoval R. Why some clades have low bootstrap frequencies and high Bayesian posterior probabilities // Israel J. Ecol. & Evol. 2014. V. 60(1). P. 41–44. https://doi.org/10.1080/15659801.2014.937900
Дополнительные материалы отсутствуют.


