Генетика, 2019, T. 55, № 2, стр. 165-173
Оценка генетического полиморфизма сливы домашней на основе анализа микросателлитных локусов
И. И. Супрун 1, *, И. В. Степанов 1, С. В. Токмаков 1, Г. В. Еремин 2
1 Северо-Кавказский федеральный научный центр садоводства, виноградарства, виноделия
350901 Краснодар, Россия
2 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова, Крымская опытно-селекционная станция
353384 Краснодарский край, Крымск, Россия
* E-mail: supruni@mail.ru
Поступила в редакцию 14.02.2018
После доработки 04.07.2018
Принята к публикации 06.06.2018
Аннотация
Проведено исследование внутривидового генетического разнообразия и структуры вида слива домашняя на основе анализа 60 сортов по восьми SSR-локусам. В выборку изучаемых образцов вошли различные подвиды сливы домашней: мирабели, ренклоды, терносливы, венгерки. Выявленное в ходе исследования число аллелей на локус варьировало от 6 до 39, всего было идентифицировано 153 аллеля. Байесовский анализ позволил выявить четыре гипотетические панмиктические популяции. При этом вклад панмиктических популяций специфичен для каждого подвида сливы домашней. Основываясь на результатах кластеризации методом UPGMA и данных PCoA, можно заключить, что наиболее генетически обособленным подвидом сливы домашней являются терносливы. Наряду с этим большинство сортов отечественной и зарубежной селекции не образуют выраженных групп при кластеризации, это свидетельствует о единстве генофонда культурных форм сливы домашней.
Немалая доля представителей рода Prunus имеет хозяйственную ценность и выращивается в качестве плодовых и декоративных растений. В странах с умеренным климатом широко распространены настоящие сливы (P. domestica subsp. domestica), терносливы (P. domestica subsp. insitita), ренклоды (P. domestica subsp. italiaca) и мирабели (P. domestica subsp. syriaca), которые объединяются в один вид – слива домашняя (Prunus domestica L.). Согласно современной таксономии слива домашняя относится к подроду Prunophora рода Prunus, семейства Rosaceae. Этот вид предположительно возник путем спонтанной гибридизации между терном (P. spinosa) и алычой (P. cerasifera) и является гексаплоидным [1, 2].
В процессе накопления научных знаний, в частности по цитогенетике, возникает некоторое количество альтернативных точек зрения по вопросу видообразования и происхождения сливы домашней. Так, Zohary предполагает аутоплоидное происхождение сливы домашней от диплоидной и тетраплоидной алычи, основываясь на отсутствии у мирабели, ренклодов и тернослив признаков терна. Однако это утверждение является весьма спорным [3–5].
Нерешенным остается вопрос о статусе терносливы, которая согласно одной из точек зрения заслуживает выделения в отдельный вид [6]. Данная точка зрения базируется на существовании устойчивых переходных форм между терном и терносливой. Однако большинство исследователей считают терносливу подвидом сливы домашней.
Появление и развитие методов молекулярной биологии позволяет по-новому взглянуть на вопросы, связанные с систематикой и филогенией вида Prunus domestica. На данный момент существует ряд работ, связанных с изучением филогении и систематики рода Prunus с использованием методов молекулярной генетики. Самые ранние работы по сливе домашней использовали мультилокусные маркеры RFLP [7] и RAPD [8].
В сравнении с другими культурами рода Prunus, такими как персик, черешня, абрикос и миндаль [9–11], в исследованиях вида слива домашняя было задействовано меньшее число SSR-маркеров. Первое исследование с использованием микросателлитных маркеров было проведено на пяти балканских сортах и 10 наиболее известных сортах мировой селекции. Десять микросателлитов, использованных в данной работе, были выделены из ядерного и хлоропластного геномов абрикоса [12]. Это исследование послужило началом изучения вида с помощью микросателлитов, определив перспективность их для изучения генофонда сливы домашней. В дальнейшем число сортов, использованных в работе, возрастало. Так, в генетическом исследовании трех видов P. domestica, P. cerasifera и P. spinosa были проанализированы 80 генотипов с помощью SSR-маркеров различного происхождения [13].
В 2014 г. опубликована работа, в которой микросателлитные маркеры были успешно применены в филогенетической оценке 24 генотипов в рамках подвида P. domestica subsp. italic [14]. В том же году осуществлен анализ сортов сливы домашней, возделываемых в Белоруссии, по 20 SSR-маркерам [15].
Также были проведены две крупные исследовательские работы на генофонде балканских и скандинавских сортов сливы домашней. В исследовании, проведенном на балканской генплазме, были изучены 62 образца, 42 из которых являются традиционными хорватскими сортами [16].
В свою очередь, в общей сложности 76 генотипов сливы домашней, включая норвежские и шведские сорта народной селекции, изучены с применением девяти SSR-маркеров [17].
В 2016 г. нами была проделана работа по предварительной генетической оценке сортов из коллекций Майкопской опытной станции и Крымской опытно-селекционной станции ВИР по восьми SSR-маркерам [18, 19].
В связи с тем, что среди большого числа микросателлитных маркеров, разработанных на видах рода Prunus, апробированные на сливе домашней составляют лишь малую часть, актуальным направлением видится проведение работ по апробации и поиску новых эффективных SSR-маркеров для P. domestica. Анализ результатов генотипирования большой выборки сортов сливы домашней может пролить свет на ряд вопросов, связанных с систематикой и внутривидовой структурой.
Целью настоящей работы послужил микросателлитный анализ обширной группы сортов, включающей различные подвиды и сортотипы сливы домашней.
МАТЕРИАЛЫ И МЕТОДЫ
В качестве объектов исследования были взяты сорта сливы домашней из коллекций Майкопской опытной станции (МОС) и Крымской опытно-селекционной станции (КОСС) Всероссийского института растениеводства им. Н.И. Вавалова (ВИР). В выбороку из 60 сортов вошли два сорта из группы мирабелей, семь ренклодов, девять тернослив, десять стародавних сортов, 32 сорта из группы венгерок (табл. 1).
Таблица 1.
Cорта сливы домашней, отобранные для исследования
| Номер п/п | Сорт | Подвид | Отбор из коллекции |
|---|---|---|---|
| 1 | Венгерка ВИРа | P. domestica subsp. domestica | МОС ВИР |
| 2 | Венгерка местная | То же | » |
| 3 | Венгерка майкопская | » | » |
| 4 | Венгерка майкопчанка | » | » |
| 5 | Венгерка Шунтукская | » | » |
| 6 | Венгерка Шунтучка | » | » |
| 7 | Мирабель маленькая | P. domestica subsp. syriaca | » |
| 8 | Слива местная | Сорт народной селекции | » |
| 9 | Исполинская | P. domestica subsp. domestica | КОСС ВИР |
| 10 | Персиковая | P. domestica subsp. italiaca | » |
| 11 | Астрахань 2 | P. domestica subsp. insitita | » |
| 12 | Гонгоша | То же | » |
| 13 | Президент | P. domestica subsp. domestica | » |
| 14 | Милена | То же | » |
| 15 | Скороспелка красная | Сорт народной селекции | » |
| 16 | Дебют | P. domestica subsp. domestica | » |
| 17 | Венгерка кавказская | То же | » |
| 18 | Стенлей | » | » |
| 19 | Баллада | » | » |
| 20 | Синяя птица | » | » |
| 21 | Кубанский карлик | » | » |
| 22 | Кабардинская ранняя | » | » |
| 23 | Кубанская легенда | » | » |
| 24 | Кубанская ранняя | » | » |
| 25 | Большой приз | » | » |
| 26 | Анна Шпет | » | » |
| 27 | Синичка | » | » |
| 28 | Ренклод Альтана | P. domestica subsp. italiaca | » |
| 29 | Ренклод зеленый | То же | » |
| 30 | Керасий кислая | P. domestica subsp. insitita | » |
| 31 | Бромптон | Сорт народной селекции | » |
| 32 | Екатерина | P. domestica subsp. domestica | МОС ВИР |
| 33 | Краснодарская ранняя | То же | КОСС ВИР |
| 34 | Ренклод Яна | P. domestica subsp. italiaca | » |
| 35 | Мирабель крупная | P. domestica subsp. syriaca | МОС ВИР |
| 36 | Ренклод фиолетовый | P. domestica subsp. italiaca | » |
| 37 | Метелка крупноплодная | Сорт народной селекции | » |
| 38 | Изюм Эрика крупноплодный | То же | » |
| 39 | Крымзы Эрика | » | » |
| 40 | Тернослива донецкая | P. domestica subsp. insitita | КОСС ВИР |
| 41 | Тернослив Цареградский | То же | » |
| 42 | Тернослив Соляновский | » | » |
| 43 | Ренклод Браги | P. domestica subsp. italiaca | » |
| 44 | Тернослив садовый | P. domestica subsp. insitita | » |
| 45 | Сочинская юбилейная | P. domestica subsp. domestica | » |
| 46 | Джефферсон | То же | » |
| 47 | Тернослив Абинский | P. domestica subsp. insitita | » |
| 48 | Изюм Эрика | Сорт народной селекции | » |
| 49 | Венгерка Ажанская | P. domestica subsp. domestica | » |
| 50 | Чачакская ранняя | То же | » |
| 51 | Венгерка обыкновенная | » | » |
| 52 | Начанская | » | » |
| 53 | Дмонисий 10 | Сорт народной селекции | » |
| 54 | Соперница | P. domestica subsp. domestica | » |
| 55 | Ренклод Дизи | P. domestica subsp. italiaca | » |
| 56 | Лазоватский тернослив | P. domestica subsp. insitita | » |
| 57 | Венгерка венская | P. domestica subsp. domestica | » |
| 58 | Зайнаб | Сорт народной селекции | » |
| 59 | Венгерка итальянская | P. domestica subsp. domestica | » |
| 60 | Очаковская желтая | Сорт народной селекции | » |
Экстракцию ДНК проводили из молодых листьев с использованием метода ЦТАБ с модификациями [20].
Для генотипирования были отобраны восемь SSR-маркеров: для шести микросателлитных локусов характерны длинные нуклеотидные мотивы повторов RPPG1-017, RPPG1-032, RPPG3-026, RPPG4-059, RPPG2-011, RPPG1-037 [21]; остальные два локуса BPPCT007 [11] и CPSCT004 [22] обладают динуклеотидными мотивами повторов. Все восемь SSR ранее были апробированы на четырех генотипах сливы домашней [23].
ПЦР проводили с выполнением предварительной оптимизации ряда параметров, таких как температура отжига праймеров, длительность циклов отжига праймеров и элонгации, общее число циклов, концентрация дезоксинуклеотидтрифосфатов, праймеров.
В состав ПЦР смеси общим объемом 25 мкл входили 50 нг ДНК, 0.25 мМ dNTPs, 0.2 мкМ каждого праймера, 2.5 мкл 10-кратного реакционного буфера (ООО “Сибэнзим”), 1 ед. Taq-полимеразы. ПЦР-программу проводили по следующей схеме: 3 мин при 94°С – начальная денатурация; следующие 35 циклов: 45 с денатурации при 94°С, 45 с отжига праймеров при 58°С, 45 с синтеза при 72°С; последний цикл синтеза 4 мин 30 с при 72°С.
Анализ размеров амплифицированных фрагментов проводили на автоматическом генетическом анализаторе ABI prism 3130. Обработку данных осуществляли в программе Gene Mapper 4.1. Для статистической обработки результатов SSR-генотипирования и анализа генетических взаимосвязей изученного генофонда использовали пакеты программ PAST version 2.17c. Для оценки генетической структуры выборки использовалась программа Structure 2.3.4. В расчете были использованы различные значения гипотетических популяций от K = 2 до K = 8 (burn-in period = 200 000; 500 000 iterations). Оптимальные значения K были установлены с помощью онлайн программы Structure Harvester (http://taylor0.biology.ucla.edu/ structureHarvester/), основанной на принципе Evanno method.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Для использованных в работе SSR-маркеров на выборке из 60 генотипов сливы домашней число установленных аллелей на локус варьировало в значениях от 6 до 39. Наиболее высокополиморфными маркерами оказались RPPG3-026 (39 аллелей) и RPPG2-011 (34 аллеля). SSR-локус CPSCT004 обладал наименьшим в наборе маркеров полиморфизмом (6 аллелей). В среднем у отобранных для работы микросателлитных маркеров было обнаружено 19.13 аллелей на локус, что сопоставимо с данными, полученными при изучении балканской (17.15 аллелей на локус) и скандинавской (18.7 аллелей на локус) генплазмы [16, 17]. В табл. 2 указана характеристика маркеров, полученная в ходе генотипирования 60 сортов: диапазон размеров аллелей маркеров и число аллелей, выявленных в изученной выборке сортов. Таким образом, совокупно по всем использованным в работе маркерам было получено 153 полиморфных аллеля. По маркерам BPPCT007 и RPPG2-011 уникальным аллельным набором обладали 59 генотипов из исследованной выборки. В свою очередь по маркеру CPSCT004 было установлено лишь девять сортов, обладающих уникальным набором аллелей. Для остальных SSR-маркеров были характерны промежуточные значения. В целом с использованием восьми маркеров получены уникальные наборы аллелей по всем сортам. Общее число аллелей по всем маркерам достаточно для проведения генетической оценки коллекции из 60 генотипов сливы домашней.
Таблица 2.
Характеристика SSR-маркеров, отобранных для генетического анализа сортов сливы домашней
| Маркер | Число аллелей | Диапазон размеров аллелей, пн | Число уникальных генотипов |
|---|---|---|---|
| RPPG1-017 | 10 | 167–183 | 24 |
| RPPG1-032 | 11 | 186–236 | 15 |
| RPPG3-026 | 39 | 183–335 | 55 |
| RPPG4-059 | 7 | 150–171 | 10 |
| RPPG2-011 | 34 | 164–231 | 59 |
| RPPG1-037 | 19 | 222–253 | 43 |
| CPSCT004 | 6 | 123–129 | 9 |
| BPPCT007 | 27 | 126–173 | 59 |
| Среднее значение | 19.13 | ||
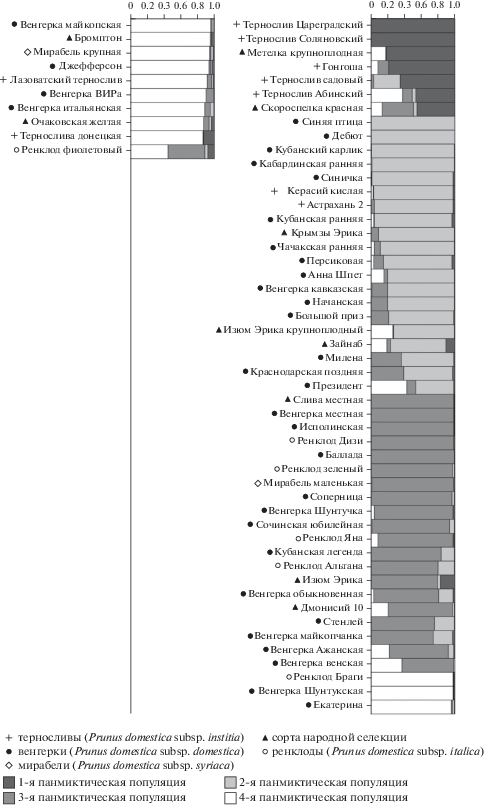
Байесовский анализ был проведен на 60 генотипах сливы домашней с использованием восьми SSR-маркеров. Применение Evanno method позволило выявить оптимальное значение K, равное 4 (рис. 1). Первая реконструированная панмиктическая популяция присутствует в соотношении от 50–100% у семи генотипов, пять из которых являются терносливами, а два – традиционными русскими сортами. У подавляющего большинства сортов вклад данной популяции не выявлен. Вторая реконструированная панмиктическая популяция широко представлена у венгерок отечественной и зарубежной селекции, а также тернослив (Керасий кислая, Астрахань 2). Третья популяция преобладает у представленных в выборке ренклодов (за исключением Ренклода Браги и Персиковой), а также встречается у ряда сортов венгерок и сортов народной селекции. Четвертая популяция выявлена у всех групп сортов включая венгерки, ренклоды и терносливы (рис. 1). Также следует отметить, что если 2-я и 4-я популяции представлены у всех подвидов, то первая популяция характерна для терносливов и двух сортов народной селекции. Значительный для большинства ренклодов вклад популяции 3 отсутствует у терносливов. Таким образом, можно судить о наличии в генетической структуре тернослив редкого генетического компонента, отсутствующего у большинства слив за исключением нескольких сортов народной селекции. В наиболее обширных исследованиях, посвященных SSR-генотипированию генофонда сливы домашней, терносливы сформированы отдельной предковой популяцией [13, 16]. Использованные в работе два генотипа мирабели происходят от двух различных гипотетических популяций, что согласуется с данными хорватских и скандинавских исследователей [16, 17] и контрастирует с результатами Horvath et al., по которым мирабели были сформированы одной популяцией [13].
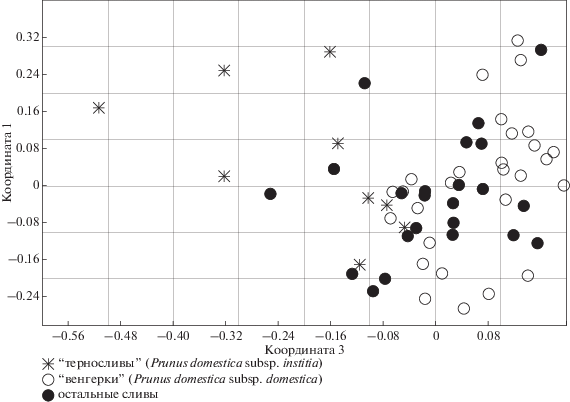
Также можно отметить тенденцию различных генотипов тернослив к образованию внешних относительно основной выборки сортов кластеров при проведении PCoA (метода главных координат) с применением программы Past. Результаты анализа отображены на рис. 2.
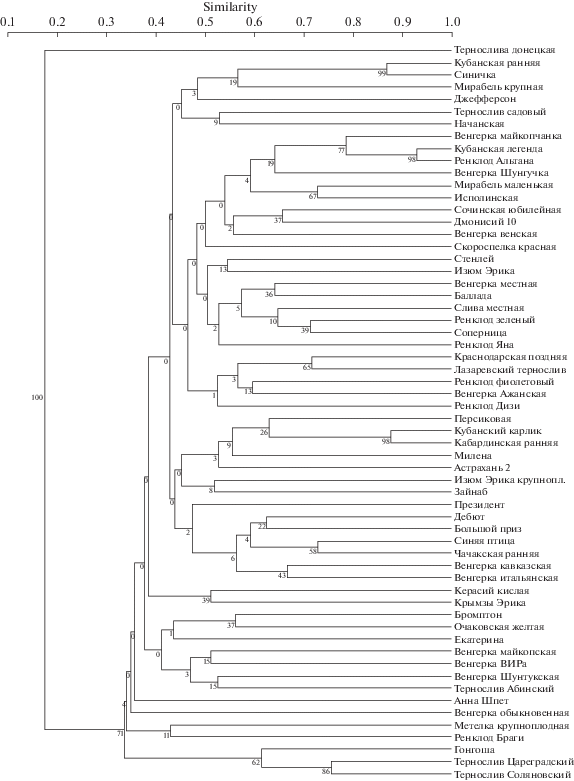
Для исследуемых генотипов сливы домашней был проведен кластерный анализ на основе полученных с помощью SSR-генотипирования данных. Кластеризацию проводили методом UPGMA c применением коэффициента Dice. Дендрограмма, построенная по результатам кластеризации отобранных для исследования генотипов, отображена на рис. 3.
Наиболее обособленные от общей выборки генотипы были представлены терносливами: Тернослива донецкая, Тернослив Соляновский, Тернослив Цареградский, Гонгоша. При этом Тернослива донецкая является самым отдаленным генотипом. Однако несколько тернослив не выделяются из общего для большинства слив кластера.
Следует отметить, что зарубежные и отечественные сорта не формируют отдельных друг от друга групп, а представлены в работе единым цельным кластером, обособленным только от большей части терносливов, это свидетельствует об общности происхождения российских и европейских сортов сливы домашней.
Примечательно, что отобранные в работе ренклоды входят в различные кластеры, в которых превалирует сортотип венгерки. Отдельных групп не образуют и крымские автохтонные сорта Изюм Эрика, Крымзы Эрика и Изюм Эрика крупноплодный. Сорт, представляющий в выборке сортотип мирабели, Мирабель крупная, отнесен к кластеру генотипов различного происхождения, из чего следует предположение о генетической близости сортов, относящихся к разным подвидам/сортотипам. Данную генетическую близость, отраженную методом кластеризации, можно объяснить распространенной практикой скрещивания сортотипов между собой.
Данные кластеризации свидетельствуют о генетической удаленности большинства терносливов от основной массы сортов. В связи с этим можно предположить, что терносливы в виду своих морфологических особенностей (размер плода, терпкий вкус и наличие колючек на ветвях) не участвовали в создании большей части сортимента востребованных сортов сливы домашней. Так как обособление генотипов терносливы неполное, не затрагивающее часть образцов, нельзя исключать генетическую связь локальных форм терносливы с возделываемыми культурными сортами сливы домашней.
Исследование выполнено при поддержке Российского фонда фундаментальных исследований – проект № 16-44-230260 р_а и Госзадания (тема № 0689-2018-0003).
Список литературы
Maynard C.K., Havanagh K., Fuernkranz H. Draw Black cherry (Prunus serotina Ehrh.) // Biotechnol. Agr. For. 1991. V. 16. P. 3–22.
Crane M.B., Lawrence W.J.C. Studies in sterility Intern. Hort. Congress. 1930. № 9. P. 100–116.
Salesses G. Some data on the cytogenetics of plums and the origin of plums // Acta Horticulturae. 1975. V. 1. № 48. P. 59–65.
Bajashvili E.I. Studies of some species of Prunus Mill. genus // Acta Horticulturae. 1990. № 283. P. 31–34. doi 10.17660/ActaHortic.1990.283.3
Zohary D. Is the European plum, Prunus domestica L., a P. cerasifera EHRH × P. spinosa L. allopolyploid // Euphytica. 1992. V. 60. P. 75–77. doi 10.1007/BF00022260
Woldring H. On the origin of plums: a study of sloe, damson, cherry plum, domestic plums and their intermediates // Palaeohistoria. 2000. № 39/40. P. 535–562.
Badenes M.L., Parfitt D.E. Phylogenetic relationships of cultivated Prunus species from an analysis of chloroplast DNA variation // Theor. Appl. Genet. 1995. V. 90. P. 1035–1041. doi 10.1007/BF00222918
Casas A.M., Igartua E., Balaguer G., Moreno M.A. Genetic diversity of Prunus rootstocks analysed by RAPD markers // Euphytica. 1999. V. 110. P. 139–149.
Downey S.L., Iezzoni A.F. Polymorphic DNA markers in black cherry Prunus serotina are identified using sequences from sweet cherry, peach and sour // J. Am. Soc. Hort. Sci. 2000. V. 125. № 1. P. 76–80.
Sosinski B., Gannavarapu M., Hager L.D. et al. Characterisation of microsatellite markers in peach Prunus persica (L.) Batsch. // Theor. Appl. Genet. 2000. V. 101. P. 421–428. doi 10.1007/s001220051499
Dirlewanger E., Cosson P., Tavaud M. et al. Development of microsatellite markers in peach Prunus persica (L.) Batsch. and their use in genetic diversity analysis in peach and sweet cherry Prunus avium L. // Theor. Appl. Genet. 2002. V. 105. № 1. P. 127–138. doi 10.1007/ s00122-002-0867-7
Decroocq V., Hagen L.S., Fave M.-G. et al. Microsatellite markers in the hexaploid Prunus domestica species and parentage lineage of three European plum cultivars using nuclear and chloroplast simplesequence repeats // Mol. Breed. 2004. V. 13. № 2. P. 135–142. doi 10.1023/ B:MOLB.0000018761.04559.b3
Horvath A., Balsemin E., Barbot J.-C. et al. Phenotypic variability and genetic structure in plum (Prunus domestica L.), cherry plum (P. cerasifera Ehrh.) and sloe (P. spinosa L.) // Sci. Horticulturae. 2011. V. 129. № 2. P. 283–293.
Gharbi O., Wünsch A., Rodrigo J. Characterization of accessions of ‘Reine Claude Verte’ plumusing Prunus SRR and phenotypictraits // Sci. Horticulturae. 2014. V. 169. P. 57–65. doi 10.1016/j.scienta.2011.03.049
Урбанович О.Ю., Кузмицкая П.В., Козловская З.А. Исследование генетического разнообразия сортов слив с помощью молекулярных маркеров SSR-типа // Докл. Национальной академии наук Беларуси. 2014. № 58(5). С. 92–97.
Kazija D.H., Jelačić T., Vujević P. Plum germplasm in Croatia and neighboring countries assessed by microsatellites and DUS descriptors // Tree Genetics & Genomes. 2014. V. 10. № 3. P. 761–778.
Sehic J., Nybom H., Hjeltnes S.H. Genetic diversity and structure of Nordic plum germplasm preserved ex situ and on-farm // Sci. Horticulturae. 2015. № 190. P. 195–202.
Степанов И.В., Супрун И.И., Лободина Е.В. Анализ SSR-полиморфизма Северо-Кавказских сортов сливы домашней // Науч. тр. СКЗНИИСиВ. 2016. Т. 9. С. 78–84.
Степанов И.В., Супрун И.И., Токмаков С.В. Оценка генетического разнообразия сортов сливы домашней селекции МОС ВИР с использованием SSR-маркеров // Междун. саммит молодых ученых: Материалы конф. Краснодар, 2016. С. 193–197.
Murray M.G., Thompson W.F. Rapid isolation of high molecular weight plant DNA // Nucl. Acids Res. 1980. V. 8(19). P. 4321–4325.
Dettori M.T., Micali S., Giovinazzi J. Mining microsatellites in the peach genome: development of new long‑core SSR markers for genetic analyses in five Prunus species // Springer Plus. 2015. V. 4. P. 337–340.
Mnejja M., Garcia-Mas J., Howad W. et al. Simple-sequece repeat (SSR) markers of Japanese plum (Prunus salicina Lindl.) are highly polymorphic and transferable to peach and almond // Mol. Ecol. Notes. 2004. V. 4(2). P. 163–166.
Супрун И.И., Степанов И.В., Токмаков С.В. Апробация SSR-маркеров вида Prunus persica для генотипирования сливы домашней // Науч. журн. КубГАУ. 2016. № 124(10).
Дополнительные материалы отсутствуют.