Генетика, 2019, T. 55, № 2, стр. 223-228
Характеристика генов-гомологов INNER NO OUTER у дикорастущих видов томата
М. А. Филюшин 1, *, М. А. Слугина 1, 2, Е. З. Кочиева 1, 2, А. В. Щенникова 1
1 Федеральный исследовательский центр “Фундаментальные основы биотехнологии”
Российской академии наук
119071 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: michel7753@mail.ru
Поступила в редакцию 02.02.2018
После доработки 03.04.2018
Принята к публикации 26.03.2018
Аннотация
Ген INNER NO OUTER принадлежит семейству факторов транскрипции YABBY и определяет идентичность абаксиального слоя клеток внешнего интегумента семязачатка. В настоящем исследовании идентифицированы и охарактеризованы полногеномные последовательности INO у 12 образцов 11 видов томата секции Lycopersicon. В белках INO всех анализируемых видов томата были идентифицированы консервативные домены цинкового пальца и YABBY. В последовательностях генов INO видов томата выявлено 243 вариабельных сайта (12.9% от выровненной длины), из которых 43 локализованы в экзонах, включая SNPs, приводящие к замещениям аминокислотных остатков в домене цинкового пальца (10), YABBY-домене (2) и внедоменной области (10). У INO S. arcanum была выявлена делеция 10 а.о. в С-концевой внедоменной области.
При всем многообразии морфологии цветок покрытосеменных растений имеет консервативную структуру, в том числе строение репродуктивных органов, функция которых – производство семени, несущего зародыш нового растения. Семя развивается из семязачатка, основные части которого – нуцеллус, окруженный одним или двумя интегументами (покровами), и семяножка [1]. Считается, что сходство организации семязачатка является результатом консервативного механизма регуляции пространственно-временной экспрессии гомологичных генов. Так, на развитие нуцеллуса существенное влияние оказывают факторы транскрипции семейств MADS-box и KNOX, а онтогенез внешнего интегумента невозможен без активности фактора транскрипции INNER NO OUTER (INO) семейства YABBY [2, 3].
Семейство генов YABBY, специфичное для растений, появилось в результате эволюционной дупликации гена-предшественника с последующей диверсификацией гомологичных генов, основная роль которых – контроль полярного развития латеральных органов. Эволюция этих генов считается одной из причин возникновения плоского листа и, позднее, цветковых органов. Наиболее полно гены YABBY изучены у модельного растения Arabidopsis thaliana [4–6]. В семействе YABBY различают “вегетативные” гены YABBY1, YABBY2, YABBY3 и YABBY5, определяющие абаксиально-адаксиальную асимметрию всех надземных листоподобных органов, и два “генеративных” гена INO и CRABS-CLAW (CRC), которые в процессе эволюции приобрели узкую специализацию. Так, INO определяет идентичность абаксиального (наружного) слоя клеток внешнего покрова семязачатка [1, 7, 8], а CRC участвует в регуляции развития завязи и нектарников [4, 9, 10].
Данные о полиморфизме генов-гомологов INO могут быть информативны в свете изучения формирования и развития семян, поскольку внешний интегумент необходим для определения положения микропиле и для последующего образования семенной кожуры, что непосредственно влияет на жизнеспособность семян. Также стоит отметить, что сам интегумент как эволюционное образование появился с возникновением семян и, соответственно, свойствен только семенным растениям. Как правило, растительный геном содержит одну копию гена INO, у некоторых видов выявлено несколько изоформ INO, что является следствием альтернативного сплайсинга [11, 12]. Как и растительные факторы транскрипции других семейств у двудольных растений, белки YABBY обладают высокой структурно-функциональной гомологией. Белок INO, как и другие члены семейства YABBY, содержит на С-конце сходный с HMG-боксом YABBY-домен, а на N-конце – домен цинкового пальца Cys2Cys2-типа [4].
Определено, что асимметричный рост интегумента семязачатка A. thaliana опосредован взаимодействием INO с SUPERMAN [5, 13], а также с ко-активатором ADA2b/PRZ1 и ко-репрессорами LEUNIG и SEUSS [3]. Мутации в гене или промоторе INO вызывают аномалии развития семязачатка вплоть до его отсутствия [5, 8]. Учитывая степень участия INO в развитии семязачатка, можно предположить связь полиморфизма генов/белков INO с числом интегументов.
Известно, что унитегмальный семязачаток как более недавний эволюционный признак характерен для всех представителей Asterids, к которому принадлежит семейство Solanaceae, включающее важные овощные культуры перец (род Capsicum), картофель (секция Petota род Solanum) и томат (секция Lycopersicon род Solanum). Секция Lycopersicon включает в себя культивируемый вид томат овощной Solanum lycopersicum и 12 дикорастущих видов [14, 15]. К настоящему времени ген INO охарактеризован только у S. lycopersicum [16, 17] и S. pimpinellifolium [18].
В настоящей работе были идентифицированы и охарактеризованы полногеномные последовательности INO 12 образцов 11 видов томата (табл. 1). ДНК выделяли из свежих листьев согласно [19]. На основе последовательности INO S. lycopersicum cv. Heinz 1706 (GeneID:101244955) были разработаны специфичные праймеры для амплификации полных последовательностей генов-гомологов INO у исследуемых видов томата – INOF (5'-AYTCTAGATYCCTCTCTCAC-3') и INOR (5'-TTGACAATRGCATCTTYAACAC-3'). Также для секвенирования были разработаны внутренние праймеры, специфичные к последовательностям четвертого (INOex4F; 5'-AGAGAGCTCCWTCKGCTTA-3') и пятого (INOex5R; 5'-AAGCTTGCTTGTGAGTCATG-3') экзонов. ПЦР-фрагменты клонировали в pGEMT-easy (Promega, Madison, CША) и секвенировали (по 4 клона) на платформе Applied Biosystems 3730 DNA Analyzer (ЦКП “Биоинженерия”). Структурно-филогенетический анализ проводили в программе MEGA 7.0. В качестве референсной использовали последовательность SlINO S. lycopersicum cv. Heinz 1706. Консервативные домены определяли с помощью программы NCBI-CDD (http://www.ncbi.nlm.nih.gov/Structure/cdd/ wrpsb.cgi) и литературных данных [16]. Влияние аминокислотных замен на структуру и функции белков предсказывали с помощью программы PROVEAN (http://provean.jcvi.org).
Таблица 1.
Характеристика экзон-интронной структуры гена INO у исследуемых видов Solanum
| Вид/сорт (кат. № ВИР) |
Номер доступа в NCBI | Экзон-интронная структура | Ген, пн | кДНК, пн | Белок, а.о. | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| экзон I | интрон I | экзон II | интрон II | экзон III | интрон III | экзон IV | интрон IV | экзон V | интрон V | экзон VI | интрон VI | экзон VII | |||||
| S. arcanum (13958) | MG873878 | 84 | 96 | 132 | 198 | 115 | 209 | 49 | 111 | 76 | 135 | 36 | 481 | 57 | 1779 | 549 | 182 |
| S. cheesmaniae (3969) | MG873879 | 84 | 86 | 132 | 204 | 115 | 209 | 49 | 125 | 76 | 134 | 66 | 450 | 57 | 1787 | 579 | 192 |
| S. chilense (4300) | MG873880 | 84 | 96 | 132 | 197 | 115 | 186 | 49 | 115 | 76 | 133 | 66 | 469 | 57 | 1775 | 579 | 192 |
| S. chmielewskii (13725) | MG873881 | 84 | 86 | 132 | 204 | 115 | 210 | 49 | 124 | 76 | 134 | 66 | 450 | 57 | 1787 | 579 | 192 |
| S. corneliomulleri (4367) | MG873882 | 84 | 97 | 132 | 198 | 115 | 185 | 49 | 113 | 76 | 135 | 66 | 490 | 57 | 1797 | 579 | 192 |
| S. galapagense (3970) | MG873883 | 84 | 86 | 132 | 204 | 115 | 209 | 49 | 125 | 76 | 134 | 66 | 450 | 57 | 1787 | 579 | 192 |
| S. habrochaites (13964) | MG873884 | 84 | 96 | 129 | 197 | 115 | 197 | 49 | 110 | 76 | 135 | 66 | 476 | 57 | 1787 | 576 | 191 |
| S. lycopersicum cv. Silvestre recordo (1580) | MG873885 | 84 | 86 | 132 | 204 | 115 | 208 | 49 | 125 | 76 | 134 | 66 | 450 | 57 | 1786 | 579 | 192 |
| S. neorickii (5033) | MG873886 | 84 | 96 | 132 | 200 | 115 | 200 | 49 | 128 | 76 | 139 | 66 | 449 | 57 | 1791 | 579 | 192 |
| S. peruvianum (4361) | MG873887 | 84 | 96 | 132 | 197 | 115 | 208 | 49 | 112 | 76 | 135 | 66 | 469 | 57 | 1796 | 579 | 192 |
| S. peruvianum var. dentatum (3966) | MG873888 | 84 | 97 | 132 | 197 | 115 | 217 | 49 | 126 | 76 | 135 | 66 | 476 | 57 | 1827 | 579 | 192 |
| S. pimpinellifolium var. racemigerum (1018) | MG873889 | 84 | 86 | 132 | 204 | 115 | 209 | 49 | 125 | 76 | 135 | 66 | 450 | 57 | 1788 | 579 | 192 |
| S. lycopersicum cv. Heinz 1706* | 101244955 | 84 | 86 | 132 | 204 | 115 | 209 | 49 | 125 | 76 | 134 | 66 | 450 | 57 | 1787 | 579 | 192 |
| S. pennellii* | 107019174 | 84 | 97 | 132 | 192 | 121 | 197 | 49 | 124 | 76 | 135 | 66 | 477 | 57 | 1807 | 585 | 194 |
| S. tuberosum* | 102594693 | 84 | 110 | 132 | 198 | 136 | 282 | 49 | 119 | 76 | 278 | 66 | 652 | 57 | 2239 | 600 | 199 |
Полные последовательности гена INO были амплифицированы, клонированы и секвенированы у всех 12 анализируемых образцов томата. Общая длина гена, включающего семь экзонов, варьировала от 1775 пн у S. chilense до 1827 пн у S. peruvianum var. dentatum (табл. 1), в основном за счет коротких инделей в интронах. Определенные генетические расстояния между исследуемыми видами томата были невелики и составляли от 0.00 (S. lycopersicum cv. Silvestre recordo – S. cheesmaniae, S. pimpinellifolium) до 0.05 (S. peruvianum var. dentatum – S. cheesmaniae, S. galapagense, S. chmielewskii). Однако они были больше генетических расстояний между последовательностями гена INO (0.00–0.03) у видов рода Capsicum [20].
Размер кДНК INO составил 579 пн, за исключением INO S. arcanum (549 пн; делеция 30 пн в экзоне VI) и S. habrochaites (576 пн; делеция 3 пн в экзоне II) (табл. 1). У S. pennellii, который считается наиболее древним видом томата [14], размер кДНК INO составляет 585 пн. У другого представителя рода Solanum, картофеля S. tuberosum, размер кДНК INO составляет 600 пн благодаря вставке 24 пн и делеции 3 пн в экзоне III (табл. 1).
Белки INO имели размер192 а.о., за исключением INO S. arcanum (182 а.о.) и S. habrochaites (191 а.о.). Для сравнения INO видов Capsicum состоит из 215 а.о. [20], а INO A. thaliana – из 262 а.о., что предполагает существенную разницу длин междоменных участков белка INO. На основании этого можно было бы связать размер междоменных областей INO с числом покровов семязачатка. Однако у видов Prunus, как уни-, так и битегмальных, INO имеет размер около 220 а.о. [21]. К тому же было показано отсутствие связи между уни- или битегмальной структурой семязачатка и экспрессией гена INO [1, 21].
В белках INO всех анализируемых видов томата были идентифицированы консервативные домены цинкового пальца (12–52 а.о.) и YABBY (108–163 а.о.) (рис. 1,а).
Рис. 1.
Сравнение аминокислотных последовательностей ортологов INO у исследуемых образцов томата с известными белками INO томата овощного S. lycopersicum cv. Heinz 1706 (SlINO; AOS88523), S. pennellii (SpINO; XP_015075232), картофеля S. tuberosum (StINO; NW_006239143), перца C. annuum (CaINO; XP_016546914) и табака N. tabacum (NtINO; XP_016511953). Домен цинкового пальца (12–52 а.о.) и домен YABBY (108–163 а.о.) подписаны под выравниванием полужирным шрифтом. Замены в консервативных доменах указаны звездочками (а); б – дендрограмма, построенная на основе анализа аминокислотных последовательностей INO у исследуемых видов томата (ML, модель Jones–Taylor–Thornton + Gamma distributed with Invariant sites). В узлах указаны значения бутстрепа на 1000 бутстреп-реплик.
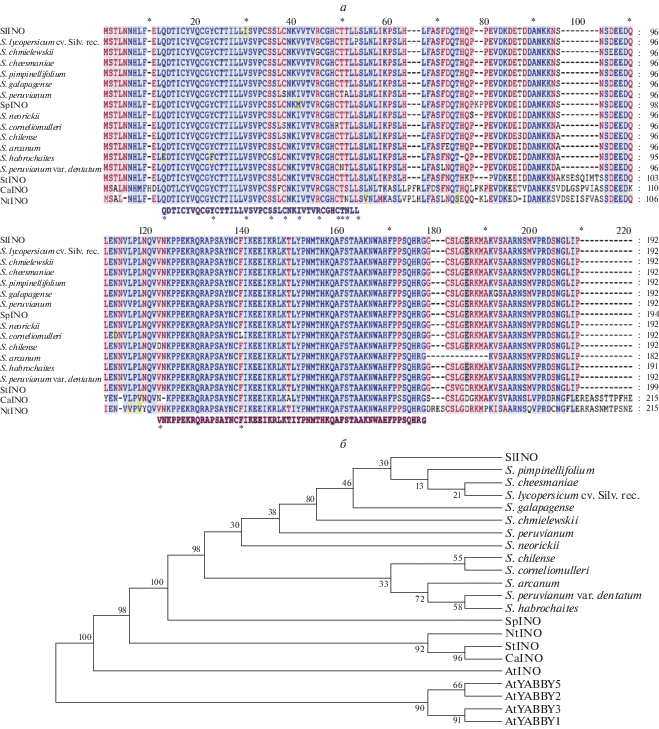
В сравнении с референсным геном SlINO в последовательностях INO исследуемых образцов томата выявлены 243 вариабельных сайта (12.9% от выровненной длины): 200 локализованы в интронах, 43 – в экзонах. Из 43 экзон-специфичных SNPs 22 были несинонимичными и приводили к замещениям аминокислотных остатков: 10 – в домене цинкового пальца (Q12E, Y22F, S34G, C37S, V40I, R44G, C48R, T49N, T50A, L52R), девять – в междоменной области (F67L, D68N, P73S, T81I, N88D, S89A, D92E, N99D, L102V), два – в YABBY-домене (V108D, F125L), и одно – на С-конце (V175G). При этом следует отметить, что четыре радикальных замещения в домене цинкового пальца (S34G, R44G, C48R, T49N) и оба радикальных замещения в YABBY-домене признаны потенциально вредными (статус “deleterious”). Интересно, что в экзоне VI INO S. arcanum была выявлена протяженная делеция 30 пн, приводящая к отсутствию 10 а.о. в С-концевой внедоменной области, сразу после YABBY-домена (рис. 1,а).
Выявленная вариабельность, в особенности потенциально вредные радикальные замещения, могут оказывать влияние на фолдинг белков INO, изменяя силу и/или специфичность связывания с ДНК-мишенями и/или белковыми партнерами. Можно предположить, что межвидовой полиморфизм INO Solanum, не изменяя основной функции белка, может приводить к морфологическому разнообразию архитектуры интегумента и отражаться на эффективности оплодотворения семязачатка и формирования оболочки семени и, как следствие, на жизнеспособности семян.
Ранее было показано, что экспрессия кДНК генов семейства YABBY A. thaliana, CRC и YABBY3, под контролем промотора гена INO (PROINO, A. thaliana) была способна восстанавливать формирование внешнего интегумента в трансгенных растениях A. thaliana с мутацией ino. Однако только экспрессия кДНК INO под контролем PROINO была ограничена базальной областью развивающихся семязачатков за счет негативной регуляции транскрипционным фактором SUPERMAN, что приводило к восстановлению правильной асимметрии внешнего интегумента [5]. Исследование отдельных областей INO A. thaliana с использованием химерных белков под контролем промоторов PROINO и 35S CaMV показало, что важная роль в правильном развитии интегумента принадлежит внедоменным областям [5]. В случае исследуемых видов томата N- и С-концевые области белка INO меньше, чем у INO A. thaliana в 4 и 2 раза соответственно. Это согласуется с тем, что сверхэкспрессия гена SlINO (S. lycopersicum) в растениях A. thaliana с мутацией ino не восстанавливала исходный фенотип семязачатка [1], что может свидетельствовать о функциональных различиях внедоменных областей ортологов INO у разных видов/семейств растений. В этой связи факт обнаружения 10 а.о. делеции в С-концевой внедоменной области INO S. arcanum может представлять интерес для дальнейших исследований.
Для того чтобы оценить, насколько эволюция INO соотносится с предполагаемой эволюцией видов томата в целом [14, 22], была построена дендрограмма по аминокислотным последовательностям INO (рис. 1,б). На дендрограмме виды томата секции Lycopersicon образовывали единый кластер (ИБ=100) в кладе INO, базальные ветви к которому формировали белки INO других Solanaceae – перца C. annuum, картофеля S. tuberosum и табака N. tabacum (рис. 1,б). Однако внутри кластера хорошего разделения между видами не наблюдалось. Не было показано даже достоверного разделения видов на более древние зеленоплодные и считающиеся эволюционно более молодыми красноплодные виды, что было выявлено при анализе других представителей семейства YABBY видов томата (YABBY1 и YABBY3) [19, 23]. Данный факт может указывать на более недавнее происхождение гена INO и/или более низкую скорость эволюции гена INO в сравнении с “вегетативными” генами YABBY.
Работа выполнена при финансовой поддержке гранта Российского научного фонда № 16-16-10022, с использованием экспериментальной установки искусственного климата ЭУИК и ЦКП “Биоинженерия”.
Список литературы
Skinner D.J., Brown R.H., Kuzoff R.K., Gasser C.S. Conservation of the role of INNER NO OUTER in development of unitegmic ovules of the Solanaceae despite a divergence in protein function // BMC Plant Biology. 2016. V. 16. Article 143. doi 10.1186/s12870-016-0835-z
Nakayama H., Yamaguchi T., Tsukaya H. Expression patterns of AaDL, a CRABS CLAW ortholog in Asparagus asparagoides (Asparagaceae), demonstrate a stepwise evolution of CRC/DL subfamily of YABBY genes // Am. J. Bot. 2010. V. 97. № 4. P. 591–600. doi 10.3732/ ajb.0900378
Simon M.K., Skinner D.J., Gallagher T.L., Gasser C.S. Integument development in Arabidopsis depends on interaction of YABBY protein INNER NO OUTER with coactivators and corepressors // Genetics. 2017. V. 207. № 4. P. 1489–1500. doi 10.1534/genetics.117.300140
Bowman J.L., Smyth D.R. CRABS CLAW, a gene that regulates carpel and nectary development in Arabidopsis, encodes a novel protein with zinc finger and helix-loop-helix domains // Development. 1999. V. 126. P. 2387–2396.
Meister R.J., Oldenhof H., Bowman J.L., Gasser C.S. Multiple protein regions contribute to differential activities of YABBY proteins in reproductive development // Plant Physiol. 2005. V. 137. № 2. P. 651–662. doi 10.1104/pp.104.055368
Finet C., Floyd S.K., Conway S.J. et al. Evolution of the YABBY gene family in seed plants // Evol. Dev. 2016. V. 18. № 2. P. 116–126. doi 10.1111/ede.12173
Yamada T., Yokota S., Hirayama Y. et al. Ancestral expression patterns and evolutionary diversification of YABBY genes in angiosperms // The Plant J. 2011. V. 67. № 1. P. 26–36. doi 10.1111/j.1365-313X.2011.04570.x
Simon M.K., Williams L.A., Brady-Passerini K. et al. Positive- and negative-acting regulatory elements contribute to the tissue-specific expression of INNER NO OUTER, a YABBY-type transcription factor gene in Arabidopsis // BMC Plant Biol. 2012. V. 12. Article 214. doi 10.1186/1471-2229-12-214
Lee J.Y., Baum S.F., Oh S.H. et al. Recruitment of CRABS CLAW to promote nectary development within the eudicot clade // Development. 2005. V. 132. P. 5021–5032.
Sun W., Huang W., Li Z. et al. Characterization of a Crabs Claw gene in basal eudicot species Epimedium sagittatum (Berberidaceae) // Int. J. Mol. Sci. 2013. V. 14. № 1. P. 1119–1131. doi 10.3390/ijms14011119
Toriba T., Harada K., Takamura A. et al. Molecular characterization the YABBY gene family in Oryza sativa and expression analysis of OsYABBY1 // Mol. Genet. Genomics. 2007. V. 277. № 5. P. 457–468.
Shchennikova A.V., Slugina M.A., Beletsky A.V. et al. The YABBY genes of leaf and leaf-like organ polarity in leafless plant Monotropa hypopitys // Intern. J. Genomics. 2018. V. 2018. Article ID 7203469. doi 10.1155/2018/7203469
Gallagher T.L., Gasser C.S. Independence and interaction of regions of the INNER NO OUTER protein in growth control during ovule development // Plant Physiol. 2008. V. 147. № 1. P. 306–315. doi 10.1104/ pp.107.114603
Peralta I.E., Spooner D.M., Knapp S. Taxonomy of wild tomatoes and their relatives (Solanum sect. Lycopersicoides, sect. Juglandifolia, sect. Lycopersicon; Solanaceae) // Systematic Botany Monographs American Society of Plant Taxonomists. USA, 2008. V. 84. 186 p.
Spooner D.M., Peralta I.E., Knapp S. Comparison of AFLPs with other markers for phylogenetic inference in wild tomatoes [Solanum L. section Lycopersicon (Mill.) Wettst.] // Taxon. 2005. V. 54. P. 43–61.
Han H.Q., Liu Y., Jiang M.M. et al. Identification and expression analysis of YABBY family genes associated with fruit shape in tomato (Solanum lycopersicum L.) // Genet. Mol. Res. 2015. V. 14. № 2. P. 7079–7091.
Ezura K., Ji-Seong K., Mori K. et al. Genome-wide identification of pistil-specific genes expressed during fruit set initiation in tomato (Solanum lycopersicum) // PLoS One. 2017. V. 12. № 7. Article e0180003. doi 10.1371/journal.pone.0180003
Huang Z., Van Houten J., Gonzalez G. et al. Genome-wide identification, phylogeny and expression analysis of SUN, OFP and YABBY gene family in tomato // Mol. Genet. Genomics. 2013. V. 288. № 3–4. P. 111–129. doi 10.1007/s00438-013-0733-0
Филюшин М.А., Слугина М.А., Щенникова А.В., Кочиева Е.З. YABBY3-ортологи дикорастущих видов томата: структура, полиморфизм и экспрессия // Acta Naturae. 2017. Т. 9. № 4 (35). С. 106–115.
Филюшин М.А., Слугина М.А., Пышная О.Н. и др. Структурный анализ генов-гомологов INNER NO OUTER (INO) у видов рода Capsicum // Генетика. 2018. Т. 54. № 6. С. 735–740.
Lora J., Hormaza J.I., Herrero M. Transition from two to one integument in Prunus species: expression pattern of INNER NO OUTER (INO), ABERRANT TESTA SHAPE (ATS) and ETTIN (ETT) // New Phytol. 2015. V. 208. № 2. P. 584–595. doi 10.1111/nph.13460
Aflitos S., Schijlen E., de Jong H. et al. Exploring genetic variation in the tomato (Solanum section Lycopersicon) clade by whole-genome sequencing // Plant J. 2014. V. 80. № 1. P. 136–148.
Филюшин М.А., Слугина М.А., Щенникова А.В., Кочиева Е.З. Идентификация и анализ экспрессии гена YABBY1 у дикорастущих видов томатов // Генетика. 2018. Т. 54. № 5. С. 535–546.
Дополнительные материалы отсутствуют.


