Генетика, 2019, T. 55, № 2, стр. 199-206
Высокое аллельное разнообразие гена DRB3 (класс II ГКГ) у сайгака (Saiga tatarica L., 1766), полученное с помощью секвенирования нового поколения
К. К. Тарасян 1, П. А. Сорокин 1, *, Н. В. Кашинина 1, М. В. Холодова 1, 2
1 Институт проблем экологии и эволюции им. А.Н. Северцова Российской академии наук
119071 Москва, Россия
2 Томский государственный университет, кафедра зоологии
634050 Томск, Россия
* E-mail: sorokin-p@yandex.ru
Поступила в редакцию 25.01.2018
После доработки 03.05.2018
Принята к публикации 27.04.2018
Аннотация
Впервые описаны нуклеотидные последовательности аллелей гена DRB3 главного комплекса гистосовместимости (класс II) сайгака. Для исследованной выборки получена высокая степень гетерозиготности. Результаты анализа согласуются с гипотезой сверхдоминирования гетерозигот. Филогенетические связи аллей гена DRB3 сайгака и других полорогих не соответствуют систематическому положению сайгака в сем. Bovidae. Предполагается, что полиморфизм аллелей гена DRB3 ГКГ сайгака в первую очередь обусловлен разнообразием возбудителей заболеваний (бактерий, простейших, гельминтов и др.), воздействующих на популяцию сайгаков в ходе эволюционной истории.
Проблема утраты генетического разнообразия в результате длительной депрессии численности животных и нарушение механизма сохранения биологического разнообразия в таких популяциях являются одной из важнейших в современной экологии и природоохранной биологии. Генетическое разнообразие в значительной степени отражает адаптивный потенциал организмов, популяций и видов [1–5]. В первую очередь это касается разнообразия функционально значимых генов и генных комплексов, обусловливающих устойчивость животных к неблагоприятным факторам среды – инфекционным агентам, резким климатическим изменениям и т.п. В связи с этим исследователи оказывают пристальное внимание генам главного комплекса гистосовместимости (ГКГ), отвечающим за иммунитет. В семействе Bovidae, к которому относится сайгак, расположение генов ГКГ было изучено на примере Bos taurus [6]. Установлено, что в этой группе млекопитающих он расположен на 23-й хромосоме [7]. Как и у остальных млекопитающих, его принято разделять на три класса генов: первый отвечает за иммунитет к внутриклеточным паразитам (как вирусного, так и бактериального происхождения), второй – за восприимчивость к внеклеточным чужеродным агентам, третий класс генов несет вспомогательную функцию по отношению к первым двум [8–11]. Наиболее информативным у копытных считают ген DRB3, относящийся ко II классу ГКГ [12]. Полиморфизм аллелей гена DRB3 критически важен для жизнеспособности популяций и видов, испытывающих длительное катастрофическое снижение численности [13]. Именно к этой категории относится популяция сайгака (Saiga tatarica) Северо-Западного Прикаспия, включенная в Красную книгу России и в Международный список видов, находящихся под угрозой исчезновения (IUCN, http://www.iucnredlist.org/ details/19834/0). Численность этой, единственной в Европе, популяции сайгака, обитающей в восточных районах Республики Калмыкия и юго-западной части Астраханской области, с конца XX в. снизилась более чем в 40 раз: с 150 000 особей до 7500 в 2012 г. [14–16], а в 2016 г., согласно экспертной оценке (В.Г. Калмыков, личное сообщение), составила всего 3500 особей. В природоохранной биологии считается, что сохранение видов позвоночных животных обеспечивается благодаря поддержанию генетического разнообразия и приспособленности к меняющимся условиям среды при необходимой пороговой величине эффективной численности в 500–5000 особей [17–20]. Исходя из этого, можно предположить, что эффективная численность популяции сайгака Северо-Западного Прикаспия в настоящее время приблизилась к ее минимальной пороговой величине. Катастрофические падения численности неоднократно отмечались и в эволюционной истории сайгака [21–23]. Кроме того, следует отметить, что в естественных условиях сайгак подвержен разнообразным заболеваниям (пастереллезу, ящуру, бруцеллезу, сибирской язве и др.), а также инвазиям разнообразных видов гельминтов [21, 24–27]. До настоящего момента исследований, посвященных полиморфизму аллелей генов ГКГ, обеспечивающих иммунный ответ у сайгаков, не проводили.
Цель данной работы – описать аллельное разнообразие гена DRB3 у сайгака (Saiga tatarica) методом полногеномного секвенирования NGS и сравнить его с полиморфизмом этого гена у других представителей семейства Bovidae.
МАТЕРИАЛЫ И МЕТОДЫ
Для работы мы использовали образцы пуповины от 12 новорожденных сайгаков, собранные на территории заповедника “Черные Земли” в Калмыкии в 2011 г. Выделение ДНК из образцов производили набором реагентов Diatom prep 100 (Изоген, Россия). Полимеразная цепная реакция (ПЦР) проводилась с помощью набора реагентов Tersus Plus PCR Kit (Евроген, Россия) с праймерами La31 (5'-GATGGATCCTCTCTCTGCAGCACATTTCCT-3') и La32 (5'-CTTGAATTCGCGCTCACCTCGCCGCTG-3'), для которых показано, что они эффективно амплифицируют ген DRB3 у разных видов копытных млекопитающих [28–31] в режиме, описанном ранее [29]. Фрагменты ПЦР нужной длины очищали методом вырезания из геля с помощью колонок Cleanup Standard (Евроген, Россия). Концентрацию ДНК измеряли на капельном спектрофотометре NanoDrop 8000 (Thermo Scientific, США). Общая концентрация ПЦР-продукта составляла 500 нг. Для приготовления ДНК-библиотеки использовали лигирование очищенного ПЦР-продукта с адапторами Gs Rapid Library (Roche, Германия), с помощью которых определялась принадлежность ПЦР-продукта к определенному образцу. Далее проводили эмульсионную ПЦР с использованием набора с эмульсионным маслом и реагентов для эмПЦР лигированных библиотек. ПЦР-продукт секвенировали с использованием реакции параллельного пирофосфатного секвенирования (технология 454) на частицах-носителях на аппарате GS Junior (Roche, США) на базе ЦКП “Геном” ИМБ РАН. За один запуск определяли последовательности ПЦР-продукта для 12 разных образцов. В результате секвенирования было получено большое количество прямых и обратных прочтений разной длины. С помощью программы СLС Genomic Workbench 8.5 (QIAGEN Aarhus A/S) отбирали последовательности с уровнем качества (PHRED) больше 30, а длина соответствовала расчетной (длина фрагмента гена, включающего праймерные и адапторные последовательности). Для каждого образца было получено от 341 до 4207 прочтений подходящего качества и длины. Последовательности, прошедшие фильтрацию, были разделены на прямые и обратные и сохранены в формате FASTA. Их выравнивание и удаление праймерных областей проводили с помощью программы Lasergene MegAlign. 7.1.0 (DNASTAR. Madison, WI). Для выявления уникальных последовательностей в каждом образце использовали сетевой ресурс FaBox [32]. Число последовательностей, описанных в программе FaBox для каждого образца, составило от 78 до 510. В каждом образце с частотой от 19.71 до 30.41% присутствовали два варианта. Помимо этого, с частотой меньше 0.7% встречались минорные последовательности. Поскольку ген DRB3 является однокопийным, то наиболее частые варианты рассматривались как истинные аллели, а минорные последовательности – как результат ошибок полимераз в процессе ПЦР.
Вычисление показателей гетерозиготности выполняли в программе Arlequin 3.5.2 [33]. Статистические расчеты проведены в пакете программ STATISTICA 8.0 (StatSoft, Inc., Tulsa, OK, USA). Проверку используемых выборок значений на нормальность распределений осуществляли с помощью теста Шапиро–Уилкса. По результатам данного теста сравнение значений наблюдаемой (HO) и ожидаемой гетерозиготности (HE) проводили с помощью Т-теста для зависимых переменных, а наблюдаемые генетические дистанции между описанными сочетаниями аллелей и остальными, теоретически возможными вариантами выполняли методом Манна–Уитни. Построение филогенетических деревьев сделано в программе MEGA 6.06 [34] методом Neighbor-Joining (NJ) с использованием модели Тамура–Нея, медианной сети аллелей гена DRB3 – в программе Network 5000 [35]. Для удаления избыточного количества промежуточных узлов использовалась функция упрощения сети (MP calculation). Для построения сети гаплотипов и NJ-дендрограмм были использованы нуклеотидные последовательности аллелей гена DRB3 и гена цитохрома b мтДНК представителей сем. Bovidae, включенные в базу NCBI (номера указаны на соответствующих рисунках).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Описание нуклеотидных последовательностей
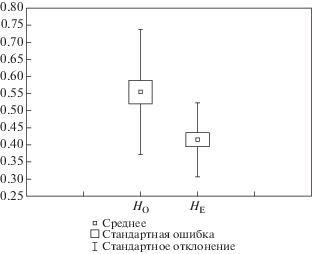
Для изученной выборки сайгаков описано семь аллелей гена DRB3. Нуклеотидные последовательности аллелей (250 пн) помещены в международную генетическую базу NCBI под номерами MF960850–MF960856. Распределение аллелей в исследованных образцах показано в табл. 1. Выявлено 29 вариабельных сайтов (замен), из которых 23 – парсимониально-информативные. Все животные были гетерозиготны по аллелям гена DRB3. Наблюдаемая гетерозиготность (HO = 1) достоверно отличалась от ожидаемой (HE = 0.83; χ2 = 18.58, р < 0.01). Дополнительно рассчитывали показатели гетерозиготности на основании отдельных изменчивых нуклеотидных позиций (замен), рассматривая их как отдельные локусы. Различия между значениями HO и HE были статистически достоверны (T = 7.77; p < 0.01; n = 29) (рис. 1). Таким образом, в обоих случаях наблюдаемая гетерозиготность по гену DRB3 была достоверно выше ожидаемой. Эти результаты согласуются с гипотезой преимущественного отбора гетерозигот, основанной на экспериментально полученном наблюдении кодоминантного типа наследования аллелей ГКГ [36]. Согласно данной гипотезе существуют два варианта действия отбора: симметричный балансирующий отбор, дающий одинаковое преимущество любым гетерозиготам [37], и дивергентное аллельное преимущество. В последнем случае наиболее успешными, с точки зрения отбора, являются гетерозиготы с наиболее сильно отличающимися аллелями [38]. Для проверки существования признаков дивергентного аллельного преимущества в нашей выборке аллелей мы рассмотрели аминокислотные последовательности аллелей гена DRB3.
Описание аминокислотных последовательностей
Полученные нуклеотидные последовательности аллелей гена DRB3 сайгака транслировали в аминокислотные последовательности (табл. 2). Исследуемая последовательность гена DRB3 кодирует 83 аминокислоты. В описанных аллелях гена DRB3 выявлено 20 вариабельных позиций (замен) аминокислот, из них 16 – парсимониально-информативные. Были обнаружены две пары разных нуклеотидных последовательностей, кодирующие идентичные последовательности аминокислот: MF960851 и MF960854, а также MF960853 и MF960856. Поэтому далее последовательность аминокислот, соответствующая каждой паре, была записана под номером одного из кодирующих ее вариантов (MF960851 и MF960853 соответственно). Оказалось, что и на уровне белковых продуктов гена DRB3 сохраняется высокий уровень гетерозиготности. Мы вычислили генетическую дистанцию (p-distance) между аминокислотными последовательностями аллелей каждого исследуемого образца. Для сравнения использовали выборку генетических расстояний, рассчитанных тем же способом между всеми возможными сочетаниями аллелей, за исключением уже описанных нами. Сравнение этих выборок не показало достоверных отличий наблюдаемых дистанций между описанными сочетаниями аллелей от остальных, теоретически возможных (U = 59.5, Z = 0.033, p = 0.97). Таким образом, на уровне аминокислотных последовательностей наши данные не позволяют подтвердить предположение о дивергентном аллельном преимуществе в популяции сайгаков. Необходимо отметить, что наши выводы не являются окончательными, поскольку основаны на анализе небольшой выборки животных. Дальнейшее развитие работы позволит внести уточнения в сделанные нами заключения.
Сравнение аллелей гена DRB3 сайгака с другими полорогими
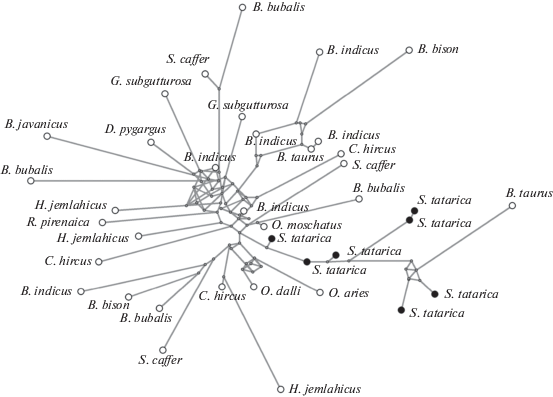
Для выявления возможных филогенетических связей аллелей гена DRB3 сайгака была построена медианная сеть аллелей с использованием гомологичных нуклеотидных последовательностей ряда видов семейства Bovidae (рис. 2). Общих гаплотипов с другими видами этого семейства обнаружено не было. Медианная сеть аллелей гена DRB3 не образовала ясной филогенетической структуры. При этом аллели гена DRB3 сайгаков объединились в три группы. Одна из этих групп, близкая к быку (Bos taurus), возможно, сформировалась под влиянием одних и тех же внешних экологических факторов.
Рис. 2.
Медианная сеть аллелей гена DRB3 сайгака и других представителей сем. Bovidae. Длина ветвей пропорциональна числу мутаций, разделяющих аллели, черным цветом выделены аллели сайгаков. Серыми точками показаны узловые гипотетические аллели.
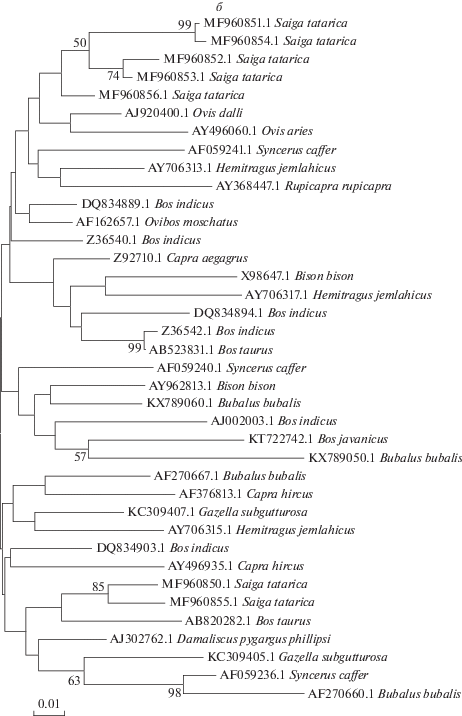
Для филогенетических деревьев (NJ), построенных по фрагментам генов DRB3 и цитохрома b мтДНК для сайгака и других видов семейства полорогих, соответствия между топологиями не обнаружено (рис. 3,а, б). Характерно, что на NJ-дендрограмме аллели гена DRB3 (рис. 3,б) формируют три группы, одна из которых существенно удалена от других, в то время как на дендрограмме, построенной по последовательностям гена цитохрома b мтДНК, сайгаки образуют единую гаплогруппу (рис. 3,а). Аналогичные результаты были получены при сравнении дендрограммы по гену DRB3 с опубликованными ранее построениями, основанными на ядерных и/или митохондриальных генах [39, 40]. Таким образом, филогенетические связи аллей гена DRB3 сайгака и других полорогих не соответствуют систематическому положению сайгака в семействе Bovidae. Вероятно, разнообразие аллелей гена DRB3 ГКГ сайгака в первую очередь обусловлено разнообразием инфекционных и инвазийных агентов, воздействующих на популяцию сайгаков в ходе эволюционной истории.
Рис. 3.
Филогенетическое дерево гаплотипов цитохрома b мтДНК, построенное методом Neighbor-Joining с эволюционной моделью Тамура–Нея (а). Филогенетическое дерево аллелей гена DRB3, построенное методом Neighbor-Joining с эволюционной моделью Тамура–Нея (б). Цифры в узлах обозначают уровень бутстреп поддержки (1000 реплик), значения менее 50% не показаны. Шкала – генетическая дистанция между гаплотипами. Перед латинским названием вида указан номер последовательности в базе NCBI (для обоих позиций рисунка).
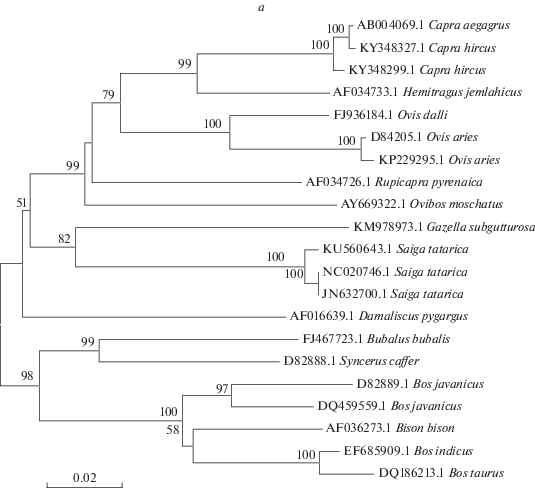
Рис. 3.
Окончание.
Наше предварительное исследование позволило впервые описать нуклеотидные последовательности аллелей гена DRB3 (ГКГ класс II) сайгака. У животных обнаружена высокая степень гетерозиготности, а также признаки соответствия сформулированной в литературе гипотезе сверхдоминирования гетерозигот. Предполагается, что полиморфизм аллелей гена DRB3 ГКГ сайгака в первую очередь обусловлен множеством возбудителей заболеваний, воздействующих на популяцию сайгаков в ходе эволюционной истории, включая Pasteurella multocida (возбудитель пастереллеза), а также более 55 видов эндопаразитов, в том числе около 10 видов простейших, кокцидий рода Eimeria, различных видов нематод и цестод [41]. Для уточнения выдвинутых нами предположений требуется дальнейшее исследование на большей выборке животных.
Авторы благодарят за содействие в организации сбора образцов А.В. Давыдова, за помощь в проведении секвенирования – специалиста по продукции ООО “Рош Диагностика Рус” Н.В. Коробан и руководителя ЦКП “Геном” ИМБ РАН А.В. Кудрявцеву, а также зав. сектором молекулярной эволюции ИППИ РАН Г.А. Базыкина за консультации по обработке данных.
Работа поддержана грантом РФФИ № 17-04-01351.
Список литературы
Soulé M.E. Viable Populations for Conservation. Cambridge: Univ. Press, 1987. 189 p. doi 10.1017/ CBO9780511623400
Luikart G., Sherwin W.B., Steele B.M., Allendorf F.W. Usefulness of molecular markers for detecting population bottlenecks via monitoring genetic change // Mol. Ecol. 1998. № 7. P. 963–974. doi 10.1046/j.1365-294x.1998.00414.x
Frankham R. Ecosystem recovery enhanced by genotypic diversity // Heredity. 2005. V. 95. P. 183. doi 10.1038/sj.hdy.6800706
Fox C.W., Reed D.H. Inbreeding depression increases with environmental stress: an experimental study and meta-analysis // Evolution. 2011. V. 65. № 1. P. 246–258. doi 10.1111/j.1558-5646.2010.01108.x
Willi Y., Van Buskirk J., Hoffmann A.A. Limits to the adaptive potential of small populations // Annu. Rev. Ecol. Evol. Syst. 2006. V. 37. P. 433−458. doi 10.1146/annurev.ecolsys.37.091305.110145
Bensaid A., Young J.R., Kaushal A., Teale A.J. Pulsed-field gel electrophoresis and its application in the physical analysis of the bovine MHC // Gene Mapping Techniques 122 and Applications / Ed. McLaren Marcel Dekker, New York, NY, USA, 1991. P. 127.
Fries R., Aggen A., Womack J.E. The bovine genome map // Mamm. Genome. 1993. V. 4. P. 405–428.
Behl J.D., Verma N.K., Tyagi N. et al. The major histocompatibility complex in Bovines: a review // ISRN Veterinary Sci. 2012. P. 1–12. doi 10.5402/2012/872710
Paracha H., Hussain T., Tahir M.Z. et al. Multifunctional DRB3, a MHC class II gene, as a useful biomarker in small ruminants: a review // J. Inf. Mol. Biol. 2015. V. 3. № 1. P. 19–23. doi 10.14737/journal.jimb/ 2015/3.1.19.23
Subramani K.V., Sankar M., Raghunatha R.R. et al. Association of genetic resistance to gastrointestinal nematodes and the polymorphism at CAHI-DQA1 exon 2 // Internat. J. Sci. Environ. Technol. 2016. V. 5. № 2. P. 678–687.
Тарасян K.K., Сорокин П.А., Холодова М.В., Рожнов В.В. Главный комплекс гистосовместимости (Major Histocompatibility Complex, МНС) у млекопитающих и его значение в изучении редких видов (на примере семейства Felidae) // Журн. общ. биол. 2014. Т. 75. № 4. С. 302–314.
Kumar S., Sangwan M.L., Ahlawat S., Barwar A. Polymorphism in DRB3 exon 2 by PCR-RFLP and its association with mastitis in Murrah baffaloes // Ind. J. Biotech. 2011. V. 10. P. 232–234.
Sommer S. The importance of immune gene variability (MHC) in evolutionary ecology and conservation // Front. Zool. 2005. V. 2. № 16. P. 1–18. doi 10.1186/ 1742-9994-2-16
Неронов В.М., Каримова Т.Ю., Лущекина А.А. Экосистемный подход и ГАП-анализ для обследования состояния популяции сайгака в Северо-Западном Прикаспии // Астрахан. вестн. экологич. образования. 2011. № 2(18). С. 151−157.
Неронов В.М., Арылова Н.Ю., Дубинин М.Ю. и др. Современное состояние и перспективы сохранения сайгака в Северо-Западном Прикаспии // Аридные экосистемы. 2013. Т. 19. Вып. 2(55). С. 5−14.
Neronov V.M., Lushchekina A.A., Karimova T.Y., Arylova N.Y. Population dynamics of a key steppe species in a changing world: the critically endangered saiga antelope // Eurasian Steppes. Ecological Problems and Livelihoods in a Changing World Plant and Vegetation 6 / Eds Werger M.J.A., van Staalduinen M.A. Springer Science+Business Media B. V. 2012. Ch. 12. P. 335–357. doi 10.1007/978-94-007-3886-7_12
Soule M.E. Threshold for survival: maintaining fitness and evolutionary potential // Conservation Biology: An Evolutionary-Ecologial Perspective / Eds Soule M.E., Wilcox B.A. Sinauer, Sunderland, 1980. P. 151–169.
Lynch M., Lande R. The critical effective size for a genetically secure population // Anim Conserv. 1998. V. 1. P. 70–72. doi 10.1111/j.1469-1795.1998.tb00229.x
Frankham R., Bradshaw C.J., Brook B.W. Genetics in conservation management: revised recommendations for the 50/500 rules, Red List criteria and population viability analyses // Biol. Conserv. 2014. V. 170. P. 56–63. doi 10.1016/j.biocon.2013.12.036
Frankham R., Bradshaw C.J., Brook B.W. 50/500 rules need upward revision to 100/1000 – response to Franklin et al. // Biol. Conserv. 2014. V. 176. P. 286. doi 10.1016/j.biocon.2014.05.006
Банников А.Г., Жирнов Л.В., Лебедева Л.С., Фандеев А.А. Биология сайгака. М.: Изд-во с-х. лит-ры, журналов и плакатов, 1961. 336 с.
Барышников Г.Ф., Дмитриева Е.Л., Крахмальная Т.В., Шер А.В. Происхождение, эволюция и систематика сайгака // Сайгак: филогения, систематика, экология, охрана и использование / Ред. Соколов В.Е., Жирнов Л.В. М., 1998. С. 9−20.
Campos P.F., Kristensen T., Orlando L. et al. Ancient DNA sequences point to a large loss of mitochondrial genetic diversity in the saiga antelope (Saiga tatarica) since the Pleistocene // Mol. Ecol. 2010. V. 19. № 22. P. 4863–4875. doi 10.1111/j.1365-294X.2010.04826.x
Байтурсинов К.К. Гельминты сайгака в Казахстане // Вестн. ПГУ им. С. Торайгырова. Сер. химико-биологич. 2005. № 2. С. 67−81.
Байтурсинов К.К. Краткие данные по биологии и зараженности гельминтами сайгака (Saiga tatarica L., 1766) в Казахстане // Вестник КазНУ. Сер. биологич. 2009. № 3(42). С. 83−87.
Петров В.С. Гельминты сайгаков и их значение в эпизоотологии гельминтами овец: Автореф. дисс. ... канд. вет. наук. М.: ВИГИС, 1985. 24 с.
Грачев Ю.А. Массовая гибель сайгаков в Казахстане – погибло около 12000 особей // Saiga news. 2010. № 11. С. 2–3.
Sigurdardottir S., Borsch C., Gustafsson K., Andersson L. Cloning and sequence analysis of 14 DBR alleles of the bovine major histocompatibility complex using the polymerase chain reaction // Animal Genet. 1991. V. 22. P. 199–209.
Mikko S., Roed K., Schmutz S., Andersson L. Monomorphism and polymorphism at MHC DRB loci in domestic and wild ruminants // Immunol. Rev. 1999. V. 167. P. 169–178. doi 10.1111/j.1600-065X.1999.tb01390.x
Kennedy L.J., Modrell A., Groves P. et al. Genetic diversity of the major histocompatibility complex class II in Alaskan caribou herds // Internat. J. Immunogenet. 2010. V. 38. P. 109–119. doi 10.1111/j.1744-313X.2010.00973.x
Taylor S.S., Jenkins D.A., Arcese P. Loss of MHC and neutral variation in Peary caribou: genetic drift is not mitigated by balancing selection or exacerbated by MHC allele distributions // PLoS One. 2012. V. 7. № 5. P. 1–11. doi 10.1371/journal.pone.0036748
Villesen P. FaBox: an online toolbox for fasta sequences // Mol. Ecol. Notes. 2007 V. 7. № 6. P. 965–968. doi 10.1111/j.1471-8286.2007.01821.x
Excoffier L., Lischer H.E.L. Arlequin suite ver. 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. 2010. V. 1. P. 564–567.
Tamura K., Stecher G., Peterson D. et al. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0 // Mol. Biol. Evol. 2013. V. 30. P. 2725–2729. doi 10.1093/molbev/mst197
Bandelt H.J., Forster P., Rohl A. Median-Joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48. doi 10.1093/oxfordjournals.molbev.a026036
Doherty P.C., Zinkernagel R.M. A biological role for the major histocompatibility antigens // Lancet. 1975. V. 1. № 7922. P. 1406–1409.
Takahata N., Nei M. Allelic genealogy under overdominant and frequency-dependent selection and polymorphism of major histocompatibility complex loci // Genetics. 1990. V. 124. № 4. P. 967–978.
Wakeland E.K., Boehme S., She J.X. et al. Ancestral polymorphisms of MHC class II genes: divergent allele advantage // Immunol. Res. 1990. V. 9. № 2. P. 115–122. doi 10.1007/BF02918202
Barmann E.V., Rossner G.E., Worheide G. A revised phylogeny of Antilopini (Bovidae, Artiodactyla) using combined mitochondrial and nuclear genes // Mol. Phylogenet. Evol. 2013. V. 67. P. 484−493. doi 10.1016/ j.ympev.2013.02.015
Khademi T.G. Evaluation of phylogenetic relationships of Antilopini and Oreotragini tribes (Bovidae: Artiodactyla) based on complete mitochondrial genomes // J. Wildlife Biodiver. 2017. V. 1. № 1. P. 1−11. doi 10.22120/JWB.2017
Бекенов А.Б., Поле С.Б., Поле С.Б. и др. Гибель сайгаков от болезней и паразитарных инвазий // Сайгак: филогения, систематика, экология, охрана и использование / Ред. Соколов В.Е., Жирнов Л.В. М., 1998. С. 247–251.
Дополнительные материалы отсутствуют.