Генетика, 2019, T. 55, № 2, стр. 192-198
К вопросу о происхождении Larix × lubarskii Sukaczev: анализ полиморфизма маркеров митохондриального генома
Е. А. Васюткина 1, *, И. Ю. Адрианова 1
1 Федеральный научный центр биоразнообразия наземной биоты Восточной Азии
Дальневосточного отделения Российской академии наук
690022 Владивосток, Россия
* E-mail: levina@biosoil.ru
Поступила в редакцию 27.03.2018
После доработки 04.07.2018
Принята к публикации 23.05.2018
Аннотация
Впервые исследованы генетическое разнообразие Larix × lubarskii Sukaczev и ее генетические взаимоотношения с другими лиственницами юга Приморского края, дальневосточными и сибирскими видами Larix по данным анализа полиморфизма длины фрагмента UBC460 митохондриального происхождения и нуклеотидных последовательностей 5'-фланкирующей области гена atpA и региона nad4(3c–4r) митохондриальной ДНК. В популяции L. × lubarskii выявлено два варианта длины фрагмента UBC460, с преобладанием короткого. Этот таксон характеризуется средним гаплотипическим (0.582) и низким нуклеотидным (0.0003) разнообразием. Сравнительный анализ нуклеотидного полиморфизма двух регионов мтДНК у лиственниц юга Приморского края выявил у L. × lubarskii три митотипа, из них только один был общим с L. olgensis A. Henry (locus classicus). Сеть генеалогических связей митотипов указывает на общность происхождения L. × lubarskii и L. olgensis и относительно недавнюю их дивергенцию, а также на более близкую связь с L. sibirica Ledeb., чем с L. gmelinii (Rupr.) Rupr.
В юго-западной части Приморского края произрастает Larix × lubarskii Sukaczev, имеющая диагностические морфологические признаки, характерные для нескольких видов, распространенных на Российском Дальнем Востоке (РДВ) и в Китае. Одни авторы [1, 2] отмечали ее морфологическое сходство с L. principis-rupprechtii Mayr. и L. gmelinii (Rupr.) Rupr. и связывали это сходство с их общим происхождением и недавней географической изоляцией. Другие авторы считали ее L. olgensis A. Henry [3] либо L. gmelinii var. olgensis (A. Henry) Ostenf. & Syrach [4–6]. Кроме того, рассматривались версии о гибридном происхождении L. × lubarskii: по одной из них [7] она является результатом давней и длительной гибридизации L. olgensis и L. principis-rupprechtii, по другой [8] – это гибридное смешение L. olgensis с гибридами L. kamtschatica (Rupr.) Carr. и L. gmelinii. В.М. Урусов [9, 10], учитывая все известные работы и опираясь на собственные исследования, предположил, что L. × lubarskii является плиоцен-раннеплейстоценовым интрогрессивным гибридом L. principis-rupprechtii, L. olgensis и L. kamtschatica, преобразованным в позднем плейстоцене гибридизацией с L. gmelinii. Таким образом, L. × lubarskii представляет собой полиморфную группу растений, основная часть ареала которой расположена на северо-востоке Китая и севере п-ова Корея и незначительная часть на Борисовском плато и прилегающей к нему территории в юго-западной части Приморского края [1, 2, 7, 10–12]. Борисовское плато относится к системе Восточно-Маньчжурских гор, наибольшие высоты (600–700 м н. у. м.) находятся на западе плато. Поверхность плато глубоко расчленена долинами рек на отдельные столообразные хребты с крутыми, часто обрывистыми склонами. Лиственница здесь растет небольшими разреженными группировками в составе хвойно-широколиственных лесов [12].
Наблюдаемое разнообразие лиственницы на юге РДВ вызвано процессами интрогрессивной гибридизации вследствие отсутствия покровного оледенения в периоды похолодания в этом регионе и наличия ритмических флуктуаций климата в геологическом прошлом [13, 14]. На территории Приморского края L. olgensis является единственным видом, самостоятельность которого признана многими авторами [2, 3, 7, 8, 15–17], однако сведения о его распространении неоднозначны. Неизменным остается описание основной части ареала L. olgensis, которая находится в юго-восточной части Приморского края, располагаясь вдоль побережья Японского моря и восточных склонов Сихотэ-Алиня между бухтой Валентин на юге и заливом Владимира на севере.
Молекулярные маркеры митохондриального генома широко используются в таксономических, филогеографических и филогенетических исследованиях рода Larix Mill. (Pinaceae). Семериков с соавт. [18] показали, что фрагмент UBC460 митохондриального происхождения способен дифференцировать популяции L. olgensis и восточную расу L. sibirica Ledeb. от западной расы L. sibirica, а также L. gmelinii, L. kaempferi Lamb., L. decidua Mill. благодаря присутствию/отсутствию нуклеотидной вставки размером 266 пн. Этот фрагмент наряду с другими маркерами митохондриальной ДНК (мтДНК) использовался при исследовании популяций L. sibirica, что позволило выявить расположение рефугиумов бореальной растительности последнего ледникового максимума, а также пути и характер послеледниковой реколонизации лиственницы [19, 20]. Анализ полиморфизма UBC460, nad5(1–2r), nad4(3c–4r) и 5'-фланкирующей области гена atpA мтДНК у лиственниц северо-восточной Азии [21] выявил высокое разнообразие митотипов на юге РДВ, где распространены гибриды L. gmelinii, L. olgensis и L. kamtschatica, и только два митотипа у L. cajanderi Mayr. и L. gmelinii. Кроме того, исследование этих авторов показало, что L. kaempferi генетически сходна с популяциями L. kamtschatica, произрастающими на Курильских островах и юге о. Сахалин; популяции северной части Сахалина оказались более близки к континентальным видам; распределение генетической изменчивости может указывать на наличие нескольких рефугиумов во время плейстоценовых ледниковых периодов. Популяционно-генетические исследования L. olgensis из разных частей основного ареала [22–24] подтвердили дифференциацию прибрежных популяций из окрестностей зал. Ольги (locus classicus) и пос. Валентин от всех остальных, которые имели общий митотип с гибридными видами Larix, что указывает на сокращение ареала L. olgensis. Исследование популяций лиственниц на севере Приморского края [25] показало, что митотип L. olgensis был обнаружен только в трех популяциях, в остальных присутствовали в разных соотношениях два “даурских” митотипа, свойственные L. gmelinii и L. cajanderi. Таким образом, достаточно подробно изучены лиственницы Сибири и РДВ, однако генетическое исследование L. × lubarskii, произрастающей на Борисовском плато в юго-западной части Приморского края, в литературе не освещено.
Цель настоящего исследования – изучение генетического разнообразия L. × lubarskii и ее генетических взаимоотношений с другими дальневосточными и восточносибирскими лиственницами на основе анализа изменчивости фрагмента UBC460 и нуклеотидного полиморфизма регионов 5'-фланкирующей области гена atpA и nad4(3c–4r) мтДНК.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследования служили 49 растений из природных популяций Larix юга Приморского края (табл. 1, рис. 1).
Таблица 1.
Происхождение образцов Larix, код популяции, генетическое разнообразие по данным 5'-atpA и nad4(3c–4r) мтДНК, номера доступа нуклеотидных последовательностей митотипов в ENA/GenBank
| Происхождение образца (число образцов) | Код | Разнообразие | Митотип | Номер доступа в GenBank | ||
|---|---|---|---|---|---|---|
| гаплотипическое (SD) | нуклеотидное (SD) | 5'-atpA | nad4(3c–4r) | |||
| Приморский край, Уссурийский р-н, верховье р. Лиственничная, Борисовское базальтовое плато (14) | LUB | 0.582 (0.092) | 0.0003 (0.0003) | H1 | LT992012 | LT992022 |
| H9 | LT992013 | LT992023 | ||||
| H10 | LT992014 | LT992024 | ||||
| Приморский край, Ольгинский р-н, зал. Ольга, каменистый склон, locus classicus L. olgensis (9) | OLG | 0.000 (0.000) | 0.0000 (0.0000) | H1 | LT992012 | LT992022 |
| Приморский край, Лазовский р-н, побережье бух. Большая Тихая (8) | VAL1 | 0.250 (0.180) | 0.0002 (0.0002) | H2 | LT992015 | LT992025 |
| H3 | LT992016 | LT992026 | ||||
| Приморский край, Лазовский р-н, 150 м от побережья бух. Большая Тихая (6) | VAL2 | 0.800 (0.122) | 0.0010 (0.0007) | H3 | LT992016 | LT992026 |
| H4 | LT992017 | LT992027 | ||||
| H5 | LT992018 | LT992028 | ||||
| Приморский край, Лазовский р-н, окр. пос. Валентин (10) | VAL3 | 0.533 (0.180) | 0.0008 (0.0005) | H2 | LT992015 | LT992025 |
| H3 | LT992016 | LT992026 | ||||
| H5 | LT992018 | LT992028 | ||||
| H6 | LT992019 | LT992029 | ||||
| Приморский край, Ольгинский р-н, исток р. Минеральная (2) | MIN | 1.000 (0.500) | 0.0004 (0.0005) | H7 | LT992020 | LT992030 |
| H8 | LT992021 | LT992031 | ||||
| L. sibirica isolate Severobaikalsk34 | SIB | – | – | H11 | FJ572170* | FJ572134* |
| L. gmelinii isolate Hatanga8 | GME | – | – | H12 | FJ572171* | FJ572135* |
| L. gmelinii isolate Vanino48 | – | – | H13 | FJ572172* | FJ572136* | |
| L. gmelinii isolate Kavalerovo12 | – | – | H14 | FJ572173* | FJ572137* | |
| L. gmelinii isolate Chuvansk1 | – | – | H15 | FJ572181* | FJ572138* | |
| L. gmelinii isolate Esso4 | – | – | H16 | FJ572182* | FJ572139* | |
| L. gmelinii isolate Oha1 | – | – | H17 | FJ572184* | FJ572140* | |
| L. kaempferi isolate Japan13 | KAE | – | – | H18 | FJ572169* | FJ572133* |
Рис. 1.
Карта-схема географического положения исследованных популяций Larix на юге Приморского края и частоты митотипов по данным 5'-atpA и nad4(3c–4r) мтДНК.

Индивидуальные препараты тотальной ДНК выделены из свежей хвои по методике [26] с небольшими модификациями. Для амплификации фрагмента UBC460 использовали праймеры, реакционные условия и температурный режим, рекомендованные для этого участка [18]. Изменчивость длины продукта оценивали с помощью электрофореза в 1%-ном агарозном геле. Амплификацию фрагмента 5'-фланкирующей области митохондриального гена atpA (5'-atpA) проводили с разработанными для данного исследования праймерами 5'-GATAAAAGGTCTACGCCAGAA-3' и 5'-CATGACCCCTTAATACCTTTC-3' при следующих условиях: 94°C – 4 мин; 35 циклов: 94°C – 30 с, 57°С – 45 с, 72°C – 2 мин и 72°C – 7 мин. Условия амплификации и секвенирования региона nad4(3c–4r) приведены в работе [24]. Циклическое секвенирование обеих цепей фрагментов ДНК осуществляли с использованием набора флуоресцентно меченых нуклеотидов Big Dye Terminator v. 3.1 (Applied Biosystems, USA). Нуклеотидные последовательности определяли на генетическом анализаторе ABI 3130 (Applied Biosystems, Foster City, USA) в Центре коллективного пользования ФНЦ Биоразнообразия ДВО РАН.
Для каждого образца нуклеотидные последовательности регионов 5'-atpA и nad4(3c–4r) мтДНК выравнивали и объединяли в одну матрицу, используя пакет программ MEGA ver. 6 [27]. Число митотипов, нуклеотидное (π) и гаплотипическое (h) разнообразие, распределение генетической изменчивости внутри и между популяциями (анализ молекулярной дисперсии, AMOVA) рассчитывали с помощью программы Arlequin v. 3.5 [28]. Степень дивергенции между популяциями на основе нуклеотидных замен (KS) определяли в программе DnaSP v. 5.0 [29]. Для уточнения генетических взаимоотношений с другими видами лиственниц Сибири и Дальнего Востока в матрицу данных были добавлены последовательности этих же регионов мтДНК из базы данных GenBank (табл. 1). Генеалогические связи митотипов анализировали методом Reduced Median (RM) в программе Network 5.0 [30].
Нуклеотидные последовательности 5'-atpA и nad4(3c–4r) исследуемых лиственниц юга Приморского края депонированы в базу данных ENA/EMBL/GenBank под номерами доступа LT992012–LT992031.
РЕЗУЛЬТАТЫ
Полиморфизм фрагмента UBC460 связан с изменчивостью его длины. В популяциях лиственницы c Борисовского плато (LUB) и побережья бух. Большой Тихой (VAL1 и VAL2) обнаружены два варианта длины фрагмента – длинный и короткий, различающиеся присутствием и отсутствием вставки 266 пн соответственно. Частота встречаемости вариантов длины фрагмента UBC460 в популяциях лиственницы Приморского края представлена в табл. 2.
Таблица 2.
Частота встречаемости вариантов длины фрагмента UBC460 в популяциях Larix юга Приморского края
| Фрагмент | Частота встречаемости, % | |||||
|---|---|---|---|---|---|---|
| LUB | OLG* | VAL1 | VAL2 | VAL3* | MIN | |
| Длинный | 42.9 | 100 | 87.5 | 33.3 | 80 | 0 |
| Короткий | 57.1 | 0 | 12.5 | 66.7 | 20 | 100 |
* Данные, полученные нами ранее [24].
Определены нуклеотидные последовательности фрагмента 5'-atpA у всех 49 растений исследуемых популяций и третьего интрона гена nad4 у представителей популяции LUB и одного образца MIN. В матрицу данных были добавлены полученные нами ранее [24] последовательности nad4(3c–4r) образцов популяций OLG, VAL1, VAL2, VAL3 и MIN. Длина 5'-atpA и nad4(3c–4r) была одинаковой у всех образцов и составила 880 и 1762 пн соответственно. Из 2642 пн объединенной матрицы двух регионов 2635 были мономорфными, из семи выявленных нуклеотидных замен (трансверсий) шесть были информативны согласно методу максимальной экономии. Во фрагменте 5'-atpA в позиции 304 выявлена нуклеотидная замена C → A, которая маркирует все образцы популяций VAL1, VAL2 и VAL3, а в позиции 557 присутствует нуклеотидная замена T → G, которая маркирует образцы MIN. У представителей L. × lubarskii в двух регионах мтДНК выявлено всего три нуклеотидные замены, не являющиеся маркерными.
Параметры генетической изменчивости исследованных популяций представлены в табл. 1. Популяция LUB L. × lubarskii характеризуется средним уровнем генетического разнообразия (h = 0.582, π = 0.0003). В целом для лиственниц юга Приморского края свойственно высокое гаплотипическое (0.790) и низкое нуклеотидное (0.0007) разнообразие. Согласно результатам AMOVA, генетическая изменчивость распределена практически в равных долях на внутри- и межпопуляционную изменчивость (ΦST = 0.48109, P < 0.0001). Выявлена довольно низкая нуклеотидная дивергенция между исследуемыми популяциями (табл. 3). Наименьшее значение KS отмечено у пары популяций OLG–LUB, а наибольшие значения KS имеет выборка MIN со всеми популяциями. Кроме того, только между MIN и всеми другими популяциями выявлены фиксированные нуклеотидные различия.
Таблица 3.
Нуклеотидная дивергенция (KS) между исследуемыми популяциями Larix по данным 5'-atpA и nad4(3c–4r) мтДНК
| Код | LUB | OLG | VAL1 | VAL2 | VAL3 | MIN |
|---|---|---|---|---|---|---|
| LUB | – | 0.571(0) | 1.554(0) | 2.810(0) | 2.043(0) | 3.929(1) |
| OLG | 0.00022 | – | 1.000(0) | 3.000(0) | 1.700(0) | 4.500(4) |
| VAL1 | 0.00059 | 0.00038 | – | 3.250(0) | 1.250(0) | 5.250(3) |
| VAL2 | 0.00106 | 0.00114 | 0.00123 | – | 3.100(0) | 2.833(1) |
| VAL3 | 0.00077 | 0.00064 | 0.00047 | 0.00117 | – | 4.800(1) |
| MIN | 0.00149 | 0.00170 | 0.00199 | 0.00107 | 0.00182 | – |
Примечание. Выше диагонали – среднее число нуклеотидных различий между популяциями (в скобках число фиксированных различий), ниже диагонали – среднее число нуклеотидных замен на один сайт между популяциями. Код популяции см. в табл. 1.
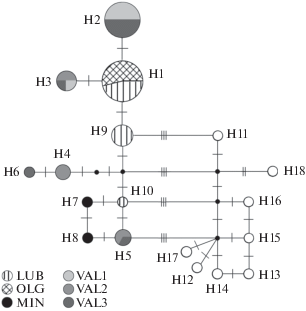
Анализ объединенной матрицы нуклеотидных последовательностей 5'-atpA и nad4(3c–4r) представителей популяций лиственниц юга Приморского края и других видов Larix Сибири и РДВ из базы GenBank выявил 18 митотипов. В популяции L. × lubarskii обнаружено три митотипа (H1, H9, H10), из них H1 с частотой встречаемости 50.7% оказался общим c популяцией OLG L. olgensis (locus classicus) (рис. 1). В VAL1 и MIN выявлено по два митотипа (H2, H3 и H7, H8 соответственно), три (H3, H4, H5) в VAL2 и четыре (H2, H3, H5, H6) в VAL3. На генеалогической сети все митотипы представителей приморских популяций (H1–H10) в основном находятся в одном мутационном шаге от соседнего и не подразделяются в соответствии с их популяционной принадлежностью (рис. 2). Образцы видов Larix из базы GenBank имеют индивидуальные митотипы (Н11–Н18). Общих митотипов у приморских популяций и других видов лиственниц не выявлено. Наличие альтернативных связей между митотипами могут указывать на высокий уровень рекомбинации или гомоплазии (повторных и обратных мутаций в мтДНК).
ОБСУЖДЕНИЕ
Настоящее исследование L. × lubarskii и популяций из ареала L. olgensis подтверждает и дополняет ранее полученные данные по распространению у видов Larix дифференцирующего фрагмента UBC460 митохондриального происхождения [20, 21, 24, 25]. Частота встречаемости длинного фрагмента UBC460, зафиксированного у L. olgensis из зал. Ольги, к югу по побережью Японского моря постепенно уменьшается и достигает 42.9% в популяциях Юго-Западного Приморья (табл. 2). В северных районах Приморского края и на остальной территории РДВ, включая Курильские острова и Сахалин, у лиственниц фиксирован короткий фрагмент, за исключением двух прибрежных популяций Сихотэ-Алинского биосферного заповедника и двух популяций из Забайкальского края и Амурской области, где отмечено редкое присутствие длинного фрагмента [19, 21]. Такое географическое распределение двух вариантов длины фрагмента UBC460 согласуется с мнением о существовании в южной части РДВ гибридных лиственниц, образованных в результате интрогрессивной гибридизации видов L. olgensis, L. gmelinii, L. cajanderi и L. kamtschatica [8].
Впервые по результатам анализа нуклеотидного полиморфизма 5'-фланкирующей области гена atpA и nad4(3c–4r) мтДНК дана генетическая оценка состояния природной популяции L. × lubarskii. Этот таксон характеризуется средним уровнем гаплотипического и низким уровнем нуклеотидного разнообразия (табл. 1). Присутствие общего митотипа H1 у L. × lubarskii и L. olgensis (рис. 1 и 2) указывает на их генетическое родство, которое ранее предполагалось на основании сходства морфологических признаков [3–8], и свидетельствует об относительно недавней дивергенции этих таксонов. Наблюдаемое отсутствие изменчивости мтДНК в популяции OLG L. olgensis (locus classicus) может быть результатом эффекта основателя, проявившегося при вытеснении вида более конкурентоспособной растительностью на периферию ареала к морскому побережью. Наличие в генеалогической сети альтернативных связей не позволяет дать однозначного ответа о генетических отношениях L. × lubarskii с другими видами Larix (рис. 2). Митотипы Н12–Н16 L. gmelinii, как одного из предполагаемых родительских видов лиственницы Любарского, оказались намного дальше от митотипов Н1, Н9, Н10 L. × lubarskii, чем митотип Н11 L. sibirica. Сходство, казалось бы, географически отдаленных лиственниц вполне вероятно, поскольку L. sibirica по ряду морфологических признаков и по данным аллозимного анализа чрезвычайно близка с L. olgensis [7, 32], которая в свою очередь является близким таксоном с L. × lubarskii. Полученные результаты согласуются с палеоботаническими исследованиями, которые указывают на существование в раннем плейстоцене в южном Приморье лиственницы Laricioxylon aff. sichotealinense Blokh., схожей по некоторым характеристикам анатомии древесины с L. sibirica и L. gmelinii [33, 34]. В этот же период L. olgensis имела более широкое распространение, достигая, вероятно, Восточно-Манчжурских гор, что могло способствовать образованию гибридных форм лиственниц, одной из которых стала L. × lubarskii.
Таким образом, результаты анализа трех митохондриальных маркеров выявили более близкое родство L. × lubarskii с L. olgensis и L. sibirica, чем с L. gmelinii.
Авторы благодарят В.Ю. Баркалова за предоставленный растительный материал, К.В. Киселева за помощь в разработке праймеров для амплификации ПЦР-продукта фрагмента 5'-фланкирующей области митохондриального гена atpA и Д.М. Атопкина за помощь в подготовке проб для секвенирования.
Работа поддержана грантами Президиума РАН (проекты № 15-I-6-030 и 15-I-6-080).
Список литературы
Сукачев В.Н. О двух новых ценных для лесного хозяйства древесных породах // Тр. и исслед. по лесн. хоз-ву и лесн. пром-сти. 1931. Вып. 10. С. 12–18.
Колесников Б.П. К систематике и истории развития лиственниц секции Pauciseriales Patshke: Материалы по истории флоры и растительности СССР. М.; Л.: Изд-во АН СССР, 1946. Вып. 2. С. 21–86.
Недолужко В.А. Конспект дендрофлоры российского Дальнего Востока. Владивосток: Дальнаука, 1995. 208 с.
Farjon A. World Checklist and Bibliography of Conifers. 2nd. ed. Kew: Royal Botanic Gardens, 2001. 309 p.
Farjon A. A Handbook of the World’s Conifers. Leiden, Boston: BRILL, 2010. 526 p.
Chang C.S., Kim H., Chang K.S. Provisional Checklist of Vascular Plants for the Korea Peninsula Flora (KPF). DESIGNPOST, 2014. P. 1–660.
Дылис Н.В. Лиственница Восточной Сибири и Дальнего Востока. М.: Изд-во АН СССР, 1961. 209 с.
Бобров Е.Г. История и систематика лиственниц // Комаровские чтения. Вып. 25. Л.: Наука, 1972. 96 с.
Урусов В.М. Гибридизация в природной флоре Дальнего Востока и Сибири (причины и перспективы использования). Владивосток: Дальнаука, 2002. 230 с.
Урусов В.М., Лобанова И.И., Варченко Л.И. Хвойные деревья и кустарники российского Дальнего Востока: география и экология. Владивосток: Дальнаука, 2004. 111 с.
Шишкин И.К. К познанию ольгинской лиственницы (L. olgensis A. Henry) // Бот. журн. СССР. 1933. Т. 18. Вып. 3. С. 162–207.
Гуков Г.В. Лиственницы и лиственничные леса российского Дальнего Востока. Владивосток: ГТС ДВО РАН, 2009. 350 с.
Голубева Л.В., Караулова Л.П. Растительность и климатостратиграфия плейстоцена и голоцена юга Дальнего Востока СССР. М.: Наука, 1983. 144 с.
Крестов П.В., Баркалов В.Ю., Омелько А.М. и др. Реликтовые комплексы растительности современных рефугиумов Северо-Восточной Азии // Комаровские чтения. 2009. Вып. 56. С. 5–63.
Сукачев В.Н. К истории развития лиственниц // Лесное дело. М.; Л.: Новая деревня, 1924. С. 12–44.
Коропачинский И.Ю. Голосеменные // Сосудистые растения советского Дальнего Востока. Т. 4. Л.: Наука, 1989. С. 9–25.
Потокин А.Ф., Иванов С.А., Орлова Л.В. Разнообразие лиственничников из Larix olgensis A. Henry в средней и южной части Приморского края // Труды XIII съезда Русск. ботан. о-ва. Т. 2. Тольятти, 2013. С. 296–297.
Semerikov V.L., Vendramin G.G., Sebastiani F., Lascoux M. RAPD-derived, PCR-based mitochondrial markers for Larix species and their usefulness in phylogeny // Conserv. Genet. 2006. V. 7. № 4. P. 621–625.
Семериков В.Л., Полежаева М.А. Структура изменчивости митохондриальной ДНК лиственниц Восточной Сибири и Дальнего Востока // Генетика. 2007. Т. 43. № 6. С. 782–789.
Semerikov V.L., Semerikova S.A., Polezhaeva M.A. et al. Southern montane populations did not contribute to the recolonisation of West Siberian Plain by Siberian larch (Larix sibirica): a range-wide analysis of cytoplasmic markers // Mol. Ecol. 2013. V. 22. № 19. P. 4958–4971. doi 10.1111/mec.12433
Polezhaeva M.A., Lascoux M., Semerikov V.L. Cytoplasmic DNA variation and biogeography of Larix Mill. in Northeast Asia // Mol. Ecol. 2010. V. 19. P. 1239–1252. doi 10.1111/j.1365-294X.2010.04552.x
Vasyutkina E.A., Adrianova I.Yu., Kozyrenko M.M. et al. Genetic differentiation of larch populations from the Larix olgensis range and their relationships with larches from Siberia and Russian Far East // For. Sci. Technol. 2007. V. 3. № 2. P. 132–138.
Васюткина Е.А., Лауве Л.С., Реунова Г.Д., Журавлев Ю.Н. Хромосомный мозаицизм лиственницы Larix olgensis A. Henry в Приморском крае // Изв. РАН. Сер. биол. 2010. № 6. С. 670–675.
Васюткина Е.А., Реунова Г.Д., Тупикин А.Е., Журавлев Ю.Н. Изменчивость митохондриальной ДНК лиственницы ольгинской (Larix olgensis A. Henry) в Приморском крае России // Генетика. 2014. Т. 50. № 3. С. 291–298. doi 10.7868/ S0016675814030102
Полежаева М.А., Семериков В.Л., Пименова Е.А. Генетическое разнообразие лиственницы на севере Приморского края и границы распространения Larix olgensis A. Henry // Генетика. 2013. Т. 45. № 5. С. 580–586. doi 10.7868/S0016675813030144
Isabel N., Tremblay L., Michaud M. et al. RAPDs as an aid to evaluate the genetic integrity of somatic embryogenesis derived populations of Picea mariana (Mill.) B.S.P. // Theor. Appl. Genet. 1993. V. 86. P. 81–87.
Tamura K., Stecher G., Peterson D. et al. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0 // Mol. Biol. Evol. 2013. V. 30. P. 2725–2729.
Excoffier L., Lischer H.E.L. Arlequin suite ver. 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. 2010. V. 10. P. 564–567. doi 10.1111/j.1755-0998.2010.02847.x
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452. doi 10.1093/bioinformatics/btp187
Bandelt H.-J., Forster P., Sykes B.C., Richards M.B. Mitochondrial portraits of human populations // Genetics. 1995. V. 141. P. 743–753.
Corander J., Marttinen P., Sirén J., Tang J. Enhanced Bayesian modelling in BAPS software for learning genetic structures of populations // BMC Bioinformat. 2008. № 9. P. 539. doi 10.1186/1471-2105-9-539
Semerikov V.L., Semerikov L.F., Lascoux M. Intra- and interspecific allozyme variability in Eurasian Larix Mill. species // Heredity. 1999. V. 82. P. 193–204.
Blokhina N.I., Bondarenko O.V. Fossil plant assemblages from the Pliocene of southern Primory’e Region (Russian Far East): implications for reconstruction of plant communities and their environments // Acta Palaeobot. 2011. V. 51. № 1. P. 19–37.
Bondarenko O.V., Blokhina N.I., Bruch A.A. et al. Quantification of Calabrian vegetation in Southern Primory’e (Far East of Russia) using multiple proxies // Palaeogeogr. Palaeoclimatol. Palaeoecol. 2017. V. 467. P. 253–264. http://dx.doi.org/10.1016/j.palaeo.2016.09.017.
Дополнительные материалы отсутствуют.