Генетика, 2019, T. 55, № 4, стр. 398-405
Филогенетический анализ штаммов Yersinia pestis кавказского подвида из очагов Кавказа и Закавказья по данным полногеномного секвенирования
К. А. Никифоров 1, *, **, Ж. В. Альхова 1, Л. М. Куклева 1, Е. А. Нарышкина 1, Е. Г. Оглодин 1, Г. А. Ерошенко 1, В. В. Кутырев 1
1 Российский научно-исследовательский противочумный институт “Микроб”
410005 Саратов, Россия
* E-mail: rusrapi@microbe.ru
** E-mail: nikiforov6666666@mail.ru
Поступила в редакцию 24.04.2018
После доработки 05.07.2018
Принята к публикации 22.08.2018
Аннотация
Нами исследована популяционная структура штаммов Yersinia pestis кавказского подвида из природных очагов чумы, расположенных на Кавказе и в Закавказье. По данным полногеномного SNP анализа двадцати восьми штаммов кавказского подвида, семь из которых секвенированы нами, и с учетом 1625 выявленных коровых SNPs определено наличие нескольких филогенетических линий и популяций Yersinia pestis ssp. caucasica, соответствующих географическому распространению штаммов. Штаммы Y. pestis кавказского подвида включают три отдельных кластера, первый из которых представлен штаммами из Восточно-Кавказского высокогорного очага чумы. В их геноме выявлено 80 уникальных SNPs, что свидетельствует об их наибольшей древности и о давней дивергенции этой ветви от общего ствола эволюции кавказских штаммов. Геномы штаммов двух других кластеров также отличаются большим количеством индивидуальных SNPs – 32 и 36 соответственно. Один из них включает штаммы из Присеванского горного, Зангезуро-Карабахского горного и Прираксинского низкогорного очагов с выделением в пределах кластера трех отдельных субкластеров: присеванского (IIa), зангезуро-карабахского (IIb) и зангезуро-карабахско-приараксинского (IIc). Еще один, третий кластер кавказского подвида состоит из штаммов северо-западной части Кавказского нагорья (Гюмрийский и Джавахетско-Ахалкалакский очаги). С помощью ПЦР с гибридизационно-флуоресцентным учетом результатов и полногеномного секвенирования доказана принадлежность двух клинических штаммов Y. pestis к кавказскому подвиду, что подтверждает способность этого подвида вызывать чуму у людей.
Бактерия Yersinia pestis – возбудитель особо опасной природно-очаговой инфекции, очаги которой встречаются на многих континентах. В Российской Федерации и других странах СНГ расположено 45 природных очагов, которые часто напоминают о себе проявлением эпизоотической и эпидемической активности. Так, в 2013 г. в Республике Кыргызстан произошел случай заражения чумой человека с летальным исходом [1]. В 2014 и 2016 гг. в Горно-Алтайском высокогорном очаге в Республике Алтай зарегистрированы случаи заражения чумой человека [2]. Заболевания чумой отмечают и в сопредельных с Россией государствах – Монголии, Китае. Помимо важного практического значения изучение возбудителя чумы представляет и значительный фундаментальный интерес, поскольку Y. pestis является примером быстрой эволюции высоковирулентной бактерии, которая произошла от почвенной бактерии Y. pseudotuberculosis в основном благодаря приобретению двух плазмид патогенности (pFra и pPst) и потери части хромосомных генов [3, 4]. По широко распространенной среди исследователей чумы в России и других странах СНГ подвидовой классификации штаммы Y. pestis делят на пять подвидов – основной и четыре неосновных, включающих кавказский, алтайский, гиссарский и улегейский. Штаммы основного подвида высоковирулентны и эпидемически значимы, штаммы неосновных подвидов избирательно вирулентны, в основном для мелких мышевидных грызунов. Неосновные подвиды дивергировали от общего ствола эволюции Y. pestis, обозначаемого как “0”, на ранних этапах (до отхождения ветвей “1”, “2”, “3” и “4” основного подвида) и поэтому их по филогенетической номенклатуре обозначают как 0.PE [5]. Недавно на основе полученных данных по полногеномному секвенированию штаммов Y. pestis из очагов России и стран СНГ нами было предложено усовершенствование подвидовой классификации возбудителя чумы, отражающее современную популяционного структуру вида. В составе вида Y. pestis выделено семь подвидов: основной, кавказский, тибетский, ангольский, центральноазиатский, цинхайский, улегейский [6].
Для некоторых неосновных подвидов зарегистрированы случаи заражения чумой человека [7]. В том числе известны случаи чумы, вызванные кавказским подвидом Y. pestis ssp. caucasica, соответствующим по современной филогенетической номенклатуре ветви 0.PE2 [8, 9]. Кавказский подвид – одна из наиболее древних ветвей эволюции Y. pestis, по ряду признаков близкая предшественнику, Y. pseudotuberculosis [5–7]. Штаммы кавказского подвида сохранили способность к ферментации диагностически значимых субстратов – рамнозы, мелибиозы, арабинозы, глицерина, а также к редукции нитратов. Отличительным генетическим признаком кавказских штаммов является отсутствие плазмиды пестициногенности pPst (синонимы pPla, pPCP1), кодирующей видовой бактериоцин – пестицин и фактор патогенности – активатор плазминогена. В то же время в их геноме присутствуют две другие резидентные для Y. pestis плазмиды – кальцийзависимости (pCad) и синтеза фракции I (pFra).
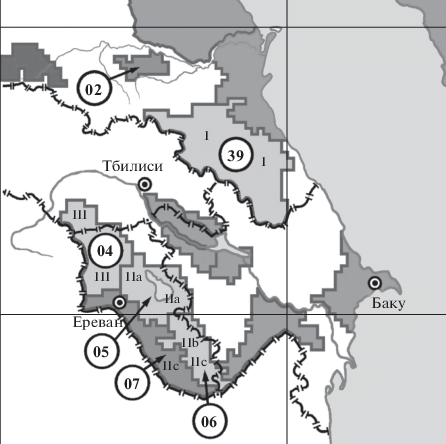
Штаммы кавказского подвида встречаются только в очагах Кавказа и Закавказья, расположенных на территории России, Армении, Азербайджана и Грузии, и не встречаются в других регионах мира. Они циркулируют в Восточно-Кавказском высокогорном (39), Ленинаканском горном (04), Присеванском горном (05), Зангезуро-Карабахском горном (06), Приараксинском низкогорном (07) очагах чумы (рис. 1). Сообщалось о случае выделения штамма кавказского подвида в Терско-Сунженском низкогорном очаге [10]. Ранее проведенный молекулярно-генетический анализ штаммов Y. pestis кавказского подвида с помощью методов MLVA25-, CRISPR- и SNP-типирования выявил наличие трех независимых филогенетических групп этих штаммов, соответствующих природным очагам чумы – 39, 04 и группе очагов 05-07. Однако только метод SNP-типирования позволил выделить из группы штаммов, выделенных в очагах 05-07, отдельную ветвь изолятов из очага 05. Отдельную и наиболее древнюю филогенетическую группу составили штаммы из Восточно-Кавказского высокогорного очага в России, для которых была выявлена большая генетическая гетерогенность [11, 12].
Рис. 1.
Распространение штаммов Yersinia pestis кавказского подвида в очагах чумы Кавказа и Закавказья: Ленинаканский горный (04), Присеванский горный (05), Зангезуро-Карабахский горный (06), Приараксинский низкогорный (07), Восточно-Кавказский высокогорный (39) очаги. В скобках приведены номера очагов по принятой в России и других странах СНГ классификации природных очагов чумы. Римскими цифрами (I, IIa, IIb, IIc, III) указано распространение штаммов кавказского подвида различных филогенетических линий в очагах.
Штаммы кавказского подвида распространены в различных природных биоценозах низкогорного, горного и высокогорного типов. Носителями кавказских штаммов являются полевки – Microtus arvalis, Microtus socialis, Microtus majori, Chionomis gud и их блохи Xenopsylla conformis, Nosopsyllus iranus, Stenoponia tripectinata [10]. В Приараксинском низкогорном одновременно циркулируют штаммы кавказского и основного подвидов. Поскольку штаммы этих двух подвидов отличаются по вирулентности и эпидемической значимости, нами был разработан способ их дифференциации с применением современных молекулярно-генетических методов [13].
Цель нашей работы заключалась в изучении современной популяционной структуры кавказского подвида возбудителя чумы и определении географических регионов распространения штаммов отдельных филогенетических линий и популяций в очагах Кавказа и Закавказья с учетом данных проведенного нами полногеномного секвенирования штаммов Y. pestis ssp. caucasica.
МАТЕРИАЛЫ И МЕТОДЫ
Штаммы Y. pestis, культивирование, выделение ДНК. Штаммы Y. pestis, использованные в этой работе, получены из Государственной коллекции патогенных бактерий РосНИПЧИ “Микроб”. Штаммы выращивали на LB-агаре и в LB-бульоне (pH 7.2) при 28°С в течение 24–48 ч. Анализ культурально-морфологических и дифференциально-диагностических признаков проводили в соответствии со стандартными методами лабораторной диагностики чумы [14]. ДНК штаммов выделяли с помощью набора PureLink™ Genomic DNA Mini Kit Invitrogen производства Thermo Fisher Scientific.
Проведение ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени. Дифференциацию штаммов Y. pestis основного и кавказского подвида выполняли методом ПЦР с гибридизационно-флуоресцентным учетом результатов в режиме реального времени (ПЦР-РВ). Реакцию проводили в одной ПЦР смеси в амплификаторе Rotor-Gene 6000 (Сorbett Research, Австралия). Условия проведения ПЦР-РВ были следующими: 1 цикл 95°С 10 мин; 40 циклов: 95°С 15 с, 57°С 40 с, 72°С 15 с. На этапе отжига праймеров проводили учет флуоресценции, уровень порога равнялся 0.05, пороговое значение Ct – менее 30.
Полногеномное секвенирование, поиск SNPs, филогенетический анализ, построение дендрограмм. Получение полногеномных нуклеотидных последовательностей штаммов Y. pestis осуществляли с использованием системы Ion PGM (Life technologies, США). Обработку данных и проведение сборки геномов de novo выполняли с помощью программы Ion Torrent Suit software package 3.4.2 и Newbler gsAssembler 2.6. Полученные последовательности ридов собирали в геномы, покрытие которых по геному референсного штамма СО92 (номер доступа в GenBank NC_003143.1) составило около 98.84% с 60-кратным прочтением. Средняя длина собранного генома составила 4.64 Мпн.
Полногеномный SNP-анализ и поиск маркерных SNPs проводили с помощью программы Wombac 2.0. Дендрограмму с применением алгоритма Maximum Likelihood строили, используя программу PhyML 3.1 на основе модели HKY85. Дендрограммы визуализировали, применяя программу FigTree 1.4.3. Бутстреп-анализ проводили, используя 500 случайных выборок.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В этой работе нами проведено полногеномное секвенирование семи штаммов Y. pestis ssp. caucasica из очагов Кавказа и Закавказья (табл. 1, отмечены звездочкой). Для анализа популяционной структуры кавказского подвида использованы полногеномные последовательности еще 21 штамма кавказского подвида из базы данных NCBI GenBank, а также последовательности девяти штаммов Y. pestis, относящихся к другим филогенетическим ветвям возбудителя чумы [15, 16].
Таблица 1.
Штаммы Y. pestis, полногеномные последовательности которых использованы в работе
| Штамм Y. pestis | Происхождение (очаг), № доступа в GenBank | Подвид, филогенетическая ветвь |
|---|---|---|
| 835 В.Р.С.* | Ленинаканский, 1958. LYOJ00000000 | Кавказский, 0.PE2 |
| 3544 Арм* | Ленинаканский, 1968. LZNH00000000 | Кавказский, 0.PE2 |
| 3551 Арм* | Присеванский, 1979. MBSJ00000000 | Кавказский, 0.PE2 |
| М-986* | Зангезуро-Карабахский, 1973. LYMO00000000 | Кавказский, 0.PE2 |
| КМ874* | Зангезуро-Карабахский, 1984. LZTG00000000 | Кавказский, 0.PE2 |
| С-741* | Восточно-Кавказский, 1998. LPTX00000000 | Кавказский, 0.PE2 |
| 1393Г * | 1979. PVMU01 | Кавказский, 0.PE2 |
| Штаммы из NCBI GenBank | ||
| SCPM-O-B-6536 | Ленинаканский, NZ_LIZE00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-7761 | Ленинаканский, NZ_LIYQ00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6990 | Ленинаканский, NZ_LIYU00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-7832 | Присеванский, LIZB00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6540 | Присеванский, LIZF00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6984 | Зангезуро-Карабахский, LIYZ00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6974 | Зангезуро-Карабахский, NZ_LIYX00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6757 | Приараксинский, NZ_LIYY00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6300 | Приараксинский, LIZC00000000.1 | Кавказский, 0.PE2 |
| SCPM-O-B-6904 | Восточно-Кавказский, NZ_LIYP00000000.1 | Кавказский, 0.PE2 |
| 14735 | Армения, NZ_AYLS00000000.1 | Кавказский, 0.PE2 |
| 1522 | Армения, Ширакская обл., NZ_CP006758.1 | Кавказский, 0.PE2 |
| 3067 | Грузия, Ниноцминда, NZ_CP006754.1 | Кавказский, 0.PE2 |
| 1670 | Грузия, Ниноцминда, NZ_AYLR00000000.1 | Кавказский, 0.PE2 |
| 8787 | Грузия, Ниноцминда, NZ_CP006748.1 | Кавказский, 0.PE2 |
| 3770 | Грузия, Ниноцминда, NZ_CP006751.1 | Кавказский, 0.PE2, |
| G8786 | Грузия, NZ_ADSG00000000.1 | Кавказский, 0.PE2 |
| 1412 | Грузия, Ниноцминда, NZ_CP006783.1 | Кавказский, 0.PE2 |
| 1413 | Грузия, Ниноцминда, NZ_CP006762.1 | Кавказский, 0.PE2 |
| Pestoides G | NZ_CP010247.1 | Кавказский, 0.PE2 |
| Pestoides F | NC_009381.1 | Кавказский, 0.PE2 |
| Pestoides A | NZ_ACNT00000000.1 | Алтайский, 0.PE4 |
| SCPM-O-B-6304 | Гиссарский, NZ_LIYS00000000.1 | Гиссарский, 0.PE4 |
| CO92 | США, NC_003143.1 | Основной, 1.ORI |
| KIM10 | Иран/Курдистан, 1968. NC_004088.1 | Основной, 2.MED |
| MGJZ12 | Монголия, NZ_ADSV00000000.1 | Основной, 4.ANT |
| MGJZ6 | Монголия, NZ_ADSX00000000.1 | Основной, 3.ANT |
| 91001 | Китай, Внутренняя Монголия, NC_005810.1 | Microtus, 0.PE4 |
| А-1815 | Таласский высокогорный, LPTY00000000.1 | Таласский, 0.PE4 |
| 620024 | Китай, Цинхай, NZ_ADPM00000000.1 | Тибетский, 0.PE7 |
Филогенетическое исследование кавказского подвида выполняли методом полногеномного SNP-анализа на основе выявления коровых SNPs в последовательностях взятых в работу штаммов. Всего выявлено 1625 коровых SNPs, с учетом которых на основе алгоритма Maximum Likelihood построена дендрограмма штаммов кавказского подвида (рис. 2).
Рис. 2.
Филогения Y. рestis кавказского подвида из природных очагов Кавказа и Закавказья по данным полногеномного SNP-анализа штаммов разных филогенетических ветвей с учетом 1625 выявленных SNPs (программа PhyML 3.1, алгоритм Maximum Likelihood). На дендрограмме представлен фрагмент филогенетического дерева, включающий только штаммы кавказского подвида.
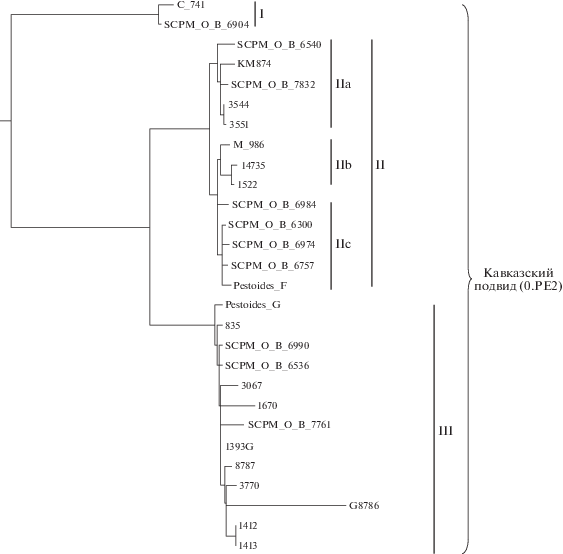
Как видно из дендрограммы, штаммы Y. pestis кавказского подвида разделились на три отдельных кластера. Первый, далеко отстоящий кластер, представлен двумя штаммами из Восточно-Кавказского высокогорного очага чумы. Этот кластер (I) дивергировал от общего ствола эволюции кавказского подвида раньше двух других кластеров и, по-видимому, достаточно давно. Об этом свидетельствует наличие 80 уникальных SNPs, не встречающихся у других штаммов Y. pestis. Один из двух штаммов из этого кластера был секвенирован нами. Он выделен в 1998 г. от блох обыкновенной полевки. Сведения по времени происхождения второго штамма (SCPM-O-B-6904) в базе данных GenBank не приводятся. Однако штаммы отличаются друг от друга и содержат в геноме 8 и 1 индивидуальных SNPs соответственно. Эти данные свидетельствуют о гетерогенности популяции штаммов кавказского подвида в Восточно-Кавказском высокогорном очаге, о чем сообщали ранее другие исследователи [11, 12].
Два других кластера (II и III) имеют 74 общих SNPs, но отличаются друг от друга также большим количеством индивидуальных SNPs. Все штаммы II кластера содержат в геноме 32 общих SNPs, однако далее их ветвь четко разделяется на два отдельных субкластера, в которых штаммы группируются по филогеографической принадлежности. В первый подкластер (IIa) входят секвенированные нами штаммы КМ874 (Зангезуро-Карабахский горный, 1984), 3544 Арм (Ленинаканский горный, 1968), 3551 Арм (Присеванский горный, 1979), а также два штамма, SCPM-О-B-6540 и SCPM-О-B-7832, из базы GenBank, выделенные в Присеванском горном очаге (рис. 2). Штаммы этого подкластера составляют отдельную линию эволюции кавказского подвида. Принадлежность штаммов из подкластера к трем разным очагам не противоречит их принадлежности к одной линии эволюции, поскольку все три очага являются мезоочагами, входящими в состав Закавказского высокогорного очага, расположенного в горах Малого Кавказа. Закавказский высокогорный очаг занимает зону центральной, северо-западной и юго-восточной частей Закавказского нагорья, территориально расположенных в Армении, Грузии и Азербайджане [10]. Преобладание в этом подкластере штаммов из Присеванского очага (три из пяти штаммов), а также отсутствие присеванских штаммов в других кластерах свидетельствуют о преимущественном распространении штаммов этой филогенетической линии на территории Присеванского горного очага и в пограничных с ним районах Ленинаканского и Зангезуро-Карабахского очагов. Распространение штаммов подкластера IIa не только на территории Присеванского горного очага, но в пограничных с ним районах других мезоочагов Закавказского высокогорного очага выявлено нами впервые в отличие от данных других исследователей [11].
Второй подкластер в составе II кластера включает еще две линии эволюции кавказских штаммов. В первую линию (IIb) входит секвенированный штамм М-986 из Зангезуро-Карабахского горного очага и еще два штамма (14 735 и 1522 из GenBank), выделенные в Армении. Эта линия имеет два уникальных SNPs, в то время как у второй линии (IIc) в геноме присутствуют шесть индивидуальных SNPs (рис. 2). Ко второй линии относятся штаммы из Зангезуро-Карабахского (SCPM-О-B-6984, SCPM-О-B-6974) и Приараксинского очагов (SCPM-О-B-6300, SCPM-О-B-6757), расположенных в Армении и Азербайджане. То, что на территории этих двух очагов циркулируют штаммы, относящиеся к одной филогенетической линии, также не удивительно, поскольку оба очага расположены рядом друг с другом и имеют протяженную общую границу. К этой же линии принадлежит штамм Pestoides F из GenBank, что позволяет предположить происхождение штамма с этой же территории.
Таким образом, в Закавказском высокогорном очаге и прилегающем Приараксинском низкогорном очаге циркулируют штаммы трех отдельных филогенетических линий, которые условно можно обозначить как присевансакая (субкластер IIa), зангезуро-карабахская (IIb) и зангезуро-карабахско-приараксинская (IIc), кластеризующихся по филогеографическому принципу. Большее генетическое разнообразие штаммов кавказского подвида в Закавказском высокогорном очаге по сравнению с данными предшествующих исследований и наличие нового субкластера штаммов IIb нам удалось установить благодаря увеличению числа включенных в филогенетический анализ штаммов из этого очага, в том числе и секвенированных нами в рамках этого исследования.
В составе кавказского подвида отдельно расположен третий кластер штаммов (III), который имеет 36 уникальных SNPs. В основании кластера лежат штаммы, выделенные в Ленинаканском горном очаге чумы, включая клинический штамм 835 “В.Р.С.”, выделенный в 1958 г., а также штаммы SCPM-О-B-6990 и SCPM-О-B-6536. Как следует из дендрограммы, последовательная смена генотипов штаммов из Ленинаканского очага привела в дальнейшем к формированию грузинской ветви кавказских штаммов, включающей штаммы 3067, 1670, 8787, 3770, 1393G, G8786, 1412, 1413. В соответствии с последними изменениями в классификации очагов Ленинаканский очаг получил обозначение Гюмрийский (Армения), который составляет одно целое с Джавахетско-Ахалкалакским очагом, расположенным в Грузии [17]. В эту же грузинскую ветвь вошел секвенированный нами штамм Y. pestis 1393G, происхождение которого точно не известно. В целом к кластеру III относятся штаммы кавказского подвида из северо-западной части Закавказского нагорья.
Таким образом, популяционная структура кавказского подвида включает несколько отдельных популяций. Безусловно, отдельную популяцию и независимую ветвь эволюции кавказских штаммов составляют штаммы из Восточно-Кавказского высокогорного очага. Эта ветвь рано дивергировала от общего ствола эволюции кавказского подвида. В геномах восточно-кавказских штаммов содержится значительное число уникальных SNPs, отделяющих эту ветвь от других кавказских штаммов.
Другая крупная ветвь эволюции кавказских штаммов (кластер II) включает три отличающиеся популяции, относящиеся к трем филогенетическим линиям. Линия IIa включает преимущественно штаммы из Присеванского горного очага, линия IIb – преимущественно штаммы из Зангезуро-Карабахского очага. Третья линия IIc включает штаммы из двух юго-восточных очагов – Зангезуро-Карабахского горного и Прираксинского низкогорного. Еще одно отдельное направление эволюции (кластер III) представлено штаммами из северо-западной части Кавказского нагорья (Лениканский очаг – современные Гюмрийский и Джавахетско-Ахалкалакский очаги).
В целом выявлено значительное генетическое разнообразие штаммов Y. pestis ssp. caucasica, связанное с древностью этого подвида и разнообразием ландшафтно-географических условий (низкогорные, горные, высокогорные очаги), в которых происходила эволюция штаммов кавказского подвида. Очаги Кавказа и Закавказья являются эндемичной территорией, на которой распространены штаммы кавказского подвида. В других регионах мира штаммы кавказского подвида отсутствуют.
Ранее нами был разработан способ дифференциации штаммов основного и кавказского подвидов методом ПЦР-РВ. Были найдены ДНК мишени “45” и “Caucasic (–91)” – участки хромосомы, содержащие у штаммов основного подвида делецию в 45 пн и у штаммов кавказского подвида – делецию в 91 пн соответственно. Этот способ может быть использован в очагах с одновременной циркуляцией штаммов двух подвидов – Y. pestis ssp. pestis и Y. pestis ssp. caucasica. К таким очагам относится Приараксинский низкогорный очаг сопряженного типа. Разработанный способ дифференциации кавказского подвида методом ПЦР-РВ может быть использован для проведения эпидемиологического мониторинга в очагах чумы сопряженного типа.
Среди исследованных нами штаммов кавказского подвида выявлено два клинических штамма, изолированных от больных с бубонной формой чумы. Это штамм Y. pestis 835 “В.Р.С.”, выделенный от больного в Ленинаканском горном очаге в 1958 г., и штамм М-986 от больного из Зангезуро-Карабахского горного очага, 1973 г. С помощью разработанного нами способа дифференциации штаммов основного и кавказского подвидов методом ПЦР-РВ с праймерами и зондами на ДНК мишени “45” и “Caucasic (–91)” [13] подтверждена принадлежность этих двух клинических штаммов Y. pestis (М-986 и 835 “В.Р.С.”) к кавказскому подвиду. Дальнейший анализ полногеномных последовательностей этих штаммов установил их принадлежность к Зангезуро-Карабахскому и Ленинаканскому кластерам штаммов соответственно.
Нами был осуществлен поиск индивидуальных SNPs, которые могли быть причиной способности этих штаммов вызывать заболевание у людей. Для штамма 835 “В.Р.С.” было найдено три специфических SNPs с координатами (по геному референсного штамма СО92): 4 228 757 (межгенное пространство pepQ-fadB), 4 309 418 (ген YPO3840), 4523124 (ген uhpA). Для штамма М-986 выявлены четыре специфических SNPs с координатами: 211 007 (ген slyD), 288 471 (ген YPO0287), 445 958 (ген hmsT), 3 739 402 (ген ydjJ). Анализ найденных SNPs показал, что они расположены в генах жизнеобеспечения и в межгенных пространствах и, по-видимому, не оказывают влияния на вирулентность этих клинических штаммов Y. pestis кавказского подвида. Скорее всего, развитие инфекционного процесса, вызванное этими штаммами, связано с ослабленным иммунитетом у заболевших людей. Тем не менее полученные результаты подтверждают данные литературы о способности штаммов кавказского подвида вызывать чуму у людей [8, 9].
Таким образом, проведенное нами полногеномное секвенирование семи штаммов Y. pestis ssp. caucasica c учетом ранее секвенированных геномов из генетической базы данных GenBank позволило выявить наличие новых кластеров штаммов в пределах ранее установленных филогенетических линий [11] штаммов кавказского подвида. Определено географическое распространение штаммов этих филогенетических линий и популяций Y. pestis ssp. сaucasica в природных очагах Кавказа и Закавказья. Впервые проведено полногеномное секвенирование двух клинических штаммов кавказского подвида.
Список литературы
Джапарова А.К., Абдикаримов С.Т., Ерошенко Г.А. и др. Оценка эффективности молекулярной диагностики Yersinia pestis в полевом материале из Сарыджазского очага в Кыргызстане // Проблемы особо опасных инфекций. 2017. Вып. 4. С. 23–26. http://dx.doi.org/10.21055/0370-1069-2017-4-23-26.
Попова А.Ю., Кутырев В.В., Балахонов С.В. и др. Координация мероприятий противочумных учреждений Роспотребнадзора по оздоровлению Горно-Алтайского высокогорного природного очага чумы в 2016 г. // Проблемы особо опасных инфекций. 2016. Вып. 4. С. 5–10. http://dx.doi.org/ 10.21055/0370-1069-2016-4-120-127.
Perry R.D., Fetherston J.D. Yersinia pestis – etiologic agent of plague // Clin. Microbiol. Rev. 1997. V. 10. P. 35–66.
Chain P.S., Carniel E., Larimer F.W. et al. Insights into the evolution of Yersinia pestis through wholegenome comparison with Yersinia pseudotuberculosis // Proc. Natl Acad. Sci. USA. 2004. V. 101. P. 13826–13831. doi 10.1073/pnas.0404012101
Achtman M., Morelli G., Zhu P. et al. Microevolution and history of the plague bacillus, Yersinia pestis // Proc. Natl Acad. Sci. USA. 2004. V. 101. P. 17837–17842. doi 10.1073/pnas.0408026101
Kutyrev V.V., Eroshenko G.A., Motin V.L. et al. Phylogeny and classification of Yersinia pestis through the lens of strains from the plague foci of Commonwealth of Independent States // Front. Microbiol. 2018. V. 9. P. 1106. doi 10.3389/fmicb.2018.01106
Cui Y., Yu C., Yan Y. et al. Historical variation in mutational rate in an epidemic pathogen Yersinia pestis // Proc. Natl Acad. Sci. USA. 2013. V. 110. P. 577–582. doi 10.1073/pnas.1205750110
Ефременко В.И., Грижебовский Г.М., Суворова А.Е. и др. О заболевании чумой людей на Кавказе // Организация эпиднадзора при чуме и меры ее профилактики. Алма-Ата, 1992. С. 18–21.
Овасапян О.В., Шехикян М.Т. Случаи заболевания бубонной чумой в высокогорном природном очаге Армении // Эпидемиология, микробиология и иммунология бактериальных и вирусных инфекций. Ростов-на-Дону, 1989. С. 193–195.
Онищенко Г.Г., Кутырев В.В., Федоров Ю.М. и др. Природные очаги чумы Кавказа, Прикаспия, Средней Азии и Сибири / Под ред. Онищенко Г.Г., Кутырева В.В. М.: Медицина, 2004. 192 с.
Кисличкина А.А., Соломенцев В.И., Благодатских С.А. и др. В полевочьих природных очагах чумы на Кавказе циркулируют три генетически различные линии штаммов Yersinia pestis subsp. microtus bv. caucasica (0.PE2) // Мол. генет., микробиол. и вирусол. 2017. № 35(4). С. 140–144. doi 10.18821/0208-0613-2017-35-4-140-144
Платонов М.Е., Евсеева В.В., Светоч Т.Э. и др. Филогеография полевочьих штаммов Yersinia pestis из природных очагов Кавказа и Закавказья // Мол. генет., микробиол. и вирусол. 2012. № 3. С. 18–21.
Никифоров К.А., Оглодин Е.Г., Куклева Л.М. и др. Подвидовая дифференциация штаммов Yersinia pestis методом ПЦР с гибридизационно-флуоресцентным учетом результатов // Журн. микробиол., эпидемиол., иммунобиол. 2017. № 2. С. 22–27.
Лабораторная диагностика опасных инфекционных болезней: Практическое руководство / Под ред. Онищенко Г.Г. и Кутырева В.В. 2-е изд., перераб. и доп. М.: ЗАО “Шико”, 2013. 560 с.
Kislichkina A.A., Bogun A.G., Kadnikova L.A. et al. Nineteen whole-genome assemblies of Yersinia pestis subsp. microtus, including representatives of biovars caucasica, talassica, hissarica, altaica, xilingolensis, and ulegeica // Genome Announc. 2015. № 3(6). pii: e01342-15. doi 10.1128/genomeA.01342-15
Zhgenti E., Johnson S.L., Davenport K.W. et al. Genome assemblies for 11 Yersinia pestis strains isolated in the Caucasus region // Genome Announc. 2015. № 3(5). pii: e01030-15. doi 10.1128/genomeA.01030-15
Кадастр эпидемических и эпизоотических проявлений чумы на территории Российской Федерации и стран ближнего зарубежья (с 1876 по 2016 г.) / Под ред. Кутырева В.В. и Поповой А.Ю. Саратов: ООО “Амирит”, 2016. 248 с.
Дополнительные материалы отсутствуют.


