Генетика, 2019, T. 55, № 4, стр. 418-425
Разработка ядерных микросателлитных маркеров с длинными (трех-, четырех-, пяти- и шестинуклеотидными) мотивами для трех видов лиственницы на основе полногеномного de novo секвенирования лиственницы сибирской (Larix sibirica Ledeb.)
Н. В. Орешкова 1, 2, *, Е. И. Бондар 1, Ю. А. Путинцева 1, В. В. Шаров 1, Д. А. Кузьмин 1, К. В. Крутовский 1, 3, 4, 5, **
1 Научно-образовательный центр геномных исследований
Сибирского федерального университета
660041 Красноярск, Россия
2 Институт леса им. В.Н. Сукачева Сибирского отделения Российской академии наук
660036 Красноярск, Россия
3 Геттингенский университет им. Георга-Августа
37077 Геттинген, Германия
4 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
5 Техасский АМ университет, Колледж Стейшн
77843-2138 Техас, США
* E-mail: oreshkova@ksc.krasn.ru
** E-mail: kkrutovsky@gmail.com
Поступила в редакцию 14.08.2018
После доработки 28.09.2018
Принята к публикации 17.10.2018
Аннотация
Лиственница сибирская Larix sibirica Ledeb. является одной из преобладающих бореальных пород в Евразии и имеет высокую экономическую и экологическую ценность. Однако, несмотря на это, разработка и использование микросателлитных маркеров для ее исследования остаются недостаточными. Микросателлитные маркеры уже долгое время являются признанным инструментом для оценки популяционной изменчивости и структуры видов. В настоящей работе был проведен поиск простых три-, тетра-, пента- и гексануклеотидных тандемных повторов в геномной референсной de novo сборке лиственницы сибирской, локусы которой легко генотипируются даже путем простого гель-электрофореза. Всего было найдено более тысячи предположительных микросателлитных локусов. На основе этих данных были разработаны и проверены 60 пар олигонуклеотидных ПЦР праймеров. По итогам тестирования праймеров на образцах ДНК из трех видов лиственницы (L. sibirica Ledeb., L. gmelinii (Rupr.) Rupr. и L. cajanderi Mayr.) были отобраны 14 наиболее перспективных полиморфных локусов, которые могут успешно применяться для изучения и идентификации не только лиственницы сибирской, но также лиственниц Гмелина и Каяндера.
Изучение генетического разнообразия и внутривидовой дифференциации лесообразующих видов хвойных, в том числе и видов рода Larix, имеет важное биосферное и ресурсное значение и является одной из важных задач популяционной биологии. Адаптивный потенциал и устойчивость популяций связаны с их генетической структурой. Для разработки эффективных стратегий селекции и консервации необходимо знание основных генетических характеристик популяций, таких как уровень генетической изменчивости и дифференциации [1]. На основании точных сведений о генетической структуре популяций хвойных, уровне их генетической изменчивости и характере ее распределения в пределах ареалов видов может быть оценен генетический потенциал видов и разработан комплекс мероприятий, направленных на максимальное сохранение генетического разнообразия в процессе их использования и воспроизводства.
Молекулярные маркеры, в частности микросателлитные локусы, или SSR-маркеры, уже долгое время являются традиционными генетическими маркерами для индивидуальной ДНК-идентификации, оценки популяционной изменчивости, анализа генетической структуры популяций и определения родства. Ввиду высокой скорости мутирования, кодоминантного характера наследования, равномерного распределения по геному и высокой воспроизводимости микросателлитные маркеры получили широкое применение в изучении растений, в том числе хвойных [2, 3]. Более того, было показано, что SSR-маркеры могут быть использованы для идентификации видов одного рода, а также для определения происхождения деревьев, что очень важно для борьбы с нелегальными рубками и нелегальным оборотом древесины и растительного материала [4–7]. К недостаткам SSR-анализа можно отнести тот факт, что микросателлитные маркеры, разработанные для одного вида, часто плохо амплифицируются в других, даже родственных видах. Для конкретного вида самые надежные результаты показывают видоспецифичные маркеры, специально разработанные для этого вида, что в случае с немодельными организмами часто является дорогостоящей процедурой. Учитывая большие размеры генома – от 10 до 30 млрд пар нуклеотидов (пн) – для хвойных данная задача становится особенно сложной. Однако развитие методов секвенирования нового поколения позволило значительно облегчить процесс, сделав его более быстрым и дешевым [8–10].
Из видов рода Larix на сегодняшний день микросателлитные маркеры были разработаны для лиственницы японской [11, 12], европейской [13], альпийской, или Лайэля [14], западной [14, 15] и сибирской [9]. В ранних исследованиях измерение генетического разнообразия и оценка популяционной структуры лиственниц сибирской, Гмелина и Каяндера проводились с использованием SSR-маркеров, разработанных для других видов лиственницы [16, 17]. В 2017 г. для L. sibirica был разработан набор из 11 динуклеотидных видоспецифичных SSR-локусов на основе полногеномного секвенирования данного вида [9], которые показали высокий уровень полиморфизма и могут применяться для популяционно-генетических исследований. Однако использование динуклеотидных маркеров предполагает в первую очередь проведение фрагментного анализа с участием капиллярного электрофореза, поскольку работать со столь короткими повторяющимися мотивами, когда фрагменты могут различаться по длине всего на пару нуклеотидов, на обычных гель-электрофорезах крайне тяжело. Это является преградой для многих лабораторий ввиду отсутствия необходимого оборудования для проведения такого рода анализа. Вместе с этим многие ведомственные генетические лаборатории, специализирующиеся на изучении лесного разнообразия, испытывают необходимость в современных методиках генетической оценки видового разнообразия и мониторинга происхождения древесины. Поэтому существует объективная потребность в разработке генетических микросателлитных маркеров с более длинными (трех-, четырех-, пяти- и шестинуклеотидными) мотивами, с которыми удобно работать в условиях среднестатистической генетической лаборатории, используя обычный гель-электрофорез. В данной работе мы восполнили этот пробел, разработав такие маркеры.
Лиственница сибирская имеет большой адаптивный потенциал как на индивидуальном, так и на популяционном уровне [16, 17]. Благодаря своей прочности, долговечности и устойчивости к гниению она широко используется в строительстве. Однако из-за своей ценной древесины она часто является объектом незаконных рубок, что в свою очередь вызывает серьезные экономические и экологические проблемы и несет угрозу лесному биоразнообразию. Несмотря на то, что лиственница сибирская является чрезвычайно важным лесообразующим видом, разработка и использование микросателлитных маркеров для ее исследования до сих пор остаются недостаточными и практически не используются в борьбе с нелегальными рубками и оборотом древесины. Мы надеемся, что предложенные маркеры изменят ситуацию в лучшую сторону.
МАТЕРИАЛЫ И МЕТОДЫ
Для поиска повторяющихся последовательностей были использованы данные полногеномного секвенирования L. sibirica, полученные на приборе Illumina HiSeq2000 в лаборатории лесной геномики Сибирского федерального университета (СФУ). Образцы ДНК для секвенирования были взяты с дерева в дендрарии Института леса им. В.Н. Сукачева СО РАН г. Красноярска. Для выделения ДНК использовали хвою. Очистка ридов, обрезка по качеству и удаление последовательностей адаптеров проводились при помощи Trimmomatic [18], черновая сборка генома выполнена при помощи программного обеспечения CLC Assembly Cell (https://www.qiagenbioinformatics.com). Контиги, принадлежащие органельным геномам, были исключены из анализа. Поиск совпадений с последовательностями митохондриального и хлоропластного геномов проводили при помощи BLAST (Basic Local Alignment Search Tool) [19].
Отбор контигов, содержащих микросателлитные повторы, проводили при помощи программы GMATo v. 1.2 [20], предназначенной для поиска тандемных повторов в геномных сборках большого размера. Среди всех контигов были отобраны те, что содержали микросателлитные повторы с трех-, четырех-, пяти- и шестинуклеотидными мотивами. При этом минимальное число повторений мотива для трехнуклеотидных было 15 раз, для четырехнуклеотидных – 10, для пяти- и шестинуклеотидных – 7 раз. Для дальнейшей разработки праймеров с сайтами отжига, фланкирующих отобранные повторы, использовали онлайн-сервис WebSat [21]. При дизайне пар праймеров учитывали желаемый размер ампликона в диапазоне 140–280 пн, поскольку при проведении гель-электрофореза различающиеся всего на несколько нуклеотидов фрагменты определяются надежнее, если имеют относительно небольшую длину. Отобранные последовательности праймеров для ядерных маркеров лиственницы сибирской синтезировали в ЗАО “Евроген” и затем тестировали в лаборатории лесной геномики СФУ.
Схема тестирования включала первоначальную проверку полимеразной цепной реакции (ПЦР) амплификации на образцах ДНК четырех деревьев L. sibirica с последующим проведением электрофореза в полиакриламидном геле и окрашиванием продуктов ПЦР при помощи раствора бромистого этидия. После оптимизации условий амплификации каждый локус тестировали на 8–10 образцах из одной выборки лиственницы сибирской с целью выявления полиморфизма и наличия нуль-аллелей. По результатам данной проверки были отобраны локусы, демонстрирующие наиболее стабильно интерпретируемые спектры.
Поскольку иногда локусы, мономорфные для одного вида, оказывались полиморфными для другого вида, изменчивость всех отобранных перспективных локусов проверяли далее на 24 образцах из четырех географически отдаленных выборок из популяций трех видов лиственницы, представляющих разные части их ареалов (табл. 1).
Таблица 1.
Географическое расположение популяций лиственницы, из которых были взяты образцы для тестирования отобранных микросателлитных локусов
| Вид | Популяция | Код популяции | Район расположения | Географические координаты | |
|---|---|---|---|---|---|
| с. ш. | в. д. | ||||
| L. sibirica | Северо-Енисейск | ЛС-СЕ | Северо-Енисейский р-н Красноярского края, Пит-Городок | 59°22′ | 93°39′ |
| Туим | ЛС-ТУ | Окрестности с. Туим, Республика Хакасия | 54°13′ | 89°59′ | |
| L. cajanderi | Якутия | ЛК-ЯК | Республика Якутия (Саха), Намский р-н, окрестности с. Хомустах-1 (Кысыл Сыр) | 62°46′ | 129°69′ |
| L. gmelinii | Чита | ЛГ-Ч | Забайкальский край, в окрестностях населенного пункта Хилок | 51°21′ | 110°27′ |
Препараты тотальной ДНК были выделены модифицированным методом с применением цетилтриметиламмониумбромида (CTAB) из образцов тканей хвои, высушенной при помощи силикагеля [22]. Для проведения ПЦР использовали готовые реакционные смеси для амплификации ДНК “GenePak PCR Core” производства ООО “Лаборатория Изоген”, содержащие ингибированную для “горячего старта” Taq-ДНК-полимеразу, дизоксинуклеозидтрифосфаты и хлорид магния. Программа амплификации включала первичную денатурацию в течение 1 мин при 94°С, затем 9 циклов “touchdown” c понижением на 1°С каждый цикл: 30 с при 94°С, 30 с при 60°С, 1 мин при 72°С, далее 24 цикла без “touchdown”: 30 с при 94°С, 30 с при 50°С, 30 с при 72°С; финальная элонгация составляла 10 мин при 72°С. Продукты амплификации разделяли путем электрофореза в 6%-ом полиакриламидном геле с использованием трис-EDTA-боратного электродного буфера в камерах для вертикального электрофореза. Окраску геля проводили в растворе бромистого этидия с последующей визуализацией в ультрафиолетовом свете. В качестве маркера стандартных длин фрагментов ДНК использовали ДНК плазмиды pBR322 E. coli, обработанную рестриктазой HpaII.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В результате поиска тандемных повторов с помощью компьютерной программы GMATo было найдено 1015 локусов с повторяющимися трех-, четырех-, пяти- и шестинуклеотидными мотивами с учетом минимального числа повторения мотивов (табл. 2). Из них были выбраны 222 микросателлитных локуса и с помощью компьютерной программы WebSat для них был выполнен дизайн праймеров (табл. 2).
Таблица 2.
Микросателлитные ядерные маркеры L. sibirica, отобранные для дизайна праймеров
| Длина мотива, пн | Минимальное число повторов мотива | Число микросателлитных локусов | ||
|---|---|---|---|---|
| найденных | выбранных для дизайна праймеров | протестированных | ||
| 3 | 15 | 423 | 78 | 27 |
| 4 | 10 | 249 | 57 | 12 |
| 5 | 7 | 132 | 36 | 12 |
| 6 | 7 | 211 | 51 | 9 |
| Всего | 1015 | 222 | 60 | |
Выравнивание контигов, содержащих отобранные повторы, на последовательности митохондрий растений выявило наличие среди отобранных контигов одного, вероятнее всего принадлежащего митохондриальному геному. Данный контиг с соответствующим ему микросателлитным повтором из дальнейшего анализа был исключен в данной работе, но может представлять ценный маркер для изучения материнских линий. Проверка на наличие хлоропластных последовательностей не выявила их присутствия среди отобранных контигов.
После проверки полученных последовательностей праймеров на копийность при помощи выравнивания на исходную сборку была выявлена 161 пара праймеров, имеющих более одного локуса в использованной референсной сборке ядерного генома. Эти праймеры были признаны неподходящими для генотипирования и удалены из дальнейшего анализа. Оставшиеся 60 пар праймеров были отобраны для дальнейшего тестирования в лаборатории.
Первичный анализ отобранных праймеров показал, что из 60 пар праймеров 14 обнаружили отсутствие амплифицированного продукта, а для 13 амплифицированный продукт был неподходящего размера. Остальные 33 пары проявили более-менее устойчивую амплификацию в подобранных условиях. Эти праймеры далее тестировались на увеличенной выборке деревьев. По результатам данной проверки было отобрано 23 локуса, демонстрировавших наиболее стабильные интерпретируемые спектры.
По результатам тестирования 23 локусов на 24 образцах из четырех географически отдаленных популяций L. sibirica, L. gmelinii и L. cajanderi (табл. 3) четыре локуса – Ls_8778872, Ls_291006, Ls_1664757 и Ls_3542003 – были признаны мономорфными для трех видов лиственницы. Локус Ls_1524449 проявил себя высокополиморфным для L. sibirica с девятью аллельными вариантами, тогда как для L. gmelinii и L. cajanderi данный маркер показал нестабильные спектры с большим числом нуль-аллелей, с отсутствием амплификации, вероятно, вследствие мутаций в зоне отжига праймеров. Также показал себя полиморфным для L. sibirica и локус Ls_951631, но в популяции лиственницы Каяндера наблюдались проблемы с генотипированием.
Таблица 3.
Результаты тестирования 23 ядерных микросателлитных локусов на выборках из четырех географически отдаленных популяций L. sibirica, L. gmelinii и L. cajanderi
| Локус | Мотив | Размер продукта, пн | L. sibirica | L. gmelinii | L. cajanderi |
|---|---|---|---|---|---|
| Ls_417667 | AAT | 238 | П | П | П |
| Ls_840190 | TAC | 232 | П | П | П |
| Ls_951631 | ATC | 150 | П | П | П |
| Ls_954234 | ATT | 202 | П | П | П |
| Ls_611965 | CAG | 236 | П | П | П |
| Ls_752897 | AAG | 252 | П | П | П |
| Ls_1247092(2) | CTT | 214 | П | П | П |
| Ls_3765334 | GAG | 270 | П | П | П |
| Ls_254200 | AAT | 252 | П | П | П |
| Ls_1664757 | TCT | 144 | М | М | М |
| Ls_3542003 | TCA | 142 | М | М | М |
| Ls_291006 | AAAG | 168 | М | М | М |
| Ls_1008427 | ATAG | 181 | П | П | П |
| Ls_1524449 | ATAG | 179 | П | П | П |
| Ls_2672894 | TTTG | 163 | П | П | П |
| Ls_2552367 | CTAT | 280 | П | П | П |
| Ls_980491 | CTAT | 228 | П | М | П |
| Ls_1898261 | ACAT | 191 | П | П | П |
| Ls_3952800 | TATG | 252 | П | П | П |
| Ls_897755 | ACCAT | 265 | П | П | П |
| Ls_305132 | GTCGGA | 219 | П | П | П |
| Ls_8778872 | TGTTGA | 221 | М | М | М |
| Ls_4040657 | TCACTT | 245 | П | П | П |
Локус Ls_254200, при первичной проверке выглядевший мономорфным на лиственницах Каяндера и Гмелина, в дальнейшем проявил слабую полиморфность для этих видов, а для L. sibirica был полиморфным, но с большим числом нуль-аллелей и нестабильной амплификацией. А для локусов Ls_1898261 и Ls_897755 обнаружено слишком большое количество неспецифической амплификации.
Остальные 14 протестированных локусов были полиморфными и пригодными для проведения популяционно-генетических исследований (табл. 4). Из них наиболее простые спектры с одной зоной активности и числом аллелей от 3 до 5 показали семь локусов (Ls_980491, Ls_2672894, Ls_4040657, Ls_1008427, Ls_417667, Ls_2552367 и Ls_3765334). Примеры электрофореграмм данных локусов показаны на рис. 1–3. Разработанные маркеры могут успешно применяться для анализа генетической изменчивости и проведения популяционно-генетических исследований не только лиственницы сибирской, но также лиственниц Гмелина и Каяндера.
Таблица 4.
Локусы, рекомендуемые для использования в генетико-популяционных исследованиях трех видов лиственницы
| Локус | Мотив и число его повторов в референсном геноме | Нуклеотидная последовательность ПЦР-праймеров | Размер ампликона, пн | Число выявленных аллелей |
|---|---|---|---|---|
| Ls_3765334 | GAG(17) | F: GTGGGATGACTAGGTGAGAAGG R: TATTGTCGATGTGCTCTGCCTA |
174–213 | 5 |
| Ls_1247092(2) | CTT(19) | F: AAACGCCAACCACAGAATTTAC R: TTATCAAACGAGGGATGCTTCT |
201–228 | 7 |
| Ls_417667 | AAT(16) | F: CAGAGGATCTCATTCCTGTTGA R: CTCGAAGGCCAATTAGGATAAA |
207–243 | 5 |
| Ls_611965 | CAG(18) | F: GCAAGTTCCGATTCAAGAGTTT R: GTTGCTGCTGAGGCATGTAG |
222–276 | 7 |
| Ls_752897 | AAG(15) | F: GCAGATGTTGATACAGTGGAGG R: CAGCTTCATTTCGTGGCTAAT |
216–264 | 12 |
| Ls_840190 | TAC(15) | F: GATTCTAGCAACTATCACACATGGA R: ATGTCCCTATCCAAAAGAAAGC |
216–249 | 7 |
| Ls_954234 | ATT(15) | F: TGGCGTTTGGCTAAGTTGTAA R: GGTTGATTTATGTGTGTGTATGTGG |
171–204 | 10 |
| Ls_3952800 | TATG(10) | F: AGGATAGAGTGCAGAAGGGTGA R: AGAGCATTGGATCACTTTGGAT |
200–264 | 9 |
| Ls_980491 | CTAT(12) | F: TAAGCATTGAGCCTTACAACCA R: TATAGGGAAAAGGGGAATCTCA |
204–240 | 3 |
| Ls_2672894 | TTTG(11) | F: CAAAGGATGGAATGTGTCTCAA R: GTTGGTATGGTTTCCCAGAGTG |
152–164 | 3 |
| Ls_2552367 | CTAT(10) | F: AAAGGTGCAATCACGTAAAGAC R: ATCGAAGCGGAAAATGTGTA |
184–196 | 4 |
| Ls_1008427 | ATAG(13) | F: CACCCCTATCCCACAAATCTTA R: ATTTATCTTTGGCCCTCATGC |
152–174 | 5 |
| Ls_4040657 | TCACTT(11) | F: TTCACTTTCACAAACCATCACC R: ACATCTGGCATTTAACCGAAGT |
194–218 | 3 |
| Ls_305132 | GTCGGA(7) | F: GCAGAGCCGTTATTCGATCTAT R: CCCTCGTTTCCTCTTCTGACTA |
210–240 | 6 |
Рис. 1.
Электрофореграмма продуктов амплификации локуса Ls_954234, содержащего трехнуклеотидный повтор (ATT), протестированных в выборках из популяций лиственницы Гмелина из Забайкалья (ЛГ-Ч), Каяндера из центральной Якутии (ЛК-ЯК) и сибирской из Туимского района (ЛС-ТУ). Генотипы указаны в длинах (пн) амплифицируемых фрагментов. HpaII – фрагменты ДНК стандартной длины.
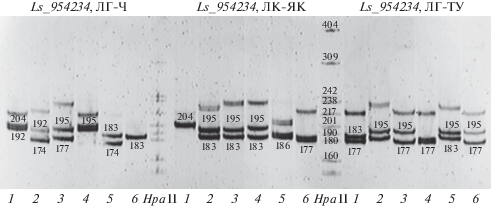
Рис. 2.
Электрофореграмма продуктов амплификации локуса Ls_2672894, содержащего тетрануклеотидный повтор (TTTG), протестированных в выборке лиственницы сибирской из Туимского района (ЛС-ТУ). Генотипы указаны в длинах (пн) амплифицируемых фрагментов. HpaII – фрагменты ДНК стандартной длины.
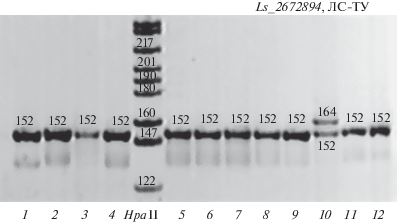
Рис. 3.
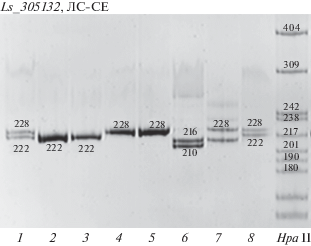
Электрофореграмма продуктов амплификации локуса Ls_305132, содержащего гексануклеотидный повтор (GTCGGA), протестированных в выборке лиственницы сибирской из Северо-Енисейского района (ЛС-СЕ). Генотипы указаны в длинах (пн) амплифицируемых фрагментов. HpaII – фрагменты ДНК стандартной длины.
Работа выполнена в рамках проекта “Геномные исследования основных бореальных лесообразующих хвойных видов и их наиболее опасных патогенов в Российской Федерации”, финансируемого Правительством РФ (договор № 14.Y26.31.0004).
Список литературы
Нечаева Ю.С., Жуланов А.А., Боронникова С.В., Пришнивская Я.В. Нуклеотидный полиморфизм адаптивно значимых генов-кандидатов в популяциях Larix sibirica Ledeb. Урала // Генетика. 2017. Т. 53. № 5. С. 587–595.
Krutovsky K.V., St. Clair J.B., Saich R. et al. Estimation of population structure in coastal Douglas-fir (Pseudotsuga menziesii (Mirb.) Franco var. menziesii) using allozyme and microsatellite markers // Tree Genetics and Genomes. 2009. V. 5. № 4. P. 641–658.
Echt C.S., Saha S., Krutovsky K.V. et al. An annotated genetic map of loblolly pine based on microsatellite and cDNA markers // BMC Genet. 2011. V. 12. № 17. doi 10.1186/1471-2156-12-17
Vlam M., de Groot G.A., Boom A. et al. Developing forensic tools for an African timber: Regional origin is revealed by genetic characteristics, but not by isotopic signature // Biol. Conserv. 2018. V. 220. P. 262–271.
Tereba A., Woodward S., Konecka A. et al. Analysis of DNA profiles of ash (Fraxinus excelsior L.) to provide evidence of illegal logging // Wood Sci. Technol. 2017. V. 51. P. 1377–1387.
Ng K.K.S., Lee S.L., Tnah L.H. et al. Forensic timber identification: a case study of a CITES listed species, Gonystylus bancanus (Thymelaeaceae) // Forensic Sci. Internat. Genet. 2016. V. 23. P. 197–209. doi 10.1016/ j.fsigen.2016.05.002
Degen B., Ward S.E., Lemes M.R. et al. Verifying the geographic origin of mahogany (Swietenia macrophylla King) with DNA-fingerprints // Forensic Sci. Internat. Genet. 2013. V. 7. P. 55–62.
Белоконь М.М., Политов Д.В., Мудрик Е.А. и др. Разработка микросателлитных маркеров сосны кедровой сибирской (Pinus sibirica Du Tour) по результатам полногеномного de novo секвенирования // Генетика. 2016. Т. 52. № 12. С. 1418–1427.
Орешкова Н.В., Путинцева Ю.А., Шаров В.В. и др. Разработка микросателлитных маркеров лиственницы сибирской (Larix sibirica Ledeb.) на основе полногеномного de novo секвенирования // Генетика. 2017. Т. 53. № 11. С. 1278–1284. doi 10.7868/ S0016675817110091
Taheri S., Lee A.T., Yusop M.R. et al. Mining and development of novel SSR markers using Next Generation Sequencing (NGS) data in plants // Molecules. 2018. V. 23. doi 10.3390/molecules23020399
Isoda K., Watanabe A. Isolation and characterization of microsatellite loci from Larix kaempferi // Mol. Ecol. Notes. 2006. V. 6. № 3. P. 664–666. doi 10.1111/j.1471-8286.2006.01291.x
Xing-Bin Chen, Yun-Hui Xie, Xiao-Mei Sun. Development and characterization of polymorphic genic-SSR markers in Larix kaempferi // Molecules. 2015. V. 20. P. 6060–6067. doi 10.3390/molecules20046060
Wagner S., Gerber S., PetitR.J. Two highly informative dinucleotide SSR multiplexes for the conifer Larix decidua (European larch) // Mol. Ecol. Res. 2012. V. 12. P. 717–725. doi 10.1111/j.1755-0998.2012.03139.x
Khasa D.P., Newton C.H., Rahman M.H. et al. Isolation, characterization, and inheritance of microsatellite loci in Alpine larch and Western larch // Genome. 2000. V. 3. № 43. P. 439–448. doi 10.1139/g99-131
Chen C., Liewlaksaneeyanawin C., Funda T. et al. Development and characterization of microsatellite loci in western larch (Larix occidentalis Nutt.) // Mol. Ecol. Res. 2009. V. 9. № 3. P. 843–845. doi 10.1111/j.1755- 0998.2008.02289.x
Орешкова Н.В., Белоконь М.М., Жамъянсурен С. Генетическое разнообразие, популяционная структура и дифференциация лиственниц сибирской, Гмелина и Каяндера по данным SSR-маркеров // Генетика. 2013. Т. 49. № 2. С. 204–213.
Babushkina E.A., Vaganov E.A., Grachev A.M. et al. The effect of individual genetic heterozygosity on general homeostasis, heterosis and resilience in Siberian larch (Larix sibirica Ledeb.) using dendrochronology and microsatellite loci genotyping // Dendrochronologia. 2016. V. 38. P. 26–37.
Bolger A.M., Lohse M., Usadel B. Trimmomatic: A flexible trimmer for Illumina sequence data // Bioinformatics. 2014. V. 30. № 15. P. 2114–2120. doi 10.1093/ bioinformatics/btu170
Bethesda M.D. BLAST // National Library of Medicine (US), National Center for Biotechnology Information. 2004. URL: https://blast.ncbi.nlm.nih.gov/Blast.cgi.
Wang X., Lu P., Luo Z. GMATo: A novel tool for the identification and analysis of microsatellites in large genomes // Bioinformation. 2013. V. 9. № 10. P. 541–544.
Martins W.S., Lucas D.C.S., Neves K.F.S., Bertioli D.J. WebSat – a web software for microsatellite marker development // Bioinformation. 2009. V. 3. № 6. P. 282–283.
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinus radiata based on RFLP, RAPD, and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. № 6. P. 673–679. doi 10.1007/BF00226088
Дополнительные материалы отсутствуют.


