Генетика, 2019, T. 55, № 4, стр. 439-448
Первый опыт исследования видового разнообразия рыб озера Ханка с использованием методов ДНК-штрихкодирования
С. В. Туранов 1, 2, *, Ю. Ф. Картавцев 1, 3, М. Е. Шаповалов 4
1 Институт биологии моря им. А.В. Жирмунского, Национальный научный центр морской биологии
Дальневосточного отделения Российской академии наук
690041 Владивосток, Россия
2 Дальневосточный государственный технический рыбохозяйственный университет,
кафедра водных биоресурсов и аквакультуры
690087 Владивосток, Россия
3 Дальневосточный федеральный университет, кафедра биоразнообразия и морских биоресурсов
690059 Владивосток, Россия
4 ФГБНУ ТИНРО-Центр
690091 Владивосток, Россия
* E-mail: sturcoal@mail.ru
Поступила в редакцию 21.05.2018
После доработки 18.09.2018
Принята к публикации 09.10.2018
Аннотация
Озеро Ханка – крупнейший водоем северо-восточной Азии, являющийся важной частью ресурсов пресноводного рыболовства. Ввиду геологической молодости, а также сообщения с крупными речными системами рек Амур и Уссури Ханка имеет низкую степень эндемизма. Список рыб озера постоянно пополняется за счет инвазий, происходящих как непреднамеренно, так и вследствие интродукции гидробионтов для максимального использования потенциала естественной продуктивности. В связи с этим озеро Ханка является подходящим полигоном для разработки и внедрения системных методов мониторинга видового разнообразия. В настоящей работе впервые представлены результаты комплексного подхода к решению данной задачи. Произведен отлов 64 экземпляров рыб от 16 видов, представляющих четыре семейства. Проанализированы таксономические отношения и дана оценка видового разнообразия с использованием как классических подходов, так и методов ДНК-штрихкодирования. Образцы генотипированы по митохондриальному гену Co-1. Сформирована и проанализирована библиотека ДНК-штрихкодов. Средние значения внутривидовых генетических расстояний, скорректированных по модели K2P, составили 0.15 ± 0.01%, расстояния между разными видами в пределах одного рода – 4.08 ± 0.01%. Филогенетический анализ выявил монофилетическое происхождение каждой видовой группы с высокой поддержкой узлов (98–100%). Таким образом, продемонстрирован консенсус морфологических и молекулярно-генетических подходов при разграничении видов и подтверждена высокая эффективность ДНК-штрихкодирования применительно к документации видового разнообразия рыб в оз. Ханка. Новые молекулярно-генетические данные в основном подтверждают описанные ранее таксономические взаимоотношения для проанализированных рыб, однако требуются дополнительные исследования для уточнения видового состава р. Acheilognathus в оз. Ханка.
Около половины известного на сегодняшний день видового разнообразия рыб сформировано пресноводными таксонами, которые, являясь значимой частью таксономического богатства позвоночных животных [1], сосредоточены на территории менее 1% от площади земной поверхности [2, 3]. Пресноводные системы, характеризующиеся уникальностью механизмов формирования и поддержания современного биоразнообразия [4], в настоящее время находятся в условиях жесткой антропогенной нагрузки, заключающейся в быстром изменении ландшафта, гидрологического режима водоемов, климата, превышении допустимых объемов вылова, загрязнении водоемов и распространении инвазивных видов [5]. В первую очередь это относится к системам, представляющим наибольшую рыбохозяйственную ценность, таким как крупные реки и озера.
Ограниченная способность к перемещению в ответ на климатические и ландшафтные изменения ввиду особенностей местообитания делает пресноводных рыб группой, наиболее подверженной риску внезапного вымирания [6, 7]. Это привлекает внимание к пресноводным рыбам в качестве популярного объекта для разработки и применения комплекса классических и современных методов изучения видового разнообразия [4, 8–10]. Мониторинг таксономического и генетического разнообразия пресноводных рыб в последнее время развивается как в качестве национальных и региональных программ [11–18], так и в виде узконаправленных проектов [19–25]. Данное направление с недавнего времени развивается и в России. К изучению привлекаются морские [26, 27] и пресноводные [16, 28, 29] рыбы, включая анализ подмены рыбных продуктов в торговой сети [30]. Отмечается успешное применение методов ДНК-штрихкодирования при идентификации рыб и изучении видового разнообразия. В то же время обращается внимание на необходимость оптимизации порога разграничения внутри- и межвидовой изменчивости [16], а также ограниченность метода в таксономических и особенно в молекулярно-филогенетических приложениях ввиду малой филогенетической информативности штрихкода Co-1 [16, 27].
ДНК-штрихкодирование [31, 32] – направление, деятельность которого сконцентрирована на инвентаризации видового разнообразия живых организмов с использованием унифицированных молекулярно-генетических методов [33] и средств биоинформатики [34, 35]. Для изучения видового разнообразия рыб в настоящее время успешно используются последовательности митохондриального гена Co-1 [33, 36–38]. Несмотря на имеющиеся недостатки, свойственные методам молекулярной систематики в целом [39, 40], ДНК-штрихкодирование зарекомендовало себя в качестве эффективной таксономической утилиты применительно к абсолютному большинству известных видов рыб [37–39] и находит широкое применение во многих смежных областях мониторинга биологического разнообразия [41–43].
Крупнейшим пресноводным водоемом на территории северо-восточной части Азии является оз. Ханка, располагающееся на границе между Китаем и Россией и принадлежащее водосборному бассейну р. Амур. Рыбы озера формируют динамическое сообщество [44], насчитывающее более 86 видов, относящихся к 60 родам и 18 семействам [45–50]. К особенностям ихтиофауны оз. Ханка можно отнести очень низкую степень эндемизма ввиду открытости и геологической молодости водоема [44], а также нарастающее из года в год по разным причинам число инвазивных видов [48], что требует внедрения комплексных методов мониторинга видового разнообразия. Целью настоящей работы является сборка и анализ библиотеки ДНК-штрихкодов, а также исследование таксономического разнообразия рыб оз. Ханка на основе последовательностей митохондриального гена Co-1.
МАТЕРИАЛЫ И МЕТОДЫ
Материал настоящей работы составили последовательности 5'-участка митохондриального гена Co-1 от 64 экземпляров рыб, принадлежащих 16 видам, 12 родам и четырем семействам. Внутривидовая выборка при этом насчитывала от 1 до 7 экземпляров. Рыбы были пойманы в 2012 г. в двух точках акватории оз. Ханка. Идентификация видовой принадлежности рыб была выполнена с использованием консенсусных данных о диагностических признаках [46, 47, 49–52] при учете номенклатурных поправок [53] (электронное приложение, табл. 1, 3 ). Для подготовки к формированию библиотеки ДНК-штрихкодов от каждого экземпляра индивидуально был взят кусочек ткани скелетной мускулатуры (100–300 мг) с последующей фиксацией в 95%-ном этиловом спирте. Каждый экземпляр был сфотографирован, подготовлен к ваучеризации (документированию) и передан в хранилище музея ННЦМБ ДВО РАН. Тотальная ДНК из зафиксированной в этаноле мышечной ткани была выделена хлороформ-фенольным методом с исключением фенола на стадии депротеинизации и добавлением при этом РНКазы [54]. Участок гена Co-1 от каждого образца был амплифицирован посредством ПЦР с использованием праймеров F1-5'-TCAACCAACCACAAAGACATTGGCAC-3' и R1-5'-TAGACTTCTGGGTGGCCAAAGAATCA-3' [55]. Реакционная смесь ПЦР объемом 25 мкл включала 1 мкл раствора ДНК (20–150 нг), 5 мкл 5× Taq-Reg-буфера (Евроген, конечная концентрация MgCl2 – 2.5 мМ), dNTPs (Евроген, 200 мкМ каждого фосфата), по 0.4 мМ прямого и обратного праймеров, а также 0.05 ед. Taq-полимеразы (Евроген). Термический алгоритм состоял из предварительного нагрева при 94°C в течение 2 мин, 35 циклов по следующей схеме: денатурация при 94°C в течение 40 с, отжиг при 52°C в течение 40 с и 1 мин элонгация при 72°C с заключительной стадией в течение 10 мин. Проверку результатов ПЦР проводили с помощью электрофореза в 1%-ном агарозном геле с последующей экспозицией в растворе бромистого этидия и просмотром под ультрафиолетовым светом с использованием системы визуализации (Хеликон). Успешно амплифицированные фрагменты были секвенированы с ПЦР-праймерами и набором BrightDye™ Terminator Cycle Sequencing Kit v3.1 (NimaGen). Капиллярный электрофорез полученных фрагментов проведен на секвенаторе ABI Prism 3130 DNA Genetic Analyzer (Applied Biosystems) в ННЦМБ ДВО РАН. Сборку консенсусных последовательностей из полученных хроматограмм осуществляли в программе ChromasPro 1.34 (ChromasPro; http://www.technelysium.com.au/ chromas.html/). Выравнивание последовательностей и корректировка рамки считывания были проведены в MEGA 6.0 [56] с использованием алгоритма MUSCLE [57]. При выполнении данной процедуры в качестве референсных последовательностей использовались наиболее близкие по значению сходства из выводных данных BLAST [58] в GenBank [59]. Последовательности вместе со всей необходимой информацией и фотографиями рыб были размещены в BOLD [34, 35] в проекте с названием FOLK (Fishes of the Lake Khanka). При расчете генетических расстояний применяли коррекцию по двухпараметрической модели Кимуры [60]. Для разграничения видов рыб, а также возможности обсуждения таксономических отношений надвидового ранга были построены филогенетические деревья с использованием следующих подходов: NJ (ближайшего соседа) и ML (максимального правдоподобия), представленных в программном пакете MEGA. Дополнительно использовали поиск оптимальной топологии с помощью байесовского метода (BI) в программе MrBayes 3.2 [61]. Модель нуклеотидных замещений была выбрана с помощью средств MEGA при использовании информационного критерия Акаике. Оценка устойчивости топологии проведена на основе 1000 реплик непараметрического бутстреп-теста (NJ, ML), а также значений апостериорной вероятности узлов ветвления (BI). При визуализации филогенетических отношений за основу взята топология ML-дерева (рис. 1). Для разрешения вопроса о таксономической принадлежности и видовом статусе филогрупп в пределах рода Acheilognathus Bleeker, 1860 понадобилось привлечение последовательностей добавочного митохондриального маркера, Cyt-b. Данный пункт в анализе обусловлен тем, что, несмотря на рекомендации по унифицированию методик ДНК-штрихкодирования рыб [36, 62], молекулярная систематика карпообразных развивалась независимо, с применением в качестве основного маркера Cyt-b, и, как следствие, сравнительный материал доступен только для этого фрагмента [63]. Из процитированной работы привлечено 16 последовательностей Cyt-b от девяти филогрупп горчаков видового уровня. Филогенетический анализ (рис. 2) выполнен по упрощенной схеме, с исключением методов NJ и BI, в остальном полностью повторяя схему, описанную выше. Совокупный набор данных Cyt-b составил 20 последовательностей длиной 1055 пн.
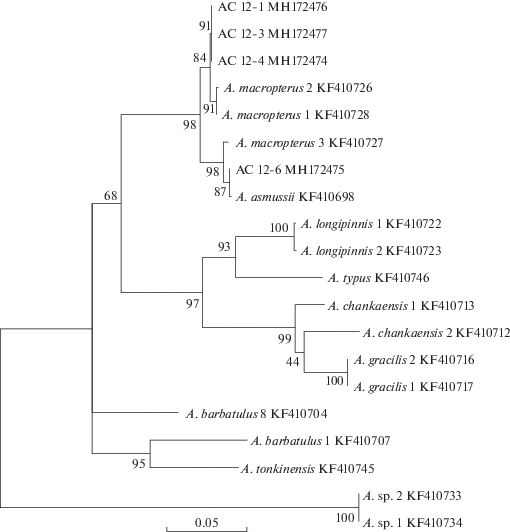
Рис. 1.
Филогенетическое дерево, сконструированное на основе последовательностей гена Co-1 рыб из оз. Ханка. Цифры в узлах ветвления показывают оценки устойчивости топологии. Укоренение проведено по средней точке. Для узлов, оценки устойчивости которых во всех алгоритмах построения совпадали, приведено единственное значение. Все видовые ветви, за исключением Acheilognathus, коллапсированы.
РЕЗУЛЬТАТЫ
Длина матрицы последовательностей Co-1 после выравнивания и корректировки рамки считывания равнялась 567 пн. Среднее значение внутривидовых генетических расстояний составило 0.15 ± 0.01%, тогда как между разными видами в пределах одного рода – 4.0811 ± 0.01% (электронное приложение, табл. 2 ). Несмотря на то, что мы не располагаем достаточной выборкой на уровне родов (более чем одним видом в выборке представлены только роды Tachysurus Lacepède, 1803, Acheilognathus и Chanodychthys Bleeker, 1859), исходя из имеющихся данных можно заключить, что все видовые ветви на филогенетическом дереве располагаются в монофилетических кластерах с поддержкой от 98 до 100%, в то время как разные виды показывают четкое топологическое разобщение (рис. 1). Исключение составляет кластер с горчаками (Acheilognathus), где филогруппа sp1 имеет поддержку 88%, причем четкая кластеризация с базальным выделением sp2 достигнута только в топологии NJ, тогда как в деревьях ML и BI (топологии не приводятся) отношения реализованы в виде политомии. Необходимо отметить, что межвидовые генетические расстояния у горчаков являются наименьшими в рассматриваемом наборе данных (2.1%). Так, расстояния между видами внутри рода Chanodichthys варьируют от 3.1 до 3.9%, в роде Hemiculter Bleeker, 1860 составляют 6.8%, а в пределах Tachysurus – 21.1%. По итогам анализа фрагмента Cyt-b с высокой поддержкой (97–98%) выделяются два макрокластера, первый из которых сформирован двумя филогруппами видового уровня с взаимной дивергенцией 2.6% и внутривидовыми расстояниями от 0 до 0.6% (рис. 2). Оригинальные последовательности, как и в случае с Co-1 (рис. 1), разделяются по принадлежности к двум филогруппам. При этом кластер с AC12-6, показывая дивергенцию по отношению к остальным горчакам оригинальной выборки, объединяется с последовательностями A. asmussii из оз. Ханка и A. macropterus из оз. Казуминаура, являющимся в данном случае парафилетическим. Второй устойчивый макрокластер сформирован пятью филогруппами с взаимной дивергенцией от 5.4 до 13.6%. В него среди прочих входит A. chankaensis, пойманный в провинции Анхой. Остальные последовательности не формируют устойчивых узлов, обнаруживая глубокую взаимную дивергенцию от 11 до 25.6%.
ОБСУЖДЕНИЕ
При оценке видового разнообразия как пресноводных, так и морских рыб стандартизированный фрагмент митохондриального гена Co-1 является наиболее часто применяемым штрихкодом, проявляя свойства, необходимые для разграничения видовых линий [12–32]. Результаты нашей работы вносят вклад в формирование глобальной референсной базы ДНК-штрихкодов пресноводных рыб, представляя собой первую попытку применения молекулярно-генетических методов для мониторинга видового разнообразия данного водоема, также демонстрируя возможность применения Co-1 для обсуждения вопросов таксономии рыб оз. Ханка надвидового уровня. Это проявляется в четкой топологической дифференцированности видовых линий на основе дистанционных методов (рис. 1). Результаты соотношения внутри- и межвидовых генетических расстояний сопоставимы с данными по изучению видового разнообразия пресноводных рыб Северной Америки [14] и Европы [15]. Консенсус топологических (рис. 1) и дистанционных (электронное приложение, табл. 2 ) критериев разграничения видовых линий позволяет подробно обсудить вопросы таксономии рыб оз. Ханка в свете новых молекулярно-генетических данных. Ввиду известных ограничений филогенетической информативности маркера Co-1 следует учитывать, что таксономические обоснования в настоящей работе делаются с допущениями, напрямую зависящими от устойчивости узлов филогении.
Cypriniformes: Cyprinidae
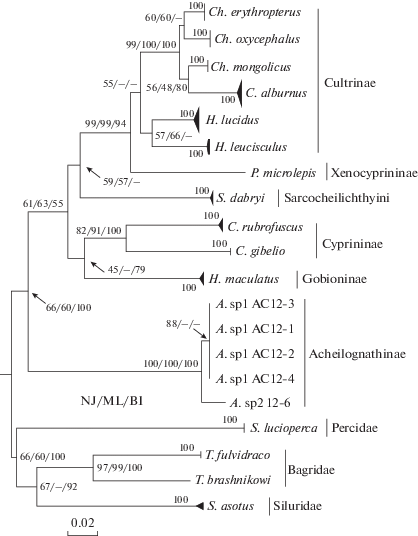
Для разработки вопросов систематики и филогении Cyprinidae в течение последних 100 лет используются не только традиционные для карповых признаки внешней морфологии и число рядов глоточных зубов, но также остеологические и эмбриологические особенности, данные кариологического анализа и современные методы биохимических и генетических исследований. Однако несмотря на то, что литература, посвященная вопросам систематики и филогении Cyprinidae, весьма обширна, единой, общепринятой и достаточно аргументированной точки зрения на число и объем его подсемейств нет [47, 64]. Так, в частности, долгое время общепринятым являлось разделение карповых на две ветви – Leuciscinae sensu lato (“безусые карповые”) и Cyprininae sensu lato (“усатые карповые”) [52, 65]. Согласно современным представлениям, усы неоднократно независимо возникали в разных группах, не относимых к настоящим Cyprininae. В соответствии с последними данными [66] подсемейство Gobionidae выделено в отдельное семейство, к которому относится род Hemibarbus. На представленной схеме (рис. 1) видно, что основная часть из исследованных карповых разделяется на собственно карповых – амурский сазан Cyprinus rubrofuscus LaCepede, 1803 и серебряный карась Carassius gibelio (Bloch, 1782), от которых отделяется конь пятнистый Hemibarbus maculatus Bleeker, 1871, и виды подсемейства Cultrinae, от которых отделяются Sarcocheilichthyini и Xenocyprininae.
Обособленной ветвью, отделившейся от представленных на схеме Cyprinidae ранее всех, располагаются виды подсемейства Acheilognathinae – горчаки. Здесь представлены колючий горчак Acheilognathus sp. 1, близкий к A. asmussii (Dybowski, 1872), и Acheilognathus sp. 2, рассматривающийся как вид-двойник ханкайского колючего горчака A. chankaensis (Dybowski, 1872) [67, 68]. Дополнительные данные анализа фрагмента Cyt-b (рис. 2) указывают на принадлежность трех горчаков из нашей выборки к предполагаемому A. macropterus, тогда как филогруппа, содержащая A. chankaensis, обнаруживает по отношению к последнему глубокую дивергенцию. Наряду с явной нехваткой референсных данных для достоверного определения видов рода Acheilognathus можно предположить, что в оз. Ханка присутствует также и третий вид этого рода. Для прояснения выявленных противоречий требуется дополнительная работа с формированием надежной референсной библиотеки для горчаков.
В недавнем исследовании филогении горчаков Европы и Азии большой группой авторов на основе молекулярно-генетических методов анализа предложено выделить горчаков в отдельное семейство Acheilognathidae в составе надсемейства Cyprinoidea [63]. Таким образом, отделение горчаков от остальных Cyprinidae в рассматриваемой схеме, по-видимому, является следствием их филогенетической обособленности.
Далее из представленной схемы следует, что ветвь пескарей рода Saurogobio Bleeker, 1870 – ящерный пескарь Saurogobio dabryi Bleeker, 1871 – является внешней по отношению к усатым и безусым карповым. Согласно принимаемой нами системе [47], Saurogobio входят в состав подсемейства Gobioninae совместно с пятнистым конем H. maculatus Bleeker, 1871. Однако в нашем случае род Hemibarbus отделяется от остальных усатых карповых значительно позже. По-видимому, на рассматриваемом дереве отражена генетическая неоднородность этого подсемейства, которое, по мнению ряда авторов [47, 69], может быть разделено на две группы родов в ранге подсемейств или триб: Sarcocheilichthyini и Gobioninae. Согласно А.М. Naseka [70], проанализировавшему имеющиеся на тот момент данные по филогении и систематике Gobioninae, в этом подсемействе выделяются четыре группы: Sarcocheilichthys group, Gobio group, Hemibarbus group, в составе которой находится H. maculatus Bleeker, 1871, и Pseudogobio group, в которую входит S. dabryi Bleeker, 1871. Таким образом, положение родов Hemibarbus и Saurogobio на данной схеме согласуется с современными представлениями о филогении Gobioninae.
Основную группу усатых карповых на рассматриваемом дереве представляют уже упомянутый выше пятнистый конь, а также типичные виды карповых – амурский сазан C. rubrofuscus LaCepede, 1803 и серебряный карась C. gibelio (Bloch, 1782), входящие в состав подсемейства Cyprininae – карповые. Вторая ветвь – безусых карповых – среди исследованных видов представлена рыбами, объединяемыми в настоящее время [47, 64] в подсемейство Cultrinae (уклееподобные): верхогляд Ch. erythropterus (Basilewsky, 1855), Ch. mongolicus (Basilewsky, 1855), горбушка Ch. oxycephalus (Bleeker, 1871), уклей – C. alburnus Basilewsky, 1855, востробрюшка уссурийская H. lucidus (Dybowski, 1872) и востробрюшка корейская H. leucisculus (Basilewsky, 1855).
Базальное положение по отношению к уклееподобным на схеме занимает мелкочешуйный желтопер P. microlepis (Bleeker, 1871), который, согласно принимаемой нами системе [47], совместно c подустом-чернобрюшкой X. macrolepis (Bleeker, 1871) входит в состав подсемейства Xenocyprininae. Однако, согласно ряду исследований [71, 72], анализ биологии размножения типичных Cultrinae, с одной стороны, и в сравнении с выделенными в группу Xenocyprininae (Banarescu, 1967) мелкочешуйным желтопером и подустом-чернобрюшкой показал, что по строению ооцитов и особенностям формирования плодовитости последние два вида заметно различаются. Таким образом, по-видимому, только морфологических признаков (в частности, отсутствия киля впереди V), а также особенностей сейсмосенсорной системы недостаточно для выделения из Cultrinae мелкочешуйного желтопера и объединение его в одно подсемейство с подустом-чернобрюшкой (выделение которого из уклееподобных, на взгляд авторов, действительно обосновано). Однако отсутствие в анализе подуста-чернобрюшки не позволяет рассмотреть этот вопрос с позиций филогенетического анализа. Проведение подобного исследования, в частности, между подсемействами Cultrinae, Leuciscinae и Xenocyprininae, позволило бы создать более обоснованную систему филогенетических отношений внутри этой ветви карповых (электронное приложение, табл. 3 ).
Siluriformes: Bagridae, Siluridae
Рыбы отряда сомообразных, рассматриваемые в данной работе, представлены двумя видами косаток-скрипунов – китайской косаткой-скрипуном T. fulvidraco (Richardson, 1846) и косаткой Бражникова T. brashnikowi (Berg, 1907) семейства Bagridae, а также амурским сомом S. asotus (Linnaeus, 1758) сем. Siluridae [47]. На рассматриваемой схеме мы видим четкое разделение рыб отряда Siluriformes на две ветви, соответствующие представленным семействам. Нельзя не отметить значительные генетические расстояния и топологическую разобщенность между видами рода Tachysurus – китайской косаткой-скрипуном и косаткой Бражникова.
Perciformes: Percidae
К отряду окунеобразных на представленной схеме относится судак – S. lucioperca (Linnaeus, 1758) сем. Percidae.
Таким образом, по результатам проведенной работы можно отметить согласованность морфологических и генетических границ выделения видовых линий, что подтверждает высокую эффективность ДНК-штрихкодирования применительно к документации видового разнообразия рыб в оз. Ханка. Новые молекулярно-филогенетические реконструкции, представленные в данной работе, в значительной степени согласуются с описанными ранее таксономическими взаимоотношениями для проанализированных рыб.
Исследование выполнено при финансовой поддержке грантов РФФИ (проект 15-29-02456) и программы Дальний Восток (проект 18-4-040).
Список литературы
Reid G.M., Contreras Macbeath T., Csatadi K. Global challenges in freshwater-fish conservation related to public aquariums and the aquarium industry // Int. Zoo Yearb. 2013. V. 47. № 1. P. 6–45. doi 10.1111/ izy.12020
Ormerod S.J. Current issues with fish and fisheries: editor’s overview and introduction // J. Appl. Ecol. 2003. V. 40. № 2. P. 204–213. doi 10.1046/j.1365-2664.2003.00824.x
Nelson J.S. Fishes of the World. Hoboken: John Wiley and Sons, 2006. 601 p.
Olden J.D., Kennard M.J., Leprieur F. et al. Conservation biogeography of freshwater fishes: Recent progress and future challenges // Divers. Distrib. 2010. V. 16. № 3. P. 496–513. doi 10.1111/j.1472-4642.2010.00655.x
Dudgeon D., Arthington A.H., Gessner M.O. et al. Freshwater biodiversity: importance, threats, status and conservation challenges // Biol. Rev. 2006. V. 81. № 2. P. 163. doi 10.1017/S1464793105006950
Ricciardi A., Rasmussen J.B. Extinction rates of North American freshwater fauna // Conserv. Biol. 1999. V. 13. № 5. P. 1220–1222.
Vié J.-C., Hilton-Taylor C., Stuart S.N. Wildlife in a changing world: an analysis of the 2008 IUCN Red List of threatened species. IUCN, 2009.
Bruton M.N. Have fishes had their chips? The dilemma of threatened fishes // Environ. Biol. Fishes. 1995. V. 43. № 1. P. 1–27. doi 10.1007/BF00001812
Harrison I.J., Stiassny M.L.J. The quiet crisis // Extinctions in near time. N.Y.: Springer Sci. and Business Med., 1999. P. 271–331.
Reynolds J.D., Webb T.J., Hawkins L.A. Life history and ecological correlates of extinction risk in European freshwater fishes // Can. J. Fish. Aquat. Sci. 2005. V. 62. № 4. P. 854–862. doi 10.1139/f05-066
Chen W., Ma X., Shen Y. et al. The fish diversity in the upper reaches of the Salween River, Nujiang River, revealed by DNA barcoding // Sci. Rep. 2015. V. 5. P. 17437. doi 10.1038/srep17437
Dahruddin H., Hadiaty R.K., Hubert N. DNA Barcoding: foundations and applications for southeast Asian freshwater fishes // Treubia. 2017. V. 43. P. 1–16. doi 10.14203/treubia.v43i0.2968
Geiger M.F., Herder F., Monaghan M.T. et al. Spatial heterogeneity in the Mediterranean Biodiversity Hotspot affects barcoding accuracy of its freshwater fishes // Mol. Ecol. Res. 2014. V. 14. № 6. P. 1210–1221. doi 10.1111/1755-0998.12257
Hubert N., Hanner R., Holm E. et al. Identifying Canadian freshwater fishes through DNA barcodes // PLoS One. 2008. V. 3. № 6. doi 10.1371/journal.pone.0002490
Knebelsberger T., Dunz A.R., Neumann D., Geiger M.F. Molecular diversity of Germany’s freshwater fishes and lampreys assessed by DNA barcoding // Mol. Ecol. Res. 2015. V. 15. № 3. P. 562–572. doi 10.1111/1755-0998.12322
Levin B.A., Simonov E., Matveyev M.P. et al. DNA barcoding of the fishes of the genus Alburnoides (Actinopterygii, Cyprinidae) from Caucasus // Mitochondrial DNA. Pt A. 2018. V. 29. № 1. P. 49–55. doi 10.1080/24701394.2016.1238900a
Mustafa K., Yerli S.V., Mangit F. et al. Fish biodiversity in inland waters of Turkey // J. Acad. Doc. Fish. Aquac. 2014. V. 1. № 3. P. 93–120.
Pereira L.H.G., Hanner R., Foresti F., Oliveira C. Can DNA barcoding accurately discriminate megadiverse Neotropical freshwater fish fauna? // BMC Genet. 2013. V. 14. P. 20. doi 10.1186/1471-2156-14-20
Barman A.S., Singh M., Pandey P.K. DNA barcoding and genetic diversity analyses of fishes of Kaladan River of Indo-Myanmar biodiversity hotspot // Mitochondrial DNA. Pt A. 2017. doi 10.1080/24701394.2017.1285290
Kartavtsev Y.P., Batischeva N.M., Bogutskaya N.G. et al. Molecular systematics and DNA barcoding of Altai osmans, oreoleuciscus (pisces, cyprinidae, and leuciscinae), and their nearest relatives, inferred from sequences of cytochrome b (Cyt-b), cytochrome oxidase c (Co-1), and complete mitochondrial genome // Mitochondrial DNA. Pt A. 2017. V. 28. № 4. P. 502–517. doi 10.3109/24701394.2016.1149822
Kondo N.I., Nakagawa M., Matsuzaki S. et al. DNA barcoding, environmental DNA and an Ongoing attempt of detecting biodiversity in lake Kasumigaura // J. Integr. F. Sci. 2016. V. 13. P. 21–29.
Maranan J.B.D., Basiao Z.U., Quilang J.P. DNA barcoding of feral tilapias in Philippine lakes // Mitochondrial DNA. 2015. V. 1736. P. 1–12. doi 10.3109/ 19401736.2015.1089478
Overdyk L.M., Braid H.E., Crawford S.S., Hanner R.H. Extending DNA barcoding coverage for Lake Whitefish (Coregonus clupeaformis) across the three major basins of Lake Huron // DNA Barcodes. 2015. V. 3. № 1. P. 59–65. doi 10.1515/dna-2015-0007
Shen Y., Guan L., Wang D., Gan X. DNA barcoding and evaluation of genetic diversity in Cyprinidae fish in the midstream of the Yangtze River // Ecol. Evol. 2016. V. 6. № 9. P. 2702–2713. doi 10.1002/ece3.2060
Yang T., Meng W., Zhang R. et al. DNA barcoding of fishes in Irtysh River China // Russ. J. Genet. 2016. V. 52. № 9. P. 969–976.
Turanov S.V., Kartavtsev Yu.Ph., Lipinsky V.V. et al. DNA-barcoding of perch-like fishes (Actinopterygii: Perciformes) from far-eastern seas of Russia with taxonomic remarks for some groups // Mitochondrial DNA. 2016. V. 27. № 2. P. 1188–1209. doi 10.3109/ 19401736.2014.945525
Гордеева Н.В., Шаховской И.Б. Применение ДНК-баркодинга для идентификации видов и филогенетических исследований летучих рыб (Exocoetidae) // Вопр. ихтиол. 2017. Т. 57. № 2. С. 212–221.
Кодухова Ю.В., Карабанов Д.П. Морфологические изменения в популяции плотвы (Rutilus rutilus, Cyprinidae) озера Плещеево в результате вселения моллюска Dreissena polymorpha (Bivalvia) // Зоол. журн. 2017. Т. 96. № 9. С. 1069–1077.
Ermakov O.A., Levina M.A., Titov S.V., Levin B.A. mtDNA-based identification of two widespread roach species (Rutilus, Cyprinidae) characterized by sympatric zone // Inland Water Biol. 2017. V. 10. № 1. P. 112–114. doi 10.1134/S1995082917010060
Nedunoori A., Turanov S.V., Kartavtsev Y.P. Fish product mislabeling identified in the Russian far east using DNA barcoding // Gene Reports. 2017. V. 8. P. 144–149. doi 10.1016/j.genrep.2017.07.006
Hebert P.D.N., Cywinska A., Ball S.L., deWaard J.R. Biological identifications through DNA barcodes // Proc. Biol. Sci. 2003. V. 270. № 1512. P. 313–321. doi 10.1098/rspb.2002.2218
Hebert P.D.N., Ratnasingham S., de Waard J.R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species // Proc. Biol. Sci. 2003. V. 270. Suppl. P. S96–S99. doi 10.1098/rsbl.2003.0025
Ivanova N.V., Zemlak T.S., Hanner R.H., Hebert P.D.N. Universal primer cocktails for fish DNA barcoding // Mol. Ecol. Notes. 2007. V. 7. № 4. P. 544–548. doi 10.1111/j.1471-8286.2007.01748.x
Ratnasingham S., Hebert P.D.N. BOLD: The barcode of life data system // Mol. Ecol. Notes. 2007. V. 7. № 3. P. 355–364. doi 10.1111/j.1471-8286.2007.01678.x
Ratnasingham S., Hebert P.D.N. A DNA-based registry for all animal species: The Barcode Index Number (BIN) system // PLoS One. 2013. V. 8. № 7. doi 10.1371/journal.pone.0066213
Becker S., Hanner R., Steinke D. Five years of FISH-BOL: Brief status report // Mitochondrial DNA. 2011. V. 22. № S1. P. 3–9. doi 10.3109/19401736.2010.535528
Ward R.D., Hanner R., Hebert P.D.N. The campaign to DNA barcode all fishes, FISH-BOL // J. Fish Biol. 2009. V. 74. № 2. P. 329–356. doi 10.1111/j.1095-8649.2008.02080.x
Ward R.D. DNA barcode divergence among species and genera of birds and fishes. // Mol. Ecol. Res. 2009. V. 9. № 4. P. 1077–1085. doi 10.1111/j.1755-0998.2009.02541.x
Collins R.A., Cruickshank R.H. The seven deadly sins of DNA barcoding. // Mol. Ecol. Res. 2013. V. 13. № 6. P. 969–975. doi 10.1111/1755-0998.12046
Meyer C.P., Paulay G. DNA barcoding: Error rates based on comprehensive sampling // PLoS Biol. 2005. V. 3. № 12. P. 1–10. doi 10.1371/journal.pbio.0030422
Ardura A., Planes S., Garcia-Vazquez E. Applications of DNA barcoding to fish landings: Authentication and diversity assessment // Zookeys. 2013. V. 365. Spec. issue. P. 49–65. doi 10.3897/zookeys.365.6409
Kress W.J., García-Robledo C., Uriarte M., Erickson D.L. DNA barcodes for ecology, evolution, and conservation // Trends Ecol. Evol. 2015. V. 30. № 1. P. 25–35. doi 10.1016/j.tree.2014.10.008
Trivedi S., Aloufi A.A., Ansari A.A., Ghosh S.K. Role of DNA barcoding in marine biodiversity assessment and conservation: An update // Saudi J. Biol. Sci. 2016. V. 23. № 2. P. 161–171. doi 10.1016/j.sjbs.2015.01.001
Барабанщиков Е.И., Шаповалов М.Е., Свирский В.Г. Динамические процессы в ихтиоцене озера Ханка // Чтения памяти Владимира Яковлевича Леванидова. 2011. № 5. С. 35–41.
Барабанщиков Е.И., Назаров В.А., Свирский В.Г. Фауна круглоротых и рыб озера Ханка // Изв. ТИНРО (Тихоокеанского научно-исследовательского рыбохозяйственного центра). 2006. Т. 146. С. 97–110.
Богуцкая Н.Г., Насека А.М. Круглоротые и рыбы бассейна озера Ханка (система реки Амур): Аннотированный список видов с комментариями по их таксономии и зоогеографии региона // Науч. тетрадь ГосНИОРХ. СПб., 1996. С. 1–45.
Богуцкая Н., Насека А.М. Каталог бесчелюстных и рыб пресных и солоноватых вод России с номенклатурными и таксономическими комментариями М.: Т-во науч. изд. КМК, 2004. 389 с.
Свирский В.Г., Барабанщиков Е.И. Биологические инвазии как элемент антропогенного давления на сообщество гидробионтов озера Ханка // Рос. журн. биологич. инвазий. 2009. № 2. С. 29–35.
Шедько С.В. Список круглоротых и рыб пресных вод побережья Приморья // Чтения памяти Владимира Яковлевича Леванидова. 2001. № 1. С. 229–249.
Шедько С.В., Шедько М.Б. Новые данные о пресноводной фауне юга Дальнего Востока России // Чтения памяти Владимира Яковлевича Леванидова. 2003. № 2. С. 319–336.
Таранец А.Я. Краткий определитель рыб советского Дальнего Востока и прилежащих вод // Изв. ТИНРО. 1937. Т. 11. С. 1–200.
Никольский Г.В. Рыбы бассейна Амура. М.: Изд-во АН СССР, 1956. 551 с.
Parin N.V., Evseenko S.A., Vasil’eva E.D. Fishes of the Rusian Seas: Annotated Catalogue. M.: KMK Scientific Press, 2014. 733 p.
Sambrook J., Fritsch E.F., Maniatis T. Molecular Cloning: A Laboratory Manual. Cold Spring Harbor Laboratory Press, 1989. P. 931–957.
Ward R.D., Zemlak T.S., Innes B.H. et al. DNA barcoding Australia’s fish species // Philos. Trans. R. Soc. Lond. Biol. Sci. 2005. V. 360. № 1462. P. 1847–1857. doi 10.1098/rstb.2005.1716
Tamura K., Stecher G., Peterson D. et al. MEGA6: Molecular evolutionary genetics analysis version 6.0 // Mol. Biol. Evol. 2013. V. 30. № 12. P. 2725–2729. doi 10.1093/molbev/mst197
Edgar R.C. MUSCLE: multiple sequence alignment with high accuracy and high throughput // Nucleic Acids Res. 2004. V. 32. № 5. P. 1792–1797. doi 10.1093/nar/gkh340
Altschul S.F., Gish W., Miller W. et al. Basic local alignment search tool // J. Mol. Biol. 1990. V. 215. № 3. P. 403–410. doi 10.1016/S0022-2836(05)80360-2
Benson D.A., Clark K., Karsch-Mizrachi I. et al. GenBank // Nucleic Acids Res. 2015. V. 43. № D1. P. D30–D35. doi 10.1093/nar/gku1216
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences // J. Mol. Evol. 1980. V. 16. № 2. P. 111–120.
Ronquist F., Teslenko M., Van Der Mark P. et al. MrBayes 3.2: Efficient bayesian phylogenetic inference and model choice across a large model space // Syst. Biol. 2012. V. 61. № 3. P. 539–542. doi 10.1093/sysbio/sys029
Weigt L.A., Driskell A.C., Baldwin C.C., Ormos A. DNA barcoding fishes // DNA Barcodes Methods Protoc. 2012. P. 109–126.
Chang C.-H., Li F., Shao K.-T. et al. Phylogenetic relationships of Acheilognathidae (Cypriniformes: Cyprinoidea) as revealed from evidence of both nuclear and mitochondrial gene sequence variation: evidence for necessary taxonomic revision in the family and the identification of cryptic species // Mol. Phylogenet. Evol. 2014. V. 81. P. 182–194. doi 10.1016/ j.ympev.2014.08.026
Богуцкая Н.Г. Объем и морфологические особенности подсемейства Leuciscinae карповых рыб (Cyprinidae) // Труды зоологического института АН СССР. 1988. Т. 181. С. 96–113.
Никольский Г.В. Частная ихтиология. М.: Сов. Наука, 1950. 436 с.
Schönhuth S., Vukić J., Šanda R. et al. Phylogenetic relationships and classification of the Holarctic family Leuciscidae (Cypriniformes: Cyprinoidei) // Mol. Phylogenet. Evol. 2018. V. 127. P. 781–799. doi 10.1016/j.ympev.2018.06.026
Naseka A.M., Bogutskaya N.G. Contribution to taxonomy and nomenclature of freshwater fishes of the Amur drainage area and the Far East (Pisces, Osteichthyes) // Zoosystematica Rossica. 2004. V. 12. № 2. P. 279–290.
Bogutskaya N.G., Naseka A.M., Shedko S.V. et al. The fishes of the Amur river: updated check-list and zoogeography // Ichtyological Explor. Freshwaters. 2008. V. 19. № 4. P. 301–366.
Насека А.М. Сравнительно-морфологические основы системы карповых рыб подсемейства пескаревых (Gobioninae, Cyprinidae) мировой фауны: Автореф. дис. … канд. биол. наук. СПб.: СПб гос. ун-т, 1998. 18 с.
Naseka A.M. Major patterns of phenotypic diversity, phylogeny and systematics of the gudgeons (Teleostei: Cypriniformes: Gobioninae) // Conference: Joint Meeting of Ichthyologists and Herpetologists. New Orleans, USA, 13–17 July, 2006. http://www.researchgate.net/publication/277721006.
Курдяева В.П., Шкарина Т.В. К биологии мелкочешуйного желтопера Plagiognathops microlepis (Bleeker) и подуста-чернобрюшки Xenocypris macrolepis Bleeker озера Ханка // Изв. ТИНРО-центра. 1998. Т. 123. С. 299–318.
Шаповалов М.Е., Курдяева В.П. Особенности размножения уклееподобных (Cultrinae, Cyprinidae) озера Ханка // Проблемы сохранения водно-болотных угодий международного значения: озеро Ханка: Тр. второй междунар. науч.-практич. конф. Спасск-Дальний, 10–11 июня 2006 г. Владивосток, 2006. С. 74–94.
Дополнительные материалы отсутствуют.