Генетика, 2019, T. 55, № 5, стр. 544-550
Сравнительный анализ изменчивости митохондриального гена cox1 и маркерного гена Y-хромосомы kl-2 1-beta dynein heavy chain у дрозофил группы virilis (Diptera: Drosophilidae)
Б. В. Андрианов 1, *, Д. А. Романов 1, С. Ю. Сорокина 2, Т. В. Горелова 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Институт биологии развития им. Н.К. Кольцова Российской академии наук
117334 Москва, Россия
* E-mail: andrianovb@mail.ru
Поступила в редакцию 04.06.2018
После доработки 30.07.2018
Принята к публикации 07.08.2018
Аннотация
Виды дрозофил группы virilis – одна из хорошо изученных моделей видообразования и микроэволюции. Мы провели сравнительный анализ изменчивости маркерных генов двух нерекомбинирующих областей генома: BOLD-фрагмента митохондриального гена cox1 и фрагмента гена dynein с целью выявления событий межвидовой гибридизации у 11 видов дрозофил. Мы выявили события переноса митохондриальной ДНК из Drosophila montana в Drosophila lacicola и перенос Y-хромосомы из Drosophila ezoana в Drosophila montana. Обсуждается вероятная связь современного процесса видообразования у Drosophila montana с геномной нестабильностью и межвидовой гибридизацией в природе.
Группа virilis относится к подроду Drosophila и имеет монофилетическое происхождение [1]. Предварительная оценка времени дивергенции предковых форм D. virilis и D. melanogaster из подрода Sophopora, сделанная на основе анализа выборки ядерных генов, оказалась равна 62.9 млн лет [2]. Уточненный анализ, включающий данные секвенирования полных геномов 12 видов дрозофилы, позволил сдвинуть оценку времени дивергенции подродов Drosophila и Sophophora к позднему эоцену – 40 млн лет назад [3]. Анализ митохондриальной изменчивости в роде Drosophila подтверждает эти оценки [4]. Общий предок группы virilis существовал от 7.5 до 8.9 млн лет назад. Принятая в настоящее время система группы virilis основана на мультилокусном анализе изменчивости шести ядерных генов (Adh, fused, Gpdh, NonA, CG9631 и CG7219) и двух митохондриальных, 12S и 16S, рРНК генов [5]. В группе выделяют несколько филад. Филада virilis включает синантропный тропический вид D. virilis, D. lummei, обитающий в умеренной зоне Европы, и два близкородственных вида из умеренной и субтропической зон Северной Америки: D. americana americana, D. americana texana, D. novamexicana. Филада montana включает циркумбореальный вид D. montana и три вида дрозофил из умеренной зоны Северной Америки: D. lacicola, D. borealis, D. flavomontana. В умеренной зоне Европы обитает D. littoralis, которую замещает в Сибири D. ezoana, а на Дальнем Востоке D. ezoana замещает D. kanekoi. Ареалы видов широко перекрываются, что создает условия для межвидовой гибридизации [6].
В данной работе проанализирована согласованность филогений группы virilis, построенных на основе анализа изменчивости BOLD-фрагмента гена cox1 и фрагмента гена kl-2 1-beta dynein heavy chain, локализованного на Y-хромосоме. Благодаря отсутствию рекомбинации единичные события межвидовой гибридизации оставляют след в генофонде вида и могут быть обнаружены при анализе индивидуальной изменчивости. Ген динеина был приобретен Y-хромосомой предка рода Drosophila перед разделением его на подроды Drosophila и Sophophora, произошедшего между 260 и 63 млн лет назад [7]. У всех секвенированных видов Drosophila ген kl-2 остался связанным с Y-хромосомой, за исключением D. pseudoobscura [7]. В случае D. pseudoobscura предковая Y-хромосома стала частью аутосомы [8]. Сравнительное исследование выборки линий дрозофил по обоим маркерам позволяет выявить события межвидовой гибридизации в филогенетической истории вида и оценить значение этого фактора эволюции в группе дрозофил virilis.
МАТЕРИАЛЫ И МЕТОДЫ
Линии дрозофил группы virilis. Линии мух D. virilis, использованные в работе, получены из Генетической коллекции линий насекомых и клеточных культур насекомых ИОГен РАН (http://vigg.ru/index.php?id=337) и Коллекции генетических линий дрозофил ИБР РАН им. Н.К. Кольцова (http:// idbras.comcor.ru/collection/Drosophila.pdf). Линии мух, использованные в работе, являются изосамочными. Исключением являются синтетические лабораторные линии D. virilis L160 и D. americana texana 422. Список линий дрозофил группы virilis, использованных в работе, и данные регистрации нуклеотидных последовательностей маркерных генов приведены в табл. 1.
Таблица 1.
Линии дрозофил группы virilis и нуклеотидные последовательности маркерных генов
| Вид дрозофилы | Шифр линии | Место и год сбора линии | GenBank ID: kl-2 1-beta dynein heavy chain | GenBank ID: cox1 |
|---|---|---|---|---|
| D. virilis (Sturtevant, 1916) | Vi-B9 | Батуми, Грузия, 1965 г. | KF600716 | MH423344 |
| Vi-1 | Ереван, Армения, 1969 г. | MH423363 | JN019869 | |
| Vi-L160 | Синтетическая лабораторная линия, 1975 г. | MH423366 | MH423345 | |
| Vi-101 | Япония, 1966 г. | MH423364 | MH423342 | |
| Vi-119 | Китай, 1993 г. | MH423365 | MH423343 | |
| D. lummei (Hackman, 1972) | Lu-200 | Москва, Россия, 1969 г. | KF600723 | JN019868 |
| Lu-1109 | Каресйоки, Финляндия, 1972 г. | MH423357 | MH423336 | |
| D. novamexicana (Patterson, 1941) | No-424 | Нью-Мексико, США, 1947 г. | KF600725 | JN019867 |
| No-402 | Аризона, США, 1953 г. | MH423361 | MH423340 | |
| D. americana americana (Spencer, 1938) | Am-405 | Южная Каролина, США, 1961 г. | KF600714 | JN019919 |
| Am-403 | Южная Каролина, США, 1961 г. | MH423346 | MH423326 | |
| D. americana texana (Patterson, 1940) | Tx-423 | Новый Орлеан, США, 1947 г. | KF600726 | JN019928 |
| Tx-422 | Синтетическая лабораторная линия | MH423362 | MH423341 | |
| D. kanekoi (Watabe, Higuchi, 1979) | Ka-1061.00 | Саппоро, Япония, 1980 г. | MH423351 | JN019866 |
| D. ezoana (Takada, Okada, 1958) | Ez-572 | Кедровая Падь, Россия, 1987 г. | KF600717 | MH423329 |
| Ez-0971.00 | Хоккайдо, Япония, 1958 г. | MH423348 | JN019864 | |
| Ez-67 OJ8 | Оуланка, Финляндия, 2008 г. | MH423350 | MH423330 | |
| Ez-170 OJ8 | Оуланка, Финляндия, 2008 г. | MH423349 | MH423328 | |
| D. littoralis (Meigen, 1830) Северная | Lt-06-17a | Рыбинский, Россия, 2006 г. | KF600721 | JN019874 |
| Lt-Zv13 | Завидово, Россия, 2013 г. | MH423356 | MH423335 | |
| Lt-202 OJ8 | Оуланка, Финляндия, 2008 г. | MH423354 | MH423333 | |
| D. littoralis (Meigen, 1830) Южная | Lt-Ab10-58 | Пицунда, Абхазия, 2010 г. | MH423355 | MH423334 |
| D. borealis (Patterson, 1952) | Bo-0961.00 | Миннесота, США, 1950 г. | KF600715 | JN019865 |
| Bo-520 | Миннесота, США, 1950 г. | MH423347 | MH423327 | |
| D. lacicola (Patterson, 1944) | La-0991.00 | Нью-Йорк, США, 1973 г. | MH423352 | JN019871 |
| La-0991.13 | Манитоба, Канада, 1949 г. | KF600720 | MH423331 | |
| La-0991.17 | Юта, США, 1985 г. | MH423353 | MH423332 | |
| D. montana montana (Stone, Griffen, Patterson, 1941) | Mo-1021.13 | Кавасаки, Япония, 1965 г. | KF600724 | MH423337 |
| Mo-1021.19 | Орегон, США, 1949 г. | MH423358 | JN019870 | |
| D. montana ovivororum (Lakovaara, Hackman, 1973) | Mo-20_OL8 | Оуланка, Финляндия, 2008 г. | MH423359 | MH423338 |
| Mo-KR1309 | Камчатка, Россия, 2013 г. | MH423360 | MH423339 |
Выделение ДНК и условия ПЦР. ДНК из индивидуальных самцов имаго дрозофил выделяли методом фенол-хлороформной экстрации по стандартной методике [9]. Выделенная тотальная ДНК была растворена в 50 мкл деионизованной воды. Концентрация ДНК определялась спектрофотометрическим методом с использованием Implen NanoPhotometer NP80. После тестирования чистоты препарата по соотношению поглощения ультрафиолета при 260/280 нм концентрацию ДНК в препаратах выравнивали до 10 нг/мкл.
Реакцию амплификации проводили в конечном объеме 25 мкл с использованием наборов для амплификации EncycloPlus PCR kit (Евроген, Россия) в соответствии с инструкцией фирмы-производителя.
BOLD-фрагмент митохондриального гена cox1 получали с помощью стандартных фолмеровских праймеров: LCO1490 (5'-GGTCAACAAATCATAAAGATATTGG-3') и HCO2198 (5'-TAAACTTCAGGGTGACCAAAAAATCA-3') [10]. Условия ПЦР: первичная денатурация – 5 мин при 94°С; 5 циклов: денатурация при 94°С – 20 с, отжиг при 45°С – 20 с, синтез при 72°С – 40 с; затем еще 35 циклов: денатурация при 94°С – 20 с, отжиг при 55°С – 20 с, синтез при 72°С – 40 с и завершающий синтез при 72°С – 7 мин.
Для амплификации последовательностей гена динеина были использованы следующие праймеры: прямой праймер Dv-dy-f 5'-GCTGCAGGCGGTAATAGAAG-3' и обратный праймер Dv-dy-r 5'-TTGCATTTGCGGATCAATAA-3'. Длина амплифицированного фрагмента составляет 429 пн.
Условия ПЦР: первичная денатурация – 5 мин при 94°С; 38 циклов с использованием Encyclo-полимеразы: денатурация при 94°С – 30 с, отжиг при 58°С – 30 с, синтез при 72°С – 60 с; завершающий синтез при 72°С – 10 мин.
Элюция продуктов амплификации. Фрагменты, полученные в результате амплификации, очищали в 1.5%-ном агарозном геле. Элюция фрагментов из геля проводилась с использованием набора для элюции Zymoclean™ Gel DNA Recovery Kit (Zymo Research, USA) в соответствии с инструкцией фирмы-производителя.
Секвенирование. Нуклеотидную последовательность ПЦР-фрагментов определяли с прямого и обратного праймеров на приборе ABI PRISM 3500 с использованием реагентов BigDye®Terminator v. 3.1 Cycle Sequencing Kit (Applied Biosystems, США) согласно рекомендациям фирмы-производителя.
Биоинформационный анализ. Анализ хроматограмм проводили с помощью программы ChromasPro 13.3 (Technelysium, Australia). Выравнивание последовательностей, полученных в результате секвенирования, с последовательностями, размещенными в базах данных, было выполнено с использованием ресурсов NCBI (http://www.ncbi.nlm.nih.gov) и программы MEGA 7.0. [11]. Для построения медианной сети гаплотипов использовалась программа POPART [12] с использованием алгоритма TCS [13].
РЕЗУЛЬТАТЫ
Для выявления событий межвидовой гибридизации в группе дрозофил virilis мы сравнили две реконструкции филогенетической истории с использованием выборки изосамочных линий 11 видов дрозофил по маркерным генам Y-хромосомы и митохондриальной ДНК. На рис. 1 представлен полученный результат. Изменчивость BOLD-фрагмента митохондриального гена cox1 в 2 раза больше, чем изменчивость гена динеина. По митохондриальной ДНК наблюдается внутривидовая изменчивость, тогда как для большинства видов группы найден только один гаплотип динеина. Кластеризация линий по обоим маркерам совпадает для всех линий дрозофил, кроме двух. Эти две линии представляют особый интерес. Митохондриальный гаплотип линии D. montana KR1309, отловленной на Камчатке, как и ожидалось, кластеризуется с митохондриальными гаплотипами других линий D. montana и D. lacicola. Наибольшее сходство наблюдается с митохондриальным гаплотипом линии D. montana 20_OL8 из Финляндии, но гаплотип по динеину линии KR1309 отличается от гаплотипов других линий D. montana и кластеризуется с четырьмя линиями D. ezoana. Эти данные указывают на гибридное происхождение линии D. montana KR1309, происходящей от скрещивания самки D. montana с самцом D. ezoana. D. ezoana имеет два гаплотипа по гену динеина. Один найден в линиях, отловленных на территории Финляндии, другой – в линиях из Сибири и Японии. Совпадение гаплотипов динеина линий D. montana KR1309 и D. ezoana 0971.00 из Японии, географически близко расположенной к Камчатке, дополнительно подтверждает возможность межвидовой гибридизации самок D. montana с самцами D. ezoana в природе. Второй линией, имеющей гибридное происхождение, является D. lacicola 0991.17 из Северной Америки. На рис. 1 эта линия выделена звездочкой. Сравнение двух филогений, представленных на рис. 1,а, б, позволяет сделать вывод о происхождении этой линии от скрещивания самки D. montana с самцом D. lacicola. Вид D. montana разделяется на два подвида: D. montana montana, обитающий в Северной Америке, и D. montana ovivororum, распространенный в Северной Евразии. Эти два подвида надежно различаются по нуклеотидной последовательности BOLD-фрагмента. Как и ожидалось, митохондриальная интрогрессия у D. lacicola происходит от симпатрически обитающей D. montana montana.
Рис. 1.
Сравнение кластеризации видов дрозофил группы virilis по данным изменчивости митохондриального cox1 гена и маркерного гена Y-хромосомы kl-2 1-beta dynein heavy chain. а – кладограмма группы virilis по данным изменчивости BOLD-фрагмента митохондриального гена cox1 длиной 658 пн. Кладограмма Neighbor-Joining, модель нуклеотидных замещений (p-distance) с bootstrap-поддержкой – 1000 реприк построена в программе MEGA 7. Величина bootstrap-поддержки кластеризации таксонов в процентах приведена рядом с узлами кладограммы. Кладограмма построена в масштабе числа нуклеотидных замен на сайт. В качестве внешней группы взята D. melanogaster GenBank ID: EU685283. Буквы в начале названий линий обозначают вид дрозофилы (Am – D. americana americana, No – D. novamexicana, Tx – D. americana texana, Mo – D. montana, La – D. lacicola, Ez – D. ezoana, Bo – D. borealis, Lt – D. littoralis, Ka – D. kanekoi, Lu – D. lummei, Vi – D. virilis), цифры – шифр линии. Характеристика линий дрозофил и GenBank ID нуклеотидных последовательностей приведены в табл. 1. Линии мух, имеющие гибридное происхождение, отмечены на филограммах рамкой и звездочкой; б – кладограмма группы virilis по данным изменчивости фрагмента гена kl-2 1‑beta dynein heavy chain длиной 429 пн. Обозначение названий линий дрозофил и их характеристика – как в позиции а.

ОБСУЖДЕНИЕ
В данном исследовании были получены новые результаты по изменчивости BOLD-фрагмента митохондриального гена cox1 и маркерного гена Y-хромосомы kl-2 1-beta dynein heavy chain у дрозофил группы virilis. Совремнная филогения группы virilis, полученная на основе данных мультилокусного анализа, не позволяет однозначно определить положение D. kanekoi [5]. Консенсусная модель дерева для анализа мультилокусных данных показывает, что D. kanekoi и D. ezoana образуют один кластер, а D. littoralis имеет более древнее происхождение. Применение другой модели для анализа мультилокусных данных с помощью алгоритма (BEST) определяет, что D. kanekoi – это первый вид, отделившийся от общего предка. В нашем исследовании были получены новые данные по изменчивости двух маркерных генов нерекомбиногенных областей генома 11 видов группы virilis. Данные по изменчивости гена динеина, который у дрозофил служит маркером Y‑хромосомы, получены нами для дрозофил группы virilis впервые. На основе этих результатов мы провели уточнение неясных вопросов филогении группы. Филогенетический анализ изменчивости динеина указывает на монофилию Y‑хромосомы в группе. На рис. 2 представлена медианная сеть взаимопревращений гаплотипов динеина, построенная с помощью алгоритма TCS. Выявлены четыре кластера, происходящих от общего предка, соответствующие филадам группы virilis. В филаде D. virilis корневым видом является D. virilis. От нее происходят адаптированные к более холодному климату D. lummei в Европе и североамериканские виды D. americana americana, D. novamexicana. В филаде D. montana также первым оформляется более теплолюбивый вид D. borealis, а затем более холодоустойчивые D. montana и D. lacicola. Филада D. littoralis представлена двумя подвидами. Первым оформляется южный подвид D. littoralis, от него происходит северный подвид. Эти данные поддерживают сделанное ранее предположение о том, что южная популяция D. littoralis представляет собой отдельный вид – D. imeretensis [14]. Четвертый кластер включает два вида, распространенных в Азии: D. kanekoi и D. ezoana. Сравнение филогений, полученных с помошью алгоритмов TCS и NJ (рис. 1 и 2), показывает неустойчивое положение D. kanekoi. Согласно реконструкции филогенетической истории Y-хромосомы, D. kanekoi является наиболее древним видом группы, тогда как филогения, построенная на данных митохондриальной изменчивости, показывает, что первым событием видообразования в группе было оформление филады D. virilis.
Рис. 2.
Медианная сеть гаплотипов Y-хромосомы дрозофил группы virilis. Медианная сеть построена на основании нуклеотидного полиморфизма фрагмента гена kl-2 1-beta dynein heavy chain длиной 429 пн. Штрихи обозначают минимальное число нуклеотидных замен, являющееся необходимым для трансформации одного гаплотипа в другой. Размер кружков пропорционален числу синонимичных гаплотипов. В том случае, если вид дрозофил имеет более одного гаплотипа, после видового имени добавлена буква, указывающая на географическую локальность, где этот гаплотип был найден. F – Финляндия; R – Сибирь и Дальний Восток; S – Закавказье, N – Европейская равнина (см. табл. 1).
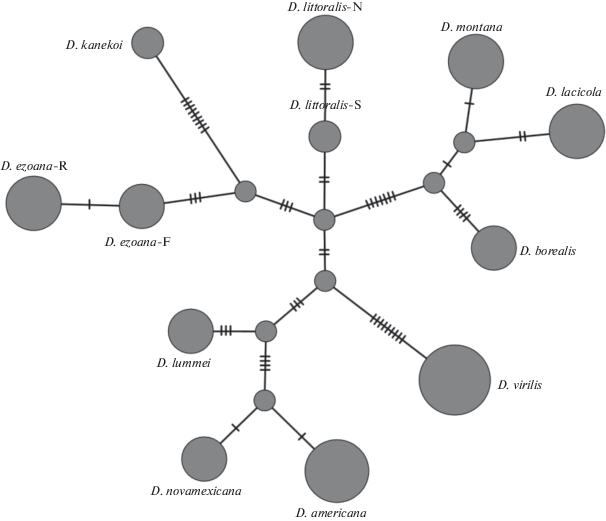
Сравнительный анализ 31 линии дрозофил, относящихся к 11 видам, позволил нам выявить одно событие митохондриальной интрогресии, возникшее при межвидовой гибридизации самок D. montana с самцами D. lacicola в Северной Америке, и одно событие замены Y-хромосомы при межвидовой гибридизации самок D. montana с самцами D. ezoana на Камчатке. В обоих случаях межвидовая гибридизация произошла с участием D. montana, что вероятно не случайно. Межвидовую гибридизацию можно рассматривать как одно из проявлений геномной нестабильности у видов, находящихся в процессе видообразования. Межвидовая гибридизация не только признак, но и причина геномной нестабильности. Существование периода геномной нестабильности в период видообразования предсказывается теорией видообразования путем прерывистого равновесия [15]. Перемещения мобильных элементов в геноме – еще один признак геномной нестабильности и процесса видообразования [16]. В случае D. montana ovivororum выполняются оба условия, необходимые для процесса видообразования. Ранее мы получили данные о недавних перемещениях ретротранспозона Tv1 в геноме евроазиатского подвида D. montana [17]. Эти данные позволяют предполагать наличие геномной нестабильности у D. montana ovivororum и идущем у этого вида процессе видообразования.
Работа выполнена при финансовой поддержке проекта (АААА-А16-116111610180-3) “Изучение изменчивости автономных генетических элементов насекомых и разработка маркеров нестабильности генома” (Договор: 0112-2016-0001) и Программы фундаментальных исследований Президиума РАН “Биоразнообразие природных систем”, раздел “Генофонды живой природы и их сохранение”.
Список литературы
van der Linde K., Houle D. A supertree analysis and literature review of the genus Drosophila and closely related genera (Diptera, Drosophilidae) // Insect System. Evol. 2008. V. 39. P. 241–267. doi https://doi.org/10.1163/ 187631208788784237
Tamura K., Subramanian S., Kumar S. Temporal patterns of fruit fly (Drosophila) evolution revealed by mutation clocks // Mol. Biol. Evol. 2004. V. 21. P. 36–44. doi https://doi.org/10.1093/molbev/msg236
Bhutkar A., Russo S.M., Smith T.F., Gelbart W.M. Genome-scale analysis of positionally relocated genes // Genome Res. 2007. V. 17. P. 1880–1887. doi https://doi.org/10.1101/ gr.7062307
Wang B.C., Park J., Watabe H.A. et al. Molecular phylogeny of the Drosophila virilis section (Diptera: Drosophilidae) based on mitochondrial and nuclear sequences // Mol. Phylogenet. Evol. 2006. V. 40. P. 484–500. doi https://doi.org/10.1016/j.ympev.2006.03.026
Morales-Hojas R., Reis M., Vieira C.P., Vieira J. Resolving the phylogenetic relationships and evolutionary history of the Drosophila virilis group using multilocus data // Mol. Phylogenet. Evol. 2011. V. 60. P. 249–258. doi https://doi.org/10.1016/j.ympev.2011.04.022
Caletka B., McAllister B. A genealogical view of chromosomal evolution and species delimitation in the Drosophila virilis species subgroup // Mol. Phylogenet. Evol. 2004. V. 33. P. 664–670. doi https://doi.org/10.1016/j.ympev.2004.08.007
Koerich L.B., Wang X., Clark A.G., Carvalho A.B. Low conservation of gene content in the Drosophila Y chromosome // Nature. 2008. V. 456. P. 949–951. doi https://doi.org/10.1038/nature07463
Carvalho A.B., Clark A.G.Y. Y Chromosome of D. pseudoobscura is not homologous to the ancestral Drosophila Y // Science. 2005. V. 307. P. 108–110. doi https://doi.org/10.1126/ science.1101675
Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии. Молекулярное клонирование. М.: Мир, 1984. 479 с.
Folmer O., Black M., Hoeh W. et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates // Mol. Mar. Biol. Biotechnol. 1994. V. 3. P. 294–299.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis Version 7.0 for Bigger Datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874. doi https://doi.org/10.1093/molbev/msw054
Leigh J.W., Bryant D. POPART: full-feature software for haplotype network construction // Methods Ecol. Evol. 2015. V. 6. P. 1110–1116. doi https://doi.org/10.1111/2041-210X.12410
Clement M., Posada D., Crandall K.A. TCS: A computer program to estimate gene genealogies // Mol. Ecol. 2000. V. 9. P. 1657–1659. doi https://doi.org/10.1046/j.1365-294x.2000.01020.x
Соколов Н.Н. Новый вид дрозофил – Drosophila imeretensis // Докл. Акад. наук СССР. 1948. Т. 59. № 5. С. 1007–1008.
Eldredge N., Gould S.J Punctuated equilibria: an alternative to phyletic gradualism // Models in Paleobiology / Ed. Schopf T.J.M. San Francisco: Freeman Co, 1972. P. 82–115.
Sverdlov E.D. Retroviruses and primate evolution // BioEssays. 2000. V. 22. P. 161–171. doi https://doi.org/10.1002/(SICI)1521-1878(200002)22:2<161::AID-BIES7>3.0.CO;2-X
Андрианов Б.В., Романов Д.А. Транспозиции сайт-специфичного ретротранспозона Tv1 маркируют события видообразования у дрозофил из группы virilis // Актуальные проблемы биологической и химической экологии. М.: Изд-во МГОУ, 2016. С. 89–93.
Дополнительные материалы отсутствуют.


