Генетика, 2019, T. 55, № 5, стр. 524-530
Морфологическая и молекулярно-генетическая верификация межвидового гибрида Salix × zhataica (Salicaceae) из Центральной Якутии
А. П. Ефимова 1, Т. А. Полякова 2, *, М. М. Белоконь 2, Ю. С. Белоконь 2, Д. В. Политов 2
1 Институт биологических проблем криолитозоны Сибирского отделения
Российской академии наук
677980 Якутск, Россия
2 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
* E-mail: tat-polyakova@yandex.ru
Поступила в редакцию 31.05.2018
После доработки 04.07.2018
Принята к публикации 05.07.2018
Аннотация
Приводятся результаты морфологического и молекулярно-генетического анализов, подтверждающие гибридную природу Salix × zhataica Efimova, Shurduk et Ahti (ива жатайская). В частности, по совокупности фенотипических признаков (на основании дискриминантного анализа) гибрид, полученный, по всей вероятности, в результате межвидовой гибридизации S. brachypoda (Trautv. et C.A. Mey.) Kom. и S. pyrolifolia Ledeb., оказался более близким S. brachypoda, что косвенно свидетельствует о возможности этого “родителя” быть материнским. Для верификации гибрида и его предполагаемых родительских видов проанализирован полиморфизм таких молекулярно-генетических маркеров, как ядерные микросателлитные локусы и нуклеотидные последовательности ITS-региона, которые показали высокую эффективность использования. Например, в ITS-регионе обнаружены две однонуклеотидные транзиции, свидетельствующие о заимствовании гибридом соответствующих нуклеотидов от обоих родительских видов. Приведенные свидетельства показывают, что S. × zhataica действительно является гибридом между S. brachypoda и S. pyrolifolia.
Ивы (Salix L., Salicaceae Mirb.) известны как одни из наиболее изменчивых представителей древесных растений, среди которых часто наблюдаются случаи естественной внутриродовой интрогрессивной гибридизации [1–6].
В 2009 г. [7] был обнародован новый межвидовой гибрид ивы, обнаруженный в бассейне Средней Лены между 62° и 63° с.ш., 129° и 130° в.д. Гибрид получил бинарное название Salix × zhataica Efimova, Shurduk et Ahti – ива жатайская. Гипотеза гибридности найденных особей и версия его родительских видов – S. brachypoda (Trautv. et C.A. Mey.) Kom. (ива коротконожковая) и S. pyrolifolia Ledeb. (грушанколистная) – были выдвинуты авторами по результатам сравнительно-морфологического анализа. Гербарный аутентик (голотип) гибрида находится в гербарии Института биологических проблем криолитозоны СО РАН в Якутске (SASY), изотипы – в H, LE.
Родительские виды принадлежат к разным довольно отдаленным секциям: S. brachypoda – к Incubaceae A. Kern., S. pyrolifolia – к Hastatae A. Kern. подрода Vetrix Dumort. [8]. Число хромосом S. pyrolifolia – 2n = 38 [9]. Данные о числах хромосом S. brachypoda в литературе отсутствуют. Гетерозисные явления не наблюдаются. S. × zhataica представлен и женскими, и мужскими особями почти в равных пропорциях. Растения образуют жизнеспособные семена: лабораторная всхожесть составляет 90%.
Гибрид спорадически встречается на естественных, практически неизмененных человеком долинных экотопах, где, как правило, симпатрично произрастают оба родительских вида. К настоящему времени установлены четыре дополнительные точки нахождения в Якутии: три – в бассейне Средней Лены, одна – в Юго-Восточной Якутии (окр. кордона Чабда в низовьях р. Маи). Среди гибридного потомства, вопреки ожиданиям, F1 не наблюдается, регистрируются лишь бэккроссы. Отсутствие первого поколения, вероятно, объясняется продолжительностью гибридизационного процесса и коротким жизненным циклом ив. В гибридных зонах “чистые” особи S. brachypoda и S. pyrolifolia внешне хорошо различимы, но часто наблюдаются единичные признаки, выходящие за рамки характерных для этих видов пределов морфологической изменчивости. Это свидетельствует о циркулировании интрогрессивных генов в “чистых” популяциях.
Закономерности наследования и экспрессии генов в гибридной зоне свидетельствуют о том, что морфологические признаки зачастую не проявляются простым аддитивным образом. В связи с этим необходимо рассматривать проблему на основе совокупности морфологических и молекулярных данных [5, 10], поэтому для более убедительного подтверждения гибридной природы S. × zhataica нами проведен молекулярно-генетический анализ.
МАТЕРИАЛЫ И МЕТОДЫ
Материал гибридных особей собран в loсus classicus в окрестностях пос. Жатай (Zhat) близ г. Якутска (Центральная Якутия), а также на месте сбора изотипов – в распадке коренного берега близ Вилюйского тракта (Vilu). “Чистые” образцы родительских видов были собраны как в окрестностях пос. Жатай, так и в удаленных от места гибридизации пунктах: Зеленом лугу (окрестности г. Якутска), близ с. Хатырык и с. Тюбятцы (Центральная Якутия, левобережье среднего течения р. Лены), на Ленских Столбах, в среднем течении р. Яны (Северо-Восточная Якутия), а также с фондовых гербарных материалов с устья р. Алдан (Центральная Якутия) и близ устья р. Олекма (Юго-Западная Якутия). Материалом для анализа микросателлитной изменчивости послужили образцы листьев индивидуальных особей Salix braсhypoda (n = 15), S. pyrolifolia (n = 15) и S. × zhataica (n = 8). Тестирование и отбор пригодных для анализа гибридизации микросателлитных локусов проводили на четырех образцах S. braсhypoda и четырех образцах S. pyrolifolia из зон аллопатрии.
Для анализа морфологических признаков в locus classicus собирались особи, отображающие весь набор фенотипических вариаций гибрида. Среди родительских видов были отобраны типичные особи. Изучены следующие внешние признаки: длина листовой пластинки; ширина листовой пластинки; длина черешка; длина прилистника; ширина прилистника; длина ножки коробочки. Эти параметры четко дифференцируют внешние особенности изучаемых видов и их гибрида. Дискриминантный анализ морфометрических данных проведен в программе Statistica [11].
Для выделения ДНК с 2-летних побегов собирались молодые неповрежденные листья и сушились в силикагеле. Выделение ДНК проводили по стандартным методикам для растительных тканей из 20–25 мг сухого растительного материала с применением цетилтриметиламмониумбромида (CTAB) [12, 13].
Тестирование проводили с использованием молекулярного маркирования образцов по 14 ядерным микросателлитным локусам [14, 15], разработанным для разных видов ив и входящим в набор тест-системы для индивидуальной идентификации представителей рода Salix: Sa54B, SB24, SB38, SB80, SB85, SB88, SB100, SB196, SB210, SB233, SB243, SB525, SB880 и SB904 [16].
Для проведения полимеразной цепной реакции использовали наборы реагентов для ПЦР-амплификации ДНК “GenePak® PCR Core” производства ООО “Лаборатория Изоген” согласно прилагаемым инструкциям. Амплификацию ядерных микросателлитных локусов проводили с использованием режима ПЦР, включавшего предварительную денатурацию ДНК при 94°С в течение 3 мин, затем 35 циклов, состоящих из 30 с денатурации при 94°С, 30 с отжига при 50°С и инкубирования 1 мин 30 с при 72°С. Финальная элонгация проводилась 15 мин при температуре 72°С. Для локуса SB38 температура отжига была 48°С. Программа завершалась охлаждением реакционной смеси до 4°С. Продукты ПЦР (амплификаты) анализировали с помощью электрофореза в 6%-ном полиакриламидном геле (ПААГ) в трис-ЭДТА-боратной буферной системе. После электрофореза гели окрашивали в растворе бромистого этидия и визуализировали в УФ-свете. Графические изображения гелей (электрофореграммы) сохраняли с помощью системы гель-документации “Doc-Print II” (Vilbet Lourmat) с дальнейшей обработкой в графических редакторах. Размер амплифицированных фрагментов определяли с помощью программного обеспечения Photo-Capt (Vilbet Lourmat). В качестве стандартного маркера длины использовали ДНК плазмиды pBR322 E. coli, обработанную эндонуклеазой рестрикции HpaII, с диапазоном длин фрагментов от 9 до 622 пар нуклеотидов (пн).
Анализ полученных данных проводили с помощью надстройки для MS Excel GenAlEx [17, 18]. Распределение генотипов с помощью метода главных компонент выполнено в программе Statistica [11].
При амплификации фрагмента ITS-оперона использовали праймеры ITS6 и ITS9, разработанные для восточноазиатских видов трибы Spiraeeae [19]. Цикл амплификации включал денатурацию при 94°С в течение 1 мин, отжиг праймеров при 58°С в течение 50 с и элонгацию при 72°С в течение 1 мин с числом циклов – 30. Полученные ПЦР-фрагменты были очищены набором реагентов для быстрой элюции ДНК из агарозных гелей Diatom DNA Elution (ООО “Лаборатория Изоген”). Секвенирование ITS-фрагментов проводили в ЗАО “Евроген” в обоих направлениях. Сиквенсы были попарно выравнены в программе BioEdit [20], множественное выравнивание выполнено в программе ClustalW2 с последующей визуальной проверкой спорных позиций на хроматограммах.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Выдвинутая ранее гипотеза, что Salix × zhataica действительно является межвидовым (межсекционным) гибридом двух видов – S. brachypoda и S. pyrolifolia [7], подтверждается нами на основе анализа полиморфизма ядерных микросателлитных локусов (nrSSR), изменчивости нуклеотидных последовательностей ITS-региона рДНК, а также совокупности диагностических морфологических признаков.
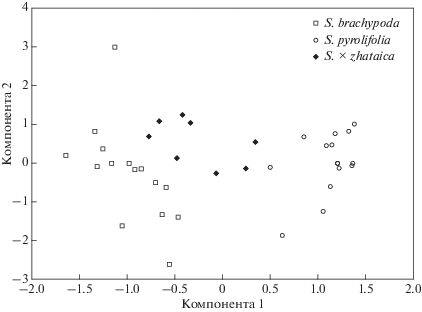
У образцов гибрида S. × zhataica и его родительских видов S. pyrolifolia, S. brachypoda изучены диагностические морфометрические признаки, характеризующие размеры листовой пластинки, черешка и прилистников, а также длину ножки коробочки. Дискриминантный анализ фенотипических признаков показал связи между гибридом и исходными видами (рис. 1). В диаграмме по осям 1 и 2 выделились три группы, соответствующие двум родительским видам и гибриду. При этом совокупность образцов гибрида расположилась между обоими видами. Некоторое смещение его “облака” относительно родительских групп показывает, что гибрид имеет собственные фенотипические очертания. При этом на диаграмме совокупность исследованных образцов гибрида находится ближе к S. brachypoda, чем к S. pyrolifolia, что косвенно свидетельствует о том, что первый является материнским “родителем”. От него унаследовано подавляющее большинство морфологических признаков, что подтверждается камеральными сравнительно-морфологическими исследованиями [7].
Рис. 1.
Диаграмма дискриминантного анализа образцов гибрида Salix × zhataica и его родительских видов S. pyrolifolia, S. brachypoda по морфометрическим признакам.
Проведенный микросателлитный анализ показал, что по большинству из 14 ядерных микросателлитных локусов спектры амплифицируемых фрагментов S. brachypoda и S. pyrolifolia перекрывались по длинам. В отдельных локусах наблюдалось отсутствие амплификации у некоторых образцов (т.н. “нуль-аллели”). В результате тестирования были отобраны три локуса – Sa54B, SB24 и SB233, позволяющие наиболее четко отличить представителей этих двух видов. Характеристики продуктов амплификации этих локусов приведены в табл. 1.
Таблица 1.
Характеристики амплифицируемых фрагментов ядерных микросателлитных локусов S. brachypoda, S. pyrolifolia и S. × zhataica
| Локус | Общее число аллелей | Длины амплифицируемых фрагментов | ||
|---|---|---|---|---|
| S. brachypoda (n = 15) | S. pyrolifolia (n = 15) | S. × zhataica (n = 8) | ||
| Sa54B | 10 | 105–131 (6) | 117–141 (7) | 117–135 (5) |
| SB24 | 22 | 143–177 (12) | 129–141, 179 (6) | 135–211 (7) |
| SB233 | 11 | 199–225 (8) | 211–221 (5) | 203–227 (5) |
Длины амплифицируемых фрагментов ядерных микросателлитных локусов S. × zhataica в большинстве случаев совпадали с фрагментами S. brachypoda или S. pyrolifolia. В локусе Sa54B был обнаружен один аллель, отсутствующий у чистых видов, а в локусе SB24 таких аллелей выявлено три. Их наличие свидетельствует о том, что в нашем материале недостаточно представлены S. brachypoda и S. pyrolifolia, и обнаруженная нами изменчивость не охватывает весь спектр генетических вариаций, присутствующих в естественных популяциях. Однако этот факт не влияет на приведенные в данном сообщении выводы.
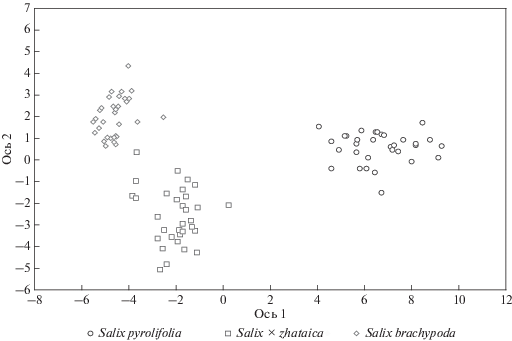
По результатам факторного анализа распределения генотипов представители видов S. brachypoda и S. pyrolifolia также четко различаются (рис. 2). Особи, отнесенные на основании морфологического анализа к S. × zhataica, на плоскости двух первых главных компонент занимают промежуточное положение между S. brachypoda и S. pyrolifolia. Причем некоторые из них обладают большей схожестью с одним из родительских видов, что позволяет предположить возможность возвратных скрещиваний S. × zhataica с S. brachypoda.
Для подтверждения гибридной природы образцов S × zhataica также были привлечены нуклеотидные последовательности ITS-региона рДНК, которые в настоящее время зарекомендовали себя наиболее востребованными маркерами для видоидентификации растений [21–24] благодаря ряду преимуществ, среди которых высокая вариабельность, консервативная протяженность, высокая копийность, а также двуродительское наследование [25], обеспечивающее идентификацию недавно возникших гибридов [26].
Данные по нуклеотидной изменчивости ITS-региона рДНК S. brachypoda, S. pyrolifolia, S × zhataica также указывают на гибридную природу последнего. В результате амплификации для каждого образца были получены фрагменты длиной около 650 пн, включавшие полноразмерный регион межгенных спейсеров ITS1 и ITS2, а также гена 5.8S. После выравнивания длина анализируемого фрагмента ITS насчитывала 585 позиций (табл. 2). Сравнительное изучение ITS-фрагмента у изученных видов и гибрида показало наличие двух точечных мутаций – транзиций (табл. 3). Обнаруженная нами относительно слабая изменчивость ITS-региона согласуется с ранее полученными данными по семейству Salicaceae [27] и роду Salix [28]. Проанализированы различия в нуклеотидном составе фрагментов ITS изучаемых видов, в том числе трех образцов гибридов. Позиция 139 в спейсере ITS1 является дискриминирующей. Несмотря на то что позиция 368 гена 5.8S рДНК представляет собой синонимичный сайт, демонстрируемые различия также указывают на гибридизацию Salix brachypoda и S. pyrolifolia и соответственно гибридное происхождение S. × zhataica. Таким образом, анализ ITS-региона рДНК для видов S. brachypoda, S. pyrolifolia и их гибрида S. × zhataica выявил специфические однонуклеотидные замены, указывающие на гибридное происхождение S. × zhataica.
Таблица 2.
Характеристики ITS1–5.8S–ITS2-региона рДНК у S. brachypoda, S. pyrolifolia и их гибрида S. × zhataica
| Показатель | ITS1 | 5.8S | ITS2 | Всего |
|---|---|---|---|---|
| Выравненная длина, пн | 221 | 167 | 197 | 585 |
| GC-состав, % | 66.2 | 55.1 | 68.6 | – |
| Число константных сайтов | 220 | 166 | 197 | 583 |
| Число вариабельных сайтов (замен) | 1 | 1 | 0 | 2 |
Таблица 3.
Однонуклеотидные замены в регионе ITS рДНК у видов S. brachypoda, S. pyrolifolia и их гибрида S.× zhataica
| Позиция | Участок ITS | S. brachypoda | S. × zhataica | S. pyrolifolia | ||
|---|---|---|---|---|---|---|
| Zhat | Vilu1 | Vilu2 | ||||
| 139 | ITS1 | T | C | C | C | C |
| 368 | 5.8S | C | T | C | C | T |
Выдвинутая ранее гипотеза, что Salix × zhataica действительно является межвидовым (межсекционным) гибридом двух видов – S. brachypoda и S. pyrolifolia [7], подтверждается нами на основе анализа полиморфизма ядерных микросателлитных локусов, изменчивости нуклеотидных последовательностей ITS-региона рДНК, а также совокупности диагностических морфометрических признаков. По результатам дискриминантного анализа гибрид по совокупности своих морфометрических признаков занимает промежуточное положение, но ближе к S. brachypoda. Анализ нуклеотидного состава ITS-региона рДНК показал наличие двух однонуклеотидных транзиций, указывающих на заимствование гибридом соответствующих нуклеотидов от обоих родительских видов. Анализ совокупности изученных морфологических и молекулярных признаков свидетельствует о гибридном происхождении S. × zhataica, а также о возвратных скрещиваниях S. × zhataica и S. brachypoda, в которых последняя выполняет функцию материнской особи.
Исследование поддержано Проектом ИБПК СО РАН VI.52.1.8 “Фундаментальные и прикладные аспекты изучения разнообразия растительного мира Северной и Центральной Якутии” (0376-2018-0001, рег. номер АААА-А17-117020110056-0); грантом РФФИ № 15-44-05103 “р_восток_a”, а также частично Программой фундаментальных исследований Президиума РАН № 32 “Эволюция органического мира. Роль и влияние планетарных процессов” (тема 0112-2018-0027 “Изучение генетических механизмов эволюции на геномном и организменном уровне: роль гибридизации, последствия глобальных экологических изменений”).
Список литературы
Беляева И.В. Новый гибрид Salix triandra и S. dasyclados (Salicaceae) // Бот. журн. 1998. Т. 83. № 11. С. 111–113.
Elven R., Karlsson T. Salix L. 1. Lycopodiaceae to Polygonaceae // Flora Nordica. Stockholm: The Bergius Foundation, 2000. P. 117–188.
Triest L. Hybridisation in staminate and pistillate Salix alba and S. fragilis (Salicaceae): morphology versus RAPDs // Plant Systemat. Evol. 2001. V. 226. P. 143–154.
Коропачинский И.Ю., Милютин Л.И. Естественная гибридизация древесных растений. Новосибирск: Гео, 2006. 223 с.
Fogelqvist J., Verkhozina A.V., Katyshev A.I. et al. Genetic and morphological evidence for introgression between three species of willows // BMC Evol. Biol. 2015. V. 15. № 193. 10 p. doi https://doi.org/10.1186/s12862-015-0461-7
Gramlich S., Sagmeister P., Dullinger S. et al. Evolution in situ: hybrid origin and establishment of willows (Salix L.) on alpine glacier forefields // Heredity. 2016. V. 116. P. 531–541.
Ефимова А.П., Шурдук И.Ф., Ахти T. Новый межвидовой гибрид рода Salix (Salicaceae) из Якутии // Бот. журн. 2009. Т. 94. № 1. С. 83–89.
Скворцов А.К. Ивы СССР: систематический и географический обзор. М.: Наука, 1968. 262 с.
Фёдорова-Саркисова О.В. О числе хромосом некоторых видов ив и тополей // Докл. АН СССР. 1946. Т. 54. № 4. С. 357–360.
Hardig T.M., Brunsfeld S.J., Fritz R.S. et al. Morphological and molecular evidence for hybridization and introgression in a willow (Salix) hybrid zone // Mol. Ecol. 2000. V. 9. P. 9–24.
StatSoft, Inc. STATISTICA for Windows [Computer program manual]. 1998. WEB: http://www.statsoft.com.
Doyle J.J., Doyle J.L. Isolation of plant DNA from fresh tissue // Focus. 1990. V. 12. P. 12–15.
Devey M.E., Bell J.C., Smith D.N. A genetic linkage map for Pinus radiata based on RFLP, RAPD, and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. P. 673–679.
Barker J.H.A., Pahlich A., Trybush S. et al. Microsatellite markers for diverse Salix species // Mol. Ecol. Notes. 2003. V. 3. № 1. P. 4–6.
King R.A., Harris S.L., Karp A., Barker J.H.A. Characterization and inheritance of nuclear microsatellite loci for use in population studies of the allotetraploid Salix alba – Salix fragilis complex // Tree Genetics & Genomes. 2010. V. 6. № 2. P. 247–258.
Политов Д.В., Белоконь М.М., Белоконь Ю.С. и др. Способ молекулярного маркирования, основанный на микросателлитных локусах, предназначенный для идентификации генотипов ивы: пат. 2630662 Рос. Федерации. 2017. Бюл. № 26.
Peakall R., Smouse P.E. GenAlEx 6: genetic analysis in Excel. Population genetic software for teaching and research // Mol. Ecol. Notes. 2006. V. 6. P. 288–295.
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update // Bioinformatics. 2012. V. 28. P. 2537–2539.
Potter D., Still S.M., Grebenc T. et al. Phylogenetic relationships in tribe Spiraeeae (Rosaceae) inferred from nucleotide sequence data // Plant Systemat. Evol. 2007. V. 266. P. 105–118.
Hall T.A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Acids Symp. 1999. Ser. 41. P. 95–98.
Baldwin B.G., Sanderson M.J., Porter J.M. et al. The ITS region of nuclear ribosomal DNA: A valuable source of evidence on angiosperm phylogeny // Annals Missouri Bot. Garden. 1995. V. 82. P. 247–277.
Рыжова Н.Н., Бурляева М.О., Кочиева Е.А., Вишнякова М.А. Использование ITS-последовательностей для оценки таксономических отношений у представителей трибы Viciae (Adans.) Bronn Сем. Fabaceae Lindl. // Экол. генетика. 2007. № 3. С. 5–14.
Polyakova T.A. Nuclear ribosomal DNA ITS region variability in the genus Spiraea from Asian Russia // Molecular Phylogenetics: Contributions to the 4th Moscow Intern. Conf. “Molecular Phylogenetics” (Moscow, Russia, September 23–26, 2014). Moscow: TORUS PRESS, 2014. P. 58.
Полякова Т.А., Шатохина А.В. Филогенетические взаимоотношения российских видов рода Spiraea L. (Rosaceae Juss.) по морфологическим и молекулярным данным // 50 лет без К.И. Мейера. XIII Моск. совещ. по филогении растений: Материалы междунар. конф. (2–6 февраля 2015 г., Москва) / Под ред. Тимонина А.К. М.: МАКС Пресс, 2015. С. 263–265.
Матвеева Т.В., Павлова О.А., Богомаз Д.И. и др. Молекулярные маркеры для видоидентификации и филогенетики растений // Экол. генетика. 2011. Т. IX. № 1. С. 32–43.
Alvarez I.A., Wendel J.F. Ribosomal ITS sequences and plant phylogenetic inference // Mol. Phylogenet. Evol. 2003. V. 29. № 3. P. 417–434.
Leskinen E., Alström-Rapaport C. Molecular phylogeny of Salicaceae and closely related Flacourtiaceae: evidence from 5.8S, ITS1 and ITS2 of the rDNA // Plant Systemat. Evol. 1999. V. 215. P. 209–227.
Ефимова А.П., Полякова Т.А., Шатохина А.В. Изменчивость нуклеотидного состава ITS-региона различных видов ив из Якутии // Проблемы изучения растительного покрова Сибири: Материалы V междунар. науч. конф. (Томск, 20–22 октября 2015 г.). Томск, 2015. С. 67–69.
Дополнительные материалы отсутствуют.