Генетика, 2019, T. 55, № 5, стр. 574-582
Структура и формирование митохондриального генофонда русского населения Восточной Европы
Б. А. Малярчук 1, *, А. Н. Литвинов 1, М. В. Деренко 1
1 Институт биологических проблем Севера Дальневосточного отделения
Российской академии наук
685000 Магадан, Россия
* E-mail: malyarchuk@ibpn.ru
Поступила в редакцию 23.05.2018
После доработки 05.07.2018
Принята к публикации 22.08.2018
Аннотация
Филогеографический анализ изменчивости целых митохондриальных геномов у русского населения Восточной Европы (n = 557) показал, что в митохондриальном генофонде русских присутствуют подгруппы мтДНК, характерные только для русских и других восточных славян (11.7%), а также для славян в целом (12.2%), славян и германцев (6.6%), славян и прибалтийских финнов (5.7%). Результаты молекулярного датирования свидетельствуют о том, что митохондриальные подгруппы, характерные для русских и славян в целом, начали формироваться 2.7–3.5 тыс. лет тому назад. Результаты исследования динамики эффективной численности популяций, основанного на байесовском анализе данных об изменчивости гаплотипов мтДНК, специфичных для русских и славян, показали, что резкое увеличение популяционных размеров произошло сразу же после периода снижения численности 4–5 тыс. лет тому назад. Полученные данные обсуждаются в сравнении с результатами анализа полиморфизма Y-хромосомы в популяциях Европы.
Исследования изменчивости митохондриальной ДНК (мтДНК) русского населения Восточной Европы, проведенные с помощью секвенирования целых митохондриальных геномов (митогеномов), позволили описать основные черты митохондриального генофонда русских [1, 2]. Русские характеризуются типично европейским набором гаплогрупп мтДНК, а вклад восточноазиатских и африканских линий в структуру генофонда – невелик (менее 2%). Анализ межпопуляционной дифференциации показал отсутствие генетических различий между исследованными русскими популяциями (Fst = 0.22%, p = 0.15) [1]. Полученные данные в целом хорошо согласуются с результатами предыдущих исследований, выполненных на менее информативном уровне (т.е. с помощью секвенирования гипервариабельных участков ГВС1 и ГВС2, а также анализа полиморфизма гаплогруппо-специфичных локусов кодирующей области мтДНК) [3–5]. Между тем полномитогеномный анализ позволил намного более детально описать структуру генофонда русских, выявить множество новых подгрупп мтДНК и провести ревизию европейских гаплогрупп мтДНК. Анализ целых митогеномов 376 русских индивидуумов позволил также получить новую информацию относительно демографической истории населения Восточной Европы. Нами было обнаружено, что наряду с картиной изменений эффективной численности популяций (Ne), выявленной и в других менее масштабных исследованиях мтДНК европейских популяций (т.е. падением Ne во время ледникового максимума и ее ростом в послеледниковое время и голоцене), в Восточной Европе имел место резкий рост Ne примерно 4.3 тыс. лет тому назад (95%-ный доверительный интервал (ДИ): 2.9–5.8 тыс. лет) [1]. Это свидетельствует о том, что экспансия населения Восточной Европы, ставшего основой для русских популяций, началась в эпоху бронзового века. Как известно по данным археологии и палеогеномики, самая значительная экспансия этого времени связана с распространением носителей ямной культуры из понто-каспийской степной зоны в соседние регионы Европы и Азии [6–8].
Полномитогеномное секвенирование позволяет на предельном для мтДНК уровне выявлять полиморфизм и находить маркеры, дающие важную информацию о генетической истории популяций. Предпринятое нами ранее исследование митохондриальных гаплогрупп, относящихся к макрогаплогруппе U, позволило зарегистрировать у русских целый ряд подгрупп мтДНК, которые характеризуются этногеографической специфичностью своего распределения [2]. Некоторые подгруппы мтДНК (примерно 11% митогеномов) обнаружены только среди русских и могут быть, таким образом, определены как подгруппы мтДНК, специфичные для русских. Столько же обнаруженных у русских подгрупп мтДНК (11%) характерны для славян в целом. Общий славяно-германский компонент U-фракции митохондриального генофонда выявлен у русских с частотой 17.4%. Частота же славяно-финского компонента составляет 6.8%. Полученные данные свидетельствуют, таким образом, о том, что, несмотря на соседство различных групп славян как с германскими, так и с финно-угорскими популяциями, интенсивность контактов (о величине которой можно судить по частоте идентичных и аналогичных гаплотипов мтДНК в сравниваемых группах населения) между славянами и германцами существенно выше. Возраст U-подгрупп мтДНК, характерных для славян отдельно или для славян и германцев, составляет от 1 до 5 тыс. лет (для более 70% датировок) [2].
Поскольку описанные выше характеристики митохондриального генофонда русских выявлены, главным образом, при исследовании гаплотипов, относящихся к макрогаплогруппе U, представляется необходимым провести более детальный анализ генетических компонентов русского митохондриального генофонда, основываясь уже на полном наборе гаплогрупп мтДНК. Цель настоящей работы – поиск и молекулярное датирование подгрупп мтДНК, характеризующихся различной этнотерриториальной специфичностью, а также анализ демографической истории (динамики Ne) отдельных компонентов русского генофонда.
МАТЕРИАЛЫ И МЕТОДЫ
В работе анализировали нуклеотидные последовательности целых митохондриальных геномов (n = 557) русского населения европейской части России по данным работ [1, 9–11], а также баз данных GenBank (www.ncbi.nlm.nih.gov), mtDNA Community (www.mtdnacommunity.org) и Logan DNA Project (http://www.ianlogan.co.uk). Проанализированы митохондриальные геномы русского населения Новгородской (n = 92), Псковской (n = 68), Тульской (n = 59), Владимирской (n = 73), Калужской (n = 21), Ярославской (n = 10), Белгородской (n = 64), Орловской (n = 48), Нижегородской (n = 6), Саратовской (n = 1), Магаданской (n = 24) областей, а также с неизвестной областной локализацией (n = 91). Принадлежность большинства митогеномов (n = 466) именно русским по материнской линии устанавливалась на основании анкетирования доноров крови во время экспедиционных работ, проведенных лабораториями генетики ИБПС ДВО РАН (Новгородская, Белгородская, Орловская, Нижегородская, Саратовская, Магаданская области) и генетики человека ИОГен РАН (Псковская, Тульская, Владимирская, Калужская и Ярославская области). Часть русских митогеномов (n = 91) обнаружена в базе данных GenBank; этническая принадлежность этих образцов установлена на основании сведений, предоставленных базой данных.
Филогенетические взаимоотношения между нуклеотидными последовательностями мтДНК реконструировали с помощью метода медианных сетей [12], реализованного в пакете компьютерных программ mtPhyl v4.015 (http://eltsov.org). Полиморфизм длины в участках мтДНК 16 180–16 193, 309–315 и 514–524, а также нуклеотидные замены в позиции 16519 при построении деревьев не учитывали. Генетические дистанции ρ (и их стандартные ошибки) между последовательностями мтДНК рассчитывали как среднее число мутаций между генотипами-основателями и производными типами ДНК, входящими в состав соответствующих филогенетических кластеров ДНК [12]. Для определения эволюционного возраста монофилетических кластеров мтДНК использовали две мутационные скорости (μ), которые определяют одну нуклеотидную замену в целом митогеноме за 3624 лет [13] и одну нуклеотидную замену в кодирующей области мтДНК за 4610 лет [14].
В настоящей работе использована классификация гаплогрупп мтДНК, предложенная разработчиками ресурса PhyloTree (www.phylotree.org). Новые подгруппы мтДНК определяли в тех случаях, когда как минимум два гаплотипа объединялись с помощью как минимум одной нуклеотидной замены, произошедшей не в “горячей” точке мутаций. Монофилетические кластеры мтДНК учитывались нами как этно-специфичные, если не менее 75% гаплотипов мтДНК в кластере были характерны только для представителей определенной этнотерриториальной группы (русских, славян, славян и германцев и т.д.). Следует отметить, что аналогичный критерий для популяционной специфичности (75%) был выбран в исследованиях митохондриального генофонда финнов [15].
Для филогеографического анализа мтДНК использовали информацию об изменчивости целых молекул мтДНК у представителей различных популяций человека, согласно данным GenBank, PhyloTree, mtDNA Community и Logan DNA Project. Славянская выборка (n = 1287) представлена в анализе русскими, белорусами, украинцами, поляками, чехами, словаками, сербами, хорватами, словенцами, боснийцами и болгарами; германская выборка (n = 1331) – немцами, австрийцами, датчанами, шведами, норвежцами, голландцами и англичанами; финно-угорская выборка (n = 684) – финнами, эстонцами, саами, венграми и мордвой; балтская (n = 34) – литовцами и латышами.
Для исследования демографической истории популяций использовали основанный на данных об изменчивости митохондриальных геномов байесовский анализ графиков эффективной численности популяций. Этот подход реализован в пакете программ BEAST 1.7.5 [16]. Наиболее оптимальная модель нуклеотидных замен (HKY + G + I), характеризующаяся наименьшими значениями байесовского информационного критерия (BIC, Bayesian Information Criterion), была выбрана с помощью пакета программ MEGA 5.05 [17]. В анализе использовали строгие молекулярные часы, значение мутационной скорости, соответствующее одной нуклеотидной замене в целом митогеноме за 3624 года [13], и протяженность байесовского анализа, составляющую 100 млн циклов с отбором каждого 10-тысячного из полученных филогенетических деревьев. Для анализа данных, сгенерированных BEAST 1.7.5, применяли пакет программ Tracer 1.4. Марковские цепи MCMC (Markov Сhain Monte Carlo) считали стабилизированными, если значение параметра ESS (Effective Sample Size) для всех статистик было более 200. Изменения эффективной численности популяций оценивали не в абсолютных значениях, а с помощью пропорционального Ne параметра Ne ⋅ μ (произведение эффективной численности популяций и мутационной скорости), вычисляемого непосредственно по данным об изменчивости мтДНК с помощью пакета программ BEAST 1.7.5.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
По результатам филогенетического анализа у русских нами обнаружено 169 гаплогрупп мтДНК. Среди них абсолютное большинство подгрупп входит в состав гаплогрупп, распространенных среди населения Западной Евразии (гаплогруппы HV, H, V, J, T, U2e, U3, U4, U5, K, U8a, R1a, N1a, N1b, W, X). Анализ показал, что 9.7% митохондриальных геномов, обнаруженных у русских, формируют подгруппы мтДНК, специфичные только для русских (табл. 1). Как нами уже отмечалось ранее, понятие такого рода популяционной специфичности является довольно условным, поскольку восточная часть Европы в отношении полногеномной изменчивости мтДНК изучена намного хуже, чем западная. Поэтому нельзя исключить, что по мере увеличения данных по популяциям Восточной Европы часть вариантов мтДНК, характерных (на нынешнем этапе исследований) для русских, перейдет в разряд специфичных для финно-угорских или тюркских популяций Восточной Европы. Это же замечание относится и к вариантам мтДНК, выявленным как у русских, так и у белорусов или украинцев (подгруппы H6a1b3c, U3b1b1, U3b3a, U5a1b1b, U5a2a1c) (табл. 2). Частота таких восточнославянских подгрупп мтДНК у русских очень мала – всего около 2%. Гораздо выше (12.2%) у русских частота вариантов мтДНК, характерных для славян в целом (табл. 2). Этот результат представляется очень важным, поскольку показывает, что довольно большая фракция гаплотипов мтДНК у русских является частью общеславянского генетического пула. Это, в свою очередь, свидетельствует о реальности существования в прошлом славянской общности, в которую входили и предки русских.
Таблица 1.
Молекулярные датировки подгрупп мтДНК, специфичных для русского населения
| Подгруппа мтДНК | Возраст подгрупп мтДНК (в тыс. лет) | Распространенность гаплотипов мтДНК1/2/3/4/5 | |
|---|---|---|---|
| скорость 1 | скорость 2 | ||
| H1cl | 3.9 (–0.5; 8.4) | 2.3 (2.3) | 2/0/0/0/0 |
| H2a5a1c | 1.3 (–1.2; 3.8) | 0 | 2/0/0/0/0 |
| H11a1a | 1.3 (–0.5; 3.1) | 1.2 (1.2) | 3/0/0/1/0 |
| H11a1b1 | 2.6 (–1.0; 6.2) | 4.6 (3.3) | 2/0/0/0/0 |
| H11a2a2b | 1.3 (–1.2; 3.8) | 0 | 2/0/0/0/0 |
| H109 | 2.6 (–0.3; 5.6) | 3.1 (2.2) | 3/0/0/0/0 |
| J1c3f1 | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 2/0/0/0/0 |
| J1c4b1 | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 2/0/0/0/0 |
| T1a1s | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 2/0/0/0/0 |
| T2b4j | 3.9 (–0.5; 8.4) | 3.5 (2) | 3/0/0/0/1 |
| T2b38 | 3.9 (–0.5; 8.4) | 2.3 (2.3) | 2/0/0/0/0 |
| V1a1c | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 2/0/0/0/0 |
| V1a1d | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 2/0/0/0/0 |
| HV9b1 | 3.2 (–1.0; 7.6) | 1.2 (1.2) | 3/0/0/0/1 |
| R1a1a1 | 4.2 (0.3; 8.1) | 5.5 (3.2) | 4/0/0/0/1 |
| R1a1a1a | 1.7 (–0.7; 4.1) | 1.5 (1.5) | 3/0/0/0/0 |
| U2e2a1d1a | 2.6 (–1.0; 6.2) | 2.3 (2.3) | 2/0/0/0/0 |
| U3b2a1b | 3.9 (–0.5; 8.4) | 4.6 (3.3) | 2/0/0/0/0 |
| U5a1a2a2 | 7.0 (1.6; 12.6) | 12.3 (4.9) | 3/0/0/0/0 |
| U5a1b1c2 | 3.2 (–0.1; 6.7) | 3.5 (2.6) | 2/0/0/0/0 |
| U5a1c2b | 2.9 (–1.0; 6.7) | 2.3 (2.3) | 2/0/0/0/0 |
| K1b2a2b | 2.6 (0.3; 4.9) | 2.8 (1.6) | 4/0/0/0/1 |
Примечание (для табл. 1 и 2). Для расчета возраста подгрупп мтДНК использованы скорость 1, основанная на изменчивости всей молекулы мтДНК [13], и скорость 2, основанная на изменчивости только кодирующей области мтДНК [14]. 1/2/3/4/5 – число гаплотипов, обнаруженных у русских (1), других славян (2), германцев (3), прибалтийских финнов (4) и в других популяциях Европы, включая образцы с неизвестной этнической принадлежностью (5).
Таблица 2.
Молекулярные датировки и распространенность подгрупп мтДНК, выявленных у русских, в различных группах населения Европы
| Подгруппа мтДНК | Возраст подгрупп мтДНК (в тыс. лет) | Распространенность гаплотипов мтДНК1/2/3/4/5 | |
|---|---|---|---|
| скорость 1 | скорость 2 | ||
| Подгруппы мтДНК, характерные для славян | |||
| H1c23a | 2.1 (0.04; 4.1) | 0.9 (0.9) | 1/3/0/0/1 |
| H1ck | 2.6 (–1.2; 6.4) | 4.6 (3.4) | 3/2/0/0/0 |
| H1b2b | 3.5 (–0.7; 7.7) | 3.1 (2.2) | 1/2/0/0/0 |
| H2a5b1 | 2.6 (0.2; 5.0) | 4.6 (3.4) | 1/4/0/0/1 |
| H5a1a | 3.4 (1.8; 5.1) | 4.2 (1.2) | 7/9/1/1/3 |
| H5e1a1 | 1.9 (0.2; 3.8) | 1.7 (1.1) | 4/4/0/0/0 |
| H6a1b3c | 2.6 (–1.0; 6.2) | 2.3 (2.3) | 1/1/0/0/0 |
| H13a1a1c1 | 1.7 (–0.7; 4.1) | 3.1 (2.2) | 2/1/0/0/0 |
| H13a1a3 | 3.5 (0.1; 6.9) | 6.1 (3.1) | 2/1/0/0/0 |
| H24a3 | 2.6 (–0.3; 5.6) | 4.6 (2.7) | 2/1/0/0/1 |
| H41a1 | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 1/1/0/0/0 |
| H55b | 6.7 (1.7; 11.8) | 4.2 (2.0) | 1/7/1/0/3 |
| H56a1 | 2.9 (0.1; 3.9) | 4.6 (1.1) | 1/1/0/0/0 |
| J1c4d | 1.7 (–0.7; 4.1) | 1.5 (1.5) | 2/1/0/0/0 |
| J1c2f | 1.6 (–0.2; 3.3) | 1.8 (1.3) | 2/2/1/0/0 |
| J1c2u | 1.7 (0.03; 3.4) | 2.3 (1.3) | 3/2/0/0/1 |
| T2a1a1a | 2.6 (–1.0; 6.2) | 2.3 (2.3) | 1/1/0/0/0 |
| N1b1a2b | 5.2 (0.1; 10.5) | 6.9 (4.0) | 1/1/0/0/0 |
| HV6a | 4.3 (0.5; 8.2) | 3.1 (2.2) | 2/1/0/0/0 |
| U3b1b | 2.6 (0.5; 4.7) | 2.8 (1.7) | 3/5/0/1/1 |
| U3b1b1 | 1.0 (–0.4; 2.5) | 0.9 (0.9) | 1/3/0/0/1 |
| U3b3a | 4.3 (0.5; 8.2) | 4.6 (2.7) | 2/1/0/0/0 |
| U4a2a3 | 5.2 (0.1; 10.5) | 9.2 (4.6) | 2/1/0/0/0 |
| U4a2a4 | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 1/1/0/0/0 |
| U4a2c | 12.8 (4.7; 21.3) | 12.9 (5.1) | 1/3/0/0/1 |
| U4a2g1 | 1.9 (–0.3; 4.2) | 2.3 (1.6) | 2/1/0/1/0 |
| U4d2b | 4.3 (–0.3; 9.1) | 1.5 (1.1) | 2/3/0/0/0 |
| U5a1b1b | 5.6 (1.6; 9.6) | 3.5 (1.4) | 5/1/2/0/0 |
| U5a1c1 | 4.8 (1.5; 8.2) | 2.7 (1.0) | 2/4/0/1/1 |
| U5a2a1c | 2.6 (–1.2; 6.4) | 1.5 (1.5) | 2/1/0/0/0 |
| U5a2b1a1 | 0.9 (–0.8; 2.6) | 0 (0) | 1/2/0/0/0 |
| U5a2b1c1 | 3.5 (0.1; 6.9) | 3.1 (2.2) | 1/2/0/0/0 |
| U5a2b1e | 3.9 (0.3; 7.6) | 3.5 (2.6) | 2/2/0/0/0 |
| U5b1a1 | 1.0 (–0.4; 2.5) | 1.8 (1.3) | 1/4/0/0/0 |
| U5b1b1f | 4.6 (0.3; 8.9) | 3.5 (2.6) | 1/2/1/0/0 |
| U5b1e1a | 1.3 (–0.2; 2.8) | 0.8 (0.8) | 1/5/0/0/1 |
| V1a1b1 | 2.6 (–1.2; 6.4) | 1.5 (1.5) | 1/1/0/0/0 |
| V3d | 1.3 (–0.5; 3.1) | 1.2 (1.2) | 3/1/0/0/0 |
| V7a* | 5.4 (2.2; 8.7) | 6.5 (2.8) | 5/4/1/0/2 |
| V7a2 | 6.6 (0.9; 12.4) | 10.0 (4.9) | 2/3/0/0/1 |
| Славяно-германские подгруппы мтДНК | |||
| H1b5 | 4.1 (0.03; 8.2) | 2.6 (1.3) | 2/2/2/1/0 |
| H1a3c1 | 4.6 (1.7; 7.6) | 7.2 (2.4) | 1/1/7/0/0 |
| H2a1f1 | 1.3 (–0.5; 3.1) | 1.2 (1.2) | 1/0/3/0/0 |
| H5a1q | 5.2 (1.0; 9.5) | 3.1 (2.2) | 1/1/1/0/1 |
| H5u1 | 1.1 (–0.1; 2.4) | 0.7 (0.7) | 4/0/3/0/0 |
| H6a1b4 | 4.0 (2.0; 6.0) | 3.5 (1.2) | 1/1/11/0/4 |
| H27g | 4.3 (–0.8; 9.6) | 3.8 (2.5) | 1/1/4/0/0 |
| H79b | 3.1 (0.6; 5.6) | 2.8 (1.6) | 2/1/2/0/0 |
| J1c2o | 3.6 (0.9; 6.4) | 4.6 (2.1) | 1/0/3/0/1 |
| V1a1e | 2.6 (–0.8; 6.0) | 0.9 (0.9) | 1/0/3/0/1 |
| V15a | 3.2 (1.2; 5.3) | 2.9 (1.3) | 1/0/5/0/2 |
| HV7 | 9.3 (4.0; 14.7) | 9.2 (3.3) | 2/0/2/0/0 |
| HV9b | 5.8 (0.2; 11.5) | 5.5 (3.9) | 3/1/2/0/0 |
| HV9a1a | 7.0 (0.6; 13.7) | 6.1 (3.8) | 1/0/2/0/0 |
| U2e1f1a | 3.5 (–0.7; 7.7) | 6.2 (3.8) | 1/0/1/0/0 |
| U4a1a1a | 1.0 (–0.4; 2.5) | 0.9 (0.9) | 1/3/2/0/1 |
| U4a1a3 | 3.5 (–0.7; 7.7) | 6.2 (3.8) | 1/0/1/0/0 |
| U5a1b1c | 7.7 (3.6; 11.8) | 9.2 (3.3) | 4/4/2/2/1 |
| U5a1b1c1 | 5.2 (2.3; 8.2) | 6.3 (2.1) | 1/4/1/2/0 |
| U5a1d2a1 | 2.9 (0.8; 5.1) | 2.3 (1.2) | 1/4/2/1/0 |
| U5a1f2 | 7.4 (1.9; 13.2) | 6.9 (4.1) | 2/2/1/0/1 |
| U5a2b1b | 3.1 (–0.4; 6.7) | 0.9 (0.9) | 2/1/2/0/0 |
| U5a2c4 | 2.6 (0.05; 5.2) | 3.5 (2.0) | 1/0/5/0/1 |
| K1d1 | 3.5 (0; 8.4) | 6.1 (4.3) | 1/0/6/0/1 |
| Славяно-финские подгруппы мтДНК | |||
| H1a2 | 1.7 (0.7; 2.7) | 1.3 (0.5) | 5/0/1/16/2 |
| H1a7 | 2.3 (0.6; 4.0) | 2.9 (1.3) | 1/1/1/5/0 |
| H1a8 | 3.2 (0.7; 5.8) | 5.2 (2.2) | 1/0/1/4/2 |
| H1n4 | 1.9 (0.8; 3.0) | 1.8 (0.8) | 2/0/0/11/2 |
| H3h1 | 1.8 (0.2; 3.4) | 2.7 (1.4) | 3/0/0/14/2 |
| H3h5a | 3.9 (–0.5; 8.4) | 4.6 (3.3) | 1/0/0/1/0 |
| H7b7 | 4.6 (0.7; 8.5) | 2.3 (1.6) | 1/0/0/2/1 |
| HV25a | 2.6 (–1.0; 6.2) | 0 (0) | 1/0/0/1/0 |
| I1a1a1 | 2.1 (–1.1; 5.3) | 0 (0) | 1/0/0/4/0 |
| I1a1a4 | 2.6 (–1.0; 6.2) | 2.3 (2.3) | 1/0/0/1/0 |
| W6a1 | 3.1 (1.2; 5.0) | 2.3 (1.0) | 2/4/0/3/1 |
| T2f1b1 | 1.3 (–1.2; 3.8) | 2.3 (2.3) | 1/0/0/1/0 |
| T2l | 4.3 (0.5; 8.2) | 6.1 (3.1) | 1/0/0/1/1 |
| K1c1c | 1.3 (–0.2; 2.8) | 1.5 (1.1) | 1/0/0/4/1 |
| U4d1a1 | 5.0 (0; 10.1) | 6.0 (4.3) | 1/0/0/5/3 |
| U4d1b | 1.3 (–0.3; 2.9) | 2.3 (1.4) | 2/2/0/3/1 |
| U5b1b1a1a | 2.6 (–0.2; 5.5) | 0.6 (0.6) | 2/0/0/5/1 |
| U8a1a1b1 | 3.7 (1.7; 5.7) | 4.2 (1.4) | 4/0/1/5/2 |
| U8a1a5 | 4.4 (–0.8; 9.6) | 7.7 (4.6) | 1/0/0/2/0 |
Частота генетического компонента, представленного гаплотипами мтДНК, присутствующими как у русских и других славян, так и в германоязычных популяциях, составляет у русских 6.6% (табл. 2). Немного ниже у русских (5.7%) частота гаплотипов, характерных для прибалтийских финнов (финнов и эстонцев) (табл. 2). Доля вариантов мтДНК, имеющих кавказское или западноазиатское происхождение, составляет у русских всего 0.9% (это гаплотипы, относящиеся к подгруппам HV4b, R1a2, T1a2c, U1a1c1d3 и U3b2d). Между тем, частота гаплотипов мтДНК, имеющих неясное происхождение, достигает 60%. К этой категории относятся гаплотипы, обнаруженные в составе подгрупп мтДНК, распространенных в различных популяциях Европы, в связи с чем на данном уровне исследований их можно определить как общеевропейские. Однако по мере расширения выборок митогеномов из различных частей Европы вполне возможно, что некоторые из гаплотипов станут популяционно-специфичными. Вместе с тем, учитывая низкий уровень межэтнической дифференциации в Европе по распределению гаплогрупп мтДНК, можно ожидать, что существенная часть разнообразия мтДНК так и останется недифференцированной в отношении этнотерриториальной специфичности.
Результаты молекулярного датирования показывают, что возраст подгрупп мтДНК, к которым принадлежат гаплотипы, выявленные только у русских, изменяется в широком диапазоне – от 1.3 до 7 тыс. лет (для мутационной скорости, основанной на изменчивости целых митохондриальных геномов) (табл. 1). В среднем эволюционный возраст этого генетического компонента составляет 2.7 ± 0.2 тыс. лет. Подгруппы мтДНК, характерные для славян, имеют возраст от 0.6 до 12.8 тыс. лет (в среднем 3.5 ± 0.3 тыс. лет) (табл. 2). Возраст славяно-германских подгрупп мтДНК варьирует в интервале от 1 до 9.3 тыс. лет (средний возраст 4.2 ± 0.4 тыс. лет) (табл. 2). Возраст славяно-финских подгрупп мтДНК изменяется в диапазоне от 1.3 до 5 тыс. лет и составляет в среднем 2.8 ± 0.2 тыс. лет (табл. 2). Таким образом, молекулярные датировки возраста славянских и славяно-германских подгрупп мтДНК соответствуют эпохе бронзового века. Согласно данным археологии и палеогеномики, в это время (примерно 4.5 тыс. лет назад) имели место масштабные миграции носителей курганной культуры из степей Восточной Европы в Центральную Европу [6–8].
Об экспансии населения Восточной Европы в эпоху бронзового века свидетельствуют и результаты проведенного нами ранее исследования динамики эффективной численности популяций в русских популяциях [1]. На основании данных о полногеномной изменчивости мтДНК, у русских был обнаружен резкий рост Ne примерно 4.3 тыс. лет назад (95% ДИ: 2.9–5.8 тыс. лет). В настоящей работе нами исследована динамика Ne на основании анализа подгрупп мтДНК, специфичных для русских и славян. Оказалось, что в обоих случаях наблюдается резкий рост величины Ne: 4-кратный рост, начавшийся примерно 4.4 тыс. лет назад (95% ДИ: 3.8–5.0 тыс. лет) для подгрупп мтДНК, специфичных для русских, и 15-кратный рост, начавшийся примерно 5.1 тыс. лет назад (95% ДИ: 4.5–5.7 тыс. лет) для славяно-специфичных подгрупп мтДНК (рис. 1). Интересно, что байесовские графики эффективной численности, полученные для популяционно-специфичных фракций митохондриального генофонда русских, отличаются от графика, полученного в результате анализа всего набора митохондриальных геномов у русских, одной важной деталью – рост численности для подгрупп мтДНК, специфичных для русских и славян, происходит после резкого (примерно 4-кратного) снижения численности, в то время как при анализе всей совокупности митохондриальных геномов выявляется момент резкого роста численности примерно 4.3 тыс. лет назад на фоне постепенного повышения Ne, начиная с послеледникового времени (рис. 1). Эти новые данные указывают на возможность того, что росту численности населения Восточной Европы в бронзовом веке предшествовал период снижения численности, начавшийся примерно 12 тыс. лет назад и достигший максимума 4–5 тыс. лет назад.
Рис. 1.
Байесовские графики динамики эффективной численности популяций во времени, построенные по данным об изменчивости целых митохондриальных геномов. 1 – значения медианы и 95%-ного доверительного интервала с наивысшей апостериорной вероятностью (сплошные линии) для данных, полученных при анализе всего набора митохондриальных геномов русского населения. График ранее был опубликован в работе [1]; 2 – значения медианы и 95%-ного доверительного интервала с наивысшей апостериорной вероятностью (пунктирные линии) для данных об изменчивости целых митохондриальных геномов, относящихся к подгруппам мтДНК, специфичным для славян. График показывает изменение параметра Ne ⋅ μ (ось Y) во времени (ось X, в тыс. лет).
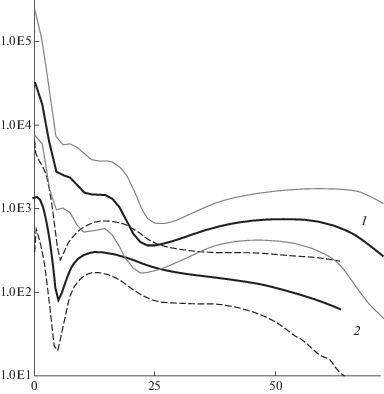
Следует отметить, что аналогичный результат в отношении динамики Ne получен и при исследовании разнообразия мтДНК у финнов [15]. Так, для фракции подгрупп мтДНК, специфичных для финнов, “бутылочное горлышко” и последующий резкий рост численности обнаружены примерно 1 тыс. лет назад, а до этого времени происходило постепенное снижение Ne, начиная с отметки в 4–5 тыс. лет назад. Вместе с тем снижение эффективной численности не наблюдалось для групп митогеномов, не относящихся к финно-специфичному компоненту. Авторы этого исследования отметили, что выявленный при исследовании финно-специфичных подгрупп мтДНК эпизод редукции популяционного размера, предшествующий периоду резкого увеличения Ne, может быть искусственным и тем самым не свидетельствовать о реально произошедшем в истории финнов периоде снижения эффективной численности, поскольку при использовании байесовского анализа динамики Ne имеются некоторые ограничения. Так, к видимым на байесовских графиках эпизодах редукции Ne может приводить наличие субструктуры популяций или отклонения при формировании выборок из популяций [18–20]. В случае анализа популяционно-специфичных подгрупп мтДНК момент редукции численности также может быть искусственным, поскольку анализируется не вся совокупность митогеномов, представляющих различные структурные компоненты генофонда, а только лишь определенная его часть, диверсификация которой началась с некоторого момента времени – как в случае подгрупп мтДНК, характерных для русских и для славян в целом. Этот вопрос требует дальнейших исследований с помощью компьютерного моделирования.
Полученные данные дают новую информацию относительно демографической истории европейцев. Так, в ряде работ отмечалось, что динамика Ne в популяциях Европы, реконструируемая по данным об изменчивости целых митохондриальных геномов и протяженных участков Y-хромосомы, существенно различается [21–23]. Для Y-хромосомы отмечался резкий рост эффективной численности, следующий после “бутылочного горлышка” 2–4 тыс. лет назад, в то время как для мтДНК в указанных выше исследованиях наблюдался только постепенный рост численности, начиная с послеледникового времени. Batini et al. [23] сделали, таким образом, вывод о том, что миграции населения с востока Европы на запад в эпоху бронзового века нашли свое отражение только в Y-хромосоме. Между тем одной из причин несоответствия демографической истории, выявляемой по мужским и женским генетическим линиям, могут быть различия в Ne, поскольку Karmin et al. [22] обнаружили, что в период 4–8 тыс. лет тому назад Nef (для женщин) превышал Nem (для мужчин) в 17 раз. Соответственно, для проведения сравнительного анализа европейских популяций размеры выборок митохондриальных геномов должны существенно превышать таковые для Y-хромосом. Однако в исследованиях Batini et al. [23, 24] размеры популяционных выборок для мтДНК и Y-хромосомы были примерно равными и маленькими (не более 20 человек на выборку). Между тем байесовский анализ Ne больших выборок мтДНК (более 250 человек на выборку в исследованиях [1, 15]) позволил, подобно исследованиям Y-хромосомы, выявить рост эффективной численности населения Восточной Европы в эпоху бронзового века, а при анализе этноспецифичных фракций митохондриальных генофондов ([15] и настоящая работа) позволил обнаружить и редукцию Ne, предшествовавшую периоду резкого увеличения популяционного размера. Таким образом, по всей видимости, более масштабные исследования разнообразия мтДНК в популяциях Европы позволят в перспективе более корректно описать и согласовать генетическую историю, реконструируемую по данным об изменчивости мтДНК и Y-хромосомы.
Проведенный в настоящей работе анализ полиморфизма мтДНК в русских популяциях показал, что митохондриальные генофонды как многокомпонентные системы хранят в себе достаточно много информации о генетической истории. Анализ митохондриального генофонда русских позволил выявить генетические компоненты, характерные для русских и славян в целом, а также общие славяно-германские и славяно-финские компоненты, сформировавшиеся, главным образом, в бронзовом и железном веках.
Работа выполнена при финансовой поддержке РФФИ (грант № 14-04-00131).
Список литературы
Malyarchuk B., Litvinov A., Derenko M. et al. Mitogenomic diversity in Russians and Poles // Forensic Sci. Intern.: Genetics. 2017. V. 30. P. 51–56. doi https://doi.org/10.1016/ j.fsigen.2017.06.003
Малярчук Б.А., Деренко М.В., Литвинов А.Н. Структура макрогаплогруппы U у русских // Генетика. 2017. Т. 53. № 4. С. 488–494. doi https://doi.org/10.7868/S0016675817020059
Malyarchuk B., Derenko M., Grzybowski T. et al. Differentiation of mitochondrial DNA and Y chromosomes in Russian populations // Hum. Biol. 2004. V. 76. P. 877–900.
Morozova I., Evsyukov A., Kon’kov A. et al. Russian ethnic history inferred from mitochondrial DNA diversity // Am. J. Phys. Anthropol. 2012. V. 147. P. 341–351. doi https://doi.org/10.1002/ajpa.21649
Kushniarevich A., Utevska O., Chuhryaeva M. et al. Genetic heritage of the Balto-Slavic speaking populations: a synthesis of autosomal, mitochondrial and Y-chromosomal data // PLoS One. 2015. V. 10. e0135820. doi https://doi.org/10.1371/journal.pone.0135820
Gimbutas M. The prehistory of Eastern Europe. Part I: Mesolithic, Neolithic and Copper Age cultures in Russia and the Baltic area. Cambridge: The Peabody Museum, 1956. 241 p.
Allentoft M.E., Sikora M., Sjogren K.G. et al. Population genomics of Bronze Age Eurasia // Nature. 2015. V. 522. P. 167–172. doi https://doi.org/10.1038/nature14507
Haak W., Lazarids I., Patterson N. et al. Massive migration from the steppe was a source for Indo-European languages in Europe // Nature. 2015. V. 522. P. 207–211. doi https://doi.org/10.1038/nature14317
Malyarchuk B.A., Grzybowski T., Derenko M. et al. Mitochondrial DNA phylogeny in Eastern and Western Slavs // Mol. Biol. Evol. 2008. V. 25. № 8. P. 1651–1658. doi https://doi.org/10.1093/molbev/msn114
Malyarchuk B., Derenko M., Grzybowski T. et al. The peopling of Europe from the mitochondrial haplogroup U5 perspective // PLoS One. 2010. V. 5. e10285. doi https://doi.org/10.1371/journal.pone.0010285
Derenko M., Malyarchuk B., Denisova G. et al. Western Eurasian ancestry in modern Siberians based on mitogenomic data // BMC Evol. Biol. 2014. V. 14. P. 217. doi https://doi.org/10.1186/s12862-014-0217-9
Bandelt H.J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. P. 37–48. doi https://doi.org/10.1093/oxfordjournals.molbev.a026036
Soares P., Ermini L., Thomson N. et al. Correcting for purifying selection: an improved human mitochondrial molecular clock // Am. J. Hum. Genet. 2009. V. 84. P. 740–759. doi https://doi.org/10.1016/j.ajhg.2009.05.001
Perego U.A., Achilli A., Angerhofer N. et al. Distinctive Paleo-Indian migration routes from Beringia marked by two rare mtDNA haplogroups // Curr. Biol. 2009. V. 19. P. 1–8. doi https://doi.org/10.1016/j.cub.2008.11.058
Översti S., Onkamo P., Stoljarova M. et al. Identification and analysis of mtDNA genomes attributed to Finns reveal long-stagnant demographic trends obscured in the total diversity // Sci. Rep. 2017. V. 7. 6193. doi https://doi.org/10.1038/s41598-017-05673-7
Drummond A.J., Suchard M.A., Xie D. et al. Bayesian phylogenetics with BEAUti and the BEAST 1.7 // Mol. Biol. Evol. 2012. V. 29. P. 1969–1973. doi https://doi.org/10.1093/molbev/mss075
Tamura K., Peterson D., Peterson N. et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods // Mol. Biol. Evol. 2011. V. 28. P. 2731–2739. doi https://doi.org/10.1093/molbev/msr121
Peter B.M., Wegmann D., Excoffier L. Distinguishing between population bottleneck and population subdivision by a Bayesian model choice procedure // Mol. Ecol. 2010. V. 19. P. 4648–4860. doi https://doi.org/10.1111/j.1365-294X.2010.04783.x
Heller R., Chikhi L., Siegismund H.R. The confounding effect of population structure on Bayesian skyline plot inferences of demographic history // PLoS One. 2013. V. 8. e62992. doi https://doi.org/10.1371/journal.pone.0062992
Mazet O., Rodríguez W., Grusea S. et al. On the importance of being structured: instantaneous coalescence rates and human evolution – lessons for ancestral population size inference? // Heredity (Edinb). 2016. V. 116. P. 362–371. doi https://doi.org/10.1038/hdy.2015.104
Lippold S., Xu H., Ko A. et al. Human paternal and maternal demographic histories: insights from high-resolution Y chromosome and mtDNA sequences // Investig. Genet. 2014. V. 5. 13. doi https://doi.org/10.1186/2041-2223-5-13
Karmin M., Saag L., Vicente M. et al. A recent bottleneck of Y chromosome diversity coincides with a global change in culture // Genome Res. 2015. V. 25. P. 459–466. doi https://doi.org/10.1101/gr.186684.114
Batini C., Hallast P., Vågene Å.J. et al. Population resequencing of European mitochondrial genomes highlights sex-bias in Bronze Age demographic expansions // Sci. Rep. 2017. V. 7. 12086. doi https://doi.org/10.1038/s41598-017-11307-9
Batini C., Hallast P., Zadik D. et al. Large-scale recent expansion of European patrilineages shown by population resequencing // Nat. Commun. 2015. V. 6. 7152. doi https://doi.org/10.1038/ncomms8152
Дополнительные материалы отсутствуют.


