Генетика, 2019, T. 55, № 5, стр. 557-573
Идентификация и филогения криптических видов комплекса Daphnia longispina (Cladocera, Daphniidae) на основе вторичной структуры промежуточного транскрибируемого спейсера 2 (ITS2) ядерной ДНК
Е. И. Зуйкова *
Институт систематики и экологии животных Сибирского отделения
Российской академии наук
630091 Новосибирск, Россия
* E-mail: zuykova@ngs.ru
Поступила в редакцию 26.06.2018
После доработки 07.08.2018
Принята к публикации 22.08.2018
Аннотация
Ветвистоусые ракообразные рода Daphnia O.F. Müller (Crustacea: Cladocera) являются типичными представителями разнотипных водоемов и используются как модельные объекты в различных областях биологии. В пределах рода отмечено значительное число близкородственных и криптических видов, идентификация которых по морфологическим признакам часто вызывает затруднения. В настоящей работе проведена идентификация видов группы Daphnia longispina – D. longispina O.F. Müller, D. hyalina Leydig, D. umbra Taylor, Hebert et Colbourne и D. cf. longispina на основе данных о предполагаемой вторичной структуре второго внутреннего транскрибируемого спейсера ITS2 ядерной ДНК. Филогенетический анализ с использованием алгоритма Profile Neighbor-Joining и анализ компенсаторных замен подтвердили видовую самостоятельность D. hyalina и D. cf. longispina. Показано, что данные о вторичной структуре ITS2 могут применяться для решения сложных таксономических задач при изучении кладоцер в целом и видовых комплексов в частности.
Криптические виды – широко распространенный феномен в пределах различных групп растений и животных. Изучение скрытого видового разнообразия и выявление границ между видами представляет собой одну из задач эволюционной биологии, которая особо актуальна в свете проблемы реальной оценки и сохранения современного биоразнообразия [1–3]. Криптические виды встречаются и среди водных беспозвоночных, в том числе низших ракообразных. Род Daphnia O.F. Müller (Crustacea: Cladocera) – один из модельных объектов в эволюционной биологии – не является исключением. Например, значительная часть таксонов из группы D. longispina s. lat. представлена криптическими видами [4–6]. Между ними к настоящему времени не выявлено морфологических различий, и систематика этой группы остается весьма проблематичной [4, 5, 7]. В ряде случаев единственно возможным методом идентификации форм/видов группы D. longispina является молекулярно-генетический анализ исследуемых особей [6, 8–12]. При этом известно и то, что некоторые криптические виды образуют гибриды, различение которых возможно только с помощью генетических методов [13].
Для установления филогенетических отношений и выявления случаев гибридизации в пределах рода Daphnia часто используют первый и второй внутренние транскрибируемые спейсеры (ITS1 и ITS2) ядерной ДНК [14, 15]. Однако при этом нередко обнаруживается несовпадение результатов анализа изменчивости митохондриальной и ядерной ДНК. Такая несогласованность чаще всего объясняется ядерной или митохондриальной интрогрессией, неполным сортингом (ISL) и т.п. [13, 16, 17]. Второй внутренний транскрибируемый спейсер ITS2, по сравнению с фланкирующими генами 5.8S и 28S рРНК, представляет собой быстро эволюционирующий регион ядерной ДНК и поэтому является подходящим маркером для реконструкции филогений на видовом и родовом уровнях [18–20].
Вторичная структура ITS2 универсальна для всех групп эукариот и обычно представлена четырьмя спиралями, хотя их число может варьировать. Несмотря на то что некоторые признаки последовательностей ITS2 высоковариабельны (в том числе и их длина), их вторичная структура остается консервативной за счет мотивов, формирующих гомологичные спирали [18, 19, 21]. Эта особенность была успешно использована для установления филогенетических отношений между близкородственными видами; а корреляция между наличием компенсаторных замен (CBC) в консервативных участках ITS2 и способностью к межвидовой гибридизации (в рамках биологической концепции вида) – надежный критерий для выявления криптических видов [18–22]. Первоначально этот метод применялся для распознавания криптических видов водорослей и грибов. Позднее он был успешно апробирован для реконструкции филогений и идентификации криптических видов в разных группах артропод [23–28].
Цель данного исследования – выявление границ между криптическими и близкородственными видами группы D. longispina и установление филогенетических отношений между этими видами с использованием данных о вторичной структуре второго внутреннего транскрибируемого спейсера ядерной ДНК (ITS2 яДНК).
МАТЕРИАЛЫ И МЕТОДЫ
Сбор проб и виды. Образцы видов группы D. longispina (D. longispina O.F. Müller, D. hyalina Leydig, D. cf. longispina и D. umbra Taylor, Hebert et Colbourne) были собраны в течение вегетационных сезонов 2003–2013 гг. в оз. Глубоком (Московская обл.), бассейне оз. Чаны (Новосибирская обл.), Телецком озере (Республика Алтай), в водоемах Тоджинской котловины (бассейн р. Большой Енисей, Республика Тыва) – озера Шурам-Холь, Борзу-Холь, постоянные безымянные пруды. Для последующего генетического анализа образцы фиксировали 96%‑ным этанолом.
Выделение ДНК и амплификация. Общую геномную ДНК экстрагировали из отдельно взятых особей с помощью 5%-ного раствора Chelex 100 resin (BioRad, США). Для амплификации фрагмента ядерной ДНК, включающего в себя часть гена 5.8S рДНК, второй промежуточный транскрибируемый спейсер ITS2 и часть гена 28S рДНК, использовали ПЦР. Амплификацию проводили в программируемом термостате М111 (ООО БИС-Н, Россия). Состав реакционной смеси, праймеры и температурные профили, использованные в процессе амплификации фрагмента ядерного спейсера, были подробно описаны ранее [29, 30]. Продукты реакции амплификации разделяли методом электрофореза в 1%-ном агарозном геле в 1× ТАЕ-буфере, окрашенном бромистым этидием, и фотодокументировали. Полученные ПЦР-продукты очищали с помощью набора реактивов “Биосилика” (Россия) и секвенировали по прямому и обратному праймерам с помощью автоматического анализатора ABI 3130xl (Applied Biosystems, США) в ЦКП “Геномика” СО РАН (http://sequest.niboch.nsc.ru) с использованием BigDye-терминатора (Applied Biosystems).
В анализе использованы 43 нуклеотидные последовательности ядерного маркера ITS2 для пяти близкородственных видов рода Daphnia. Многокопийные последовательности ITS2 в анализ не включали. Нуклеотидные последовательности D. galeata из оз. Шурам-Холь и оз. Чаны использовались в качестве внешней группы. В анализ также была добавлена последовательность ITS2 для D. umbra из базы данных GenBank (#AY730403, Канада) [16].
Анализ и прогнозирование вторичной структуры ITS2. Для удаления фланкирующих участков генов 5.8S и 28S в ядерном маркере полная последовательность ITS2 была аннотирована с помощью инструмента “ITS2-Annotation” в ITS2 Database при следующих параметрах: E < 0.01, скрытой Марковской модели (hidden Markov model, HHM) для Metazoa [31]. Эта процедура необходима для установления гибридизации между 5.8S и 28S генами [32]. Предполагаемые модели вторичной структуры ITS2, характеризующиеся минимальной свободной энергией (MFE), получены с помощью RNAfold WebServer (http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi; настройки по умолчанию при 37°С). Поиск тандемных повторов в последовательностях ITS2 выполнен с помощью ресурса Microsatellite Repeat Finder с настройками по умолчанию (http://sgdp.iop.kcl.ac.uk/ nikammar/repeatfinder.html).
Визуализация вторичных структур ITS2 выполнена с помощью PseudoViewer v. 3 Web Application (http://pseudoviewer.inha.ac.kr/). Оригинальные нуклеотидные последовательности 5.8S–ITS2–28S согласно выявленным гаплотипам депонированы в международную базу данных GenBank под номерами MH399707–MH399728.
Анализ компенсаторных замен. Выравнивание нуклеотидных последовательностей ITS2 с гомологичными вторичными структурами выполнено с помощью встроенного в программу 4SALE v. 1.7 алгоритма ClustalW [33, 34]. Матрицу компенсаторных замен (CBC, нескорректированные p-дистанции) также получали с помощью 4SALE v. 1.7 и анализировали в программе CBCAnalyzer v. 1.0.3 [35]. Результаты выравнивания с информацией о вторичной структуре ITS2 были сохранены для последующего филогенетического анализа [20].
Филогения. Филогенетический анализ выполнен в программе ProfDistS v. 0.9.8 с использованием алгоритма “RNA/DNA structure Profile Neighbor-Joining” при числе репликаций 1000 для модели General Time Reversible и матрице Q = Q_ITS2.txt как специфической модели замен для ITS2 [36, 37]. Результирующее дерево визуализировано с помощью программ NJplot v. 2.4 [38] и FigTree v. 1.4.3 (http://tree.bio.ed.ac.uk). В качестве альтернативного подхода полученная в программе ProfDistS матрица длины ветвей между группами видов использована для реконструкции сети с помощью алгоритма “equal angle” в программе SplitsTree4 [39]. Для сравнения результатов дополнительные филогенетические деревья реконструированы без учета вторичной структуры ITS2 на основе модели GTR в программах MEGA v. 7 [40] и MRBAYES v. 3.2 [41, 42]. В программе MEGA v. 7 дерево реконструировано с помощью метода максимального правдоподобия (ML). Байесовский анализ реализован при числе генераций равном 1000000 и частоте записи параметров 500. Оценка эффективного размера выборки параметров (ESS > 500) для проверки конвергенции Марковских цепей выполнена при помощи Tracer v. 1.6 [43]. Генетические p-дистанции в пределах видов и между ними, доля GC-нуклеотидов рассчитаны в программе MEGA v. 7. Полиморфизм изучаемых последовательностей оценивался с помощью программы DnaSP v. 6 [44].
РЕЗУЛЬТАТЫ
Анализ ITS2 последовательностей
Длина нуклеотидных последовательностей сильно различалась как в пределах форм/видов дафний, так и между ними. Общая длина полученных последовательностей составила 1006–1038 пар нуклеотидов (далее – пн) при длине аннотированного фрагмента ITS2 624–693 пн. Участки фланкирующих генов 5.8S и 28S длиной по 25 пн каждый были высококонсервативными, и все последовательности соответствовали критерию гибридизации [32]. Такое взаимодействие между ними свидетельствует о способности образовывать аннотированный участок ITS2 на основе скрытой Марковской модели (HMM) и о том, что ITS2-последовательности не являются псевдогенами [24].
Последовательности ITS2 с самой вариабельной длиной отмечены у D. longispina, а самая длинная – у D. hyalina (табл. 1). Длина последовательностей ITS2 зависела от числа тандемных повторов в мотивах: GAGAGA, CGCGCG, UUUUUU, UCUCUC, GUGUGU и UAUAUA (Приложение). Доля GC-нуклеотидов варьировала в пределах 48.9–51.1%, и последовательности ITS2 для D. umbra характеризовались более высоким их содержанием (табл. 1).
Таблица 1.
Полиморфизм фрагмента ITS2 яДНК видов группы D. longispina
| Вид | Водоем | Длина оперона, пн | GC, % | n | h | S | k | Hd | π |
|---|---|---|---|---|---|---|---|---|---|
| D. longispina | Бассейн оз. Чаны, Телецкое озеро, озера Тоджинской котловины | 624–687 | 48.9–49.8 | 24 | 12 | 63 | 6.043 | 0.717 ± 0.103 | 0.0099 ± 0.0277 |
| D. hyalina | Оз. Глубокое | 691–693 | 49.5–49.8 | 5 | 3 | 3 | 1.200 | 0.700 ± 0.218 | 0.0017 ± 0.0007 |
| D. umbra | Оз. Борзу-Холь | 675–681 | 50.3–51.1 | 8 | 2 | 4 | 1 | 0.250 ± 0.180 | 0.0015 ± 0.0011 |
| D. cf. longispina | Бассейн оз. Чаны | 665–666 | 49.6–49.8 | 4 | 4 | 10 | 5.667 | 1.000 ± 0.177 | 0.0085 ± 0.0082 |
Высокое число ITS2-гаплотипов зарегистрировано для вида D. longispina, который был представлен бóльшим числом популяций из разных районов Сибири (табл. 1). Этот вид также характеризуется высокими значениями гаплотипического и нуклеотидного разнообразия. Высокие значения этих показателей зарегистрированы для всех видов, кроме D. umbra, а для D. hyalina отмечен высокий уровень гаплотипического разнообразия при низком нуклеотидном разнообразии.
Предполагаемые модели вторичной структуры ITS2
Анализ вторичных структур последовательностей ITS2 выявил несколько типов предполагаемых моделей, соответствующих разным формам/видам дафний группы D. longispina (рис. 1–3). Для образцов вида D. longispina выявлено три типа моделей вторичной структуры ITS2, и их изменчивость проявлялась в числе и длине отдельных спиралей (рис. 1,а–д). Тип вторичной структуры ITS2 для D. hyalina практически полностью соответствовал самому распространенному типу предполагаемой вторичной структуры D. longispina (рис. 1,в). Предполагаемые модели вторичной структуры для ITS2-последовательностей D. umbra соответствовали двум слабо различающимся типам (рис. 2). Для образцов D. cf. longispina выявлено три типа таких моделей, при этом первая модель (рис. 3,а) наиболее сильно отличалась от остальных (рис. 3,б, в). В целом различия между всеми предполагаемыми моделями вторичных структур ITS2 для каждой формы/вида дафний были обусловлены длиной тандемных повторов (рис. 1–3). Кроме того, во всех моделях довольно четко можно было определить только две канонические структурные единицы – 2-ю (по наличию пиримидин-пиримидинового несоответствия) и 3-ю (с характерным мотивом в верхней части) спирали, а для образца D. longispina Brb2-L2 2-я спираль также была плохо различимой (рис. 1,д).
Рис. 1.
Схемы предполагаемых вторичных структур ITS2 D. longispina: а – PL16 (тип 1), б – PL15 (тип 2), в – GlH1 (тип 1), г – DdL13 (тип 1), д – Brb2-L2 (тип 3). II и III – 2-я и 3-я спирали; стрелки – пиримидин-пиримидиновые совпадения для 2-й спирали.
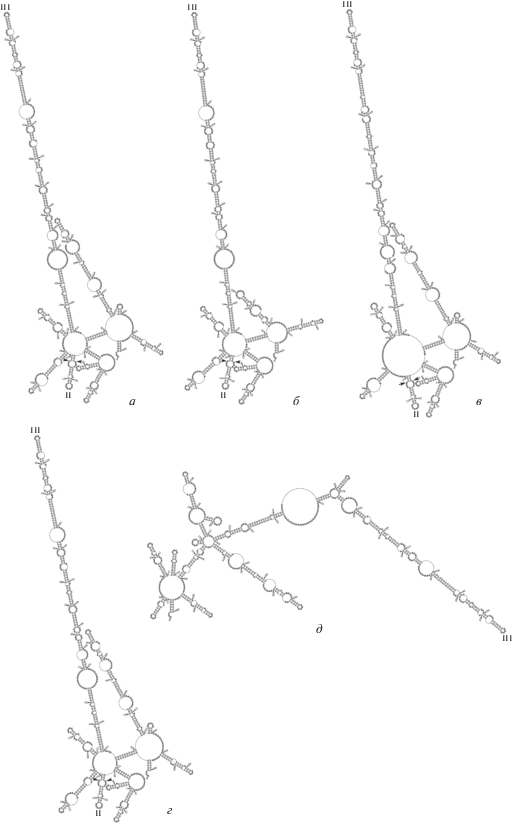
Рис. 2.
Схемы предполагаемых вторичных структур ITS2 D. umbra: а – BrU15, б – D. umbra #AY730403. Остальные обозначения см. рис. 1.
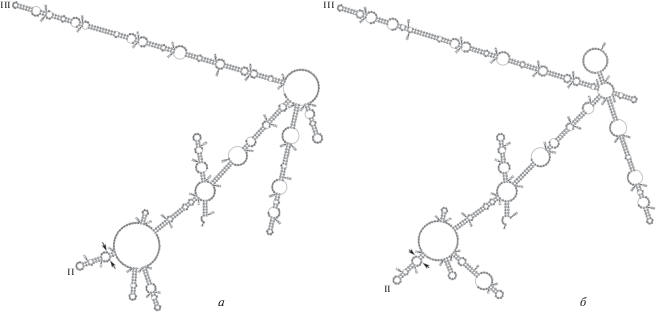
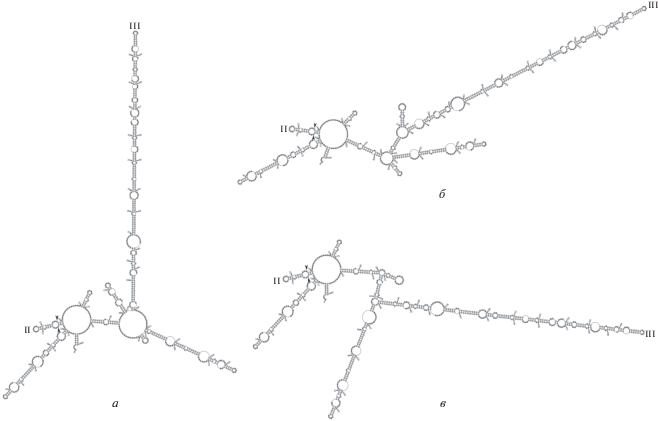
Рис. 3.
Схемы предполагаемых вторичных структур ITS2 D. cf. longispina: а – Brb1-Dsp2, б – Brb1-Dsp10, в – Brb1-Dsp12. Остальные обозначения см. рис. 1.
Анализ компенсаторных замен
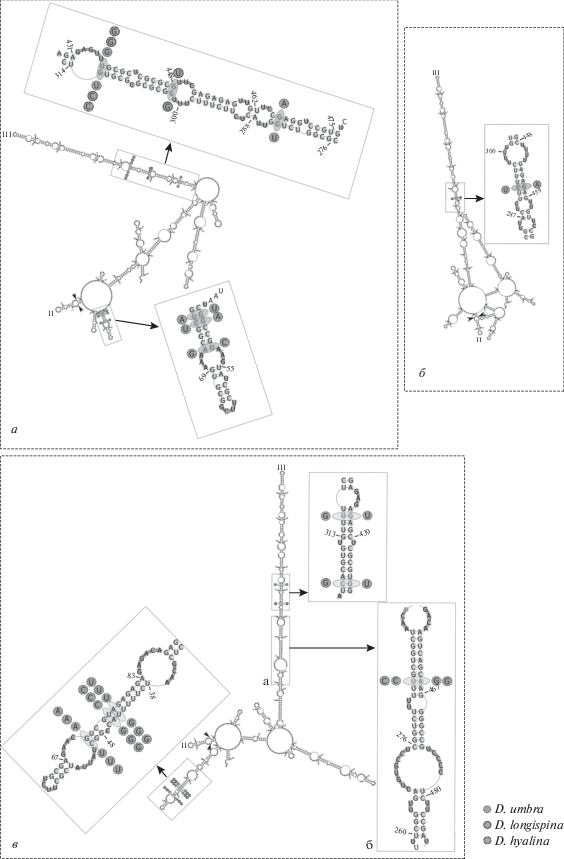
Как и предполагалось, компенсаторные замены были обнаружены при попарном сравнении моделей вторичных структур ITS2 D. longispina с предполагаемыми моделями D. umbra и D. cf. longispina (табл. 2; рис. 4,а, в). Число CBC-замен при сравнении D. umbra с другими формами/видами варьировало от 1 (D. umbra – D. hyalina) до 5 (D. umbra – D. cf. longispina). Наибольшее число CBC-замен зарегистрировано при попарном сравнении моделей вторичных структур ITS2 D. longispina и D. hyalina с D. cf. longispina (рис. 4,в). Компенсаторные замены при попарном сравнении вторичных структур ITS2 D. longispina и D. hyalina обнаружены только для одного образца D. longispina Brb2-L2 (рис. 4,б). Все CBC-замены обнаружены либо во 2-й и 3-й спиралях одновременно, либо только в 3-й спирали (D. longispina – D. hyalina).
Таблица 2.
Общее число компенсаторных (CBC) замен между предполагаемыми вторичными структурами ITS2 для анализируемых форм/видов группы D. longispina
| DL | Brb2-L2 | PL17 | DH | DU | Brb1-Dsp2 | Brb1-Dsp10 | |
|---|---|---|---|---|---|---|---|
| DL | |||||||
| Brb2-L2 | 0 | ||||||
| PL17 | 0 | 0 | |||||
| DH | 0 | 1 | 0 | ||||
| DU | 2 | 2 | 1 | 1 | |||
| Brb1-Dsp2 | 4 | 4 | 3 | 3 | 4 | ||
| Brb1-Dsp10 | 5 | 5 | 4 | 3 | 5 | 0 | |
| Brb1-Dsp12 | 3 | 3 | 3 | 3 | 4 | 0 | 0 |
Рис. 4.
CBC-анализ для вторичных структур ITS2: а – D. umbra с D. longispina/D. hyalina и D. cf. longispina (показана структура D. umbra), б – D. hyalina c D. longispina (показана структура D. hyalina), в – D. cf. longispina c D. umbra и D. longispina/D. hyalina (показана структура D. cf. longispina Brb1-Dsp2; а, б – участки структур Brb1-Dsp2 и Brb1-Dsp12/Brb1-Dsp10 соответственно).
Филогения
Реконструкция филогенетических взаимоотношений на основе последовательностей ITS2 между видами группы D. longispina с помощью различных методов и алгоритмов выявила сходную топологию деревьев (рис. 5, 6). Во всех случаях анализируемые виды сформировали отдельные кластеры со значимой поддержкой ветвей. На филогенетическом дереве, полученном с учетом вторичной структуры ITS2 с использованием алгоритма Profile Neighbor-Joining, кластеры для видов D. hyalina, D. umbra и D. cf. longispina выявились более четко с высокой поддержкой ветвей (рис. 5,а). Простирающаяся сеть на основе длин ветвей между ITS2-гаплотипами (также с учетом вторичной структуры ITS2) показала, что группа лонгиспино-подобных особей D. cf. longispina наиболее сильно удалена от остальных видов (рис. 5,б). Этот результат хорошо согласуется с данными об эволюционной дивергенции между последовательностями ITS2 – самые высокие значения зарегистрированы при попарном сравнении последовательностей всех видов с последовательностями D. cf. longispina (табл. 3).
Рис. 5.
Филогенетические деревья, построенные с учетом вторичной структуры ITS2 для видов рода Daphnia. а – дерево, реконструированное с помощью профиля присоединения ближайшего соседа (Profile Neighbor-Joining, PNJ) в программе ProfDistS на основе модели GTR. Дерево визуализировано в программе NJplot, показаны бутстрэп-значения при 1000 репликаций. Профили получены на основе “Pi” – идентичности пороговой величины (identity threshold), “Pb” – порогового бутстрэп-значения (bootstrap threshold). Профили: D. galeata (profile 1) – KgG28, ShG11; D. hyalina (profile 2) – GlH1-GlH3; D. longispina (profile 3) – DdL14, DdL15, TlL9, TlL11, KgL, ZdL1, ZdL2, Brb1-L9, Brb2-L2, Brb2-L4, PL15; D. umbra (profile 4) – BrU15, BrU18-BrU20; D. cf. longispina (profile 5) – Brb1-Dsp2, Brb1-Dsp16; б – простирающееся филогенетическое дерево, визуализированное в программе SplitsTree с помощью алгоритма “equal angle” на основе длины ветвей между кладами. Масштаб – число ожидаемых замен на сайт.
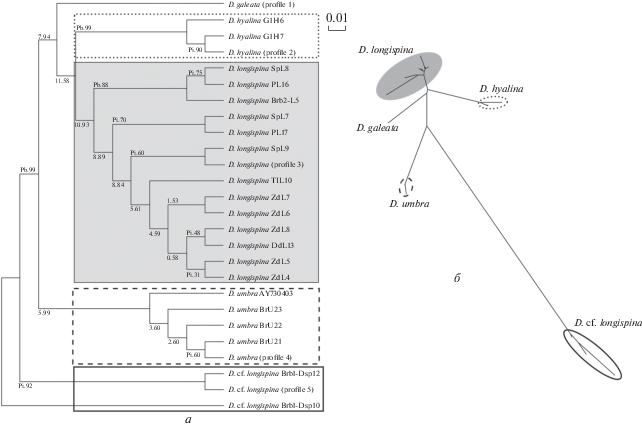
Рис. 6.
Филогенетические деревья, реконструированные без учета вторичной структуры ITS2. а – Байесовский метод, BI; б – метод максимального правдоподобия, ML. В узлах ветвления указаны апостериорная вероятность (BI, %) и бутстрэп-значения (ML, %). Масштаб – число ожидаемых замен на сайт.
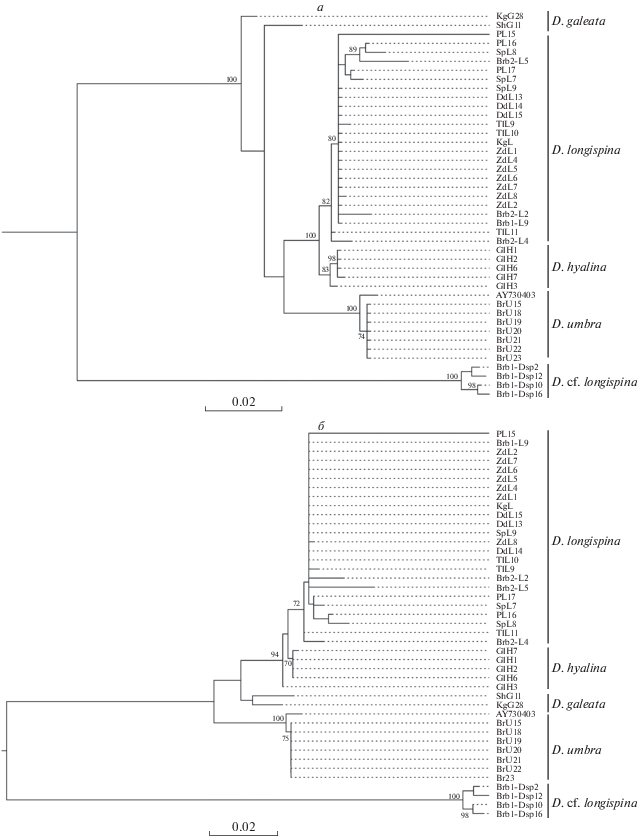
Таблица 3.
Оценка эволюционной дивергенции в пределах и между группами последовательностей ITS2 ядерной ДНК для форм/видов группы D. longispina (p-дистанции, %)
| Вид | В пределах группы | D. longispina | D. hyalina | D. umbra | D. cf. longispina | D. galeata |
|---|---|---|---|---|---|---|
| D. longispina | 1.0 ± 0.1 | – | 0.4 | 0.6 | 1.6 | 0.6 |
| D. hyalina | 0.2 ± 0.1 | 1.3 | – | 0.6 | 0.4 | 0.5 |
| D. umbra | 0.2 ± 0.1 | 3.0 | 2.5 | – | 1.6 | 0.6 |
| D. cf. longispina | 0.9 ± 0.3 | 18.0 | 16.9 | 17.2 | – | 1.6 |
| D. galeata | 2.0 ± 0.6 | 2.8 | 2.3 | 2.7 | 17.2 | – |
ОБСУЖДЕНИЕ
Анализ второго промежуточного спейсера ITS2 у криптических и близкородственных видов группы D. longispina в первую очередь выявил высокую вариабельность фрагмента. Последовательности различались по уровню полиморфизма и длине, которая зависела от числа и длины тандемных повторов. Наиболее вариабельными оказались последовательности типичных особей D. longispina (идентифицированных по морфологическим признакам и митохондриальной ДНК). Длина и CG-насыщение последовательностей ITS2 у дафний оказались сопоставимыми с таковыми у некоторых других артропод, клещей [23] и амфипод [27], но сильно отличаются по этим показателям от копепод [28, 45]. Очевидно, вследствие высокой вариабельности последовательностей ITS2 основные четыре спирали, характеризующие вторичную структуру эукариот, оказались плохо распознаваемыми, что, впрочем, отмечалось в некоторых подобных исследованиях вторичной структуры ITS2 у артропод [19, 21, 23, 27].
Анализ компенсаторных замен во вторичных структурах ITS2 подтверждает видовую самостоятельность криптических видов группы D. longispina, которые не отличаются (D. cf. longispina) или слабо отличаются (D. umbra, см. [46]) от D. longispina по морфологическим признакам. Ранее было показано, что даже одна CBC-замена указывает на принадлежность особей к разным видам с точностью до 93%, однако их отсутствие также не может являться признаком принадлежности особей к одному виду [22, 26]. При сравнении вторичных структур ITS2 видов D. longispina и D. hyalina обнаружена единственная полукомпенсаторная замена в одном попарном сравнении, что, скорее всего, свидетельствует в пользу их видовой самостоятельности. Наличие компенсаторных замен во вторичной структуре ITS2 D. cf. longispina подтверждает предположение о видовом статусе особей этой дивергентной митохондриальной линии из временного водоема бассейна оз. Чаны (Новосибирская обл.). Эти longispina-подобные дафнии обитают симпатрично с типичной формой D. longispina, и между ними не выявлено явных морфологических различий, за исключением некоторых различий в форме тела [10, 47]. Результаты CBC-анализа согласуются с филогенией видов группы D. longispina. В филогенетических деревьях, реконструированных как с учетом вторичной структуры ITS2, так и без нее, все образцы D. hyalina формируют отдельный, хорошо поддерживаемый кластер. Возможно, такое сходство вторичных структур ITS2 у D. longispina и D. hyalina является следствием недавнего видообразования, либо неполного сортинга. Подобное сходство между вторичными структурами ITS2 ранее отмечено при изучении копепод [28]. Кроме того, этот результат может быть следствием гибридизации с последующей интрогрессией ядерной ДНК, которая характерна для видов группы D. longispina [17, 48].
К криптическим видам группы D. longispina применима когезионная концепция вида [49, 50], поскольку, несмотря на гибридизацию и интрогрессию, они сохраняют видовую обособленность (например, D. longispina – D. hyalina). Каждый криптический вид в этой группе дафний имеет собственную эволюционную направленность, если обсуждать наши результаты в свете эволюционной теории вида [49]. Обнаруженный нами криптический вид D. cf. longispina не является близкородственным по отношению к другим видам группы D. longispina, поскольку монофилия между ними не поддерживается ни одним из анализов, и его можно отнести к так называемым видам-двойникам. Морфологическое сходство между D. longispina и D. cf. longispina вполне объясняется гипотезой морфологической конвергенции между эволюционно далекими видами, которая появляется вследствие воздействия сходных режимов отбора [51]. Однако возможна и ситуация, когда различия между таксонами будут найдены, например при сопоставлении признаков взрослых самцов, которые оказываются обычно более “полезными” для систематики дафний разных групп видов [7, 46].
В целом ядерная филогения на основе последовательностей ITS2 для криптических видов группы D. longispina соответствует результатам митохондриальной филогении, полученной нами ранее на основе изменчивости генов 16S и 12S [9–11], что не подтверждает недавно полученные данные об их несогласованности [17]. Как было показано ранее, использование информации о вторичной структуре ITS2 позволяет более точно реконструировать филогенетические отношения между видами в пределах рода и выявить границы между криптическими видами и дивергентными митохондриальными линиями даже в начальной стадии их радиации [24, 52].
Результаты настоящего исследования показали, что использование информации о вторичной структуре второго промежуточного транскрибируемого спейсера ITS2 ядерной ДНК позволяет более точно установить филогенетические связи между криптическими и близкородственными видами группы D. longispina. Основные клады, полученные на основе анализа изменчивости ядерного маркера, согласуются с кладами митохондриальной филогении (16S, 12S, ND2). Использование информации о вторичной структуре ITS2 для видов группы D. longispina значительно повышает уровень разрешения между кладами. Анализ компенсаторных замен (CBC) во вторичной структуре ITS2 позволяет с высокой степенью надежности дифференцировать близкородственные и криптические виды группы D. longispina, которые морфологически не отличаются (или плохо отличаются) друг от друга. Таким образом, данный подход может применяться для решения схожих задач при таксономических исследованиях низших ракообразных в целом и отдельных видовых комплексов.
Автор выражает благодарность А.А. Котову (ИПЭЭ РАН, г. Москва) и Н.А. Бочкареву (ИСиЭЖ СО РАН, г. Новосибирск) за ценные замечания при подготовке рукописи и помощь в сборе проб зоопланктона.
Исследование поддержано Программой фундаментальных научных исследований (ФНИ) государственных академий наук на 2013–2020 гг., проект № VI.51.1.9. АААА-А16-116121410119-4.
ПРИЛОЖЕНИЕ.
Нуклеотидный состав, положение и длина тандемных повторов в последовательностях ITS2 для форм/видов группы D. longispina
| Вид (образец) | Позиция | Число повторов | Мотив |
|---|---|---|---|
| D. longispina | 78 | 3 | GAGAGA |
| (PL15) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 7 | GAGAGAGAGAGAGA | |
| 449 | 3 | GAGAGA | |
| 529 | 3 | GUGUGU | |
| 611 | 3 | UAUAUA | |
| D. longispina | 78 | 3 | GAGAGA |
| (PL16) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 296 | 3 | GCGCGC | |
| 305 | 3 | UCUCUC | |
| 326 | 5 | CUCUCUCUCU | |
| 342 | 3 | GUGUGU | |
| 394 | 4 | AGAGAGAG | |
| 419 | 6 | GAGAGAGAGAGA | |
| 445 | 3 | GAGAGA | |
| 525 | 3 | GUGUGU | |
| 601 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (PL17) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 438 | 3 | CGCGCG | |
| 449 | 3 | GAGAGA | |
| 529 | 3 | GUGUGU | |
| 605 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (SpL7) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 601 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (SpL8) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 296 | 3 | GCGCGC | |
| 326 | 5 | CUCUCUCUCU | |
| 342 | 3 | GUGUGU | |
| 394 | 4 | AGAGAGAG | |
| 419 | 7 | GAGAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 479 | 3 | CUCUCU | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (SpL9) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 7 | GAGAGAGAGAGAGA | |
| 449 | 3 | GAGAGA | |
| 529 | 3 | GUGUGU | |
| 605 | 3 | AUAUAU | |
| D. longispina(DdL13, DdL14, DdL15, TlL9, KgL, ZdL1, ZdL2, ZdL4, ZdL5, ZdL6, ZdL7, Brb1-L9) | 78 | 3 | GAGAGA |
| 114 | 3 | CGCGCG | |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (ZdL8) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 371 | 3 | CUGCUGCUG | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (TlL10) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 528 | 3 | GUGUGU | |
| 604 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (TlL11) | 114 | 3 | CGCGCG |
| 171 | 3 | AAAAAA | |
| 178 | 3 | UUUUUU | |
| 292 | 3 | GCGCGC | |
| 301 | 3 | UCUCUC | |
| 322 | 5 | CUCUCUCUCU | |
| 338 | 3 | GUGUGU | |
| 390 | 4 | AGAGAGAG | |
| 415 | 9 | GAGAGAGAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (Brb2-L2) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (Brb2-L4) | 114 | 3 | CGCGCG |
| 171 | 3 | AAAAAA | |
| 178 | 3 | UUUUUU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 328 | 5 | CUCUCUCUCU | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. longispina | 78 | 3 | GAGAGA |
| (Brb2-L5) | 114 | 3 | CGCGCG |
| 177 | 3 | UUUUUU | |
| 238 | 3 | UGUGUG | |
| 256 | 3 | UGUGUG | |
| 263 | 3 | GUGUGU | |
| 298 | 3 | GCGCGC | |
| 307 | 3 | UCUCUC | |
| 323 | 7 | UCUCUCUCUCUCUC | |
| 344 | 3 | GUGUGU | |
| 396 | 4 | AGAGAGAG | |
| 421 | 6 | GAGAGAGAGAGA | |
| 447 | 3 | GAGAGA | |
| 527 | 3 | GUGUGU | |
| 603 | 3 | AUAUAU | |
| D. hyalina | 78 | 3 | GAGAGA |
| (GlH1, GlH2, GlH3, GlH7) | 114 | 3 | CGCGCG |
| 171 | 3 | AAAAAA | |
| 178 | 3 | UUUUUU | |
| 235 | 3 | GUGUGU | |
| 303 | 3 | GCGCGC | |
| 312 | 3 | UCUCUC | |
| 333 | 5 | CUCUCUCUCU | |
| 349 | 3 | GUGUGU | |
| 401 | 4 | AGAGAGAG | |
| 426 | 5 | GAGAGAGAGA | |
| 441 | 3 | CGCGCG | |
| 452 | 3 | GAGAGA | |
| 534 | 3 | GUGUGU | |
| 610 | 3 | AUAUAU | |
| D. hyalina | 78 | 3 | GAGAGA |
| (GlH6) | 114 | 3 | CGCGCG |
| 171 | 3 | AAAAAA | |
| 178 | 3 | UUUUUU | |
| 235 | 3 | GUGUGU | |
| 303 | 3 | GCGCGC | |
| 312 | 3 | UCUCUC | |
| 333 | 5 | CUCUCUCUCU | |
| 349 | 3 | GUGUGU | |
| 401 | 4 | AGAGAGAG | |
| 426 | 5 | GAGAGAGAGA | |
| 441 | 3 | CGCGCG | |
| 452 | 3 | GAGAGA | |
| 536 | 3 | GUGUGU | |
| 612 | 3 | AUAUAU | |
| D. umbra(BrU15, BrU20, BrU21, BrU22, BrU23) | 25 | 4 | UUUUUUUU |
| 80 | 3 | GAGAGA | |
| 116 | 3 | CGCGCG | |
| 173 | 3 | AAAAAA | |
| 180 | 3 | UUUUUU | |
| 238 | 3 | GUGUGU | |
| 301 | 5 | CGCGCGCGCG | |
| 332 | 5 | CUCUCUCUCU | |
| 348 | 3 | GUGUGU | |
| 400 | 4 | AGAGAGAG | |
| 425 | 4 | GAGAGAGA | |
| 442 | 3 | CGCGCG | |
| 451 | 3 | GAGAGA | |
| 603 | 3 | AUAUAU | |
| 657 | 6 | AUAUAUAUAUAU | |
| D. umbra AY730403 | 25 | 4 | UUUUUUUU |
| 71 | 3 | AAAAAA | |
| 84 | 3 | GAGAGA | |
| 120 | 3 | CGCGCG | |
| 177 | 3 | AAAAAA | |
| 184 | 3 | UUUUUU | |
| 242 | 3 | GUGUGU | |
| 305 | 5 | CGCGCGCGCG | |
| 336 | 5 | CUCUCUCUCU | |
| 352 | 3 | GUGUGU | |
| 406 | 4 | AGAGAGAG | |
| 431 | 4 | GAGAGAGA | |
| 448 | 3 | CGCGCG | |
| 457 | 3 | GAGAGA | |
| 609 | 3 | AUAUAU | |
| 663 | 6 | AUAUAUAUAUAU | |
| D. cf. longispina | 76 | 5 | AGAGAGAGAG |
| (Brb1-Dsp2, Brb1-Dsp12) | 116 | 3 | CGCGCG |
| 247 | 3 | UGUGUG | |
| 280 | 3 | UUUUUU | |
| 308 | 3 | GUGUGU | |
| 350 | 3 | GUGUGU | |
| 400, 401 | 5 | AGAGAGAGAG | |
| 425, 526 | 6 | GAGAGAGAGAGA | |
| 647, 428 | 3 | AAAAAA | |
| D. cf. longispina | 76 | 5 | AGAGAGAGAG |
| (Brb1-Dsp10) | 116 | 3 | CGCGCG |
| 235 | 4 | GUGUGUGU | |
| 247 | 3 | UGUGUG | |
| 280 | 3 | UUUUUU | |
| 307 | 3 | GUGUGU | |
| 349 | 3 | GUGUGU | |
| 400 | 5 | AGAGAGAGAG | |
| 425 | 6 | GAGAGAGAGAGA | |
| 646 | 3 | AAAAAA | |
| D. cf. longispina | 76 | 5 | AGAGAGAGAG |
| (Brb1-Dsp16) | 116 | 3 | CGCGCG |
| 235 | 4 | GUGUGUGU | |
| 247 | 3 | UGUGUG | |
| 280 | 3 | UUUUUU | |
| 307 | 3 | GUGUGU | |
| 400 | 5 | AGAGAGAGAG | |
| 425 | 6 | GAGAGAGAGAGA | |
| 646 | 3 | AAAAAA |
Список литературы
Petrusek A., Kořínek V., Černý M. Analysis of Cladoceran cryptic species: what approaches can we take? // Acta Facultatis Ecol. 2003. V. 10. S. 1. P. 105–107.
Bickford D., Lohman D.J., Sodhi N.S. et al. Cryptic species as a window on diversity and conservation // Trends Ecol. Evol. 2006. V. 22(3). doi https://doi.org/10.1016/ j.tree.2006.11.004
Kordbacheh A., Garbalena G., Walsh E.J. Population structure and cryptic species in the cosmopolitan rotifer Euchlanis dilatata // Zool. J. Linn. Soc. 2017. V. XX. P. 1–21. doi https://doi.org/10.1093/zoolinnean/zlx027
Котов А.А. Морфология и филогения Anomopoda (Crustacea: Cladocera). М.: КМК, 2013. 638 с.
Котов А.А. Фаунистические комплексы Cladocera (Crustacea, Branchiopoda) Восточной Сибири и Дальнего Востока России // Зоол. журн. 2016. Т. 95. № 7. С. 748–768. doi https://doi.org/10.7868/S0044513416070059
Petrusek A., Thielsch A., Schwenk K. Mitochondrial sequence variation suggests extensive cryptic diversity within the Western Palearctic Daphnia longispina complex // Limnol. Oceanogr. 2012. V. 57. № 6. P. 1838–1845. doi https://doi.org/10.4319/lo.2012.57.6.1838
Kotov A.A. A critical review of the current taxonomy of the genus Daphnia O.F. Müller, 1785 // Zootaxa. 2015. V. 3911. P. 184–200. doi https://doi.org/10.11646/zootaxa.3911.2.2
Möst M., Petrusek A., Sommaruga R. et al. At the edge and on the top: molecular identification and ecology of Daphnia dentifera and D. longispina in high-altitude Asian lakes // Hydrobiologia. 2013. V. 715. P. 51–62. doi https://doi.org/10.1007/s10750-012-1311-x
Zuykova E.I., Bochkarev N.A., Katokhin A.V. Identification of the Daphnia species (Crustacea: Cladocera) in the lakes of the Ob and Yenisei River basins: morphological and molecular phylogenetic approaches // Hydrobiologia. 2013. V. 715. P. 135–150. doi https://doi.org/10.1007/ s10750-012-1423-3
Зуйкова Е.И., Бочкарев Н.А., Катохин А.В. Молекулярно-генетическая диагностика и филогения видов рода Daphnia (Crustacea: Cladocera) из водоемов бассейна озера Чаны // Генетика. 2013. Т. 49. № 2. С. 235–243. doi https://doi.org/10.7868/S0016675812120181
Зуйкова Е.И., Бочкарев Н.А., Шевелева Н.Г. Генетический полиморфизм, распространение гаплотипов и филогения видов рода Daphnia (Cladocera: Anomopoda) из некоторых водоемов России по результатам секвенирования гена 16S мтДНК // Генетика. 2016. Т. 52. № 6. С. 672–684. doi https://doi.org/10.7868/ S0016675816040135
Ventura M., Petrusek A., Miró A. et al. Local and regional founder effects in lake zooplankton persist after thousands of years despite high dispersal potential // Mol. Ecol. 2014. V. 23. P. 1014–1027. doi https://doi.org/10.1111/ mec.12656
Dlouhá Ś., Thielsch A., Kraus R.H.S. et al. Identifying hybridizing taxa within the Daphnia longispina species complex: a comparison of genetic methods and phenotypic approaches // Hydrobiologia. 2010. V. 643. P. 107–122. doi https://doi.org/10.1007/s10750-010-0128-8
Keller B., Wolinska J., Tellenbach C., Spaak P. Reproductive isolation keeps hybridizing Daphnia species distinct // Limnol. Oceanogr. 2007. V. 52. № 3. P. 984–991.
Gießler S., Englbrecht C.C. Dynamic reticulate evolution in a Daphnia multispecies complex // J. Exp. Zool. 2009. V. 311. P. 531–549. doi https://doi.org/10.1002/jez.550
Taylor D.J., Sprenger H.L., Ishida S. Geographic and phylogenetic evidence for dispersed nuclear introgression in a daphniid with sexual propagules // Mol. Ecol. 2005. V. 14. P. 525–537. doi https://doi.org/10.1111/j.1365-294X.2005.02415.x
Thielsch A., Knell A., Mohammadyari A. et al. Divergent clades or cryptic species? Mitonuclear discordance in a Daphnia species complex // BMC Evol. Biol. 2017. V. 17(227). doi https://doi.org/10.1186/s12862-017-1070-4
Coleman A.W. ITS2 is a double-edged tool for eukaryote evolutionary comparisons // Trends Genet. 2003. V. 19. P. 370–375. doi https://doi.org/10.1016/S0168-9525(03)00118-5
Schultz J., Maisel S., Gerlach D. et al. A common core of secondary structure of the internal transcribed spacer 2 (ITS2) throughout the Eukaryota // RNA. 2005. V. 11. P. 361–364. http://www.rnajournal.org/cgi/doi/ 10.1261/rna.7204505
Schultz J., Wolf M. ITS2 sequence-structure analysis in phylogenetics: A how-to manual for molecular systematics // Mol. Phyl. Evol. 2009. V. 52. P. 520–523. doi https://doi.org/10.1016/j.ympev.2009.01.008
Coleman A.W. Pan-eukaryote ITS2 homologies revealed by RNA secondary structure // Nucl. Acids. Res. 2007. V. 35. № 10. P. 3322–3329. doi https://doi.org/10.1093/nar/gkm233
Müller T., Philippi N., Dandekar T. et al. Distinguishing species // RNA. 2007. V. 13. P. 1469–1472. http:// www.rnajournal.org/cgi/doi/10.1261/rna.617107
Hlinka O., Murrell A., Barker C. Evolution of the secondary structure of the rRNA internal transcribed spacer 2 (ITS2) in hard ticks (Ixodidae, Arthropoda) // Heredity. 2002. V. 88. P. 275–279.
Wiemers M., Keller A., Wolf M. ITS2 secondary structure improves phylogeny estimation in a radiation of blue butterflies of the subgenus Agrodiaetus (Lepidoptera: Lycaenidae: Polyommatus) // BMC Evol. Biol. 2009. doi https://doi.org/10.1186/1471-2148-9-300
Ullrich B., Reinhold K., Niehuis O., Misof B. Secondary structure and phylogenetic analysis of the internal transcribed spacers 1 and 2 of bush crickets (Orthoptera: Tettigoniidae: Barbitistini) // J. Zool. Syst. Evol. Res. 2009. doi https://doi.org/10.1111/j.1439-0469.2009.00553.x
Ruhl M.W., Wolf M., Jenkins T.M. Compensatory base changes illuminate morphologically difficult taxonomy // Mol. Phyl. Evol. 2010. V. 54. P. 664–669. doi https://doi.org/10.1016/ j.ympev.2009.07.036
Kornobis E., Pálsson S. The ITS region of groundwater amphipods: length, secondary structure and phylogenetic information content in Crangonyctoids and Niphargids // J. Zool. Syst. Evol. Res. 2012. doi https://doi.org/10.1111/jzs.12006
Di Capua I., Maffucci F., Pannone R. et al. Molecular phylogeny of Oncaeidae (Copepoda) using nuclear ribosomal internal transcribed spacer (ITS rDNA) // PLoS One. 2017. doi https://doi.org/10.1371/journal.pone.0175662
Zuykova E.I., Bochkarev N.A., Semenova A.S., Katokhin A.V. Morphological differentiation, mitochondrial and nuclear DNA variability between geographically distant populations of Daphnia galeata and Daphnia cucullata (Anomopoda, Daphniidae) // J. Sib. Fed. Univ. Biol. 2010. V. 4. № 3. P. 434–453.
Зуйкова Е.И., Симонов Е.П., Бочкарев Н.А. Сравнительный морфологический и генетический анализ популяций и видов рода Daphnia O.F. Müller, 1785 (Crustacea, Daphniidae) из озер Глубокое и Чаны // Изв. РАН. Серия биологическая. 2017. № 3. С. 262–275. doi https://doi.org/10.7868/S0002332917030134
Koetschan C., Förster F., Keller A. et al. The ITS2 Database III – sequences and structures for phylogeny // Nucl. Acids Res. 2009. doi https://doi.org/10.1093/nar/gkp966
Keller F., Schleicher T., Schultz J. et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation // Gene. 2009. V. 430. P. 50–57. doi https://doi.org/10.1016/ j.gene.2008.10.012
Seibel P.N., Müller T., Dandekar T. et al. 4SALE – a tool for synchronous RNA sequence and secondary structure alignment and editing // BMC Bioinformatics. 2006. V. 7. P. 498. doi https://doi.org/10.1186/1471-2105-7-498
Seibel P.N., Müller T., Dandekar T., Wolf M. Synchronous visual analysis and editing of RNA sequence and secondary structure alignments using 4SALE // BMC Res. Notes. 2008. V. 1. P. 91. doi https://doi.org/10.1186/1756-0500-1-91
Wolf M., Friedrich J., Dandekar T., Muller T. CBCAnalyzer: inferring phylogenies based on compensatory base changes in RNA secondary structures // In Silico Biol. 2005. V. 5. P. 291–294.
Friedrich J., Dandekar T., Wolf M., Muller T. ProfDists: a tool for the construction of large phylogenetic trees based on profile distances // Bioinformatics. 2005. V. 21. P. 2108–2109. doi https://doi.org/10.1093/bioinformatics/bti289
Wolf M., Ruderisch B., Dandekar T. et al. ProfDistS: profiledistance based phylogeny on sequence-structure alignments // Bioinformatics. 2008. V. 24. P. 2401–2402. doi https://doi.org/10.1093/bioinformatics/btn453
Perrière G., Gouy M. WWW-Query: An on-line retrieval system for biological sequence banks // Biochimie. 1996. V. 78. P. 364–369.
Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies // Mol. Biol. Evol. 2006. V. 23. № 2. P. 254–267. doi https://doi.org/10.1093/molbev/msj030
Kumar S., Stecher G., Tamura K. MEGA7. Molecular Evolutionary Genetics Analysis version 7.0 // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874. doi https://doi.org/10.1093/molbev/msw054
Huelsenbeck J.P., Ronquist F. MRBAYES: Bayesian inference of phylogeny // Bioinformatics. 2001. V. 17. P. 754–755.
Ronquist F., Huelsenbeck J.P. MRBAYES 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. P. 1572–1574. doi https://doi.org/10.1093/bioinformatics/btg180
Rambaut A., Suchard M.A., Xie D., Drummond A.J. Tracer V. 1.6. 2014. Available from http:// tree.bio.ed.ac.uk/software/tracer/.
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J. C., Guirao-Rico S. et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets // Mol. Biol. Evol. 2017. V. 34. P. 3299–3302. doi 10.1093/molbev/msx248
Zagoskin M.V., Lazareva V.I., Grishanin A.K., Mukha D.V. Phylogenetic information content of copepoda ribosomal DNA repeat units: ITS1 and ITS2 // BioMed Res. Int. 2014. doi https://doi.org/10.1155/2014/926342
Zuykova E.I., Simonov E.P., Bochkarev N.A. et al. Resolution of the Daphnia umbra problem (Crustacea: Cladocera) using an integrated taxonomic approach // Zool. J. Linn. Soc. 2018. doi https://doi.org/10.1093/zoolinnean/zly015
Зуйкова Е.И., Бочкарев Н.А. Популяционная и межвидовая морфологическая изменчивость видов рода Daphnia O.F. Müller. 1785 (Cladocera, Daphniidae) // Зоол. журн. 2016. Т. 95. № 5. С. 1–10. doi https://doi.org/10.7868/S0044513416050160
Ishida S., Takahashi A., Matsushima N. et al. The long-term consequences of hybridization between the two Daphnia species, D. galeata and D. dentifera, in mature habitats // BMC Evol. Biol. 2011. V. 11. doi https://doi.org/10.1186/ 1471-2148-11-209
Боркин Л.Я., Литвинчук С.Н., Розанов Ю.М., Скоринов Д.В. О криптических видах (на примере амфибий) // Зоол. журн. 2004. Т. 83. № 8. С. 936–960.
Templeton A.R. Using phylogeographic analyses of gene trees to test species status and processes // Mol. Ecol. 2001. V. 10. P. 779–791.
Fišer C., Robinson C.T., Malard F. Cryptic species as a window into the paradigm shift of the species concept // Mol. Ecol. 2017. doi https://doi.org/10.1111/mec.14486
Stocsits R.R., Letsch H., Hertel J. et al. Accurate and efficient reconstruction of deep phylogenies from structured RNAs // Nucl. Acids. Res. 2009. V. 37. № 18. P. 6184–6193. doi https://doi.org/10.1093/nar/gkp600
Дополнительные материалы отсутствуют.


