Генетика, 2019, T. 55, № 6, стр. 675-683
Участие микроРНК в регуляции гормональной зависимости клеток рака простаты
В. А. Тарасов 1, А. В. Набока 1, М. А. Махоткин 1, И. Е. Чикунов 3, М. Г. Тютякина 1, Д. А. Чеботарев 1, *, Е. Н. Черкасова 1, М. И. Коган 2, М. Б. Чибичян 2, Д. Г. Матишов
1 Южный научный центр Российской академии наук
344006 Ростов-на-Дону, Россия
2 Ростовский государственный медицинский университет, кафедра урологии и репродуктивного здоровья
344006 Ростов-на-Дону, Россия
3 Южно-Российский государственный политехнический университет им. М.И. Платова
346428 Новочеркасск, Россия
* E-mail: last-che@yandex.ru
Поступила в редакцию 28.06.2018
После доработки 02.10.2018
Принята к публикации 17.10.2018
Аннотация
С помощью метода масштабного параллельного секвенирования получен спектр дифференциально экспрессирующихся микроРНК в клетках здоровых тканей простаты, гормон-зависимых опухолей рака простаты, а также в клеточных линиях LNCaP и DU145. В опухолях и клеточных линиях рака предстательной железы (РПЖ) идентифицирован набор микроРНК, экспрессия которых значимо изменялась по сравнению с экспрессией в нормальных тканях простаты. В опухолевых тканях выявлено 27 аберрантно экспрессирующихся микроРНК, из которых 10 также показали значимое изменение экспрессии в клетках LNCaP и DU145. При этом семь из них продемонстрировали однонаправленное изменение экспрессии во всех образцах опухолей, а также в клеточных линиях РПЖ. Экспрессия miR-148a изменялась в клетках DU145 в противоположном направлении по сравнению с клетками LNCaP и опухолями. Кроме того, направление экспрессии let-7c, let-7b, miR-99a, miR-125b-2, miR-100, miR-10a и miR-31, не показавших значимого изменения в клетках опухолей, менялось на противоположное при переходе от клеток LNCaP к клеткам DU145. Оказалось, что мишенью miR-148a, let-7b и микроРНК, включенных в кластер miR-99a/let-7c/miR-125b-2, экспрессия которых увеличивается в клетках LNCaP и снижается в DU145, является ген рецептора инсулиноподобного фактора роста 1 (IGF1R). Полученные в работе результаты дают основание полагать, что различие в действии микроРНК в клеточных линиях связано с их репрессирующим влиянием на экспрессию IGF1R в клетках гормон-зависимой линии LNCaP и отсутствие этого влияния в клетках гормон-независимой линии DU145.
МикроРНК представляют собой короткие, размером 19–23 нуклеотида, молекулы РНК, которые, ассоциируясь с белками-аргонавтами (AGO1–4), образуют комплекс RISC (RNA-induced silencing complex). Функция зрелой микроРНК заключается в распознавании комплексом RISC целевых информационных РНК. Взаимодействие этого комплекса с иРНК приводит либо к ее деградации, либо к подавлению трансляции [1, 2]. По разным оценкам, от 30 до 60% генов человека находится под прямым контролем микроРНК [3]. МикроРНК играют ключевую роль в регуляции основных генетических процессов, таких как клеточное деление [4], апоптоз [5], репарация ДНК [6], дифференцировка [7, 8], ответ клетки на стрессовые воздействия [9].
Мутации и, как показано в последние 15–20 лет, эпигенетические нарушения онкогенов и генов-супрессоров опухоли приводят к злокачественному перерождению клеток различных органов, включая простату. В развитых странах рак предстательной железы занимает второе место среди онкологических заболеваний у мужчин. Хорошо известно, что клетки нормальной ткани, так же как и опухоли простаты, на ранних этапах развития нуждаются в андрогенах [10]. Однако в процессе развития злокачественная опухоль простаты часто приобретает способность к независимому от гормонов росту и прогрессии. Это ограничивает возможности гормональной терапии, которая широко используется при лечении рака простаты.
Несмотря на интенсивные исследования, механизм возникновения гормон-независимости опухолей простаты остается не до конца понятным и на сегодняшний день роль микроРНК в этом процессе не ясна. Ранее обнаружены микроРНК, экспрессия которых уменьшается в клетках гормон-независимых линий рака простаты по сравнению с клетками гормон-зависимых линий. К ним относятся микроРНК семейства miR-99 (miR-99a-99b-100) [11], miR-148a [12], let-7c [13] и другие, такие как miR-34a,c, miR-146a, miR-124, miR-31, miR-200b, miR-185 [14].
Известно, что IGF1R-сигнальный путь играет существенную роль в развитии рака простаты. Уровень экспрессии гена IGF1R в клетках гормон-независимой линии DU145 значительно превосходит таковой в клетках гормон-зависимой линии LNCaP [15, 16]. Однако роль микроРНК в гиперэкспрессии IGF1R в клетках DU145 остается не ясной. Данная работа посвящена анализу возможной роли микроРНК-опосредованной регуляции IGF1R в изменении зависимости клеток рака простаты от гормонов.
МАТЕРИАЛЫ И МЕТОДЫ
Образцы тканей. В работе использованы образцы гормон-зависимых опухолей локального рака предстательной железы (РПЖ) и нормальные ткани предстательной железы, предоставленные НИИ урологии и нефрологии Ростовского государственного медицинского университета. Нормальные ткани простаты, используемые в качестве контроля, были получены в ходе радикальной цистэктомии. Информированное добровольное согласие получено от всех пациентов, образцы которых были включены в исследование. Ни один из пациентов не получал гормональную терапию или химиотерапию до операции. Образцы тканей на 24 ч помещали в стабилизирующий раствор RNAlater (Ambion, США) сразу после операции, а затем измельчали и замораживали для последующего выделения РНК. Все образцы опухолей охарактеризованы морфологически в соответствии с системой TNM [17] и шкалой Глисона [18] (табл. 1). Исследование одобрено комитетом по этике Ростовского государственного медицинского университета.
Таблица 1.
Клиническая информация об образцах ткани РПЖ
| № | Возраст (лет) | Классификация по системе TNM | Оценка по шкале Глисона | Сумма по шкале Глисона | Андрогеновые рецепторы (+/–) | ПСА, нг/мл | |||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 61 | T2 | pN0 | M0 | 3 | 4 | 7 | + | 8.7 |
| 2 | 55 | T2a | pNx | M0 | 3 | 3 | 6 | + | 10.0 |
| 3 | 53 | T1c | pNx | M0 | 3 | 3 | 6 | + | 12.0 |
Иммуногистохимия. Для морфологической характеристики образцов ткани и оценки статуса андрогеновых рецепторов проводили иммуногистохимическое окрашивание. Из формалин-парафин-фиксированных образцов тканей получали пять последовательных срезов толщиной 4 мкм и монтировали на предварительно подготовленные стекла. Иммуноокрашивание проводили с использованием системы Ventana HX BenchMark™ (Ventana Medical Systems, США) в соответствии с инструкциями производителя. Использовали моноклональные мышиные антитела против андрогенного рецептора в разведении 1/50 (клон AR441, DAKO, США). В качестве хромогенного субстрата использовали диаминобензидин. Для контрастной окраски использовали гематоксилин и эозин.
Клеточные линии. Клетки гормон-зависимой линии рака простаты LNCaP культивировали в среде RPMI-1460, а клетки гормон-независимых линий DU145 – в среде EMEM. К основной среде добавляли 10 объемных процентов эмбриональной бычьей сыворотки. Клетки культивировали при 37°C в присутствии 5% CO2 во флаконах площадью 75 см2 (с интервалом времени между последовательными пересевами, равным 3 сут).
Выделение РНК. Для выделения тотальной РНК из образцов ткани простаты брали фрагмент объемом 0.5 см3, измельчали в жидком азоте и помещали в лизирующий буфер QIAzol (Qiagen, США). Для выделения РНК из клеточных линий использовали 2 × 106 клеток. Тотальную РНК выделяли с использование реагентов miRNeasy Mini Kit (Qiagen, США) по протоколу производителя. Для выделения фракции малых РНК использовали miRNeasy Mini Kit (Qiagen, США), RNeasy MinElute Cleanup Kit (Qiagen, США). Количественное определение тотальной РНК измеряли на приборе Qubit 3.0 Fluorometer (Life Science Technologies, США). Для качественного анализа тотальной РНК проводили электрофорез в денатурирующем 1%-ном агарозном геле с формальдегидом.
Масштабное параллельное секвенирование. Экспрессию микроРНК оценивали при помощи масштабного параллельного секвенирования на платформе Illumina. Для оценки первой повторности DU145 и LNCaP применяли систему MiSeq (Illumina Inc., США), для второй повторности этих клеточных линий и образцов опухолевых и нормальных тканей – HiSeq (Illumina Inc., США). В первой серии опытов для DU145 и LNCaP применяли систему MiSeq (Illumina Inc., США), для остальных случаев – HiSeq (Illumina Inc., США). Создание библиотеки кДНК и проведение масштабного параллельного секвенирования осуществляли в соответствии с методикой, описанной нами ранее [19].
Статистический анализ. При идентификации микроРНК секвенированные последовательности кДНК с помощью программы Bowtie, v. 1.2.0 [20] сравнивали с каноническими последовательностями микроРНК (miRBase, v. 21). При этом допускали замену одного нуклеотида в секвенированных последовательностях кДНК зрелой микроРНК. При нормализации уровня экспрессии сравниваемых вариантов использовали метод ТММ, реализованный в компьютерной программе edgeR [21]. В анализ включены микроРНК, зрелые формы которых представлены в анализируемой выборке не менее чем 25 копиями в пробе перевиваемой линии либо образцах ткани простаты и которые показали изменение экспрессии при доле ложных определений (FDR) ≤ 0.05.
РЕЗУЛЬТАТЫ
С использованием метода масштабного параллельного секвенирования получен спектр дифференциальной экспрессии микроРНК в клетках трех образцов гормон-зависимых опухолей РПЖ и трех образцов нормальной ткани простаты, а также в клетках линий LNCaP и DU145 в двух повторностях. Сравнивали экспрессию микроРНК в клетках каждого образца опухоли и в клетках LNCaP и DU145 с экспрессией микроРНК в клетках каждого образца нормальной ткани простаты. В результате для опухолей проведено девять, а для линий LNCaP и DU145 – шесть сравнений для каждой. Анализируемые микроРНК показали статистически значимое (FDR ≤ 0.05) однонаправленное изменение экспрессии при каждом сравнении. 27 микроРНК (табл. 2) были идентифицированы в клетках опухолей, в то время как в клетках LNCaP – 116 микроРНК, а в клетках DU145 – 87 микроРНК (рис. 1).
Таблица 2.
Изменение экспрессии микроРНК в клетках опухолей рака простаты
| микроРНК | Опухоль 1 | Опухоль 2 | Опухоль 3 | ||||||
|---|---|---|---|---|---|---|---|---|---|
| К1 | К2 | К3 | К1 | К2 | К3 | К1 | К2 | К3 | |
| Уменьшение экспрессии* | |||||||||
| miR-126-3p | –1.9 | –1.2 | –2.6 | –3.0 | –2.5 | –4.1 | –2.3 | –1.8 | –3.2 |
| miR-144-5p | –3.7 | –3.3 | –3.0 | –3.0 | –2.9 | –2.6 | –3.4 | –3.2 | –2.8 |
| miR-150-5p | –2.0 | –1.4 | –1.6 | –4.3 | –4.0 | –4.3 | –2.8 | –2.5 | –2.6 |
| miR-15b-5p | –1.8 | –1.7 | –2.2 | –1.1 | –1.2 | –1.8 | –1.9 | –1.9 | –2.4 |
| miR-16-5p | –1.1 | –0.7 | –1.0 | –1.6 | –1.5 | –2.0 | –1.2 | –1.0 | –1.3 |
| miR-21-5p | –0.8 | –0.5 | –1.0 | –1.8 | –1.8 | –2.4 | –1.5 | –1.5 | –1.9 |
| miR-223-3p | –3.4 | –3.7 | –3.3 | –3.0 | –3.5 | –3.2 | –2.4 | –2.9 | –2.4 |
| miR-301a-3p | –1.2 | –0.6 | –1.1 | –1.2 | –0.8 | –1.5 | –1.3 | –1.0 | –1.4 |
| miR-378a-3p | –1.3 | –1.2 | –1.2 | –1.1 | –1.2 | –1.4 | –1.8 | –1.9 | –1.9 |
| miR-378a-5p | –1.9 | –2.2 | –1.2 | –4.0 | –4.5 | –3.7 | –3.4 | –3.9 | –2.9 |
| miR-424-5p | –1.3 | –0.9 | –2.1 | –1.9 | –1.7 | –3.0 | –1.8 | –1.6 | –2.8 |
| miR-451a | –1.8 | –1.9 | –3.6 | –2.4 | –2.8 | –4.6 | –3.1 | –3.5 | –5.1 |
| miR-454-3p | –1.0 | –0.7 | –0.9 | –1.1 | –1.0 | –1.3 | –1.0 | –0.9 | –1.0 |
| miR-486-5p | –4.0 | –3.9 | –2.8 | –2.8 | –3.0 | –2.0 | –4.1 | –4.2 | –3.1 |
| Увеличение экспрессии* | |||||||||
| miR-1260a | 2.1 | 1.9 | 2.6 | 1.5 | 1.1 | 1.6 | 1.7 | 1.4 | 2.0 |
| miR-143-3p | 2.6 | 3.2 | 5.5 | 3.7 | 4.0 | 6.3 | 3.1 | 3.5 | 5.9 |
| miR-148a-3p | 4.6 | 4.4 | 3.4 | 3.7 | 3.4 | 2.2 | 2.0 | 1.6 | 0.6 |
| miR-148a-5p | 1.8 | 1.8 | 3.4 | 2.5 | 2.2 | 3.7 | 1.4 | 1.1 | 2.8 |
| miR-148b-3p | 2.7 | 2.9 | 1.3 | 3.0 | 3.0 | 1.3 | 2.2 | 2.2 | 0.7 |
| miR-152-3p | 0.9 | 1.0 | 0.9 | 2.3 | 2.2 | 1.9 | 1.9 | 1.7 | 1.7 |
| miR-196b-5p | 2.8 | 3.5 | 3.8 | 3.1 | 3.5 | 3.7 | 3.0 | 3.5 | 3.8 |
| miR-21-3p | 4.3 | 7.4 | 1.0 | 5.5 | 8.4 | 1.8 | 4.5 | 7.4 | 1.0 |
| miR-26b-5p | 1.2 | 1.9 | 2.0 | 1.1 | 1.5 | 1.5 | 0.7 | 1.2 | 1.4 |
| miR-30a-3p | 2.2 | 2.8 | 2.4 | 3.6 | 3.9 | 3.5 | 1.8 | 2.1 | 1.8 |
| miR-30c-2-3p | 3.5 | 4.0 | 3.8 | 5.3 | 5.6 | 5.3 | 3.8 | 4.1 | 3.9 |
| miR-582-3p | 5.2 | 5.2 | 4.4 | 5.7 | 5.4 | 4.5 | 3.4 | 3.1 | 2.4 |
| miR-99a-3p | 2.2 | 2.7 | 4.1 | 3.4 | 3.7 | 5.0 | 2.4 | 2.7 | 4.2 |
Рис. 1.
МикроРНК, однонаправлено экспрессирующиеся в исследуемых линиях и опухолях РПЖ. a – пересечение спектров микроРНК с однонаправленной повышенной экспрессией в клетках LNCaP, DU145 и опухолей РПЖ; б – пересечение спектров микроРНК с однонаправленной сниженной экспрессией в клетках LNCaP, DU145 и опухолей РПЖ.
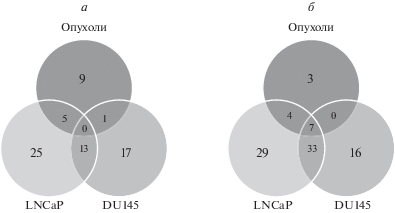
Сравнение однонаправленно экспрессирующихся микроРНК в гормон-зависимых клетках опухолей и линии LNCaP показало, что одновременно изменилась экспрессия только 16 микроРНК. При этом у семи из них экспрессия изменялась в том же направлении и в клетках DU145 (табл. 3). Экспрессия этих микроРНК отражает изменения метаболизма в злокачественных клетках, не связанные с их зависимостью от гормонов.
Таблица 3.
Однонаправленное изменение экспрессии микроРНК в клетках опухолей и обеих клеточных линий РПЖ
| микроРНК | Опухоли** | Клеточная линия | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LNCaP | DU145 | ||||||||||||
| эксперимент I | эксперимент II | эксперимент I | эксперимент II | ||||||||||
| К1 | К2 | К3 | К1 | К2 | К3 | К1 | К2 | К3 | К1 | К2 | К3 | ||
| Изменение экспрессии микроРНК* | |||||||||||||
| miR-126-3p | ↓ | –4.6 | –4.1 | –5.1 | –6.2 | –5.4 | –7.0 | –4.2 | –3.7 | –4.8 | –5.9 | –5.4 | –6.8 |
| miR-144-5p | ↓ | –8.8 | –8.0 | –7.0 | –10.0 | –9.5 | –9.1 | –8.7 | –7.9 | –6.9 | –10.0 | –9.7 | –9.2 |
| miR-150-5p | ↓ | –11.7 | –10.7 | –10.1 | –12.9 | –12.2 | –12.3 | –11.6 | –10.6 | –10.1 | –12.9 | –12.4 | –12.3 |
| miR-223-3p | ↓ | –10.8 | –10.6 | –9.5 | –12.1 | –12.1 | –11.7 | –10.8 | –10.6 | –9.4 | –12.1 | –12.3 | –11.7 |
| miR-378a-3p | ↓ | –3.4 | –3.5 | –3.1 | –3.3 | –3.1 | –3.4 | –2.4 | –2.5 | –2.2 | –2.2 | –2.3 | –2.4 |
| miR-451a | ↓ | –13.7 | –13.4 | –14.3 | –14.9 | –14.9 | –16.5 | –13.6 | –13.3 | –14.3 | –14.9 | –15.0 | –16.5 |
| miR-486-5p | ↓ | –2.2 | –2.3 | –0.8 | –2.7 | –2.6 | –1.7 | –4.8 | –4.9 | –3.5 | –5.6 | –5.7 | –4.6 |
Для четырех из них – miR-150-5р [22, 23], miR-223-3р [24], miR-378а-3р [25], miR-486-5p [26] – ранее показано участие в контроле рака простаты. Прямые данные об участии в регуляции канцерогенеза простаты трех других микроРНК, входящих в эту группу, отсутствуют. Однако известно, что miR-451а участвует в контроле колоректального рака и рака молочной железы [27, 28], miR-126-3p – рака груди [29] и рака щитовидной железы [30], miR-144-5p – рака мочевого пузыря [31].
В клеточных линиях у восьми микроРНК направление изменения экспрессии менялось на противоположное при переходе от клеток LNCaP к клеткам DU145, при этом у miR-148a уровень экспрессии менялся и в клетках опухолей (табл. 4). Для всех пяти микроРНК, показавших увеличение экспрессии в клетках LNCaP и соответственно ее уменьшение в клетках DU145, мишенью является ген IGF1R.
Таблица 4.
Разнонаправленное изменение экспрессии микроРНК в клетках опухолей и обеих клеточных линий РПЖ
| микроРНК | Опухоли** | Клеточная линия | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| LNCaP | DU145 | ||||||||||||
| эксперимент I | эксперимент II | эксперимент I | эксперимент II | ||||||||||
| К1 | К2 | К3 | К1 | К2 | К3 | К1 | К2 | К3 | К1 | К2 | К3 | ||
| Изменение экспрессии микроРНК* | |||||||||||||
| miR-99a-5p | н.з. | 1.2 | 0.8 | 6.5 | 1.6 | 1.5 | 6.5 | –8.0 | –8.4 | –2.8 | –8.1 | –8.5 | –3.3 |
| let-7c-5p | н.з. | 2.0 | 2.0 | 6.7 | 2.5 | 2.7 | 6.8 | –8.9 | –8.9 | –4.3 | –7.4 | –7.4 | –3.1 |
| miR-125b-2-3p | н.з. | 3.2 | 3.5 | 6.6 | 3.2 | 3.7 | 6.3 | –8.2 | –7.3 | н.з. | –6.6 | –6.3 | н.з. |
| let-7b-5p | н.з. | 2.4 | 2.3 | 2.1 | 2.0 | 2.1 | 1.3 | –1.1 | –1.2 | –1.5 | –0.7 | –0.8 | –1.5 |
| miR-148a-3p | ↑ | 3.1 | 2.8 | 2.1 | 1.1 | 1.1 | н.з. | –0.6 | –1.0 | –1.7 | –3.1 | –3.4 | –4.5 |
| miR-10a-5p | н.з. | –2.0 | –2.0 | –1.2 | –2.4 | –2.2 | –2.0 | 2.0 | 2.0 | 2.7 | 1.7 | 1.6 | 2.0 |
| miR-100-3p | н.з. | –10.9 | –10.0 | –8.5 | –12.2 | –11.5 | –10.6 | 4.3 | 4.5 | 5.7 | 1.0 | 1.3 | 2.1 |
| miR-31-5p | н.з. | –9.2 | –8.9 | –12.0 | –6.2 | –6.2 | –10.3 | 5.6 | 5.3 | 1.8 | 4.9 | 4.6 | 0.7 |
ОБСУЖДЕНИЕ
Ключевую роль в развитии рака простаты играет андрогенный рецептор (АR), взаимодействие которого с андрогенами приводит к его активации [32, 33]. Активированный AR приобретает способность перемещаться из цитоплазмы в ядро клетки [34]. Его взаимодействие с AREs (androgen response elements) – каноническими последовательностями ДНК в промоторных участках генов – приводит к подавлению или активации их транскрипционной активности [35]. Однако активация AR и его перемещение в ядро клетки может происходить и в отсутствие андрогенов при включении сигнального пути IGF-1 [36]. Мишенями андрогенного рецептора являются свыше 400 генов [37]. К ним относится и ген микроРНК miR-148а [38], направление экспрессии которой меняется на противоположное при переходе от клеток LNCaP к DU145 (табл. 4). Прямыми мишенями этой микроРНК являются ДНК-метилтрансферазы DNMT3b [39, 40] и DNMT1 [41], отвечающие за метилирование de novo и поддерживающие метилирование соответственно (рис. 2).
Рис. 2.
МикроРНК-зависимая регуляция экспрессии IGF1R в клетках линий LNCaP и DU145. а – схема регуляции в клетках LNCaP; б – схема регуляции в клетках DU145. 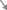 – усиление экспрессии гена или активация белка;
– усиление экспрессии гена или активация белка;  – подавление экспрессии;
– подавление экспрессии;  – отсутствие (ослабление) регулирующего действия;
– отсутствие (ослабление) регулирующего действия;  – увеличенная экспрессия гена;
– увеличенная экспрессия гена;  – уменьшенная экспрессия гена;
– уменьшенная экспрессия гена;  – предсказанная регуляция;
– предсказанная регуляция;  – верифицированная регуляция.
– верифицированная регуляция.
Кроме того, по данным miRDB (http:// mirdb.org) и TargetScan (http://www.targetscan.org) к предсказанным мишеням miR-148а относится и ген KDM2B, кодирующий лизин-специфическую деметилазу 2B. KDM2B входит в состав поликомб-комплекса 1 (PRC1). Ее связывание с полностью не метилированной ДНК CpG-островков приводит к моноубиквитинированию лизина в положении 119, гистона 2A (H2AK119 Ub1) и рекрутированию к месту связывания поликомб-комплекса 2 (PRC2), компонентом которого является гистон-метилтрансфераза 2 (EZH2). Это приводит к триметилированию лизина в 27-м положении гистона 3 (H3K27me3) и, соответственно, к гистон-зависимому подавлению транскрипционной активности генов, промоторы которых содержат CpG-островки. Кроме того, H3K27me3 является молекулярной меткой для метилирования ДНК de novo [42].
Известно, что промоторный участок гена микроРНК let-7b, мишенью которой является гистон-метилтрансфераза EZH2 [43], содержит CpG-островки и его транскрипция зависит от уровня метилирования ДНК [44]. На примере эпигенетической регуляции деления клеток эмбриональных фибробластов мыши [43] и миелоидных клеточных линий [45] показано, что активация KDM2B приводит к активации EZH2 и уменьшению экспрессии let-7b, и, напротив, уменьшение экспрессии KDM2B сопровождается уменьшением экспрессии EZH2 и увеличением экспрессии let-7b. В свою очередь Сан и соавт. [46] показали, что активность EZH2 подавляет активность гена MIR99AHG. Транскрипт этого гена содержит кластер микроРНК miR-99а/let-7c/miR-125b-2. Мишенью микроРНК, входящих в этот кластер, так же как miR-148а и let-7b, является ген IGF1R.
Для miR-148a, показавшей увеличение экспрессии в клетках LNCaP, мы также наблюдаем увеличение экспрессии в гормон-зависимых клетках опухолей. Различия в экспрессии микроРНК в клетках LNCaP и клетках опухолей касаются let-7c, let-7b, miR-99a и miR-125b. Природа этих различий остается не ясной, однако можно полагать, что она связана с изменениями в экспрессии генов, белки которых участвуют в дифференциальном созревании микроРНК, например, LIN – 28 [13, 47]. В настоящее время показано, что IGF1R играет существенную роль в развитии гормональной независимости клеток рака простаты [48]. Это может быть, в частности, связано с независимой от андрогенов активацией андрогенных рецепторов [36]. Однако в работе Алимира и соавт. [49] показано, что в гормон-независимых клетках DU145 и PC3 снижена активность гена AR по сравнению с таковой в гормон-зависимых клетках LNCaP. В этой связи не исключено, что в основе деления и выживаемости гормон-независимых клеток, культивируемых in vitro, лежит зависимый от IGF1R, но не от AR, обходной сигнальный путь [50, 51].
Полученные нами результаты дают основания полагать, что различия в действии микроРНК в двух изученных клеточных линиях в основном связаны с их репрессирующим влиянием на экспрессию IGF1R в клетках гормон-зависимой линии LNCaP и отсутствием этого влияния в клетках гормон-независимой линии DU145.
Работа выполнена в рамках Государственного задания ЮНЦ РАН, номер государственной регистрации 01201363192.
Список литературы
Lin S., Gregory R.I. MicroRNA biogenesis pathways in cancer // Nat. Rev. Cancer. 2015. V. 15. № 6. P. 321–333. https://doi.org/10.1038/nrc3932
Kim Y.K., Kim B., Kim V.N. Re-evaluation of the roles of DROSHA, Exportin 5, and DICER in microRNA biogenesis // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 13. E1881–1889. https://doi.org/10.1073/pnas.1602532113
Friedman R.C., Farh K.K.H., Burge C.B., Bartel D.P. Most mammalian mRNAs are conserved targets of microRNAs // Genome Res. 2009. V. 19. № 1. P. 92–105. https://doi.org/10.1101/gr.082701.108
Shenoy A., Blelloch R.H. Regulation of microRNA function in somatic stem cell proliferation and differentiation // Nat. Rev. Mol. Cell Biol. 2014. V. 15. № 9. P. 565–576. https://doi.org/10.1038/nrm3854
Lima R.T., Busacca S., Almeida G.M. et al. MicroRNA regulation of core apoptosis pathways in cancer // Eur. J. Cancer. 2011. V. 47. № 2. P. 163–174. https://doi.org/10.1016/ j.ejca.2010.11.005
Landau D.A., Slack F.J. MicroRNAs in mutagenesis, genomic instability, and DNA repair // Semin. Oncol. 2011. V. 38. № 6. P. 743–751. https://doi.org/10.1053/j.seminoncol.2011.08.003
Ivey K.N., Srivastava D. MicroRNAs as regulators of differentiation and cell fate decisions // Cell Stem Cell. 2010. V. 7. № 1. P. 36–41. https://doi.org/10.1016/j.stem.2010.06.012
Turner M.L., Schnorfeil F.M., Brocker T. MicroRNAs regulate dendritic cell differentiation and function // J. Immunol. 2011. V. 187. № 8. P. 3911–3917. https://doi.org/10.4049/jimmunol.1101137
Leung A.K., Sharp P.A. MicroRNA functions in stress responses // Mol. Cell. 2010. V. 40. № 2. P. 205–215. https://doi.org/10.1016/j.molcel.2010.09.027
Cucchiara V., Yang J.C., Mirone V. et al. Epigenomic regulation of androgen receptor signaling: potential role in prostate cancer therapy // Cancers. 2017. V. 9. № 1. P. 9. https://doi.org/10.3390/cancers9010009
Sun D., Lee Y.S., Malhotra A. et al. miR-99 family of MicroRNAs suppresses the expression of prostate-specific antigen and prostate cancer cell proliferation // Cancer Res. 2011. V. 71. № 4. P. 1313–1324. https://doi.org/10.1158/0008-5472.can-10-1031
Fujita Y., Kojima K., Ohhashi R. et al. miR-148a attenuates paclitaxel resistance of hormone-refractory, drug-resistant prostate cancer PC3 cells by regulating MSK1 expression // J. Biol. Chem. 2010. V. 285. № 25. P. 19076–19084. https://doi.org/10.1074/jbc.M109.079525
Nadiminty N., Tummala R., Lou W. et al. MicroRNA let-7c suppresses androgen receptor expression and activity via regulation of Myc expression in prostate cancer cells // J. Biol. Chem. 2012. V. 287. № 2. P. 1527–1537. https://doi.org/10.1074/jbc.M111.278705
Li F., Mahato R.I. MicroRNAs and drug resistance in prostate cancers // Mol. Pharm. 2014. V. 11. № 8. P. 2539–2552. https://doi.org/10.1021/mp500099g
Iwamura M., Sluss P.M., Casamento J.B., Cockett A.T. Insulin- like growth factor I: action and receptor characterization in human prostate cancer cell lines // Prostate. 1993. V. 22. № 3. P. 243–252. https://doi.org/10.1002/ pros.2990220307
Rochester M.A., Riedemann J., Hellawell G.O. et al. Silencing of the IGF1R gene enhances sensitivity to DNA-damaging agents in both PTEN wild-type and mutant human prostate cancer // Cancer Gene Ther. 2005. V. 12. № 1. P. 90–100. https://doi.org/10.1038/sj.cgt
Schröder F.H., Hermanek P., Denis L. et al. The TNM classification of prostate cancer // Prostate Suppl. 1992. V. 21. № 4. P. 129–138. https://doi.org/10.1002/pros.2990210521
Mellinger G.T., Gleason D., Bailar J. The histology and prognosis of prostatic cancer // J. Urol. 1967. V. 97. № 2. P. 331–337. https://doi.org/10.1016/S0022-5347(17)63039-8
Тарасов В.А., Бойко Н.В., Махоткин М.А. и др. Зависимость индуцированной митомицином С аберрантной экспрессии микроРНК от метилирования ДНК // Генетика. 2016. Т. 52. № 11. С. 1233–1240. https://doi.org/10.7868/S0016675816110151
Langmead B., Trapnell C., Pop M., Salzberg S.L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome // Genome Biol. 2009. V. 10. № 3. R25. https://doi.org/10.1186/gb-2009-10-3-r25
Robinson M.D., McCarthy D.J., Smyth G.K. edgeR: a Bioconductor package for differential expression analysis of digital gene expression data // Bioinformatics. 2010. V. 26. № 1. P. 139–140. https://doi.org/10.1093/bioinformatics/btp616
Dezhong L., Xiaoyi Z., Xianlian L. et al. miR-150 is a factor of survival in prostate cancer patients // J. BUON. 2015. V. 20. № 1. P. 173–179.
Liu D.Z., Zhang H.Y., Long X.L. et al. MIR-150 promotes prostate cancer stem cell development via suppressing p27Kip1 // Eur. Rev. Med. Pharmacol. Sci. 2015. V. 19. № 22. P. 4344–4352.
Kurozumi A., Goto Y., Matsushita R. et al. Tumor-suppressive microRNA-223 inhibits cancer cell migration and invasion by targeting ITGA3/ITGB1 signaling in prostate cancer // Cancer Sci. 2016. V. 107. № 1. P. 84–94. https://doi.org/10.1111/cas.12842
Avgeris M., Stravodimos K., Scorilas A. Loss of miR-378 in prostate cancer, a common regulator of KLK2 and KLK4, correlates with aggressive disease phenotype and predicts the short-term relapse of the patients // Biol. Chem. 2014. V. 395. № 9. P. 1095–1104. https://doi.org/10.1515/hsz-2014-0150
Zhang X., Zhang T., Yang K. et al. miR-486-5p suppresses prostate cancer metastasis by targeting snail and regulating epithelial–mesenchymal transition // Onco. Targets Ther. 2016. V. 9. P. 6909–6914. https://doi.org/10.2147/ ott.s117338
Bitarte N., Bandres E., Boni V. et al. MicroRNA-451 is involved in the self-renewal, tumorigenicity, and chemoresistance of colorectal cancer stem cells // Stem Cells. 2011. V. 29. № 11. P. 1661–1671. https://doi.org/10.1002/ stem.741
Kovalchuk O., Filkowski J., Meservy J. et al. Involvement of microRNA-451 in resistance of the MCF-7 breast cancer cells to chemotherapeutic drug doxorubicin // Mol. Cancer Ther. 2008. V. 7. № 7. P. 2152–2159. https://doi.org/10.1158/1535-7163.mct-08-0021
Zhu N., Zhang D., Xie H. et al. Endothelial-specific intron-derived miR-126 is down-regulated in human breast cancer and targets both VEGFA and PIK3R2 // Mol. Cell. Biochem. 2011. V. 351. № 1–2. P. 157–164. https://doi.org/10.1007/s11010-011-0723-7
Xiong Y., Kotian S., Zeiger M.A. et al. miR-126-3p inhibits thyroid cancer cell growth and metastasis, and is associated with aggressive thyroid cancer // PLoS One. 2015. V. 10. № 8. e0130496. https://doi.org/10.1371/journal.pone.0130496
Matsushita R., Seki N., Chiyomaru T. et al. Tumour-suppressive microRNA-144-5p directly targets CCNE1/2 as potential prognostic markers in bladder cancer // Br. J. Cancer. 2015. V. 113. № 2. P. 282–289. https://doi.org/10.1038/bjc.2015.195
Liu L.L., Xie N., Sun S. et al. Mechanisms of the androgen receptor splicing in prostate cancer cells // Oncogene. 2014. V. 33. № 24. P. 3140–3150. https://doi.org/10.1038/onc.2013.284
Liao R.S., Ma S., Miao L. et al. Androgen receptor-mediated non-genomic regulation of prostate cancer cell proliferation // Transl. Androl. Urol. 2013. V. 2. № 3. P. 187–196. https://doi.org/10.3978/j.issn.2223-4683.2013.09.07
Watson P.A., Arora V.K., Sawyers C.L. Emerging mechanisms of resistance to androgen receptor inhibitors in prostate cancer // Nat. Rev. Cancer. 2015. V. 15. № 12. P. 701–711. https://doi.org/10.1038/nrc4016
Denayer S., Helsen C., Thorrez L. et al. The rules of DNA recognition by the androgen receptor // Mol. Endocrinol. 2010. V. 24. № 5. P. 898–913. https://doi.org/10.1210/ me.2009-0310
Wu J.D., Haugk K., Woodke L. et al. Interaction of IGF signaling and the androgen receptor in prostate cancer progression // J. Cell. Biochem. 2006. V. 99. № 2. P. 392–401. https://doi.org/10.1002/jcb.20929
Liu S., Kumari S., Hu Q. et al. A comprehensive analysis of coregulator recruitment, androgen receptor function and gene expression in prostate cancer // eLife. 2017. V. 6. e28482. https://doi.org/10.7554/eLife.28482
Hamilton M.P., Rajapakshe K.I., Bader D.A. et al. The landscape of microRNA targeting in prostate cancer defined by AGO-PAR-CLIP // Neoplasia. 2016. V. 18. № 6. P. 356–370. https://doi.org/10.1016/j.neo.2016.04.008
Li Y., Deng X., Zeng X., Peng X. The role of mir-148a in cancer // J. Cancer. 2016. V. 7. № 10. P. 1233–1241. https://doi.org/10.7150/jca.14616
Duursma A.M., Kedde M., Schrier M. et al. miR-148 targets human DNMT3b protein coding region // RNA. 2008. V. 14. № 5. P. 872–877. https://doi.org/10.1261/ rna.972008
Braconi C., Huang N., Patel T. MicroRNA dependent regulation of DNMT-1 and tumor suppressor gene expression by Interleukin-6 in human malignant cholangiocytes // Hepatology. 2010. V. 51. № 3. P. 881–890. https://doi.org/10.1002/hep.23381
Wang W., Qin J.J., Voruganti S. et al. Polycomb group (PcG) proteins and human cancers: multifaceted functions and therapeutic implications // Med. Res. Rev. 2015. V. 35. № 6. P. 1220–1267. https://doi.org/10.1002/med.21358
Tzatsos A., Paskaleva P., Lymperi S. et al. Lysine-specific demethylase 2B (KDM2B)-let-7-enhancer of zester homolog 2 (EZH2) pathway regulates cell cycle progression and senescence in primary cells // J. Biol. Chem. 2011. V. 286. № 38. P. 33061–33069. https://doi.org/10.1074/jbc.M111.257667
Vrba L., Muñoz-Rodríguez J.L., Stampfer M.R., Futscher B.W. miRNA gene promoters are frequent targets of aberrant DNA methylation in human breast cancer // PLoS One. 2013. V. 8. № 1. e54398. https://doi.org/10.1371/ journal.pone.0054398
Karoopongse E., Yeung C., Byon J. et al. The KDM2B-let-7b-EZH2 axis in myelodysplastic syndromes as a target for combined epigenetic therapy // PLoS One. 2014. V. 9. № 9. e107817. https://doi.org/10.1371/journal.pone.0107817
Sun D., Layer R., Mueller A.C. et al. Regulation of several androgen-induced genes through the repression of the miR-99a/let-7c/miR-125b-2 miRNA cluster in prostate cancer cells // Oncogene. 2014. V. 33. № 11. P. 1448–1457. https://doi.org/10.1038/onc.2013.77
Nadiminty N., Tummala R., Lou W. et al. MicroRNA let-7c is downregulated in prostate cancer and suppresses prostate cancer growth // PLoS One. 2012. V. 7. № 3. e32832. https://doi.org/10.1371/journal.pone.0032832
Denduluri S.K., Idowu O., Wang Z. et al. Insulin-like growth factor (IGF) signaling in tumorigenesis and the development of cancer drug resistance // Genes Dis. 2015. V. 2. № 1. P. 13–25. https://doi.org/10.1016/j.gendis.2014.10.004
Alimirah F., Chen J., Basrawala Z. et al. DU-145 and PC-3 human prostate cancer cell lines express androgen receptor: implications for the androgen receptor functions and regulation // FEBS Lett. 2006. V. 580. № 9. P. 2294–2300. https://doi.org/10.1016/j.febslet.2006.03.041
Ferraldeschi R., Welti J., Luo J. et al. Targeting the androgen receptor pathway in castration-resistant prostate cancer: progresses and prospects // Oncogene. 2015. V. 34. № 14. P. 1745–1757. https://doi.org/10.1038/onc.2014.115
Edwards J., Bartlett J. The androgen receptor and signal-transduction pathways in hormone-refractory prostate cancer. Pt 2: Androgen-receptor cofactors and bypass pathways // BJU Int. 2005. V. 95. № 9. P. 1327–1335. https://doi.org/10.1111/j.1464-410X.2005.05527.x
Дополнительные материалы отсутствуют.


