Генетика, 2019, T. 55, № 7, стр. 798-810
Тандемные повторы в геноме свиньи Sus scrofa, их локализация в хромосомах и ядрах клеток сперматогенного ряда
Н. Г. Иванова 1, *, В. Н. Стефанова 1, Д. И. Остромышенский 1, О. И. Подгорная 1, 2, 3
1 Институт цитологии Российской академии наук
194064 Санкт-Петербург, Россия
2 Санкт-Петербургский государственный университет, кафедра цитологии и гистологии
199034 Санкт-Петербург, Россия
3 Дальневосточный федеральный университет, Школа биомедицины
690922 Владивосток, Россия
* E-mail: Nadyaxs@gmail.com
Поступила в редакцию 18.11.2018
После доработки 16.01.2019
Принята к публикации 30.01.2019
Аннотация
Методы биоинформатики позволяют найти и аннотировать большие тандемные повторы (ТП) в сборках геномов разных организмов. В настоящей работе анализировали геномную сборку свиньи Sus scrofa (в базе данных NCBI Sscrofa10.2) и выявили набор ТП, характерных для вида, а также картировали некоторые из них методом FISH на метафазных хромосомах с помощью рассчитанных нами коротких олигонуклеотидных зондов (Sscrf_335A, Sscrf_243A, Sscrf_15A, Sscrf_50A). Все ТП, присутствующие в базе данных Repbase для свиньи, выявляются с помощью биоинформатических методов, но кроме них найдено еще 18 новых семейств ТП в сборке Sscrofa10.2. Набор найденных ТП в сборке Sscrofa10.2 по сравнению с человеком и мышью отличается бóльшей длиной мономеров. Зонды Sscrf_15А и Sscrf_243А выявляют набор акроцентрических (Ас) хромосом, зонд Sscrf_50А – набор суб/метацентрических (Мс) хромосом, зонд Sscrf_335А метит все хромосомы. Использовали зонды для того, чтобы проследить за укладкой центромерного района при сперматогенезе. При использовании всех зондов в профазе I мейоза наблюдали образования в виде колец, которые не описаны в литературе. Мы показали следующее: 1) сконструированные нами зонды выявляют Мс- и Ас-наборы хромосом; 2) в сперматозоидах существует характерный паттерн для каждого зонда; 3) вероятно, центромерные зонды дают возможность наблюдать сборку центромерного района.
Развитие методов секвенирования и сборки геномов, а также развитие методов обработки данных высокопроизводительного секвенирования позволяют найти и аннотировать большие тандемные повторы (ТП), которые также называют сателлитной ДНК (сатДНК), в сборках геномов различных организмов. Особенности организации строения ТП затрудняют сборку, аннотирование и картирование гетерохроматиновых районов хромосом, которые содержат ТП как основной компонент [1]. Ранее в нашей лаборатории был разработан биоинформатический метод, позволяющий найти ТП в геномных сборках [2]. Термин “большие тандемные повторы” (ТП), который мы используем в настоящей работе, поддается формализации, так как все характеристики ТП имеют численное выражение. Исторически сложилось, что для некоторых последовательностей ТП используют термин “сателлитная ДНК” (SSAT), например, SSAT_A и SSAT_B свиньи в Repbase.
Поиски цитогенетических маркеров центромерных районов хромосом свиньи ведутся давно. Разрешение, получаемое методом FISH, не дает возможности определить точную локализацию зонда в области первичной перетяжки – центромерную или прицентромерную. Поэтому локализацию большинства зондов описывают как центромерную в широком смысле, т.е. в области первичной перетяжки хромосомы без уточнения позиции.
Для классификации хромосом в цитогенетических исследованиях используют дифференциальное окрашивание, хромосом-специфичные зонды, полученные с помощью микродиссекции или цитофлуорометрической сортировки хромосом и последующей ПЦР-амплификации. Хромосом-специфичные зонды расположены в сперматозоидах свиньи неслучайным образом [3]. Однако при таком подходе не выявляются гетерохроматиновые участки, определяющие пространственную организацию хромосом в ядрах, кроме того, не видны хромосомные ассоциации в интерфазном ядре (степень близости хромосом в ядре сперматозоида). Ставя задачу настоящей работы, мы предполагали, что предложенные нами и используемые впервые зонды позволят прояснить вопрос о положении гетерохроматиновых районов хромосом в сперматогенезе.
В сперматозоидах млекопитающих ДНК достигает максимальной степени компактизации в почти кристаллическую структуру [4, 5]. Хромосомы упакованы в тороиды, которые, плотно прилегая, располагаются друг над другом [6]. Хромосомные территории организованы неслучайно и в отличие от соматических клеток здесь хромосомы свернуты так, что их плечи p- и q- соприкасаются. Их теломеры тяготеют к периферии, а центромеры агрегируют в один или несколько хромоцентров ближе к центру ядра [7, 8]. Сперматозоидам свиньи свойственна определенная форма, напоминающая весло с плоской головкой высотой около 3 мкм и вытянутой вдоль на 12 мкм, которая отличает их от яйцевидной формы сперматозоидов человека или сперматозоидов мыши, похожих на крюк. Расположение хромосом в сперматозоидах разной формы важно для последующего развития. В сперматозоидах свиньи теломеры локализованы в центре ядра как несколько конгломератов, центромеры Ас- и Мс-хромосом формируют отдельные кластеры [9]. Эти данные были получены с использованием синтетических олигонуклеотидов, рассчитанных на основании клонированных и тестированных ТП свиньи [9].
В настоящей работе мы провели анализ геномной сборки Sscrofa10.2 свиньи S. scrofa и выявили набор ТП, характерных для этого вида, часть из которых тестировали методами цитогенетики. Мы поставили следующие задачи: 1) биоинформатическими методами выявить семейства ТП в геномной сборке Sscrofa10.2; 2) подобрать к ним зонды; 3) сравнить полученные семейства ТП с ранее известными из литературных данных; 4) определить позиции зондов в клетках сперматогенного ряда. Для решения последней задачи мы использовали FISH на препаратах, не обработанных специально для сохранения 3D-структуры. В работе Фостер с соавт. [3] была показана возможность исследования преимущественной локализации хромосом в ядрах сперматозоидов, не обработанных специальным образом для сохранения 3D-структуры. ДНК сперматозоидов и сперматид упакованы настолько плотно, что полностью распластанные ядра получить невозможно. Можно только разрыхлить хроматин для того, чтобы разобщить и разрешить сближенные FISH-сигналы. При конфокальной микроскопии таких ядер мы получили несколько серийных оптических срезов, из которых представлена максимально информативная проекция. Задача построения 3D-модели расположения ТП в нашей работе не ставилась. В будущем мы планируем провести 3D FISH для создания более полной картины распределения ТП в клетках сперматогенного ряда.
МАТЕРИАЛЫ И МЕТОДЫ
Поиск больших тандемных повторов в геномных сборках основывался на методике, предложенной в работе Комиссарова с соавт. [2]. ТП искали в геномной сборке свиньи S. scrofa породы Дюрок Sscrofa10.2, скаченной с сайта www.ncbi.nlm.nih.gov. Для поиска ТП использовали программу TRF (Tandem repeats finder, [10]). Параметры работы программы аналогичны тем, которые описаны в работе [2]: “mismatch” (вес несовпадения) равен 5; максимальная длина мономера равна 2 тпн, остальные значения выставлены по умолчанию. Для обработки результатов работы TRF использовали оригинальную программу, написанную на языке Python (http://github.com/DmitriiOstr/ tandem-repeat-family-finder). Программа была отредактирована, но логика программы осталась прежней и была проверена по сходимости данных.
Из результатов работы TRF удаляли вложенные повторы, а среди повторов, имеющих одинаковые координаты, оставляли только повтор с наименьшей длиной мономера. Затем оставшиеся повторы фильтровали для удаления микро- и минисателлитов, а также повторов со слишком коротким полем. Кандидатов в большие ТП определяли по следующим параметрам (критериям): длина мономера больше 4 пн, длина поля больше 2 тпн, содержание GC – от 20 до 80%, информационная энтропия поля больше 1.76.
Для поиска семейств ТП сравнивали поля ТП друг с другом, используя программу BLASTN со значением параметра E-value 10e-16 и параметра DUST = “no”. Затем строили граф, в котором вершинами служили поля ТП. Для полученного графа искали компоненты связности. Поля ТП, соответствующие вершинам в каждом компоненте связности графа, считали принадлежащими к одному семейству ТП. Поля с комбинированными мономерами, которые объединяли неродственные семейства, были изъяты из анализа.
Для поиска совпадений с известными повторами использовали программу BLASTN со значением E-value 10e-10 и DUST = “no”. Для поиска совпадений с диспергированными повторами сравнивали поля найденных семейств ТП с повторами китообразных и парнокопытных из базы данных Repbase версии 21.01.
Для оценки содержания ТП в геноме свиньи использовали программу Bowtie2 с параметром чувствительности “local”. С помощью этой программы исходные риды чтения генома выравнивали на все поля ТП, принадлежащих к одному семейству. Доля ридов (%), выравненных на поля ТП, служила оценкой содержания в геноме семейства ТП. Выравнивания проводили на риды из рана ERR173170_1, хранящиеся в архиве: http://www.ebi.ac.uk/ena/data/view/ERX149133. Качество чтения последовательностей в каждом файле проверено программой FASTQC (http:// www.bioinformatics.babraham.ac.uk/projects/fastqc/) и при необходимости последовательности прочтений (ридов) обрезаны с помощью программы Trimmomatic (http://www.usadellab.org/cms/ ?page=trimmomatic).
Номенклатура тандемных повторов. Устоявшейся номенклатуры для ТП не существует, поэтому при выборе названия для новых семейств ТП мы использовали схему, предложенную ранее [2]. Название семейства ТП состоит из двух частей: в первой указывается аббревиатура видового названия животного, в геноме которого обнаружено семейство ТП, во второй части цифра указывает на размер минимального мономера (пн) среди полей ТП семейства, а следующая за ней буква используется для дифференциации семейств ТП с одинаковой длиной мономера. Видовое название не приводится в таблицах и подписях под рисунками.
Расчет коротких олигонуклеотидных зондов для метода FISH. Для FISH подобрали короткие одноцепочечные олигонуклеотидные зонды к каждому из выбранных семейств ТП. Подбор осуществляли собственной программой на языке Python со следующим алгоритмом: 1) все поля, принадлежащие к семейству ТП, выбрали из полногеномной сборки; 2) для полей рассчитывали частоты всех возможных К-меров с длиной слова (К), равной 12 [11]; 3) для создания пробы брали наиболее часто встречающиеся К-меры; 4) для подбора оптимальных праймеров увеличивали длину до 20–25 пн. Контроль качества нуклеотидов (например, отсутствие вторичной структуры) осуществляли программой Primer3. Зонды подбирали для максимально гомогенного поля, чтобы на начальном этапе охватить все возможные поля ТП. Названия зондов соответствуют названию семейств ТП. Видовое название не приводится в таблицах и подписях под рисунками.
Получение препаратов метафазных пластинок хромосом из лимфоцитов периферической крови и препаратов из семенников хряков. Материалом для исследования служили клетки крови, полученной от свиней Sus scrofa domesticus из свинокомплексов Ленинградской области, а также семенники, полученные при кастрации хряков породы Ландрас. Эксперименты проведены в соответствии с Гарантийным регистрационным номером F18-00380 Института цитологии РАН (период действия 12.10.2017–31.10.2022) по защите животных, которых выращивают на экспериментальных фермах и используют в научных целях. Препараты метафазных хромосом для анализа получены из лейкоцитов периферической крови по стандартному протоколу, разработанному для хромосом человека [12].
Гомогенат семенников центрифугировали и отмывали фосфатным буфером, выдерживали в гипотоническом растворе (0.075 М KCl) 20 мин и фиксировали осадок клеток смесью метанол–уксусная кислота в соотношении 3 : 1.
Флуоресцентная гибридизация in situ (FISH). Для гибридизации использованы синтезированные олигонуклеотиды (Синтол, Бигль, Россия), модифицированные по 5'- и 3'-концам биотином. Для флуоресцентной гибридизации in situ использовали метод, разработанный для хромосом свиньи [13]. Гибридизацию проводили во влажной камере при 37°С в течение 16–18 ч. Для детекции биотина использовали стрептавидин, конъюгированный с Alexa 546 (ThermoFisher Scientific, USA), для усиления сигнала – биотинилированный антистрептавидин (Vector Laboratories, USA), затем снова стрептавидин, конъюгированный с Alexa 546; все концентрации соответствовали прописи производителя. В заключение наносили раствор антифейда Prolong Gold с флуорохромом DAPI (ThermoFisher Scientific, USA). Последовательности олигонуклеотидных зондов, использованных для FISH-анализа, представлены в табл. 1.
Таблица 1.
Последовательности зондов, использованные для постановки FISH
| Зонд | Соответствует в Repbase | Последовательность |
|---|---|---|
| 15A | SSRS2 | TGCAGTGGATTTTATGCAGC |
| 335A | SSAT | GCAAGCGTTGCTTTCCTGA |
| 50A | SAT-1_SSc | TCGACCAGCTGTCCCGGCC |
| 243A | AGACTGCAGGTGCTCCAGATGAGCC |
Препараты просматривали с помощью лазерного сканирующего конфокального микроскопа LEICA TCS SP5 (ИНЦ РАН) или LEICA DM4000B (ЦКП “Хромас”).
FISH на клетках сперматогенного ряда. Эксперименты проводили как описано в работе [14]. Ключевыми моментами являются обработка 0.2%-ным Tween-20 в PBS в течение 5 мин и выдерживание клеток на стеклах 30 мин в растворе гепарина (0.1 мг/мл), содержащем 1 мМ DTT в PBS, которые делают компактный хроматин клеток, главным образом сперматозоидов, доступным для зонда.
РЕЗУЛЬТАТЫ
Характеристика ТП in silico и их картирование на метафазных хромосомах
Для поиска ТП использовали сборку генома свиньи S. scrofa (Sscrofa10.2). Значение метрики N50 контигов показывает наибольшую длину контига N, такую, что сумма длин контигов ≥N дает не менее 50% длины сборки. Величина метрики N50 и ее применимость для оценки качества сборки зависят в первую очередь от размера генома, и для геномов близких размеров значение N50 вполне может использоваться для сравнения качества сборок [15]. В базе данных значение N50 для контигов Sscrofa10.2 составляет 69 474 пн. Эта же метрика N50 для сборок генома человека и мыши на порядок выше. Сборка Sscrofa10.2 является наиболее законченной сборкой по сравнению с другими сборками свиньи. В табл. 2 приведены обнаруженные ТП в убывающем порядке по значению содержания ТП в геноме.
Таблица 2.
Семейства ТП в геномной сборке Sscrofa10.2
| Семейство ТП | Содержание в геноме, % | Количество полей, шт. | GC% | Repbase | Расположение на хромосомах in silico | Расположение in situ |
|---|---|---|---|---|---|---|
| 15A | 0.56625 | 37 | 44 | SSRS2 | 12, 16, 6, 3 | Цен Ac |
| 335A | 0.06963 | 69 | 47 | SSAT | 1, 2, 3, 4, 6, 7, 9, 13, 17, 18 | Цен Мс, Х |
| 50A | 0.02912 | 23 | 65 | SAT-1_SSc | 1, 2, 4, 5, 8, 9, 11 | Цен Мс, Х |
| 1985A | 0.01702 | 2 | 43 | SSRS1 | 18 | |
| 50B | 0.00309 | 1 | 55 | MER41B_SS-LTR | 1 | |
| 84A | 0.00244 | 4 | 41 | 1, 2, 8, 14 | ||
| 190A | 0.00157 | 3 | 59 | L1_SS | 2, 5 | |
| 1559A | 0.00101 | 2 | 50 | X | ||
| 23A | 0.00088 | 2 | 49 | 3 | ||
| 768A | 0.00067 | 2 | 49 | 2 | ||
| 39A | 0.00041 | 2 | 50 | 4 | Мс | |
| 120A | 0.00033 | 1 | 63 | 7 | ||
| 234A | 0.00024 | 1 | 50 | 12 | ||
| 63B | 0.00022 | 2 | 56 | 14 | ||
| 853A | 0.00019 | 2 | 64 | 9 | Цен Ас | |
| 366A | 0.00015 | 1 | 58 | 14 | ||
| 291A | 0.00007 | 1 | 55 | 4 | ||
| 63A | 0.00006 | 2 | 52 | 6 | ||
| 99A | 0.00005 | 1 | 56 | 2 | ||
| 37A | 0.00003 | 1 | 42 | 5 | ||
| 456A | 0.00003 | 1 | 58 | 13 | ||
| 818A | 0.00002 | 1 | 54 | 2 | ||
| 243A | 0.00002 | 2 | 59 | 5 | Цен Ас | |
| 1311A | 0.00002 | 2 | 59 | 15 | ||
| 128A | 0.00001 | 1 | 51 | 6 | ||
| 29A | 0.00001 | 1 | 52 | 8 | ||
| 30A | 0.00001 | 1 | 52 | 18 | ||
| 327A | 0.00001 | 1 | 73 | 4 | ||
| 32A | 0.00001 | 2 | 43 | 1 | ||
| 96A | 0.00001 | 2 | 61 | 18 | ||
| 82A | 0 | 1 | 52 | 7 | ||
| 28A | 0 | 1 | 56 | 10 | ||
| 141A | 0 | 1 | 49 | 6 | ||
| 38A | 0 | 1 | 64 | MSAT-1_SSc | 11 | |
| 288A | 0 | 1 | 34 | 9 | ||
| 27A | 0 | 1 | 74 | 13 | ||
| 65A | 0 | 1 | 43 | 5 |
Примечание. GC% – усредненное содержание GC в ТП в процентах; сходство ТП по Repbase; позиции ТП на хромосомах, предсказанные in silico и выявленные in situ. Ас – акроцентрические хромосомы, Мс – метацентрические хромосомы, Цен – центромера. Полужирным выделены ТП, для которых проведена FISH на метафазных пластинах.
Хромосомный набор у свиньи состоит из 18 пар аутосом (12 пар Мс-хромосом и 6 пар Ас-хромосом) и двух половых хромосом (Х и Y) [16]. Окрашивание флуорохромами показало, что центромеры всех Ас-хромосом ярко окрашиваются DAPI (обогащены АТ-парами оснований ДНК), а Мс-хромосомы окрашиваются хромомицином А3 (обогащены ГЦ-парами ДНК) [17]. Молекулярно-цитогенетическими методами на Мс-хромосомах (1–12) и Х-хромосоме было выявлено Мс1-семейство сатДНК, а на Ас-хромосомах (13–18) – семейство Ас2 [18]. Эти описанные два мажорных семейства сатДНК были обнаружены после рестрикции тотальной ДНК и последующего клонирования и картирования фрагментов Мс1 и Ас2 [18]. Mc1 соответствует SSAT_A и SSAT_B по базе Repbase и Sscrf_335A по табл. 2, в которой названия ТП приведены в соответствии с нашей номенклатурой. Ac2 соответствует SSRS2 по Repbase и Sscrf_15А по табл. 2. Найденное ранее подсемейство Мс2 соответствует Ssat-1_Ssc, которое находится в геномной сборке на хромосомах 1, 2, 4, 5, 8, 9, 11; соответствует Sscrf_50A – в табл. 2. Клонированный повтор MSAT-1_SSc считают специфичным для хромосомы 11 [19] (Sscrf_38A в табл. 2). В литературе для Мс-хромосом описаны еще несколько семейств сатДНК [20, 21]. Праймеры на основе Мс1 и Ас2 использовали для мечения центромер соответствующих групп хромосом in situ методом PRINS [22]. Эти сатДНК заложены в базу данных повторов Repbase [21, 23]. Из космиды был клонирован фрагмент размером ~3 тпн, который оказался специфичным для хромосомы 9 [21]. Хромосомная специфичность достигается за счет повторов высшего порядка (HOR, high order repeats) и зонд должен быть длинным. Для мажорных ТП неудивительно наличие HOR и соответственно хромосом-специфичных вариантов [2].
Все ТП у свиньи, присутствующие в Repbase, выявляются методом, который используется в нашей лаборатории, но кроме них найдено еще 30 новых семейств ТП в сборке Sscrofa10.2. Таким образом, найдены отсутствующие в Repbase новые ТП, которые не описаны и не картированы. По набору найденных ТП свинья отличается от человека и мыши бóльшей длиной мономеров [24].
Самым представленным повтором в геномной сборке Sscrofa10.2 является семейство Sscrf_15A. Его значительно больше (~0.6%), чем остальных семейств ТП. Это семейство ТП имеет высокую степень сходства с клонированным прицентромерным повтором SSRS2. Следующее семейство по представленности в ридах – семейство ТП Sscrf_335A (69 полей в сборке Sscrofa10.2). Это семейство обладает сходством с клонированным ранее центромерным повтором SSAT [21]. Клонированный повтор локализовали на центромере хромосомы 9, что предполагает его хромосомную специфичность за счет HOR-структуры [21], однако in silico мы нашли его на бóльшем числе хромосом (табл. 2).
Из вновь выявленных повторов мы выбрали повтор Sscrf_243А, относительно богатый по содержанию GC (59%), не имеющий сходства с диспергированными повторами, но с возможной хромосомной специфичностью, определенной in silico на 5-й хромосоме.
Подсемейство Mc2 (SAT-1_SSc по Repbase и Sscrf_50A по табл. 2) было определено как специфичное для хромосомы 11 и дающее более слабый сигнал в центромерных районах других Мс-хромосом [19]. Поиск по геномной сборке in silico показал, что семейство Sscrf_50A присутствует на Мс-хромосомах 1, 2, 4, 5, 8, 9 и 11, хотя не на всех. In silico это семейство не обнаружено на хромосомах 3, 6, 7, 10 и 12. Семейство Sscrf_50A богато содержанием GC (65%). Это семейство имеет относительно высокое содержание в геноме (табл. 2). Для 11-й хромосомы in silico найдено специфичное для нее семейство ТП (Sscrf_38A), которое имеет одно поле. Это семейство присутствует в базе данных Repbase и обозначено MSAT-1_SSc (табл. 2).
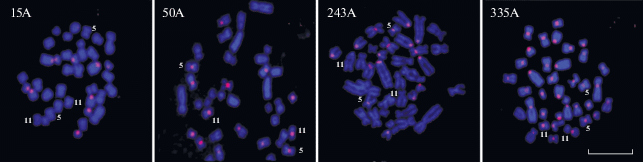
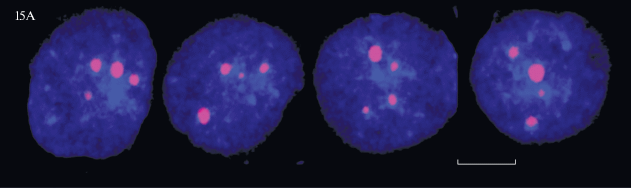
In silico обнаружено много (31) семейств ТП, присутствующих только на одной хромосоме (табл. 2). Семейств ТП, локализующихся исключительно на одной хромосоме in silico, нет всего у четырех хромосом – у двух маленьких Ас-хромосом (16 и 17), Y-хромосомы и хромосомы 3. На Y‑хромосоме не удалось обнаружить ни одного семейства больших ТП. Данные FISH на метафазных хромосомах показывают, что из использованных зондов, обнаруженных в геномной сборке на одной или нескольких хромосомах, два (Sscrf_15A и Sscrf_243A) гибридизовались в центромерном районе Ас-хромосом, один (Sscrf_335А) – на всех хромосомах и один (Sscrf_50A) – на Мс-хромосомах (рис. 1). Таким образом, данные in silico недостаточны для предсказания позиций зонда и обязательно нуждаются в экспериментальной проверке.
Рис. 1.
Гибридизация зондов (приведены на каждой панели) на метафазных пластинках лимфоцитов свиньи. Зонд 15A дает сигналы на Ас-хромосомах, зонд 335A дает сигналы на всех хромосомах, хотя и с разной интенсивностью. 50A дает сигналы на Мс-хромосомах; зонд 243A дает сильные сигналы на Ас-хромосомах, есть более слабые сигналы на некоторых Мс-хромосомах, например на 5-й. Отмечены хромосомы 5 и 11. Масштабный отрезок 10 мкм.
Центромерный ТП для свиньи не известен. В процессе анализа мы нашли повтор Sscrf_1985A, хорошо представленный в сборке и сходный с SSRS1 (табл. 2). Повтор SSRS1 по классификации Repbase не относится к сатДНК (ТП), но специфичен для Y-хромосомы. Повтор SSRS1 длиной 3832 пн выравнивается практически по всему полю повтора Sscrf_1985A (на хромосоме 18 поле этого ТП составляет 13517 пн, а на некартированном скэффолде – 9695 пн) с невысокой степенью идентичности (около 67%). Вероятно, несмотря на известную уникальную структуру ТП половых хромосом [25], этот ТП ошибочно принимали за повтор, специфичный для Y-хромосомы. Sscrf_1985A может оказаться вариантом центромерного ТП, специфичного для Y-хромосомы, подобно Y-хромосомному варианту минорного центромерного сателлита мыши [26]. До экспериментальной проверки предположения мы считаем использованные в настоящей работе зонды для ТП прицентромерными.
ТП в клетках сперматогенного ряда
Для гибридизации с ДНК клеток сперматогенного ряда мы использовали четыре зонда (Sscrf_15A, Sscrf_335A, Sscrf_50A, Sscrf_243A). Теломерный зонд дает 6—16 сигналов на ядро, что говорит об ассоциации хромосом в сперматозоидах [9]. Зонд Sscrf_335А (для всех хромосом) дает тот же порядок числа сигналов. В предельном случае они слиты вместе в центре ядра в хромоцентр, но чаще распадаются на 6–10 отдельных сигналов. Группа сигналов регулярно находится в нижней части ядра (рис. 2, зонд Sscrf_335А, ряд sp). Число сигналов в сперматозоидах зависит от природы зонда, специфичного для определенного подтипа хромосом. Ас-центромеры сливаются преимущественно в один или два хромоцентра (рис. 2, зонды Sscrf_15А и Sscrf_243А, ряд sp), тогда как Мс-центромеры ассоциируют в меньшие и более многочисленные кластеры (рис. 2, зонд Sscrf_50А, ряд sp).
Рис. 2.
Гибридизация (FISH) клеток сперматогенного ряда с зондами ТП, указанными на каждой панели. SI – ядра сперматоцитов 1-го порядка (профаза 1-го деления мейоза), SII – ядра сперматоцитов 2-го порядка, sp – сперматозоиды. Масштабный отрезок для всех изображений 10 мкм. ДНК окрашена DAPI (синий), зонд красный.
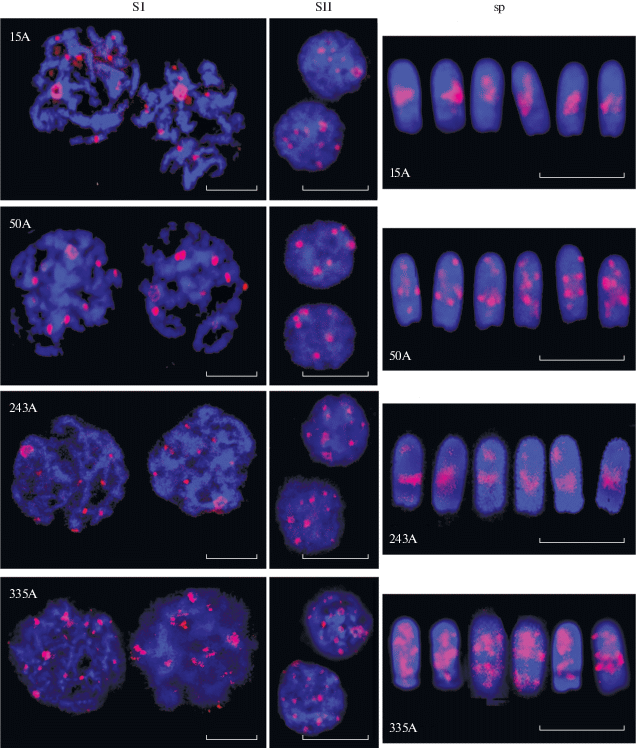
Зонды Sscrf_15А и Sscrf_243А (Ас) дают сходную картину – обширный сигнал на середине ядра (рис. 2, зонды Sscrf_15А и Sscrf_243А, ряд sp). Второй сигнал в верхней части ядра при использовании зонда Sscrf_243А, видимо, принадлежит Мс-хромосомам, которые выявляются этим зондом на метафазных пластинках (рис. 1, зонд Sscrf_243А). Если на метафазной пластинке зонд Sscrf_50А выявляет семь пар Мс-хромосом, то в сперматозоидах – четыре ярких сигнала в области экватора и два сигнала в верхней и нижней частях ядра, т.е. в среднем по одному сигналу на одну центромеру. Результаты FISH в целом совпадают с данными, опубликованными другими авторами [9].
Стадии сперматогенеза в процессе мейотического деления использовали, чтобы проследить, как образуется укладка хроматина сперматозоида (рис. 3, зонд Sscrf_335А). Сигнал зондов часто наблюдали в виде кольца, особенно у сперматоцитов I порядка. Кольцевые структуры наблюдали со всеми использованными зондами (рис. 2, Sscrf_15А, Sscrf_50А и Sscrf_243А; рис. 3). Кольца расположены в районах, сравнительно слабо окрашенных DAPI. Кольца могут быть результатом объединения центромер разных хромосом, а могут принадлежать каждой отдельной хромосоме. Чтобы проверить эти предположения, мы учитывали число сигналов в сперматоцитах и сперматозоидах по отношению к числу сигналов на метафазных пластинках (табл. 3). Число сигналов варьирует в зависимости от эффективности гибридизации, и слабые сигналы проявляются не регулярно, а общий результат числа сигналов не дает возможности отдать полное предпочтение какой-либо из двух гипотез, однако не исключает возможности того, что мы наблюдаем именно сборку отдельных центромер (табл. 3, рис. 4).
Рис. 3.
Центромеры на разных стадиях укладки, гибридизация с зондом 335А. SI – сперматоциты 1-го порядка, SII – сперматоциты 2-го порядка, sp – сперматозоиды. 1 – профаза 1-го митоза, пахитена; 2 – профаза 2-го мейоза; 3 – ряд сперматид; 4 – сперматозоиды. На панели 2 и 3 приведено черно-белое изображение хроматина ядер (окраска DAPI). Зонд к Sscrf_335A.
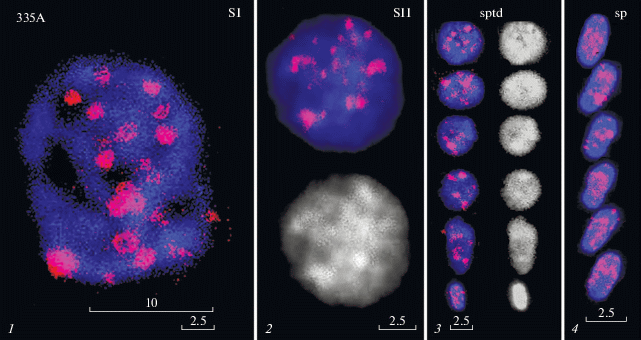
Таблица 3.
Среднее число сигналов для метафаз, клеток сперматогенного ряда и сперматозоидов
| Зонд | Среднее число сигналов | |||
|---|---|---|---|---|
| метафазы (М) |
сперматоциты I порядка | сперматозоиды (C) | отношение С/М |
|
| 15А | 10.8 | 8.1 | 2.4 | 0.22 |
| 50А | 14.0 | 7.7 | 6.3 | 0.45 |
| 243А | 18.0 | 9.0 | 3.9 | 0.21 |
| 335А | 34.9 | 16.3 | 8.1 | 0.23 |
Рис. 4.
Кольцевые структуры, характерные для клеток SI сперматогенного ряда. Зонды указаны на каждой панели. Масштабный отрезок 1 мкм.
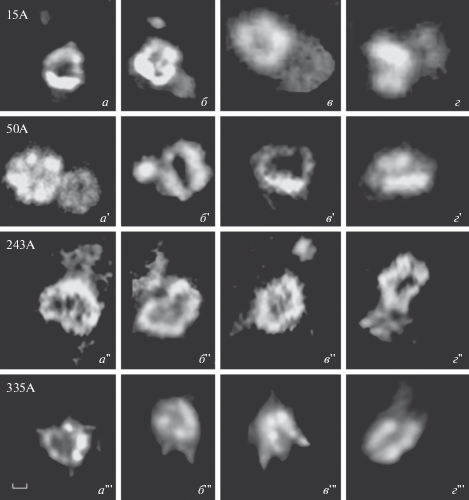
Число сигналов в сперматоцитах 1-го порядка в среднем в 2 раза меньше, чем таковое для метафазных пластинок. Сигналы Ас-хромосом (зонды Sscrf_15А и Sscrf_243А) проявляют большую тенденцию к слиянию, чем сигналы Мс-хромосом (зонд Sscrf_50А). Можно думать, что в профазе 1-го деления мейоза (2n) мы наблюдаем сигналы отдельных хромосом (рис. 2, ряд SI), а кольцо отражает образование центромеры одной хромосомы.
Центромеры в виде двух параллельных линий (рис. 4,в''', г', г''') наблюдали при иммунофлуоресценции [27], однако изображений кольцевых образований на мейотических хромосомах нам не удалось найти в доступной литературе. Двойные кольца (рис. 4,в, а', а'') мы наблюдали чаще на стадии пахитены. Стадию пахитены определяли по морфологическим критериям состояния хроматина, когда уже произошло сближение гомологов [28]. Полагаем, что наш набор зондов, расположенных в прицентромерных районах, позволяет проследить как образование отдельных центромер, так и объединение гомологичных бивалентов.
ОБСУЖДЕНИЕ
Два семейства прицентромерных сателлитных повторов Мс1 и Ас2 свиньи, которые ранее описаны как специфичные для Мс- и Ас-хромосом соответственно [18], при анализе геномной сборки Sscrofa10.2 биоинформатическими методами оказались менее строго распределенными по подгруппам хромосом. Для семейства ТП Sscrf_335А (гомологичное Мс1) in silico находятся поля, локализованные не только на Мс-хромосомах, но также на Ас-хромосомах 13, 17 и 18 (табл. 2). Данные о том, что это семейство повторов характерно только для Мс-хромосом, получены цитологическим методом [18], в то время как анализ in silico более чувствителен. Однако известно, что экспериментально клонированные повторы семейства Мс1 (Sscrf_335А) при мягких условиях гибридизации находили на всех хромосомах, кроме Y‑хромосомы [20]. Семейство Sscrf_15A, которое гомологично Ас2, т.е. клонировано как семейство, характерное для Ас-хромосом, in silico обнаруживается только на одной Ас-хромосоме (16) и еще на трех Мс-хромосомах – 3, 6 и 12 (табл. 2). Это может объясняться тем, что, во-первых, возможны ошибки сборки; во-вторых, эти семейства могут быть еще более вариабельными, чем предполагалось ранее [18], и иметь дивергировавшие повторы как на Ас-, так и на Мс-хромосомах.
Наличие семейств Мс1 на Ас-хромосомах и Ас2 на Мс-хромосомах согласуется с гипотезой о недавнем происхождении Ас-хромосом свиньи [29]. Методы биоинформатики, разрешение которых выше, чем у метода FISH, свидетельствуют о расселении ТП по разным типам хромосом.
Зонды Sscrf_335A и Sscrf_15A, рассчитанные по найденным in silico ТП, оказались похожими на использованные в других исследованиях [9, 20, 22]. Наши олигонуклеотидные зонды подобраны к максимально часто встречающимся К-мерам этих семейств и, соответственно, чаще встречаются в сборке Sscrofa10.2, чем зонды, использованные в цитированных работах (Sscrf_335А/SSCSR2A (SSAT_A) соответствует 591/380; Sscrf_15A/Ac6 (SSRS2) соответствует 6391/4851). FISH с зондами, сконструированными на основании данных, полученных in silico, в основном согласуются с данными из литературы [9].
Организация хромосом при сперматогенезе
Организация хроматина в сперматозоидах кардинально отличается от соматических клеток, хотя слияние центромер хромосом в хромоцентрах можно наблюдать и в специализированных клетках (рис. 5). Литературные данные работ, сделанных на сперматозоидах разных видов, свидетельствуют о том, что хромосомные территории различимы в сперматозоидах и организованы неслучайным образом [4, 8]. Достигнут значительный прогресс в понимании важности организации ядра сперматозоида; известно, что нарушение этой организации ведет к нарушениям развития зиготы и снижает фертильность. В отношении клеток человека (как максимально изученного вида) пришли к выводу, что организация хромосомных территорий в лимфоцитах и сперматозоидах сходна и зависит от величины хромосомы и плотности генов в ней [30].
Рис. 5.
Объединение центромер Ас-хромосом в интерфазных специализированных клетках (клетках Сертоли). В присутствии сигнала на семи парах хромосом на метафазной пластине всегда выделяются четыре хромоцентра, но не во всех хромоцентрах присутствует сигнал. Клетки Сертоли определены по состоянию хроматина (окраска DAPI), характерного для активно синтезирующих клеток.
В сперматозоидах быка самая большая (1-я) и самая маленькая (29-я) хромосомы тяготеют к противоположным концам ядра: хромосома 1 преимущественно локализована в акросомной части, тогда как хромосома 29 находится в базальной части ядра [8]. В сперматозоидах свиньи наблюдали кластеризацию центромер в зависимости от типа хромосом [9]. Нет корреляции между величиной хромосомы и ее позицией в ядре: маленькие хромосомы 11, 18 и Y находят как у края, так и внутри ядра сперматозоида, большие хромосомы 1, 6 и 13 тяготеют к внутренней части ядра [3]. На основе литературных данных можно сделать вывод, что длинные хромосомы вытянуты по длинной оси ядра – показано для хромосомы 1 [31] и хромосом 14, 15 [32–34]. Тенденцию к распределению хромосом вдоль длинной оси наблюдали в работе [35]: Мс-хромосомы 11 и 10 обнаружили ближе к верхней части ядра, а Ас-хромосому 18 и Мс‑хромосому 1 – ближе к хвостовой части. Согласно работе [36], Ас-хромосома 13 занимает промежуточное положение, ближе к хвостовой части. Эти работы [35, 36] выполнены с использованием в качестве зондов бактериальных искусственных хромосом (ВАС) или хромосомных красок (отдельных сортированных хромосом, меченых с помощью DOP-праймера). Такие зонды, в отличие от использованных в настоящей работе, далеко не всегда гибридизуются с прицентромерным районом [37, 38].
Использование зондов ТП позволяет следить именно за центромерными районами и их перемещением при сперматогенезе. По литературным данным центромеры Ас-хромосом кластеризуются в один или два хромоцентра, а центромеры Мс-хромосом – в более многочисленные, но и более мелкие хромоцентры [9]. Зонд, выявляющий центромеры всех хромосом в настоящей работе (Sscrf_335А), используется впервые (рис. 2). Он позволяет видеть, как центромерные районы распределяются по длине сперматозоида (рис. 3). Картина соответствует схеме, предложенной в работе [39] для сперматозоидов человека, где подчеркивается внутреннее, но не центральное, расположение центромер без тотального слияния в хромоцентр в отличие от ранее предложенной схемы [4, 40].
Зонд Sscrf_50А позволяет видеть, что при кластеризации Мс-хромосом в ядрах сперматозоидов видны шесть сигналов, что соответствует попарной кластеризации (рис. 1, рис. 2). Зонд Sscrf_243А дает два дополнительных сигнала по сравнению с зондом Sscrf_15А, дающим в среднем два сигнала. Вероятно, дополнительные сигналы от зонда Sscrf_243А по сравнению с зондом Sscrf_15А связаны с Мс-хромосомами, которые также содержат этот ТП (рис. 1). Хромосомы 5 и 11 занимают максимально периферическое положение в ядре сперматозоида у свиньи [3]. На метафазных пластинках (рис. 1) они определены по сходству с таковыми стандартного кариотипа свиньи [41]. Именно хромосома 5 (Мс) метится зондом Sscrf_243А дополнительно к набору Ас-хромосом (рис. 1). Мы планируем проверить это предположение с помощью зондов, специфичных для хромосомы 5. Зонды, полученные на основании методов биоинформатики и проверенные в настоящей работе, при гибридизации в основном подтверждают литературные данные и пригодны для дальнейшей работы.
Организация центромерного района
Не вызывает сомнения, что в основе укладки хромосом, и соответственно центромер, лежит петлевая организация [4, 28]. Прицентромерные ТП организованы в большие поля, которые, например, у мыши и человека составляют до 10% генома. Известны транскрипты ТП, обнаруженные в раннем эмбриональном развитии разных организмов [42, 43] и в клетках опухолей [1]. Для клеток подавляющего большинства высших эукариот известны лишь несколько мажорных ТП. Даже бурное развитие техник секвенирования и сборки геномов не дало алгоритмов сборки ТП. В сборках геномов большинства высших эукариот в центромерной (прицентромерной) области находится незаполненный промежуток — Golden Path Gap (GPG). Геном свиньи не является исключением, поэтому невозможно оценить истинный размер полей найденных ТП. Максимально длинное поле ТП, найденное в геномной сборке Sscrofa10.2, составляет 47 744 пн для семейства Sscrf_15А, что практически соответствует экспериментальной оценке величины петель ДНК сперматозоида – 20–50 тпн [7]. Петли в районе центромеры могут быть меньше [44] и соответствовать величине даже меньшим полям ТП. Петли, выступающие за ось хромосом с торца хромосомы, выглядят как розетка [45]. Можно предполагать, что именно такие поля, организованные в виде розетки петель, мы наблюдаем в процессе организации мейотических хромосом. Это предположение может быть проверено при использовании в качестве зонда ТП строго для одной хромосомы; такие ТП по нашим предварительным данным обнаружены у хомяков. У мыши ассоциацию как бивалентов, так и отдельных хромосом наблюдали в районе гетерохроматина [46]. Мы предполагаем использовать метод иммуноFISH, т.е. антитела против элементов синаптонемного комплекса, и зонды к ТП для того, чтобы проверить, справедливо ли это в случае двойных колец (рис. 4, Sscrf_15А, в, и зонд Sscrf_50A, а').
Некартированные на хромосомах и неаннотированные поля ТП присутствуют в контигах баз данных. Использование методов биоинформатики позволило нам выявить набор ТП в геноме свиньи домашней S. scrofa (объекта, важного для народного хозяйства) и определить специфичность изготовленных зондов. Мы показали следующее: 1) короткие олигонуклеотидные зонды выявляют Мс- и Ас-наборы хромосом; 2) в сперматозоидах существует характерный паттерн для каждого зонда; 3) центромерные зонды вероятно, дают возможность наблюдать сборку центромерного района.
Авторы признательны А.Ф. Сайфитдиновой и И.Л. Трофимовой за плодотворную дискуссию, И.А. Гамалей за бесценную помощь в редактировании текста статьи, а также сотрудникам ЦКП “Хромас” (С.-Петербургский университет) за методическую помощь.
Работа выполнена при финансовой поддержке РФФИ (18-04-00238-мол_а), гранта Президиума РАН “Молекулярная и клеточная биология” и РНФ 19-74-20102.
Авторы заявляют, что у них нет конфликта интересов.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Список литературы
Подгорная О.И., Остромышенский Д.И., Енукашвили Н.И. Кому он нужен, этот мусор, или темная материя генома // Биохимия. 2018. Т. 83. № 4. С. 610–628.
Komissarov A.S., Gavrilova E.V., Demin S.J. et al. Tandemly repeated DNA families in the mouse genome // BMC Genomics. 2011. V. 12. № 531. P. 1–21.
Foster H.A., Abeydeera L.R., Griffin D.K., Bridger J.M. Non-random chromosome positioning in mammalian sperm nuclei, with migration of the sex chromosomes during late spermatogenesis // J. Cell Sci. 2005. V. 116. P. 1811–1820. https://doi.org/10.1242/jcs.02301
Zalensky A., Zalenskaya I. Organization of chromosomes in spermatozoa: an additional layer of epigenetic information? // Biochem. Soc. Transact. 2007. V. 35. № 3. P. 609–611.
Ward W.S. Organization of sperm DNA by the nuclear matrix // Am. J. Clin. Exptl Urol. 2018. V. 6(2). P. 87–92.
Govin J., Escoffier E., Rousseaux S. et al. Pericentric heterochromatin reprogramming by new histone variants during mouse spermiogenesis // J. Cell Biol. 2007. V. 176. P. 283–294. https://doi.org/10.1083/jcb.200604141
Ward W.S. Function of sperm chromatin structural elements in fertilization// Mol. Human Reprod. 2010. V. 16. № 1. P. 30–36. https://doi.org/10.1093/molehr/gap080
Chagin V., Zalensky A., Nazarov I., Mudrak O. Preferable location of chromosomes 1, 29, and X in bovine spermatozoa // AIMS Genet. 2018. V. 5(2). P. 113–123. https://doi.org/10.3934/genet.2018.2.113
Acloque H., Bonnet-Garnier A., Mompart F. et al. Sperm nuclear architecture is locally modified in presence of a robertsonian translocation t(13;17) // PLoS One. 2013. V. 8. I. 10. P. 1–12. https://doi.org/10.1371/journal.pone.0078005
Benson G. Tandem repeats finder: a program to analyze DNA sequences // Nucl. Acids Res. 1999. V. 27. № 2. P. 573.
Rizk G., Lavenier D., Chikhi R. DSK: k-mer counting with very low memory usage // Bioinformatics. 2013. V. 29. № 5. P. 652–653.
Moorhead P.S., Nowell P.C., Mellman W.J. et al. Chromosome preparations of leukocytes cultured from human peripheral blood // Exptl Cell Res. 1960. V. 20. P. 613–616.
Chowdhary B.P., de la Sena C., Harbitz I. et al. FISH on metaphase and interphase chromosomes demonstrates the physical order of the genes for GPI, CRC, and LIPE in pigs // Cytogenet. Cell Genet. 1995. V. 71. № 2. P. 175–178.
Zalenskaya I., Zalensky A. Non-random positioning of chromosomes in human sperm nuclei // Chromosome Res. 2004. V. 12(2). P. 163–173.
Earl D., Bradnam K., John J.S. et al. A competitive assessment of de novo short read assembly methods // Genome Research. 2011. V. 21. № 12. P. 2224–2241. doi/https://doi.org/10.1101/gr.126599.111
Gustavsson I. Standard karyotype of the domestic pig // Hereditas. 1988. V. 109. P. 151–157.
Schnedl W., Abraham R., Forster M., Schweizer D. Differential fluorescent staining of porcine heterochromatin by chromomycin A3/distamycin A/DAPI and D 287/170 // Cytogenet. Cell Genet. 1981. V. 31(4). P. 249–253.
Jantsch M., Hamilton B., Mayr B., Schweizer D. Meiotic chromosome behaviour reflects levels of sequence divergence in Sus scrofa domestica satellite DNA // Chromosoma. 1990. V. 99. P. 330–335.
Riquet J., Mulsant P., Yerle M. et al. Sequence analysis and genetic mapping of porcine chromosome 11 centromeric S0048 marker // Cytogenet. Cell. Genet. 1996. V. 74. P. 127–132.
Miller J.R., Hindkjaer J., Thomsen P.D. A chromosomal basis for the differential organization of a porcine centromere-specific repeat // Cytogenet. Cell. Genet. 1993. V. 62. P. 37–41.
Janzen M.A., Buoen L.B., Zhao F., Louis C.F. Characterization of a swine chromosome-specific centromeric higher-order repeat // Mammalian Genome. 1999. V. 10. P. 579–584.
Rogel-Gaillard C., Bourgeaux N., Save J.C. et al. Construction of a swine YAC library allowing an efficient recovery of unique and centromeric repeat sequences // Mammalian Genome. 1997. V. 8. P. 186–192.
Akamatsu M., Chen Z., Dziuk P.J., McGraw R.A. A highly repeated sequence in the domestic pig: A gernder-neutral probe // Nucl. Acids Res. 1989. V. 17. № 23. P. 10120.
Ostromyshenskii D.I., Chernyaeva E.N., Kuznetsova I.S., Podgornaya O.I. Mouse chromocenters DNA content: sequencing and in silico analysis // BMC Genomics. 2018. V. 19. P. 159. doi.org/https://doi.org/10.1186/s12864-018-4534-z
O’Neill M.J., O’Neill R.J. Sex chromosome repeats tip the balance towards speciation // Mol. Ecol. 2018. P. 1–16. https://doi.org/10.1111/mec.14577
Pertile M.D., Graham A.N., Choo K.H., Kalitsis P. Rapid evolution of mouse Y centromere repest DNA belies recent sequence stability // Genome Res. 2009. V. 19. № 12. P. 2202–2213.
Qiao H., Chen J.K., Reynolds A. et al. Interplay between synaptonemal complex, homologous recombination, and centromeres during mammalian meiosis // PLoS Genet. 2012. V. 8. № 6. P. 1–17. https://doi.org/10.1371/journal.pgen.1002790
Muller H., Scolari V.F., Agier N. et al. Characterizing meiotic chromosomes’ structure and pairing using a designer sequence optimized for Hi-C // Mol. Systems Biol. 2018. V. 14. P. 1–19. https://doi.org/10.15252/msb.20188293
Tortereau F., Servin B., Frantz L. et al. A high density recombination map of the pig reveals a correlation between sex-specific recombination and GC content // BMC Genomics. 2012. V. 13. № 586. P. 1–12.
Manvelyan M., Hunstig F., Bhatt S. et al. Chromosome distribution in human sperm – a 3D multicolor banding-study // Mol. Cytogenet. 2008. № 1. P. 25. https://doi.org/10.1186/1755-8166-1-25
Sembon S., Fuchimoto D., Iwamoto M. et al. A simple method for producing tetraploid porcine parthenogenetic embryos // Theriogenology. 2011. V. 76. P. 598–606. https://doi.org/10.1016/j.theriogenology.2011.03.010
Orsztynowicz M., Pawlak P., Ole D. et al. Low incidence of chromosome aberrations in spermatozoa of fertile boars // Original Res. 2011. V. 11. № 3. P. 224–235.
Massip K., Berland H., Bonnet N. et al. Study of inter- and intra-individual variation of meiotic segregation patterns in t(3;15)(q27;q13) boars // Theriogenology. 2008. V. 70. P. 655–661. https://doi.org/10.1016/j.theriogenology.2008.04.026
Massip K., Bonnet N., Calgaro A. et al. Male meiotic segregation analyses of peri- and paracentric inversions in the pig species // Cytogenet. Genome Res. 2009. V. 125. P. 117–124. https://doi.org/10.1159/000227836
Bonnet-Garnier A., Guardia S., Pinton A. et al. Analysis using sperm-FISH of a putative interchromosomal effect in boars carrying reciprocal translocations // Cytogenet. Genome Res. 2009. V. 126. P. 194–201. https://doi.org/10.1159/000245920
Barasc H., Ferchaud S., Mary N. et al. Cytogenetic analysis of somatic and germinal cells from 38,XX/38,XY phenotypically normal boars // Theriogenology. 2013. V. XXX. P. 1–5. https://doi.org/10.1016/j.theriogenology.2013.10.006
Telenius H., Carter N.P., Bebb C.E. et al. Degenerate oligonucleotide-primed PCR: General amplification of target DNA by a single degenerate primer // Genomics. 1992. V. 13. P. 718–725.
Kuznetsova I.S., Ostromyshenskii D.I., Komissarov A.S. et al. LINE related component of mouse heterochromatin and complex chromocenters’ composition // Chromosome Res. 2016. V. 24(3). P. 309–323.
Ioannou D., Millan N.M., Jordan E., Tempest H.G. A new model of sperm nuclear architecture following assessment of the organization of centromeres and telomeres in three-dimensions// Nature Scientific Reports. 2017. V. 7. № 41585. P. 1–14. https://doi.org/10.1038/ srep41585
Zalensky A.O., Allen M.J., Kobayashi A. et al. Well-defined genome architecture in the human sperm nucleus // Chromosoma. 1995. V. 103. P. 577–590.
Hansen-Melander E., Melander Y. The karyotype of the pig // Hereditas. 1974. V. 77. P. 149–158.
Enukashvily N.I., Malashicheva A.B., Waisertreiger I.S.-R. Satellite DNA spatial localization and transcriptional activity in mouse embryonic E-14 and IOUD2 stem cells // Cytogenet Genome Res. 2009. V. 124. P. 277–287. doi .https://doi.org/10.1159/000218132
Probst A.V., Okamoto I., Casanova M. et al. A strand-specific burst in transcription of pericentric satellites is required for chromocenter formation and early mouse development // Dev. Cell. 2010. V. 19. P. 625–638. https://doi.org/10.1016/j.devcel.2010.09.002
Kuznetsova I., Podgornaya O., Ferguson-Smith M.A. High-resolution organization of mouse centromeric and pericentromeric DNA // Cytogenet. Genome Res. 2006. V. 112. P. 248–255. https://doi.org/10.1159/000089878
Kalitsis P., Zhang T., Marshall K.M. et al. Condensin, master organizer of the genome // Chromosome Res. 2017. P. 1–16. https://doi.org/10.1007/s10577-017-9553-0
Berríos S., Manterola M., Prieto Z. et al. Model of chromosome associations in Mus domesticus spermatocytes // Biol. Res. 2010. V. 43. P. 275–295.
Дополнительные материалы отсутствуют.


