Генетика, 2019, T. 55, № 7, стр. 788-797
leidyi – новая группа DD41D-транспозонов в геноме мнемиопсиса Mnemiopsis leidyi
М. В. Пузаков 1, *, Л. В. Пузакова 1
1 Институт морских биологических исследований
им. А.О. Ковалевского Российской академии наук
299011 Севастополь, Россия
* E-mail: puzakov@ngs.ru
Поступила в редакцию 23.10.2018
После доработки 26.12.2018
Принята к публикации 22.01.2019
Аннотация
Суперсемейство мобильных генетических элементов (МГЭ) IS630/Tc1/mariner является одним из наиболее многочисленных и широко распространенных среди ДНК-транспозонов. МГЭ IS630/Tc1/mariner делятся на восемь семейств: IS630, Tc1, mariner, pogo, maT, mosquito, plants и rosa. Представители семейства rosa (DD41D-транспозоны) были выделены в отдельную группу сравнительно недавно и на данный момент подробно охарактеризованы только у насекомых. Известно, что семейство rosa включает в себя подсемейства Crmar-like и LTIR. В настоящей работе впервые описаны DD41D-транспозоны гребневиков Mnemiopsis leidyi и Pleurobrachia bachei. Установлено, что элементы мнемиопсиса представляют новую, неизвестную ранее группу, которая была названа нами leidyi. Представители этого подсемейства сочетают в себе признаки, присущие двум другим подсемействам DD41D-транспозонов. У P. bachei выявлены элементы rosa, принадлежащие подсемейству LTIR и имеющие более протяженные DDE/D-домены.
Мобильными генетическими элементами (МГЭ) являются последовательности ДНК, способные интегрироваться в новые участки генома внутри клетки хозяина. Их активность может приводить к изменениям в первичной структуре кодирующих или регуляторных последовательностей и, как следствие, приводить к увеличению спектра генетического разнообразия. Следовательно, МГЭ могут играть значительную роль в адаптации организмов к окружающей среде и эволюции генома [1–3]. Классификация МГЭ основана на структурно-функциональных различиях. Все описанные к настоящему времени мобильные генетические элементы поделены на два класса: ретротранспозоны (класс I) и ДНК-транспозоны (класс II) [4].
Одно из наиболее многочисленных и широко распространенных суперсемейств ДНК-транспозонов – IS630/Tc1/mariner. Протяженность МГЭ IS630/Tc1/mariner обычно составляет от 1 до 3 тыс. пар нуклеотидов (пн). Последовательность элементов фланкирована концевыми инвертированными повторами (TIR) длиной от 20 до 1900 пн [5, 6]. Кроме того, у некоторых ДНК-транспозонов IS630/Tc1/mariner присутствуют предконцевые инвертированные повторы (SIR) длиной от 175 до 1403 пн [6]. В центральной части элементов расположена открытая рамка считывания (ОРС), которая кодирует фермент транспозазу протяженностью в среднем 350 аминокислотных остатков (а.о.). Данный фермент обладает как ДНК-связывающей, так и эндонуклеазной активностью, благодаря которой он осуществляет инсерции элемента в характерный для данного суперсемейства дуплицируемый сайт встраивания ТА [7–9].
Несмотря на то что элементы суперсемейства IS630/Tc1/mariner обнаружены практически во всех геномах эукариот [10, 11], только незначительное их количество имеют функциональную транспозазу и обладают транспозиционной активностью [12–15]. Это явление связывают с тем, что пик активности преобладающей доли ДНК-транспозонов IS630/Tc1/mariner был далеко в прошлом и их “жизненный цикл” пришел к завершению [16]. Поэтому у большинства МГЭ последовательности, кодирующие транспозазу, нарушены делециями, сдвигами рамки считывания и аминокислотными заменами.
На данный момент описаны восемь семейств МГЭ, принадлежащих к суперсемейству IS630/Tc1/mariner: DDxE (IS630), DD34E (Tc1), DD34D (mariner), DDхD (pogo), DD37D (maT), DD37E (mosquito), DD39D (plants) и DD41D (rosa) [17–21].
Первые представители семейства rosa (DD41D-транспозоны) были описаны почти два десятка лет назад [21], но их биология и эволюция до недавнего времени оставались в значительной степени неизвестными. При исследовании DD41D-транспозонов, до настоящего времени выявленных только в геномах насекомых, было обнаружено, что эта группа МГЭ включает в себя два подсемейства [6, 22]. Первое подсемейство (Crmar-like) объединяет элементы, характеризующиеся относительно невысокой протяженностью (~1300 пн) и длиной TIR от 15 до 45 пн. Элементы второго подсемейства (LTIR) имеют бóльшую протяженность (~1500–4400 пн) и TIR до 1900 пн. У некоторых представителей LTIR были обнаружены SIR [6, 22].
У гребневиков (Ctenofora) представленность и эволюция элементов IS630/Tc1/mariner (в частности, семейства rosa) на данный момент не исследованы [23]. Актуальность изучения МГЭ у гребневиков обусловлена еще и удивительной приспособляемостью, свойственной некоторым представителям этого таксона. Так, в начале 1980-х гг. мнемиопсис (Mnemiopsis leidyi A. Agassiz, 1865) стал стабильным компонентом экосистемы Черного моря, а затем распространился в Азовское, Мраморное, Эгейское моря, Восточное Средиземноморье и, наконец, Каспийское море [24]. Столь успешная адаптация мнемиопсиса к новым условиям обитания вызывает закономерный вопрос о его эволюционной пластичности и роли МГЭ в формировании этой пластичности.
В настоящей работе исследовались представленность и эволюционная история DD41D-транспозонов мнемиопсиса, а также DD41D-подобных транспозонов другого гребневика – Pleurobrachia bachei A. Agassiz, 1860.
МАТЕРИАЛЫ И МЕТОДЫ
Поиск DD41D-транспозонов
Для поиска ДНК-транспозонов с каталитическим доменом DD41D был использован tBLASTn со стандартными настройками [25]. В качестве матриц для поиска были взяты аминокислотные последовательности, кодируемые ОРС элементов Crmar2.5 (AAK61417) и Apismar5.1 [22]. Нуклеотидные последовательности геномной ДНК гребневиков Mnemiopsis leidyi (MneLei_Aug2011, GCA_000226015) и Pleurobrachia bachei (P.bachei_draft_genome_v.1.1, GCA_000695325) были взяты из базы данных NCBI (http://www.ncbi.nlm.nih.gov/). Первый геном составляет 155.9 млн пн, объединенных в 5100 скаффолдов; второй состоит из 156.1 млн пн, организованных в 21 979 скаффолдах [26, 27]. Для того чтобы определить полные нуклеотидные последовательности каждого элемента, фрагменты, обладающие наибольшей гомологией к аминокислотным последовательностям транспозаз Crmar2.5 и Apismar5.1, были извлечены из соответствующих скаффолдов вместе с фланкирующими участками протяженностью 5000 пн. В каждой извлеченной последовательности был осуществлен поиск инвертированных повторов (TIR и SIR) с помощью BLASTn [28]. Каждая полная последовательность нового выявленного элемента затем использовалась для определения количества присутствующих в геноме копий и уточнения границ элемента. Последовательности протяженностью менее 10% от длины полноразмерного МГЭ исключались из анализа. Консенсусные последовательности были получены с использованием правила относительного большинства.
Анализ последовательностей
Границы предполагаемых ОРС определяли с помощью ORF Finder (https://www.ncbi.nlm.nih.gov/ orffinder/) и далее уточняли визуально. Последовательность сигнала ядерной локализации (NLS) выявляли с помощью ScanProsite [29]. ДНК-связывающий мотив HTH определяли, используя PSIPRED v3.3 [30]. Мотив GRPR-типа, а также DDE/D-домен идентифицировали визуально.
Филогенетический анализ
Для филогенетического анализа и анализа DDE/D-доменов, помимо изучаемых последовательностей, были взяты транспозазы 29 элементов, представляющих различные группы суперсемейства IS630/Tc1/mariner, и 19 DD41D-транспозонов подсемейств Crmar-like и LTIR (табл. 1). Множественное выравнивание аминокислотных последовательностей было выполнено с помощью MUSCLE [31] с использованием стандартных настроек. Поиск наилучшей модели для филогенетического анализа и филогенетический анализ проводили с использованием программного обеспечения MEGA7.0 [32].
Таблица 1.
ДНК-транспозоны IS630/Tc1/mariner, используемые в исследовании
| Семейство | Элемент | Источник | Семейство | Элемент | Источник |
|---|---|---|---|---|---|
| mariner, DD34D |
Dmmar1 Hsmar1 Famar1 Bytmar1 Apismar1.1 |
X78906 U52077 AY155492 CAD45367 [22] |
pogo, DDxD |
PogoR11 Tigger1 Fot1 Tan1 Flipper |
S20478 U49973 Q00832 U58946 U74294 |
| Tc1, DD34E |
Impala Tc1 Bari-1 Passport Quetzal |
AF282722 X01005 X67681 CAB51371 AAB02109 |
rosa, DD41D, Crmar-like |
Crmar2.5 rosa_Ae rosa_Ap rosa_Cf rosa_Dy rosa_Rp Apismar4.1 Apismar4.2 Apismar4.3 Mariner-4_CGi Mariner-12_CGi Mariner-1_RAu |
AAK61417 [6] [6] [6] [6] [6] [22] [22] [22] Repbase Repbase Repbase |
| maT, DD37D |
Bmmar1 Bmmar6 Cemar6 CbmaT4 |
U47917 AF461149 LK928390 AC084524 |
|||
| mosquito, DD37E |
Ae-atropalpus1 An-gambiae1 PrDD37E1 |
AF377999 AF378002 DQ138288 |
|||
| plants, DD39D |
Soymar1 Br-oleracea Ca-sativa Phyllostachys edulis Pisum sativum |
AF078934 XP013589454 XP010462775 ADP24264 AAX51974 |
rosa, DD41D, LTIR |
Lsra_Ap Lsra_Bt Lsra_Cf Lsra_Hs Lsra_Nv Apismar5.1 Apismar5.2 |
[6] [6] [6] [6] [6] [22] [22] |
| IS630, DDxE |
IS630Ss IS630Se |
X05955 NP_073225 |
Выявление случаев горизонтального переноса
Консенсусные нуклеотидные последовательности DD41D-транспозонов, выявленных в настоящей работе, были использованы для идентификации возможных случаев горизонтального переноса. С помощью BLASTn осуществлен поиск по базам данных нуклеотидных (nr) и полногеномных (WGS) последовательностей, доступных в NCBI. Как было предложено ранее [33], случаи возможного горизонтального переноса рассматривались когда элементы имели более 75% идентичности к более 90% последовательности матрицы для поиска.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
DD41D-транспозоны в геноме M. leidyi
Для идентификации ДНК-транспозонов семейства rosa у мнемиопсиса в качестве матрицы для поиска были взяты аминокислотные последовательности транспозаз элемента Crmar2.5 (подсемейство Crmar-like) и элемента Apismar5.1 (подсемейство LTIR). В результате анализа геномной ДНК M. leidyi с помощью tBLASTn выявлено около ста локусов, имеющих высокую гомологию с транспозазами элементов Crmar2.5 и Apismar5.1. Выявленные нуклеотидные последовательности были фрагментами делетированных и полноразмерных копий трех DD41D-транспозонов, которые согласно номенклатуре Repbase [34] названы Mariner-2_MLe, Mariner-3_MLe и Mariner-N1_MLe (табл. 2).
Таблица 2.
ДНК-транспозоны семейства rosa в геномах гребневиков M. leidyi и P. bachei
| Элемент | Длина, пн | TIR, пн | SIR, пн | Транспозаза, а.о. | Число копий1 | DDD-домен |
|---|---|---|---|---|---|---|
| Mariner-2_MLe | 1323 | 31/31 | 15/15 | 350 | 21 (129) | D92D41D |
| Mariner-3_MLe | 1322 | 30/30 | 19/19 | 310 | 17 (143) | D92D41D |
| Mariner-N1_MLe | 940 | 26/26 | н | >209 | 0 (8) | D92D41D |
| Mariner-1_PBaс | 2110 | 87/87 | н | 426 | 1 (7) | D127D55D |
| Mariner-2_PBaс2 | 2302 | 377/366 | н | 375 | 0 (89) | D105D45D |
Для элементов Mariner-2_MLe и Mariner-3_MLe обнаружены потенциально функциональные копии, тогда как для Mariner-N1_MLe были найдены только делетированные варианты. При сравнении структур исследуемых элементов с известными DD41D-транспозонами установлено, что по длине TIR и общей протяженности они имеют сходство с элементами подсемейства Crmar-like (рис. 1). Кроме того, у элементов Mariner-2_MLe и Mariner-3_MLe были выявлены SIR, что обусловливает их сходство с МГЭ подсемейтва LTIR, в структуре которых SIR могут присутствовать [6]. У Mariner-N1_MLe SIR не были обнаружены.
Рис. 1.
Размерные характеристики DD41D-транспозонов. пн – пары нуклеотидов; а.о. – аминокислотные остатки.
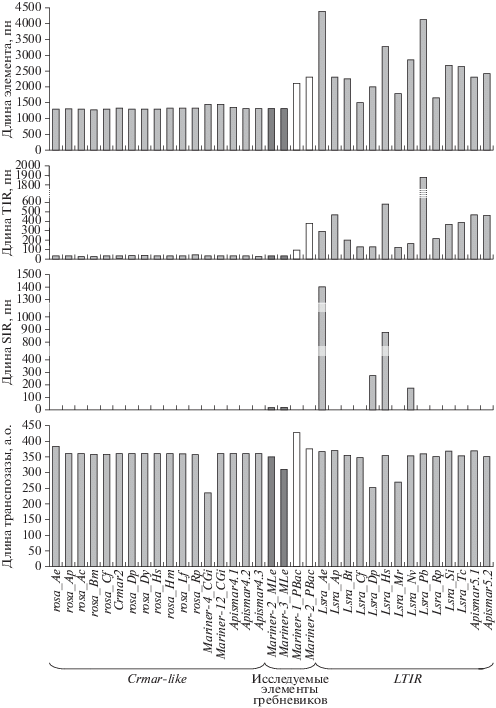
В результате анализа с помощью BLASTn обнаружено 129 копий элемента Mariner-2_MLe, 143 копии Mariner-3_MLe и восемь копий Mariner-N1_MLe (табл. 2). При этом были выявлены 21 и 17 полноразмерных вариантов элементов Mariner-2_MLe и Mariner-3_MLe соответственно. Оценка идентичности полноразмерных копий к консенсусной последовательности показала, что у транспозона Mariner-2_MLe эта характеристика варьирует от 92 до 96%. У элемента Mariner-3_MLe уровень идентичности был гораздо шире (78–96%). Здесь же следует отметить, что среди полноразмерных копий данного элемента не было ни одной с потенциально функциональной транспозазой. Это дает основания полагать, что Mariner-3_MLe – более древний элемент, чем Mariner-2_MLe, и в ходе эволюции генома мнемиопсиса подвергся вертикальной инактивации. У Mariner-N1_MLe не было выявлено ни одной полноразмерной копии. Отсутствие потенциально функциональных вариантов и малое число копий в целом свидетельствуют о том, что либо Mariner-N1_MLe является еще более древним транспозоном и большая часть его копий была элиминирована из генома, либо он подвергся ранней инактивации и не смог широко распространиться по геному.
Особенности транспозаз DD41D-транспозонов M. leidyi
Активные mariner-подобные транспозазы содержат ДНК-связывающий мотив HTH и мотив GRPR-типа (известный как AT-крюк) в N-концевой части белка, а также каталитический домен (DDE/D-домен) в С-концевой части. Кроме того, у некоторых mariner-подобных транспозаз выявляют последовательность сигнала ядерной локализации (NLS), которая, как правило, расположена в петле, разделяющей HTH и DDE/D-домен. Однако участие NLS в функциональности фермента пока не подтверждено [35].
Для анализа транспозаз Mariner-2_MLe и Mariner-3_MLe были выбраны копии с наиболее полной ОРС, без стоп-кодонов и сдвигов рамки считывания. У Mariner-N1_MLe сохранились только копии, ОРС которых содержали участок, кодирующий С-конец транспозазы с DDE/D-доменом. Анализ транспозаз Mariner-2_MLe и Mariner-3_MLe показал, что в обоих белках присутствуют мотив HTH (в позиции 70 и 30 соответственно) и АТ-крюк (в позиции 63 и 23 соответственно). Последовательность NLS нами не была выявлена ни в одной из транспозаз исследуемых элементов мнемиопсиса. В работе Буалег с соавт. [22] NLS также не были обнаружены в транспозазах элементов Crmar-like и LTIR.
Каталитический домен ДНК-транспозонов имеет три маркерных аминокислотных остатка: два аспартата (D) и третий либо глутамат (Е), либо аспартат, в связи с чем эта триада называется DDE/D-домен. Этот домен отвечает за разрезание цепей ДНК и лигирование в процессе инсерции. Для сохранения ферментативной активности необходимо наличие всех трех маркерных аминокислотных остатков, поэтому данная область обладает большей консервативностью по сравнению с N-концевой частью транспозазы [35].
При анализе DD41D-транспозонов M. leidyi каталитический домен был идентифицирован в транспозазах всех трех МГЭ, но в аминокислотной последовательности Mariner-3_MLe третий аспартат DDE/D-домена был замещен на аспарагин (N). Таким образом, только фермент, кодируемый ОРС Mariner-2_MLe, имеет НТН-мотив, АТ-крюк и полноценный DDE/D-домен, наличие которых характерно для активных транспозаз, что свидетельствует о его возможной функциональности.
leidyi – новое подсемейство DD41D-транспозонов
Различия в протяженности района транспозазы между вторым и третьим маркерными аминокислотными остатками (аспартат и глутамат/аспартат) каталитического домена являются классификационным критерием для элементов суперсемейства IS630/Tc1/mariner. По этому признаку IS630/Tc1/mariner-транспозоны подразделяются на восемь семейств: DDxE (IS630) [17], DD34E (Tc1) [18], DD34D (mariner) [18], DDхD (pogo) [19], DD37D (maT) [20], DD37E (mosquito) [20], DD39D (plants) [20] и DD41D (rosa) [21]. Такое деление подтверждается и филогенетическими исследованиями [6, 22, 36], но встречаются исключения – недавно было описано подсемейство TRT, которое филогенетически относится к транспозонам Tc1, но обладает DD37E-доменом [37]. Сравнение консервативных областей каталитичеcкого домена транспозаз 19 элементов подсемейств Crmar-like и LTIR (табл. 1) и элементов мнемиопсиса показало, что DDE/D-домены Mariner-2_MLe, Mariner-3_MLe и Mariner-N1_MLe имеют явные отличия (рис. 2). Консенсусные последовательности областей маркерных аспартатов (D) элементов M. leidyi имеют уникальные, характерные только для этих МГЭ аминокислотные остатки (на рис. 2 отмечены стрелками). Данные различия дают основания предполагать, что исследуемые транспозоны могут представлять новую, не описанную ранее группу ДНК-транспозонов с каталитическим доменом DD41D, названную нами leidyi. В пользу этой гипотезы свидетельствуют особенности структуры МГЭ M. leidyi, в соответствии с которыми их нельзя отнести ни к подсемейству Crmar-like (из-за наличия SIR), ни к LTIR (из-за коротких TIR и относительно небольшой длины МГЭ) (рис. 1).
Рис. 2.
Множественное выравнивание каталитических доменов ДНК-транспозонов rosa. Символами “/ /” указаны неотображенные на рисунке участки аминокислотных последовательностей. Черным цветом выделены маркерные аспартаты DDD-доменов; серым цветом обозначены гомологичные участки среди элементов подсемейств; стрелками указаны отличия в консенсусных последовательностях DDD-доменов элементов leidyi от Crmar-like и LTIR.

Для того чтобы проверить гипотезу о принадлежности МГЭ M. leidyi к новому подсемейству, мы провели филогенетический анализ, в который были включены ДНК-транспозоны семейства rosa как мнемиопсиса, так и описанные в работах Жанг с соавт. [6] и Буалег с соавт. [22], а также представители других семейств суперсемейства IS630/Tc1/mariner (рис. 3). Все три элемента мнемиопсиса сформировали отдельную от Crmar-like и LTIR ветвь с высоким бутстреп-значением (88). Таким образом, обнаруженные нами МГЭ в геноме M. leidyi действительно представляют новое подсемейство DD41D-транспозонов.
Рис. 3.
Филогения ДНК-транспозонов суперсемейства IS630/Tc1/mariner. Дерево было сгенерировано в MEGA7 с использованием метода максимального правдоподобия с использованием модели WAG+G+F. Указаны только бутстреп-значения выше 60%. Семейства и подсемейства указаны справа от дерева. Стрелками указаны исследуемые в данной работе DD41D-транспозоны.
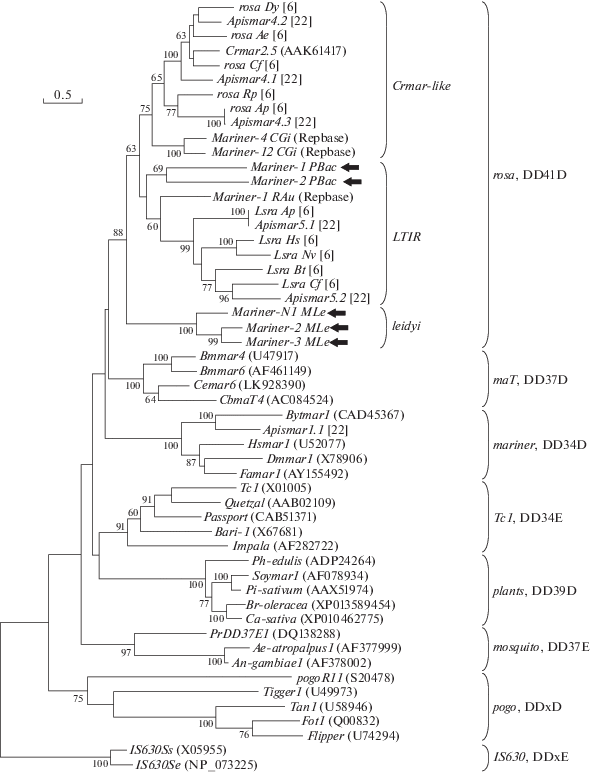
В связи с тем, что для элементов суперсемейства IS630/Tc1/mariner горизонтальный перенос – достаточно распространенное явление [38], мы предприняли поиск последовательностей, обладающих идентичностью свыше 75% к не менее чем 90% протяженности элементов Mariner-2_MLe, Mariner-3_MLe и Mariner-N1_MLe. В результате данного анализа не было установлено случаев возможного горизонтального переноса между мнемиопсисом и представителями других таксонов. И если подобные события имели место, то они были далеко в прошлом. Поскольку геномные исследования последних лет позволили установить, что гребневики – это наиболее древняя ветвь среди многоклеточных животных [26, 27, 39], то, вероятно, все три элемента являются “потомками” древнего DD41D-транспозона, проникшего в геном гребневика и эволюционировавшего вместе с ним.
DD41D-транспозоны в геноме Pleurobrachia bachei
Помимо M. leidyi полная нуклеотидная последовательность геномной ДНК была определена у P. bachei (гребневика из отряда Cydippida) [27]. Для того чтобы выяснить, ограничивается ли присутствие транспозонов подсемейства leidyi геномом M. leidyi (отрядом Lobata) или распространяется и на другой отряд гребневиков, мы провели поиск DD41D-транспозонов в геноме P. bachei. В качестве матрицы для поиска с помощью tBLASTn были взяты аминокислотные последовательности транспозаз элементов Crmar2.5 и Mariner-2_MLe. Нами найдены два МГЭ, гомологичных DD41D-транспозонам, которые были названы согласно номенклатуре Repbase [34] Mariner-1_PBaс и Mariner-2_PBaс (табл. 1). В результате анализа с помощью BLASTn обнаружено семь копий элемента Mariner-1_PBaс и среди них только одна полноразмерная. У элемента Mariner-2_PBaс выявлено 89 делетированных копий, полноразмерная копия была реконструирована нами из двух делетированных. Оба элемента имеют протяженность свыше 2000 пн и длину TIR 87 (Mariner-1_PBaс) и 377 пн (Mariner-2_PBaс), характерные для транспозонов LTIR (рис. 1). SIR обнаружены не были, что, однако, не протеворечит данным, полученным ранее, поскольку не все элементы LTIR обладают SIR [6, 22]. Транспозазы обоих элементов имели HTH-мотив, АТ-крюк и DDE/D-домен. Несмотря на высокую гомологию обоих элементов P. bachei DD41D-транспозонам, их каталитические домены имеют большую протяженность как между первым и вторым и маркерными аспартатами, так и между вторым и третьим – D127D55D у Mariner-1_PBaс и D105D45D у Mariner-2_PBaс (рис. 2). В то же время области маркерных аспартатов более всего гомологичны элементам подсемейства LTIR (рис. 2). Филогенетический анализ также показал, что оба элемента P. bachei являются представителями подсемейства LTIR (рис. 3). Таким образом, в геноме P. bachei не обнаружено МГЭ подсемейства leidyi, что может быть свидетельством ограниченного распространения данных транспозонов среди гребневиков.
Интерес представляют каталитические домены элементов P. bachei, необычные для LTIR. Поскольку и у Mariner-1_PBaс, и у Mariner-2_PBaс было обнаружено только по одной копии, содержащей DDE/D-домен, невозможно установить, является ли этот признак характерным для данных элементов или отличия в протяженности обусловлены мутационными событиями в конкретной сохранившейся копии. Подобный случай наблюдался в геноме тихоокеанской устрицы Crassostrea gigas, в котором был обнаружен элемент Mariner-21_CGi с DD83D-доменом, имеющий 80 копий, среди которых только одна полноразмерная [40].
На сегодняшний день известно достаточно большое количество генов, возникших от транспозонов, хотя биологическая роль большинства из них остается неясной [41]. Отдельные случаи “приручения” белков МГЭ геномом рассматриваются как адаптация к различным эволюционным конфликтам. Некоторые белки МГЭ были перепрофилированы как часть защитных систем, которые ограждают геном хозяина от инфекционных или инвазивных агентов, включая вирусы и сами МГЭ. Предполагается, что “приручение” белков МГЭ часто является единственным эволюционным путем к уменьшению ущерба, причиняемого транспозиционной активностью [42]. В результате “приручения” используемые геномом функциональные домены МГЭ как правило сохраняются в исходной или несколько измененной форме, тогда как остальные структуры утрачиваются [43]. На данный момент предложено несколько критериев, по которым можно предположить происхождение генов от МГЭ [10]: 1) элементы, которые были “приручены”, существуют в геномах как одиночные копии; 2) в структуре таких генов отсутствуют молекулярные особенности ДНК-транспозонов, такие как инвертированные повторы или дуплицируемые сайты встраивания; 3) ортологи обнаруживаются у далеких видов. У многоклеточных уже известны три независимых случая “приручения” pogo-подобных транспозаз [44]. Mariner-1_PBaс и Mariner-2_PBaс соответствуют первому из предложенных критериев, поэтому можно предположить, что эти МГЭ не просто подверглись инактивации и элиминации, но вовлечены в генетические процессы хозяина, что, возможно, объясняет и наличие более протяженных каталитических доменов.
В настоящей работе впервые описаны DD41D-транспозоны гребневика M. leidyi. Было установлено, что данные МГЭ представляют новую, неизвестную ранее кладу элементов суперсемейства IS630/Tc1/mariner, которая была названа нами leidyi. Представители этого подсемейства сочетают в себе признаки, присущие для двух других подсемейств DD41D-транспозонов – Crmar-like и LTIR. Подсемейство leidyi не является характерным для всех гребневиков, что было показано при исследовании генома P. bachei. Мы также идентифицировали DD41D-подобные транспозоны, принадлежащие к подсемейству LTIR, которые имеют более протяженные каталитические домены. Данные, полученные в работе, послужат основой для будущих исследований, направленных на понимание роли мобильных генетических элементов в процессах эволюции и адаптации, а также механизмов “приручения” МГЭ геномом-носителем.
Исследование ДНК-транспозонов M. leidyi выполнено при финансовой поддержке РФФИ и города Севастополь в рамках научного проекта № 18-44-920002. Изучение МГЭ P. bachei проведено в рамках государственного задания ФГБУН ИМБИ “Функциональные, метаболические и токсикологические аспекты существования гидробионтов и их популяций в биотопах с различным физико-химическим режимом”, номер гос. регистрации НИОКТР АААА-А18-118021490093-4.
Авторы заявляют, что у них нет конфликта интересов.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Список литературы
Fontdevila A. Hybrid genome evolution by transposition // Cytogenet. Genome Res. 2005. V. 110. № 1–4. P. 49–55. https://doi.org/10.1159/000084937
Васильева Л.А., Антоненко О.В., Захаров И.К. Роль мобильных генетических элементов в геноме Drosophila melanogaster // Вавилов. журн. генетики и селекции. 2011. Т. 15. № 2. С. 225–260.
Piacentini L., Fanti L., Specchia V. et al. Transposons, environmental changes, and heritable induced phenotypic variability // Chromosoma. 2014. V. 123. P. 345–354.
Kapitonov V.V., Jurka J. A universal classification of eukaryotic transposable elements implemented in Repbase // Nat. Rev. Genet. 2008. V. 9. № 5. P. 411–412. https://doi.org/10.1038/nrg2165-c1
Claudianos C., Brownlie J., Russell R. et al. maT: a clade of transposons intermediate between mariner and Tc1 // Mol. Biol. Evol. 2002. V. 19. P. 2101–2109.
Zhang H.H., Shen Y.H., Xiong X.M. et al. Identification and evolutionary history of the DD41D transposons in insects // Genes Genom. 2016. V. 38. P. 109–117. https://doi.org/10.1007/s13258-015-0356-4
van Luenen H.G., Plasterk R.H. Target site choice of the related transposable elements Tc1 and Tc3 of Caenorhabditis elegans // Nucl. Acids Res. 1994. V. 22. № 3. P. 262–269.
Lohe A., Sullivan D., Hartl D. Genetic evidence for subunit interactions in the transposase of the transposable element mariner // Genetics. 1996. V. 144. P. 1087–1095.
Pietrokovski S., Henikoff S. A helix-turn-helix DNA-binding motif predicted for transposases of DNA transposons // Mol. Gen. Genet. 1997. V. 254. P. 689–695.
Feschotte C., Pritham E.J. DNA transposons and the evolution of eukaryotic genomes // Annu. Rev. Genet. 2007. V. 41. P. 341–368.
Liu Y., Yang G. Tc1-like transposable elements in plant genomes // Mobile DNA. 2014. V. 5. P. 17.
Emmons S.W., Yesner L., Ruan K., Katzenberg D. Evidence for a transposon in Caenorhabditis elegans // Cell. 1983. V. 32. P. 55–65.
Franz G., Savakis C. Minos, a new transposable element from Drosophila hydei, is a member of the Tc1-like family of transposons // Nucl. Acids Res. 1991. V. 19. P. 6646.
Langin T., Capy P., Daboussi M.J. The transposable element impala, a fungal member of the Tc1-mariner superfamily // Mol. Gen. Genet. 1995. V. 246. P. 19–28.
Clark K.J., Carlson D.F., Leaver M.J. et al. Passport, a native Tc1 transposon from flatfish, is functionally active in vertebrate cells // Nucl. Acids Res. 2009. V. 37. P. 1239–1247.
Schaack S., Gilbert C., Feschotte C. Promiscuous DNA: horizontal transfer of transposable elements and why it matters for eukaryotic evolution // Trends Ecol. Evol. 2010. V. 25. P. 537–546.
Doak T.G., Doerder F.P., Jahn C.L., Herrick G. A proposed superfamily of transposase genes: transposon-like elements in ciliated protozoa and a common “D35E” motif // Proc. Natl Acad. Sci. USA. 1994. V. 91. № 3. P. 942–946.
Capy P., Vitalis R., Langin T. et al. Relationships between transposable elements based upon the integrase-transposase domains: is there a common ancestor? // J. Mol. Evol. 1996. V. 42. P. 359–368.
Smit A.F.A., Riggs A.D. Tiggers and other DNA transposon fossils in the human genome // Proc. Natl Acad. Sci. USA. 1996. V. 93. P. 1443–1448.
Shao H., Tu Z. Expanding the diversity of the IS630-Tc1-mariner superfamily: discovery of a unique DD37E transposon and reclassification of the DD37D and DD39D transposons // Genetics. 2001. V. 159. № 3. P. 1103–1115.
Gomulski L.M., Torti C., Bonizzoni M. et al. A new basal subfamily of mariner elements in Ceratitis rosa and other tephritid flies // J. Mol. Evol. 2001. V. 53. P. 597–606.
Bouallègue M., Filée J., Kharrat I. et al. Diversity and evolution of mariner-like elements in aphid genomes // BMC Genomics. 2017. V. 18. P. 494. https://doi.org/10.1186/s12864-017-3856-6
Пузаков М.В., Пузакова Л.В. Морские беспозвоночные как новый объект исследования транспозиционной активности мобильных генетических элементов // Успехи соврем. биологии. 2017. Т. 137. № 5. С. 450–457. https://doi.org/10.7868/S0040364417050027
Kideys A.E. Fall and rise of the Black Sea ecosystem // Science. 2002. V. 297. Iss. 5586. P. 1482–1484. https://doi.org/10.1126/science.1073002
Altschul S.F., Madden T.L., Schäffer A.A. et al. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs // Nucl. Acids Res. 1997. V. 25. P. 3389–3402.
Ryan J.F., Pang K., Schnitzler C.E. et al. The genome of the ctenophore Mnemiopsis leidyi and its implications for cell type evolution // Science. 2013. V. 342. P. 1242592. https://doi.org/10.1126/science.1242592
Moroz L.L., Kocot K.M., Citarella M.R. et al. The ctenophore genome and the evolutionary origins of neural systems // Nature. 2014. V. 510. P. 109–114. https://doi.org/10.1038/nature13400
Zhang Z., Schwartz S., Wagner L., Miller W. A greedy algorithm for aligning DNA sequences // J. Comput. Biol. 2000. V. 7. № 1–2. P. 203–214.
De Castro E., Sigrist C.J.A., Gattiker A. et al. ScanProsite: detection of PROSITE signature matches and ProRule-associated functional and structural residues in proteins // Nucl. Acids Res. 2006. V. 34. W362–W365.
Buchan D.W.A., Minneci F., Nugent T.C.O. et al. Scalable web services for the PSIPRED Protein Analysis Workbench // Nucl. Acids Res. 2013. V. 41(W1). W340–W348.
Edgar R.C. MUSCLE: multiple sequence alignment with high accuracy and high throughput // Nucl. Acids Res. 2004. V. 32. P. 1792–1797.
Kumar S., Stecher G., Tamura K. MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874.
Filée J., Rouault J.D., Harry M., Hua-Van A. Mariner transposons are sailing in the genome of the blood-sucking bug Rhodnius prolixus // BMC Genomics. 2015. V. 16. P. 1061. https://doi.org/10.1186/s12864-015-2060-9
Jurka J. Repeats in genomic DNA: mining and meaning // Curr. Opin. Struct. Biol. 1998. V. 8. P. 333–337.
Brillet B., Bigot Y., Auge-Gouillou C. Assembly of the Tc1 and mariner transposition initiation complexes depends on the origins of their transposase DNA-binding domains // Genetica. 2007. V. 130. P. 105–120.
Gao B., Chen W., Shen D. et al. Characterization of autonomous families of Tc1/mariner transposons in neoteleost genomes // Marine Genomics. 2017. V. 34. P. 67–77. https://doi.org/10.1016/j.margen.2017.05.003
Zhang H.H., Li G.Y., Xiong X.M. et al. TRT, a vertebrate and protozoan Tc1-like transposon: current activity and horizontal transfer // Genome Biol. Evol. 2016. V. 8. P. 2994–3005. https://doi.org/10.1093/gbe/evw213
Wallau G.L., Capy P., Loreto E. et al. VHICA, a new method to discriminate between vertical and horizontal transposon transfer: Application to the mariner family within Drosophila // Mol. Biol. Evol. 2016. V. 34. P. 1094–1109.
Jekely G., Paps J., Nielsen C. The phylogenetic position of ctenophores and the origin(s) of nervous systems // Evodevo. 2015. V. 6. P. 1–8. https://doi.org/10.1186/2041-9139-6-1
Puzakov M.V., Puzakova L.V., Cheresiz S.V. An analysis of IS630/Tc1/mariner transposons in the genome of a Pacific oyster, Crassostrea gigas // J. Mol. Evol. 2018. V. 86. Iss. 8. P. 566–580. https://doi.org/10.1007/s00239-018-9868-2
Sinzelle L., Izsv Z., Ivics Z. Molecular domestication of transposable elements: From detrimental parasites to useful host genes // Cell. Mol. Life Sci. 2009. V. 66. Iss. 6. P. 1073–1093. https://doi.org/10.1007/s00018-009-8376-3
Jangam D., Feschotte C., Betrán E. Transposable element domestication as an adaptation to evolutionary conflicts // Trends Genet. 2017. V. 33. Iss. 11. P. 817–831. https://doi.org/10.1016/j.tig.2017.07.011
Bourque G., Burns K.H., Gehring M. et al. Ten things you should know about transposable elements // Genome Biol. 2018. V. 19. P. 199. https://doi.org/10.1186/s13059-018-1577-z
Mateo L., González J. Pogo-like transposases have been repeatedly domesticated into CENP-B-related proteins // Genome Biol. Evol. 2014. V. 6. Iss. 8. P. 2008–2016. https://doi.org/10.1093/gbe/evu153
Дополнительные материалы отсутствуют.


