Генетика, 2019, T. 55, № 9, стр. 1031-1042
Молекулярно-генетический анализ родственных связей и происхождения малоротой корюшки (Hypomesus, Osmeridae) – нового представителя фауны Баренцева моря
Л. А. Скурихина 1, *, А. Г. Олейник 1, А. Д. Кухлевский 1, 2, А. П. Новоселов 3, 4, Д. С. Сендек 5
1 Национальный научный центр морской биологии им. А.В. Жирмунского Дальневосточного отделения
Российской академии наук
690041 Владивосток, Россия
2 Дальневосточный федеральный университет, кафедра клеточной биологии и генетики
690090 Владивосток, Россия
3 Северный филиал Полярного научно-исследовательского института рыбного хозяйства
и океанографии им. Н.М. Книповича
163002 Архангельск, Россия
4 Федеральный исследовательский центр комплексного изучения Арктики
им. академика Н.П. Лаверова Российской академии наук
163000 Архангельск, Россия
5 Государственный научно-исследовательский институт озерного
и речного рыбного хозяйства
199053 Санкт-Петербург, Россия
* E-mail: skurikhina@gmail.com
Поступила в редакцию 23.12.2018
После доработки 04.02.2019
Принята к публикации 11.02.2019
Аннотация
Исследованы родственные связи и происхождение популяции малоротых корюшек рода Hypomesus, недавно обнаруженной на значительном удалении от западной границы ареала этого рода в Баренцевом море. Проведенный анализ свидетельствует о принадлежности исследованной популяции к обыкновенной малоротой корюшке H. olidus, а также о ее филогенетической близости и общей демографической истории с H. olidus из бассейна Берингова моря. Показатели генетической дифференциации указывают на длительную изоляцию популяций, относящихся к тихоокеанскому и баренцевоморскому бассейнам. Статистические оценки исторических демографических процессов, соотнесенные с палеогеографическими данными арктических районов России, допускают вероятность выживания отдельных популяций H. olidus в ледовых рефугиумах Полярного Урала. H. olidus могла неоднократно проходить через “бутылочное горлышко” как в прошлом на рефугиальной стадии, так и в современное время из-за суровых условий обитания в арктическом регионе.
Hypomesus olidus (Pallas, 1814) обыкновенная малоротая корюшка относится к циркумполярным видам, пережившим в своем прошлом глобальные климатические изменения. Исследование таких видов может не только пролить свет на ряд аспектов их эволюции, но одновременно дать более полное представление о роли оледенений и связанных с ними изменениях гидрографического режима на обширных территориях в формировании современной генетической структуры северных морских и эстуарных видов рыб и расселении их по ареалу. В настоящее время это направление вызывает большой научный интерес [1–5 и др.]. H. olidus, которая представлена озерно-речной и проходной формами, широко распространена в прибрежьях северной части Тихого океана и арктических морей, омывающих берега России и Северной Америки [6–9]. Недавно малоротые корюшки были обнаружены за пределами западной границы ареала в юго-восточной части Баренцева моря [10]. Цель данного исследования заключается в генетическом анализе выборки малоротых корюшек из Варандейской губы Баренцева моря для установления родственных связей и происхождения популяции.
МАТЕРИАЛЫ И МЕТОДЫ
Работа основана на оригинальном материале из коллекции авторов. В анализ включены как опубликованные ранее [11], так и новые результаты. Данные о географической локализации популяций, распределении по ареалу и объеме выборок приведены в табл. 1. Для более полного представления об уровне дивергенции были использованы данные по нуклеотидным последовательностям из GenBank: H. japonicus (FJ205564/FJ010863, FJ205565/FJ010864), H. nipponensis (FJ205569/FJ010868), Osmerus dentex (KP257674/KP257752, KP257626/KP257704, KP257650/KP257728).
Таблица 1.
Данные об исследованных выборках из популяций Hypomesus olidus
| Популяция, выборка | Объем выборки | Место сбора | Широта/долгота | Гаплотипы и их количество (n) в выборках | Ссылка |
|---|---|---|---|---|---|
| 1. Варандейская губа, HolVar | 18 | Баренцево море, юго-восточная часть |
68°48′ N/58°16′ E | V01 (8), V39 (5), V40 (3), V41 (1), V42 (1) |
Новые данные |
| 2. Оз. Азабачье, HolAz | 20 | Берингово море, п-ов Камчатка, восточное побережье |
56°10′ N/161°47′ E | V01 (12), V06 (3), V10 (2), V11 (2), V17 (1) | [11] |
| 3. Р. Саранная, HolSar | 21 | Берингово море, Командорские о-ва | 55°32′ N/166°13′ E | H1 = V01 (15), H2 = V43 (6) | [36] |
| 4. Оз. Глухое, HolGlu | 11 | Охотское море, северное побережье |
59°44′ N/149°55′ E | V02 (8), V18 (1), V19 (1), V20 (1) |
[11] |
| 5. Устье р. Амур, HolAm | 5 | Японское море, северо- западная часть |
52°59′ N/141°02′ E | V14 (1), V16 (1), V36 (1), V37 (1), V38 (1) |
[11], новые данные |
| 6. Оз. Карасевое, HolKa | 14 | Охотское море, о-в Сахалин, северо- восточное побережье |
53°01′ N/143°07′ E | V04a (4), V05 (3), V09 (1), V12 (2), V21 (1), V22 (1), V23 (1), V24 (1) |
[11] |
| 7. Набильский зал., HolNab | 8 | Охотское море, о-в Сахалин, восточное побережье |
51°35′ N/143°17′ E | V04b (1), V05 (1), V25 (1), V26 (1), V27 (1), V28 (1), V29 (1), V30 (1) |
[11] |
| 8. Р. Погиби, HolPo | 15 | Японское море, о-в Сахалин, северо- западное побережье |
52°13′ N/141°39′ E | V03 (6), V05 (1), V07 (4), V09 (1), V31 (1), V32 (1), V33 (1) |
[11] |
| 9. Р. Самарга, HolSa | 7 | Японское море, западное побережье, северный район Приморского края |
59°44′ N/149°55′ E | V08 (3), V13 (2), V34 (1), V35 (1) |
[11] |
| Общее число | 119 | 43 |
Фрагменты мтДНК амплифицировали методом полимеразной цепной реакции, используя следующие праймеры: для Cytb – 5'-ACC ACC GTT GTT ATT CAA CTA CAA GAA C-3', 5'-AAC CTC CGA CAT CCG GCT TAC AAG ACC G-3' [12]; для CoI – 5'-TCA ACC AAC CAC AAA GAC ATT GGC AC-3', 5'-TAG ACT TCT GGG TGG CCA AAG AAT CA-3' [13]. Затем ампликоны очищали и секвенировали в прямом и обратном направлениях с использованием набора BigDye Terminator v. 3.1 (Applied Biosystems, США). Последовательности нуклеотидов определяли в автоматическом анализаторе ABI Prism 3500 (Applied Biosystems). Первичную обработку и множественное выравнивание нуклеотидных последовательностей проводили в программах SeqScape v. 3 (Applied Biosystems) и Geneious v. 8.1 (www.geneious.com). Новые нуклеотидные последовательности депонированы в базу данных GenBank/NCBI под номерами доступа CoI: MK038781–MK038807, Cytb: MK038813–MK038836.
Статистический анализ объединенных последовательностей, включая расчет основных показателей разнообразия выборок, p-дистанций [14], числа мигрантов Nm между популяциями [15] проводили в программе DnaSP v. 6 [16]. Степень генетической дифференциации между парами популяций определяли на основе критерия FST [17, 18], методом одноуровневой и многоуровневой AMOVA [19], используя пакет программ Arlequin vers. 3.5 [20]. Существенность иерархических компонентов дисперсии и соответствующих Φ‑статистик оценивали из непараметрического распределения псевдовероятностного теста [19].
Исторические демографические процессы тестировали по распределению нуклеотидных замен между всеми парами гаплотипов (mismatch-распределение) [21, 22] в Arlequin vers. 3.5. Соответствие между наблюдаемым частотным распределением парных различий и ожидаемым распределением при модели “взрывной” экспансии (быстрого расширения ареала) [23] определялось для каждого тестируемого набора данных из непараметрического распределения вероятностей [19]. Достоверность модели оценивалась при сравнении сумм квадратов отклонений (SSD) между наблюдаемым распределением и распределением, полученным для каждого итерационного набора данных. Кроме того, рассчитывался raggedness-индекс Харпендинга (Rag) [24], показывающий равновесие mismatch-распределения на основе различий между стабильной популяцией и популяцией, увеличивающей численность. Для индекса определялась вероятность появления более высоких величин по сравнению с наблюдаемыми значениями при нулевой гипотезе демографической или пространственной экспансии P(Rag). Для распределений, которые несущественно отличаются от модели (Р > 0.05), рассчитывали параметр времени экспансии (τ) [25]. Значения τ переводили в оценки времени экспансии через уравнение τ = 2ut, где u – скорость мутаций на поколение, t – время с момента экспансии, выраженное в поколениях. В свою очередь, u = 2μk, где μ – скорость мутации на нуклеотид, k – число нуклеотидов в анализируемых гаплотипах. При расчетах использовали значение скорости нуклеотидных замен в 1% [26] и 1.77% [27] за 1 млн лет; средний возраст поколения 3.8, вычисленный ранее для корюшковых рыб [28].
Для представления филогенетических отношений между гаплотипами использовали метод медианных сетей (алгоритм MJ) [29], реализованный в пакете DnaSP v. 6. Дополнительно были построены дендрограммы – NJ и ML, с тестированием топологии при помощи бутстрэп-анализа (1000 реплик) [30]. В качестве оптимальной на основе AIC-критерия была выбрана TrN93 + G модель нуклеотидных замещений [31]. Статистический и филогенетический анализы проводили в программе MEGA7 [32]. В качестве внешней группы использовали Osmerus dentex.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
В работе исследовано 122 особи малоротых корюшек, в том числе 119 экз. H. olidus из девяти популяций (табл. 1). Общая длина нуклеотидных последовательностей мтДНК составила 1668 пн. В объединенной последовательности (1–588 пн CoI и 589–1668 пн Cytb) у малоротых корюшек рода Hypomesus выявлены 344 вариабельных сайта (241 информативных) и 46 гаплотипов мтДНК. Для филогруппы H. olidus выявлены 57 вариабельных сайтов (35 информативных) и 43 гаплотипа.
На исследованном участке мтДНК в выборке из Варандейской губы обнаружено пять гаплотипов, четыре из которых уникальные. Особенностью данной выборки является наличие двух гаплотипов (V01, V39), встречающихся с высокой частотой. Гаплотип V01 также доминирует в выборках из оз. Азабачьего (п-ов Камчатка) и р. Саранной (Командорские о-ва). На NJ-дереве (рис. 1) все выявленные в Баренцевом море гаплотипы формируют общий кластер H. olidus с высокой степенью поддержки (100%). При этом уровень внутривидовой дивергенции мтДНК H. olidus достаточно низкий (Dxy = 0.0001 ± 0.0001–0.0024 ± 0.0010). В то же время выборка из Варандейской губы показывает одинаковые оценки дивергенции с другими выборками H. olidus относительно маркерных препаратов H. japonicus (Dxy = 0.1331 ± 0.0076), H. nipponensis (Dxy = 0.1277 ± 0.0071) и внешней группы O. dentex (Dxy = 0.1323 ± 0.0080). Полученные оценки соответствуют уровням внутри- и межвидовой изменчивости корюшковых рыб [33, 34].
Рис. 1.
Филогенетические отношения (ML-дерево) гаплотипов комбинированных нуклеотидных последовательностей генов CoI и Cytb мтДНК корюшковых рыб рода Hypomesus. Цифры в узлах соответствуют значениям бутстрэп-поддержки (BS > 80%) для ML- и NJ-деревьев соответственно. Внешняя группа – Osmerus dentex.

Для H. olidus, как и для других корюшковых рыб [34, 35], характерны высокие показатели молекулярного разнообразия мтДНК. Средние значения гаплотипического и нуклеотидного разнообразия H. olidus на исследованном ареале оказались равны 0.9004 ± 0.0225 и 0.0107 ± 0.0059 соответственно. Оценки гаплотипического разнообразия в выборке из Варандейской губы (h = = 0.7320 ± 0.0732) несколько ниже средних по ареалу, показатели нуклеотидного разнообразия достоверно ниже (π = 0.0024 ± 0.0019). Анализ объединенных выборок H. olidus свидетельствует, что показатели нуклеотидного разнообразия выборок Охотского и Японского морей на порядок выше, чем соответствующие значения для выборок Баренцева и Берингова морей (табл. 2).
Таблица 2.
Основные показатели изменчивости мтДНК Hypomesus olidus
| Популяция | N | n | Число полиморфных сайтов (S) |
Оценка различий между гаплотипами (θπ) | Гаплотипическое разнообразие (h) |
Нуклеотидное разнообразие (π) |
|---|---|---|---|---|---|---|
| 1. Баренцево море | 18 | 5 | 4 | 1.0392 ± 0.7252 | 0.7320 ± 0.0732 | 0.0024 ± 0.0019 |
| 2. Берингово море | 41 | 6 | 6 | 0.6829 ± 0.5311 | 0.5476 ± 0.0848 | 0.0016 ± 0.0014 |
| 3. Охотское море | 53 | 29 | 44 | 5.8229 ± 2.8285 | 0.9514 ± 0.0150 | 0.0138 ± 0.0075 |
| 4. Японское море | 7 | 4 | 6 | 2.7619 ± 1.6584 | 0.8095 ± 0.1298 | 0.0065 ± 0.0045 |
| По ареалу | 119 | 43 | 57 | 4.5347 ± 2.2461 | 0.9004 ± 0.0225 | 0.0107 ± 0.0059 |
Оценка подразделенности полиморфизма мтДНК с помощью критерия FST подтвердила дифференциацию между выборками H. olidus. Минимальные существенные различия наблюдаются между выборками H. olidus из Варандейской губы и оз. Азабачьего (0.1966, Р < 0.01). Попарные FST-оценки статистически значимы (Р < 0.01), за исключением нескольких пар сравнений выборок из бассейна Охотского моря, в которых отсутствие гетерогенности, вероятно, определяется ошибкой выборочности. Для объединенных выборок, представляющих бассейны морей, все попарные FST‑оценки статистически значимы (Р < 0.01) (табл. 3). Тем не менее значения параметров FST и Nm подтверждают прекращение современных генных потоков между выборками географических регионов (табл. 3).
Таблица 3.
Индексы генетической дифференциации объединенных выборок Hypomesus olidus: критерий FST [17, 18], поток мигрантов Nm [15], среднее число нуклеотидных различий на сайт между группами [14]
| Пара сравнения | FST | Nm | Dxy ± SE |
|---|---|---|---|
| Баренцево–Берингово | 0.2277*** | 1.90 | 0.0007 ± 0.0002 |
| Баренцево–Охотское | 0.3433*** | 0.68 | 0.0036 ± 0.0004 |
| Баренцево–Японское | 0.7320*** | 0.24 | 0.0035 ± 0.0009 |
| Берингово–Охотское | 0.3932*** | 0.67 | 0.0034 ± 0.0005 |
| Берингово–Японское | 0.8040*** | 0.23 | 0.0033 ± 0.0009 |
| Охотское–Японское | 0.1719** | 1.60 | 0.0034 ± 0.0005 |
Согласно AMOVA [19], для тестируемых выборок H. olidus характерны не только высокое генетическое разнообразие внутри популяций (57% дисперсии, Р < 0.001), но и высокая структурированность между популяциями (43%) (вариант I, табл. 4). В то же время объединение выборок по принадлежности к бассейнам морей существенно не изменило распределения компонентов дисперсии (вариант II, табл. 4). Разложение молекулярной изменчивости мтДНК на три иерархических уровня, согласно принадлежности к географическим регионам, подтвердило выявленные характерные особенности распределения: 1) высокая внутрипопуляционная доля дисперсии (51%) и высокое разнообразие (38%) между группами (вариант III, табл. 4). Все компоненты дисперсии статистически высоко существенны (Р < 0.001), что свидетельствует о наличии у H. olidus географической структурированности.
Таблица 4.
Иерархический анализ межпопуляционных и внутрипопуляционных гаплотипических различий Hypomesus olidus на основе комбинированных нуклеотидных последовательностей генов CoI и Cytb мтДНК
| Уровень разнообразия | d.f. | Дисперсия, % | P |
|---|---|---|---|
| Вариант I (популяции) | |||
| Между популяциями | 8 | 43.22 | <0.001 |
| Внутри популяций | 110 | 56.78 | |
| Вариант II (объединенные выборки: Баренцево, Берингово, Охотское, Японское моря) | |||
| Между группами | 3 | 39.44 | <0.001 |
| Внутри групп | 115 | 60.56 | |
| Вариант III (объединенные выборки: Баренцево, Берингово, Охотское, Японское моря) | |||
| Между группами | 3 | 38.21 | <0.002 |
| Между популяциями внутри групп | 7 | 11.14 | <0.001 |
| Внутри популяций | 133 | 50.66 | <0.001 |
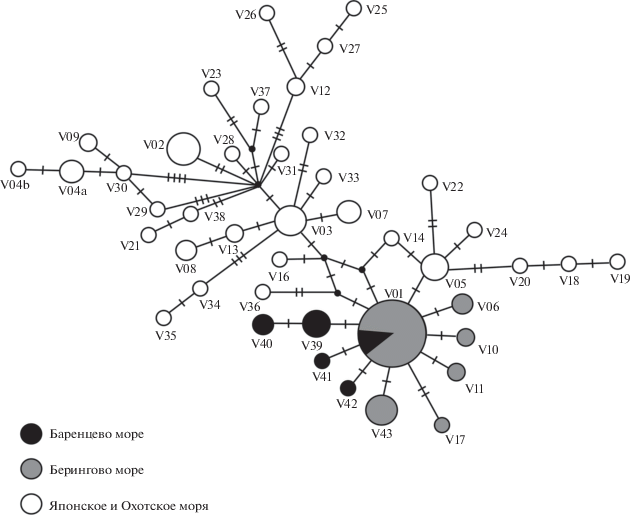
Реконструированная генеалогия гаплотипов объединенных нуклеотидных последовательностей генов CoI и Cytb H. olidus имеет радиальную структуру с тремя филогруппами (рис. 2). Особенностями MJ-сети H. olidus являются небольшая протяженность, которая определяется низким нуклеотидным разнообразием выборок, и наличие нескольких центров радиации гаплотипов. Такая генеалогия соответствует опубликованным ранее результатам [11, 36]. Внутреннее положение занимает филогруппа с центральным гаплотипом V03, объединяющая восемь гаплотипов, обнаруженных в выборках пролива Невельского и Японского моря. Обращает внимание, что все гаплотипы малоротых корюшек из Баренцева и Берингова морей формируют самостоятельный центр радиации. Внутренним гаплотипом этой филогруппы является V01, доминирующий в трех популяциях (оз. Азабачье, р. Саранная, Варандейская губа) и не встречающийся в выборках из Охотского и Японского морей. Гаплотип V01 также связан с гаплотипами из выборок северной части о. Сахалин и устья р. Амур. Гаплотипы, обнаруженные в выборках из Охотского моря, входят в разные структуры MJ-сети, генеалогические расстояния между которыми оказываются очень большими.
Рис. 2.
Генеалогическая сеть (MJ-анализ) гаплотипов комбинированных нуклеотидных последовательностей генов CoI и Cytb мтДНК Hypomesus olidus. На ветвях штрихами указано число нуклеотидных замен; размер окружностей пропорционален абсолютным частотам гаплотипов. Все мутации имеют равный вес, интервал поиска медианных векторов (черные кружки в узлах) ε равен нулю. Обозначение гаплотипов приведено согласно первому описанию [11, 36].
Распределения попарных различий [21] между 43 гаплотипами мтДНК, выявленных в суммарной выборке H. olidus, статистически существенно отклоняются от унимодального распределения (рис. 3). Присутствие нескольких пиков, разделенных значительными разрывами, указывает на высокую генетическую гетерогенность географических выборок. Подобного рода кривые распределения наблюдаются и для выборок малоротых корюшек Охотского и Японского морей (не показаны на рисунке). В то же время mismatch-распределения для объединенных выборок бассейна Баренцева и Берингова морей близки к унимодальному (рис. 3) и соответствуют модели исторической “взрывной” демографической экспансии или пространственной экспансии с высоким уровнем миграции между соседними популяциями (P(Rag) > 0.05; P(SSD) > 0.05) (табл. 5). Наличие основных пиков в области минимальных различий между сайтами свидетельствует об историческом уменьшении эффективной численности предковых популяций и/или эффекте основателя.
Рис. 3.
Распределение числа нуклеотидных замен между гаплотипами комбинированных нуклеотидных последовательностей генов CoI и Cytb мтДНК Hypomesus olidus в выборках: суммарно по ареалу (а), Баренцева моря (б), Берингова моря (в), в объединенной выборке из Баренцева и Берингова морей (г). По оси абсцисс – число попарных различий. Сплошная линия – наблюдаемая частота попарных различий, пунктирная линия – теоретически ожидаемая частота [21–23].
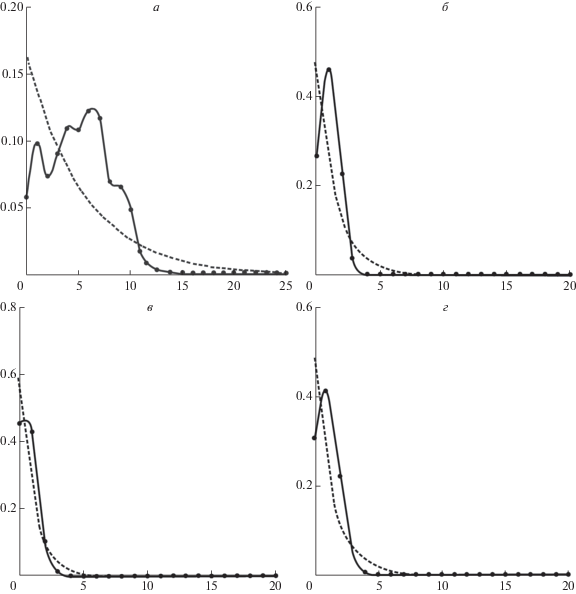
Таблица 5.
Параметры mismatch-распределения для объединенных выборок Hypomesus olidus
| Группа | Тестирование модели демографической экcпансии [23] | |||||||
|---|---|---|---|---|---|---|---|---|
| τ | θ0 | θ1 | t1 | t2 | P(SSD) | Rag | P(Rag) | |
| 1. Баренцево море | 1.0 | 0 | 3407 | 59 370 | 34 000 | 0.2235 | 0.1313 | 0.2147 |
| 2. Берингово море | 1.0 | 0 | 3407 | 59 370 | 34 000 | 0.4177 | 0.1138 | 0.3855 |
| 3. Охотское море | 6.6 | 0.16 | 25.84 | 391 880 | 224 000 | 0.5210 | 0.0139 | 0.4625 |
| 4. Японское море | 6.4 | 0 | 4.36 | 380 000 | 217 100 | 0.2659 | 0.1904 | 0.4948 |
| Общее по ареалу | 6.6 | 0.02 | 12.56 | 391 870 | 224 000 | 0.9355 | 0.0069 | 0.9593 |
| Группа | Тестирование модели пространственной экcпансии [22] | |||||||
| τ | θ0 | М | t1 | t2 | P(SSD) | Rag | P(Rag) | |
| 1. Баренцево море | 0.7 | 0.71 | 5099 | 41 560 | 23 750 | 0.07165 | 0.1312 | 0.25076 |
| 2. Берингово море | 0.7 | 0.01 | 3655 | 53 440 | 30 540 | 0.09218 | 0.1138 | 0.3855 |
| 3. Охотское море | 5.0 | 1.38 | 18 | 296 880 | 169 640 | 0.68324 | 0.01398 | 0.79776 |
| 4. Японское море | 4.4 | 2.15 | 1 | 261 250 | 149 300 | 0.19971 | 0.19048 | 0.69424 |
| Общее по ареалу | 3.5 | 2.75 | 11 | 207 800 | 118 750 | 0.64547 | 0.00689 | 0.98100 |
Примечание. τ – возраст экспансии; θ0, θ1 – мутационные параметры на начальном и конечном этапах роста численности популяции; P(SSD) – вероятность максимального отклонения между наблюдаемым и ожидаемым mismatch-распределением при нулевой гипотезе, постулирующей экспансию; Rag – значение индекса Харпендинга [24]; P(Rag) – вероятность появления более высоких величин индекса Харпендинга по сравнению с наблюдаемыми значениями при нулевой гипотезе демографической или пространственной экспансии; M – число мигрантов; t1– время экспансии при скорости нуклеотидных замен 1% за 1 млн лет; t2– время экспансии при скорости нуклеотидных замен 1.77% за 1 млн лет.
Обращает на себя внимание совпадение величин τ у двух пар объединенных выборок, дающих приблизительную оценку времени начала предполагаемой экспансии (табл. 5). При этом самые высокие значения τ наблюдаются у H. olidus из Охотского и Японского морей, а самые низкие – из Баренцева и Берингова морей. В пересчете на временные характеристики это означает, что наиболее ранняя экспансия H. olidus имела место в историческом прошлом в южных регионах, в то время как филогеографические группы H. olidus северных участков ареала вида пережили период, соответствующий его расширению, позднее. Временные границы демографических процессов H. olidus для Баренцева и Берингова морей, полученные при использовании разных констант в уравнении трансформации показателей τ при оценке времени экспансии, отличаются, но не выходят за границы позднего плейстоцена (табл. 5). В соответствии с принятыми допущениями демографическая экспансия для Баренцева и Берингова морей датируется одним временем 34 000 (95%-ный доверительный интервал: 14 000–85 000) – 59 370 (24 000–148 000) лет назад и, согласно нашим данным, предшествует пространственной экспансии.
Сравнительные характеристики основных показателей генетической изменчивости малоротых корюшек, включающих выборку из Баренцева моря, подтверждают ее несомненную принадлежность к виду H. olidus. Результаты генеалогического анализа сопряженного географического распространения и mismatch-распределения гаплотипов мтДНК указывают на филогенетическую близость и общую демографическую историю обыкновенной малоротой корюшки Баренцева и Берингова морей (рис. 2, 3). Высокое генетическое сходство между выборками H. olidus наблюдается на фоне значительной географической удаленности популяций.
Следует отметить, что для H. olidus в последнее время границы распространения были откорректированы [9, 37]. При этом западную границу видового ареала по-прежнему определяло оз. Круглое (бассейн р. Кара, Байдарацкая губа Карского моря) [8, 38, 39]. Относительно недавно в бассейне р. Байдарата были обнаружены новые популяции H. olidus [40–42], а в 2016 г. на значительном удалении от западной границы – в районе выхода протоки оз. Песчанка-То в Баренцево море и непосредственно в акватории Варандейской губы юго-восточной части Баренцева моря [10]. Логично, что сразу возник вопрос о происхождении популяций в этой части ареала.
Изучению вклада арктических рефугиумов в сохранение и последующее расселение видов в настоящее время придается все большее значение [4, 43], в том числе и в отношении корюшковых рыб [44]. По мнению ряда ихтиологов [38, 39, 45, 46], во время последнего ледникового периода, 15–22 тыс. лет назад [47], обыкновенная малоротая корюшка могла выжить не только в Беренгийском рефугиуме, но и в небольших рефугиумах Полярного Урала. Согласно гипотезе [48], опирающейся на анализ литературных источников, H. olidus относится к видам, обладающим способностью к высокой скорости расселения. Тем не менее существенным препятствием, затрудняющим объяснение постледникового распространения H. olidus вдоль арктического побережья, является наличие на ее ареале двух протяженных разрывов: около 3000 км на североазиатской части ареала от р. Алазея до водоемов Полярного Урала [39] и 1100 км на североамериканской части [49].
Генетико-статистическая оценка демографических процессов H. olidus соответствует модели демографической экспансии и пространственной экспансии из Берингова в Баренцево море (табл. 5). Временные границы расширения ареала имеют большой доверительный интервал, но превышают датировки окончательного открытия Берингова пролива (около 11 000 лет назад) [50]. Большая статистическая ошибка обычна для подобного рода оценок, что связано со стохастичностью генетических процессов. В целом проведенный нами анализ и сопоставление полученных результатов с палеогеологическими данными не отвергают возможности выживания обыкновенной малоротой корюшки во время последнего ледникового периода в рефугиумах Полярного Урала. Пока на территории от Байдарацкой губы Карского моря до р. Алазея не обнаружены популяции H. olidus, огромный разрыв в современном ареале вида также служит фактическим подтверждением переживания малоротой корюшкой неблагоприятных условий существования в приледниковых убежищах на стыке между Азией и Европой.
Палеогеографические данные свидетельствуют о том, что предпоследний ледовый покров прибрежных территорий Евразии полностью растаял 60–45/41 тыс. лет назад [47, 51]. Затем наступил теплый межстадиал, во время которого H. olidus могла широко расселиться вдоль арктических побережий. Последнее оледенение начало зарождаться в Скандинавии около 30 тыс. лет назад и на территории России достигло своего максимума 21 тыс. лет назад. К этому времени произошло слияние Скандинавского ледового покрова с Баренцевоморско-Карским, восточное крыло которого достигло Таймыра. Следствием этих процессов стало разделение западных и восточных популяций корюшек. Однако юго-восточную часть Баренцева моря и юго-западную часть Карского моря ледники не закрыли, и район оказался изолированным от сибирских морей. Полярный Урал этими ледниками также не был захвачен. Последние исследования [52] показали, что после 50–60 тыс. лет назад сплошного покровного оледенения на Урале не было, а основные речные долины и озера были свободными ото льда начиная с 45–40 тыс. лет назад. В совокупности эти данные подтверждают существование морских и пресноводных полярных рефугиумов в обсуждаемом регионе во время последнего максимального оледенения.
Таким образом, сравнительный анализ малоротых корюшек на основе данных об изменчивости нуклеотидных последовательностей митохондриальных генов свидетельствует о том, что экземпляры корюшек, выловленные в Варандейской губе Баренцева моря, относятся к H. olidus. Результаты генеалогического анализа гаплотипов мтДНК подтверждают филогенетическую близость малоротых корюшек Баренцева и Берингова морей. Тем не менее практическое отсутствие современных генных потоков и результаты AMOVA указывают на длительную изоляцию удаленных географических популяций H. olidus. Форма mismatch-распределения и пониженный уровень разнообразия мтДНК малоротых корюшек Баренцева и Берингова морей, по сравнению с южными популяциями, могут быть связаны как с историческими, так и с современными суровыми климатическими условиями существования в северной части ареала вида. Популяции H. olidus могли неоднократно проходить через “бутылочное горлышко” как в прошлом на рефугиальной стадии, так и в современное время из-за сложных условий обитания в Заполярье. Статистические оценки исторических демографических процессов у H. olidus также допускают вероятность выживания отдельных популяций в ледовых рефугиумах Полярного Урала, наличие которых подтверждаются палеогеографическими исследованиями.
Работа выполнена при финансовой поддержке программы “Дальний Восток” 2018–2020 (проект № 18-4-042) и программы Минобрнауки (проект № АААА-А19-119011500368-9).
Авторы заявляют, что у них нет конфликта интересов.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Список литературы
Weider L.J., Hobæk A. Phylogeography and Arctic biodiversity: a review // Ann. Zool. Fennici. 2000. V. 37. P. 217–231.
Hewitt G.M. Genetic consequences of climatic oscillations in the Quaternary // Philos. Trans. R. Soc. Lond. B. 2004. V. 359. P. 183−195. https://doi.org/10.1098/rstb.2003.1388
Mecklenburg C.W., Møller P.R., Steinke D. Biodiversity of arctic marine fishes: taxonomy and zoogeography // Mar. Biodiv. 2011. V. 41. P. 109–140. https://doi.org/10.1007/ s12526-010-0070-z
Hardy S.M., Carr C.M., Hardman M. et al. Biodiversity and phylogeography of Arctic marine fauna: insights from molecular tools // Mar. Biodiv. 2011. V. 41. P. 195–210. https://doi.org/10.1007/s12526-010-0056-x
Layton K.K.S., Corstorphine E.A., Hebert P.D.N. Exploring Canadian echinoderm diversity through DNA barcodes // PLoS One. 2016. V. 11. № 11. e0166118. https://doi.org/10.1371/journal.pone.0166118
Клюканов В.А. Происхождение, расселение и эволюция корюшковых (Osmeridae) // Основы классификации и филогении лососевидных рыб. Л.: Изд-во Зоол. ин-та АН СССР, 1977. С. 13–27.
Lee D.S., Gilbert C.R., Hocutt C.H. et al. Atlas of North American Freshwater Fishes. North Carolina Biological Survey: Raleigh, NC, 1980. 868 p.
Черешнев И.А., Волобуев В.В., Шестаков А.В., Фролов С.В. Лососевидные рыбы Северо-Востока России. Владивосток: Дальнаука, 2002. 496 с.
Sawatzky C.D., Michalak D., Reist J.D. et al. Distributions of Freshwater and Anadromous Fishes from the Mainland Northwest Territories, Canada. Can. Manuscr. Rep. Fish. Aquat Sci. 2793, 2007. 239 p.
Новоселов А.П., Кондаков А.В., Гофаров М.Ю., Болотов И.Н. Обыкновенная малоротая корюшка Hypomesus olidus (Osmeridae) – новый вид для фауны Баренцева моря // Вопр. ихтиологии. 2019. Т. 59. № 1. С. 25–30. https://doi.org/10.1134/S0042875219010090
Скурихина Л.А., Кухлевский А.Д., Железнова К.О., Ковалев М.Ю. Анализ изменчивости митохондриальной ДНК обыкновенной малоротой корюшки Hypomesus olidus (Osmeridae) // Генетика. 2012. Т. 48. № 7. С. 844–854.
Sevilla R.G., Diez A., Noren M. et al. Primers and polymerase chain reaction conditions for DNA barcoding teleost fish based on the mitochondrial cytochrome b and nuclear rhodopsin genes // Mol. Ecol. Not. 2007. V. 7. Iss. 5. P. 730–734.
Ward R.D., Zemlak T.S., Innes B.H. et al. DNA barcoding Australia’s fish species // Philosoph. Trans Royal Soc. B. Biological Sci. 2005. V. 360. № 1462. P. 1847–1857.
Nei M., Kumar S. Molecular Evolution and Phylogenetics. N.Y.: Oxford Univ. Press, 2000. 333 p.
Hudson R.R., Slatkin M., Maddison W.P. Estimation of levels of gene flow from DNA sequence data // Genetics. 1992. V. 132. № 2. P. 583–589.
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C. et al. DnaSP 6: DNA sequence polymorphism analysis of large datasets // Mol. Biol. Evol. 2017. V. 34. P. 3299–3302.
Cockerham C.C. Analysis of gene frequencies // Genetics. 1973. V. 74. P. 679–700.
Weir B.S., Cockerham C.C. Estimating F statistics for the analysis of population structure // Evolution. 1984. V. 38. P. 1358–1370.
Excoffier L., Smouse P.E., Quattro J.M. Analysis of molecular variance inferred from metric distances among DNA haplotypes: application to human mitochondrial DNA restriction data // Genetics. 1992. V. 131. P. 479–491.
Excoffier L., Lischer H.E.L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. 2010. V. 10. P. 564–567.
Rogers A.R., Harpending H. Population growth makes waves in the distribution of pairwise genetic differences // Mol. Biol. Evol. 1992. V. 9. P. 552–569.
Excoffier L. Patterns of DNA sequence diversity and genetic structure after a range expansion: lessons from the infinite-island model // Mol. Ecol. 2004. V. 13. P. 853–864.
Rogers A. Genetic evidence for a Pleistocene population explosion // Evolution. 1995. V. 49. P. 608–615.
Harpending R.C. Signature of ancient population growth in a low-resolution mitochondrial DNA mismatch distribution // Hum. Biol. 1994. V. 66. № 4. P. 591–600.
Schneider S., Excoffier L. Estimation of demographic parameters from the distribution of pairwise differences when the mutation rates vary among sites: Application to human mitochondrial DNA // Genetics. 1999. V. 152. P. 1079–1089.
Smith G.R. Introgression in fishes: significance for paleontology, cladistics, and evolutionary rates // Syst. Biol. 1992. V. 41. P. 41–57.
Ilves K.L., Taylor E.B. Evolutionary and biogeographical patterns within the smelt genus Hypomesus in the North Pacific Ocean // J. Biogeogr. 2008. V. 35. № 1. P. 48–64. https://doi.org/10.1111/j.1365-2699.2007.01782.x
Dodson J.J., Tremblay S., Colombani F. et al. Trans-Arctic dispersals and the evolution of a circumpolar marine fish species complex, the capelin (Mallotus villosus) // Mol. Ecol. 2007. V. 16. Iss. 23. P. 5030–5043. https://doi.org/10.1111/j.1365-294X.2007.03559.x
Bandelt H.-J., Foster P., Rohl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. P. 37–48.
Felsenstein J. Confidence limits on phylogenies: An approach using the bootstrap // Evolution. 1985. V. 39. P. 783–791.
Tamura K., Nei M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees // Mol. Biol. Evol. 1993. V. 10. P. 512–526.
Kumar S., Stecher G., Tamura K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets // Mol. Biol. Evol. 2016. V. 33. P. 1870–1874. https://doi.org/10.1093/molbev/msw054
Skurikhina L.A., Kukhlevsky A.D., Kovpak N.E. Relationships of osmerid fishes (Osmeridae) of Russia: divergence of nucleotide sequences of mitochondrial and nuclear genes // Genes Genom. 2013. V. 35. P. 529–539. https://doi.org/10.1007/s13258-013-0099-z
Скурихина Л.А., Олейник А.Г., Кухлевский А.Д. и др. Генетическая дифференциация тихоокеанской корюшки Osmerus mordax dentex по данным анализа митохондриальной ДНК // Генетика. 2015. Т. 51. № 12. С. 1410–1422.
Скурихина Л.А., Олейник А.Г., Ковпак Н.Е. Изменчивость митохондриальной ДНК тихоокеанской мойвы (Mallotus villosus catervarius) Охотского моря по данным PCR-RFLP-анализа // Генетика. 2008. Т. 44. № 7. С. 929–937.
Мельникова М.Н., Сенчукова А.Л., Павлов С.Д., Малютина А.М. Новые данные о вариабельности мтДНК обыкновенной малоротой корюшки Hypomesus olidus (Osmeridae) с Командорских островов в сравнении с другими популяциями вида // Изв. РАН. Сер. Биол. 2018. № 1. С. 16–21. https://doi.org/10.1134/S1062359018010089
Черешнев И.А., Шестаков А.В., Фролов С.В. К систематике малоротых корюшек рода Hypomesus (Osmeridae) залива Петра Великого Японского моря // Биология моря. 2001. Т. 27. № 5. С. 340–346.
Иванова Е.И. О нахождении малоротой корюшки на Европейском Севере // Тр. Всесоюз. гидробиол. об-ва. 1952. № 4. С. 252–259.
Клюканов В.А. Систематика и родственные отношения корюшек родов Osmerus и Hypomesus (Osmeridae) и их расселение // Зоол. журн. 1975. Т. 54. № 4. С. 590–595.
Богданов В.Д., Богданова Е.Н., Гаврилов А.Л. и др. Биоресурсы водных экосистем Полярноrо Урала. Екатеринбург: УрО РАН, 2004. 168 с.
Мельниченко И.П., Богданов В.Д. Ихтиофауна малых рек приуральского берега Байдарацкой губы // Вестник АГТУ. Сер. Рыбное хозяйство. 2016. № 1. С. 30–35.
Богданов В.Д., Мельниченко И.П., Кижеватов Я.А., Богданова Е.Н. Структура населения рыб бассейна р. Байдаратаяхи // Вестник АГТУ. Сер. Рыбное хоз-во. 2017. № 2. С. 33–44.
Stewart J.R., Lister A.M., Barnes I., Dalén L. Refugia revisited: individualistic responses of species in space and time // Proc. R. Soc. B. 2010. V. 277. P. 661–671. https://doi.org/10.1098/rspb.2009.1272
Skurikhina L.A., Oleinik A.G., Kukhlevsky A.D. et al. Phylogeography and demographic history of the Pacific smelt Osmerus dentex inferred from mitochondrial DNA variation // Pol. Biol. 2018. V. 41. P. 877–896. https://doi.org/10.1007/s00300-018-2250-4
McPhail J.D., Lindsey C.C. Freshwater fishes of Northwestern Canada and Alaska // Fish. Res. Board Canada Bull. 1970. V. 173. 381 p.
Кудерский Л.А. Пути формирования северных элементов ихтиофауны севера европейской территории СССР // Труды ГосНИОРХ. 1987. Вып. 258. С. 102–121.
Svendsen J.I., Alexanderson H., Astakhov V.I. et al. Late Quaternary ice sheet history of northern Eurasia // Quat. Sci. Rev. 2004. V. 23. P. 1229–1271. https://doi.org/10.1016/j.quascirev.2003.12.008
Махров А.А., Лайус Д.Л. Послеледниковое вселение рыб и миноги из Тихого океана в моря севера Европы // Сиб. экол. журн. 2018. Т. 11. № 3. С. 265–279. https://doi.org/10.1134/S1995425518030071
Degraaf D.A. Aspects of the life history of the pond smelt (Hypomesus olidus) in the Yukon and Northwest Territories // Arctic. 1986. V. 39. № 3. P. 260–263.
Jakobsson M., Pearce C., Cronin T.M. et al. Post-glacial flooding of the Bering Land Bridge dated to 11 cal ka BP based on new geophysical and sediment records // Clim. Past. 2017. V. 13. P. 991–1005. https://doi.org/10.5194/cp-13-991-2017
Hughes A.L.C., Gyllencreutz R., Lohne Ø.S. et al. The last Eurasian ice sheets – a chronological database and time-slice reconstruction, DATED-1 // Boreas. 2016. V. 45. P. 1–45. https://doi.org/10.1111/bor.12142
Svendsen J.I., Faærseth L.M.B., Gyllencreutz R. et al. Glacial and environmental changes over the last 60 000 years in the Polar Ural Mountains, Arctic Russia, inferred from a high-resolution lake record and other observations from adjacent areas // Boreas. 2018. https://doi.org/10.1111/bor.12356
Дополнительные материалы отсутствуют.


