Генетика, 2019, T. 55, № 9, стр. 1021-1030
Полный геном Bifidobacterium angulatum GT102. II. Системы сигнальной трансдукции и адаптивные гены
В. З. Незаметдинова 1, *, Н. В. Захаревич 1, О. В. Аверина 1, М. С. Чекалина 1, М. Г. Алексеева 1, В. Н. Даниленко 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
* E-mail: veneranez@rambler.ru
Поступила в редакцию 28.01.2019
После доработки 12.03.2019
Принята к публикации 26.03.2019
Аннотация
Вид Bifidobacterium angulatum встречается в кишечной микробиоте человека гораздо реже по сравнению с преобладающими видами Bifidobacterium longum и Bifidobacterium adolescentis. Вероятно, это связано с различной способностью данных видов к колонизации кишечника и взаимодействию с клетками организма-хозяина. Предметом данного исследования были гены систем сигнальной трансдукции и адаптивные гены в геноме B. angulatum GT102. Обнаружена уникальная для вида B. angulatum гистидиновая киназа HK10, принадлежащая к QseC семейству киназ, среди которых встречаются бактериальные адренергические рецепторы. Показана существенная разница в структуре сенсорного С-концевого домена видоспецифической серин-треониновой протеинкиназы Pkb2 у B. angulatum и у видов B. adolescentis и B. longum, что может указывать на взаимодействие данного домена с различными лигандами. У белка-адгезина FN3 B. angulatum, содержащего два фибронектиновых домена типа 3 с мотивами цитокиновых рецепторов, аминокислотная последовательность второго мотива цитокинового рецептора уникальна и отличается от мотивов, характерных для других видов бифидобактерий человека, что может быть причиной различий во взаимодействии вида B. angulatum с иммунной системой хозяина. В отличие от B. adolescentis и B. longum в геноме B. angulatum GT102 не были найдены гены, кодирующие системы токсин–антитоксин II типа суперсемейств MazEF, RelBE и V-apBC, отвечающих за адаптацию бактерий к меняющимся условиям окружающей среды. Сравнительный анализ генов, дающих бактерии конкурентные преимущества и участвующих во взаимосвязи бифидобактерий с клетками хозяина, указывает на меньшие возможности у B. angulatum для успешной колонизации кишечника по сравнению с доминирующими видами бифидобактерий B. adolescentis и B. longum.
Представленная работа является второй частью статьи, посвященной сравнительному геномному анализу штамма Bifidobacterium angulatum GT102, двух других штамов B. angulatum, а также видов B-ifidobacterium adolescentis и Bifidobacterium longum, родственных виду B. angulatum [1]. Согласно литературным данным, B. angulatum относят либо к группе B. longum, либо к группе B. adolescentis [2–4].
В кишечной микробиоте (КМ) взрослого человека доминирующими видами бифидобактерий являются B. adolescentis и B. longum (в основном Bifidobacterium longum subsp. longum). Намного реже встречаются представители вида B. angulatum [5]. Неизвестно, что определяет конкурентные преимущества у доминирующих видов бифидобактерий в отличие от редких видов. Целью исследования была идентификация и сравнительный анализ генов и генных кластеров, участвующих в колонизации кишечника человека и взаимосвязи с клетками организма-хозяина, у редкого вида B. angulatum и доминирующих видов B. longum и B. adolescentis. Предметом сравнительного геномного анализа данной части исследования являются генные локусы и отдельные гены, кодирующие системы сигнальной трансдукции, а также адаптивные гены.
Коммуникация бактерий КМ с организмом человека осуществляется двунаправленно. Бактерии могут синтезировать различные активные соединения, способные оказывать влияние на работу пищеварительной, эндокринной, иммунной и нервной систем человека [6]. С другой стороны, бактерии кишечной микробиоты должны реагировать на изменения условий обитания и узнавать сигналы организма хозяина. Этот процесс осуществляется сенсорными системами сигнальной трансдукции, которые обычно являются трансмембранными (ТМ) белками, имеющими как внеклеточные сенсорные модули, так и внутриклеточную часть. Сигнальные системы представлены однокомпонентными системами (1КС), в частности, серин-треониновыми протеинкиназами (СТПК), а также двухкомпонентными системами (2КС) [7, 8]. Двухкомпонентные системы представляют собой гистидиновые киназы (ГК) и работающие с ними в комплексе белки – регуляторы ответа (РО), которые обычно являются регуляторами генной экспрессии [9]. Эти клеточные системы осуществляют специфическое фосфорилирование белков и, по-видимому, способны взаимодействовать друг с другом, обеспечивая тем самым дополнительные уровни регуляции клеточного процесса, что может быть важно для адаптации бактерий к различным нишам обитания и специфическим условиям [10, 11].
В данной работе также были описаны гены, ответственные за адаптацию к постоянно изменяющимся условиям внешней среды и выживание в условиях стресса: гены систем токсин–антитоксин (ТА) II типа [12] и гены, кодирующие глобальные регуляторы транскрипции WhiB-like семейства [13].
МАТЕРИАЛЫ И МЕТОДЫ
Бактериальные штаммы, среды и условия культивирования. В работе использовали штамм B. angulatum GT102 [14, 15]. Штамм выделен из кишечника взрослого здорового человека. Видовая принадлежность штамма определена с помощью секвенирования гена 16S рРНК. Штамм депонирован в международной коллекции ВКПМ (VKPM) (http:// eng.genetika.ru/service-offer/vkpm/). Международный номер депонирования: VKPM AC-1973.
Бифидобактерии выращивали на MRS-агаре и в MRS-бульоне (HiMedia, Индия) с добавлением цистеина (0.5 г/л). Культивирование проводили в анаэробных условиях (HiAnaerobic System – Mark III, A-naeroHiGas Pack 3. 5L; HiMedia, Индия) при температуре 37°С в течение 24–48 ч.
Секвенирование и сборка генома. Геном штамма B. angulatum GT102 был секвенирован на платформе 454 GS Junior (Roche, Швейцария), депонирован в базе данных GenBank (NCBI) и аннотирован [1]. Идентификационный номер генома B. angulatum GT102 в базе данных GenBank: CP014241 (NZ_CP014241.1/CP014241.1).
Биоинформатический анализ. Последовательности геномов, генов и белков бифидобактерий, обсуждаемые в статье, были получены из базы данных GenBank (NCBI). Анализ генов и генных кластеров проводили с использованием следующих ресурсов: базы данных NCBI; онлайн веб-сервера BLAST NCBI (https://blast.ncbi.nlm.nih.gov/ Blast.cgi); программы Clustal Omega (https:// www.ebi.ac.uk/Tools/msa/clustalo/); программы SMART (http://smart.embl-heidelberg.de/); программы PHOBIUS (http://phobius.sbc.su.se/); программы TMHMM v. 2.0 (http://www.cbs.dtu.dk/services/TMHMM/); веб-приложения WebLogo [16] (https://weblogo.berkeley.edu/logo.cgi). Иллюстратор IBS версии 1.0.1 был использован для визуализации доменной архитектуры анализируемых протеинкиназ и структурной организации кластеров генов [17]. Для расчета молекулярного веса (mw) и изоэлектрической точки (pI) белков использовали PROTEIN CALCULATOR v. 3.4 (http:// protcalc.sourceforge.net/).
Поиск генов двухкомпонентных сигнальных систем в геноме штамма B. angulatum GT102 проводили по аннотации и исходя из информации о 2КС у других видов Bifidobacterium с использованием двух баз данных: P2CS – Prokaryotic 2-Component Systems [18] (http://www.p2cs.org) и MiST2.2 – Microbial Signal Transduction database [19] (http:// mistdb.com/).
РЕЗУЛЬТАТЫ
Гены сенсорных сигнальных систем в геноме B. angulatum GT102
В геноме B. angulatum GT102 были идентифицированы гены, кодирующие белки двухкомпонентных сенсорных сигнальных систем (ГК и РО), и гены, кодирующие белки однокомпонентных сенсорных сигнальных систем (СТПК).
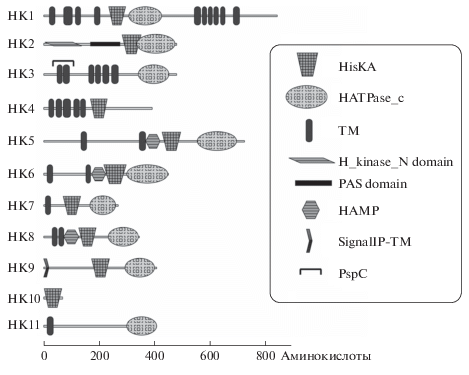
Гены, кодирующие гистидиновые протеинкиназы. Геномы различных видов бифидобактерий содержат разное число генов, кодирующих 2КС: B. angulatum – 12 ГК/11 РО, B. adolescentis – 14 2КС, B. longum subsp. longum – 9–11 ГК/13–15 РО, B. longum subsp. infantis – 18 ГК/23 РО (по данным баз данных P2CS и MiST). В геноме B. angulatum GT102 были идентифицированы гены одиннадцати 2КС. Гены и соответствующие белки были обозначены: гены гистидиновых киназ hk1–hk11, гистидиновые киназы HK1–HK11; гены регуляторов ответа rr1–rr11; регуляторы ответа RR1– RR11 (Приложение, табл. 1). Доменная организация гистидиновых киназ, кодируемых соответствующими генами, представлена на рис. 1.
Таблица 1.
Группы сенсорных гистидиновых киназ, кодируемые генами B. angulatum GT102
| Группы гистидиновых киназ | Название гистидиновых киназ и locus_tag | Идентичность аминокислотных последовательностей ГК у разных видов бифидобактерий | Локализация в клетке | ||
|---|---|---|---|---|---|
| B. angulatum | B. longum | B. adolescentis | |||
| Уникальная для штамма B. angulatum GT102 |
HK4 Bang102_002585 |
– | – | – | Трансмембранный белок |
| Уникальная
для вида B. angulatum |
HK6 Bang102_007225 |
96–100% | – | – | » |
| HK7 Bang102_006705 |
– | – | » | ||
| HK10 Bang102_007220 |
– | – | Нет данных | ||
| Специфичная для групп видов B. longum и B. adolescentis | HK1 Bang102_006975 |
94–97% | Трансмембранный белок | ||
| Присутствует у всех видов рода Bifidobacterium | HK2 Bang102_007605 |
~80% | Цитоплазматический белок | ||
| HK8 Bang102_007385 |
Трансмембранный белок | ||||
| HK3 Bang102_000565 |
52–78% | » | |||
| HK11 Bang102_000485 |
» | ||||
| HK5 Bang102_001380 |
» | ||||
| HK9 Bang102_003285 |
Внеклеточный секретируемый белок | ||||
Рис. 1.
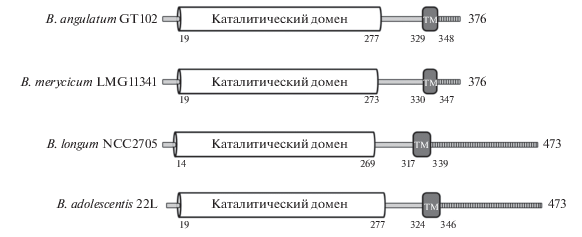
Доменная структура гистидиновых киназ B. angulatum GT102. На шкале цифры указывают длину белка в аминокислотных остатках. Сокращения: HisKA – сигнальная гистидиновая киназа, сайт димеризации и фосфоакцепторный домен; HATPase_c – каталитический домен гистидиновых киназ, осуществляющий перенос фосфата с АТФ на домен HisKA; ТМ – трансмембранный домен; H_kinase_N domain – N-концевой домен сигнальной гистидиновой киназы; PAS – сигнальный сенорный домен; HAMP – домен HAMP; SignalP-TM – сигнальный пептид; PspC – фаговый шоковый белок, N-концевой домен.
Был проведен сравнительный анализ аминокислотных последовательностей ГК штамма B. angulatum GT102 и двух видов бифидобактерий: B. longum и B. adolescentis (табл. 1).
HK2 и HK8 обнаружены у всех видов рода Bifidobacterium с идентичностью ~80% и могут быть отнесены к консервативным белкам. HK3, HK5, HK9, HK11 обнаружены у всех видов бифидобактерий и характеризуются высокой степенью дивергенции (межвидовая идентичность ~52–78%), они могут рассматриваться как видоспецифические киназы. Интересно, что у B. angulatum ген hk9 кодирует киназу HK9, имеющую сигнальный пептид и, по-видимому, являющуюся секретируемым белком; а у видов B. longum и B. adolescentis гены-ортологи кодируют киназы, имеющие трансмембранный домен, но не имеющие сигнального пептида. Очевидно, что функции этих киназ у B. angulatum и у других видов бифидобактерий различны. HK1 имеет ортологи у некоторых видов бифидобактерий из групп B. longum и B. adolescentis.
Киназы HK6, HK7, HK10 уникальны для вида B. angulatum и не имеют ортологов как у видов B. longum и B. adolescentis (табл. 1), так и у всех других видов бифидобактерий (неопубликованные данные). HK10 является сенсорной киназой семейства QseC. Среди киназ данного семейства могут встречаться бактериальные рецепторы для адреналина/норадреналина, продуцируемого организмом-хозяином [20, 21]. Ген hk10 B. angulatum GT102, по-видимому, имеет делецию и кодирует укороченный белок (71 а.о.). Штамм B. angulatum JCM7096 имеет ортолог этого гена, кодирующий секретируемую гистидинкиназу длиной 123 а.о. (идентичность 96%). Ген hk10 B. angulatum GT102 имеет стартовый кодон GTG [22], но промоторный участок не содержит RBS [23].
Гистидиновая киназа HK4 не имеет ортологов ни в штаммах B. angulatum JCM7096 и LMG11039, ни у всех других видов бифидобактерий (табл. 1) – эта уникальная киназа обнаружена только в штамме B. angulatum GT102.
Возможные функции гистидиновых киназ описаны в приложении (Приложение, табл. 1).
Гены, кодирующие серин-треониновые протеинкиназы. Ранее нами были идентифицированы и охарактеризованы шесть серин-треониновых протеинкиназ у представителей рода Bifidobacterium: одна видоспецифичная (Pkb2), одна уникальная (Pkb4) и четыре консервативных СТПК (Pkb1, Pkb3, Pkb5, Pkb6) [24–26]. Пять из шести генов, кодирующих STPK (pkb1, pkb2, pkb3, pkb5, pkb6), были аннотированы в геноме В. angulatum GT102 и кодировали Pkb1 (Bang102_005380), Pkb2 (Bang102_000205), Pkb3 (Bang102_001935), Pkb5 (Bang102_001740), Pkb6 (Bang102_001735). Аминокислотные последовательности каталитических доменов СТПК B. angulatum GT102 сравнили с последовательностями СТПК штаммов B. angulatum JCM7096, B. adolescentis ATCC15703 и BBMN23, штаммов B. longum DJO10A, NCC2705 и ATCC15697. Последовательности консервативных СТПК демонстрировали высокую степень сходства между различными видами бифидобактерий (идентичность от 76 до 90%). Степень идентичности аминокислотной последовательности видоспецифичной Pkb2 из B. angulatum GT102 и последовательности Pkb2 из других видов составляет <50%. Стоит отметить, что степень сходства аминокислотных последовательностей Pkb2 из разных штаммов, принадлежащих к одному и тому же виду, очень высока (идентичность 98–100%). Описанные закономерности согласуются с данными, полученными нами ранее [24].
В представленной статье мы сосредоточились на рассмотрении доменной структуры видоспецифической киназы Pkb2. СТПК Pkb2, помимо видоспецифичности, интересна тем, что ее предполагаемый ортолог у микобактерий (у которых СТПК хорошо изучены) представляет собой киназу PknK, которая участвует в механизмах адаптации клетки к условиям внешней среды [27, 28]. Протеинкиназа Pkb2 содержит каталитический домен, трансмембранный домен, внеклеточный С-концевой регион (рис. 2). С-концевые области являются менее консервативными областями, чем каталитические домены. Внеклеточная С-концевая область действует как сенсорный компонент. Предсказанный ТМ домен представляет собой трансмембранную альфа-спираль с гидрофобными аминокислотными остатками. Мы исследовали аминокислотные последовательности С-концевой области Pkb2, включая ТМ домен, у всех доступных секвенированных геномов штаммов B. angulatum, B. adolescentis и B. longum. Кроме того, вид Bifidobacterium merycicum был включен в анализ как наиболее близкий к виду B. angulatum [2]. С‑концевые области Pkb2 B. angulatum и B. merycicum короче, чем соответствующие области протеинкиназ Pkb2 у B. adolescentis и B. longum (рис. 2). B. angulatum и B. merycicum продемонстрировали абсолютную идентичность С-концевой области Pkb2. Между видами B. adolescentis и B. longum наблюдается межвидовая идентичность C-терминальной области до 60%. Нет гомологии между С-концевыми областями Pkb2 B. angulatum, B. merycicum и С-концевыми областями Pkb2 у видов B. adolescentis и B. longum (Приложение, рис. 1).
Кластер генов PFNA, потенциально участвующий в коммуникации бифидобактерий с иммунной системой хозяина
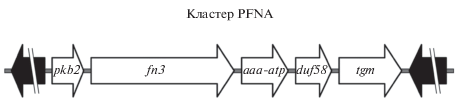
Ближайшее генетическое окружение гена pkb2 высококонсервативно у представителей рода Bifidobacterium. Проведенный нами ранее биоинформатический анализ генов, окружающих pkb2 у различных видов рода Bifidobacterium, выявил видоспецифический кластер генов, транскрибирующихся в одном направлении, и показал, что число генов кластера, обозначенного нами PFNA, может варьировать от 5 до 8 в зависимости от вида [29]. У вида B. angulatum кластер состоит из пяти генов: ген серин-треониновой протеинкиназы (pkb2); ген, кодирующий белок, содержащий фибронектиновые домены FN3 (fn3); ген ATPазы семейства MoxR (aaa-atp); ген, кодирующий белок, содержащий домен DUF58 (duf58), и ген трансглутаминазы (tgm) (рис. 3). Аналогичная организация кластера характерна для вида B. adolescentis. Для вида B. longum характерен кластер, состоящий из восьми генов [29].
Рис. 3.
Структурная организация кластера генов PFNA у B. angulatum GT102. Генам, показанным на рисунке, соответствуют следующие номера locus_tag: pkb2 – Bang102_000205; fn3 – Bang102_000210; aaa-atp – Bang102_000215; duf58 – Bang102_000220; tgm – Bang102_000225. Длина и направление стрелок коррелируют с длиной и направлением транскрипции гена.
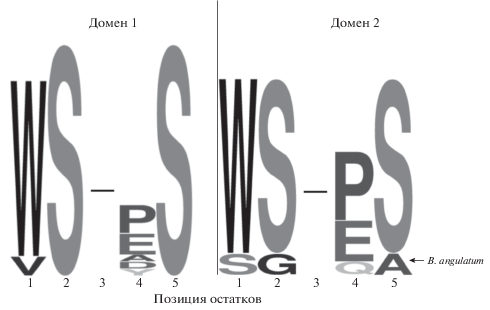
В кластере PFNA особый интерес вызывает ген fn3 – он кодирует белок, который содержит два фибронектиновых домена типа 3 (домены FN3) в С-концевой области. В каждом из FN3 доменов данного белка аннотируется мотив цитокинового рецептора. На рис. 4 представлены мотивы цитокиновых рецепторов, характерные для FN3 доменов рассматриваемого белка, для видов бифидобактерий, обитающих в кишечнике человека: B. longum, B. adolescentis, B. bifidum, B. breve, B. catenulatum, B. pseudocatenulatum, B. dentium, B. angulatum, B. kashiwanohense и B. gallicum. Для большинства рассматриваемых видов бифидобактерий мотивы довольно консервативны: WS-PS, WS-ES, WS-DS или WS-YS. Во 2-м FN3 домене описанного белка у вида B. angulatum аминокислотная последовательность мотива цитокинового рецептора SG-QA уникальна и отличается от мотивов других представленных видов.
Рис. 4.
Диаграмма Logo, отображающая структуру мотивов цитокиновых рецепторов двух фибронектиновых доменов белка FN3 для бифидобактериальных видов, населяющих кишечник человека. Мотив, обнаруженный во втором фибронектиновом домене белка FN3, характерный только для вида B. angulatum, отмечен на рисунке стрелкой. В положении 3 может находиться любая аминокислота.
Гены адаптации B. angulatum GT102
Бифидобактерии подвергаются действию различных стрессов [30]. Поэтому гены, ответственные за передачу сигнала и адаптацию к стрессовым условиям, имеют решающее значение для выживания бактерий в агрессивной среде. В данной работе мы проанализировали геном B. angulatum GT102 на наличие генов, кодирующих белки WhiB-like семейства и систем ТА II типа.
Семейство WhiB-подобных белков. Белки семейства WhiB-подобных белков выявляются только у актинобактерий, к которым относятся и бифидобактерии [31]. Они являются регуляторами транскрипции и участвуют в реакции бактерии на различные стрессовые факторы [32, 33]. При анализе генома В. angulatum GT102 было выявлено наличие двух консервативных whiB-генов: ген wblE (Bang102_007600), кодирующий белок WblE размером 92 а.о., и ген whiB2 (Bang102_007575), кодирующий белок WhiB2 размером 99 а.о. Ортологи wblE и whiB2 были найдены во всех исследуемых геномах видов В. adolescentis и B. longum [13]. Выравнивание аминокислотных последовательностей открытых рамок считывания генов для белков WhiB2 и WblE среди геномов различных штаммов B. angulatum показало высокую идентичность (100%). Идентичность 83% для WhiB2 и 96% для WblE наблюдалась при сравнении В. angulatum GT102 со штаммами В. adolescentis и 82% для WhiB2 и 97% для WblE – при сравнении со штаммами B. longum. Гены, кодирующие другие WhiB-подобные белки, были обнаружены в геномах B. adolescentis и B. longum [13], но не выявлены в геноме B. angulatum GT102. Ортологи whiB2 и wblE генов B. angulatum GT102 были найдены в геномах бифидобактерий других видов [13] и имели схожее генетическое окружение. Стоит отметить, что на филогенетическом дереве ортологов wblE, построенном ранее для бифидобактерий [13], штамм B. angulatum JCM7096 располагается на одной ветви с кластером штаммов вида B. longum.
Токсин–антитоксин системы II типа. Другими важными регуляторными белками, ответственными за адаптацию к постоянно изменяющимся условиям окружающей среды и условиям стресса, являются белки ТА II типа [34, 35]. Белки, относящиеся к важным и наиболее распространенным MazEF, RelBE и VapBC суперсемействам [36], были обнаружены у большинства видов бифидобактерий [37]. Однако они не были выявлены в геномах B. angulatum. В геноме штамма В. angulatum GT102 был идентифицирован только ген Bang102_001820, кодирующий белок токсина (103 а.о.), принадлежащий к ParE-toxin белкам, первоначально выявленным на плазмиде RK2 и отвечающим за стабильность наследования плазмиды [38]. Эти белки относятся к суперсемейству RelE/ParE, которое включает также семейства токсинов RelE, YoeB и ParE. Система ТА II типа состоит из двух генов токсина и антитоксина, организованных в оперон. Ген Bang102_001820 кодирует потенциальный токсин (putative addiction module killer protein) и расположен рядом с геном Bang102_001825, кодирующим антитоксин-подобный белок размером 97 а.о. (addiction module antitoxin). Оба гена организованы в оперон, в котором первый ген кодирует белок токсина, второй ген кодирует белок антитоксина. Ортологи этих белков были выявлены в других геномах B. angulatum (BIFANG_02205 и BIFANG_02206 в штамме JCM7096; BIANG_1422 и BIANG_1421 в штамме LMG11039). Выравнивание аминокислотных последовательностей этих белков показало 98% идентичности. При поиске ортологов в геномах других видов бифидобактерий были обнаружены гены с очень высоким уровнем идентичности (94–98%) в штаммах видов B. kashiwanohense и B. breve.
В геноме B. angulatum GT102 также был идентифицирован ген Bang102_005610, кодирующий белок токсина размером 83 а.о., также принадлежащий к семейству ParE-toxin. Этот токсин не имел парного антитоксина и не имел ортологов в других штаммах B. angulatum, доступных в базе данных NCBI. Но в геномах других видов B. adolescentis и B. longum были найдены ортологи с высоким уровнем идентичности.
ОБСУЖДЕНИЕ
В представленном исследовании был проведен сравнительный анализ генов и генных кластеров, участвующих в колонизации кишечника человека и взаимосвязи с клетками организма-хозяина, у редкого вида B. angulatum и доминирующих видов B. longum и B. adolescentis. Предметом сравнительного геномного анализа во второй части исследования были генные локусы и отдельные гены, кодирующие системы сигнальной трансдукции, а также адаптивные гены.
В геноме B. angulatum GT102 были идентифицированы гены одиннадцати 2КС. Экспериментальные данные о функциях 2КС у бифидобактерий очень ограничены. Существует лишь несколько экспериментальных исследований функций 2КС [39–41]. Мы можем делать предположения о функциях 2КС штамма B. angulatum GT102 только на основании сходства с изученными 2КС бифидобактерий, генетического окружения генов 2КС и аннотации белков. Наиболее интересным представляется ген гистидиновой киназы HK10. Данный ген является уникальным для вида B. angulatum и не имеет ортологов у других видов бифидобактерий. HK10 является сенсорной киназой семейства QseC. Сенсорная киназа QseC может являться бактериальным рецептором для адреналина/норадреналина, продуцируемого организмом-хозяином, а также гормоноподобных аутоиндукторов “quorum sensing” механизма, продуцируемых желудочно-кишечной микрофлорой [20, 21]. Не исключено, что HK10 может быть потенциальным рецептором, участвующим в коммуникации с клетками хозяина. Ген hk10, по-видимому, имеет делецию и кодирует укороченный белок (71 а.о.). Штамм B. angulatum JCM7096 имеет ортолог этого гена, кодирующего секретируемую гистидинкиназу длиной 123 а.о. (идентичность 96%). Неясно, экспрессируется ли ген hk10 и продуцируется ли белок в штамме B. angulatum GT102, поскольку ген имеет стартовый кодон GTG [22], но промоторный участок не содержит RBS [23].
При изучении генов 1КС в геноме B. angulatum GT102 мы сосредоточились на исследовании C-терминальной области видоспецифической СТПК Pkb2. Внеклеточная С-концевая область СТПК является сенсорным компонентом, имеет вариабельную структуру и может взаимодействовать с различными сигнальными молекулами из внешней среды (лигандами) [42, 43]. Мы исследовали аминокислотные последовательности С-концевой области Pkb2 из всех доступных штаммов видов B. angulatum, B. adolescentis и B. longum. Была обнаружена существенная разница в длине С-концевой области Pkb2 и низкая степень сходства между этим локусом у B. angulatum (и B. merycicum) и у двух других анализируемых видов. Это может указывать на то, что у B. angulatum и у видов B. adolescentis и B. longum С-концевой домен протеинкиназы Pkb2 взаимодействует с различными лигандами.
У вида B. angulatum видоспецифический кластер генов PFNA состоит из пяти генов (рис. 3). Аналогичная организация кластера характерна для вида B. adolescentis. Для вида B. longum характерен кластер, состоящий из восьми генов [29]. В представленной работе мы рассматривали первые два гена кластера: ген серин-треониновой протеинкиназы (pkb2) и ген, кодирующий белок-адгезин, содержащий фибронектиновые домены FN3 (fn3).
Белок FN3 содержит два фибронектиновых домена типа 3 (домены FN3) в С-концевой области. Экспериментально показано, что белок FN3 участвует в адгезии бифидобактерий на клетках эпителия кишечника человека [44]. В каждом из FN3 доменов данного белка аннотируется мотив цитокинового рецептора. Известно, что у E. coli существует белок – рецептор цитокинов, имеющий пространственную структуру, сходную со структурой FN3 домена [45]. Возможно, что белок FN3, кодируемый геном fn3 из кластера PFNA, также способен участвовать в процессах взаимодействия бифидобактерий с иммунной системой хозяина. При сравнении мотивов цитокиновых рецепторов у ряда видов бифидобактерий, обитающих в кишечнике человека, выяснилось, что для большинства рассматриваемых видов бифидобактерий мотивы достаточно консервативны (рис. 4). Однако у вида B. angulatum во втором FN3 домене описанного белка последовательность мотива цитокинового рецептора SG-QA уникальна и отличается от мотивов других представленных видов, что может быть причиной различий во взаимодействии этого вида с иммунной системой хозяина.
Важными белками, ответственными за выживание бифидобактерий в организме человека, являются белки семейства WhiB-подобных белков, которые являются регуляторами транскрипции и участвуют в основных клеточных процессах, включая реакцию на внешние и внутренние факторы стресса [32], и белки, относящиеся к системам ТА II типа, которые принимают участие в процессах трансляции и вовлечены в бактериальную адаптацию к постоянно меняющимся условиям окружающей среды [46]. Как и в геномах различных видов бифидобактерий [13], у штамма В. angulatum GT102 были найдены два консервативных гена, кодирующие белки WblE и WhiB2, но не были найдены другие whiB-подобные гены, несущие адаптивную функцию. Белок WblE является компонентом 2КС и вместе с консервативной гистидиновой киназой HK2 участвует в адаптивном ответе бактериальной клетки на окислительный стресс.
В отличие от бифидобактерий видов B. adolescentis и B. longum [37] в геноме B. angulatum GT102 из белков ТА II типа была обнаружена только одна пара, относящаяся к суперсемейству белков, участвующих в стабилизации плазмиды (plasmid stabil superfamily), но не были найдены системы ТА II типа, относящиеся к суперсемействам MazEF, RelBE и VapBC [36]. Именно белки этих суперсемейств отвечают за адаптацию бактерий к меняющимся условиям окружающей среды.
Таким образом, сравнительный анализ геномов, отдельных генов и генных кластеров штаммов различных видов указывает, что B. angulatum с большей вероятностью относится к группе B. adolescentis. Также, сравнительный анализ генов, дающих бактерии конкурентные преимущества и участвующих во взаимосвязи бифидобактерий с клетками хозяина, указывает на меньшие возможности у B. angulatum для успешной колонизации кишечника по сравнению с доминирующими видами бифидобактерий B. adolescentis и B. longum.
Исследование выполнено в рамках Государственного задания по теме 0112-2019-0002 “Генетические технологии в биологии, медицине, сельскохозяйственной и природохозяйственной деятельности” и при частичной финансовой поддержке РФФИ в рамках научного проекта № 18-34-00645 мол_а.
Авторы заявляют, что у них нет конфликта интересов.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Список литературы
Захаревич Н.В., Аверина О.В., Незаметдинова В.З., Даниленко В.Н. Полный геном Bifidobacterium angulatum GT102. I. Потенциальные гены и генные кластеры, участвующие во взаимосвязи с клетками хозяина // Генетика. 2019. Т. 55. № 8. С. 876–886.
Lugli G.A., Milani C., Turroni F. et al. Investigation of the evolutionary development of the genus Bifidobacterium by comparative genomics // Appl. Environ. M-icrobiol. 2014. V. 80. № 20. P. 6383–6394. https://doi.org/10.1128/AEM.02004-14
Milani C., Lugli G.A., Duranti S. et al. Genomic encyclopedia of type strains of the genus Bifidobacterium // Appl. Environ. Microbiol. 2014. V. 80. № 20. P. 6290–6302. https://doi.org/10.1128/AEM.02308-14
Sun Z., Zhang W., Guo C. et al. Comparative genomic analysis of 45 type strains of the genus Bifidobacterium: a snapshot of its genetic diversity and evolution // PLoS One. 2015. V. 10. № 2. e0117912. https://doi.org/10.1371/journal.pone.0117912
Turroni F., Peano C., Pass D.A. et al. Diversity of Bifidobacteria within the infant gut microbiota // PLoS One. 2012. V. 7. № 5. e36957. https://doi.org/10.1371/journal.pone.0036957
Аверина О.В., Даниленко В.Н. Микробиота кишечника человека: роль в становлении и функционировании нервной системы // Микробиология. 2017. Т. 86. № 1. С. 5–24. https://doi.org/10.7868/S0026365617010050
Mascher T., Helmann J.D., Unden G. stimulus perception in bacterial signal-transducing histidine kinases // Microbiol. Mol. Biol. Rev. 2006. V. 70. № 4. P. 910–938. https://doi.org/10.1128/MMBR.00020-06
Huang X., Pinto D., Fritz G., Mascher T. Environmental sensing in Actinobacteria: a comprehensive survey on the signaling capacity of this phylum // J. Bacteriol. 2015. V. 197. № 15. P. 2517–2535. https://doi.org/10.1128/JB.00176-15
Mattos-Graner R.O., Duncan M.J. Two-component signal transduction systems in oral bacteria // J. Oral M-icrobiol. 2017. V. 9. № 1. P. 1400858. https://doi.org/10.1080/20002297.2017.1400858
Shi L., Pigeonneau N., Ravikumar V. et al. Cross-phosphorylation of bacterial serine/threonine and tyrosine protein kinases on key regulatory residues // Front. M-icrobiol. 2014. V. 5. P. 495. https://doi.org/10.3389/fmicb.2014.00495
Mijakovic I., Grangeasse C., Turgay K. Exploring the diversity of protein modifications: special bacterial phosphorylation systems // FEMS Microbiol. Rev. 2016. V. 40. № 3. P. 398–417. https://doi.org/10.1093/femsre/fuw003
Averina O., Alekseeva M., Shkoporov A., Danilenko V. Functional analysis of the type II toxin-antitoxin systems of the MazEF and RelBE families in Bifidobacterium longum subsp. infantis ATCC 15697 // Anaerobe. 2015. V. 35. P. 59–67. https://doi.org/10.1016/j.anaerobe.2015.07.007
Averina O.V., Zakharevich N.V., Danilenko V.N. Identification and characterization of WhiB-Like family proteins of the Bifidobacterium genus // Anaerobe. 2012. V. 18. № 4. P. 421–429. https://doi.org/10.1016/j.anaerobe.2012.04.011
Dyachkova M.S., Klimina K.M., Kovtun A.S. et al. Draft genome sequences of Bifidobacterium angulatum GT102 and Bifidobacterium adolescentis 150: focusing on the genes potentially involved in the gut-brain axis // G-enome Announc. 2015. V. 3. № 4. e00709-15. https://doi.org/10.1128/genomeA.00709-15
Yunes R.A., Poluektova E.U., Dyachkova M.S. et al. GABA production and structure of gadB/gadC genes in Lactobacillus and Bifidobacterium strains from human microbiota // Anaerobe. 2016. V. 42. P. 197–204. https://doi.org/10.1016/j.anaerobe.2016.10.011
Crooks G.E., Hon G., Chandonia J.M., Brenner S.E. WebLogo: A sequence logo generator // Genome Res. 2004. V. 14. № 6. P. 1188–1190. https://doi.org/10.1101/gr.849004
Liu W., Xie Y., Ma J. et al. IBS: an illustrator for the presentation and visualization of biological sequences // Bioinformatics. 2015. V. 31. № 20. P. 3359–3361. https://doi.org/10.1093/bioinformatics/btv362
Barakat M., Ortet P., Whitworth D.E. P2CS: a database of prokaryotic two component systems // Nucleic Acids Res. 2011. V. 39 (Database issue). P. D771–D776. https://doi.org/10.1093/nar/gkq1023
Ulrich L.E., Zhulin I.B. The MiST2 database: a comprehensive genomics resource on microbial signal transduction // Nucl. Acids Res. 2010. V. 38 (Database issue). P. D401–D407. https://doi.org/10.1093/nar/gkp940
Clarke M.B., Hughes D.T., Zhu C. et al. The QseC sensor kinase: A bacterial adrenergic receptor // Proc. Natl Acad. Sci. USA. 2006. V. 103. № 27. P. 10420–10425. https://doi.org/10.1073/pnas.0604343103
Yang K., Meng J., Huang Y.C. et al. The role of the QseC quorum-sensing sensor kinase in epinephrine-enhanced motility and biofilm formation by Escherichia coli // Cell Biochem. Biophys. 2014. V. 70. № 1. P. 391–398. https://doi.org/10.1007/s12013-014-9924-5
Bottacini F., O’Connell-Motherway M., Kuczynski J. et al. Comparative genomics of the Bifidobacterium breve taxon // BMC Genom. 2014. V. 15. P. 170. https://doi.org/10.1186/1471-2164-15-170
He J., Sakaguchi K., Suzuki T. Determination of the ribosome-binding sequence and spacer length between binding site and initiation codon for efficient protein expression in Bifidobacterium longum 105-A // J. Biosci. Bioeng. 2012. V. 113. № 4. P. 442–444. https://doi.org/10.1016/j.jbiosc.2011.11.019
Nezametdinova V.Z., Zakharevich N.V., Alekseeva M.G. et al. Identification and characterization of the serine/threonine protein kinases in Bifidobacterium // Arch. Microbiol. 2014. V. 196. № 2. P. 125–136. https://doi.org/10.1007/s00203-013-0949-8
Алексеева М.Г., Мавлетова Д.А., Колчина Н.В. и др. Выделение и очистка рекомбинантных белков серин-треониновых протеинкиназ у штамма Bifidobacterium longum B379M и изучение их активности // Биохимия. 2015. Т. 80. № 10. С. 1578–1587.
Zakharevich N.V., Averina O.V., Klimina K.M. et al. Complete genome sequence of Bifidobacterium longum GT15: Identification and characterization of unique and global regulatory genes // Microb. Ecol. 2015. V. 70. № 3. P. 819–834. https://doi.org/10.1007/s00248-015-0603-x
Malhotra V., Okon B.P., Clark-Curtiss J.E. Mycobacterium tuberculosis protein kinase K enables growth adaptation through translation control // J. Bacteriol. 2012. V. 194. № 16. P. 4184–4196. https://doi.org/10.1128/JB.00585-12
Захаревич Н.В., Даниленко В.Н. Серин-треониновые протеинкиназы бактерий – потенциальная мишень для регуляции состава микробиоты человека // Вестник РГМУ. 2017. № 2. С. 20–29. https://doi.org/10.24075/brsmu.2017-02-03
Nezametdinova V.Z., Mavletova D.A., Alekseeva M.G. et al. Species-specific serine-threonine protein kinase Pkb2 of Bifidobacterium longum subsp. longum: Genetic environment and substrate specificity // Anaerobe. 2018. V. 51. P. 26–35. https://doi.org/10.1016/j.anaerobe.2018.03.003
Tannock G.W. Analysis of bifidobacterial populations in bowel ecology studies // Bifidobacteria: Genomics and Molecular Aspects. Norfolk, UK: Caister Academic Press, 2010. P. 1–17.
Gao B., Paramanathan R., Radhey S.G. Signature proteins that are distinctive characteristics of Actinobacteria and their subgroups // Antonie van Leeuwenhoek. 2006. V. 90. № 1. P. 69–91. https://doi.org/10.1007/s10482-006-9061-2
Geiman D.E., Raghunand T.R., Agarwal N., Bishai W.R. Differential gene expression in response to exposure to antimycobacterial agents and other stress conditions among seven Mycobacterium tuberculosis whiB-like genes // Antimicrob. Agents Chemother. 2006. V. 50. № 8. P. 2836–2841. https://doi.org/10.1128/AAC.00295-06
Zheng F., Long Q., Xie J. The function and regulatory network of WhiB and WhiB-like protein from comparative genomics and systems biology perspectives // Cell Biochem. Biophys. 2012. V. 63. № 2. P. 103–108. https://doi.org/10.1007/s12013-012-9348-z
Krügel H., Klimina K.M., Mrotzek G. et al. Expression of the toxin-antitoxin genes yefM(Lrh), yoeB(Lrh) in human Lactobacillus rhamnosus isolates // J. Basic Microbiol. 2015. V. 55. № 8. P. 982–991. https://doi.org/10.1002/jobm.201400904
Rocker A., Meinhart A. Type II toxin: antitoxin systems. More than small selfish entities? // Curr. Genet. 2016. V. 62. № 2. P. 287–290. https://doi.org/10.1007/s00294-015-0541-7
Gerdes K., Christensen S.K., Lobner-Olsen A. Prokaryotic toxin-antitoxin stress response loci // Nat. Rev. Microbiol. 2005. V. 3. № 5. P. 371–382. https://doi.org/10.1038/nrmicro1147
Аверина О.В., Алексеева М.Г., Абилев С.К. и др. Распространение генов систем токсин–антитоксин семейств MazEF и RelBE у бифидобактерий кишечной микробиоты человека // Генетика. 2013. Т. 49. № 3. С. 315–327. https://doi.org/10.7868/S0016675813030028
Dalton K.M., Crosson S. A conserved mode of protein recognition and binding in a ParD-ParE toxin-antitoxin complex // Biochemistry. 2010. V. 49. № 10. P. 2205–2215. https://doi.org/10.1021/bi902133s
Alvarez-Martin P., Fernández M., O’Connell-Motherway M. et al. A conserved two-component signal transduction system controls the response to phosphate starvation in Bifidobacterium breve UCC2003 // Appl. Environ. Microbiol. 2012. V. 78. № 15. P. 5258–5269. https://doi.org/10.1128/AEM.00804-12
Alvarez-Martin P., O’Connell-Motherway M., Turroni F. et al. A two-component regulatory system controls autoregulated serpin expression in Bifidobacterium breve UCC2003 // Appl. Environ. Microbiol. 2012. V. 78. № 19. P. 7032–7041. https://doi.org/10.1128/AEM.01776-12
Ruiz L., Zomer A., O’Connell-Motherway M. et al. Discovering novel bile protection systems in Bifidobacterium breve UCC2003 through functional genomics // Appl. Environ. Microbiol. 2012. V. 78. № 4. P. 1123–1131. https://doi.org/10.1128/AEM.06060-11
Pereira S.F., Goss L., Dworkin J. Eukaryote-like serine/threonine kinases and phosphatases in bacteria // Microbiol. Mol. Biol. Rev. 2011. V. 75. № 1. P. 192–212. https://doi.org/10.1128/MMBR.00042-10
Ruggiero A., De Simone P., Smaldone G. et al. Bacterial cell division regulation by Ser/Thr kinases: a structural perspective // Curr. Protein. Pept. Sci. 2012. V. 13. № 8. P. 756–766. https://doi.org/10.2174/138920312804871201
Westermann C. Analysis of potential host-colonization factors in Bifidobacterium bifidum S17: Diss. cand. sci. biol. Germany: Ulm Univ., 2015.
Moriel D.G., Heras B., Paxman J.J. et al. Molecular and structural characterization of a novel Escherichia coli interleukin receptor mimic protein // mBio. 2016. V. 7. № 2. e02046-15. https://doi.org/10.1128/mBio.02046-15
Yamaguchi Y., Park J.H., Inouye M. Toxin–antitoxin systems in bacteria and archaea // Ann. Rev. Genet. 2011. V. 45. P. 61–79. https://doi.org/10.1146/annurev-genet-110410-132412
Дополнительные материалы отсутствуют.