Генетика, 2020, T. 56, № 1, стр. 67-77
ГЕНЕТИЧЕСКАЯ ДИФФЕРЕНЦИАЦИЯ ПОПУЛЯЦИЙ ВОЛКА Canis lupus L. СИБИРИ ПО МИКРОСАТЕЛЛИТНЫМ ЛОКУСАМ
М. С. Талала 1, *, А. Я. Бондарев 2, Е. С. Захаров 3, 4, 5, Д. В. Политов 1, **
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Контрольный информационно-аналитический центр охотничьих животных
и среды их обитания
107140 Москва, Россия
3 Институт биологических проблем криолитозоны Сибирского отделения
Российской академии наук
677980 Якутск, Россия
4 Институт естественных наук Северо-Восточного федерального университета
им. М.К. Аммосова
677000 Якутск, Россия
5 Якутская государственная сельскохозяйственная академия
677007 Якутск, Россия
* E-mail: maiiatalala@gmail.com
** E-mail: dmitri_p@inbox.ru
Поступила в редакцию 09.01.2018
После доработки 22.03.2019
Принята к публикации 15.05.2019
Аннотация
Исследованы образцы 270 особей волка Canis lupus L. из различных регионов Сибири (Алтайский край, Республика Алтай, Республика Тыва, Красноярский край, Республика Бурятия, Забайкальский край, Якутия) с помощью семи микросателлитных локусов. Обнаружен высокий уровень ожидаемой гетерозиготности (HE = 0.676), при этом статистически значимых различий показателей внутрипопуляционного аллельного и генного разнообразия между выборками не выявлено. Низкие попарные значения FST (0.012–0.084, в среднем 0.036) свидетельствуют о том, что популяции волка Сибири связывает активный поток генов. Многомерный анализ по матрице генетических дистанций (PCoA) показал группировку выборок в соответствии с их географическим происхождением (Западная, Средняя и Восточная Сибирь). Кластеризация многолокусных гаплотипов в программе STRUCTURE v. 2.3.4 также выявила наличие пространственной подразделенности – изменение вклада различных генетических кластеров (К = 4) в генофонд изученных выборок волка как с запада на восток, так и с юга на север. Обсуждается в свете полученных генетических данных вопрос о выделении и распространении подвидов волка.
Волк Canis lupus L. (Mammalia: Carnivora: Canidae) – широко распространенный крупный хищник. Его восстановленный ареал охватывает большую часть суши Северного полушария [1], где этот вид играет важную роль в природных экосистемах и имеет важное социально-экономическое значение. Испокон веков существует конфликт между человеком и волком. Хищничество волка оказывает непосредственное давление на популяции диких и домашних видов животных, нанося ощутимый урон охотничьему хозяйству и животноводству и вынуждая человека на ответные меры. В результате этого противостояния в течение последних двух веков произошло общее снижение численности и сокращение ареала хищника, как вследствие прямого преследования человеком, так и по причине фрагментации естественных местообитаний [1], что в целом ряде случаев привело к формированию изолированных популяций. Для волка Северной Америки и Европы, обитающего в антропогенно нарушенных экосистемах, было показано снижение генетического разнообразия вследствие прохождения таких популяций через “бутылочное горлышко” и дальнейшего инбридинга [2–10]. Результатом исследований стало внедрение программ по восстановлению волка в местах, где он был истреблен или его численность значительно сократилась [9, 11]. Например в Европе проведение охранных мероприятий вместе с восстановлением кормовой базы и созданием коридоров для распространения способствовали постепенному росту численности волка, и в настоящее время она оценивается в 12 тыс. особей [12].
В России волк широко распространен, а его численность, согласно данным зимних маршрутных учетов, составляет около 60 тыс. особей [13]. В XX в. на данной территории волк также испытывал давление со стороны человека. Неоднократно предпринимались попытки минимизации численности хищника, но устойчивого сокращения ареала это не вызывало, напротив, наблюдалось активное распространение вида в восточную часть страны вслед за человеком [14], а в последние годы и восстановление численности в Европейской части России. В настоящее время волк включен в перечень охотничьих видов России, и его добыча ограничена сезоном. Ежегодно в стране добывается от семи до десяти тысяч волков [15]. Повысить эффективность мер контроля численности и в то же время способствовать сохранению генофонда волка поможет лучшее понимание внутривидовой структуры хищника, которая для данной территории изучена недостаточно.
Для волка, обитающего на территории России, описан ряд подвидов и географических популяций (“группировок”), связанных с приуроченностью к определенным экологическим условиям и трофической специализацией [16]. Однако причины пространственной дифференциации по морфологическим признакам до сих пор не ясны. Связаны ли они с адаптивным характером вариаций размера и окраски или указывают на наличие устойчивых наследственно обусловленных различий? Это нуждается в проверке и дополнении результатами других видов исследований, в первую очередь молекулярно-генетических, масштаб которых на территории России до сих пор был незначителен. Некоторые зарубежные исследования включают анализ генетического материала российских волков [17–19], однако такие выборки немногочисленны, локальны и в основном происходят из Европейской части страны, что не позволяет оценить все разнообразие вида на российской части ареала. Тем не менее, вместе с российскими работами по изучению волка Алтая [20, 21], такие исследования говорят в пользу относительно высокого уровня генетической изменчивости российских волков.
Волк, обитающий на территории Сибири, остается наименее изученным с помощью генетических методов. В то же время Сибирь с ее малонарушенными природными местообитаниями представляет собой идеальный “полигон” для изучения естественных популяций волка. Низкая плотность населения человека позволяет волку поддерживать высокую численность даже в условиях ее регулирования. Изучение относительно незатронутых человеком популяций волка необходимо для понимания эволюционной истории и факторов формирования и поддержания внутривидовой структуры этого хищника на ареале.
В рамках данного исследования мы оценили уровень генетического разнообразия волка Сибири, а также проанализировали популяционно-генетическую структуру вида на данной части ареала с помощью молекулярных маркеров ядерной локализации – микросателлитных локусов.
МАТЕРИАЛЫ И МЕТОДЫ
Характеристика биоматериала
В работе использовали образцы (сушеные фрагменты невыделанных шкур и ткани, фиксированные в спирту) 270 волков Canis lupus L., добытых охотниками в 2007–2014 гг. в следующих регионах России (число образцов приведено в скобках): Алтайский край (64), Республика Алтай (11), Республика Тыва (43), Красноярский край (12), Республика Бурятия (76), Забайкальский край (21), Республика Саха (Якутия) (43). Волков добывали охотники в рамках программ по регулированию численности, проводимых в регионах. Образцы получали через охотуправления (управления по охране и рациональному использованию животного мира субъектов Федерации) или непосредственно от охотников. Для всех образцов были известны дата и район добычи, для некоторых также были указаны возраст и пол особей. Индивидуальные образцы были сгруппированы в 21 географическую выборку (табл. 1, рис. 1). Приведенные географические координаты соответствуют средним значениям координат районов добычи животных, входящих в состав выборок.
Таблица 1.
Характеристика сформированных выборок
| № п/п | Код выборки | Число особей | Субъект РФ | Районы | Широта, ° с.ш. |
Долгота, ° в.д. |
|---|---|---|---|---|---|---|
| 1 | Alt_N | 5 | Алтайский край | Ельцовский, Заринский | 53.58 | 85.78 |
| 2 | Alt_W | 34 | То же | Егорьевский, Михайловский, Угловский | 51.55 | 80.24 |
| 3 | Alt_E | 25 | » | Алтайский, Курьинский, Солонешенский, Усть-Канский, Чарышский | 51.78 | 85.03 |
| 4 | R_Alt | 11 | Республика Алтай | Онгудайский, Чемальский, Чойский | 51.33 | 86.56 |
| 5 | Tv_NW | 9 | Республика Тыва | Бай-Тайгинский | 51.18 | 89.44 |
| 6 | Tv_SW | 10 | То же | Монгун-Тайгинский | 50.18 | 89.82 |
| 7 | Tv_E | 24 | » | Тоджинский | 52.43 | 96.23 |
| 8 | Kr_Sh | 3 | Красноярский край | Шушенский | 53.09 | 91.77 |
| 9 | Kr_Ke | 4 | То же | Кежемский | 58.78 | 100.37 |
| 10 | Kr_Tur_N | 2 | » | Туруханский | 67.11 | 86.89 |
| 11 | Kr_Tur_S | 3 | » | Туруханский | 62.42 | 89.06 |
| 12 | Bur_SW | 14 | Республика Бурятия | Джидинский, Закаменский, Кабанский, Селенгинский | 51.12 | 105.15 |
| 13 | Bur_SE | 21 | То же | Бичурский, Кяхтинский, Мухоршибирский |
50.57 | 107.62 |
| 14 | Bur_Cntrl | 9 | » | Заиграевский, Кижингинский, Хоринский |
52.10 | 109.55 |
| 15 | Bur_Ervn | 19 | » | Еравнинский | 52.97 | 112.58 |
| 16 | Bur_Bnt | 13 | » | Баунтовский | 54.20 | 113.69 |
| 17 | Zab_W | 13 | Забайкальский край | Красночикойский | 50.07 | 109.23 |
| 18 | Zab_E | 8 | То же | Кыринский | 49.76 | 111.85 |
| 19 | Ya_Olkm | 14 | Республика Саха (Якутия) | Олекминский | 60.25 | 119.63 |
| 20 | Ya_Nrb | 15 | То же | Нюрбинский | 63.28 | 118.36 |
| 21 | Ya_Olnk | 14 | » | Оленекский | 68.50 | 112.50 |
Рис. 1.
Географическое положение сформированных выборок, диаметр точек пропорционален размеру выборок.
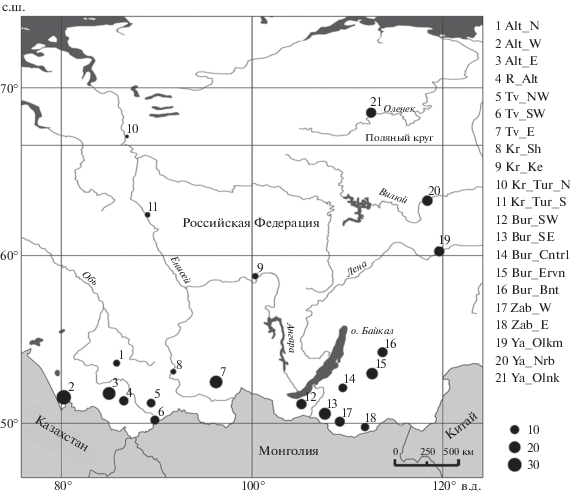
В связи с низкой численностью (менее восьми особей) выборок из Красноярского края и северных районов Алтайского края, и по причине достаточной удаленности этих районов друг от друга и от других мест сбора материала, возникла необходимость сохранения отдельных малочисленных выборок: Alt_N, Kr_Sh, Kr_Ke, Kr_Tur_N, Kr_Tur_S (далее – “малочисленные выборки”), которые не объединялись с другими, но в дальнейшем использовались лишь в некоторых видах анализа, менее чувствительных к численностям выборок.
Выделение и анализ ДНК
Выделение ДНК из сухих невыделанных шкур и фиксированных этанолом тканей мышц и внутренних органов проводили набором реагентов для выделения ДНК DiatomTM DNAPrep 100 (ООО “Лаборатория Изоген”, Москва, Россия). В качестве генетических маркеров мы отобрали разработанные ранее для домашней собаки семь микросателлитных локусов, содержащих тетрануклеотидные повторы: 2001, 2006, 2054, 2062, 2100, 2109, 2018 [22], для амплификации которых с помощью полимеразной цепной реакции (ПЦР) использовали семь пар специфических олигонуклеотидных праймеров, синтезированных в ЗАО “Евроген” (Москва, Россия) (табл. 2). В анализ были также включены ранее полученные генотипические данные для 109 образцов алтайских волков по локусам 2054, 2062 [20] (генотипы по остальным пяти локусам для указанных образцов были получены в настоящем исследовании).
Таблица 2.
Характеристика использованных олигонуклеотидных праймеров (из [11])
| Локус | Мотив повтора | Последовательность прямого праймера (5'–3') |
Последовательность обратного праймера (5'–3') |
|---|---|---|---|
| 2001 | (GATA)8 | TCCTCCTCTTCTTTCCATTGG | TGAACAGAGTTAAGGATAGACACG |
| 2006 | (GAAT)7 | TGGGGGCGTTAAGAGTAATG | CTAGGCCTAAACCCCTGAGC |
| 2109 | (GATT)8 | CAATCCAGCAACCCTCATCT | CAGGGATTGAGTCCCACATC |
| 2100 | (GATT)8 | AGTCCCACATCAGGCTTCC | TCATCTAGCTGAGCTCTTCTCA |
| 2054 | (GATA)16 | GCCTTATTCATTGCAGTTAGGG | ATGCTGAGTTTTGAACTTTCCC |
| 2062 | (GAAT)9 | GGCTTCTGGAGACAGGCAT | CAGAACGCTGTCTAGCCCTT |
| 2018 | (ATGA)7(ATTA)7 | TTGCTTTACCGCTTCATTTATT | GAGCCTGCTTCTCCCTCTG |
Выделенную тотальную геномную ДНК разводили до концентрации 50–100 нг/мкл и использовали как матрицу для последующей амплификации с помощью ПЦР. Для амплификации использовали наборы реагентов для ПЦР-амплификации ДНК GenePakTM PCR Core (ООО “Лаборатория Изоген”). Смесь прямого и обратного праймеров для каждого из исследуемых локусов готовили из расчета конечной концентрации смеси 1 пкмоль/мкл. В реакционную смесь для ПЦР (объемом 20 мкл) добавляли 5 мкл смеси праймеров. Режим ПЦР (ДНК-амплификатор BioRad Dyad T100 Thermal Cycler, Hercules, CA, USA) включал: 1) начальную денатурацию 95°С в течение 5 мин, 2) 32 цикла, состоящих из денатурации при 94°C (1 мин), отжиг для всех пар праймеров при 58°C (1 мин) и элонгации 72°C (1 мин), заключительная элонгация 72°C в течение 3 мин, сохранение продукта при 4°C.
Для определения длин амплифицированных фрагментов проводили вертикальный электрофорез продуктов ПЦР в блоках 6%-ного полиакриламидного геля (ПААГ) с использованием камер для вертикального электрофореза VE-20 (ООО “Компания Хеликон”, Москва, Россия) и трис-ЭДТА-боратной буферной системы (TBE). Выявление фрагментов ДНК на геле осуществляли с помощью окраски бромистым этидием с последующей визуализацией в УФ-свете и перехватом цифрового фотоизображения (система гель-регистрации Vilber-Lourmat, Collégien, France).
Длины амплифицированных фрагментов определяли с помощью программы PhotoCapt. Для более надежной идентификации аллелей проводили дополнительные проверочные электрофорезы. Для образцов, у которых не выявлялось специфических продуктов амплификации, проводили повторное выделение ДНК, ПЦР и электрофорез. Если амплификация по отдельным локусам по-прежнему отсутствовала, делали вывод о наличии гомозигот по “нуль-аллелям” (отсутствие амплификации из-за нуклеотидных замен в зоне отжига праймера).
Статистическую обработку данных для оценки уровней внутрипопуляционного генного и аллельного разнообразия и межпопуляционной генетической дифференциации проводили с помощью надстройки для электронной таблицы MS Excel – GenAlEx 6.5 [23, 24]. Для оценки наличия и расчета частот “нуль-аллелей” использовали программу GENEPOP 4.4 [25].
Оценку числа генотипических кластеров (К) по микросателлитным локусам осуществляли с помощью алгоритма Байесовской кластеризации в программе STRUCTURE v. 2.3.4 [26]. Для каждого предполагаемого значения K (от 2 до 5) было проведено пять независимых реплик симуляций с количеством итераций, равных 100 000 с предшествующим периодом “разогрева” в 10 000 итераций с использованием следующих опций: популяционной привязки данных LOCPRIOR=1; смешанной модели (admixture model), которая допускает наличие потока генов между выборками; независимых частот аллелей (independent allele frequency) между выборками, что рекомендовано для нашего типа данных. Оценку наиболее вероятного числа кластеров проводили в онлайн-программе Structure Harvester [27, 28]. Последующую обработку результатов для наиболее вероятного K производили в программе CLUMPP v. 1.1.2 [29]. Визуализацию результатов, полученных в CLUMPP, осуществляли в программе Distruct [30].
РЕЗУЛЬТАТЫ
Внутрипопуляционное генетическое разнообразие волка Сибири
Были получены генотипы для 270 особей по семи локусам. Для локуса 2006 была зафиксирована значительная частота нуль-аллелей, вследствие чего мы исключили данный локус из дальнейшего анализа. Для остальных шести локусов не обнаружено статистически значимых отклонений от равновесия Харди–Вайнберга. Исключение составляет локус 2062, по которому для выборок Alt_W, Tv_E и Bur_Bnt наблюдаются значимые (P < 0.05, P < 0.01) отклонения, как в сторону дефицита, так и эксцесса гетерозигот.
Средние показатели изменчивости, полученные по всем выборкам для каждого из шести локусов, представлены в табл. 3. Для исследованных микросателлитных локусов выявлен высокий уровень полиморфизма, среднее число аллелей на локус Na варьирует от 5 до 9. Приватных аллелей для изученных выборок не обнаружено. Средние значения показателей генетической изменчивости и стандартные ошибки для всех выборок, усредненные по шести локусам, представлены в табл. 4. Результаты, полученные для малочисленных выборок (в табл. 4 выделены серым фоном), далее в этом подразделе не приводятся.
Таблица 3.
Средние показатели генетической изменчивости и подразделенности по семи микросателлитным локусам в популяционных выборках волка
| Локус | Na | HE | HO | Размер аллелей, пн |
Частота нуль-аллелей |
FIS | FST |
|---|---|---|---|---|---|---|---|
| 2001 | 8 | 0.705 | 0.727 | 130–158 | 0.034 | –0.031 | 0.113 |
| 2006* | 6 | 0.624 | 0.555 | 191–211 | 0.092 | 0.112 | 0.113 |
| 2109 | 6 | 0.523 | 0.606 | 168–188 | 0.010 | –0.158 | 0.073 |
| 2100 | 5 | 0.614 | 0.677 | 160–176 | 0.039 | –0.103 | 0.131 |
| 2054 | 9 | 0.669 | 0.666 | 146–178 | 0.030 | 0.004 | 0.120 |
| 2062 | 6 | 0.611 | 0.680 | 128–148 | 0.028 | –0.113 | 0.115 |
| 2018 | 9 | 0.772 | 0.813 | 144–176 | 0.056 | –0.053 | 0.097 |
| По шести локусам для всех выборок | Среднее | –0.076 | 0.108 | ||||
| SE | 0.024 | 0.008 | |||||
| По шести локусам для 16 выборок | Среднее | –0.053 | 0.070 | ||||
| SE | 0.017 | 0.007 | |||||
Примечание. Na – среднее число аллелей на локус; HE – ожидаемая гетерозиготность; HO – наблюдаемая гетерозиготность; FIS – коэффициент инбридинга особи относительно субпопуляции; FST – коэффициент инбридинга субпопуляции относительно всей популяции; SE – ошибка среднего. * Локус исключен из дальнейшего анализа.
Таблица 4.
Средние показатели генетического разнообразия всех выборок (серым фоном выделены малочисленные выборки N < 8)
| Выборка | Объем выборки | Na | Ne | HO | HE | F |
|---|---|---|---|---|---|---|
| Alt_N | 5 | 2.833 | 2.269 | 0.600 | 0.510 | –0.152 |
| 0.307 | 0.328 | 0.103 | 0.069 | 0.119 | ||
| Alt_W | 34 | 5.500 | 3.558 | 0.647 | 0.676 | 0.044 |
| 0.563 | 0.670 | 0.061 | 0.050 | 0.044 | ||
| Alt_E | 25 | 5.167 | 3.376 | 0.693 | 0.669 | –0.039 |
| 0.477 | 0.537 | 0.048 | 0.048 | 0.031 | ||
| R_Alt | 11 | 4.667 | 3.271 | 0.697 | 0.680 | –0.028 |
| 0.333 | 0.301 | 0.051 | 0.031 | 0.065 | ||
| Tv_NW | 9 | 5.333 | 3.672 | 0.815 | 0.703 | –0.175 |
| 0.615 | 0.499 | 0.079 | 0.039 | 0.121 | ||
| Tv_SW | 10 | 5.333 | 3.783 | 0.717 | 0.671 | –0.085 |
| 0.667 | 0.843 | 0.054 | 0.063 | 0.062 | ||
| Tv_E | 24 | 5.333 | 3.796 | 0.688 | 0.710 | 0.035 |
| 0.919 | 0.635 | 0.074 | 0.032 | 0.089 | ||
| Kr_Sh | 3 | 2.667 | 2.221 | 0.500 | 0.519 | 0.055 |
| 0.211 | 0.243 | 0.143 | 0.059 | 0.231 | ||
| Kr_Ke | 4 | 3.500 | 2.771 | 0.625 | 0.557 | –0.132 |
| 0.563 | 0.585 | 0.107 | 0.087 | 0.091 | ||
| Kr_Tur_N | 2 | 2.833 | 2.600 | 0.750 | 0.583 | –0.244 |
| 0.307 | 0.333 | 0.171 | 0.053 | 0.255 | ||
| Kr_Tur_S | 3 | 3.667 | 2.962 | 0.778 | 0.648 | –0.199 |
| 0.211 | 0.251 | 0.070 | 0.034 | 0.077 | ||
| Bur_SW | 14 | 5.000 | 3.718 | 0.738 | 0.708 | –0.055 |
| 0.730 | 0.501 | 0.035 | 0.036 | 0.070 | ||
| Bur_SE | 21 | 5.333 | 3.113 | 0.730 | 0.638 | –0.152 |
| 0.843 | 0.422 | 0.068 | 0.062 | 0.036 | ||
| Bur_Cntrl | 9 | 4.500 | 2.949 | 0.648 | 0.603 | –0.086 |
| 0.847 | 0.540 | 0.073 | 0.065 | 0.093 | ||
| Bur_Ervn | 19 | 5.167 | 3.595 | 0.711 | 0.681 | –0.056 |
| 0.749 | 0.583 | 0.049 | 0.051 | 0.071 | ||
| Bur_Bnt | 13 | 5.833 | 3.599 | 0.718 | 0.669 | –0.084 |
| 0.703 | 0.562 | 0.081 | 0.073 | 0.061 | ||
| Zab_W | 13 | 5.667 | 3.654 | 0.654 | 0.680 | 0.044 |
| 0.667 | 0.556 | 0.076 | 0.064 | 0.047 | ||
| Zab_E | 8 | 5.000 | 3.198 | 0.688 | 0.650 | –0.089 |
| 0.365 | 0.462 | 0.063 | 0.053 | 0.114 | ||
| Ya_Olkm | 14 | 5.333 | 3.608 | 0.786 | 0.690 | –0.156 |
| 0.667 | 0.519 | 0.049 | 0.047 | 0.081 | ||
| Ya_Nrb | 15 | 4.833 | 3.448 | 0.700 | 0.681 | –0.012 |
| 0.477 | 0.407 | 0.084 | 0.051 | 0.079 | ||
| Ya_Olnk | 13.833 | 4.833 | 3.639 | 0.712 | 0.703 | –0.014 |
| 0.167 | 0.543 | 0.420 | 0.082 | 0.039 | 0.102 | |
| Для всех выборок | 12.849 | 4.683 | 3.276 | 0.695 | 0.649 | –0.075 |
| 0.717 | 0.147 | 0.110 | 0.018 | 0.012 | 0.023 | |
| Для 16 выборок | 15.802 | 5.177 | 3.498 | 0.709 | 0.676 | –0.057 |
| 0.706 | 0.155 | 0.127 | 0.016 | 0.012 | 0.019 |
Примечание. F – коэффициент инбридинга; верхняя строка – среднее значение, нижняя – ошибка среднего (SE); другие обозначения см. в примечании к табл. 3.
Уровень ожидаемой гетерозиготности HE относительно высок и не имеет статистически значимых различий между выборками. Среднее значение ожидаемой гетерозиготности HE составляет 0.676. Среднее значение коэффициента инбридинга F равное –0.057 свидетельствует о незначительном избытке гетерозигот в целом для изученных выборок.
Межпопуляционное разнообразие волка Сибири
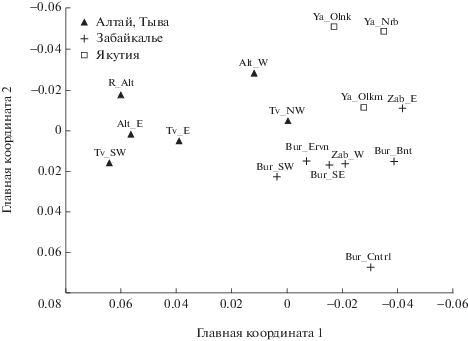
Среднелокусные значения показателя межпопуляционной подразделенности FST равны 0.108 и 0.070 для всех выборок и для 16 выборок соответственно (табл. 3). Результаты анализа главных координат (PCoA), проведенного по матрице попарных значений FST (0.012–0.084, среднее 0.036) для 16 выборок, представлены на рис. 2. Ординация в пространстве двух первых главных координат показала наличие кластеризации выборок в соответствии с их взаимным географическим расположением: 1) Алтай + Тыва, 2) Забайкалье, 3) Якутия.
Рис. 2.
Результаты анализа главных координат (PCoA) по матрице значений FST по микросателлитным локусам.
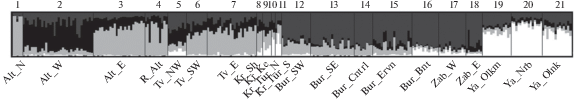
Результаты анализа генетической структуры для всех исследуемых выборок в комплексе программ STRUCTURE представлены на рис. 3. Наиболее вероятное (по методу Эванно) число исходных генетических кластеров К оказалось равно четырем. Вклад выявленных кластеров в генотипы отдельных особей визуализирован с помощью различных оттенков серого цвета. Принимая за обозначения кластеров К1, К2, К3 и К4 цвета светло-серый, темно-серый, белый и черный соответственно, можно в первую очередь отметить явное изменение характера вклада обнаруженных кластеров в генофонды исследуемых выборок как в широтном, так и в меридиональном направлениях. Кластер К1 (светло-серый) составляет большую часть генофонда волков из юго-западной части исследуемой территории, определяя генетическое сходство выборок из Алтая и Тывы. Для Забайкалья характерно наибольшее присутствие кластера К2 (темно-серый). По мере продвижения на север, наблюдается преобладание кластера К3, обозначенного белым цветом. Его вклад максимален для выборок из Якутии, но также наблюдается для некоторых юго-западных выборок и северных выборок из Красноярского края. Кластер К4 (черный цвет) выделяет выборку из Алтайского края Alt_W среди прочих юго-западных выборок. Вклад кластера К4 в разной степени характерен для всех западных выборок, а для выборок из Забайкалья и Якутии его вклад в генофонд минимален. Также для некоторых выборок можно отметить присутствие нехарактерных по соотношению вкладов различных кластеров особей.
ОБСУЖДЕНИЕ
Внутрипопуляционное генетическое разнообразие волка Сибири
За четыре десятилетия, минувших с начала изучения генетического разнообразия волка с помощью молекулярных методов, опубликовано множество работ, в том числе основанных на анализе микросателлитных локусов. Однако единую методику, которая бы позволяла давать адекватную сравнительную оценку результатам работы разных групп исследователей еще предстоит разработать [31]. Разные наборы используемых маркеров и определенная субъективность метода не позволяют напрямую сравнивать значения, получаемые в разных лабораториях. Тем не менее, мы приведем сравнительную оценку нашим результатам, опираясь на опубликованные данные.
В изученных выборках волка Сибири показан относительно высокий уровень генетической изменчивости по исследованным микросателлитным локусам. Для шести локусов наблюдается высокое аллельное разнообразие, среднее число обнаруженных аллелей для всех выборок составляет 4.68, при исключении выборок c N < 8 – 5.18 аллелей. Уровень ожидаемой гетерозиготности HE для всех выборок с N > 8 относительно высок и не имеет статистически значимых различий между выборками, среднее значение составляет 0.676. Значения показателей разнообразия, полученные для волков антропогенно нарушенных изолированных популяций Западной Европы, оказались в целом более низкими, чем в настоящем исследовании. Для волков Пиренейского полуострова HE по восьми исследованным тетрануклеотидным микросателлитам составила 0.599 [32]. Для 294 особей волка Апеннин по 12 микросателлитным локусам HE равна 0.620, среднее число обнаруженных аллелей составило 4.9, а для 141 волка Альп HE = 0.560, а Na= 3.5 [33]. Низкий уровень гетерозиготности (0.540) и аллельного разнообразия (Na= 3.8) для 12 микросателлитных локусов показаны в выборке из 97 волков Скандинавии [34]. Также для некоторых популяций волка Скандинавии отмечена высокая степень инбридинга: F = 0.00–0.41 [35], в то время как для изученных нами 16 выборок (N > 7) волка Сибири наблюдаются статистически недостоверно отличающиеся от нуля, как положительные, так и отрицательные значения коэффициента инбридинга. Полученные в нашей работе значения ожидаемой гетерозиготности для многочисленных выборок сравнимы со значением данного показателя для 29 волков Беловежской Пущи (N = 29, HE = 0.697), для которых был зафиксирован в целом высокий уровень генетического разнообразия [6].
Таким образом, можно сделать вывод об относительно высоком уровне внутрипопуляционной генетической изменчивости волков из разных регионов Сибири. Низкая плотность населения человека, отсутствие непреодолимых барьеров для распространения и разнообразие естественных местообитаний способствуют поддержанию уровня генетического разнообразия внутри популяций. Тем не менее, высокие показатели генетического разнообразия не исключают значительного сокращения численности волка и временной изоляции каких-либо групп особей на данных территориях в прошлом. Так, на примере волка Скандинавии был продемонстрирован эффект “генетического спасения”, который заключается в том, что иммиграция небольшого числа особей в стаю с низким уровнем гетерозиготности способна в течение непродолжительного времени восстановить относительно высокое значение данного показателя изменчивости [34].
Популяционно-генетическая структура волка Сибири и вопрос о распространении подвидов
Результаты различных типов анализа межпопуляционной дифференциации сибирских волков указывают на наличие определенной генетической структуры среди исследованных выборок.
Результаты анализа главных координат (PCoA) (рис. 2), проведенного по матрице значений FST, демонстрируют группирование географически смежных выборок между собой. Наблюдается кластеризация выборок в виде трех групп: 1) Алтай + Тыва; 2) Забайкалье; 3) Якутия. При этом наиболее западная алтайская выборка Alt_W удалена от остальных выборок с Алтая и вместе с выборкой Tv_NW сближается c двумя другими группами, а наиболее южная выборка из Якутии Ya_Olkm оказывается ближе других выборок из Якутии к выборкам из Забайкалья, занимая промежуточное положение.
Результаты анализа структуры популяций сибирского волка по всем выборкам с числом предполагаемых кластеров К = 4 (рис. 3) в программе STRUCTURE также демонстрируют группирование особей изученных выборок между собой, географически соответствующее границам Западной, Средней и Восточной Сибири.
Для обсуждения наблюдаемой генетической структуры обратимся к имеющимся литературным данным по динамике численности и распространению подвидов сибирских волков на исследованных территориях. А.П. Суворов и Н.Н. Кириенко [16] подробно рассмотрели вопрос о подвидах волка, обитающих на территории России, а также предложили выделение ряда географических популяций, претендующих на статус подвида. При выделении таких популяций авторы опирались на особенности экологии вида в различных условиях. Для крупных хищных уже было показано, что пищевая специализация и климатические условия лучше объясняют генетические различия, чем географические дистанции между популяциями [35]. Так, границы подвидовых ареалов волка Сибири определяет распространение связанных с ним видов диких копытных. Ареал тундрового волка (Canis lupus albus Kerr, 1792) совпадает с южной границей зимнего распространения таймырской тундровой популяции дикого северного оленя, среднетаежного волка (C. l. lupus Linnaeus, 1758) – с лосем, C. l. var. оrientalis Dubovski, 1922 – с ареалом лесного северного оленя, саянского горно-таежного (C. l. altaicus Noak, 1911) – с ареалом марала, лесостепного синантропного (возможно, является потомком гибридизации степного волка С. l. сampestris Dwigubski, 1804 с лесным волком) – с зимним распространением косули.
Описанное географическое распределение подвидов сибирского волка в большей части соответствует полученным нами данным по его генетической структуре. Можно предположить, что кластер К4 (черный цвет), вклад которого максимален для выборки Alt_W и присутствует среди всех выборок из Алтая и Тывы, представляет собой генофонд лесостепного синантропного волка, обитающего преимущественно на юго-западе Алтайского края (Canis l. sibiricus Koslow, 1966). Генофонд горно-таежного волка Алтая и Саян C. l. altaicus представлен преимущественно кластером К1 (светло-серый цвет). Выборки из Забайкалья характеризуются максимальным вкладом кластера К2 (темно-серый цвет), который вероятно составляет основу генофонда горно-таежной байкальской географической популяции. Присутствие кластера К1 в юго-западных выборках из Бурятии может быть связано с обитанием здесь волков преимущественно в лесах хребтов Хамар-Дабана, который сообщается с Восточными Саянами, Западными Саянами и Алтаем. Вероятно, данные популяции связывает поток генов. Кластер К3 (белый цвет) составляет генетическую основу выборок из Якутии, позволяя предположить его соответствие генофонду северных форм волка. Выборки из Якутии попадают в зону распространения горно-лесотундрового волка (C. l. turuchanensis Ognev, 1923) и лесного восточносибирского волка (Canis l. var. orientalis), однако существенных различий между якутскими выборками по характеру вклада генетических кластеров нет. Этот же кластер имеет значительную представленность в выборках с севера Красноярского края, что говорит о генетической близости изученных северных волков Средней и Восточной Сибири.
Таким образом, на основании полученных данных можно сделать вывод об относительно высоком уровне генетического разнообразия сибирского волка, а также о наличии популяционно-генетической структуры, которая в большей части соответствует литературным данным о распространении подвидовых форм. Тем не менее, необходимо отметить, что приведенное нами соответствие обнаруженных генетических кластеров ранее выделенным на основании морфологии и экологии подвидам волка носит предварительный характер. Для более определенного решения вопроса о правомерности выделения тех или иных подвидов, их составе и распространении, а также об их соответствии генетически дифференцированным популяциям, необходим комплексный анализ экологических, морфологических, а также шире представленных генетических данных. Важно также отметить, что истребление в прошлом таймырского полярного волка с применением авиации, а также степного волка и последующее восстановление этих группировок за счет гибридизации с лесным волком [16] привело к размыванию тех географических и экологических границ, на основе которых ранее выделялись и наиболее морфологически отличающиеся подвиды. Способность волка, как хорошо адаптированного к широкому спектру условий обитания хищника, восстанавливать почти утраченные популяции путем иммиграции особей из соседних и даже удаленных группировок делает вопрос о его внутривидовой структуре особенно актуальным в теоретическом плане. Насколько описанные подвиды являются результатом древнего расселения и экологической специализации, а в какой мере их своеобразие определяется современным отбором из общего генного пула связанных потоком генов группировок, происходящим на контрастном экофоне – еще предстоит выяснить.
Отметим также практическую сторону вопроса, а именно эффективность мер по регуляции численности волка, которая может быть невысокой без понимания популяционной структуры вида и анализа миграционной активности волка. Лишь получив представление о генетических процессах, происходящих в популяциях волка, можно организовать как охрану вида в регионах, где его популяции находятся в состоянии временной депрессии, так и регуляцию численности там, где наблюдается ее устойчивый рост, приводящий к конфликтам с животноводством и другим последствиям возросшей в последние десятилетия синантропности волка.
Работа выполнена при поддержке проекта Российского фонда фундаментальных исследований № 18-04-01300 и Программы фундаментальных исследований Президиума РАН № 41 “Биоразнообразие природных систем и биологические ресурсы России” (раздел “Генофонды живой природы и их сохранение”, тема 0112-2018-0025), а также темы государственного задания 0112-2019-0001.
Авторы благодарят за предоставленный материал по Забайкалью И.Н. Белова (Сохондинский природный биосферный заповедник), по Тыве – Н.Д. Карташова (Государственный природный заповедник “Азас”) и А.Д. Пояркова (ИПЭЭ РАН), за ценные консультации – А.А. Банникову (биологический факультет МГУ им. М.В. Ломоносова), за помощь и поддержку – М.М. Белоконь и Ю.С. Белоконя (ИОГен РАН).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Wolves: Behaviour, Ecology, and Conservation / Eds. Mech L.D., Boitani L. Chicago: University of Chicago Press, 2003. 428 p.
Aspi J., Roininen E., Ruokonen M. et al. Genetic diversity, population structure, effective population size and demographic history of the Finnish wolf population // Mol. Ecol. 2006. V. 15. № 6. P. 1561–1576. https://doi.org/10.1111/j.1365-294X.2006.02877.x
Ellegren H. Inbreeding and relatedness in Scandinavian grey wolves Canis lupus // Hereditas. 1999. V. 130. № 3. P. 239–244. https://doi.org/10.1111/j.1601-5223.1999.00239.x
Flagstad O., Walker C.W., Vila C. et al. Two centuries of the Scandinavian wolf population: patterns of genetic variability and migration during an era of dramatic decline // Mol. Ecol. 2003. V. 12. № 4. P. 869–880. https://doi.org/10.1046/j.1365-294X.2003.01784.x
Gomercic T., Sindicic M., Galov A. et al. High genetic variability of the grey wolf (Canis lupus L.) population from Croatia as revealed by mitochondrial DNA control region sequences // Zool. Studies. 2010. V. 49. № 6. P. 816–823. https://doi.org/10.1371/journal.pone.0093828
Jędrzejewski W., Branicki W., Veit C. et al. Genetic diversity and relatedness within packs in an intensely hunted population of wolves Canis lupus // Acta Theriologica. 2005. V. 50. № 1. P. 3–22. https://doi.org/10.1007/bf03192614
Leonard J.A., Vila C., Wayne R.K. Legacy lost: genetic variability and population size of extirpated US grey wolves (Canis lupus) // Mol. Ecol. 2005. V. 14. № 1. P. 9–17. https://doi.org/10.1111/j.1365-294X.2004.02389.x
Lucchini V., Galov A., Randi E. Evidence of genetic distinction and long-term population decline in wolves (Canis lupus) in the Italian Apennines // Mol. Ecol. 2004. V. 13. № 3. P. 523–536. https://doi.org/10.1046/j.1365-294X.2004.02077.x
vonHoldt B.M., Stahler D.R., Bangs E.E. et al. A novel assessment of population structure and gene flow in grey wolf populations of the Northern Rocky Mountains of the United States // Mol. Ecol. 2010. V. 19. № 20. P. 4412–4427. https://doi.org/10.1111/j.1365-294X.2010.04769.x
Wayne R.K., Lehman N., Allard M.W., Honeycutt R.L. Mitochondrial DNA variability of the gray wolf: genetic consequences of population decline and habitat fragmentation // Conservation Biol. 1992. V. 6. № 4. P. 559–569. https://doi.org/10.1046/j.1523-1739.1992.06040559.x
Randi E. Genetics and conservation of wolves Canis lupus in Europe // Mammal Review. 2011. V. 41. № 2. P. 99–111. https://doi.org/10.1111/j.1365-2907.2010.00176.x
Chapron G., Kaczensky P., Linnell J.D.C. et al. Recovery of large carnivores in Europe’s modern human-dominated landscapes // Science. 2014. V. 346. № 6216. P. 1517–1519. https://doi.org/10.1126/science.1257553
Ломанова Н.В., Борисов Б.П., Володина О.А. и др. Состояние охотничьих ресурсов в Российской Федерации в 2008–2010 гг. Информационно-аналитические материалы // Охотничьи животные России (биология, охрана, ресурсоведение, рациональное использование). 2011. № 9. С. 219.
Волк. Происхождение, систематика, морфология, экология / Под ред. Бибикова Д.И. М.: Наука, 1985. 606 с.
Бондарев А.Я., Дежкин А.В., Павлов П.М. Регулирование популяций волка: проблемы и решения // Териофауна России и сопредельных территорий. Международное совещание (X). Съезд Териологического общества при РАН. М.: Товарищество научных изданий КМК, 2016. С. 52.
Суворов А.П., Кириенко Н.Н. К вопросу о внутривидовом полиморфизме волка (Canis lupus), обитающего на территории России // Вестник Красноярского гос. агр. ун-та. 2008. № 3. С. 6.
Aspi J., Roininen E., Kiiskilä J. et al. Genetic structure of the northwestern russian wolf populations and gene flow between Russia and Finland // Conservation Genet. 2009. V. 10. № 4. P. 815–826. https://doi.org/10.1007/s10592-008-9642-x
Fan Z., Silva P., Gronau I. et al. Worldwide patterns of genomic variation and admixture in gray wolves // Genome Res. 2016. V. 26. № 2. P. 163–173. https://doi.org/10.1101/gr.197517.115
Sastre N., Vilà C., Salinas M. et al. Signatures of demographic bottlenecks in European wolf populations // Conservation Genet. 2010. V. 12. № 3. P. 701–712. https://doi.org/10.1007/s10592-010-0177-6
Воробьевская Е.А., Балдина С.Н. Исследование молекулярной филогеографии волка (Canis lupus L.) Алтая методом микросателлитных маркеров // Вестн. Моск. ун-та. 2011. Т. 66. № 2. С. 53–54.
Бондарев А.Я., Воробьевская Е.А., Политов Д.В. О генетическиой дифференциации волка Сибири // Вестник АГАУ. 2013. Т. 9. № 107. С. 49–57.
Francisco L.V., Langston A.A., Mellersh C.S. et al. A class of highly polymorphic tetranucleotide repeats for canine genetic mapping // Mammalian Genome. 1996. V. 7. № 5. P. 359–362. https://doi.org/10.1007/s003359900104
Peakall R.O.D., Smouse P.E. GenAlEx 6: genetic analysis in Excel. Population genetic software for teaching and research // Mol. Ecol. Notes. 2006. V. 6. № 1. P. 288–295. https://doi.org/10.1111/j.1471-8286.2005.01155.x
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research–an update // Bioinformatics. 2012. V. 28. № 19. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Rousset F. GENEPOP’007: a complete re-implementation of the GENEPOP software for Windows and Linux // Mol. Ecol. Resour. 2008. V. 8. № 1. P. 103–106. https://doi.org/10.1111/j.1471-8286.2007.01931.x
Pritchard J.K., Matthew S., Peter D. Inference of population structure using multilocus genotype data: Linked loci and correlated allele frequencies // Genetics. 2000. V. 164. № 4. P. 1567–1587. https://doi.org/10.3410/f.1015548.197423
Earl D.A., vonHoldt B.M. STRUCTURE HARVESTER: a website and program for visualizing STRUCTURE output and implementing the Evanno method // Conservation Genet. Res. 2011. V. 4. № 2. P. 359–361. https://doi.org/10.1007/s12686-011-9548-7
Evanno G., Regnaut S., Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE: a simulation study // Mol. Ecol. 2005. V. 14. № 8. P. 2611–2620. https://doi.org/10.1111/j.1365-294X.2005.02553.x
Jakobsson M., Rosenberg N.A. CLUMPP: a cluster matching and permutation program for dealing with label switching and multimodality in analysis of population structure // Bioinformatics. 2007. V. 23. № 14. P. 1801–1806. https://doi.org/10.1093/bioinformatics/btm233
Rosenberg N.A. Distruct: a program for the graphical display of population structure // Mol. Ecol. Notes. 2003. V. 4. № 1. P. 137–138. https://doi.org/10.1046/j.1471-8286.2003.00566.x
De Groot G.A., Nowak C., Skrbinsek T. et al. Decades of population genetic research reveal the need for harmonization of molecular markers: the grey wolf Canis lupus as a case study // Mammal Review. 2016. V. 46. № 1. P. 44–59. https://doi.org/10.1111/mam.12052
Godinho R., Llaneza L., Blanco J.C. et al. Genetic evidence for multiple events of hybridization between wolves and domestic dogs in the Iberian Peninsula // Mol. Ecol. 2011. V. 20. № 24. P. 5154–5166. https://doi.org/10.1111/j.1365-294X.2011.05345.x
Fabbri E., Miquel C., Lucchini V. et al. From the Apennines to the Alps: colonization genetics of the naturally expanding Italian wolf (Canis lupus) population // Mol. Ecol. 2007. V. 16. № 8. P. 1661–1671. https://doi.org/10.1111/j.1365-294X.2007.03262.x
Vila C., Sundqvist A.K., Flagstad O. et al. Rescue of a severely bottlenecked wolf (Canis lupus) population by a single immigrant // Proc. Biol. Sci. 2003. V. 270. № 1510. P. 91–97. https://doi.org/10.1098/rspb.2002.2184
Liberg O., Andren H., Pedersen H.C. et al. Severe inbreeding depression in a wild wolf (Canis lupus) population // Biol. Lett. 2005. V. 1. № 1. P. 17–20. https://doi.org/10.1098/rsbl.2004.0266
Дополнительные материалы отсутствуют.