Генетика, 2020, T. 56, № 1, стр. 21-34
Влияние структуры микроРНК животных на их биогенез
П. С. Ворожейкин 1, *, И. И. Титов 1, 2
1 Новосибирский национальный исследовательский государственный университет,
кафедра информатики ВКИ
630090 Новосибирск, Россия
2 Федеральный исследовательский центр Институт цитологии и генетики
Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
* E-mail: pavel.vorozheykin@gmail.com
Поступила в редакцию 06.02.2019
После доработки 07.03.2019
Принята к публикации 24.04.2019
Аннотация
МикроРНК — это малые некодирующие РНК, которые участвуют в посттранскрипционном регулировании экспрессии большинства генов в разных организмах. Рассматриваются современные достижения в понимании роли первичной и вторичной структур микроРНК-предшественников животных для разных путей биогенеза и на разных стадиях созревания микроРНК. Описываются эффекты генетической изменчивости и вариаций границ микроРНК, которые представляют собой часть эпитранскриптомики, а также ошибки аннотации.
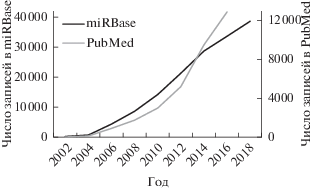
В настоящее время у животных обнаружено большое количество малых РНК, направленных на подавление нежелательного генетического материала или транскриптов. Эти РНК характеризуются своей небольшой длиной (20–30 нуклеотидов) и связью с семейством белков Argonaute (AGO- и PIWI-белки). Выделяют три класса малых РНК – микроРНК (от англ. miRNA), киРНК(короткие интерферирующие РНК, от англ. siRNA) и пиРНК (от англ. piRNA) [1]. Наиболее изученный класс, микроРНК, характеризуется длиной последовательностей в ~22 нуклеотида, которые получаются в результате разрезания первичного транскрипта РНКазами III Dicer и Drosha [2]. Зрелые микроРНК связываются с одним из AGO-белков и мишенями в мРНК, таким образом способствуя расщеплению последней или блокируя трансляцию с нее [3]. Одна микроРНК может адресовать несколько разных мишеней, что дает возможность контролировать активность большого количества белков и биологических процессов организма. Неудивительно, что блокирование генов микроРНК в животных приводит к появлению фенотипических изменений, а также к возникновению разных болезней [4]. Благодаря этой вовлеченности во многие регуляторные процессы в клетке микроРНК стремительно набирают популярность в качестве объекта исследований (рис. 1).
Рис. 1.
Динамика числа записей в базе данных miRBase и в базе данных PubMed, содержащих упоминания микроРНК в названии или аннотации.
В этом обзоре мы систематизируем наблюдения о влиянии первичной и вторичной структур предшественников микроРНК животных на их функции и биогенез. Также затрагиваются проблемы генетической и биохимической изменчивости микроРНК. Представленные данные будут полезны для понимания организации, регуляции и эпигенетики микроРНК.
ТРАНСКРИБИРОВАНИЕ прай-миРНК
Процесс созревания микроРНК животных начинается (рис. 2) с транскрибирования РНК-полимеразой II (или РНК-полимеразой III для некоторых микроРНК) длинного транскрипта (первичной микроРНК, прай-миРНК), который содержит одну или несколько шпилек предшественников микроРНК (пре-миРНК), m7G-кэп и поли(А)-хвост [5, 6]. Поли(А)-хвост может отсутствовать в случаях, когда обработка прай-миРНК микропроцессорным комплексом начинается раньше окончания транскрибирования [7].
Рис. 2.
Общая схема путей биогенеза микроРНК животных. Предшественники микроРНК (прай-миРНК) транскрибируются РНК-полимеразами II или III. Канонический путь биогенеза включает в себя процессинг первичных РНК комплексами Drosha и Dicer. Неканонические пути биогенеза включают в себя Dicer- и Drosha-независимые шаги созревания микроРНК.

Гены микроРНК находятся в различных геномных областях: в интронах некодирующих и белок-кодирующих транскриптов, экзонах или межгенных областях; гены транскрибируются независимо или как часть белок-кодирующих генов-хозяев [8]. Многие последовательности микроРНК обнаружены на небольшом расстоянии друг от друга (~3–50 тпн), некоторые из них формируют полицистронные транскрипционные единицы (например, miR-100/let-7/miR-125, кластер miR-17/92) [8, 9], а другие (например, miR-30a/miR-30c-2) – нет [10]. Некоторые микроРНК находятся в обеих цепях ДНК и комплементарны друг другу, например miR-3120 и miR-214 [11]. Те микроРНК, которые располагаются рядом, могут транскрибироваться вместе. Однако посттранскрипционно они могут функционировать и подвергаться регулированию как вместе, так и независимо через внешние механизмы, проявляя разную активность в разных тканях и стадиях созревания организма [12, 13]. Помимо внешнего регулирования изменения последовательности одной из микроРНК кластера могут приводить к изменению уровней экспрессии соседних микроРНК [14].
Как и белок-кодирующие гены, транскрипция микроРНК может регулироваться транскрипционными факторами (ТФ), усиливающими или блокирующими процессинг прай-миРНК [15]. При этом, когда экспрессия самих транскрипционных факторов находится под контролем микроРНК, образуются регуляторные контуры [16, 17]. Эти контуры являются частью общей сети регуляции экспрессии генов [18].
Помимо ТФ процессинг прай-миРНК может зависеть от статуса метилирования промоторов генов, модификаций гистонов или модификаций концов РНК [19–21]. Изменения нуклеотидной последовательности мРНК, такие как замены A>I, мутации или варианты однонуклеотидного полиморфизма, оказывают влияние на процессинг прай-/пре-/микроРНК через трансформацию структуры предшественника и стабильности связывания с мишенями [21, 22].
ПРОЦЕССИНГ прай-миРНК В ЯДРЕ
После транскрибирования в каноническом пути биогенеза (рис. 2) прай-миРНК животных разрезается микропроцессорным комплексом из РНКазы III Drosha и РНК-связывающего белка DGCR8 (Pasha у D. melanogaster и C. elegans) [3]. Комплекс связывается с шпилечной структурой и вырезает шпильку пре-миРНК длиной ~65–70 нуклеотидов (нт) на расстоянии ~11 нт от однонитевых хвостов РНК и на расстоянии ~22 нт от терминальной петли [23]. Границы между одно- и двунитевыми фрагментами РНК выступают сигналами для процессинга Drosha. Каждый из доменов РНКазы III (RIIIDa и RIIIDb), входящих в Drosha, разрезает одну из двух ветвей шпильки таким образом, что получается пре-миРНК с 3'-выступающим концом [24]. Каждое разрезание определяет один из двух концевых нуклеотидов будущих микроРНК.
Превращение прай-миРНК в пре-миРНК может регулироваться, во-первых, взаимодействием белков с компонентами микропроцессорного комплекса. Например, белок р53 в кооперации с другими белками (р68, p72 и др.) регулирует вырезание пре-миРНК mir-16, mir-143, mir-145 и других [25, 26]. Во-вторых, регуляторами могут выступать структура прай-миРНК, а также другие РНК [22, 27]. В самой прай-миРНК присутствуют следующие регуляторные элементы: терминальная петля, олигонуклеотидные мотивы в основании шпильки, в стебле и в однонитевых концах РНК (рис. 3). Одним из наиболее изученных элементов является терминальная петля. 14% прай-миРНК человека содержат консервативные нуклеотиды в терминальной петле [28]. Связываясь с терминальной петлей, белок hnRNP A1 может как облегчать процессинг прай-миРНК (увеличивая размер внутренней петли, pri-mir-18a), так и блокировать (pri-let-7) [29, 30]. Белок KSRP связывается с G-богатым участком в терминальной петле и способствует вырезанию pre-let-7, pre-mir-196a, pre-mir-21 и других пре-миРНК из транскрипта [31]. Конкуренция разнонаправленно действующих факторов KSRP и hnRNP A1 за связывание с терминальной петлей pre-let-7 определяет уровень экспрессии микроРНК [30]. Белок TDP-43 связывается с UG-богатыми терминальными петлями (но не с областью последовательности двунитевой РНК у pre-mir-143 и pre-mir-574) и помогает позиционированию микропроцессорного комплекса на пре-миРНК [32]. Белок YB-1 предпочитает связываться с мотивом UYAUC в терминальной петле pri/pre-mir-29b-2 человека, блокируя соединение микропроцессора и Dicer с предшественником [33]. Часть микропроцессорного комплекса, DGCR8, связывается с UGUG-мотивом в терминальной петле и способствует позиционированию всего комплекса на шпильке предшественника и ее разрезанию [34, 35]. Связывание белков с терминальной петлей прай-миРНК может быть частью регуляторных контуров: например, белок LIN28 и микроРНК let-7 регулируют друг друга, образуя отрицательную обратную связь [36].
Рис. 3.
Структурные особенности канонического предшественника микроРНК. Обычно прай-миРНК содержит два одноцепочечных конца, стебель с небольшими петлями и терминальную петлю. МикроРНК формируют дуплекс с двунуклеотидными 3'-выступающими концами (выделен серым). Также показаны олигонуклеотидные мотивы, которые могут регулировать процессинг и повышать точность разрезания Dicer или Drosha. Стрелками условно показан разброс в позициях разрезания.
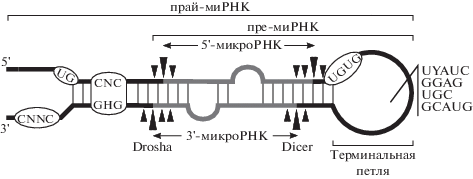
Кроме терминальной петли прай-миРНК содержат и другие сигналы для белков. Белки R‑SMAD содействуют функции Drosha через мотив R-SBE (CAGAC) в двунитевом участке шпильки пре-миРНК [37]. Белок BRCA1 связывается с основанием шпильки пре-миРНК и подавляет созревание микроРНК miR-155; с другой стороны, подобное связывание ускоряет процессинг let-7a-1, miR-16-1, miR-145 и miR-34 [38].
Факторы сплайсинга SF2/ASF связываются со стеблем прай-миРНК и помогают обрабатывать pri-mir-7; в свою очередь, miR-7 блокирует экспрессию этих факторов [39]. Другой фактор сплайсинга SRp20 (также известный как SRSF3) связывается с CNNC-мотивом (где N – любой нуклеотид из A, U, G, C). Мотив расположен на расстоянии 16–18 нт от сайта разрезания Drosha (рис. 3) и увеличивает уровень экспрессии пре-миРНК [35]. Этот мотив также требуется для РНК-хеликазы p72, которая соединяет микропроцессор с другими белками и тем самым способствует процессингу [40]. Еще два регуляторных мотива, способствующих процессингу, обнаружены в прай-миРНК (рис. 3): UG (~13–14 нт от 5'-конца 5'-микроРНК) и GHG (где H – это A, или C, или U; в двунитевом участке ~11 нт до сайта разрезания Drosha) [41, 42].
Перечисленные выше мотивы (UG и CNNC, GHG и UGUG) в прай-миРНК помогают ориентировать микропроцессорный комплекс относительно прай-миРНК с тем, чтобы резать возле основания шпильки, а не терминальной петли, а также могут компенсировать небольшие структурные дефекты пре-миРНК (маленькие петли и отклонение стебля от оптимальной длины) [34, 42]. Эти мотивы широко распространены: CNNC/UG во фланкирующих однонитевых РНК-сегментах или UGUG в терминальной петле встречаются в 79% прай-миРНК человека [41]. При этом у млекопитающих достаточно присутствия не всех, а только некоторых мотивов [43].
РНК-РНК-взаимодействия также регулируют созревание микроРНК. Зрелая микроРНК мыши miR-709 связывается с однонитевым участком вблизи пре-миРНК pri-mir-15a/pri-mir-16-1, блокируя процессинг прай-миРНК [44]. Прай-миРНК let-7 и ее зрелая микроРНК связаны положительной обратной связью, когда let-7 связывается с консервативным комплементарным сайтом в 3'‑конце транскрипта прай-миРНК [45].
Микропроцессорный комплекс может напрямую деградировать мРНК: Drosha блокирует экспрессию FSTL1-белка путем вырезания шпильки пре-миРНК pre-mir-198 в 3'-нетранслируемом регионе (НТР) мРНК белка [46]. В 5'-НТР мРНК DGCR8 (ко-фактора Drosha) находится шпилька предшественника pre-mir-1306, которая является частью обратной регуляторной петли между Drosha и DGCR8 [47]. Избыток микроРНК miR-128-3p приводит к уменьшению уровней присутствия Drosha и Dicer, тем самым подавляя процессинг всех микроРНК и приводя к развитию рака легких [48]. Таким образом, miR-1306/miR-198/miR-128 могут быть как функциональными микроРНК, так и дополнительными цис-регуляторными элементами.
В целом микропроцессорный комплекс создает богатый ассортимент шпилек, часть из которых отфильтровывается на следующих стадиях созревания микроРНК, в частности по их неподходящей длине при транспортировке из ядра в цитоплазму.
ТРАНСПОРТИРОВКА ИЗ ЯДРА В ЦИТОПЛАЗМУ
После процессинга в ядре полученная шпилька пре-миРНК (рис. 2) перемещается в цитоплазму с помощью белка Exportin-5 (XPO5) и его ко-фактора Ran-GTP [49]. В некоторых случаях в транспортировке могут участвовать и другие комплексы, например Exportin-1 (XPO1) для mir-320/mir-484 и других пре-миРНК с m7G-кэпом на 5'-конце, сформированном непосредственно транскрипцией [50, 51]. Эти комплексы не только перемещают шпильку пре-миРНК, но и защищают ее от деградации клеточными экзонуклеазами. Транспортный комплекс специфичен к форме предшественника и захватывает его как “бейсбольная перчатка”, связываясь с двойной спиралью, терминальной петлей и выступающим 3'-концом [52, 53]. Таким образом транспортный комплекс может перемещать любые малые шпилечные РНК (включая вирусные или случайные), а эти шпилечные РНК – регулировать экспрессию микроРНК через конкуренцию с пре-миРНК за транспортный комплекс [54]. При генетических повреждениях элементов транспортного комплекса его структура не соответствует структуре предшественника, а сам он не способен захватывать и перемещать пре-миРНК, что, в свою очередь, приводит к накоплению пре-миРНК в ядре и уменьшению уровня экспрессии микроРНК [55]. Также вирусные микроРНК могут блокировать транспорт, как конкурируя с реальными микроРНК, так и адресуясь к мРНК элементов транспортного комплекса, например Ran-GTP [56].
ПРОЦЕССИНГ пре-миРНК DICER И ЕГО КО-ФАКТОРАМИ
После перемещения в цитоплазму пре-миРНК связывается с белковым комплексом Dicer (рис. 2), который разрезает шпильку предшественника возле терминальной петли и производит дуплекс микроРНК–микроРНК с выступающими 3'-концами [3]. Таким образом, на этом этапе формируется второй конец микроРНК. Обычно Dicer состоит из следующих доменов: хеликаза, PAZ, α-спираль и две РНКазы III [57, 58]. Хеликаза формирует зажимоподобную структуру, которая прилегает к РНКазам, облегчает распознавание терминальной петли пре-миРНК, перемещает и фиксирует шпильку предшественника [58]. Домен PAZ содержит два кармана для связывания с концами пре-миРНК [57, 59]. Каждая из двухРНКаз III разрезает одну из двух ветвей пре-миРНК и освобождает дуплекс микроРНК от терминальной петли. PAZ и РНКазы III соединяются α-спиралью, длина которой устанавливает расстояние между этими доменами и, таким образом, размер производимого дуплекса микроРНК [57].
Существенно, что Dicer может процессировать короткие шпилечные РНК независимо от их нуклеотидной последовательности [60]. Связывающие карманы в домене PAZ располагаются на расстоянии, которое соответствует длине двух нуклеотидов 3'-выступающего конца, как раз такого, который производит Drosha. Однако, задействовав только один карман, Dicer может обрабатывать шпильки без выступающего 3'-конца или с выступающим 5'-концом.
Строение Dicer определяет расположение сайта разрезания. Для большинства пре-миРНК этот сайт располагается на фиксированной дистанции от 3'-выступающего конца (так называемое “3'-counting rule”) [59]. Dicer может распознавать также фосфатную группу 5'-конца шпильки и разрезать пре-миРНК на расстоянии ~22 нт от этого конца [61]. Это так называемое “5'-counting rule” наблюдается в основном тогда, когда стебель у основания шпильки не замыкается сильной парой G-C [61]. Наблюдаемые отклонения от этих двух правил обусловлены модификациями пре-миРНК, такими как изменение длины концов пре-миРНК, которое сдвигает сайт разрезания Dicer и сид-регион микроРНК. Модификации 3'-концов происходят чаще, нежели 5'-концов, что делает “5'-countig rule” более важным для пре-миРНК с 3'-модифицированным концом.
Как и у Drosha, процессинг у Dicer регулируется разными способами. Важным цис-регуляторным элементом как активации, так и репрессии этапа разрезания Dicer является терминальная петля пре-миРНК. Например, белки KSRP и TDP-43 контролируют активность Dicer путем связывания с терминальной петлей [31, 32]. Белки LIN28A и LIN28B, распознавая мотив GGAG в терминальной петле (например, у let-7, miR-107, miR-143, miR-200c и других), притягивают к ней белок из семейства терминальных уридилтрансфераз (например, TUT4 или TUT7) [62]. Трансфераза добавляет олигонуклеотидный U-хвост к 3'-концу пре-миРНК: это блокирует процессинг Dicer и приводит к деградации пре-миРНК, в частности, когда выступающий конец до своего наращивания имел каноническую длину два нуклеотида [63]. Подобный способ блокирования процесса созревания микроРНК через уридилирование 3'-конца шпильки присутствует и у Drosophila [64], при этом трансфераза Tailor распознает и уридилирует пре-миРНК с гуанином на 3'-конце, что может быть причиной избегания гуанина на этом конце в наблюдаемых микроРНК [65]. И наоборот, для однонуклеотидного конца TUT4 восстанавливает его каноническую длину (например, у let-7 и miR-105), что в дальнейшем способствует их каноническому процессингу [66].
Другой мотив внутри терминальной петли пре-миРНК, UGC, связывается с белком MBNL1, что помогает в разрезании предшественника [67]. MBNL1 конкурирует с LIN28 за связывание с терминальной петлей и таким образом защищает пре-миРНК от наращивания 3'-конца [67]. Мотив GCAUG (рис. 3) в терминальной петле связывается с белками Rbfox и подавляет процесс созревания микроРНК человека miR-20b, miR-107 и других [68].
Помимо белков на процессинг Dicer могут влиять и различные РНК: в C. elegans некодирующая РНК rncs-1 может заменять эндогенные двунитевые РНК для связывания с Dicer [69]. МикроРНК могут связываться с мРНК Dicer и таким образом регулировать свою собственную экспрессию через обратные связи (например, let-7, miR-BART6-5p для Dicer человка) [70].
Последовательность и структура пре-миРНК определяют не только скорость, но и качество разрезания предшественника. Точность разрезания Dicer подчиняется так называемому правилу “loop-counting rule”: разрезание 3'-ветви пре-миРНК точнее, когда на расстоянии два нуклеотида выше от сайта разрезания в 3'–5'-направлении располагается петля [71]. В других случаях Dicer производит продукты с вариабельными 5'‑концами 3'-микроРНК [71].
ОБРАЗОВАНИЕ miRISC, AGO-ПРОЦЕССИНГ
После образования дуплекса микроРНК–микроРНК он вместе с одним из белков семейства аргонавтов (AGO) и ко-факторами создает микроРНК-индуцируемый комплекс выключения гена (от англ. miRISC). Этот комплекс разворачивает дуплекс и выбирает ту из его ветвей, которая впоследствии свяжется с мРНК [72]. В процессе разворачивания дуплекса одна из его ветвей (так называемая “направляющая”) чаще используется для формирования зрелого комплекса miRISC, в то время как другая, “пассажирская”, ветвь расщепляется или быстро деградирует. Соотношение фракций “направляющей” и “пассажирской” микроРНК определяется термодинамической стабильностью и концевым нуклеотидом или может варьироваться в зависимости от стадии развития организма, его пола, ткани (в которых происходит экспрессия) или ориентации дуплекса при загрузке в комплекс [60, 73]. Белки AGO предпочитают ветвь с нуклеотидами A или U на 5'-конце или ту ветвь дуплекса, у которой замыкающая пара на этом конце менее стабильна [74]. С другой стороны, смена ветви может вызываться дестибилизирующим [75] редактированием дуплекса, причем это редактирование чаще наблюдается в сид-регионе микроРНК (у человека и мыши, но не у D. melanogaster) [76]. Также ветвь может смениться из-за смещения пре-миРНК в мРНК, так называемого “сдвига шпильки” [77].
AGO-белки и выполняемые ими функции различаются в зависимости от организма [78]. У некоторых организмов есть специализация AGO-белков по виду малых РНК в зависимости от характеристик их дуплексов. Например, у Drosophila Ago1 предпочитает связываться с дуплексом, в котором на 5'-конце “направляющей” микроРНК находится урацил, в то время как Ago2 предпочитает дуплекс микроРНК с цитозином на этом же конце [79]. У человека связывание микроРНК c белками семейства Argonaute (Ago2 и Ago3) коррелирует с содержанием тетрануклеотидов YRHB вблизи их 3'-конца, а связывание микроРНК с белком Ago2 коррелирует с содержанием тетрануклеотидов RHHK в центре последовательности [80]. Петли в 9–10-й позициях “направляющей” микроРНК Drosophila направляют дуплекс микроРНК непосредственно в Ago1, но не позволяют ему обрабатываться Ago2 [81]. То есть при отсутствии петель “пассажирская” микроРНК разрезается, а “направляющая” включается в комплекс с Ago2. В обратном случае при наличии петель в центре дуплекса “пассажирская” ветвь деградирует, а “направляющая” – включается в Ago1. Однако у человека влияние петли в середине дуплекса микроРНК на выбор AGO-белка отсутствует [82]. Смена ветви может закрепиться эволюционно с новыми функциями микроРНК [77].
НЕКАНОНИЧЕСКИЕ ПУТИ БИОГЕНЕЗА
Кроме описанного выше канонического пути биогенеза, микроРНК образуются и другими, Dicer- и Drosha-независимыми путями (рис. 2). Большинство из неканонических микроРНК составляют миртроны, которые пропускают шаг процессирования мРНК Drosha, а их предшественники образуются через сплайсинг, расплетение петли интрона и перестройку в каноническую шпилечную структуру [83]. Некоторые из миртронов содержат дополнительные нуклеотиды на одном или обоих концах шпильки, которые отличают структуру от канонической. Впоследствии эти нуклеотиды удаляются экзонуклеазами, а созревание микроРНК продолжается по каноническому пути [83].
Хотя к настоящему моменту обнаружены сотни миртронов [84, 85], особенности их созревания изучены слабее, чем для канонических микроРНК. В среднем шпилька пре-миРНК миртронов немногим длиннее, чем канонические предшественники [85]. После сплайсинга большинство из предшественников миртронов содержат длинный выступающий конец (так называемые “tailed-миртроны”) из нуклеотидов между сайтом сплайсинга и основанием шпильки пре-миРНК [84]. Укорачивание экзонуклеазами хвостов этих миртронов может смещать границы микроРНК [86]. Во многих миртронах без выступающих концов и в миртронах с 3'-выступающим концом (так называемых “3'-tailed миртронах”) с 5'-конца дополнительно удаляется гуанин, который находится на границе интрона и экзона [85]. Это удаление гуанина может быть дополнительным механизмом повышения точности разрезания пре-миРНК РНКазой III Dicer [87].
Типичная структура предшественников миртронов несет отпечатки особенностей их биогенеза и отличается от структуры канонических пре-миРНК: выступающий конец в основании шпильки чаще состоит из одного, а не двух нуклеотидов [87]. По сравнению с каноническими микроРНК у миртронов выше плотность вариантов однонуклеотидного полиморфизма (ОНП), а сам ОНП обратно ассоциирован с болезнями. Вероятно, миртроны находятся под положительным (движущим) отбором, в то время как большинство из канонических микроРНК – под отрицательным (стабилизирующим); миртроны могут являться имманентным источником изменчивости, которая способствует возникновению болезней. Интересно, что участок точки ветвления при сплайсинге чаще располагается ровно на том месте, чтобы не быть экранированным структурой предшественников миртронов [87]. Таким образом миртроны могут легко возникать из случайных шпилек подходящего размера недалеко от 3'-сайта сплайсинга. В результате миртроны чаще появляются и быстрее исчезают, чем канонические микроРНК, даже если те и другие генерируют функционально идентичные регуляторные РНК [88].
Биогенез микроРНК млекопитающих miR-1225 и miR-1228, первоначально охарактеризованных как миртроны, на самом деле не зависит от сплайсинга [89]. Но их дальнейший биогенез не требует канонических компонент DGCR8, Dicer, Exportin-5 или AGO, а только Drosha [90]. Этот класс микроРНК назван симтронами (от англ. simtron, сплайсинг-независимые миртроно-подобные РНК). Некоторые интроны содержат одновременно миртроны и симтроны, а получившиеся микроРНК – результат выбора одного из путей биогенеза [89].
Совсем недавно был обнаружен еще один сплайсинг-зависимый класс микроРНК – аготроны (от англ. agotron), чей биогенез включает интеграцию микроРНК в miRISC и взаимодействие с белками семества Argonaute, но не требует шага разрезания Dicer [91].
Другой Dicer-независимый путь наблюдается для семейства микроРНК miR-451, в котором каталитическую функцию Dicer берет на себя белок Ago2 [92, 93]. Drosha порождает предшественника микроРНК pre-mir-451 со слишком коротким для процессирования Dicer, ~18 нуклеотидным, стеблем. Белок Ago2 разрезает этот стебель в середине 3'-ветви и производится ~30-нуклеотидный РНК-продукт [92, 93]. Затем поли(А)-специфичная рибонуклеаза PARN отрезает 3'-конец у этого продукта и освобождает зрелую 5'-микроРНК, при этом разницы между уровнем активности этой микроРНК и изначального 30-нуклеотидного продукта не обнаружено [94]. Также в противовес другим пре-миРНК терминальная петля не влияет на созревание miR-451 [94]. Белок Ago2 может заменить Dicer не только для коротких шпилек, но и при его отсутствии; и наоборот, предшественник miR-451 с восстановленной канонической длиной стебля может процессироваться посредством Dicer [95]. Таким образом Ago2- и Dicer-пути оказываются для miR-451 взаимозаменяемыми.
МУТАЦИИ, ОДНОНУКЛЕОТИДНЫЙ ПОЛИМОРФИЗМ, РЕДАКТИРОВАНИЕ A>I
Изменения в последовательности прай-/пре-/ микроРНК могут влиять на вторичную структуру предшественника и таким образом изменять уровень экспрессии и функции микроРНК. Нуклеотидные замены чаще наблюдаются в центре микроРНК и в окрестности ее границ в предшественнике, а районы, отвечающие за адресацию микроРНК, так называемый сид-регион (позиции 2–8 микроРНК) и дополнительный сид-регион (позиции 13–16), более консервативны [96]. Эти два района характеризуются также наименьшим присутствием петель [87, 96]: подобная связь интенсивности мутагенеза со вторичной структурой характерна для структурных РНК, например тРНК [97].
Однонуклеотидный полиморфизм – один из типов замен в ДНК, который влияет на процесс созревания и функцию микроРНК. ОНП в пре-миРНК человека может подавлять или повышать экспрессию микроРНК на шаге разрезания Drosha (например, miR-30c, miR-125a, miR-146a, miR-510 и др.) или Dicer (например, miR-196a, miR-196a и др.) [22, 98]. Замена C>T в первой позиции мотива CNNC замедляет процессинг Drosha pri-mir-16-1 [41]. Кроме того, ОНП внутри микроРНК (особенно внутри сид-региона) или внутри мишени влияет на функцию микроРНК [99]. Замена в первом нуклеотиде miR-934-5p человека приводит к изменению сайтов разрезания Dicer и Drosha, а также к смене ветви дуплекса, задействованной в комплексе miRISC [100].
Мутации в генах белков комплексов процессинга микроРНК (например, Dicer, Drosha или Exportin-5) могут нарушать функции этих комплексов [55, 101].
Еще один способ регулировать созревание микроРНК – это редактирование РНК [102]. Фермент ADAR заменяет аденозин (A) на инозин (I), что может как блокировать разрезание Drosha или Dicer, так и способствовать ему [103]. Модификации последовательности прай-/пре-миРНК (например, в pri-mir-151 и в кластере miR-376 человека и мыши) приводят к изменению стабильности структуры прай-миРНК, мишеней микроРНК и могут блокировать шаги разрезания Dicer [76, 104, 105]. Кроме того, такое редактирование одной ветви дуплекса микроРНК может приводить к выбору второй в качестве функциональной [76].
Замечательно, что, хотя некоторые из записей в базах данных ОНП являются на самом деле событиями редактирования РНК [106], плотность редактирования A>I ведет себя антибатно относительно плотности ОНП и чаще наблюдается в сид-регионе для микроРНК человека и мыши (но не D. melanogaster) [76].
НЕОДНОРОДНОСТЬ ГРАНИЦ И ОШИБКИ АННОТАЦИИ микроРНК
В каноническом биогенезе Drosha и Dicer часто режут одну и ту же шпильку предшественников в нескольких соседних позициях (рис. 3) и производят таким образом микроРНК с разными границами, так называемые изомиры (от англ. isoMiRs) [107]. Точность разрезания Drosha чувствительна к мотивам последовательности прай-миРНК и контролируется DGCR8 [35]. Домен RIIIDa Dicer режет более точно, и он более чувствителен как ко вторичной структуре, так и к нуклеотидной последовательности своего субстрата, чем домен RIIIDb [65, 71]. В результате 5'‑граница микроРНК (та граница, что прилегает к сид-региону) оказывается более однородной, а неоднородность разрезания проявляется в основном в вариабельности длин выступающих 3'-концов [65, 108]. Сходство строения Dicer и Drosha [109], а также распределение длин выступающих концов дуплекса микроРНК [87] позволяют предположить, что ровно то же самое верно и для Drosha. Длины выступающих концов взаимозависимы, что послужило основанием для двухрычажной модели динамики режущего комплекса Dicer [87].
Смещение границ микроРНК в предшественнике может приводить к изменению мишеней, тем самым расширяя функциональный репертуар микроРНК [110, 111]. Выступающие концы дополнительно могут изменяться в обе стороны аденилированием, уридилированием и активностью экзонуклеаз, что приводит к дополнительной неоднородности (в большинстве случаев 3') границ микроРНК [112–114]. Таким образом, 5'-граница микроРНК, которая отвечает за выбор мишени, оказывается более однородной. Однако для некоторых микроРНК, например miR‑79, miR‑193, miR‑210 у D. melanogaster и miR‑124, miR‑133a, miR‑223 у M. musculus [77], два и более фрагмента микроРНК (изомира) наблюдаются на уровнях, сравнимых с главной формой. Смена главной формы, вызванная смещением 5'-границы (рис. 4), так называемый сдвиг сид-региона, приводит к смене мишеней и иногда наблюдается в эволюции [115]. Стоит отметить, что только около трети аннотированных микроРНК из базы miRBase (релиз 21.0) идентичны главной форме своих изомиров, при этом около 37% микроРНК из базы (рис. 4) никогда не были самыми наблюдаемыми из изомиров [116].
Рис. 4.
Пример смены главной формы изомиров и сдвига сид-региона в D. melanogaster. Данные получены из базы miRBase. В последовательности пре-миРНК серым цветом выделена аннотированная микроРНК. Последовательности ридов подчеркнуты, приведены только фракции с минимальным количеством наблюдений 10 000. В dme-mir-79 наблюдаются фракции ридов с разными начальными позициями. Каждая из этих позиций эволюционно зафиксирована в предшественниках dme-mir-9c и dme-mir-4 в виде микроРНК с соответствующим сдвигом сид-региона на один нуклеотид. Заметим, что в miRBase главная фракция ридов не всегда идентична аннотированной микроРНК (например, dme-mir-9c-3p и dme-mir-4-3p), что может быть ошибками базы miRBase.

В отличие от канонических микроРНК граница миртронов более вариабельна и служит дополнительным фактором гетерогенности зрелого продукта. В результате сплайсинга у миртронов могут образовываться дополнительные выступающие концы, которые в дальнейшем активно укорачиваются 3'–5'- и 5'–3'-экзонуклеазами [84]. В большинстве миртронов шпилька пре-миРНК находится у 3'-сайта сплайсинга и потому имеет длинный выступающий 5'-конец, отрезание которого определяет 5'-границу 5'-микроРНК и выбор ее мишени. У остальных миртронов 5'-граница их 5'-микроРНК часто определяется сплайсингом и последующей потерей концевого гуанина [85]. На противоположной ветви пре-миРНК терминальная уридилтрансфераза Tailor распознает гуанин на 3'-конце сайта сплайсинга (3'‑AG) и уридилирует этот конец предшественника, что блокирует дальнейшее созревание микроРНК в Drosophila [64]. Таким образом, хотя в биогенезе миртронов отсутствует Drosha, они, так же как и канонические пре-миРНК [65], избегают гуанина на 5' (3')-конце своих 5' (3')-микроРНК, соответственно. Это позволяет предположить, что гуанин на конце пре-миРНК оказывает влияние на обработку предшественника Dicer.
Изучение уровня экспрессии микроРНК и уровня деградации РНК в разных тканях показывает, что микроРНК, добавленные в последних релизах базы miRBase, демонстрируют повышенную экспрессию при высокой степени деградации РНК и поэтому могут быть артефактами [117]. Такие ложноположительные последовательности, а также неоднородность границ микроРНК приводят к появлению ошибок аннотации в базах данных. Определение и верификация микроРНК осложнены их малым размером и некодирующим происхождением. Развитие методов глубокого секвенирования и биоинформатических алгоритмов для обработки их результатов привело к быстрому росту числа кандидатов микроРНК, в том числе и ложных. Для отсечения ложноположительных микроРНК применяют следующие критерии: эволюционная консервативность, экспериментальная подтвержденность дуплекса микроРНК с выступающими двухнуклеотидными 3'‑концами, структурное подобие последовательностей, однородность 5'-края микроРНК [118]. Этим критериям удовлетворяет только одна треть микроРНК человека и около одной шестой части микроРНК животных из базы miRBase: из них была сформирована база MirGeneDB с условно-достоверными микроРНК [118]. Описанным выше критериям не удовлетворяют почти все неканонические микроРНК.
Ложноположительные микроРНК могут порождаться шумом транскрипции, другими видами малых РНК, тРНК и транспозонами. Еще одной причиной ошибки является загрязнение данных при эксперименте, в том числе присутствие микроРНК из другого организма (так называемые экзогенные микроРНК, или “ксеномиры”, от англ. xenomiR), например растений у мыши и человека [119]. До сих пор ксеномиры не наблюдались в воспроизводимых экспериментах, а результаты тех экспериментов, в которых они наблюдались, отсутствуют в свободном доступе, что не дает возможности независимо контролировать их чистоту [120].
ЗАКЛЮЧЕНИЕ
К настоящему моменту стало известно многое о микроРНК, их биогенезе и функциях, однако некоторые вопросы остаются открытыми. Вхождение микроРНК в различные регуляторные контуры, а также влияние их первичной и вторичной структур на биогенез и функции дают богатый репертуар способов контроля над клеточными процессами и, очевидно, мы знаем только малую их часть. Требуют дальнейшего изучения редкие биогенезы микроРНК, структурные изменения прай-/пре-/микроРНК и их последствия, а также ксеномиры и изомиры. Неизвестно значение избыточного количества последовательностей микроРНК при том, что каждая из микроРНК может присутствовать в нескольких слабо различающихся формах. Имманентная биохимическая вариабельность микроРНК расширяет их функциональный репертуар и может приводить к генетической изменчивости самих микроРНК и систем контроля над ними, в частности механизмов редактирования. Благодаря своим свойствам микроРНК занимают важное, возможно центральное, место в эпигенетике РНК, иначе известной как эпитранскриптомика.
Работа поддержана государственным бюджетным проектом 0324-2019-0040.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Kutter C., Svoboda P. miRNA, siRNA, piRNA: Knowns of the unknown // RNA Biology. 2008. V. 5. № 4. P. 181–188. https://doi.org/10.4161/rna.7227
Lee Y., Jeon K., Lee J.-T. et al. MicroRNA maturation: stepwise processing and subcellular localization // EMBO J. 2002. V. 21. № 17. P. 4663–4670.
O’Brien J., Hayder H., Zayed Y. et al. Overview of microRNA biogenesis, mechanisms of actions, and circulation // Frontiers in Endocrinology. 2018. V. 9. № 402. P. 1–12. https://doi.org/10.3389/fendo.2018.00402
Vidigal J.A., Ventura A. The biological functions of miRNAs: lessons from in vivo studies // Trends in Cell Biology. 2015. V. 25. № 3. P. 137–147. https://doi.org/10.1016/j.tcb.2014.11.004
Borchert G.M., Lanier W., Davidson B.L. RNA polymerase III transcribes human microRNAs // Nat. Struct. Mol. Biol. 2006. V. 13. № 12. P. 1097–1101. https://doi.org/10.1038/nsmb1167
Cai X., Hagedorn C.H., Cullen B.R. Human microRNAs are processed from capped, polyadenylated transcripts that can also function as mRNAs // RNA. 2004. V. 10. № 12. P. 1957–1966. https://doi.org/10.1261/rna.7135204
Ballarino M., Pagano F., Girardi E. et al. Coupled RNA processing and transcription of intergenic primary microRNAs // Mol. Cell. Biol. 2009. V. 29. № 20. P. 5632–5638. https://doi.org/10.1128/MCB.00664-09
Marco A., Ninova M., Griffiths-Jones S. Multiple products from microRNA transcripts // Biochem. Soc. Trans. 2013. V. 41. № 4. P. 850–854. https://doi.org/10.1042/BST20130035
Titov I.I., Vorozheykin P.S. Analysis of miRNA duplication in the human genome and the role of transposon evolution in this process // Russian Journal of Genetics: Applied Research. 2011. V. 1. № 4. P. 308–314. https://doi.org/10.1134/S2079059711040083
Chang T.-C., Pertea M., Lee S. et al. Genome-wide annotation of microRNA primary transcript structures reveals novel regulatory mechanisms // Genome Res. 2015. V. 25. № 9. P. 1401–1409. https://doi.org/10.1101/gr.193607.115
Scott H., Howarth J., Lee Y.B. et al. MiR-3120 is a mirror microRNA that targets heat shock cognate protein 70 and auxilin messenger RNAs and regulates clathrin vesicle uncoating // J. Biol. Chem. 2012. V. 287. № 18. P. 14726–14733. https://doi.org/10.1074/jbc.M111.326041
Abasi M., Kohram F., Fallah P. et al. Differential maturation of miR-17 ~92 cluster members in human cancer cell lines // Appl. Biochem. Biotechnol. 2017. V. 182. № 4. P. 1540–1547. https://doi.org/10.1007/s12010-017-2416-5
Wang Y., Luo J., Zhang H. et al. MicroRNAs in the same clusters evolve to coordinately regulate functionally related genes // Mol. Biol. Evol. 2016. V. 33. № 9. P. 2232–2247. https://doi.org/10.1093/molbev/msw089
Lataniotis L., Albrecht A., Kok F.O. et al. CRISPR/ Cas9 editing reveals novel mechanisms of clustered microRNA regulation and function // Scientific Reports. 2017. V. 7. № 8585. P. 1–14. https://doi.org/10.1038/s41598-017-09268-0
Tong Z., Cui Q., Wang J. et al. TransmiR v2.0: an updated transcription factor-microRNA regulation database // Nucl. Acids Res. 2019. V. 47. № D1. P. D253–D258. https://doi.org/10.1093/nar/gky1023
Ben-Ami O., Pencovich N., Lotem J. et al. A regulatory interplay between miR-27a and Runx1 during megakaryopoiesis // Proc. Natl. Acad. Sci. USA. 2009. V. 106. № 1. P. 238–243. https://doi.org/10.1073/pnas.0811466106
Wang Y., Liang H., Zhou G. et al. HIC1 and miR-23~27~24 clusters form a double-negative feedback loop in breast cancer // Cell Death & Differentiation. 2017. V. 24. № 3. P. 421–432. https://doi.org/10.1038/cdd.2016.136
Shalgi R., Lieber D., Oren M. et al. Global and local architecture of the mammalian microRNA–transcription factor regulatory network // PLoS Computat. Biol. 2007. V. 3. № 7. P. 1291–1304. https://doi.org/10.1371/journal.pcbi.0030131
Barros-Silva D., Costa-Pinheiro P., Duarte H. et al. MicroRNA-27a-5p regulation by promoter methylation and MYC signaling in prostate carcinogenesis // Cell Death & Disease. 2018. V. 9. № 167. P. 1–15. https://doi.org/10.1038/s41419-017-0241-y
Munoz-Tello P., Rajappa L., Coquille S. et al. Polyuridylation in eukaryotes: a 3'-end modification regulating RNA life // BioMed Res. Intern. 2015. V. 2015. P. 1–12. https://doi.org/10.1155/2015/968127
Zhao B.S., Roundtree I.A., He C. Post-transcriptional gene regulation by mRNA modifications // Nat. Rev. Mol. Cell Biol. 2017. V. 18. № 1. P. 31–42. https://doi.org/10.1038/nrm.2016.132
Fernandez N., Cordiner R.A., Young R.S. et al. Genetic variation and RNA structure regulate microRNA biogenesis // Nat. Communicat. 2017. V. 8. № 15114. P. 1–12. https://doi.org/10.1038/ncomms15114
Nguyen T.A., Jo M.H., Choi Y.-G. et al. Functional anatomy of the human microprocessor // Cell. 2015. V. 161. № 6. P. 1374–1387. https://doi.org/10.1016/j.cell.2015.05.010
Kwon S.C., Nguyen T.A., Choi Y.-G. et al. Structure of Human DROSHA // Cell. 2016. V. 164. № 1–2. P. 81–90. https://doi.org/10.1016/j.cell.2015.12.019
Suzuki H.I., Yamagata K., Sugimoto K. et al. Modulation of microRNA processing by p53 // Nature. 2009. V. 460. № 7254. P. 529–533. https://doi.org/10.1038/nature08199
Connerty P., Ahadi A., Hutvagner G. RNA binding proteins in the miRNA pathway // Intern. J. Mol. Sci. 2015. V. 17. № 31. P. 1–16. https://doi.org/10.3390/ijms17010031
Treiber T., Treiber N., Plessmann U. et al. A compendium of RNA-binding proteins that regulate microRNA biogenesis // Mol. Cell. 2017. V. 66. № 2. P. 270–284. https://doi.org/10.1016/j.molcel.2017.03.014
Michlewski G., Guil S., Semple C.A. et al. Posttranscriptional regulation of miRNAs harboring conserved terminal loops // Mol. Cell. 2008. V. 32. № 3. P. 383–393. https://doi.org/10.1016/j.molcel.2008.10.013
Jean-Philippe J., Paz S., Caputi M. hnRNP A1: The swiss army knife of gene expression // Intern. J. Mol. Sci. 2013. V. 14. № 9. P. 18999–19024. https://doi.org/10.3390/ijms140918999
Michlewski G., Cáceres J.F. Antagonistic role of hn‑RNP A1 and KSRP in the regulation of let-7a biogenesis // Nat. Structural & Mol. Biol. 2010. V. 17. № 8. P. 1011–1018. https://doi.org/10.1038/nsmb.1874
Trabucchi M., Briata P., Garcia-Mayoral M. et al. The RNA-binding protein KSRP promotes the biogenesis of a subset of microRNAs // Nature. 2009. V. 459. № 7249. P. 1010–1014. https://doi.org/10.1038/nature08025
Kawahara Y., Mieda-Sato A. TDP-43 promotes microRNA biogenesis as a component of the Drosha and Dicer complexes // Proc. Natl. Acad. Sci. USA. 2012. V. 109. № 9. P. 3347–3352. https://doi.org/10.1073/pnas.1112427109
Wu S.-L., Fu X., Huang J. et al. Genome-wide analysis of YB-1-RNA interactions reveals a novel role of YB-1 in miRNA processing in glioblastoma multiforme // Nucl. Acids Res. 2015. V. 43. № 17. P. 8516–8528. https://doi.org/10.1093/nar/gkv779
Nguyen T.A., Park J., Dang T.L. et al. Microprocessor depends on hemin to recognize the apical loop of primary microRNA // Nucl. Acids Res. 2018. V. 46. № 11. P. 5726–5736. https://doi.org/10.1093/nar/gky248
Kim K., Nguyen T.D., Li S. et al. SRSF3 recruits DROSHA to the basal junction of primary microRNAs // RNA. 2018. V. 24. № 7. P. 892–898. https://doi.org/10.1261/rna.065862.118
Viswanathan S.R., Daley G.Q. Lin28: A microRNA regulator with a macro role // Cell. 2010. V. 140. № 4. P. 445–449. https://doi.org/10.1016/j.cell.2010.02.007
Davis B.N., Hilyard A.C., Nguyen P.H. et al. Smad proteins bind a conserved RNA sequence to promote microRNA maturation by Drosha // Mol. Cell. 2010. V. 39. № 3. P. 373–384. https://doi.org/10.1016/j.molcel.2010.07.011
Kawai S., Amano A. BRCA1 regulates microRNA biogenesis via the DROSHA microprocessor complex // The J. Cell Biol. 2012. V. 197. № 2. P. 201–208. https://doi.org/10.1083/jcb.201110008
Wu H., Sun S., Tu K. et al. A splicing-independent function of SF2/ASF in microRNA processing // Mol. Cell. 2010. V. 38. № 1. P. 67–77. https://doi.org/10.1016/j.molcel.2010.02.021
Ha M., Kim V.N. Regulation of microRNA biogenesis // Nat. Rev. Mol. Cell Biol. 2014. V. 15. № 8. P. 509–524. https://doi.org/10.1038/nrm3838
Auyeung V.C., Ulitsky I., McGeary S.E. et al. Beyond secondary structure: primary-sequence determinants license pri-miRNA hairpins for processing // Cell. 2013. V. 152. № 4. P. 844–858. https://doi.org/10.1016/j.cell.2013.01.031
Bartel D.P. Metazoan microRNAs // Cell. 2018. V. 173. № 1. P. 20–51. https://doi.org/10.1016/j.cell.2018.03.006
Fromm B., Domanska D., Hackenberg M. et al. MirGeneDB2.0: the curated microRNA Gene Database. 2018. https://doi.org/10.1101/258749
Tang R., Li L., Zhu D. et al. Mouse miRNA-709 directly regulates miRNA-15a/16-1 biogenesis at the posttranscriptional level in the nucleus: evidence for a microRNA hierarchy system // Cell Res. 2012. V. 22. № 3. P. 504–515. https://doi.org/10.1038/cr.2011.137
Zisoulis D.G., Kai Z.S., Chang R.K. et al. Autoregulation of microRNA biogenesis by let-7 and Argonaute // Nature. 2012. V. 486. № 7404. P. 541–544. https://doi.org/10.1038/nature11134
Sundaram G.M., Common J.E.A., Gopal F.E. et al. ‘See-saw’ expression of microRNA-198 and FSTL1 from a single transcript in wound healing // Nature. 2013. V. 495. № 7439. P. 103–106. https://doi.org/10.1038/nature11890
Han J., Pedersen J.S., Kwon S.C. et al. Posttranscriptional crossregulation between Drosha and DGCR8 // Cell. 2009. V. 136. № 1. P. 75–84. https://doi.org/10.1016/j.cell.2008.10.053
Frixa T., Sacconi A., Cioce M. et al. MicroRNA-128-3p-mediated depletion of Drosha promotes lung cancer cell migration // Carcinogenesis. 2018. V. 39. № 2. P. 293–304. https://doi.org/10.1093/carcin/bgx134
Yi R., Qin Y., Macara I.G. et al. Exportin-5 mediates the nuclear export of pre-microRNAs and short hairpin RNAs // Genes Dev. 2003. V. 17. № 24. P. 3011–3016. https://doi.org/10.1101/gad.1158803
Büssing I., Yang J.-S., Lai E.C. et al. The nuclear export receptor XPO-1 supports primary miRNA processing in C. elegans and Drosophila // The EMBO J. 2010. V. 29. № 11. P. 1830–1839. https://doi.org/10.1038/emboj.2010.82
Xie M., Li M., Vilborg A. et al. Mammalian 5'-capped microRNA precursors that generate a single microRNA // Cell. 2013. V. 155. № 7. P. 1568–1580. https://doi.org/10.1016/j.cell.2013.11.027
Zeng Y. Structural requirements for pre-microRNA binding and nuclear export by Exportin 5 // Nucl. Acids Res. 2004. V. 32. № 16. P. 4776–4785. https://doi.org/10.1093/nar/gkh824
Okada C., Yamashita E., Lee S.J. et al. A high-resolution structure of the pre-microRNA nuclear export machinery // Science. 2009. V. 326. № 5957. P. 1275–1279. https://doi.org/10.1126/science.1178705
Bennasser Y., Chable-Bessia C., Triboulet R. et al. Competition for XPO5 binding between Dicer mRNA, pre-miRNA and viral RNA regulates human Dicer levels // Nat. Structural & Mol. Biol. 2011. V. 18. № 3. P. 323–327. https://doi.org/10.1038/nsmb.1987
Melo S.A., Moutinho C., Ropero S. et al. A genetic defect in exportin-5 traps precursor microRNAs in the nucleus of cancer cells // Cancer Cell. 2010. V. 18. № 4. P. 303–315. https://doi.org/10.1016/j.ccr.2010.09.007
Singh C.P., Singh J., Nagaraju J. A baculovirus-encoded microRNA (miRNA) suppresses its host miRNA biogenesis by regulating the Exportin-5 cofactor Ran // J. Virology. 2012. V. 86. № 15. P. 7867–7879. https://doi.org/10.1128/JVI.00064-12
MacRae I.J., Zhou K., Doudna J.A. Structural determinants of RNA recognition and cleavage by Dicer // Nat. Structural & Mol. Biol. 2007. V. 14. № 10. P. 934–940. https://doi.org/10.1038/nsmb1293
Lau P.-W., Guiley K.Z., De N.et al. The molecular architecture of human Dicer // Nat. Structural & Mol. Biol. 2012. V. 19. № 4. P. 436–440. https://doi.org/10.1038/nsmb.2268
MacRae I.J. Structural basis for double-stranded RNA processing by Dicer // Science. 2006. V. 311. № 5758. P. 195–198. https://doi.org/10.1126/science.1121638
MacRae I.J., Li F., Zhou K. et al. Structure of Dicer and mechanistic implications for RNAi // Cold Spring Harbor Symp. Quantitative Biol. 2006. V. 71. P. 73–80. https://doi.org/10.1101/sqb.2006.71.042
Park J.-E., Heo I., Tian Y. et al. Dicer recognizes the 5' end of RNA for efficient and accurate processing // Nature. 2011. V. 475. № 7355. P. 201–205. https://doi.org/10.1038/nature10198
Thornton J.E., Chang H.-M., Piskounova E. et al. Lin28-mediated control of let-7 microRNA expression by alternative TUTases Zcchc11 (TUT4) and Zcchc6 (TUT7) // RNA. 2012. V. 18. № 10. P. 1875–1885. https://doi.org/10.1261/rna.034538.112
Newman M.A., Thomson J.M., Hammond S.M. Lin-28 interaction with the Let-7 precursor loop mediates regulated microRNA processing // RNA. 2008. V. 14. № 8. P. 1539–1549. https://doi.org/10.1261/rna.1155108
Bortolamiol-Becet D., Hu F., Jee D. et al. Selective suppression of the splicing-mediated microRNA pathway by theterminal uridyltransferase Tailor // Mol. Cell. 2015. V. 59. № 2. P. 217–228. https://doi.org/10.1016/j.molcel.2015.05.034
Starega-Roslan J., Witkos T., Galka-Marciniak P. et al. Sequence features of Drosha and Dicer cleavage sites affect the complexity of isomiRs // Intern. J. Mol. Sci. 2015. V. 16. № 12. P. 8110–8127. https://doi.org/10.3390/ijms16048110
Heo I., Ha M., Lim J. et al. Mono-uridylation of pre-microRNA as a key step in the biogenesis of group II let-7 microRNAs // Cell. 2012. V. 151. № 3. P. 521–532. https://doi.org/10.1016/j.cell.2012.09.022
Rau F., Freyermuth F., Fugier C. et al. Misregulation of miR-1 processing is associated with heart defects in myotonic dystrophy // Nat. Structural & Mol. Biol. 2011. V. 18. № 7. P. 840–845. https://doi.org/10.1038/nsmb.2067
Chen Y., Zubovic L., Yang F. et al. Rbfox proteins regulate microRNA biogenesis by sequence-specific binding to their precursors and target downstream Dicer // Nucl. Acids Res. 2016. V. 44. № 9. P. 4381–4395. https://doi.org/10.1093/nar/gkw177
Hellwig S., Bass B.L. A starvation-induced noncoding RNA modulates expression of Dicer-regulated genes // Proc. Natl. Acad. Sci. USA. 2008. V. 105. № 35. P. 12897–12902. https://doi.org/10.1073/pnas.0805118105
Iizasa H., Wulff B.-E., Alla N.R. et al. Editing of Epstein-Barr virus-encoded BART6 microRNAs controls their dicer targeting and consequently affects viral latency // J. Biol. Chem. 2010. V. 285. № 43. P. 33358–33370. https://doi.org/10.1074/jbc.M110.138362
Gu S., Jin L., Zhang Y. et al. The loop position of shRNAs and pre-miRNAs is critical for the accuracy of Dicer processing in vivo // Cell. 2012. V. 151. № 4. P. 900–911. https://doi.org/10.1016/j.cell.2012.09.042
Okamura K., Nakanishi K. Argonaute Proteins. N.Y.: Springer, 2018. 260 p. https://doi.org/10.1007/978-1-4939-7339-2
Pinhal D., Bovolenta L.A., Moxon S. et al. Genome-wide microRNA screening in Nile tilapia reveals pervasive isomiRs’ transcription, sex-biased arm switching and increasing complexity of expression throughout development // Sci. Reports. 2018. V. 8. № 8248. P. 1–18. https://doi.org/10.1038/s41598-018-26607-x
Suzuki H.I., Katsura A., Yasuda T. et al. Small-RNA asymmetry is directly driven by mammalian Argonautes // Nat. Structural & Mol. Biol. 2015. V. 22. № 7. P. 512–521. https://doi.org/10.1038/nsmb.3050
Wright D.J., Rice J.L., Yanker D.M. et al. Nearest neighbor parameters for inosine·uridine pairs in RNA duplexes // Biochemistry. 2007. V. 46. № 15. P. 4625–4634. https://doi.org/10.1021/bi0616910
Li L., Song Y., Shi X. et al. The landscape of miRNA editing in animals and its impact on miRNA biogenesis and targeting // Genome Res. 2018. V. 28. № 1. P. 132–143. https://doi.org/10.1101/gr.224386.117
Berezikov E. Evolution of microRNA diversity and regulation in animals // Nat. Rev. Genet. 2011. V. 12. № 12. P. 846–860. https://doi.org/10.1038/nrg3079
Hutvagner G., Simard M.J. Argonaute proteins: key players in RNA silencing // Nat. Rev. Mol. Cell Biol. 2008. V. 9. № 1. P. 22–32. https://doi.org/10.1038/nrm2321
Ghildiyal M., Xu J., Seitz H. et al. Sorting of Drosophila small silencing RNAs partitions microRNA* strands into the RNA interference pathway // RNA. 2010. V. 16. № 1. P. 43–56. https://doi.org/10.1261/rna.1972910
Ponomarenko M.P., Suslov V.V., Ponomarenko P.M. et al. Abundances of microRNAs in human cells can be estimated as a function of the abundances of YRHB and RHHK tetranucleotides in these microRNAs as an ill-posed inverse problem solution // Frontiers in Genet. 2013. V. 4. P. 1–13. https://doi.org/10.3389/fgene.2013.00122
Okamura K., Liu N., Lai E.C. Distinct mechanisms for microRNA strand selection by Drosophila Argonautes // Mol. Cell. 2009. V. 36. № 3. P. 431–444. https://doi.org/10.1016/j.molcel.2009.09.027
Shin C. Cleavage of the star strand facilitates assembly of some microRNAs into Ago2-containing silencing complexes in mammals // Cell. 2008. № 26. P. 308–313.
Curtis H.J., Sibley C.R., Wood M.J.A. Mirtrons, an emerging class of atypical miRNA: Mirtrons, an emerging class of atypical miRNA // Wiley Interdisciplinary Rev.: RNA. 2012. V. 3. № 5. P. 617–632. https://doi.org/10.1002/wrna.1122
Ladewig E., Okamura K., Flynt A.S. et al. Discovery of hundreds of mirtrons in mouse and human small RNA data // Genome Res. 2012. V. 22. № 9. P. 1634–1645. https://doi.org/10.1101/gr.133553.111
Wen J., Ladewig E., Shenker S. et al. Analysis of nearly one thousand mammalian mirtrons reveals novel features of Dicer substrates // PLoS Computational Biol. / Ed. Rigoutsos I. 2015. V. 11. № 9. P. 1–29. https://doi.org/10.1371/journal.pcbi.1004441
Yang L. Splicing noncoding RNAs from the inside out: splicing noncoding RNAs from the inside out // Wiley Interdisciplinary Rev.: RNA. 2015. V. 6. № 6. P. 651–660. https://doi.org/10.1002/wrna.1307
Titov I.I., Vorozheykin P.S. Comparing miRNA structure of mirtrons and non-mirtrons // BMC Genomics. 2018. V. 19. № S3. P. 92–102. https://doi.org/10.1186/s12864-018-4473-8
Berezikov E., Liu N., Flynt A.S. et al. Evolutionary flux of canonical microRNAs and mirtrons in Drosophila // Nat. Genet. 2010. V. 42. № 1. P. 6–9. https://doi.org/10.1038/ng0110-6
Havens M.A., Reich A.A., Duelli D.M. et al. Biogenesis of mammalian microRNAs by a non-canonical processing pathway // Nucl. Acids Res. 2012. V. 40. № 10. P. 4626–4640. https://doi.org/10.1093/nar/gks026
Abdelfattah A.M., Park C., Choi M.Y. Update on non-canonical microRNAs // Biomolecular Concepts. 2014. V. 5. № 4. P. 275–287. https://doi.org/10.1515/bmc-2014-0012
Stagsted L.V.W., Daugaard I., Hansen T.B. The agotrons: Gene regulators or Argonaute protectors? // BioEssays. 2017. V. 39. № 4. P. 1–6. https://doi.org/10.1002/bies.201600239
Cheloufi S., Dos Santos C.O., Chong M.M.W. et al. A dicer-independent miRNA biogenesis pathway that requires Ago catalysis // Nature. 2010. V. 465. № 7298. P. 584–589. https://doi.org/10.1038/nature09092
Cifuentes D., Xue H., Taylor D.W. et al. A novel miRNA processing pathway independent of Dicer requires Argonaute2 catalytic activity // Science. 2010. V. 328. № 5986. P. 1694–1698. https://doi.org/10.1126/science.1190809
Yoda M., Cifuentes D., Izumi N. et al. Poly(A)-specific ribonuclease mediates 3'-end trimming of Argonaute2-cleaved precursor microRNAs // Cell Reports. 2013. V. 5. № 3. P. 715–726. https://doi.org/10.1016/j.celrep.2013.09.029
Yang J.-S., Maurin T., Lai E.C. Functional parameters of Dicer-independent microRNA biogenesis // RNA. 2012. V. 18. № 5. P. 945–957. https://doi.org/10.1261/rna.032938.112
Wheeler B.M., Heimberg A.M., Moy V.N. et al. The deep evolution of metazoan microRNAs // Evolution & Development. 2009. V. 11. № 1. P. 50–68. https://doi.org/10.1111/j.1525-142X.2008.00302.x
Kolchanov N.A., Titov I.I., Vlassova I.E. et al. Chemical and computer probing of RNA structure // Progr. Nucl. Acid Res. and Mol. Biol. 1996. V. 53. P. 131–196. https://doi.org/10.1016/S0079-6603(08)60144-0
Slezak-Prochazka I., Durmus S., Kroesen B.J. et al. MicroRNAs, macrocontrol: Regulation of miRNA processing // RNA. 2010. V. 16. № 6. P. 1087–1095. https://doi.org/10.1261/rna.1804410
Gong J., Tong Y., Zhang H.-M. et al. Genome-wide identification of SNPs in microRNA genes and the SNP effects on microRNA target binding and biogenesis // Hum Mutat. 2012. V. 33. № 1. P. 254–263. https://doi.org/10.1002/humu.21641
Sun G., Yan J., Noltner K. et al. SNPs in human miRNA genes affect biogenesis and function // RNA. 2009. V. 15. № 9. P. 1640–1651. https://doi.org/10.1261/rna.1560209
Hill D.A., Ivanovich J., Priest J.R. et al. DICER1 mutations in familial pleuropulmonary blastoma // Science. 2009. V. 325. № 5943. P. 965–965. https://doi.org/10.1126/science.1174334
Nishikura K. A-to-I editing of coding and non-coding RNAs by ADARs // Nat. Rev. Mol. Cell Biol. 2016. V. 17. № 2. P. 83–96. https://doi.org/10.1038/nrm.2015.4
Tomaselli S., Bonamassa B., Alisi A. et al. ADAR Enzyme and miRNA Story: A Nucleotide that Can Make the Difference // Intern. J. Mol. Sci. 2013. V. 14. № 11. P. 22796–22816. https://doi.org/10.3390/ijms141122796
Kawahara Y., Zinshteyn B., Sethupathy P. et al. Redirection of silencing targets by adenosine-to-inosine editing of miRNAs // Science. 2007. V. 315. № 5815. P. 1137–1140. https://doi.org/10.1126/science.1138050
Kawahara Y., Zinshteyn B., Chendrimada T.P. et al. RNA editing of the microRNA-151 precursor blocks cleavage by the Dicer–TRBP complex // EMBO Reports. 2007. V. 8. № 8. P. 763–769. https://doi.org/10.1038/sj.embor.7401011
Zhang F., Lu Y., Yan S. et al. SPRINT: an SNP-free toolkit for identifying RNA editing sites // Bioinformatics / Ed. Hofacker I. 2017. V. 33. № 22. P. 3538–3548. https://doi.org/10.1093/bioinformatics/btx473
Neilsen C.T., Goodall G.J., Bracken C.P. IsomiRs – the overlooked repertoire in the dynamic microRNAome // Trends in Genetics. 2012. V. 28. № 11. P. 544–549. https://doi.org/10.1016/j.tig.2012.07.005
Starega-Roslan J., Galka-Marciniak P., Krzyzosiak W.J. Nucleotide sequence of miRNA precursor contributes to cleavage site selection by Dicer // Nucl. Acids Res. 2015. V. 43. № 22. P. 10939–10951. https://doi.org/10.1093/nar/gkv968
Li S., Patel D.J. Drosha and Dicer: Slicers cut from the same cloth // Cell Res. 2016. V. 26. № 5. P. 511–512. https://doi.org/10.1038/cr.2016.19
Ma M., Yin Z., Zhong H. et al. Analysis of the expression, function, and evolution of miR-27 isoforms and their responses in metabolic processes // Genomics. 2018. https://doi.org/10.1016/j.ygeno.2018.08.004
Yu F., Pillman K.A., Neilsen C.T. et al. Naturally existing isoforms of miR-222 have distinct functions // Nucl. Acids Res. 2017. V. 45. № 19. P. 11371–11385. https://doi.org/10.1093/nar/gkx788
Han B.W., Hung J.-H., Weng Z. et al. The 3'-to-5' Exoribonuclease Nibbler Shapes the 3' Ends of MicroRNAs Bound to Drosophila Argonaute1 // Curr. Biol. 2011. V. 21. № 22. P. 1878–1887. https://doi.org/10.1016/j.cub.2011.09.034
Liu N., Abe M., Sabin L.R. et al. The exoribonuclease nibbler controls 3' end processing of microRNAs in Drosophila // Curr. Biol. 2011. V. 21. № 22. P. 1888–1893. https://doi.org/10.1016/j.cub.2011.10.006
Norbury C.J. Cytoplasmic RNA: a case of the tail wagging the dog // Nature Rev. Mol. Cell Biol. 2013. V. 14. № 10. P. 643–653. https://doi.org/10.1038/nrm3645
Tan G.C., Dibb N. IsomiRs have functional importance // Malays J Pathol. 2015. V. 37. № 2. P. 73–81.
McCall M.N., Kim M.-S., Adil M. et al. Toward the human cellular microRNAome // Genome Res. 2017. V. 27. № 10. P. 1769–1781. https://doi.org/10.1101/gr.222067.117
Ludwig N., Becker M., Schumann T. et al. Bias in recent miRBase annotations potentially associated with RNA quality issues // Sci. Reports. 2017. V. 7. № 5162. P. 1–11. https://doi.org/10.1038/s41598-017-05070-0
Fromm B., Billipp T., Peck L.E. et al. A uniform system for the annotation of vertebrate microRNA genes and the evolution of the human microRNAome // Annual Rev. Genet. 2015. V. 49. № 1. P. 213–242. https://doi.org/10.1146/annurev-genet-120213-092023
Hou D., He F., Ma L. et al. The potential atheroprotective role of plant MIR156a as a repressor of monocyte recruitment on inflamed human endothelial cells // The J. Nutritional Biochem. 2018. V. 57. P. 197–205. https://doi.org/10.1016/j.jnutbio.2018.03.026
Fromm B., Kang W., Rovira C. et al. Plant microRNAs in human sera are likely contaminants // The J. Nutritional Biochem. 2018. https://doi.org/10.1016/j.jnutbio.2018.07.019
Дополнительные материалы отсутствуют.


