Генетика, 2020, T. 56, № 10, стр. 1205-1214
Бактериальные гены биомаркерных метаболитов расстройства аутистического спектра в кишечной микробиоте детей раннего возраста: выявление методами количественной ПЦР и анализом in silico
О. В. Аверина 1, 3, *, А. С. Ковтун 1, 2, В. Н. Даниленко 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Московский физико-технический институт (Государственный университет)
141701 Московская область, Долгопрудный, Россия
3 Российский национальный исследовательский медицинский университет им. Н.И. Пирогова
117997 Москва, Россия
* E-mail: olgavr06@mail.ru
Поступила в редакцию 10.12.2019
После доработки 31.01.2020
Принята к публикации 04.02.2020
Аннотация
С использованием двух различных методов: количественной ПЦР и in silico анализа было проведено исследование кишечных метагеномов детей с расстройством аутистического спектра (РАС) и здоровых детей раннего возраста по количественному содержанию бактериальных генов, кодирующих ключевые ферменты для продукции биомаркерных метаболитов. В результате анализа массива опубликованных данных для исследования были отобраны биомаркерные для РАС метаболиты: п‑крезол, индол, пропионовая кислота, Д-молочная кислота. Для количественной ПЦР и транскриптомного анализа были использованы разработанные специфические праймеры, подобранные к консервативным участкам генов, кодирующих ключевые ферменты, участвующие в продукции этих бактериальных метаболитов. Для метагеномного in silico анализа использованы сборки метагеномов и подобранный каталог нуклеотидных последовательностей регионов, специфичных для исследуемых генов. Проведенный сравнительный анализ выявил снижение уровня генов для исследуемых ферментов в метагеномах детей с РАС как в данных ПЦР анализа, так и в данных биоинформатического анализа метагеномов. Вместе с тем, с помощью количественной ПЦР было выявлено значительное увеличение уровня генов, кодирующих р-гидроксифенилацетат декарбоксилазу и Д‑лактат дегидрогеназу из бактерий Bacteroides fragilis и Alistipes finegoldii, метилмалонил-CoA-декарбоксилазу из бактерий B. fragilis, Alistipes shahii, Eubacterium rectali. Однако для всех этих генов был выявлен низкий уровень транскриптов в общей РНК из микробиоты ребенка с тяжелой формой РАС.
Кишечная микробиота, включающая все микроорганизмы пищеварительного тракта человека, находится в двунаправленной коммуникации с мозгом человека и ее влияние на мозг в основном осуществляется через действие продуцируемых нейроактивных метаболитов [1]. Различные клинические и экспериментальные данные свидетельствуют о влиянии кишечной микробиоты на широкий спектр поведенческих аспектов, включая социальное, эмоциональное и тревожное поведение [2]. Значительные изменения в динамичном взаимодействии микробиоты с хозяином в критические периоды раннего детства приводят к глубокому нарушению в сигнализации между кишечником и мозгом и могут привести к нарушениям в развитии мозга [3–5]. Расстройство аутистического спектра (РАС) является одним из заболеваний, возникших в результате нарушений в развитии мозга ребенка. Характеризуется определенными нарушениями в вербальной и невербальной коммуникации, социальных взаимодействий. РАС сегодня является распространенным ранним неврологическим заболеванием, которое выявляется у детского населения с частотой 1–2% и продолжает расти из года в год. Это подтверждает, что большинство случаев аутизма не связано с нарушениями генома, и позволяет рассматривать неблагополучное влияние окружающей среды как основу патогенеза большей части случаев аутизма [5]. Различные независимые исследования показали специфические изменения в составе кишечной микробиоты у пациентов с РАС [6] и изменения в уровнях ряда метаболитов, выделяя их как специфические биомаркеры, связанные с заболеванием [7]. Одной из причин, негативно влияющей на композицию микробиоты и ее функциональность, сегодня рассматривается чрезмерное употребление в раннем детстве антибиотиков [8].
В данной работе объектом исследования были биомаркерные метаболиты, продуцируемые бактериями кишечной микробиоты человека: пропионовая кислота (пропионат), п-крезол, индол, Д-молочная кислота. В фекалиях детей с РАС по сравнению с контрольной группой здоровых детей были обнаружены высокие концентрации общего пропионата [9]. Пропионат способен модифицировать социальное поведение. У людей с нарушениями метаболизма пропионата наблюдаются аномалии в развитии нервной системы, сходные с симптомами аутизма [10]. Помимо прямого воздействия на мозг, пропионат модулирует секрецию серотонина в кишечнике и снижает уровень серотонина и дофамина в головном мозге [11]. В образовании пропионовой кислоты в микробиоте людей с РАС могут участвовать бактерии Clostridia, Sutterella и Bacteroidetes, уровень которых выше в сравнении с микробиотой в норме [12]. Уровень фенольных соединений, в том числе п-крезола, выше в кале и моче детей с РАС [13]. П-крезол (4-метилфенол) является мочевым биомаркером аутизма человека [14]. П-крезол продуцируется анаэробными протеолитическими бактериями, большей частью Bacteroides, Clostridium, уровень которых увеличен в микробиоте детей с РАС [14]. У пациентов с РАС также был повышен уровень индола – бактериального продукта метаболизма триптофана [13]. Увеличение индола связывают с микробной активностью в кишечнике. Увеличение количества Д-молочной кислоты у больных с РАС может усиливать у них когнитивные нарушения [15]. Целью данной работы было проведение сравнительного in silico анализа и количественного ПЦР анализа метагеномов детей с РАС и здоровых детей по уровню генов, участвующих в продукции биомаркерных метаболитов, из геномов доминирующих видов бактерий кишечной микробиоты. А также выявление уровня транскриптов исследуемых генов в составе общей РНК микробиоты ребенка с РАС.
Поиск бактериальных ферментов, участвующих в метаболических путях синтеза отобранных биомаркерных нейроактивных соединений, и генов их кодирующих, проводился с использованием поисковых систем Google Академия, PubMed, NCBI, KEGG, InterPro и др. С помощью базы данных Protein, доступной на сайте Национального центра биотехнологической информации США, отбирались геномы бактерий кишечной микробиоты, содержащие ключевые гены, кодирующие ферменты, участвующие в продукции биомаркерных соединений. С использованием программ Clustal Omega (http://www.ebi.ac.uk/Tools/msa/clustalo/) и MEGA v. 6.0 (http://www.megasoftware.net/) проведен сравнительный и филогенетический анализ аминокислотных последовательностей продуктов выявленных генов. Подбор праймеров к консервативным нуклеотидным последовательностям отобранных генов проводили с помощью программы NCBI/Primer-BLAST (www.ncbi.nlm.nih.gov/tools/ primer-blast/). Праймеры синтезировали в фирме “Синтол”.
Объектом исследования была кишечная микробиота детей с РАС и здоровых детей без симптомов РАС возрастом 3–5 лет. Отбирали пробы фекалий, используя стандартные методики. До использования в экспериментах образцы хранили замороженными при –80°С. Тотальную бактериальную геномную ДНК выделяли из навесок замороженного кала при помощи набора реагентов QIAamp Fast DNA Stool Mini kit (Qiagen, Германия) по протоколу, рекомендованному производителем. Концентрацию выделенной ДНК определяли с помощью прибора Qubit (Invitrogen, США). Амплификацию ДНК проводили на приборе CFX96 Touch thermal cycler (Bio-Rad). ПЦР-смеси (конечный объем 25 мкл) содержали 10 мкл смеси SYBR Green PCR (Syntol), 2 мкл ДНК, 4 мкМ MgCl2 и 5 мкМ прямого и обратного праймера. ПЦР продукты визуализировали с помощью электрофореза в 1%-ном агарозном геле.
Тотальную бактериальную геномную РНК выделяли из навесок замороженного кала, которую ресуспендировали в 100 мл ТЕ буфера (30 мм/1 Tris-HCl, 1 мМ/л этилендиаминтетрауксусная кислота (ЭДТА), рН 8.0 с лизоцимом (20 мг/мл) и инкубировали в течение 10 мин при 37°С. 400 мкл лизирующего буфера (4.5 моль/л гуанидин/HCl, 50 мM/л Tris-HCl, 30% Тритон X-100, pH 6.6) добавили в клеточную суспензию и далее гомогенезировали на приборе Speed Mill Plus homogenizer (Analytik Jena AG, Germany). Для выделения РНК использовали коммерческий набор RNAse Mini Kit (Qiagen) с использованием методических рекомендаций производителя. Удаление остатков ДНК проводили обработкой RNase-free DNase I (Qiagen). Концентрацию выделенной РНК определяли с помощью прибора Qubit (Invitrogen, США).
Синтез кДНК проводили с общей РНК с использованием random hexamer primer и SuperScript II Reverse Transcriptase (Invitrogen). ПЦР в реальном времени проводили с использованием рабочих праймеров, специфичных для исследуемых генов, на приборе CFX96 Touch thermal cycler (Bio-Rad). ПЦР-смеси (конечный объем 25 мкл) содержали 10 мкл смеси SYBR Green PCR (Syntol), 3 мкл кДНК, 4 мкМ MgCl2 и 5 мкМ прямого и обратного праймера. Отсутствие контаминации ДНК в образцах РНК подтверждалось с помощью ПЦР анализа с использованием праймеров к определенному гену. В каждом запуске ПЦР использовали отрицательные контроли без кДНК. Анализ кривой плавления показывал отсутствие неспецифических продуктов.
Проводили секвенирование общей ДНК, которую использовали и для количественной ПЦР. Пробоподготовку ДНК осуществляли как описано в статье А.С. Ковтун с соавт. [16]. Сквенирование проводили на двух дорожках прибора Illumina HiSeq 4000 (151 цикл с каждого конца фрагментов) с использованием реактивов HiSeq 4000 sequencing kit version 1 (Illumina). Подготовку образцов и запуск осуществляли согласно протоколам Illumina. Файлы FASTQ были получены с помощью ПО bcl2fastq v2.20 Conversion Software (Illumina). Формат записи строки данных о качестве – Phred 33. Тримминг осуществлялся с помощью программы trimmomatic. Очистка от контаминации человеческой ДНК проводилась при помощи картирования метагеномных ридов на человеческий геном программой bowtie2. Финальная оценка качества и оценка параметров каждого сиквенса после контроля качества выполнялась при помощи программ fastqc и fastx. В результате секвенирования было отсеяно до 15% ридов. Минимальная длина рида составила 50 пн, максимальная – 140 пн. Для сборки метагеномов использовалась программа metaSPAdes. Сборка проводилась на серверной вычислительной машине, установленной в лаборатории системной биологии и вычислительной генетики Института общей генетики РАН.
Для метагеномного анализа in silico использовались сборки метагеномов и подобранный каталог нуклеотидных последовательностей регионов, специфичных для исследуемых генов, а также программа blastn.
В результате количественного ПЦР-РВ анализа общей ДНК из микробиоты от детей с РАС (5) и здоровых детей (3) с использованием разработанных праймеров, которые указаны в табл. 1, получены данные о количественном содержании 25 генов, кодирующих ферменты, участвующие в продукции метаболитов: р-крезола, индола, пропионовой кислоты, Д-молочной кислоты. Данные табл. 1 показывают разницу в среднестатистических данных количества генов различных комменсальных бактерий в метагеномах детей с РАС (МАД) и здоровых детей (МЗД). Представленные данные указывают на значительное повышение в МАД уровня генов, кодирующих ферменты: р-гидроксифенилацетат декарбоксилазу из бактерий Bacteroides fragilis и Alistipes finegoldii, участвующую в продукции п-крезола; метилмалонил-CoA-декарбоксилазу из бактерий B. fragilis, Alistipes shahii, Eubacterium rectali, участвующую в продукции пропионовой кислоты; Д-лактат дегидрогеназу из бактерий B. fragilis, A. finegoldii, участвующую в продукции Д-молочной кислоты. Однако для всех этих генов было низкое содержание транскриптов в общей РНК из микробиоты пробы № 72 от ребенка с тяжелой формой РАС (табл. 1). Количественное содержание остальных генов (60%) из различных видов бактерий было выше в МЗД. Более высокое содержание исследуемых генов (среднестатистические величины) в составе 17 метагеномов здоровых детей по сравнению с метагеномами 17 детей с РАС было выявлено методом in silico (Приложение). На рис. 1 представлена теплокарта количественного распределения четырех генов из семи различных комменсальных бактерий в составе сравниваемых групп метагеномов (вошли только гены, наиболее распространенные во всех исследуемых метагеномах с 70–100% встречаемости (Приложение)). Эти данные отличаются от данных, полученных с использованием количественного ПЦР метода. Это может быть связано с невысокой специфичностью биоинформатического анализа. Хотя нуклеотидные последовательности, найденные в метагеномах данным методом, имели гомологию не ниже 90% с исследуемыми последовательностями, их небольшая длина (в среднем 250 нуклеотидов) привела к избыточно высокой чувствительности метода.
Таблица 1.
Среднестатистический уровень генов в метаболитах детей с РАС и метагеномах здоровых детей, выявленный методом количественного ПЦР анализа
| № | Бактерия, обозначение гена | ПЦР
п-кт пн |
Олигонуклеотиды | 72 кДНК | 72 ДНК | Среднее значение у МАД | Среднее значение у МЗД | % встречаемости в МАД/МЗД | |
|---|---|---|---|---|---|---|---|---|---|
| П-гидроксифенилацетат декарбоксилаза (большая субъединица) для п-крезола | |||||||||
| 1 | Bacteroides fragilis | pcrBf | 221 | F 5'-GCGAGAAACACCGCAATCT R 5'-TTCAGGGCAGCAAGGGTAT |
39.00 | 34.10 | 29.72 ± 6.11 | 33.49 ± 0.56 | 100/100 |
| 2 | Alistipes finegoldii | pcrAf | 218 | F 5'-GTATTCACCCGCACCGTTAC R 5'-CCCAGGAAGATACGCTGTTC |
39.69 | 19.12 | 20.38 ± 2.09 | 22.78 ± 3.08 | 100/100 |
| 3 | Ruminococcus torques | pcrRt | 226 | F 5'-GAGAAGGGTGCGACAGGAT R 5'-CACGGCACTTGTTCAGGTC |
26.64 | 27.63 | 28.65 ± 1.34 | 26.55 ± 6.51 | 100/100 |
| Триптофаназа для индола | |||||||||
| 4 |
Bacteroides uniformis | indBu | 168 | F 5'-AACTCCCATTTCGACACCAC R 5'-CGGTATCCGTTCGCTATGTT |
32.17 | 17.77 | 18.12 ± 1.33 | 17.48 ± 0.69 | 100/100 |
| 5 | Bacteroides ovatus | indBo | 173 | F 5'-GGAGCGACCTCATTCTTCAA R 5'-ATGTGCCCTTTGGTGGTGT |
34.86 | 19.54 | 19.81 ± 2.89 | 18.93 ± 0.47 | 100/100 |
| Пропиональдегиддегидрогеназа для пропионовой кислоты | |||||||||
| 6 | Roseburia inulinivorans | prdRi | 245 | F 5'-TAGAGCATGGCAACCGTCAT R 5'-TCTGATGCAAAGGCTGTCTG |
32.28 | 32.36 | 27.86 ± 3.10 | 24.73 ± 0.56 | 100/100 |
| 7 | Ruminococcus torques | prdRt | 213 | F 5'-CCCAGAAGATCGTGGCTAAG R 5'-CTCCTGACCATGCCTGTGTA |
26.05 | 28.31 | 28.71 ± 2.16 | 27.19 ± 7.28 | 70/80 |
| 8 | Eubacterium hallii | prdEh | 207 | F 5'-TATCCCATTCCGCCAACAC R 5'-GATGCCTATCCTTCCTATCG |
28.84 | 28.37 | 28.74 ± 1.65 | 27.88 ± 1.96 | 100/100 |
| Метилмалонил-CoA-декарбоксилаза для пропионовой кислоты | |||||||||
| 9 | Bacteroides fragilis | MmdBf | 170 | F 5'-GACGACCTGCTGAACGAGAT R 5'-ACCACACCTACCGATTGTCC |
– | 36.38 | 27.71 ± 6.58 | 34.58 ± 0.12 | 70/60 |
| 10 | Bacteroides ovatus | MmdBBo | 208 | F 5'-GCGTTATCCTTCATGGTGCT R 5'-TCGCGTGCGTACAATACTTC |
28.10 | 22.73 | 20.8 ± 2.53 | 19.82 ± 0.64 | 100/100 |
| 11 | Bacteroides vulgates | MmdBv | 193 | F 5'-TCTTCTTCCGTCTTGGCTGT R 5'-GCGGAGCTGTTTACTCTCCT |
29.46 | 20.45 | 19.72 ± 1.56 | 20.10 ± 1.67 | 100/100 |
| 12 | Alistipes shahii | MmdAs | 210 | F 5'-CTGATGCACACCACCAAGTC R 5'-GGTCATGTCGTAGGGCTTGT |
31.35 | 18.73 | 20.53 ± 1.62 | 22.10 ± 1.47 | 100/100 |
| 13 | Alistipes putredinis | MmdAp | 240 | F 5'-TCGACAAGCAACACGAGAAG R 5'-TCGAAAGCGACAGAGAACCT |
32.62 | 19.04 | 24.88 ± 6.87 | 20.70 ± 3.07 | 100/80 |
| 14 | Clostridium bolteae | MmdCb | 200 | F 5'-ATGTGCAGCGAGAGAAGGAT R 5'-GCCTCAGACGGATTCATCAT |
27.40 | 25.89 | 25.47 ± 2.14 | 24.69 ± 2.38 | 100/100 |
| 15 | Roseburia intestinalis | MmdRint | 186 | F 5'-AGGTTTACTTTCGGGCAGGT R 5'-GACAGCAAGAGGATGCAACA |
25.79 | 29.28 | 25.84 ± 2.81 | 25.02 ± 2.11 | 100/100 |
| 16 | Roseburia inulinivorans | MdRinu | 247 | F 5'-CCTGCCAAATCCGTCTAATG R 5'-TCTTGCAGACACAGACACAC |
39.72 | 26.35 | 23.62 ± 2.07 | 24.04 ± 4.11 | 100/100 |
| 17 | Eubacterium rectali | MmdEr | 213 | F 5'-GATTACAGGCGAGGCTTACG R 5'-GCTGCTGCTGATGAAACAGA |
34.02 | 24.03 | 24.69 ± 0.59 | 26.32 ± 5.12 | 100/100 |
| 18 | Eubacterium hallii | MmdEh | 176 | F 5'-AGATGCCTGTGCCTGCTTAT R 5'-TTGGAGGTTCCGTAGGTGAG |
39.62 | 29.99 | 29.59 ± 2.17 | 28.29 ± 1.71 | 100/100 |
| Д-лактат дегидрогеназа для Д-молочной кислоты | |||||||||
| 19 | Bacteroides fragilis | D-ldBf | 234 | F 5'-CCCTTCGTTGTGCTGGTTAT R 5'-CGATGATACCGGCTGTCTTT |
39.62 | 34.02 | 20.45 ± 6.09 | 34.36 ± 1.26 | 30/100 |
| 20 | Bacteroides ovatus | D-ldBo | 150 | F5'-GACGCAGAAGTAATCCGTGTG R 5'-TGCAACGGCATAAGGAGAGT |
– | 35.34 | 29.11 ± 5.35 | 28.14 ± 8.43 | 100/100 |
| 21 | Bacteroides vulgatus | D-ldBv | 238 | F 5'-TGACCAAAGGAGTGGATGTG R 5'-GGAATCTTGCGGTTGAGTGA |
29.23 | 19.28 | 19.56 ± 1.42 | 20.28 ± 1.87 | 100/100 |
| 22 | Alistipes putredinis | D-ldAp | 195 | F 5'-GTGGGAGAATGCCTGAATGT R 5'-GGGCTGGATGTCTATGAGGA |
31.52 | 19.75 | 25.52 ± 6.56 | 21.62 ± 2.33 | 100/80 |
| 23 | Alistipes finegoldii | D-ldAf | 177 | F 5'-TCGTAAGCCAGCACCTTCAT R 5'-GCTCAACCGCAAAGTCCAC |
32.29 | 18.59 | 19.51 ± 1.96 | 21.95 ± 2.89 | 100/100 |
| 24 | Roseburia hominis | D-ldRh | 236 | F 5'-CTCGATTCCCTCGATGATGT R 5'-GGATTCTGGCATACAGCAGA |
29.04 | 25.15 | 25.15 ± 1.32 | 24.77 ± 2.19 | 70/100 |
| 25 | Eubacterium eligens | D-ldEe | 172 | F 5'-GGAGCGGATTTACAGCAGAT R 5'-GCTGCCTCAAGGTCAACACT |
36.03 | 19.88 | 22.78 ± 5.48 | 20.41 ± 1.2 | 100/100 |
Рис. 1.
Теплокарта среднестатистического уровня генов, кодирующих ферменты для продукции биомаркерных метаболитов, в 17 метагеномах детей с РАС и в 17 метагеномах здоровых детей (контрольная группа). Данные, полученные после in silico-анализа метагеномов.
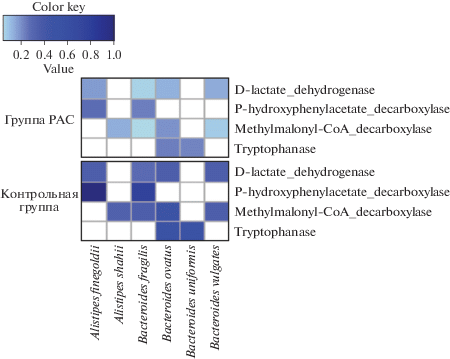
Полученные данные сравнительного анализа исследуемых метагеномов указывают на снижение уровня большинства генов, участвующих в продукции биомаркерных метаболитов, в метагеномах детей с РАС. Однако в биоматериале (крови, моче и фекалиях) от детей с РАС (по сравнению с контрольной группой здоровых детей) были обнаружены более высокие концентрации общего пропионата [9], индола [13], п-крезола [13, 14] и Д-молочной кислоты [15]. Возможно в повышении концентрации данных метоболитов задействованы другие комменсальные бактерии или клетки хозяина. Поиск новых знаний о причинах возникновения РАС продолжается [17]. Большой интерес вызывают эпигенетические процессы, в которые могут включаться различные бактериальные соединения [4, 5], гены устойчивости к антибиотикам с дополнительной функциональностью [8] и другие.
Целью данной работы было выявление разницы в количественном содержании исследуемых генов нейроактивных метаболитов в составе метагеномов детей сравниваемых групп при использовании различных методов исследования. Данные количественного метода ПЦР РВ анализа оказались более точными и достоверными по сравнению с данными in silico анализа. Полученные результаты указывают на необходимость использования комбинации различных методов для анализа бактериальных генов в составе микробиома, включая данные транскиптомного и метаболомного анализа, которые дадут более полную характеристику нейрометаболической функциональности кишечной микробиоты, коррелирующей с РАС.
Работа выполнена при финансовой поддержке гранта Российского научного фонда (проект № 17-15-01488).
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Averina O.V., Danilenko V.N. The human gut microbiota: Role in the formation and functioning of nervous system // Microbiology. 2017. V. 86. № 1. P. 1–19. https://doi.org/10.1134/S0026261717010040
Dinan T.G., Cryan J.F. Gut-brain axis in 2016: Brain-gut-microbiota axis – mood, metabolism and behaviour // Nat. Rev. Gastroenterol. Hepatol. 2017. V.14. № 2. P. 69–70. https://doi.org/10.1038/nrgastro.2016.200
Borre Y.E., Moloney R.D., Clarke G. et al. The impact of microbiota on brain and behavior: Mechanisms & therapeutic potential // Microbial Endocrinology: The Microbiota-Gut-Brain Axis in Health and Disease. N.Y.: Springer, 2014. P. 373–403.
Poletaev A.B., Shenderov B.A. Autism and autoimmunity: Genetics or epigenetics // Clin. Pathophysiology. 2016. V. 4. P. 1–14.
Poletaev A.B., Shenderov B.A. Autism: Genetics or epigenetics? // ARC J. Immun. and Vaccines. 2016. V. 1. I. 2. P. 1–7.
Kovtun A.S., Averina O.V., Alekseeva M.G., Danilenko V.N. Antibiotic resistance genes in the gut microbiota of children with autistic spectrum disorder as possible predictors of the disease // Microbial Drug Resistance. 2020. (в пeчaти)
Hughes H.K., Rose D., Ashwood P. The gut microbiota and dysbiosis in autism spectrum disorders // Curr. Neurol. Neurosci. Rep. 2018. V. 18. № 81. P. 1–15. https://doi.org/10.1007/s11910-018-0887-6
Kang D.W., Ilhan Z.E., Isern N.G. et al. Differences in fecal microbial metabolites and microbiota of children with autism spectrum disorders // Anaerobe. 2018. V. 49. P. 121–131. https://doi.org/10.1016/j.anaerobe.2017.12.007
Wang L., Christophersen C.T., Sorich M.J. et al. Elevated fecal short chain fatty acid and ammonia concentrations in children with autism spectrum disorder // Diges. Dis. Scien. 2012. V. 54. № 1. P. 1–7. https://doi.org/10.1007/s10620-012-2167-7
Macfabe D.F. Short-chain fatty acid fermentation products of the gut microbiome: Implications in autism spectrum disorders // Microb. Ecol. Health. Dis. 2012. V. 23. P. 1–25. https://doi.org/10.3402/mehd.v23i0.19260
Reigstad C.S., Salmonson C.E., Rainey J.F. et al. Gut microbes promote colonic serotonin production through an effect of short-chain fatty acids on enterochromaffin cells // FASEB J. 2015. V. 29. № 4. P. 1395–1403. https://doi.org/10.1096/fj.14-259598
MacFabe D.F., Cain N.E., Boon F. et al. Effects of the enteric bacterial metabolic product propionic acid on object-directed behavior, social behavior, cognition, and neuroinflammation in adolescent rats: Relevance to autism spectrum disorder // Behav. Brain. Res. 2011. V. 217. P. 47–54. https://doi.org/10.1016/j.bbr.2010.10.005
De Angelis M., Piccolo M., Vannini L. et al. Fecal microbiota and metabolome of children with autism and pervasive developmental disorder not otherwise specified // PLoS One. 2013. V. 8. № 10. P. e76993. https://doi.org/10.1371/journal.pone. 0076993
Persico A.M., Napolioni V. Urinary p-cresol in autism spectrum disorder // Neurotoxicol. Teratol. 2013. V. 36. P. 82–90. https://doi.org/10.1016/j.ntt.2012.09.002
Mack D.R. D(–)-lactic acid-producing probiotics, D(–)-lactic acidosis and infants// Can. J. Gastroenterol. 2004. V. 18. № 11. P. 671–675. https://doi.org/10.1155/2004/342583
Ковтун А.С., Алексеева М.Г., Аверина О.В., Даниленко В.Н. Идентификация аминогликозидфосфотрансфераз клинических штаммов бактерий в микробиоте жителей России // Вестник РГМУ. 2017. Т. 2. С. 14–19. https://doi.org/10.24075/brsmu.2017-02-02
Srikantha P., Mohajeri M.H. The possible role of the microbiota-gut-brain-axis in autism spectrum disorder // Int. J. Mol. Sci. 2019. V. 20. № 9. P. E2115. https://doi.org/10.3390/ijms20092115
Дополнительные материалы отсутствуют.


