Генетика, 2020, T. 56, № 12, стр. 1349-1365
Оценка актуальности молекулярно-генетического тестирования мутаций в генах BRCA у пациентов со злокачественными новообразованиями яичника и молочной железы
В. А. Лемешко 1, 2, 3, *, Т. С. Тепцова 1, 3, С. С. Ратушняк 3, Н. З. Мусина 1, 3, 4
1 Первый Московский государственный медицинский университет им. И.М. Сеченова
119435 Москва, Россия
2 Научно-исследовательский финансовый институт
127006 Москва, Россия
3 Центр экспертизы и контроля качества медицинской помощи
109028 Москва, Россия
4 Санкт-Петербургский государственный химико-фармацевтический университет
Министерства здравоохранения Российской Федерации
197376 Санкт-Петербург, Россия
* E-mail: lera.lemeschko@yandex.ru
Поступила в редакцию 25.12.2019
После доработки 04.02.2020
Принята к публикации 17.03.2020
Аннотация
Генетическая предрасположенность является одним из основных факторов риска развития рака молочной железы (РМЖ) и рака яичников (РЯ). Нами был проведен анализ эпидемиологии мутаций в генах BRCA1 и BRCA2 в российской популяции и проанализирована отечественная и международная клиническая практика молекулярно-генетического тестирования пациентов с РМЖ и/или РЯ на наличие мутаций в генах BRCA1 и BRCA2. Эффективность выявления мутаций во многом зависит от выбранной технологии молекулярно-генетического тестирования (МГТ). Наиболее распространенными в российской популяции BRCA-мутациями являются 5382insC, 4153delA, 185delAG, 300T>G (в гене BRCA1) и 6174delT (в гене BRCA2). Сравнение отечественных и зарубежных клинических рекомендаций показало, что область применения результатов МГТ на BRCA-мутации в международной клинической практике шире, чем в российской. Важным вопросом для клинической практики является выбор технологии МГТ. Основным преимуществом технологии NGS является возможность определения мутации в любой части генов BRCA1/2.
В Российской Федерации в 2018 г. число случаев впервые выявленного рака молочной железы (РМЖ) составило 64 544, при этом в 26.5% случаев была диагностирована I стадия заболевания, в 44.7% случаев – II стадия, в 20.6% случаев – III стадия и в 7.8% – IV стадия. Летальность на первом году жизни с момента постановки диагноза составила 5.8%. На учете в онкологических учреждениях на конец 2018 г. находились 692 297 пациентов, при этом 60.9% из них находились на учете от 5 лет и более [1]. Заболеваемость РМЖ у женщин в 2018 г. в стандартизированных показателях составила 51.63 случай на 100 тыс. населения, а среднегодовой темп прироста с 2008 по 2018 год составил 1.97. Смертность от РМЖ среди женщин в 2017 г. в стандартизированных показателях составила 14.02 случаев на 100 тыс. населения [2]. Число случаев впервые выявленного рака яичников (РЯ) составило 13 701, при этом в 27.8% случаев была диагностирована I стадия заболевания, в 12.5% случаев – II стадия, в 38.0% случаев – III стадия и в 20.0% – IV стадия. Летальность на первом году жизни с момента постановки диагноза составила 20.3%. На учете в онкологических учреждениях на конец 2018 г. находились 111 915 пациентов, при этом 62.1% из них находились на учете от 5 лет и более [1]. Заболеваемость РЯ у женщин в 2018 г. в стандартизированных показателях составила 11.14 случаев на 100 тыс. населения, а среднегодовой темп прироста с 2008 по 2018 год составил 0.45. Смертность от РЯ среди женщин в 2018 г. в стандартизированных показателях составила 4.92 случаев на 100 тыс. населения [2]. В структуре онкологической заболеваемости среди обоих полов РМЖ занимает второе место и составляет 11.4%, при этом на рак яичников приходится доля 2.3% [2].
Генетическая предрасположенность является одним из основных факторов риска развития РМЖ и РЯ [3]. От 5 до 10% случаев РМЖ и от 10 до 15% случаев РЯ (особенно высокой степени злокачественности) являются наследственными [4]. Наследственный РМЖ связан с нарушениями в генах репарации ДНК различной степени пенетрантности. Мутации в генах BRCA1/2 характеризуются высокой пенетрантностью и обусловливают 20–50% наследственных форм РМЖ и до 90–95% наследственных форм РЯ у женщин, а также 4–40% РМЖ у мужчин. Помимо РМЖ и РЯ, у носителей мутаций в генах BRCA1/2 может развиваться рак фаллопиевых труб, рак желудка, рак простаты и рак поджелудочной железы [4].
Гены BRCA1 и BRCA2 кодируют аминокислотные последовательности ядерных белков, которые участвуют в регуляции репарации повреждений ДНК и размножения клеток. Мутации генов BRCA1 и BRCA2 приводят к хромосомной нестабильности и злокачественной трансформации клеток молочной железы, яичников и других органов [5].
По данным зарубежной статистики, средние кумулятивные риски для носителей мутаций в гене BRCA1 в возрасте 70 лет составляют 57–65% в случае РМЖ и 39–40% – в случае РЯ. Риск развития РМЖ у носителей мутаций в гене BRCA2 составляет 45–49%, тогда как риск РЯ не превышает 11–18% [6]. Во многих популяциях наблюдается так называемый эффект основателя (“founder” эффект), который представляет собой преобладание нескольких мутаций в генах BRCA1 и BRCA2, специфичных для этнической группы. Выявление широко распространенных мутаций в генах BRCA1/2, характерных для определенной популяции, лежит в основе внедрения соответствующих молекулярных скрининговых программ и оптимизации генетического тестирования [3]. Таким образом, важным аспектом оценки распространенности мутаций в генах BRCA1/2 является обнаружение эффекта основателя для определенных этнических групп населения.
Взаимосвязь между наличием мутаций в генах BRCA1/2 и риском развития наследственного синдрома РМЖ/РЯ свидетельствует о возможности использования результатов молекулярно-генетического тестирования (МГТ) на наличие данных мутаций для выявления носителей BRCA-мутации до развития заболевания и проведения ранней профилактики и мониторинга их состояния. Кроме того, выявление мутаций в генах BRCA1/2 у пациентов или родственников пациентов с РМЖ/РЯ необходимо для формирования специфического подхода к ведению таких пациентов, в частности выбор методов диагностики и назначение таргетной терапии ингибиторами поли(АДФ-рибоза)-полимеразы (англ. poly (ADP-ribose) polymerase inhibitors, PARP inhibitors, PARP-ингибиторы) [7].
В настоящий момент в практике МГТ распространение получили методы секвенирования нового поколения (англ. Next Generation Sequencing, NGS), благодаря которым стало возможно быстро и с меньшими финансовыми затратами анализировать множество участков генома одновременно, а также всю последовательность ДНК (весь геном). Данные технологии также нашли свое применение в определении клинически значимых последовательностей ДНК (таргетное NGS), в том числе и экзонов генов BRCA1/2. Каждый метод обладает рядом преимуществ и ограничений, однако актуальным остается вопрос о возможности нахождения патологических мутаций в любом месте гена с учетом этнических различий, а также о вероятности наличия новых патологических мутаций, что может привести к ложноотрицательным результатам тестирования при использовании традиционных технологий секвенирования [8, 9].
ЭПИДЕМИОЛОГИЯ МУТАЦИЙ В ГЕНАХ BRCA1 И BRCA2 В РОССИЙСКОЙ ПОПУЛЯЦИИ
С целью оценки распространенности мутаций в генах BRCA1/2 в российской популяции пациентов со злокачественными новообразованиями яичников и молочной железы был проведен поиск эпидемиологических исследований, в результате которого были найдены 16 эпидемиологических исследований, посвященных оценке распространенности мутаций в генах BRCA1/2 в российской популяции пациентов со злокачественными новообразованиями молочной железы, и 11 эпидемиологических исследований в российской популяции пациентов со злокачественными новообразованиями яичников.
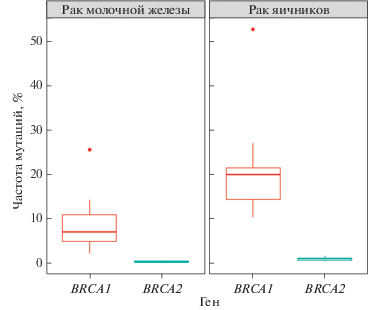
Согласно результатам найденных исследований, частота встречаемости мутаций в генах BRCA1/2 у пациентов с РМЖ в российской популяции составляет от 0.1 до 25.6% [10–22]. При этом мутации в гене BRCA1 возникают с частотой от 2.02 до 25.6% [10–22], а в гене BRCA2 – от 0.1 до 0.5% [10, 15, 17, 19–21]. Частота встречаемости мутаций в генах BRCA1/2 у пациентов с РЯ в российской популяции составляет от 0.3 до 35% [15, 23–30]. При этом частота встречаемости мутации в гене BRCA1 составила от 10.29 до 27% [15, 23–30], а в гене BRCA2 – от 0.3 до 8% [15, 23–25, 27, 30]. Стоит отметить, что согласно результатам исследования О. Федоровой и др. [28] у пациентов с первично-множественными злокачественными новообразованиями (ЗНО) с повреждением яичников частота встречаемости мутаций в генах BRCA1/2 составила 52.63%, при этом мутации в гене BRCA2 отсутствовали (рис. 1). Таким образом, в структуре частоты встречаемости мутаций в генах BRCA1/2 для российской популяции пациентов с РМЖ преобладают мутации в гене BRCA1.
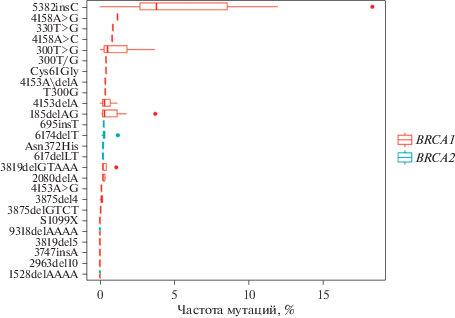
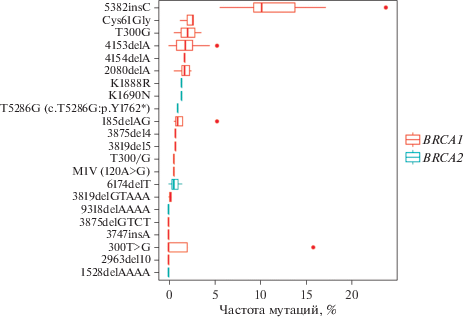
По результатам проведенного обзора распространенности мутаций в генах BRCA1/2 у пациентов со злокачественными новообразованиями яичника и молочной железы наиболее распространенными мутациями в российской популяции являются мутации в гене BRCA1 5382insC, 4153delA, 185delAG и 300T>G (рис. 2 и 3). Мутации в гене BRCA2 встречаются реже, чем в гене BRCA1, при этом наиболее распространенной мутацией в гене BRCA2 является 6174delT. Полученные в результате анализа публикаций исследований выводы о преобладающей распространенности мутации 5382insC в гене BRCA1 соотносятся с данными, полученными в исследованиях, оценивающих распространенность BRCA-мутаций в странах Восточной Европы [31–33].
Несмотря на наличие исследований, в которых оценивалась распространенность мутаций в генах BRCA1/2 для российской популяции пациентов и были выявлены мутации с эффектом основателя, данные о статистически значимых доказательствах в корреляции этих мутаций и предрасположенности к наследственному синдрому РМЖ/РЯ отсутствовали.
С точки зрения диагностики важным является обнаружение так называемых мутаций с эффектом основателя, специфических для различных этнических популяций. Однако с учетом этнического разнообразия населения России и ограниченности исследований, посвященных данному вопросу, актуальным является проведение масштабных эпидемиологических исследований с целью более полной оценки распространенности данных мутаций среди населения, а также выявления диагностически значимых мутаций.
ОТЕЧЕСТВЕННАЯ И МЕЖДУНАРОДНАЯ КЛИНИЧЕСКАЯ ПРАКТИКА МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ТЕСТИРОВАНИЯ ПАЦИЕНТОВ С РМЖ И/ИЛИ РЯ НА НАЛИЧИЕ МУТАЦИЙ В ГЕНАХ BRCA1 И BRCA2
С целью анализа степени внедрения молекулярно-генетического тестирования пациентов с РМЖ/РЯ на наличие мутаций в генах BRCA1/2 в отечественную и международную клиническую практику, а также области применения их результатов, был проведен поиск российских и зарубежных клинических рекомендаций по диагностике и лечению пациентов с РМЖ/РЯ. Поиск российских клинических рекомендаций проводился в рубрикаторе клинических рекомендаций Минздрава России http://cr.rosminzdrav.ru, а также в сети интернет. В обзор включались публикации российских клинических рекомендаций по диагностике и лечению пациентов с РМЖ или РЯ. Поиск зарубежных клинических рекомендаций по диагностике и лечению РМЖ и РЯ проводился в библиографической базе данных Medline (http:// www.ncbi.nlm.nih.gov/pubmed/), ряде других баз данных и на сайтах ассоциаций (Международной сети клинических рекомендаций, Женевского фонда медицинского образования и исследований, Национального института охраны здоровья и совершенствования медицинской помощи, Европейского общества медицинской онкологии, Американского общества клинической онкологии). Временной диапазон поиска был не ограничен. Дата осуществления поиска: 30.04.2019. В обзор включались актуальные (действующие) версии клинических рекомендаций по диагностике и лечению РМЖ и/или РЯ на английском языке, в которых был проведен контекстный поиск по ключевым словам (“BRCA”, “genetic mutation”, “genetic testing, genetic counsel”), с целью отбора публикаций, содержащих рекомендацию об обязательном проведении МГТ пациентов на наличие мутаций в генах BRCA1/2, а также информации о влиянии BRCA-статуса на дальнейшую тактику лечения.
Всего в результате поиска было найдено пять публикаций российских клинических рекомендаций по диагностике и лечению РМЖ и РЯ [41–45]. В рамках лабораторной диагностики РМЖ рекомендовано проведение консультации медицинского генетика и молекулярно-генетического тестирования на наличие мутаций в генах BRCA1/2 у ограниченной группы пациентов, соответствующих определенным критериям. Информация о влиянии BRCA-статуса на дальнейшую тактику ведения пациентов с РМЖ отсутствует. В рамках лабораторной диагностики РЯ рекомендовано проведение генетического консультирования и МГТ на наличие мутаций в генах BRCA1/2 у подгруппы пациентов с серозной и эндометриоидной карциномой высокой степени злокачественности. При этом положительный BRCA-статус влияет на ведение данных пациентов, в частности в случае платиночувствительного рецидива заболевания им рекомендовано назначение таргетного препарата олапариба.
В результате проведенного поиска также было найдено 763 публикации зарубежных клинических рекомендаций по диагностике и лечению РМЖ и/или РЯ, при этом по результатам дополнительного контекстного поиска в данный обзор были включены 19 актуальных публикаций зарубежных клинических рекомендаций [46–64]. В рамках лабораторной диагностики РМЖ при соответствии пациентов определенным критериям рекомендуется проведение генетического консультирования и МГТ на наличие мутаций в генах BRCA1/2, при этом BRCA-статус влияет на дальнейшую тактику ведения пациенток. В частности, рекомендуется назначение таргетного препарата олапариб пациенткам с BRCA-ассоциированным тройным негативным РМЖ, с BRCA-ассоциированным метастатическим и BRCA-ассоциированным распространенным РМЖ. По сравнению с российскими клиническими рекомендациями критерии для назначения и проведения генетического консультирования и МГТ представлены более широко.
В рамках лабораторной диагностики РЯ в некоторых зарубежных клинических рекомендациях рекомендовано проведение генетического консультирования и МГТ на наличие мутаций в генах BRCA1/2 в более широкой группе пациентов, например в клинических рекомендациях, разработанных Шотландской межуниверситетской сетью по клиническим рекомендациям, проведение МГТ рекомендовано всем пациентам с РЯ, раком маточных труб и первичным раком брюшины. Однако в некоторых клинических рекомендациях проведение тестирования, так же, как и в российских, показано узкой группе пациентов с серозными и эндометриоидными карциномами высокой степени злокачественности. При этом как в российских, так и в зарубежных клинических рекомендациях положительный BRCA-статус влияет на ведение данных пациентов, в частности в случае рецидива заболевания им рекомендовано назначение таргетного препарата олапариб или нирапариб (последний упоминается только в зарубежных клинических рекомендациях).
По результатам анализа отечественных и зарубежных клинических рекомендаций, а также нескольких отечественных статей, посвященных проблемам профилактики развития РМЖ/РЯ, в настоящее время в странах Европы и США сформирован комплексный подход к выявлению группы пациентов (мужчин и женщин) с высоким риском развития наследственного РМЖ/РЯ на основании их BRCA-статуса, их дальнейшему мониторингу и проведению профилактики развития злокачественных новообразований. При этом в России в рамках существующих клинических рекомендаций отсутствуют упоминания о необходимости проведения МГТ родственников пациентов с BRCA-ассоциированным РМЖ. В случае РЯ данная рекомендация существует, однако отсутствует информация о дальнейших мерах мониторинга и профилактики.
Это свидетельствует об актуальности вопроса раннего мониторинга и профилактики лиц высокого риска в российской клинической практике и потребности в формировании единообразных принципов и подходов к реализации данных мер.
НОВЫЕ ТЕХНОЛОГИИ ПРОВЕДЕНИЯ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКОГО ТЕСТИРОВАНИЯ
Следует отметить, что одним из главных факторов, влияющих на частоту выявляемых мутаций, являются критерии направления пациентов на генетическое тестирование. При этом эффективность выявления мутаций как с эпидемиологической, так и с клинической точки зрения, в свою очередь, во многом зависит от выбранной технологии МГТ. Так, в случае мутаций в генах BRCA необходимо учитывать большой размер генов – размер гена BRCA2 более чем в 2 раза больше гена BRCA1, при этом мутации в обоих генах могут быть расположены по всей их длине. Следовательно, чтобы точно определить, существует ли мутация в генах BRCA1/2, поиска конкретных мутаций может быть недостаточно (таргетное секвенирование), в таком случае их необходимо секвенировать полностью [9]. Выбор метода МГТ является важным этапом при проведении диагностики пациентов, так как достоверность полученного результата влияет на дальнейший сценарий терапии или выбор мер профилактики. Истинно положительные результаты МГТ пациентов с семейным анамнезом или родственников носителей мутации в генах BRCA1/2 указывают на то, что в гене была идентифицирована патогенная или потенциально патогенная мутация. При этом отрицательные результаты означают, что у человека был отрицательный результат теста на наличие патогенной мутации, которая, как известно, присутствует в его семье. В таких случаях считается, что риск РМЖ и/или РЯ снижается до уровня населения в целом. Неинформативные результаты возникают, когда МГТ пробанда не выявляет наличия патогенной мутации. Данные результаты, которые также включают как отсутствие обнаружения патогенной мутации (ложноотрицательные), так и обнаружение мутации неизвестного клинического значения, являются неоднозначными, поскольку в таких случаях нельзя исключать наследственный характер заболевания или наследственный риск его возникновения [9].
Выделяют несколько основных причин возникновения ложноотрицательных результатов при МГТ на наличие мутации в генах BRCA1/2 [9]: возникновение заболевания у родственника носило случайный характер (спорадическая форма заболевания); мутация расположена в регуляторной части гена (около 5–10% мутаций BRCA1 и BRCA2, по-видимому, происходят в регуляторной части гена, и они не обнаруживаются при обычном секвенировании генов); технические ограничения технологии МГТ (например, методы, используемые для поиска мутаций) обычно фокусируются на геномной ДНК и основаны на амплификации (полимеразной цепной реакции, ПЦР) и позволяют обнаруживать изменения последовательности, такие как точечные мутации, небольшие делеции и вставки, при этом обнаружить потерю частичных или полных экзонов данными методами невозможно, так как присутствие гомологичного нормального аллеля приводит к положительному результату ПЦР.
В настоящее время активное распространение получили технологии МГТ, основанные на новых поколениях методов анализа нуклеотидной последовательности ДНК, т.е. на методах секвенирования [65]. Все методы секвенирования, появившиеся после первого поколения, называются секвенированием нового поколения или технологиями NGS (англ. new generation sequencing) [66]. NGS позволяет выявлять не только мутации BRCA1/2, но и другие мутации, ассоциированные с наследственным РМЖ и/или РЯ.
Всего выделяют три основных поколения технологий секвенирования ДНК: 1) первое поколение: секвенирование по Сэнгеру (англ. Sanger sequencing), секвенирование по методу Максама–Гилберта (англ. Maxam–Gilbert sequencing); 2) второе поколение (массовое параллельное консенсусное секвенирование): пиросеквенирование (англ. pyrosequencing), синтез с обратимой терминацией (англ. reversible termination), секвенирование через лигирование (англ. ligation), ионное полупроводниковое секвенирование (англ. ion semiconductor sequencing); 3) третье поколение (массовое параллельное одномолекулярное секвенирование): одномолекулярное секвенирование в режиме реального времени (англ. single molecule real time sequencing, SMRT), нанопоровое секвенирование (англ. nanopore sequencing).
Выделяют следующие области применения платформ NGS в научно-исследовательской практике [67]:
− определение последовательности всей ДНК – полногеномное секвенирование (англ. whole-genome sequencing, WGS);
− таргетное секвенирование:
• определение последовательности белок-кодирующих участков генома – полноэкзомное секвенирование (англ. whole-exome sequencing, WES);
• определение последовательности интересующих генов (сюда относятся многие коммерческие решения – от “клинических экзомов”, включающих около 5000 медицински значимых генов, до малых таргетных панелей, анализирующих 1–3 гена);
− секвенирование транскриптома – РНК-секвенирование (RNA-seq), которое часто используется в онкологии для классификации опухолей, нахождения неоантигенов, поиска новых химерных генов и т.д.
В клинической практике технологии NGS являются эффективным методом поиска новых и редких соматических мутаций. Полногеномное, полноэкзомное и даже таргетное секвенирование (с использованием мультигенных таргетных генетических панелей) могут быть использованы для поиска новых генетических аберраций и связанных с ними потенциальных терапевтических мишеней для различных локализаций злокачественных опухолей. При этом секвенирование нового поколения позволяет обнаружить редкие генетические варианты и протестировать одновременно большое число генов предрасположенности (MLH1, MSH2, TP53, CHEK2, PALB2, PTEN, NBS1, ATM, BRIP1, RAD50, BLM, FGFR2), ассоциированных с повышенным риском развития РМЖ и/или РЯ, на наличие в них клинически значимых мутаций в короткие сроки, в отличие от традиционных молекулярно-генетических методов (первого поколения) [3, 68].
К секвенированию первого поколения относится метод ферментативного секвенирования по Сэнгеру (метод “обрыва” цепи или метод терминантов). Он основан на использовании радиоактивно меченных нуклеотидов и ДНК-полимеразы (фрагмента Кленова ДНК-полимеразы I) и в настоящее время является “золотым” стандартом секвенирования. Основными преимуществами данного метода являются дешевизна (в случае секвенирования небольшого участка ДНК и небольшого числа образцов), точность и простота автоматизации. При этом он обладает рядом недостатков: трудность прочтения длинных последовательностей, состоящих из одного вида нуклеотидов; ограниченная способность обрабатывать и анализировать частоты аллелей; сборка de novo коротких ридов, содержащих повторы, невозможна без использования физических карт высокого разрешения; трудоемкий протокол; низкая производительность (ограниченный объем и скорость секвенирования); дороговизна (в случае большого участка ДНК – за счет стоимости одной килобазы) [69–71]. Метод секвенирования Максама–Гилберта также относится к первому поколению, однако основан на химическом гидролизе меченого фрагмента ДНК (только по одному концу) под действием специфических реагентов и применим к фрагментам ДНК до 250 нуклеотидов. Особенностью данного метода является отсутствие этапа амплификации. При этом одним из основных его недостатков является использование токсичных и радиоактивных химических веществ, в связи с чем данный метод не получил широкого распространения [66].
Ко второму поколению относятся секвенирующие платформы массового параллельного консенсусного секвенирования, основанные на принципе дробного секвенирования (англ. shotgun sequencing) и позволяющие секвенировать большое количество молекул ДНК одновременно. В целом такие технологии секвенирования основаны на процессе определения последовательности нуклеотидов в геномной ДНК или в совокупности информационных РНК (транскриптоме) с помощью амплификации множества коротких участков генов. Это разнообразие генных фрагментов охватывает всю совокупность целевых генов или, при необходимости, весь геном (или транскриптом как совокупность молекул РНК). Для данных методов характерны многие черты секвенирования по Сэнгеру, например, в них используются полимеразы для синтеза, модифицированные нуклеотиды и флуоресцентное детектирование. Кроме того, они включают этап амплификации ДНК (клонирования) с образованием кластеров идентичной ДНК с высокой плотностью перед секвенированием. Отличием от метода Сэнгера является то, что в нем реакция секвенирования происходит в жидкой фазе, в то время как в платформах второго поколения молекулы ДНК связаны с твердой поверхностью, на которой происходят амплификация и секвенирование. Производительность таких платформ позволяет генерировать от 500 миллионов до миллиарда пар оснований за один прогон, что позволяет установить присутствие сотен и тысяч генных мутаций или вариантов в исследуемых клетках за относительно короткий период времени (от нескольких часов до нескольких суток) [3, 6, 8–10].
Основными этапами, характерными для всех технологий секвенирования второго поколения, являются [10, 11]: 1) подготовка библиотеки (фрагментирование ДНК или мРНК до 300–500 нуклеотидных пар, лигирование (присоединение) сиквенсовых адаптеров (синтезированные олигонуклеотиды с известной последовательностью); 2) амплификация (клонирование этих коротких последовательностей посредством большого числа специфических ДНК-зондов и с применением мультиплексной ПЦР); 3) высокопроизводительное прочтение нуклеотидных последовательностей генных фрагментов (секвенирование и анализ данных).
Основными преимуществами технологий секвенирования второго поколения являются [66]: осуществление миллионов коротких операций чтения параллельно; большая скорость секвенирования по сравнению с первым поколением; низкая стоимость секвенирования.
Пиросеквенирование
Пиросеквенирование основано на методике секвенирования с помощью синтеза (англ. sequencing-by-synthesis technique, SBS), однако вместо измерения растущей цепи ДНК в данном методе проводится обнаружение побочного продукта реакции синтеза для контроля включения меченых оснований [72]. В первую очередь, молекулы ДНК гибридизируются на специальных шариках (твердая фаза), количество которых должно быть достаточным для того, чтобы одна молекула ДНК связывалась с одной гранулой. Иммобилизированные фрагменты ДНК далее подвергаются амплификации с помощью эмульсионной ПЦР. Реакция секвенирования начинается с добавления универсального праймера, ДНК-полимеразы и одного из четырех нуклеотидов. При внедрении первого добавленного нуклеотида ДНК-полимеразой в растущую цепь ДНК высвобождается неорганический фосфат-ион, который, в свою очередь, инициирует ферментативную каскадную реакцию (с ферментами АТФ-сульфурилазой и люциферазой), которая приводит к выбросу вспышки света (хемилюминесцентного сигнала). Излучаемый световой сигнал детектируется CCD-камерой и пропорционален количеству добавленных идентичных нуклеотидов. Избыток нуклеотида затем смывают и добавляют следующий, соответствующий другому основанию, после чего цикл повторяется. Добавление нуклеотидов по отдельности и последовательно позволяет соотнести хемилюминесцентный сигнал с конкретным нуклеотидом. Вся пластинка визуализируется после добавления каждого нуклеотида, что позволяет регистрировать местоположение и интенсивность генерируемого света. Затем данные анализируются для создания последовательности чтения [72].
Основным недостатком пиросеквенирования являются трудности в секвенировании отрезков идентичных нуклеотидов (гомополимерных областей) длиннее пяти нуклеотидов, это связано с тем, что идентификация размера гомополимеров должна осуществляться по интенсивности света, испускаемого пиросеквенированием. Однако сигналы слишком высокой или слишком низкой интенсивности приводят к занижению или завышению числа нуклеотидов, что вызывает ошибки идентификации нуклеотидов [3, 8].
Синтез с обратимой терминацией
Данный метод, так же как и пиросеквенирование, относится к типу секвенирования с помощью синтеза (SBS) и включает в себя три основных этапа. На первом этапе проводится фрагментация молекул ДНК на случайные последовательности, после чего с обоих концов полученных фрагментов лигируются адаптеры. Затем адаптеры прикрепляются к соответствующим комплементарным праймерам, закрепленным на специальной пластине. На втором этапе каждый прикрепленный фрагмент амплифицируется с помощью твердофазной ПЦР (“мостиковая” амплификация). В результате образуется набор копий каждого конкретного фрагмента – кластер, содержащий около миллиона копий. На последнем этапе проводится определение нуклеотидной последовательности фрагментов ДНК. Для этого добавляются четыре модифицированных нуклеотида (с флуоресцентной меткой), секвенирующие праймеры и ДНК-полимераза. Праймеры гибридизируются с фрагментами ДНК, после чего с помощью ДНК-полимеразы они удлиняются за счет модифицированных нуклеотидов. Далее кластеры подвергаются воздействию лазера для испускания светового сигнала, специфичного для каждого нуклеотида, который улавливается CCD-камерой [3, 9].
К преимуществам метода относятся высокая точность, универсальность, доступность программного обеспечения для обработки и анализа результатов, а также наименьшая цена получаемых данных (в расчете на нуклеотид).
Одним из основных недостатков данного метода является высокое требование к контролю загрузки образцов, поскольку перегрузка может привести к перекрытию кластеров и низкому качеству секвенирования. Общая частота ошибок этой технологии секвенирования составляет около 1%. Кроме того, к недостаткам относятся: высокая цена реагентов, проблемы с секвенированием матриц с низкой сложностью и большая длительность прогона [4, 12].
Секвенирование через лигирование
В основе данного метода, в отличие от предыдущих, лежит не реакция ДНК синтеза, а лигирование нуклеотидных зондов с флуоресцентными метками, что позволяет устанавливать последовательность матричной цепи ДНК, состоящей из двух нуклеотидов одновременно. Первым этапом является фрагментация матричной цепи ДНК на короткие последовательности с последующем легированием нуклеотидных адаптеров двух типов. После чего происходит иммобилизация одноцепочечных фрагментов на твердых шариках, которая происходит с помощью отжига праймера (образования дуплексных комплексов адаптеров и комплементарных им праймеров, расположенных на шарике) с последующей амплификацией фрагментов ДНК методом эмульсионной ПЦР. Секвенирование осуществляется с помощью лигирования восьми-нуклеотидных зондов с флуоресцентной меткой, у которых первые два основания используются для определения нуклеотидной последовательности фрагмента, а остальные шесть являются вырожденными. Присоединение зонда осуществляется с помощью ДНК-лигазы, после чего избыточные праймеры вымываются и регистрируется флуоресцентный сигнал. Затем три основания праймера с прикрепленной меткой удаляются и процесс повторяется многократно в рамках одного цикла. После этого синтезированная цепь денатурируется и проводится еще несколько циклов секвенирования (всего 5), при этом в каждом последующем используется праймер, укороченный на одно основание. Таким образом, нуклеотидная последовательность матричной ДНК преобразуется в последовательность флуоресцентных сигналов [3, 6, 8, 9].
Недостатком данного метода является вероятность возникновения ошибок секвенирования, в частности наиболее характерными ошибками для данного метода являются замены. Риск возникновения ошибок при использовании данного метода может быть связан со следующими факторами [70]: иммобилизация на одном шарике смешанных фрагментов ДНК, а не уникальных (снижение качества проводимых реакций, а также фильтрации данных); маленькое расстояние между шариками (ошибки прочтения); снижение интенсивности сигнала и проблемы с удалением флуоресцентных меток (увеличение количества ошибок по мере проведения циклов).
Ионное полупроводниковое секвенирование
В отличие от всех описанных выше методов второго поколения, использующих оптический способ обнаружения для расшифровки последовательности ДНК, данный метод предполагает использование электрического детектирования. Принцип ионного полупроводникового секвенирования похож на пиросеквенирование, однако в отличие от него не предполагает использования нуклеотидов с флуоресцентной меткой. Общими стадиями являются: фрагментация матричной ДНК, лигирование олигонуклеотидных адаптеров, амплификация на шариках с помощью эмульсионной ПЦР. Присоединение праймеров с помощью ДНК-полимеразы в процессе секвенирования сопровождается выделением иона водорода, присутствие которого фиксируется специальными датчиками и преобразовывается в электрический сигнал [4, 9].
Преимуществами данного метода являются большая длина ридов, по сравнению с другими методами, и быстрота секвенирования (от 2 до 8 ч), а также относительно низкая цена за запуск. При этом основными недостатками являются сложность интерпретации гомополимерных последовательностей (более 6 пн), что приводит к ошибке вставки и удаления независимо от нелинейного электрического ответа, а также низкая производительность [66].
Методы массового параллельного консенсусного секвенирования основаны на разных методиках с точки зрения биохимии, однако они имеют много общего. Этапы подготовки библиотеки начинаются со случайной фрагментации ДНК с последующим лигированием платформо-специфических адаптеров на конце каждого фрагмента. Эти адаптеры затем используются для амплификации фрагмента на твердой поверхности с помощью ДНК-полимеразы. Продукты амплификации пространственно сгруппированы на матрице перед началом секвенирования. Процесс расшифровки нуклеотидной последовательности ДНК выполняется с помощью нескольких этапов сбора биохимических и флуоресцентных сигналов, полученных в результате секвенирования. Только системы, основанные на методе ионного полупроводникового секвенирования, используют электрический метод обнаружения. Все платформы второго поколения также имеют возможность считывать оба конца фрагмента ДНК. Методы секвенирования второго поколения способны расшифровывать в несколько раз больше данных, чем традиционное секвенирование по Сэнгеру. Технологии массового параллельного секвенирования позволяют одновременно определять нуклеотидные последовательности множества различных нитей ДНК при осуществлении процессов синтеза или лигирования ДНК, что обеспечивает чтение миллиардов нуклеотидов в день [73].
К секвенированию третьего поколения относятся методы массового параллельного одномолекулярного секвенирования, позволяющие избежать амплификации матрицы ДНК до процесса секвенирования. Целью данных технологий является последовательность отдельных молекул ДНК, предпочтительно в режиме реального времени. Потенциальными преимуществами использования одномолекулярного секвенирования являются [73]: минимальное количество требуемой исходной ДНК; отсутствие риска возникновения ошибок при амплификации; асинхронный синтез; быстрота выполнения работы; способность исследовать характеристики отдельных молекул ДНК.
Одномолекулярное секвенирование в режиме реального времени
Принцип данной технологии заключается в одномолекулярном секвенировании в режиме реального времени с использованием нуклеотидов с флуоресцентными метками. При этом, в отличие от методов предыдущего поколения, отсутствует этап амплификации нуклеотидов. Детектирующие сигналы обнаруживаются в режиме реального времени, так как они испускаются непосредственно при встраивании нуклеотидов. Для проведения секвенирования используется структура, состоящая из множества ячеек (нанофотонных камер), каждая из которых содержит тысячи волноводов с нулевой модой (англ. zero-mode waveguide, ZMW), на дне каждого из которых иммобилизирована молекула ДНК-полимеразы. Во время реакции секвенирования в фрагмент ДНК с помощью ДНК-полимеразы встраиваются нуклеотиды с флуоресцентными метками (разные цвета для каждого типа). При встраивании нуклеотид выпускает световой сигнал, который регистрируется датчиками. Обнаружение меченых нуклеотидов позволяет определить последовательность ДНК [66].
К преимуществам данного метода относятся высокая производительность, быстрая подготовка образца (от 4 до 6 ч вместо дней), высокая производительность (в среднем ~10 кбит/с, в отдельных случаях может достигать 60 кбит/с). При этом к недостаткам можно отнести высокий уровень ошибок – около 13%, с преобладанием ошибок вставок и удалений [66].
Нанопоровое секвенирование
Данный метод был разработан на основе исследований по транслокации ДНК через различные искусственные нанопоры и, в отличие от большинства современных секвенсоров ДНК, не подразумевает использования нуклеотидных меток и этапа обнаружения.
Секвенирование ДНК с помощью данного метода основано на преобразовании электрического сигнала нуклеотидов путем прохождения фрагмента ДНК через нанопору, которая представляет собой пору из α-гемолизина, ковалентно связанного с молекулой циклодекстрина, встроенного в синтетическую мембрану с высоким электрическим сопротивлением. Нанопоры располагаются в отдельных лунках, в процессе трансляции молекулы ДНК постепенно расщепляются экзонуклеазой, при этом происходит изменение ионного тока, который блокируется отщепленным нуклеотидом. Все изменения ионного тока регистрируются, а периоды его блокировки позволяют определить нуклеотидную последовательность ДНК. Кроме того, данный метод также позволяет идентифицировать характеристики и параметры (диаметра, длины и конформации) ДНК молекулы [4, 9].
К преимуществам данного метода относятся низкая стоимость и маленький размер секвенатора. При этом для него характерна высокая частота ошибок (около 12%, приблизительно 3% составляют несовпадения, 4% – вставки и 5% – удаления) [66].
В табл. 1 представлены основные платформы для секвенирования первого, второго и третьего поколений и их основные характеристики.
Таблица 1.
Основные платформы для секвенирования первого, второго и третьего поколений и их основные характеристики [3, 8, 13, 14]
| Поколение | Платформа | Метод секвенирования | Подготовка образца | Способ детектирования | Основной тип ошибок | Длина рида, пн | Время работы | Производительность (за запуск) |
|---|---|---|---|---|---|---|---|---|
| Первое | Секвенирвоание по Сэнгеру | Ферментативная терминация цепи | Клонирование/ ПЦР | Оптический | Замена | 900 | н.д. | н.д. |
| Второе | Roche GS Junior | Пиросеквенирование | Эмульсионная ПЦР | Оптический | Вставка | 400 | 10 ч | 35 Мб |
| Roche GS FLX Titanium XL | До 1000 | 23 ч | 700 Мб | |||||
| Roche GS FLX Titanium XLR 70 | До 600 | 10 ч | 450 Мб | |||||
| Illumina HiSeq 1000 | Синтез с обратимой терминацией | Мостиковая амплификация | Оптический | Замена | 1 × 35 2 × 50 2 × 100 |
1.5 дня 4.5 дня 8.5 дней | 47–52 Гб 135–150 Гб 270–300 Гб |
|
| Illumina HiSeq 2000 | 1 × 36 2 × 50 2 × 100 |
1.5 дня 4.5 дня 8.5 дней | 95–105 Гб 270–300 Гб 540–600 Гб |
|||||
| Illumina Genome Analyzer IIx | 1 × 35 2 × 50 2 × 75 2 × 100 2 × 150 |
2 дня 5 дней 7 дней 9.5 дней 14 дней | 10–12 Гб 25–30 Гб 37.5–45 Гб 54–60 Гб 85–95 Гб |
|||||
| Illumina Mi-Seq | 1 × 35 2 × 100 2 × 150 |
4 ч 19 ч 27 ч |
120 Мб 680 Мб 1 Гб |
|||||
| SOLiD 5500 System | Лигирование | Эмульсионная ПЦР | Оптический | Замена | Метод спаренных концов: 2 × 60, метод парных прочтений: 75 × 35, фрагментация: 75 |
2–7 дней в зависимости от образца | 7–9 Гб/день | |
| SOLiD 5500xl System | 10–15 Гб/день | |||||||
| Ion Torrent PGM | Ионное полупроводниковое секвенирование | Эмульсионная ПЦР | Электронный | Вставка | 100 | 1–2 ч | 1 Гб | |
| Третье | PacBio RS II | Одномолекулярное секвенирование в режиме реального времени | Амплификация отсутствует | Оптический | Вставка; удаление | 10 000–15 000 | До 240 мин | 2 Гб |
| PacBio Sequel System | 10 000–15 000 | До 240 мин | 20 Гб | |||||
| MinION (Oxford Nanopore Technologies) | Нанопоровое секвенирование | н.д. | Оптический/ электронный | Несовпадение; вставка; удаление | до 2 млн | До 2 ч | До 50 Гб | |
| PromethION (Oxford Nanopore Technologies) | До 72 ч | 220 Гб |
Выделяют следующие области применения платформ NGS в научно-исследовательской практике [10, 11]:
1) определение последовательности всей ДНК – полногеномное секвенирование (англ. whole-genome sequencing, WGS);
2) таргетное секвенирование: определение последовательности белок-кодирующих участков генома – полноэкзомное секвенирование (англ. whole-exome sequencing, WES); определение последовательности интересующих генов (сюда относятся многие коммерческие решения – от “клинических экзомов”, включающих около 5000 медицински значимых генов, до малых таргетных панелей, анализирующих 1–3 гена);
3) секвенирование транскриптома – РНК-секвенирование (RNA-seq), которое часто используется в онкологии для классификации опухолей, нахождения неоантигенов, поиска новых химерных генов и т.д.
Полногеномное, полноэкзомное и даже таргетное секвенирование (с использованием мультигенных таргетных генетических панелей) могут быть использованы для поиска новых генетических аберраций и связанных с ними потенциальных терапевтических мишеней для различных локализаций злокачественных опухолей. При этом секвенирование нового поколения позволяет обнаружить редкие генетические варианты и протестировать одновременно большое число генов предрасположенности на наличие в них клинически значимых мутаций в короткие сроки, в отличие от традиционных молекулярно-генетических методов (первого поколения) [8].
С целью анализа эффективности технологий МГТ был проведен поиск научных публикаций по оценке эффективности применения генетических тестов у пациентов с РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 в следующих базах данных: кокрановская библиотека и библиографическая база данных Medline (http:// www.ncbi.nlm.nih.gov/pubmed/). Временной диапазон поиска не был ограничен. Поиск был проведен по запросу: BRCA[All Fields] AND testing[All Fields]. В данный анализ включались публикации по результатам исследований, в которых проводилась оценка эффективности технологий МГТ на наличие мутаций в генах BRCA1/2 у пациентов с РМЖ/РЯ по следующим критериям эффективности: чувствительность, специфичность.
В результате проведенного систематического поиска были найдены 22 публикации, в которых оценивалась эффективность технологий МГТ у пациентов с РМЖ/РЯ на наличие мутации в генах BRCA1/2.
В 16 публикациях исследований [74–89], найденных по данному поисковому запросу, были представлены результаты эффективности методов таргетного NGS в сравнении с секвенированием по методу Сэнгера, в трех публикациях – результаты оценки эффективности технологий тестирования на основе ПЦР: DHPLC-метода [90], анализа плавления с высоким разрешением (технология HRM-анализа) [91] и метода ПЦР с использованием капиллярного электрофореза [92] по сравнению с секвенированием по методу Сэнгера (данные технологии рассматриваются в качестве методов предварительного скрининга для выявления вариантов (аномалий) генетической последовательности с последующим секвенированием [9, 93]). В трех публикациях исследований представлены результаты оценки эффективности технологий генетического тестирования (NGS) [94–96] и анализа плавления с высоким разрешением (технология HRM-анализа) по сравнению с методом мультиплексной амплификации лигированных зондов (англ. multiplex ligation-dependent probe amplification, MLPA). С целью соблюдения методологически корректного подхода к интерпретации результатов данные сравнения были выделены в отдельную группу, так как несмотря на то что согласно исследованию A. Germani et al. [95] метод MLPA, так же, как и секвенирование по методу Сэнгера, признан “золотым стандартом” определения мутаций в генах BRCA1/2 и используется в рутинной практике, невозможно рассматривать данные методы как сопоставимый контроль.
Эффективность генетических тестов у пациентов с РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 оценивалась по таким критериям, как точность, чувствительность, специфичность и конкордантность.
По результатам проведенного обзора чувствительность методов таргетного NGS по сравнению с секвенированием по методу Сэнгера у пациентов с РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляет от 95 до 100%. В 71% найденных исследований (10 из 14) чувствительность составила более 99%. При этом специфичность методов таргетного NGS по сравнению с секвенированием по методу Сэнгера у пациентов с РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляет от 91 до 100%. В 79% найденных исследований (11 из 14) специфичность составила более 99%.
Согласно анализу результатов найденных исследований, чувствительность метода ПЦР с использованием капиллярного электрофореза на платформе Experion Automated Electrophoresis system (Bio-Rad Laboratories) по сравнению с секвенированием по методу Сэнгера у пациентов с РЯ при определении наличия мутации в генах BRCA1/2 составляет 100%, при этом чувствительность DHPLC-метода по сравнению с секвенированием по методу Сэнгера у пациентов с синдромом наследственного РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляет 95%.
Чувствительность и специфичность метода ПЦР с использованием HRM-анализа по сравнению с секвенированием по методу Сэнгера у пациентов с синдромом наследственного РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляют 100 и 98.1% соответственно.
Чувствительность и специфичность метода таргетного NGS на платформе HiSeq2500 (Illumina) с использованием инструмента DECoN по сравнению с методом MLPA у пациентов с РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляет 100 и 99% соответственно.
Чувствительность, специфичность и точность метода таргетного NGS на платформе Ion Torrent PGM (Thermo Fisher Scientific) по сравнению с методом MLPA и/или методом MAQ у пациентов с РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляет 100, 100 и 100% соответственно. Чувствительность и конкордантность HRM-анализа на основе ПЦР по сравнению с методом MLPA у пациентов с синдромом наследственного РМЖ/РЯ при определении наличия мутации в генах BRCA1/2 составляют 100 и 100% соответственно.
ЗАКЛЮЧЕНИЕ
Наиболее распространенными в российской популяции BRCA-мутациями являются 5382insC, 4153delA, 185delAG, 300T>G (в гене BRCA1) и 6174delT (в гене BRCA2). При этом отсутствуют доказательства статистически значимой корреляции между наличием данных мутаций и предрасположенности к наследственному РМЖ/РЯ. С точки зрения диагностики важным является обнаружение так называемых мутаций с эффектом основателя, специфических для различных этнических популяций. Однако с учетом этнического разнообразия населения России и ограниченности исследований, посвященных данному вопросу, актуальным является проведение масштабных эпидемиологических исследований с целью более полной оценки распространенности данных мутаций среди населения, а также выявления диагностически значимых мутаций.
Сравнение отечественных и зарубежных клинических рекомендаций показало, что область применения результатов МГТ на BRCA-мутации в международной клинической практике шире, чем в российской. В отличие от отечественных рекомендаций в некоторых странах проведение таргетной терапии PARP-ингибиторами рекомендовано более широкой подгруппе пациентов с РМЖ и РЯ. С точки зрения использования BRCA-статуса для ранней диагностики и профилактики в отечественной практике отсутствуют какие-либо рекомендации на этот счет, при этом в странах Европы и США сформирован системный подход к выявлению группы пациентов повышенного риска развития наследственного РМЖ/РЯ, а также их мониторингу и мерам профилактики.
Важный вопрос для клинической практики – выбор технологии МГТ. Несмотря на возможную вероятность возникновения ошибки при использовании технологий NGS, их преимуществом является возможность определения мутации в любой части генов BRCA1/2, в отличие от других технологий.
Таким образом, актуально проведение эпидемиологических исследований с целью более полной оценки распространенности BRCA-мутаций среди населения, а также выявление диагностически значимых мутаций. Кроме того, актуальным является рассмотрение возможности более широкого внедрения молекулярно-генетического тестирования в клиническую практику ведения пациентов с РМЖ и РЯ, а также создания точного подхода к ранней диагностике, профилактике и терапии группы пациентов с положительным BRCA-статусом.
Исследование проведено без финансовой поддержки.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Каприн А.Д., Старинский В.В., Петрова Г.В. Состояние онкологической помощи населению России в 2018 году. М.: МНИОИ им. П.А. Герцена, филиал ФГБУ “НМИЦ радиологии” Минздрава России, 2019. 236 с.
Каприн А.Д., Старинский В.В., Петрова Г.В. Злокачественные новообразования в России в 2018 году (заболеваемость и смертность). М.: МНИОИ им. П.А. Герцена, филиал ФГБУ “НМИЦ радиологии” Минздрава России, 2019. 250 с.
Любченко Л.Н., Батенева Е.И., Абрамов И.С. и др. Наследственный рак молочной железы и яичников // Злокачественные опухоли. 2013. Т. 6. № 2. С. 53–61. https://doi.org/10.18027/2224-5057-2013-2-53-61
Лаптиев С.А., Корженевская М.А., Соколенко А.П. и др. Медико-генетическое консультирование при наследственных формах рака молочной железы и рака яичников // Уч. записки СПбГМУ им. И.П. Павлова. 2018. Т. 25. № 2. С. 7–18. https://doi.org/10.24884/1607-4181-2018-25-2-7-18
Бит-Сава Е.М., Белогурова М.Б. Наследственный рак молочной железы // Сиб. онкологич. журн. 2013. Т. 55. № 1. С. 75–81.
Chen S., Parmigiani G. Meta-analysis of BRCA1 and BRCA2 penetrance // J. Clinical Oncology : Official J. Am. Soc. Clin. Oncology. 2007. V. 25. № 11. P. 1329–1333. https://doi.org/10.1200/JCO.2006.09.1066
Høberg-Vetti H., Bjorvatn C., Fiane B.E. et al. BRCA1/2 testing in newly diagnosed breast and ovarian cancer patients without prior genetic counselling: the DNA-BONus study // Eur. J. Hum. Genet.: EJHG. 2016. V. 24. № 6. P. 881–888. https://doi.org/10.1038/ejhg.2015.196
Новикова Е.И., Снигирева Г.П. Секвенирование “Нового поколения” (NGS): применение для молекулярно-генетических исследований в онкологии // Вестник Рос. науч. центра рентгенорадиологии Минздрава России. 2016. Т. 16. № 1.
Bellosillo B., Tusquets I. Pitfalls and caveats in BRCA sequencing // Ultrastructural Pathology. 2006. V. 30. № 3. P. 229–235. https://doi.org/10.1080/01913120500521281
Sokolenko A.P., Rozanov M.E., Mitiushkina N.V. et al. Founder mutations in early-onset, familial and bilateral breast cancer patients from Russia // Familial Cancer. 2007. V. 6. № 3. P. 281–286. https://doi.org/10.1007/s10689-007-9120-5
Автомонов Д.Е., Пароконная А.А., Любченко Л.Н. и др. Анамнестические особенности BRCA1-ассоциированного рака молочной железы у женщин молодого возраста // Опухоли женской репродуктивной системы. 2012. № 3–4. С. 54–57. https://doi.org/10.17650/1994-4098-2012-0-3-4-54-57
Часовникова О.Б., Митрофанов Д.В., Демченко Д.О. и др. Анализ встречаемости девяти мутаций в генах BRCA1 и BRCA2 у больных раком молочной железы в Сибирском регионе // Сиб. онкологич. журн. 2010. Т. 41. № 5. С. 32–35.
Шмидт А.В. Мутация гена BRCA как фактор риска развития рака молочной железы на примере жительниц Челябинской области // Лучшая студенческая статья. 2017. С. 162–165.
Снигирева Г.П., Румянцева В.А., Новикова Е.И. и др. Алгоритм молекулярно-генетического обследования для выявления наследственного BRCA-ассоциированного рака молочной железы // Альманах клинич. медицины. 2019. Т. 47. № 1. С. 54–65. https://doi.org/10.18786/2072-0505-2019-47-002
Батенева Е.И., Филиппова М.Г., Тюляндина А.С. и др. Результаты генетического скрининга герминальных мутаций в генах BRCA1 и BRCA2 у больных раком молочной железы и больных раком яичников в российской популяции // Онкогинекология. 2015. № 3. С. 34–39.
Богомолова О.А., Шатова Ю.С., Верескунова М.И. и др. Герминальные мутации в генах BRCA1 и BRCA2 у пациенток Юга России с клиническими признаками наследственного рака молочной железы // Соврем. проблемы науки и образования. 2017. № 5. С. 114.
Зикиряходжаев А.Д., Новикова О.В., Сухотько А.С. и др. Частота мутаций в генах BRCA1 или BRCA2 у больных раком молочной железы // Междунар. научно-практ. конф. “РЕПРОДУКТИВНЫЕ ТЕХНОЛОГИИ В ОНКОЛОГИИ”. 2015. С. 70.
Марковский А.В., Страмбовская Н.Н., Попикова С.А. Полиморфизм генов BRCA 1/2 и белков фолатного цикла у больных раком молочной железы в Забайкалье // Забайкальский мед. вестник. 2015. № 1. С. 116–120.
Мартинков В.Н., Силин А.Е., Надыров Э.А. и др. Генетическое тестирование мутаций BRCA1, BRCA2 и CHEK2 у пациенток с первично-множественным раком молочной железы // Мол. и прикладная генетика. 2015. № 19. С. 64–69.
Налетов А.А., Сабиров А.Х., Федоров Н.М. и др. Оптимизация диагностики, индивидуализация лечения и диспансерного наблюдения больных с BRCA-обусловленными формами рака молочной железы // Мед. наука и образование Урала. 2013. Т. 14. № 1. С. 137–139.
Наседкина Т.В., Громыко О.Е., Емельянова М.А. и др. Определение герминальных мутаций в генах BRCA1, BRCA2 и CHEK2 с использованием биочипов у больных раком молочной железы // Мол. биология. 2014. Т. 48. № 2. С. 243–250.
Фролов И.М., Телышева Е.Н., Новикова Е.И. и др. Первично-множественный рак молочной железы (комплексная лучевая и молекулярно-генетическая диагностика) // Вестник Рос. науч. центра рентгенорадиологии Минздрава России. 2015. Т. 1. № 4.
Smirnova T.Y., Pospekhova N.I., Lyubchenko L.N. et al. High incidence of mutations in BRCA1 and BRCA2 genes in ovarian cancer // Bul. Experim. Biol. Medicine. 2007. V. 144. № 1. P. 83–85. https://doi.org/10.1007/s10517-007-0261-1
Suspitsin E.N., Sherina N.Y., Ponomariova D.N. et al. High frequency of BRCA1, but not CHEK2 or NBS1 (NBN), founder mutations in Russian ovarian cancer patients // Hereditary Cancer Clin. Practice. 2009. V. 7. № 1. P. 5. https://doi.org/10.1186/1897-4287-7-5
Батенева Е.И., Филиппова М.Г., Тюляндина А.С. и др. Высокая частота мутаций в генах BRCA1, BRCA2, CHEK2, NBN, BLM у больных раком яичников в российской популяции // Опухоли женской репродуктивной системы. 2014. № 4. С. 51–56.
Григорук О.Г., Пупкова Е.Э., Базулина Л.М., Лазарев А.Ф. Проведение молекулярно-генетических исследований с использованием ДНК клеток опухоли, полученных из цитологических препаратов // Лабораторная служба. 2017. Т. 6. № 1. С. 23–28. https://doi.org/10.17116/labs20176123-28
Коломиец Л.А., Чернышова А.Л., Чуруксаева О.Н. и др. Оптимизация современных подходов в лечении гинекологического рака основных локализаций // Злокачественные опухоли. 2017. Т. 7. № 3. Спецвыпуск 1. С. 66–72. https://doi.org/10.18027/2224-5057-2017-7-3-66-72
Федорова О.Е., Любченко Л.Н., Паяниди Ю.Г. и др. Использование биочипов при изучении распространенных мутаций в генах BRCA1/2 и CHEK2 у больных органоспецифическим раком яичников и первично-множественными злокачественными новообразованиями с поражением яичников (российская популяция) // Мол. биология. 2007. Т. 41. № 1. С. 37–42.
Хохлова С.В., Горбунова В.А., Любченко Л.Н., Имянитов Е.Н. BRCA-ассоциированный рак яичников (опыт отделения химиотерапии ФГБУ “РОНЦ им. Н.Н. Блохина” Минздрава России) // Соврем. онкология. 2016. Т. 18. № 1. С. 37–44.
Tyulyandina A., Gorbunova V., Khokhlova S. et al. Profile of BRCA1/BRCA2 mutations in Russian ovarian cancer population detected by NGS and MLPA analysis: Interim results of OVATAR study // Proc. Am. Association Cancer Res. 2018. V. 59. P. 320.
Janiszewska H., Haus O., Lauda-Swieciak A. et al. Frequency of three BRCA1 gene founder mutations in breast/ovarian cancer families from the Pomerania–Kujawy region of Poland // Clin. Genet. 2003. V. 64. № 6. P. 502–508. https://doi.org/10.1046/j.1399-0004.2003.00178.x
Perkowska M., BroZek I., Wysocka B. et al. BRCA1 and BRCA2 mutation analysis in breast ovarian cancer families from northeastern Poland // Hum. Mutat. 2003. V. 21. № 5. P. 553–554. https://doi.org/10.1002/humu.9139
Csokay B., Tihomirova L., Stengrevics A. et al. Strong founder effects in BRCA1 mutation carrier breast cancer patients from Latvia // Hum. Mutat. 1999. V. 14. № 1. P. 92. https://doi.org/10.1002/(sici)1098-1004(1999)14:1<92:: aid-humu23>3.0.co;2-2
Anisimenko M.S., Mitrofanov D.V., Chasovnikova O.B. et al. BRCA1 gene mutations frequency estimation by allele-specific real-time PCR of pooled genomic DNA samples // Breast (Edinburgh, Scotland). 2013. V. 22. № 4. P. 532–536. https://doi.org/10.1016/j.breast.2012.12.007
Sokolenko A.P., Mitiushkina N.V., Buslov K.G. et al. High frequency of BRCA1 5382insC mutation in Russian breast cancer patients // Eur. J. Cancer. 2006. V. 42. № 10. P. 1380–1384. https://doi.org/10.1016/j.ejca.2006.01.050
Митрофанов Д.В., Часовникова О.В., Коваленко С.П., Ляхович В.В. Метод выявления мутации 5382insc в гене BRCA1 человека с помощью флуоресцентномеченых олигонуклеотидов // Мол. биология. 2009. Т. 43. № 6. С. 999–1005.
Чердынцева Н.В., Писарева Л.Ф., Панферова Е.В. и др. Этнические аспекты наследственного рака молочной железы в регионе Сибири // Вестник Рос. академии мед. наук. 2014. Т. 69. № 11–12. С. 72–79.
Виллерт А.Б., Коломиец Л.А., Иванова А.А. и др. Рак яичников у больных при наличии мутации BRCA1 5382insC // Междунар. научно-практ. конф. “РЕПРОДУКТИВНЫЕ ТЕХНОЛОГИИ В ОНКОЛОГИИ”. 2015. С. 53.
Brovkina O.B., Shigapova L., Chudakova D.A. et al. The ethnic-specific spectrum of germline nucleotide variants in DNA damage response and repair genes in hereditary breast and ovarian cancer patients of tatar descent // Frontiers in Oncology. 2018. V. 8. P. 421. https://doi.org/10.3389/fonc.2018.00421
Krylova N.Y., Lobeiko O.S., Sokolenko A.P. et al. BRCA1 4153delA founder mutation in Russian ovarian cancer patients // Ereditary Cancer in Clin. Practice. 2006. V. 4. № 4. P. 193–196. https://doi.org/10.1186/1897-4287-4-4-193
Клинические рекомендации Минздрава России “Рак молочной железы” КР379. Ассоциация онкологов России, Российское общество клинической онкологии. 2018. 50 с.
Клинические рекомендации по диагностике и лечению больных раком молочной железы. Общероссийский союз общественных объединений, Ассоциация онкологов России. 2014. 43 с.
Стенина М.Б., Жукова Л.Г., Королева И.А. и др. Практические рекомендации по лекарственному лечению инвазивного рака молочной железы // Злокачественные опухоли: Практические рекомендации RUSSCO (электронное издание). 2018. Т. 3s2. № 8. С. 113–144. https://doi.org/10.18027/2224-5057-2019-9-3s2-128-163
Клинические рекомендации Минздрава России “Рак яичников/рак маточной трубы/первичный рак брюшины” КР547. Ассоциация онкологов России, Российское общество клинической онкологии. 2018. 44 с.
Тюляндин С.А., Коломиец Л.А., Морхов К.Ю. и др. Практические рекомендации по лекарственному лечению рака яичников, первичного рака брюшины и рака маточных труб // Злокачественные опухоли: Практические рекомендации RUSSCO (электронное издание). 2018. Т. 3s2. № 8. С. 145–155.
Khatcheressian J.L., Hurley P., Bantug E. et al. Breast cancer follow-up and management after primary treatment: American society of clinical oncology clinical practice guideline update // J. Clin. Oncol. 2013. V. 31. № 7. P. 961–965. https://doi.org/10.1200/JCO.2012.45.9859
NICE. Familial breast cancer: classification, care and managing breast cancer and related risks in people with a family history of breast cancer (CG164). 2017. 48 p.
Paluch-Shimon S., Pagani O., Partridge A.H. et al. 3rd ESO-ESMO international consensus guidelines for breast cancer in young women (BCY3) // Breast. 2017. V. 35. P. 203–217. https://doi.org/10.1016/j.breast.2017.07.017
Marth C., Hubalek M., Petru E. et al. AGO Austria recommendations for genetic testing of patients with ovarian cancer // Wien Klin Wochenschr. 2015. V. 127. P. 652–654. https://doi.org/10.1007/s00508-015-0814-7
BC Guidelines. Breast Cancer: Management and Follow-Up. 2013. 8 p.
London Cancer Alliance. LCA Breast Cancer Clinical Guidelines. 2013. 47 p.
Cancer Australia. Recommendations for the management of early breast cancer in women with an identified BRCA1 or BRCA2 gene mutation or at high risk of a gene mutation. 2014. 45 p.
Llort G., Chirivella I., Morales R. et al. SEOM clinical guidelines in hereditary breast and ovarian cancer // Clin. Transl. Oncol. 2015. V. 17. № 12. P. 956–961. https://doi.org/10.1007/s12094-015-1435-3
BC Guidelines. Breast Disease and Cancer: Diagnosis. 2013. 12 p.
The Royal Marsden NHS Foundation/The Institute of Cancer Research. Protocol 2 BRCA1 and BRCA2 mutation testing. 2017. 1 p.
Gradishar W.J., Anderson B.O., Abraham J. et al. Breast cancer // NCCN Clin. Pract. Guidel. Oncol. 2019. Version 1. 226 p.
Armstrong D.K., Alvarez R.D., Bakkum-Gamez J.N. et al. Ovarian cancer including fallopian tube cancer and primary peritoneal cancer // NCCN Clin. Pract. Guidel. Oncol. 2019. Version 1. 127 p.
Scottish Intercollegiate Guidelines Network (SIGN). Management of epithelial ovarian cancer A national clinical guideline. 2018. № 135. 67 p.
NICE. Early and locally advanced breast cancer: diagnosis and management (NG101). 2018. 61 p.
Cardoso F., Senkus E., Costa A. et al. 4th ESO – ESMO International Consensus Guidelines for Advanced Breast Cancer (ABC 4) // Ann. Oncol. 2018. V. 29. № 8. P. 1634–1657. https://doi.org/10.1093/annonc/mdy192
Guidelines of the AGO Breast Committee. Diagnosis and Treatment of Patients with Primary and Metastatic Breast Cancer. 2018. 569 p.
Senkus E., Kyriakides S., Ohno S. et al. Primary breast cancer: ESMO Clinical Practice Guidelines for diagnosis, treatment and follow-up // Ann. Oncol. 2015. V. 26. Suppl. 5. P. 8–30. https://doi.org/10.1093/annonc/mdv298
Kreienberg R., Albert U.S., Follmann M. et al. Interdisciplinary GoR level III Guidelines for the diagnosis, therapy and follow-up care of breast cancer short version – AWMF Registry No.: 032-045OL // Geburtsh Frauenheilk. 2013. V. 73. № 6. P. 556–583. https://doi.org/10.1055/s-0032-1328689
NICE. Technology appraisal guidance. Olaparib for maintenance treatment of relapsed, platinum-sensitive, BRCA mutation-positive ovarian, fallopian tube and peritoneal cancer after response to second-line or subsequent platinum-based chemotherapy. 2016. 45 p.
Heather J.M., Chain B. The sequence of sequencers: The history of sequencing DNA // Genomics. Elsevier, 2016. V. 107. № 1. P. 1–8. https://doi.org/10.1016/j.ygeno.2015.11.003
Kchouk M., Gibrat J.-F., Elloumi M. Generations of sequencing technologies: From first to next generation // Biol. Med. HATASO Enterprises LLC. 2017. V. 9. № 3. https://doi.org/10.4172/0974-8369.1000395
Бархатов И.М., Предеус А.В., Чухловин А.Б. Секвенирование нового поколения и области его применения в онкогематологии // Онкогематология. ООО “Издательский дом “АБВ-пресс”, 2016. № 4.
Anaya J.M., Shoenfeld Y., Rojas-Villarraga A. et al. Autoimmunity: from bench to bedside. El Rosario Univ. Press, 2013.
Janitz M. Next-generation genome sequencing: towards personalized medicine. John Wiley & Sons, 2011.
Ari Ş., Arikan M. Next-generation sequencing: advantages, disadvantages, and future // Plant Omics: Trends and Applications. Springer, 2016. P. 109–135. https://doi.org/10.1007/978-3-319-31703-8_5
Stranneheim H., Lundeberg J. Stepping stones in DNA sequencing // Biotechnol. J. Wiley Online Library, 2012. V. 7. № 9. P. 1063–1073. https://doi.org/10.1002/biot.201200153
Костюк С.А. Секвенирование-методы расшифровки нуклеотидной последовательности ДНК. Сообщение 1 // Медицинские новости. 2017. № 6.
Wright D.C. Next steps in the sequence: the implications of whole genome sequencing for health in the UK. PHG Foundation, 2011.
D’Argenio V., Esposito M.V., Telese A. et al. The molecular analysis of BRCA1 and BRCA2: Next-generation sequencing supersedes conventional approaches // Clin. Chim. Acta. Elsevier. 2015. V. 446. P. 221–225. https://doi.org/10.1016/j.cca.2015.03.045
Dougherty B.A., Lai Z., Hodgson D.R. et al. Biological and clinical evidence for somatic mutations in BRCA1 and BRCA2 as predictive markers for olaparib response in high-grade serous ovarian cancers in the maintenance setting // Oncotarget. Impact J. LLC. 2017. V. 8. № 27. P. 43653. https://doi.org/10.18632/oncotarget.17613
Chan M., Ji S.M., Yeo Z.X. et al. Development of a next-generation sequencing method for BRCA mutation screening: a comparison between a high-throughput and a benchtop platform // J. Mol. Diagnostics. Elsevier. 2012. V. 14. № 6. P. 602–612. https://doi.org/10.1016/j.jmoldx.2012.06.003
Strom C.M., Rivera S., Elzinga C. et al. Development and validation of a next-generation sequencing assay for BRCA1 and BRCA2 variants for the clinical laboratory // PLoS One. Publ. Library Sci. 2015. V. 10. № 8. P. e0136419.
Shin S., Kim Y., Chul Oh S. et al. Validation and optimization of the Ion Torrent S5 XL sequencer and Oncomine workflow for BRCA1 and BRCA2 genetic testing // Oncotarget. Impact J. LLC. 2017. V. 8. № 21. P. 34858. https://doi.org/10.18632/oncotarget.16799https://doi.org/10.1371/journal.pone.0136419
Badoer C., Garrec C., Goossens D. et al. Performance of multiplicom’s BRCA MASTR Dx kit on the detection of BRCA1 and BRCA2 mutations in fresh frozen ovarian and breast tumor samples // Oncotarget. Impact J. LLC. 2016. V. 7. № 49. P. 81357. https://doi.org/10.18632/oncotarget.12877
Weren R.D.A., Mensenkamp A.R., Simons M. et al. Novel BRCA1 and BRCA2 tumor test as basis for treatment decisions and referral for genetic counselling of patients with ovarian carcinomas // Hum. Mutat. Wiley Online Library. 2017. V. 38. № 2. P. 226–235.
Park H.S., Park S.J., Kim J.Y. et al. Next-generation sequencing of BRCA1/2 in breast cancer patients: potential effects on clinical decision-making using rapid, high-accuracy genetic results // Ann. Surg. Treat. Res. 2017. V. 92. № 5. P. 331–339. https://doi.org/10.4174/astr.2017.92.5.331
Feliubadaló L., Lopez-Doriga A., Castellsagué E. et al. Next-generation sequencing meets genetic diagnostics: development of a comprehensive workflow for the analysis of BRCA1 and BRCA2 genes // Eur. J. Hum. Genet. Nature Publ. Group, 2013. V. 21. № 8. P. 864. https://doi.org/10.1038/ejhg.2012.270
Kechin A., Kechin A., Khrapov E. et al. BRCA-analyzer: Automatic workflow for processing NGS reads of BRCA1 and BRCA2 genes // Comput. Biol. Chem. Elsevier. 2018. V. 77. P. 297–306. https://doi.org/10.1016/j.compbiolchem.2018.10.012
Mafficini A., Simbolo M., Parisi A. et al. BRCA somatic and germline mutation detection in paraffin embedded ovarian cancers by next-generation sequencing // Oncotarget. Impact J., LLC. 2016. V. 7. № 2. P. 1076. https://doi.org/10.18632/oncotarget.6834
Neveling K., Mensenkamp A.R., Derks R. et al. BRCA testing by single-molecule molecular inversion probes // Clin. Chem. 2017. V. 63. № 2. P. 503–512. https://doi.org/10.1373/clinchem.2016.263897
Ermolenko N.A., Boyarskikh U.A., Kechin A.A. et al. Massive parallel sequencing for diagnostic genetic testing of BRCA genes – a single center experience // Asian Pac J. Cancer Prev. 2015. V. 16. № 17. P. 7935–7941. https://doi.org/10.7314/apjcp.2015.16.17.7935
Michils G., Hollants S., Dehaspe L. et al. Molecular analysis of the breast cancer genes BRCA1 and BRCA2 using amplicon-based massive parallel pyrosequencing // J. Mol. Diagnostics. Elsevier. 2012. V. 14. № 6. P. 623–630. https://doi.org/10.1016/j.jmoldx.2012.05.006
Ruiz A., Llort G., Yagüe C. et al. Genetic testing in hereditary breast and ovarian cancer using massive parallel sequencing // Biomed Res. Int. Hindawi. 2014. V. 2014. https://doi.org/10.1155/2014/542541
Morgan J.E., Carr I.M., Sheridan E. et al. Genetic diagnosis of familial breast cancer using clonal sequencing // Hum. Mutat. Wiley Online Library. 2010. V. 31. № 4. P. 484–491. https://doi.org/10.1002/humu.21216
Pilato B., De Summa S., Danza K. et al. DHPLC/SURVEYOR nuclease: a sensitive, rapid and affordable method to analyze BRCA1 and BRCA2 mutations in breast cancer families // Mol. Biotechnol. Springer. 2012. V. 52. № 1. P. 8–15. https://doi.org/10.1007/s12033-011-9468-5
van der Stoep N., van Paridon C.D., Janssens T. et al. Diagnostic guidelines for high resolution melting curve (HRM) analysis: An interlaboratory validation of BRCA1 mutation scanning using the 96 well Light-ScannerTM // Hum. Mutat. Wiley Online Library. 2009. V. 30. № 6. P. 899–909. https://doi.org/10.1002/humu.21004
De Bonis M., Minucci A., Scaglione G.L. et al. Capillary electrophoresis as alternative method to detect tumor genetic mutations: the model built on the founder BRCA1 c. 4964_4982del19 variant // Fam. Cancer. Springer. 2019. V. 18. № 1. P. 29–35. https://doi.org/10.1007/s10689-018-0094-2
Obul J., Itoga S., Abliz M. et al. High-resolution melting analyses for gene scanning of APC, MLH1, MSH2, and MSH6 associated with hereditary colorectal cancer // Genet. Test. Mol. Biomarkers. 2012. V. 16. № 5. P. 406–411. https://doi.org/10.1089/gtmb.2011.0166
Fowler A., Mahamdallie S., Ruark E. et al. Accurate clinical detection of exon copy number variants in a targeted NGS panel using DECoN // Wellcome Open Res. 2016. V. 1. https://doi.org/10.12688/wellcomeopenres.10069.1
Germani A., Libi F., Maggi S. et al. Rapid detection of copy number variations and point mutations in BRCA1/2 genes using a single workflow by ion semiconductor sequencing pipeline // Oncotarget. Impact J. LLC. 2018. V. 9. № 72. P. 33648. https://doi.org/10.18632/oncotarget.26000
Minucci A., De Paolis E., Concolino P. et al. Competitive PCR-High Resolution Melting Analysis (C-PCR-HRMA) for large genomic rearrangements (LGRs) detection: A new approach to assess quantitative status of BRCA1 gene in a reference laboratory // Clin. Chim. Acta. Elsevier. 2017. V. 470. P. 83–92. https://doi.org/10.1016/j.cca.2017.04.026
Дополнительные материалы отсутствуют.