Генетика, 2020, T. 56, № 4, стр. 401-407
Профиль экспрессии генов FAS, YABBY2 и YABBY5 коррелирует с различным числом семенных камер у томата
М. А. Слугина 1, М. А. Филюшин 1, *, А. В. Щенникова 1, Е. З. Кочиева 1, 2, К. Г. Скрябин 1, 2
1 Федеральный исследовательский центр “Фундаментальные основы биотехнологии”
Российской академии наук
119071 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
* E-mail: michel7753@mail.ru
Поступила в редакцию 28.03.2019
После доработки 20.05.2019
Принята к публикации 31.05.2019
Аннотация
Анализ 30 сортов томата с различным числом камер в плоде выявил четыре многокамерных сорта с нуль-мутацией в гене SlFAS. Показано, что сорта с числом камер больше двух содержат в локусе locule number два SNPs, сцепленных с признаком многокамерности. У трех сортов томата, контрастных по числу камер в плоде, определен профиль экспрессии генов SlFAS, SlYABBY2 и SlYABBY5 в бутоне и на трех стадиях развития плода. У сорта Бычье сердце (≥10 камер) показано отсутствие транскрипции гена SlFAS во всех анализируемых тканях. При этом уровень транскрипции SlYABBY2 и SlYABBY5 в бутоне значительно снижен, а в плодах, напротив, выше, чем у сортов Ред Черри (2 камеры) и Викинг (5–6 камер).
Доместикация плодовых и овощных растений сопровождалась ростом их урожайности, в том числе за счет значительного увеличения размера плода. Пример тому – томат овощной Solanum lycopersicum, сочные плоды которого у некоторых сортов достигают иногда в тысячу раз большего веса по сравнению с плодами дикорастущих видов томата [1]. Благодаря существующему многообразию современных сортов томата и его дикорастущих родственников (секция Lycopersicon рода Solanum), различающихся морфофизиологией плода [2], S. lycopersicum стал основным модельным объектом для молекулярно-генетических исследований механизмов формирования и развития сочного многосемянного плода. Размер плода S. lycopersicum на 50% зависит от числа семенных камер в нем [3]. Для многих сортов томата овощного характерны многокамерные (>4) крупные плоды, в то время как у всех дикорастущих видов томата плод состоит из двух семенных камер и имеет небольшой размер [2, 3].
Количество семенных камер плода определяется числом плодолистиков в пестике цветка томата. Приобретение признака многокамерности плода S. lycopersicum в процессе доместикации связывают, главным образом, с двумя локусами количественных признаков (QTLs) locule number (lc; хромосома 2) и fasciated (fas; хромосома 11), влияющих на число плодолистиков пестика, при этом эффект от fas больше, чем от lc [1, 4]. Локус lc представляет собой некодирующую и нетранскрибируемую область (1608 пн) и считается обязательным для проявления многокамерности [5]. С одной стороны локуса lc расположен ген WUSCHEL (SlWUS), центральный регулятор развития апикальной меристемы, поддерживающий популяцию стволовых клеток [6, 7]. С другой стороны – ген WD40-белка, гомолог субъединицы хроматин-ремодулирующего фактора 1, положительно регулирующего экспрессию WUS в апикальной меристеме [5, 8]. В процессе доместикации томата в локусе lc S. lycopersicum var. cerasiforme возникли два полиморфизма (SNPs), T686C и A692G, сцепленных с числом камер, в результате чего двухкамерный плод стал четырех- и даже шестикамерным, и позже у этих растений появилась мутация fas, которая на фоне lc привела к еще большему числу камер плода [5]. Считается, что локусы lc и fas совместно регулируют сигнальный путь признака многокамерности [5].
В связи с этим возникает вопрос о взаимодействии lc и fas, поскольку в отличие от lc в локусе fas расположен ген FASCIATED (FAS), кодирующий фактор транскрипции семейства YABBY [4]. Показано, что многокамерность (>6) плодов томата связана с функционально неполноценным SlFAS из-за присутствия преждевременного стоп-кодона вследствие однонуклеотидной вставки в экзоне V [4] или же с нуль-мутацией fas, возникшей в результате инверсии участка хромосомы 11 размером 294 тыс. пн с одной из точек разрыва в интроне I SlFAS [9].
Ген FAS относится к кладе YABBY2 “вегетативных” генов YABBY, включающих также клады YABBY1/YABBY3 и YABBY5 [10]. У томата овощного S. lycopersicum ген SlFAS является ближайшим гомологом гена SlYABBY2, а также эволюционно близок к генам SlYABBY5 [11–14]. Данные гены участвуют в корректном развитии и дифференцировке апикальной меристемы побега посредством регуляции асимметричного развития всех формируемых меристемой латеральных органов, включая цветковые [10, 15, 16], и могут, следовательно, прямо или опосредованно взаимодействовать с фактором транскрипции WUS. Кроме того, гены YABBY, связанные с эволюцией плоского листа, считаются причастными также к разнообразию форм гинецея [17, 18]. В отличие от YABBY1/YABBY3, влияющих прежде всего на абаксиально-адаксиальную асимметрию латеральных органов, функция YABBY2 и YABBY5, работающих в составе гетеродимера, больше связана с регуляцией разрастания органов [19, 20].
Целью данного исследования стало определение экспрессии генов SlFAS, SlYABBY2 и SlYABBY5 на различных стадиях формирования плода – от инициации плодолистиков в меристеме цветка (стадия ранней бутонизации) до зрелого плода (созревший красный плод) – у сортов томата с различным числом плодовых камер.
МАТЕРИАЛЫ И МЕТОДЫ
Для работы были отобраны 30 сортов томата с различным числом семенных камер в плоде (табл. 1) из коллекции Федерального научного центра овощеводства (ФНЦО, Московская обл., пос. ВНИИССОК), геномная ДНК которых была использована для анализа возможных мутаций гена SlFAS. На основе последовательности FAS S. lycopersicum (NM_001247461) были разработаны специфичные праймеры для ПЦР-амплификации: 1) полногеномной последовательности SlFAS (6200 пн), 2) фрагмента “экзон I–экзон II” гена SlFAS (для определения наличия протяженной вставки в интроне I; 2200 пн без вставки) и 3) фрагмента “экзон III–экзон V” (500 пн; для поиска возможной однонуклеотидной инсерции в экзоне IV) (табл. 2). Секвенирование последовательностей ДНК проводили на платформе Applied Biosystems 3730 DNA Analyzer (ЦКП Биоинженерия, ФИЦ Биотехнологии РАН). Суммарную РНК и синтезированную на ее основе кДНК (GoScript, Promega, США) использовали для определения экспрессии генов SlFAS, SlYABBY2 и SlYABBY5a методом количественной ПЦР в реальном времени (РВ-ПЦР) (табл. 2). РВ-ПЦР проводили с использованием набора “Реакционная смесь для проведения РВ-ПЦР в присутствии SYBR GreenI и ROX” (ЗАО “Синтол”, Москва, Россия) в следующих условиях: 95°C – 5 мин; 40 циклов (95°C – 15 с, 60°C – 50 с). Для оценки уровня относительной экспрессии в качестве референсных использовали гены Expressed (SGN-U346908) и Actin 2/7 (NM_001330119.1) [21, 22] (табл. 2). Диапазон Ct для пар праймеров, использованных для РВ-ПЦР, приведен в табл. 3. Статистическую обработку полученных данных проводили с помощью программного обеспечения GraphPad Prism v. 7.02 (https://www.graphpad.com). Филогенетический анализ известных белковых последовательностей вегетативной группы семейства YABBY (YABBY1/YABBY3, YABBY2, YABBY5) S. lycopersicum и Arabidopsis thaliana был проведен с использованием программы MEGA7 (https:// www.megasoftware.net/).
Таблица 1.
Результаты скрининга 30 сортов томата с разработанными парами праймеров FASF/FASR, FASex1F/ FASex2R и FASex3F/FASex5R (“+” – фрагмент амплифицируется, “–” – фрагмент не амплифицируется)
| Сорт | Число семенных камер | FASF/FASR | FASex1F/FASex2R | FASex3F/FASex5R |
|---|---|---|---|---|
| Августин | 3 | + | + | + |
| Северянка | 4 | + | + | + |
| Отрадный | 2–3 | + | + | + |
| Ред Черри | 2 | + | + | + |
| Чародей | 4–6 | + | + | + |
| Дубок | ≥10 | – | – | + |
| Викинг | 5–6 | + | + | + |
| Оттава | 2 | + | + | + |
| Грунтовый Грибовский | 4–5 | + | + | + |
| Чаровница | 2–3 | + | + | + |
| Гном | 2–3 | + | + | + |
| Штамбовый Алпатьева | 4–5 | + | + | + |
| Грот | 3 | + | + | + |
| Лотос | 4 | + | + | + |
| Челнок | 2 | + | + | + |
| Росинка | 3 | + | + | + |
| Евгения | 4–5 | + | + | + |
| Алтайский оранжевый | ≥10 | – | – | + |
| Финиш | 4 | + | + | + |
| Фитоус | 4 | + | + | + |
| Титан | 3–4 | + | + | + |
| Яблонька России | 4 | + | + | + |
| Москвич | 4 | + | + | + |
| Черный принц | ≥10 | – | – | + |
| Талалихин | 3–4 | + | + | + |
| Динар | 5–6 | + | + | + |
| Глория | 4–5 | + | + | + |
| Бычье сердце | ≥10 | – | – | + |
| Гурман | 5–6 | + | + | + |
| Камея | 5–6 | + | + | + |
Таблица 2.
Последовательности праймеров, использованные в работе
| Ген | Последовательности праймеров |
|---|---|
| Праймеры для РВ-ПЦР | |
| SlFAS | FAS-rtF 5'-CTCAGAACGCGTTTGTTACTTGC-3' FAS-rtR 5'-TGAACATCTTGAAGGGGTAGAGC-3' |
| SlYABBY2 | YB2-rtF 5'-CCTCGGAACGTGTTTGCTATG-3' YB2-rtR 5'-AATGGGGAGAGACTGAAGGGA-3' |
| SlYABBY5a | YB5-rtF 5'-GTGACTGTTCGTTGTGGACAT-3' YB5-rtR 5'-GACAATCCTCTCCTCGTGAACA-3' |
| Expressed | Expressed-F 5'-GCTAAGAACGCTGGACCTAATG-3' Expressed-R 5'-TGGGTGTGCCTTTCTGAATG-3' |
| Actin 2/7 | Actin 2/7-F 5'-CATTGTGCTCAGTGGTGGTTC-3' Actin 2/7-R 5'-TCTGCTGGAAGGTGCTAAGTG3' |
| Праймеры для амплификации гена SlFAS | |
| SlFAS (на весь ген) | FASF 5'-CTTTGAATAGGCTTTTTCTTAATTC-3' FASR 5'-GAAAACAACATAAAAGACATGCA-3' |
| SlFAS (на фрагмент экзон I–экзон II) | FASex1F 5'-CTTCACCTTCCTCAGAACGC-3' FASex2R 5'-GAAARCAWATTTGCACAATGC-3' |
| SlFAS (на фрагмент экзон III–экзон V) | FASex3F 5'-CTARAATCCCTYCGATTCGT-3' FASex5R 5'-GCTTCACGATGGSTAATATCA-3' |
Таблица 3.
Диапазон Ct для пар праймеров, использованных для РВ-ПЦР
| Сорт | Орган | YABBY2 | YABBY5 | FAS |
|---|---|---|---|---|
| Ред Черри | Бутон | 22.02–22.17 | 23.11–23.24 | 22.89–23.20 |
| Плод 1 | 24.20–24.24 | 27.83–28.27 | 29.23–29.43 | |
| Плод 2 | 21.65–21.73 | 24.8–24.97 | 25.64–25.91 | |
| Плод 3 | 26.37–26.46 | 28.2–28.60 | 32.09–32.42 | |
| Викинг | Бутон | 21.02–21.11 | 22.26–22.75 | 22.52–22.7 |
| Плод 1 | 28.24–28.58 | 27.69–28.19 | 29.59–31.55 | |
| Плод 2 | 27.35–27.37 | 28.21–28.36 | 28.79–31.39 | |
| Плод 3 | 26.00–26.11 | 29.48–29.86 | 30.33–31.23 | |
| Бычье сердце | Бутон | 26.19–26.68 | 28.14–29.66 | Нет экспрессии |
| Плод 1 | 22.53–22.59 | 23.97–24.16 | То же | |
| Плод 2 | 22.73–23.21 | 25.18–25.38 | » | |
| Плод 3 | 24.72–24.99 | 25.24–25.42 | » |
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
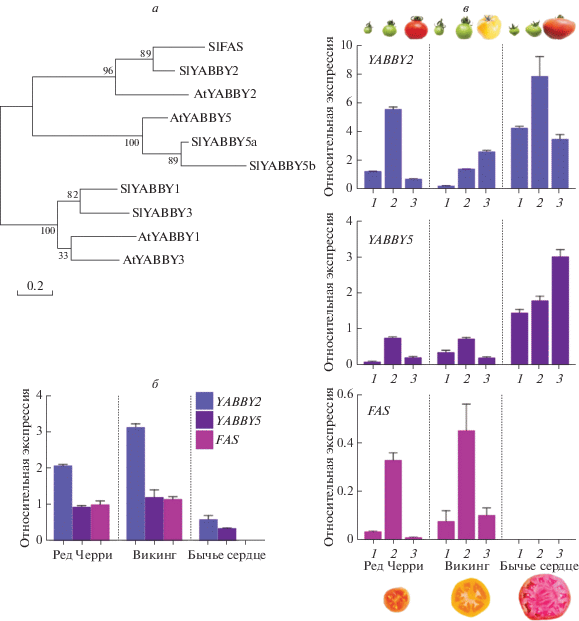
На дендрограмме SlFAS формирует единый субкластер с SlYABBY2, сестринский по отношению к SlYABBY5 (рис. 1,а). Можно предположить, что ген SlFAS возник в результате дупликации SlYABBY2 и, по всей вероятности, SlFAS сохранил функции, свойственные SlYABBY2, а именно участие в регуляции асимметрии и разрастании латеральных органов [12], но при этом, возможно, приобрел новые функции, что следует из эффекта, производимого нуль-мутацией fas [9]. Проведенный структурный анализ YABBY-последовательностей выявил вариабельность междоменных областей белков FAS и YABBY2 (идентичность 41%), в то время как консервативные домены – цинкового пальца Cys2Cys2-типа и YABBY, характерные для всех YABBY-белков [17, 23], отличались всего шестью и одним, соответственно, нейтральными замещениями аминокислотных остатков. Возможно, что именно изменение междоменных областей, отвечающих за фолдинг белка, могло привести к неофункционализации SlFAS.
Рис. 1.
Структурно-экспрессионный анализ генов SlFAS, SlYABBY2 и SlYABBY5. а – дендрограмма, основанная на аминокислотных последовательностях белков YABBY S. lycopersicum (SlFAS (мРНК NM_001247461), SlYABBY1 (XM_004229745), SlYABBY2 (XM_010324148), SlYABBY3 (XM_004245689), SlYABBY5а (XM_004242730), SlYABBY5b (XM_004251674)) и A. thaliana (AtYABBY1 (NM_130082), AtYABBY2 (NM_001084021), AtYABBY3 (NM_116235), AtYABBY5 (NM_179749)). Существенные значения бутстрэп указаны в основании ветвей; б – анализ экспрессии генов SlFAS, SlYABBY2 и SlYABBY5 в молодых бутонах сортов Ред Черри, Викинг и Бычье сердце; в – анализ экспрессии генов SlFAS, SlYABBY2 и SlYABBY5 на трех стадиях развития плода (завязавшийся плод (1), растущий зеленый плод (2) и созревший плод (3)) у сортов Ред Черри, Викинг и Бычье сердце. Над диаграммой приведено схематичное изображение плодов, соответствующих анализируемым стадиям, под диаграммой приведены фотографии зрелого плода каждого из трех анализируемых сортов в разрезе.
Скрининг ДНК 30 сортов томата с помощью разработанных праймеров выявил отсутствие у четырех сортов (Дубок, Алтайский оранжевый, Черный принц, Бычье сердце) продуктов амплификации полноразмерного гена и фрагмента “экзон I–экзон II”, что предположительно связано с нуль-мутацией fas [9]. Выявленные сорта имели многокамерные (≥10) плоды (табл. 1). У сортов томата с числом камер в плодах 2–6 амплифицировались все три фрагмента гена SlFAS, свидетельствуя об отсутствии нуль-мутации fas. У сортов с 5–6-камерными плодами число камер может быть связано с наличием в локусе lc двух SNPs, сцепленных с многокамерностью [5]. Фрагмент, содержащий экзон IV гена SlFAS, был секвенирован у всех анализируемых сортов, однако однонуклеотидной инсерции, описанной ранее Cong с соавт. [4], выявлено не было.
Для дальнейшей работы среди анализируемых образцов томата были отобраны три сорта, формирующие плоды разной камерности – Ред Черри (двухкамерный плод), Викинг (шестикамерный плод) и Бычье сердце (плод состоит из ≥10 камер) (табл. 1). Был проведен скрининг последовательности локуса lc выбранных сортов томата на предмет наличия SNPs T686C и A692G, сцепленных с повышенным числом камер. Конкретные участки локуса были амплифицированы с помощью праймеров, предложенных [5]. Секвенирование фрагментов выявило, что сорта Викинг и Бычье сердце содержат искомые SNPs, а Ред Черри – нет. Корреляция между присутствием SNPs в lc и увеличенным числом плодолистиков/семенных камер подтверждается ранее показанным фактом, что данные SNPs изменяют последовательность CArG-бокса cis-регуляторного элемента 3'‑нетранскрибируемой области гена SlWUS [24]. Таким образом, данные SNPs могут негативно влиять на связывание фактора транскрипции AGAMOUS (AG) с CArG-боксом и, как следствие, на его способность подавлять экспрессию SlWUS [24, 25]. В этом случае повышение экспрессии SlWUS может приводить к увеличению числа плодолистиков вследствие разрастания меристемы цветка и дополнительной индукции гена AG, который определяет идентичность гинецея [6, 7, 26].
Выключение SlFAS способно дополнительно к lc повысить экспрессию SlWUS, что приводит к еще большему разрастанию меристемы цветка и увеличению числа цветковых органов, включая плодолистики (камеры плода), что и было показано для томата с нуль-мутацией fas [1, 27].
Известно, что у томата с двухкамерными плодами гены SlFAS, SlYABBY2 и SlYABBY5 экспрессируются во всех надземных латеральных органах [11, 12], при этом у томата с 5–6-камерным плодом была изучена только экспрессия SlFAS [25]. Однако экспрессия данных трех генов у сортов томата с многокамерными плодами (включая ≥10-камерные) и возможная корреляция с числом камер в плоде ранее не изучались.
Нами была определена экспрессия генов SlFAS, SlYABBY2 и SlYABBY5 у сортов Ред Черри (2 камеры), Викинг (5–6) и Бычье сердце (≥10) в молодом бутоне (дифференцированная меристема цветка с примордиями всех органов, включая плодолистики) и плоде (на трех стадиях развития – завязавшийся плод (1), растущий зеленый плод (2) и созревший плод (3)) в двух биологических и трех технических повторах (рис. 1,б, в).
Проведенный экспрессионный анализ показал отсутствие транскриптов гена SlFAS в бутоне и плодах сорта Бычье сердце, что подтвердило наличие нуль-мутации fas (рис. 1,б, в).
При этом интересно, что анализ бутон-специфичной ко-экспрессии SlYABBY2 и SlYABBY5 выявил, что уровень экспрессии этих генов у сорта Бычье сердце оказался существенно ниже, чем у сортов Ред Черри и Викинг (рис. 1,б). Полученные данные можно объяснить тем, что наблюдаемая инактивация SlFAS у многокамерного сорта Бычье сердце предположительно приводит к росту уровня транскрипции SlWUS, продукт которого может участвовать в репрессии генов SlYABBY2 и SlYABBY5 в молодых бутонах после закладки меристемы цветка.
У сортов Ред Черри и Викинг транскрипты генов SlFAS, SlYABBY2 и SlYABBY5 были выявлены на всех анализируемых стадиях развития плода (рис. 1,в). Сохранение экспрессии YABBY-генов по мере развития плода подтверждает их участие в регуляции асимметричного развития и разрастания плода. Интересно, что в плодах сорта Бычье сердце, при отсутствии экспрессии SlFAS, уровень экспрессии генов SlYABBY2 (максимум на стадии 2) и SlYABBY5 (максимум на стадии 3) был высок, значимо превышая таковой в плодах сортов Ред Черри и Викинг (рис. 1,в). Это может свидетельствовать о возможной компенсации активности отсутствующего гена SlFAS в процессе развития многокамерных плодов томата.
Таким образом, в данной работе были проанализированы 30 сортов томата, формирующих плоды с различным числом семенных камер. У четырех многокамерных сортов выявлена нуль-мутация гена SlFAS. Показано, что сорта с числом камер больше двух содержат в локусе lc два SNPs, сцепленных с признаком многокамерности. Для трех сортов томата, контрастных по числу камер в плоде, определен профиль экспрессии генов SlFAS, SlYABBY2 и SlYABBY5 в бутонах и на трех стадиях развития плода. У сорта Бычье сердце (≥10 камер) показано отсутствие транскрипции гена SlFAS во всех анализируемых тканях. При этом в бутонах сорта Бычье сердце уровень транскрипции SlYABBY2 и SlYABBY5 значительно снижен, а в плодах, напротив, выше, чем у сортов Ред Черри (2 камеры) и Викинг (5–6 камер). Последнее может свидетельствовать о компенсации активности отсутствующего гена SlFAS в процессе развития плода сорта Бычье сердце.
Работа выполнена с использованием экспериментальной установки искусственного климата ЭУИК (Институт биоинженерии ФИЦ Биотехнологии РАН) при поддержке гранта РФФИ (№ 18-29-07007) и Министерства науки и высшего образования Российской Федерации.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Barrero L.S., Tanksley S.D. Evaluating the genetic basis of multiple-locule fruit in a broad cross section of tomato cultivars // Theor. Appl. Genet. 2004. V. 109. P. 669–679.
Peralta I.E., Spooner D.M., Knapp S. Taxonomy of wild tomatoes and their relatives (Solanum sect. Lycopersicoides, sect. Juglandifolia, sect. Lycopersicon; Solanaceae) // Systematic Botany Monographs American Society of Plant Taxonomists. USA, 2008. 186 p.
Tanksley S.D. The genetic, developmental, and molecular bases of fruit size and shape variation in tomato // Plant Cell. 2004. V. 16. № Sl. P. S181–S189.
Cong B., Barrero L.S., Tanksley S.D. Regulatory change in YABBY-like transcription factor led to evolution of extreme fruit size during tomato domestication // Nat. Genet. 2008. V. 40. № 6. P. 800–804. https://doi.org/10.1038/ng.144
Muños S., Ranc N., Botton E. et al. Increase in tomato locule number is controlled by two single-nucleotide polymorphisms located near WUSCHEL // Plant Physiol. 2011. V. 156. № 4. P. 2244–2254. https://doi.org/10.1104/pp.111.173997
Clark S.E. Cell signalling at the shoot meristem // Nat. Rev. Mol. Cell Biol. 2011. V. 2. P. 276–284.
Lenhard M., Bohnert A., Jürgens G., Laux T. Termination of stem cell maintenance in Arabidopsis floral meristems by interactions between WUSCHEL and AGAMOUS // Cell. 2001. V. 105. № 6. P. 805–814.
Leyser H.M.O., Furner I.J. Characterization of 3 shoot apical meristem mutants of Arabidopsis thaliana // Development. 1992. V. 116. P. 397–403.
Huang Z., van der Knaap E. Tomato fruit weight 11.3 maps close to fasciated on the bottom of chromosome 11 // Theor. Appl. Genet. 2011. V. 123. № 3. P. 465–474. https://doi.org/10.1007/s00122-011-1599-3
Finet C., Floyd S.K., Conway S.J. et al. Evolution of the YABBY gene family in seed plants // Evol. Dev. 2016. V. 18. № 2. P. 116–126. https://doi.org/10.1111/ede.12173
Huang Z., Van Houten J., Gonzalez G. et al. Genome-wide identification, phylogeny and expression analysis of SUN, OFP and YABBY gene family in tomato // Mol. Genet. Genomics. 2013. V. 288. № 3–4. P. 111–129. https://doi.org/10.1007/s00438-013-0733-0
Han H.Q., Liu Y., Jiang M.M. et al. Identification and expression analysis of YABBY family genes associated with fruit shape in tomato (Solanum lycopersicum L.) // Genet. Mol. Res. 2015. V. 14. № 2. P. 7079–7091. https://doi.org/10.4238/2015.June.29.1
Филюшин М.А., Слугина М.А., Щенникова А.В., Кочиева Е.З. YABBY3-ортологи дикорастущих видов томата: структура, полиморфизм и экспрессия // Acta Naturae. 2017. Т. 9. № 4(35). С. 106–115.
Филюшин М.А., Слугина М.А., Щенникова А.В., Кочиева Е.З. Идентификация и анализ экспрессии гена YABBY1 у дикорастущих видов томатов // Генетика. 2018. Т. 54. № 5. С. 535–546.
Yang C., Ma Y., Li J. The rice YABBY4 gene regulates plant growth and development through modulating the gibberellin pathway // J. Exp. Bot. 2016. V. 67. № 18. P. 5545–5556.
Shchennikova A.V., Slugina M.A., Beletsky A.V. et al. The YABBY genes of leaf and leaf-like organ polarity in leafless plant Monotropa hypopitys // Int. J. Genomics. 2018. V. 15. Article ID 7203469. https://doi.org/10.1155/2018/7203469
Bowman J.L. The YABBY gene family and abaxial cell fate // Curr. Opin. Plant Biol. 2000. V. 3. № 1. P. 17–22.
Sarojam R., Sapp P.J., Goldshmidt A. et al. Differentiating Arabidopsis shoots from leaves by combined YABBY activities // Plant Cell. 2010. V. 22. P. 2113–2130. https://doi.org/10.1105/tpc.110.075853
de Almeida A.M., Yockteng R., Schnable J. et al. Co-option of the polarity gene network shapes filament morphology in angiosperms // Sci. Rep. 2014. V. 4. Article 6194. https://doi.org/10.1038/srep06194
Stahle M., Kuehlich J., Staron L. et al. YABBYs and the transcriptional corepressors LEUNIG and LEUNIG_HOMOLOG maintain leaf polarity and meristem activity in Arabidopsis // Plant Cell. 2009. V. 21. P. 3105–3118.
Expósito-Rodríguez M., Borges A.A., Borges-Pérez A., Pérez J.A. Selection of internal control genes for quantitative real-time RT-PCR studies during tomato development process // BMC Plant Biol. 2008. V. 8. Article 131. https://doi.org/10.1186/1471-2229-8-131
Bemer M., Karlova R., Ballester A.R. et al. The tomato FRUITFULL homologs TDR4/FUL1 and MBP7/FUL2 regulate ethylene-independent aspects of fruit ripening // Plant Cell. 2017. V. 24. № 11. P. 4437–4451. https://doi.org/10.1105/tpc.112.103283
Kanaya E., Nakajima N., Okada K. Non-sequence-specific DNA binding by the FILAMENTOUS FLOWER protein from Arabidopsis thaliana is reduced by EDTA // J. Biol. Chem. 2002. V. 277. № 14. P. 11957–11964.
Li H., Qi M., Sun M. et al. Tomato transcription factor SlWUS plays an important role in tomato flower and locule development // Front. Plant Sci. 2017. V. 8. Article 457. https://doi.org/10.3389/fpls.2017.00457
van der Knaap E., Chakrabarti M., Chu Y.H. et al. What lies beyond the eye: the molecular mechanisms regulating tomato fruit weight and shape // Front. Plant Sci. 2014. V. 5. Article 227.
Yanofsky M.F., Ma H., Bowman J.L. et al. The protein encoded by the Arabidopsis homeotic gene agamous resembles transcription factors // Nature. 1990. V. 346. P. 35–39.
Lippman Z., Tanksley S.D. Dissecting the genetic pathway to extreme fruit size in tomato using a cross between the small-fruited wild species Lycopersicon pimpinellifolium and L. esculentum var. Giant Heirloom // Genetics. 2001 V. 158(1). P. 413–422.
Дополнительные материалы отсутствуют.


