Генетика, 2020, T. 56, № 4, стр. 408-420
Последовательности ITS1-5.8S рДНК-ITS2 и trnL-trnF как маркеры при изучении видового разнообразия ковылей Алтая
П. А. Матейкович 1, Е. О. Пунина 1, *, Ю. О. Копылов-Гуськов 2, Н. Н. Носов 1, П. Д. Гудкова 3, 4, А. А. Гнутиков 1, 5, Э. М. Мачс 1, Ю. В. Михайлова 1, Е. Е. Крапивская 1, А. В. Родионов 1, 6
1 Ботанический институт им. В.Л. Комарова Российской академии наук
197376 Санкт-Петербург, Россия
2 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
3 Алтайский государственный университет
656049 Барнаул, Россия
4 Томский государственный университет
634050 Томск, Россия
5 Федеральный исследовательский центр Всероссийский институт генетических ресурсов растений
им. Н.И. Вавилова
190000 Санкт-Петербург, Россия
6 Санкт-Петербургский государственный университет
199034 Санкт-Петербург, Россия
* E-mail: elizaveta_punina@mail.ru
Поступила в редакцию 07.03.2019
После доработки 09.11.2019
Принята к публикации 05.12.2019
Аннотация
Секвенирована последовательность ITS1-5.8S рДНК-ITS2 генов 35S рРНК 16 видов ковылей и двух видов ковылечков флоры Республики Алтай и Алтайского края (Stipa baicalensis, S. borysthenica, S. capillata, S. consanguinea, S. dasyphylla, S. desertorum, S. glareosa, S. grandis, S. korshinskyi, S. krylovii, S. lessingiana, S. orientalis, S. pennata, S. praecapillata, S. pulcherrima, S. zalesskii, Ptilagrostis junatovii, P. mongholica), четырех видов ковылей из других регионов России (S. pontica, S. rubens, S. tirsa, S. ucrainica), а также хлоропластная последовательность trnL-trnF у S. capillata, S. borysthenica, S. glareosa, S. krylovii, S. lessingiana, S. orientalis, S. pulcherrima и S. zalesskii. Показано, что район trnL-trnF так же как и 5.8S рДНК у ковылей высоко консервативен и не может быть использован для дифференциации секций и видов в пределах рода. Последовательности ITS1 и ITS2 напротив имеют ряд парсимонично информативных замен и инделей, позволяющих выявить группы риботипов, соответствующие секциям, а внутри некоторых секций – и отдельным видам. Хорошо выделяются клады, соответствующие секциям Stipa и Leiostipa, виды секций Barbatae и Subbarbatae попали в одну кладу, а секция Hemibarbatae оказалась полифилетичной. Только в секции Stipa не выявлено видоспецифичных нуклеотидных замен и инделей. Показана обособленность вида S. desertorum, таксономический ранг которого ранее был неясен. Обсуждается вопрос о происхождении полиморфных сайтов в ITS-последовательностях ковылей как возможных свидетельствах отдаленной гибридизации.
Род ковыль (Stipa L.) – один из крупнейших родов семейства злаки (Poaceae). До конца XIX в. род Stipa понимался широко, в частности ряд видов Achnatherum P. Beauv., а в некоторых случаях и виды Ptilagrostis Griseb., рассматривались в составе рода Stipa [1–5]. Всего в род Stipa включали от 400 до 600 видов, по современным представлениям относимых к 28 родам, объединяемых в трибу Stipeae [6–8]. В настоящее время к роду Stipa относят около 150 видов [9]. Одни виды являются доминантами и эдификаторами степных растительных сообществ, занимая обширные площади, другие, наоборот, очень редки и нуждаются в охране. Вопрос о количестве и взаимоотношениях видов внутри рода Stipa остается открытым. Это связано, с одной стороны, с высокой степенью внутривидового полиморфизма, а с другой – с относительно малым количеством таксономически значимых морфологических признаков. Дивергенция широко применяемых в молекулярной систематике растений хлоропластных последовательностей ДНК, а также ядерной последовательности ITS1-5.8S рДНК-ITS2 считается недостаточной для разграничения видов рода Stipa [10–12]. Однако ковыли флоры России в этом отношении до сих пор были изучены очень фрагментарно.
Мы исследовали виды рода Stipa и два вида рода ковылечек (Ptilagrostis), произрастающих на территории Алтайского края и Республики Алтай; для сравнения нами также были исследованы некоторые представители ковылей из Тывы, Хакасии, Европейской части России и Монголии. Алтай является одним из центров разнообразия ковылей (наряду с Кавказом и Крымом), при этом видовое разнообразие ковылей в этом регионе определяется перекрыванием ареалов: для части видов – это восточная граница их ареала, для других – западная. Согласно “Флоре Сибири”, на Алтае произрастает 13 видов и один подвид рода Stipa [13], однако ботанические находки последних лет добавили к агростофлоре этого региона еще два вида ковылей – S. pulcherrima K. Koch и S. grandis P.A. Smirn. [14–17]. По-видимому единственный эндемичный для Русского Алтая вид – это S. consanguinea Trin. & Rupr. [13–15].
Цель предпринятого исследования – секвенировать ITS-последовательности ранее неизученных в этом отношении видов рода Stipa флоры России, изучить возможности применения для идентификации (с точностью до вида или секции) ковылей Алтайской горной страны, обычно используемые при ДНК-штрихкодировании растений последовательности ядерных генов 35S рРНК (район ITS1-5.8S рДНК-ITS2) и район trnL-trnF генома хлоропластов [18–20]. Показано, что районы trnL-trnF и 5.8S рДНК у ковылей высоко консервативны и не могут использоваться для дифференциации секций и видов в пределах рода. Последовательности же ITS1 и ITS2, напротив, имеют ряд парсимонично информативных замен и инделей, позволяющих выявить группы риботипов, соответствующие секциям, а внутри некоторых секций – и отдельным видам.
МАТЕРИАЛЫ И МЕТОДЫ
Гербарные образцы, послужившие материалом для исследования, в основном были собраны авторами во время экспедиций в 2007–2018 гг. Они хранятся в гербарии лаборатории биосистематики и цитологии БИН РАН. Четыре образца взяты из гербария MW (Гербарий МГУ им. М.В. Ломоносова). Ваучерные образцы, у которых ITS-последовательности и последовательности trnL-trnF были секвенированы нами, приведены в приложении. Списки исследованных видов для хлоропластных и ядерных последовательностей не идентичны, поскольку хлоропластные последовательности всех видов ковылей оказались практически одинаковы, что согласуется и с данными других авторов.
ДНК из сухих листьев гербарных образцов выделяли при помощи набора DiamondDNA Plant Kit (“Алтайбиотех”, Россия) согласно протоколу производителя или методом CTAB [21] c некоторыми модификациями. Для образцов Alt13-165, Alt16-28, Alt16-19, Alt16-24, Rost1-131, Rost1-132, Kur43-9, Kur43-10, MW0200213, MW0230130, MW0225114, Kr97-1, Kr97-2, Kr110-1, Kr110-2, Alt10-105, Alt-sc1 для амплификации участка ITS1-5.8S-ITS2 были использованы праймеры ITS5 и ITS4 [22]. Для всех остальных образцов при амплификации участка ITS1-5.8S-ITS2 использовали пары праймеров ITS1P [23] и ITS2, ITS3 и ITS4 [22]. Для амплификации хлоропластных последовательностей trnL-trnF были взяты пары праймеров tabC, tabD, tabE (B49873) и tabF [24].
Первичную обработку полученных после секвенирования хроматограмм и анализ последовательностей ДНК выполняли с помощью программ MEGA X [25] и Chromas (http://technelysium.com.au/ wp/chromas/). Выравнивание последовательностей проводилось с помощью алгоритма Muscle [26], включенного в пакет программ MEGA X, затем, при необходимости, выравнивание корректировалось вручную, после чего было проведено простое кодирование инделей [27] с помощью программы SeqState 1.4.1 [28]. Полученная матрица из “0” и “1” далее анализировалась как тип данных “restriction” программой Mr. Bayes при помощи F81-подобной модели для бинарных состояний (binary data) [29]. Нуклеотидное выравнивание и матрицу инделей анализировали одновременно, но как независимые блоки. В анализ также были включены некоторые ITS-последовательности Stipa из GenBank (табл. 1).
Таблица 1.
Последовательности ITS1-5.8S рДНК-ITS2, импортированные из базы данных GenBank и добавленные в матрицу данных при построении филогенетического дерева
| Вид | Номер GenBank, происхождение образца, авторы |
|---|---|
| Stipa baicalensis | FN434508, Mongolia: NW Ulan Bator Selenga Aimak [10] |
| S. capillata | FN434524, Russia: Baschkiria, Baymak, SW of Kolk Sumar [10] |
| S. capillata | FN434525, Italy: Aosta [10] |
| S. capillata | FN434526, Germany: Nehlitz near Halle [10] |
| S. dasyphylla | HE586634, Hungary + HE586637, Germany [10] |
| S. krylovii | FN434558, Russia: Jakutsk Botanical Garden [10] |
| S. lessingiana | FN434562, Turkey: Konya, South of Kiziloeren, 1550 m [10] |
| S. lessingiana | KC129480, Turkey: Dongancal [27] |
| S. stenophylla | FN434599, Russia: Bashkiria [10] |
| S. tirsa | FN434605, Germany: Goettingen Botanical Garden [10] |
| S. zalesskii | KF445688, Ukraine, Lugansk Province Striltzivsky Step Nat. Res. [8] |
| S. zalesskii | FN434576, Germany: Halle Botanical Garden, seeds from Kiev Botanical Garden [10] |
| Achnatherum confusum (Litv.) Tzvelev | FN434534, Mongolia: Bogd Kul [10] |
| A. confusum | JF697690, Kazakhstan, South Altai [11] |
| A. sibiricum (L.) Keng ex Tzvelev | FN434596, Mongolia: Gurvan Sajdan [10] |
| A. sibiricum | FJ766186, South Korea, Kim, Y.-D., Kim, K.-J., Kim, S.-H. and Lee, J.-H., неопубл. |
| A. sibiricum | FN434595, West Himalaya near Bhardarwah [10] |
| A. splendens (Trin.) Nevski | FN434478, China: Tibet, Dulan area, 3400 m [10] |
| A. splendens | FN434477, Mongolia [10] |
| Ptilagrostis junatovii Grubov | GU254619, Russia, West Altai [11] |
| P. mongholica (Turcz. ex Trin.) Griseb. | GU254621, Kyrgyzstan [11] |
| P. mongholica | EU489168, Kazakhstan [28] |
| Jarava ichu Ruiz & Pav. | AF019803, Hsiao et al., неопубл. |
Молекулярно-филогенетический анализ проводили с помощью программы Mr. Bayes 3.0 [29], генетические расстояния рассчитывали по модели GTR + I + G, 2 млн генераций, первые 25% деревьев были исключены как “burn-in”. Эволюционная модель была выбрана с помощью критериев AIC, BIC и lnL в программах MEGA X и jModelTest 2.1.6 [30]. Модель учитывает характерные особенности эволюции внутренних транскрибируемых спейсеров в рДНК – разную скорость нуклеотидных замен, высокое отношение числа транзиций к числу трансверсий (R = 5.76) и наличие инвариабельных сайтов [31].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ.
Анализ хлоропластных последовательностей ДНК trnL-trnF
Участок хлоропластного генома trnL-trnF был секвенирован у 16 образцов рода Stipa, относящихся к восьми видам, собранным на Алтае: S. borysthenica, S. capillata, S. glareosa, S. krylovii, S. lessingiana, S. orientalis, S. pulcherrima, S. zalesskii. (приложение). Общая длина секвенированного участка составила 941–944 пн, включая: 3'trnL экзон – 11 пн, trnL интрон – 524 пн, 5'trnL экзон – 49 пн, межгенный спейсер trnL-trnF – 354–357 пн. У всех образцов последовательности trnL-trnF начинаются с мотива 5'-CGCTACGGACT-3', соответствующего 3'-экзону гена trnL, и заканчиваются мотивом 5'-GTCAGGATAGC-3', соответствующим окончанию IGS trnL-trnF. Различия в длине спейсера trnL-trnF между образцами обусловлены числом нуклеотидов в поли-С-треке, что в данном случае нельзя считать видоспецифичными различиями. Показано, что длина поли-С-треков варьирует при использовании разных Taq-полимераз, разных концентрациях ДНК-матрицы в растворе и даже при выровненных условиях при повторной амплификации одного и того же образца (см., например: [32, 33]).
Все последовательности, включая 16, полученных в нашей работе, и восемь, взятых из GenBank, оказались практически идентичны и совпадали на 99.6–100%. Таким образом, район trnL-trnF, один из широко используемых в работе с ботаническими объектами при ДНК-штрихкодировании маркеров [18–20], не может быть применен при идентификации видов рода Stipa [34, 35]. Эти данные согласуются с недавними результатами полнопластомного секвенирования 19 видов ковылей, показавших крайне высокую гомогенность их хлоропластной ДНК; идентичность их хлоропластных геномов составила 99.7–100% [36]. Поскольку считается, что хлоропластная ДНК злаков передается по материнской линии [37, 38], то можно предположить, что исследованные виды ковылей унаследовали хлоропластные геномы от общего материнского предка.
Анализ ядерных последовательностей ITS1-5.8S рДНК-ITS2
Последовательность ядерных генов 35S рРНК (район ITS1-5.8S рДНК-ITS2) была секвенирована у 58 образцов, относящихся к 20 видам рода ковыль (Stipa) и двум видам рода ковылечек (Ptilagrostis) (приложение, происхождение образцов, секвенированных другими авторами, взятых нами в анализ, приведено в табл. 1). Нами впервые секвенированы последовательности ITS1-5.8S рДНК-ITS2 у Stipa consanguinea, S. korshinskyi, S. pulcherrima, S. praecapillata, S. ucrainica и, по-видимому, S. baicalensis. Известная ранее последовательность этого района образца, определенного как S. baicalensis из Монголии [10], содержит много неоднозначно прочтенных нуклеотидов и, за их исключением, идентична последовательностям ITS S. praecapillata и определенно отличается от секвенированных нами ITS типичных по морфологии [5, 13] образцов S. baicalensis из Монголии и Алтая.
Впервые секвенированы последовательности ITS нескольких видов флоры России, ранее известные только для образцов, собранных вне нашей страны. Это: S. borysthenica, S. pontica, S. tirsa (три образца из Воронежской, Ростовской и Курской областей Европейской части России – ранее была известна одна последовательность ITS образца из Ботсада в Германии [10]), S. lessingiana (ранее секвенированы ITS двух растений из Турции [10, 34] и одного из Греции [34]), S. pennata (ранее известны ITS растения из Польши [12] и с Украины [11]), два образца S. glareosa с Алтая (ранее известны ITS двух растений из Монголии [10]), S. grandis (ранее известна ITS одного образца из Монголии [10]), S. dasyphylla (ранее была известна последовательность ITS1 растения из Венгрии и ITS2 растения из Германии [10], нами секвенирован полноразмерный район ITS двух растений этого вида, собранных в Курской области и ITS растения, собранного в Алтайском крае), S. desertorum (два образца из Горного Алтая; последовательность ITS FN434530, ранее секвенированная для растения из Киргизии [10], отличается от наших пятью транзициями и одной трансверсией).
Ранее не был секвенирован ни один образец рода Stipa из такого центра разнообразия злаков как Алтайский край и Республика Алтай. В частности среди таких видов надо упомянуть S. capillata. Нами секвенировано восемь образцов из Алтая, два из Хакасии и ITS одного растения, собранного нами в Волгоградской области. Ранее были известны ITS-последовательности одного образца из Германии, одного из Италии и одного из Башкирии [10]. В данный момент S. capillata – это по-видимому единственный вид ковыля, для которого секвенированы ITS более чем десяти растений из разных частей ареала. Нужно также отметить, что имеющиеся в базе данных GenBank ITS-последовательности S. borysthenica (FN434512, образец из Чехии), S. krylovii (FN434508, Якутск), S. lessingiana (FN434561, Германия), S. pontica (FN434579, Германия), S. tirsa (FN434605, Германия) были секвенированы из образцов, полученных в виде семян из ботанических садов [10]. Такой материал всегда требует проверки. Так Н.П. Авдулов [39] приводит 27 случаев, когда семена злаков, присланные ему из ботанических садов, относились к другому даже не виду, а роду, нежели тот, что значился на этикетке, и добавляет, что приведенный список – лишь небольшой процент от встретившихся ему случаев ошибок в этикетировании образцов злаков, присланных из ботанических садов. Следует также помнить, что злаки, выращиваемые в ботанических садах на газонах и альпийских горках в самых невероятных комбинациях видов, никак не защищены от случайных межвидовых скрещиваний – это известная проблема ex situ-коллекций растений в ботанических садах [40, 41].
Общая длина выравнивания района ITS1-5.8S рДНК-ITS2 с учетом инделей составила 601 пн, включая ITS1 (221 пн), ген, кодирующий 5.8S рРНК (160 пн), ITS2 (220 пн). Среднее попарно рассчитанное р-расстояние для данной выборки было 0.032 (p = nd/n, где nd – число позиций, по которым сравниваемые последовательности различаются, а n – длина сравниваемых позиций). Вариации для образцов, относимых к одному виду, были менее 1%: для S. capillata 0.000–0.007, для S. krylovii 0.000–0.003, S. glareosa 0.002–0.010.
Участок 5.8S рДНК начинается с мотива 5'-GACTCTCGGC-3', а заканчивается мотивом 5'-GGСGTCАCGC-3' у всех исследованных образцов. У всех секвенированных нами образцов рода Stipa последовательности, кодирующие ген 5.8S рРНК, высоко консервативны и идентичны представленным в GenBank, за одним примечательным исключением: обращает на себя внимание, что все 140 помещенных в GenBank последовательностей района ITS видов рода Stipa и нескольких близких родов, секвенированных Хамаша с соавт. [10], в том числе 85 последовательностей 66 видов и подвидов рода Stipa, имеют полиморфный сайт C/T в позиции 289 нашего выравнивания (ген 5.8S рРНК) – мутация, не встречающаяся в наших последовательностях. По-видимому это указание на то, что секвенированные нами районы ITS видов рода Stipa с использованием пар праймеров ITS1P/ITS2 [22, 23], ITS3/ITS4 [23]. ITS5/ITS4 [23] и последовательности этого же района генома, секвенированные Хамаша с соавт. [10] с помощью праймеров ITS-A, -B, -C и -D, разработанных Блаттнером [42], представляют паралоги множественных генов 35S рРНК исследуемых видов, что конечно затрудняет интерпретацию результатов, полученных в разных лабораториях. Тем не менее видно, что последовательности 5.8S рДНК ковылей отличаются от таковых у представителей наиболее близких родов Achnatherum и Ptilagrostis одной однонуклеотидной заменой C/T в 352-й позиции у Achnatherum splendens и в 353-й позиции у прочих Achnatherum и Ptilagrostis; оба представителя Ptilagrostis отличаются от прочих ковылевых заменой G/A в позиции 342.
Длина спейсера ITS1 составила 221 пн. Началом спейсера у всех исследуемых образцов является мотив 5'-TCGTGACCCT-3', окончанием 5'‑TCTAATCCAC-3'. Длина спейсера ITS2 – 220 пн, началом спейсера является мотив 5'-CAAAACACGCT-3', окончанием – 5'-CCGCGACCCC-3' У представителей рода Stipa в последовательности ITS1 мы выявили 14, а в ITS2 – 17 вариабельных позиций (однонуклеотидные замены, полиморфные сайты и индели), которые, включая полиморфные сайты, можно считать парсимонично информативными (табл. 2).
Таблица 2.
Нуклеотидные замены в районе ITS1 у исследованных нами видов алтайских ковылей
| Position | 13 | 34 | 46 | 51 | 55 | 61 | 67 | 100 | 111 | 137 | 149 | 167 | 172 | 197 | 199 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Consensus | Section* | G | C | С | G | С | C | C | C | G | G | G | G | C | С | C |
| S. ucrainica Kr110-1, Kr110-2 | S | T | T | |||||||||||||
| S. pontica Kr97-1, Kr97-2 | S | T | T | |||||||||||||
| S. dasyphylla HE586634 + HE586637 | S | T | T | |||||||||||||
| S. dasyphylla Kur43-9, Kur43-10 | S | T | C+T | T | T | |||||||||||
| S. dasyphylla Alt16-30 | S | T | T | T | ||||||||||||
| S. rubens Dag18-76 нетипичный | S | T | T | |||||||||||||
| S. zalesskii Alt16-28, FN434576 | S | T | T | |||||||||||||
| S. zalesskii Alt13-33, Alt13-165, Alt16-19, Alt16-24 | S | C+T | T | T | ||||||||||||
| S. zalesskii KF445688 | S | |||||||||||||||
| S. tirsa MW0200213, MW0230130, MW0225114 | S | |||||||||||||||
| S. pulcherrima Alt14-624 | S | |||||||||||||||
| S. pennata Alt16-17 | S | |||||||||||||||
| S. pennata Alt14-339 | S | C + T | C + T | |||||||||||||
| S. rubens Rost1-131, Rost1-132 | S | C + T | C + T | |||||||||||||
| S. zalesskii Alt14-674 нетипичный | S | A | C + T | C + T | C + T | |||||||||||
| S. borysthenica Alt14-413 | S | G + A | T | C + T | C + T | C + G | ||||||||||
| S. capillata нетипичный Kha09-170, Alt13-338 | L | G + A | T | C + T | C + T | C + T | ||||||||||
| S. capillata Alt18-14, Alt797, Alt945, Alt18-73, Kha09-118, Alt-Sc1, FN434525, FN434524 | L | A | – | A | T | |||||||||||
| S. capillata Vol16-26, Alt18-199 | L | G + A | – | A | T | |||||||||||
| S. capillata FN434526 | L | – | A | T | ||||||||||||
| S. capillata нетипичный Alt09-55 | L | A | G + A | C + T | C + T | G + A | C + T | |||||||||
| S. baicalensis Alt15-373 | L | G + A | C + T | C + A | C + T | G + A | C + T | |||||||||
| S. baicalensis Mon18-1 | L | A | T | |||||||||||||
| S. baicalensis FN434508 | L | C + T | C + T | C + A | T | |||||||||||
| S. grandis Alt16-416, Alt16-415, Kha09-91 | L | A | T | |||||||||||||
| S. krylovii Alt13-89 | L | G + A | T | C + T | C + T | G + A | C + T | . | ||||||||
| S. krylovii Alt12-122 | L | G + A | T | C + T | C + T | C + T | ||||||||||
| S. krylovii Ty09-129, Alt16-351, Alt16-386, FN434558 | L | T | C + T | T | ||||||||||||
| S. praecapillata Alt16-240, Alt16-233 | L | T | T | |||||||||||||
| S. consanguinea Alt16-343 | H | T | C + T | T | ||||||||||||
| S. orientalis Alt13-346, Alt1306 | B | T | T | T | A | |||||||||||
| S. glareosa Alt13-285, Alt16-344 | Sm | A | T | T | ||||||||||||
| S. desertorum Alt16-363, Alt10-105 | Sm | T | T | T | ||||||||||||
| S. lessingiana Alt14-516, Alt16-9, FN434562, KC129480 | Su | |||||||||||||||
| S. korshinskyi Alt16-34 | H |
На филогенетическом древе алтайских представителей трибы Stipeae все виды рода Stipa образуют единую кладу (рис. 1). В отдельных кладах оказались также представители родов Ptilagrostis и Achnatherum, за исключением вида A. splendens, что полностью согласуется с результатами, ранее полученными другими исследователями [7, 10–12, 35, 43].
Рис. 1.
Филогенетическое дерево видов рода Stipa, построенное на основании сравнения последовательностей района ITS1-5.8S rDNA-ITS2 генов 35S в программе Mr. Bayes 3.0. Полужирным шрифтом выделены виды, ITS-районы которых были секвенированы нами в ходе настоящей работы. Над ветвями дерева указана апостериорная вероятность (в процентах).
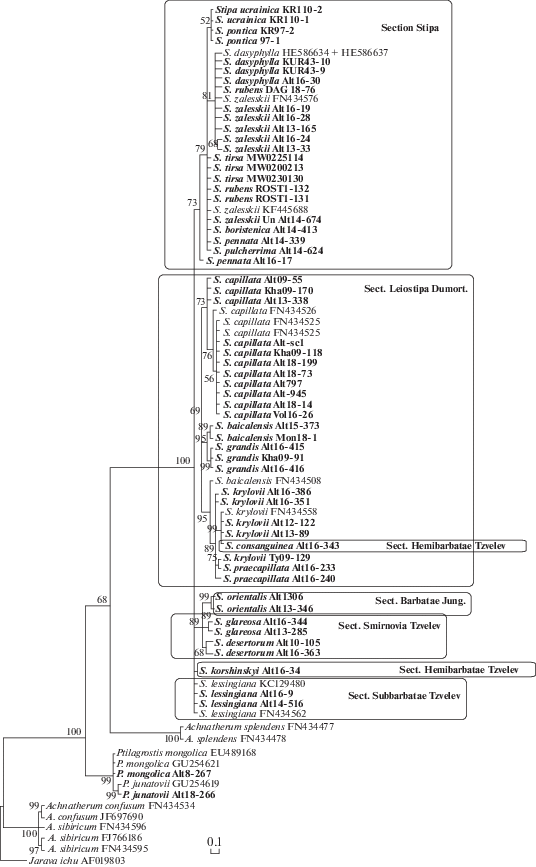
Участок ITS1-5.8S рДНК-ITS2 широко используется при молекулярно-филогенетическом анализе у цветковых растений и в частности у злаков [44, 45], позволяя решать многие таксономические вопросы на внутриродовом уровне. Однако ранее считалось, что род ковыль является, в данном случае скорее исключением, всегда демонстрируя монофилетичную, но неразрешимую кладу. При этом даже хорошо морфологически различаемые виды не показывали достоверных различий на уровне каких-либо субклад [10, 11].
В нашем исследовании нам удалось выявить субклады, которые соответствуют секциям, группам сходных видов внутри секций и даже отдельным видам. Мы полагаем, что это связано с тем, что мы учитывали не только замены нуклеотидов, но также индели. Эти же субклады соответствуют риботипам, которые можно выявить в исследуемых последовательностях (табл. 2 и 3, рис. 1).
Таблица 3.
Нуклеотидные замены в районе ITS2 у исследованных нами видов алтайских ковылей
| Position | 401 | 402 | 404 | 414 | 415 | 435 | 452 | 528 | 531 | 535 | 539 | 544 | 551 | 558 | 579 | 581 | 585 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| consensus | С | С | — | G | G | C | C | C | — | C | T | G | C | — | C | C | C |
| S. pulcherrima Alt14-624 S. tirsa MW0200213, MW0230130, MW0225114 S. borysthenica Alt14-413 S. pennata Alt14-339 S. zalesskii Alt14-674, Alt13-165, Alt16-28, Alt16-19, Alt13-33, Alt16-24, FN434576, KF445688 S. rubens Dag18-76, Rost1-131, Rost1-132 S. dasyphylla Alt16-30, Kur43-9, Kur43-10 S. pontica Kr97-1, Kr97-2 S. ucrainica Kr110-1, Kr110-2 |
— | T | — | T | — | ||||||||||||
| S. pennata Alt16-17 | C | — | T | — | |||||||||||||
| S. dasyphylla HE586634 + HE586637 | — | T | A | T | C + T | — | |||||||||||
| S. lessingiana FN434562, KC129480 | C | C | — | ||||||||||||||
| S. lessingiana Alt14-516, Alt16-9 | C | C + T | C | C + T | — | ||||||||||||
| S. korshinskyi ALT16-34 | C | C | — | ||||||||||||||
| S. desertorum Alt16-363, Alt10-105 | T | C | C | — | |||||||||||||
| S. glareosa Alt13-285, Alt16-344 | T | C | T | C | — | ||||||||||||
| S. orientalis Alt13-346, Alt1306 | T | C | C | A | — | ||||||||||||
| S. praecapillata Alt16-240, Alt16-233 | — | — | C | A | T | C | |||||||||||
| S. grandis Alt16-416, Alt16-415, Kha09-91 | — | — | C | — | G | ||||||||||||
| S. baicalensis Alt15-373, Mon18-1 | — | — | C | — | T | ||||||||||||
| S. baicalensis FN434508 | — | — | C | T | C | ||||||||||||
| S. consanguinea Alt16-343 S. krylovii Alt12-122, Alt13-89, FN434558 |
T | — | — | T | C | A | T | C | |||||||||
| S. krylovii Ty09-129, Alt16-351, Alt16-386 | — | — | C | A | T | C | |||||||||||
| S. capillata Vol16-12, Alt18-199, Alt18-14, Alt797, Alt945, Alt18-73, Kha09-118, Alt-Sc1, Kha09-170, Alt13-338, Alt09-55, FN434525, FN434524, FN434526 | — | — | C | — | T |
Так, отдельный риботип и клада “Stipa” соответствуют признаваемой большинством исследователей секции Stipa [5, 46–48 и др.]. Эта секция объединяет более 60 видов Северной Африки и Евразии и характеризуется в первую очередь длинными перистыми остями. Отношения видов/подвидов внутри секции до сих пор остаются не ясными. Наши данные показывают, что у видов этой секции последовательности ITS2 почти идентичны, но хорошо отличаются при этом от последовательности ITS2 всех прочих проанализированных видов (табл. 3). В последовательности ITS1 суммарно выявляется девять позиций, содержащих замены, в большинстве случаев это транзиции С‑T. Для целей ДНК-идентификации видов существенно, что ITS S. zalesskii определенно отличается от ITS S. pennata, с которым его ранее предлагалось объединить (в ранге подвида) [38]. От вида S. zalesskii по морфологии трудно отличить S. ucrainica и S. rubens [5, 46]. В частности собранный в Дагестане образец Dag18-76, обладающий промежуточным набором признаков, с определенным сомнением, но был отнесен нами к виду S. rubens (приложение, рис. 1). Однако по ITS – это типичный S. zalesskii. S. ucrainica по ITS определенно отличается от S. zalesskii (рис. 1, табл. 2), что может быть использовано при ДНК-штрихкодировании. Напротив, морфологически S. zalesskii и S. dasyphylla хорошо различимы, однако у них две общие, характерные только для них, транзиции в ITS1 и сходного состава полиморфный сайт в том же спейсере. Возможно родство между этими видами больше, чем предполагалось ранее. Все виды типовой секции Stipa отличают от представителей других секций делеция и замена в ITS2 A529C–CGGT. A529CCCGGC – удобный район для конструирования секция-специфичного праймера.
Следующая большая клада, объединяющая ковыли с волосовидными не перистыми остями, в целом соответствует признаваемой большинством исследователей секции Leiostipa Dumort. [46]. Виды этой секции имеют специфическую однонуклеотидную делецию в ITS2 – C410GGGG–AG, у остальных – C410GGGGGAG. В этой кладе хорошо выделяются субклады, соответствующие нескольким риботипам. К риботипу “Capillata” относятся все проанализированные нами и взятые из GenBank образцы S. capillata, большинство из них характеризуется видоспецифичной однонуклеотидной делецией в ITS1 (позиция 109) и транзициями G/A (п. 165) в ITS1 и C/T (п. 576) в ITS2. К риботипу “Baicalensis-Grandis” можно отнести S. baicalensis и S. grandis, причем S. grandis имеет и видоспецифичную трансверсию C/G в ITS2 (п. 582). Риботип и субклада “Krylovii-Praecapillata” соответствуют видам S. krylovii и S. praecapillata, но последовательности ITS1 и ITS2 этих видов оказались практически идентичны. Характерный для только для них и S. consanguinea мотив G550TGGCCCCCT, а у остальных видов – G550CGGCCCC–T. Подчеркнем, что в этом случае S. krylovii и S. praecapillata имеют одинаковые ITS, несмотря на то, что эти виды хорошо различаются морфологически, и их ареалы не перекрываются. Их роднит то, что оба вида имеют хорошо развитую коронку волосков у основания ости, что давало основание Н.Н. Цвелеву думать, что S. krylovii “участвовал в происхождении” S. praecapillata [46]. Может быть, в этом причина сходства их ITS-последовательностей. В эту же субкладу попадает и эндемичный алтайский вид S. consanguinea, который, по мнению Н.Н. Цвелева [5] также возможно имеет гибридное происхождение S. krylovii × S. desertorum или S. krylovii × S. glareosa [5]. Позднее Н.Н. Цвелев, на том основании, что ости у этого вида покрыты мелкими волосками менее 1 мм длиной, отнес этот вид в секцию Hemibarbatae Tzvelev, отмечая при этом его обособленное положение в секции (у других видов секции ости дважды коленчато согнуты, а у S. consanguinea – один) и думая, что он вероятно, заслуживает выделения в особую секцию [46].
Виды с перистыми, но относительно короткими остями – S. orientalis, S. glareosa и S. desertorum – сформировали хорошо обособленную субкладу (риботип “Orientalis-Glareosa-Desertorum”), хотя их принято относить к разным секциям: Barbatae Junge и Smirnovia Tzvelev [46, 47, 49, 50]. Основное морфологическое различие видов этих секций – наличие двух или одного коленчатого изгиба остей. Наше исследование также подтверждает обособленность вида S. desertorum, самостоятельность которого до сих пор являлась дискуссионной [51].
Неопределенное положение в кладе ковылей занимают виды S. lessingiana и S. korshinskyi, причем первый относят к секции Subbarbatae Tzvelev, а второй относили в разное время к разным секциям: Leiostipa [5], позже по наличию завязи с тремя/четырьмя рыльцами – к секции Barbatae [47], а в последнее время – к секции Hemibarbatae Tzvelev [46], так как этот вид имеет коротко опушенные ости, так же как у S. consanguinea.
Недавно группой польских исследователей было показано, что межгенный спейсер IGS, а особенно его часть, прилегающая к 26S рДНК может нести достаточный филогенетический сигнал и является наиболее вариабельным участком во всем кластере рДНК ковылей [12]. По этому участку было проанализировано 36 видов ковылей, среди которых были и виды, встречающиеся на Алтае. На филогенетическом дереве, построенном по методу максимальной парсимонии, многие виды сформировали субклады, как и в нашем случае вполне согласующиеся с секционным делением. Так, хорошо обособленные клады сформировали виды секции Stipa (S. zalesskii, S. dazyphylla, S. pennata, S. tirsa) и секции Leiostipa (S. capillata, S. krylovii, S. capillata, S. baicalensis, S. grandis). Также в одну субкладу попали S. orientalis и S. glareosa. И в то же время в одной группе оказались объединены виды S. lessingiana и S. richteriana Kar. & Kir., относимые к разным секциям, соответственно Subbarbatae и Нemibarbatae.
Исследованные нами виды ковылей секции Hemibarbatae с короткоопушенными остями (S. consanguinea и S. korshinskyi) оказались в разных филогенетических ветвях рода Stipa (рис. 1). Обоснованность выделения этой секции можно подтвердить или опровергнуть только после того как будут исследованы ITS двух других видов секции – кавказских видов S. sosnowskyi Seredin и S. daghestanica Grossh. Как уже сказано, Н.Н. Цвелев не был уверен в принадлежности S. consanguinea к секции Нemibarbatae [46], предполагая гибридогенную природу этого вида [5].
Вообще вопрос о распространенности межвидовой гибридизации у ковылей и ее роли в видообразовании до сих пор неясен. Н.Н. Цвелев [52] предполагал, что у ковылей, в отличие от других родов злаков, она встречается редко, и лишь отдельные случаи можно считать фактом межвидовой гибридизации. Другие же исследователи, напротив, полагают, что межвидовая гибридизация, как внутри- так и межсекционная у ковылей встречается очень часто [50, 53, 54]. Отметим, что до сих пор в роде Stipa sensu stricto достоверно неизвестны никакие другие хромосомные числа, кроме 2n = 44, следовательно межвидовые гибриды ковылей, если они существуют, должны быть отнесены к гомоплоидным гибридам.
В связи с этим представляет интерес анализ полиморфных сайтов (PS) в составе ITS-последовательностей ковылей. Принято считать, что несмотря на многокопийность рибосомных генов в геноме, эти последовательности достаточно однородны; и у межвидовых гибридов как и у аллополиплоидов быстро происходит их изогенизация или утрата рДНК одного из родителей. В результате при секвенировании ITS мы обнаруживаем последовательность только одного из родительских видов [44, 55–57]. Однако имеются примеры длительно существующего внутригеномного полиморфизма рДНК. В этом случае при секвенировании по Сэнгеру на хроматограммах выявляются двойные пики (PS), свидетельствующие о присутствии в геноме рДНК разного происхождения. Самый яркий пример – это дикорастущие и культивируемые пионы [58–60]. У межвидовых гибридов пионов и видов гибридного происхождения в позициях нуклеотидов, по которым отличались ITS видов-родителей, регистрируются PS. Исследования некоторых аллополиплоидных злаков и дикорастущих межродовых гибридов из круга родства рода Elymus L. (пырейник) показало более сложную картину [61]. В этом случае часть PS у гибридов соответствовала различающимся нуклеотидам в последовательностях предполагаемых родителей, а часть по-видимому возникла de novo как результат высокой мутабильности или отсутствия гомогенизации рДНК у нестабильного композитного генома межвидового гибрида [62].
Обращает на себя внимание, что у ковылей почти все выявленные нами PS сконцентрированы в районе ITS1 (табл. 2). Существенное количество PS (от трех до шести) найдено у всех не типичных по морфологии образцов (S. zalesskii Alt14-674, S. capillata Kha09-170, Alt13-338 и Alt09-55), а также у вполне типичных образцов S. krylovii Alt13-89, Alt12-122, S. baicalensis Alt15-373 и S. borysthenica Alt14-413, которые теоретически могли иметь предков из других видов ближайшего круга родства. Вместе с тем у видов, относимых Н.Н. Цвелевым в секцию Hemibarbatae (S. consanguinea и S. korshinskyi), которые гипотетически могли бы возникнуть в результате более отдаленной, межсекционной гибридизации, мы практически не выявляем PS. Однако если последовательности другого родителя все же присутствуют у гибрида или гибридогенного вида в минорном количестве, то при секвенировании по Сэнгеру PS зарегистрированы не будут. Для их выявления необходимы методы NGS-секвенирования, позволяющие выявить весь спектр вариантов повторяющихся последовательностей и долю каждого из них [63]. Очевидно, что вопрос о природе внутригеномного полиморфизма рДНК и роли межвидовой гибридизации в эволюции ковылей требует дальнейших исследований.
Авторы благодарят А.И. Шмакова, А.П. Шалимова, А.А. Кечайкина, М.С. Раутиан, А.А. Родионову, А.Н. Черепанова и А.В. Караблина за помощь в организации и проведении полевых работ.
Работа выполнена на оборудовании ЦКП “Клеточные и молекулярные технологии изучения растений и грибов”. При поддержке грантов РФФИ № 18-04-01040, РФФИ № 18-34-20112, в рамках госзаданий МГУ им. М.В. Ломоносова (тема АААА-А16-116021660045-2) и БИН РАН (тема АААА-А18-118040290161-3).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Крылов П.Н. Род Stipa // Флора Западной Сибири. Томск: Изд-во “Красное знамя”, 1928. Вып. 2. С. 137–385.
Рожевиц Р.Ю. Род Stipa L. // Флора СССР. Л.: АН СССР, 1934. Т. 2. С. 79–112.
Смирнов П.А. Род Stipa // Флора Юго-Востока Европейской части СССР. Л.: [б. и.], 1928. С. 98–118.
Смирнов П.А. Ковыли СССР // Бюл. МОИП. 1936. Т. 43. № 2. С. 104–118.
Цвелев Н.Н. Злаки СССР. Л.: Наука, 1976. С. 567–595.
Barkworth M.E., Arriaga M.O., Smith J.F. et al. Molecules and morphology in South American Stipeae (Poaceae) // Syst. Bot. 2008. V. 33. P. 719–731. https://doi.org/10.1600/036364408786500235
Romaschenko K., Garcia-Jacas N., Peterson P.M. et al. Miocene-Pliocene speciation, introgression, and migration of Patis and Ptilagrostis (Poaceae: Stipeae) // Mol. Phylogenet. Evol. 2014. V. 70. P. 244–259. https://doi.org/10.1016/j.ympev.2013.09.018
Soreng R.J., Peterson P.M., Romaschenko K. et al. A worldwide phylogenetic classification of the Poaceae (Gramineae) // J. Syst. Evol. 2015. V. 53. P. 117–137. https://doi.org/10.1111/jse.12150
Olonova M.V., Barkworth M.E., Gudkova P.D. Lemma micromorphology and the systematics of Siberian species of Stipa (Poaceae) // Nord. J. Bot. 2016. V. 34. P. 319–328. https://doi.org/10.1111/njb.00881
Hamasha H.R, von Hagen K.B., Roser M. Stipa (Poaceae) and allies in the Old World: Molecular phylogenetics realigns genus circumscription and gives evidence on the origin of American and Australian lineages // Plant. Syst. Evol. 2012. V. 298. P. 351–367. https://doi.org/10.1007/s00606-011-0549-5
Romaschenko K., Peterson P.M., Soreng R.J. et al. Systematics and evolution of the needle grasses (Poaceae: Pooideae: Stipeae) based on analysis of multiple chloroplast loci, ITS, and lemma micromorphology // Taxon. 2012. V. 61. P. 18–44.
Krawczyk K., Nobis M., Nowak A. et al. Phylogenetic implications of nuclear rRNA IGS variation in Stipa L. (Poaceae) // Sci. Reports. 2017. V. 7. P. 11506. https://doi.org/10.1038/s41598-017-11804-x
Ломоносова М.Н. Stipa L. – Ковыль // Флора Сибири. Новосибирск: Наука, 1990. Т. 2. С. 222–230.
Ломоносова М.Н. Stipa L. – Ковыль // Определитель растений Алтайского края. Новосибирск: Изд. СО РАН, 2003. Т. 2. 579 с.
Пунина Е.О., Носов Н.Н., Гнутиков А.А. и др. Находки видов Stipa в Республике Алтай и Алтайском крае // Turczaninowia. 2018. Т. 21. № 3. С. 77–80.
Золотов Д.В., Таран Г.С. Флористические находки в Алтайском крае // Бюлл. МОИП. Отд. биол. 2008. Т. 113. № 3. С. 83–84.
Гудкова П.Д., Нобис М., Олонова М.В. Stipa pulcherrima С. Koch в Алтайском крае – таксономия и распространение // Проблемы ботаники Южной Сибири и Монголии. 2014. № 13. С. 64–66.
Hollingsworth P.M., Graham S.W., Little D.P. Choosing and using a plant DNA barcode // PLoS One. 2011. V. 6(5): e19254. https://doi.org/10.1371/journal.pone.0019254
Жохова Е.В., Родионов А.В., Повыдыш М.Н. и др. Современное состояние и перспективы использования ДНК-штрихкодирования и ДНК-фингерпринтинга для анализа качества лекарственного растительного сырья и лекарственных растительных препаратов // Усп. современ. биол. 2019. Т. 139. № 1. С. 25–40.
Шнеер В.С., Родионов А.В. ДНК-штрихкоды растений // Усп. современ. биол. 2018. Т. 138. № 6. С. 531–538.
Doyle J.J., Doyle J.L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue // Phytochem. Bull. 1987. V. 19. P. 11–15.
White T.J., Bruns T., Lee S., Taylor J. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics // PCR Protocols: A Guide to Methods and Applications / Eds Innis M.A., Gelfand D.H., Sninsky J.J., White T.J. San Diego: Academic Press, 1990. P. 315–322.
Ridgway K.P., Duck J.M., Young J.P.W. Identification of roots from grass swards using PCR-RFLP and FFLP of the plastid trnl (UAA) intron // BMC Ecol. 2003. V. 3. P. 8. https://doi.org/10.1186/1472-6785-3-8
Taberlet P., Gielly L., Pautou G., Bouvet J. Universal primers for amplification of three non-coding regions of chloroplast DNA // Plant Mol. Biol. 1991. V. 17. P. 1105–1109.
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: Molecular evolutionary genetics analysis across computing platforms // Mol. Biol. Evol. 2018. V. 35. P. 1547–1549. https://doi.org/10.1093/molbev/msw054
Edgar R.C. MUSCLE: Multiple sequence alignment with high accuracy and high throughput // Nucl. Acids Res. 2004. V. 32. P. 1792–1797. https://doi.org/10.1186/1471-2105-5-113
Simmons M.P., Ochoterena H. Gaps as characters in sequence-based phylogenetic analyses // Syst. Biol. 2000. V. 49. P. 369–381.
Müller K. SeqState: Primer design and sequence statistics for phylogenetic DNA datasets // Appl. Bioinformatics. 2005. V. 4. P. 65–69.
Huelsenbeck J.P., Ronquist F. MRBAYES: Bayesian inference of phylogenetic trees // Bioinformatics. 2001. V. 17. P. 754–755.
Darriba D., Taboada G.L., Doallo R., Posada D. jModelTest 2: More models, new heuristics and parallel computing // Nat. Methods. 2012. V. 9. P. 772. https://doi.org/10.1038/nmeth.2109
Sullivan J., Joise P. Model selection in phylogenetics // Annu. Rev. Ecol. Syst. 2005. V. 36. P. 445–466. https://doi.org/10.1146/annurev.ecolsys.36.102003.152633
Seo S.B., Jang B.S., Zhang A. et al. Alterations of length heteroplasmy in mitochondrial DNA under various amplification conditions // J. Forensic Sci. & Criminal Inves. 2010. V. 55. P. 719–722.
Maki M., Yamashiro T., Dohzono I., Suzuki K. Molecular phylogeny of Isodon (Lamiaceae) in Japan using chloroplast DNA sequences: Recent rapid radiations or ancient introgressive hybridization? // Plant Species Biol. 2010. V. 25. P. 240–248.
Syme A.E., Murphy D.J., Holmes G.D. et al. An expanded phylogenetic analysis of Austrostipa (Poaceae: Stipeae) to test infrageneric relationships // Aust. Syst. Bot. 2012. V. 25. P. 1–10. https://doi.org/10.1071/SB10049
Romaschenko K., Peterson P.M., Soreng R.J. et al. Molecular phylogenetic analysis of the American Stipeae (Poaceae) resolves Jarava sensu lato polyphyletic: Evidence for a new genus, Pappostipa // J. Bot. Res. Inst. Texas. 2008. V. 2. P. 165–192.
Krawczyk K., Nobis M., Myszczyński K. et al. Plastid super-barcodes as a tool for species discrimination in feather grasses (Poaceae: Stipa) // Sci. Reports. 2018. V. 8. P. 1924. https://doi.org/10.1038/s41598-018-20399-w
Corriveau J.L., Coleman A.W. Rapid screening method to detect potential biparental inheritance of plastid DNA and results for over 200 angiosperm species // Am. J. Bot. 1988. V. 75. P. 1443–1458. https://doi.org/10.1002/j.1537-2197.1988.tb11219.x
Primavesi L.F., Wu H., Mudd E.A. et al. Visualisation of plastid degradation in sperm cells of wheat pollen // Protoplasma. 2017. V. 254. P. 229–237. https://doi.org/10.1007/s00709-015-0935-x
Авдулов Н.П. Карио-систематическое исследование семейства злаков // Тр. по прикладной ботанике, генетике и селекции. Приложение 44. Л.: 1931. С. 57.
Maunder M., Hughes C., Hawkins J.A., Culham A. Hybridization in ex situ plant collections: Conservation concerns, liabilities, and opportunities // Ex situ Plant Conservation: Supporting Species Survival in the Wild / Eds Guerrant E.O. Jr., Havens K., Maunder M.. Washington, Covelo, London: Island Press, 2004. P. 3–20.
Smirnov S., Skaptsov M., Shmakov A. et al. Spontaneous hybridization among Allium tulipifolium and A. robustum (Allium subg. Melanocrommyum, Amaryllidaceae) under cultivation // Phytotaxa. 2017. V. 301. P. 155–164.
Blattner F.R. Direct amplification of the entire ITS region from poorly preserved plant material using recombinant PCR // Biotechniques. 1999. V. 27. P. 1180–1186.
Jacobs S., Bayer R., Everett J. et al. Systematics of the tribe Stipeae (Gramineae) using molecular data // Aliso. 2007. V. 23. P. 349–361.
Rodionov A.V., Gnutikov A.A., Kotsinyan et al. ITS1-5.8S rDNA-ITS2 sequence in 35S rRNA genes as marker during reconstruction of phylogeny of cereals (Poaceae family) // Biol. Bull. Reviews. 2017. V. 7. № 2. P. 85–102.
Cialdella A.M., Sede S.M., Romaschenko K. et al. Phylogeny of Nassella (Stipeae, Pooideae, Poaceae) based on analyses of chloroplast and nuclear ribosomal DNA and morphology // Syst. Bot. 2014. V. 39. P. 814–828. https://doi.org/10.1600/036364414X681419
Цвелев Н.Н. Заметки о трибе Ковылевых (Stipeae Dumort., Poaceae) // Новости сист. высш. раст. 2012. Т. 43. С. 22–29.
Freitag H. The genus Stipa (Gramineae) in Southwest and South Asia // Notes Roy. Bot. Gard. Edinburgh. 1985. V. 42. P. 355–489.
Vázquez P.F.M., Gutiérrez E.M. Classification of species of Stipa with awns having plumose distal segments // Telopea. 2011. V. 13. № 1–2. P. 155–176.
Gonzalo P., Aedo C., Garcia M.A. Taxonomic revision of Stipa section Smirnovia and section Subsmirnovia (Poaceae) // Bot. J. Linn. Soc. 2011. V. 167. P. 394–448.
Nobis M. Taxonomic revision of the Stipa lipskyi group (Poaceae: Stipa section Smirnovia) in the Pamir Alai and Tian-Shan Mountains // Plant Syst. Evol. 2013. V. 299. P. 1307–1354.
Гудкова П.Д., Эбель А.Л., Нобис М.М., Верхозина А.В. Stipa glareosa Р.А. Smirn. (Poaceae) в Прибайкалье // Интеграция ботанических исследований и образования: традиции и перспективы. Тр. Межд. научно-практич. конф., посвященной 125-летию каф. ботаники. Томск. 12–15 ноября 2013 г. Томск: Изд-во Томского университета, 2013. С. 38–41.
Цвелёв Н.Н. О некоторых гибридогенных таксонах в роде ковыль (Stipa L., Poaceae) // Новости систематики высших растений. 2014. Т. 45. С. 5–8.
Котухов Ю.А. Конспект ковылей (Stipa L.) и ковылечков (Ptilagrostis Griseb.) Восточного Казахстана (Казахстанский Алтай, Зайсанская котловина и Приалтайские хребты) // Бот. исследования Сибири и Казахстана. 2002. Т. 8. С. 3–16.
Nobis M., Gudkova P.D. Taxonomic notes on feather grasses (Poaceae: Stipa) from Eastern Kazakhstan with typification of seven names and one new combination // Phytotaxa. 2016. V. 245. P. 31–42.
Kotseruba V., Gernand D., Meister A., Houben A. Uniparental loss of ribosomal DNA in the allotetraploid grass Zingeria trichopoda (2n = 8) // Genome. 2003. V. 46. P. 156–163.
Malinska H., Tate J.A., Matyasek R. et al. Similar patterns of rDNA evolution in synthetic and recently formed natural populations of Tragopogon (Asteraceae) allotetraploids // BMC Evol. Biol. 2010. V. 10. P. 291–308. https://doi.org/10.1186/1471-2148-10-291
Родионов А.В., Носов Н.Н., Ким Е.С. и др. Происхождение полиплоидных геномов мятликов (Poa L.) и феномен потока генов между Северной Пацификой и субантарктическими островами // Генетика. 2010. Т. 46. № 12. С. 1598–1608.
Sang T., Crawford D.J., Stuessy T.F. Documentation of reticulate evolution in peonies (Paeonia) using internal transcribed spacer sequences of nuclear ribosomal DNA: Implications for biogeography and concerted evolution // Proc. Natl Acad. Sci. USA. 1995. V. 92. P. 6813–6817.
Пунина Е.О., Мачс Э.М., Крапивская Е.Е. и др. Межвидовая гибридизация в роде Paeonia (Paeoniaceae): полиморфные сайты в транскрибируемых спейсерах генов 45S рРНК как индикаторы происхождения природных и искусственных гибридов пионов // Генетика. 2012. Т. 48. № 7. С. 812–826.
Пунина Е.О., Мачс Э.М., Крапивская Е.Е., Родионов А.В. Полиморфные сайты в транскрибируемых спейсерах генов 35S рРНК пионов как индикатор происхождения сортов // Генетика. 2017. Т. 53. № 2. С. 181–191. https://doi.org/10.7868/S0016675817010118
Родионов А.В. Добрякова К.С., Пунина Е.О. Полиморфные сайты в районе ITS1-5.8S рДНК-ITS2 у гибридогенного рода ×Elyhordeum и предполагаемых межвидовых гибридов Elymus (Poaceae: Triticeae) // Генетика. 2018. Т. 54. № 9. С. 999–1014. https://doi.org/10.1134/S0016675818090126
Родионов А.В., Амосова А.В., Беляков Е.А. и др. Генетические последствия межвидовой гибридизации, ее роль в видообразовании и фенотипическом разнообразии растений // Генетика. 2019. Т. 55. № 3. С. 255–272. https://doi.org/10.1134/S0016675819030159
Rodionov A.V., Krainova L., Gnutikov A.A. et al. Intragenomic polymorphism of internal transcribed spacer ITS1 in the locus 35S rRNA of polyploid Avena species // Plant Genet., Genom., Bioinformatics, and Biotechnology (PlantGen 2019). The Fifth Intern. Sci. Conf. Novosibirsk, Russia, 2019. P. 166.
Дополнительные материалы
- скачать Mateikovich.doc
- Приложение. Образцы растений, последовательности trnL-trnFи ITS1-5.8SрДНК-ITS2 которых были секвенированы в данной работе.


