Генетика, 2020, T. 56, № 5, стр. 550-560
Анализ транскриптома линий Drosophila melanogaster с нарушением контроля транспозиции ретротранспозона gypsy
И. В. Кукушкина 1, П. А. Махновский 2, Л. Н. Нефедова 1, *, П. А. Миляева 1, И. В. Кузьмин 1, А. Р. Лавренов 1, А. И. Ким 1, **
1 Московский государственный университет им. М.В. Ломоносова
119234 Москва, Россия
2 Институт медико-биологических проблем Российской академии наук
123007 Москва, Россия
* E-mail: lidia_nefedova@mail.ru
** E-mail: aikim57@mail.ru
Поступила в редакцию 13.05.2019
После доработки 25.06.2019
Принята к публикации 02.07.2019
Аннотация
Для исследования причин нарушения контроля активности мобильных генетических элементов в линиях с фенотипом flamenco SS (w, мутант flamenco) и MS (w, мутант flamenco, активная копия gypsy) проведено секвенирование транскриптомов этих линий. В качестве контроля была использована линия Д32 (лабораторная линия дикого типа). Разработан алгоритм для поиска аминокислотных замен в данных высокопроизводительного секвенирования РНК, использующий для анализа триплетный код. При помощи разработанного алгоритма обнаружено семь нонсенс-мутаций. Методом аллель-специфичной ПЦР подтверждено наличие пяти из семи найденных in silico нонсенс-мутаций. Однако обнаруженные нонсенс-мутации не связаны с фенотипом flamenco. Проведен поиск мутаций в 89 генах системы РНК-интерференции в линиях SS и МS относительно референсного генома BDGP6 и линии дикого типа Д32. Не обнаружено делеций, вставок, нонсенс-кодонов и других нарушений, которые могут однозначно приводить к изменению функции гена. Для выявления генов со специфической экспрессией для линий с фенотипом flamenco сравнивали транскриптомы линий SS и MS c контрольными линиями Д32, OregonR, w1118. Выделен набор из 25 генов с дифференциальной экспрессией, среди которых у двух генов, sosie и CR45822, значительно изменена экспрессия в линиях SS и MS. Оба гена прямо или косвенно вовлечены в оогенез. Таким образом, экспрессия генов sosie и CR45822 может быть использована в качестве маркера фенотипа flamenco в линиях SS и MS.
В геноме Drosophila melanogaster присутствуют разнообразные мобильные генетические элементы (МГЭ) [1], повышенная транспозиционная активность которых приводит к геномной нестабильности [2]. При этом МГЭ вносят значительный вклад в генетическую изменчивость и эволюцию геномов [3–5].
Для регуляции активности МГЭ в клетке существуют механизмы, которые действуют на транскрипционном уровне, через модификацию хроматина и на посттранскрипционном уровне [6]. Для подавления экспрессии МГЭ на посттранскрипционном уровне используется несколько путей РНК-интерференции, на каждом из которых функционируют разные гены и разные источники антисмысловой РНК [7]. В клетках зародышевого пути у самок дрозофилы активность широкого спектра МГЭ подавляется скоординированным действием генов aubergine (aub), argonaute 3 (AGO3) и piwi через образование piРНК из транскриптов локуса 42AB в ходе циклического механизма, имеющего название “пинг-понг” [8]. В фолликулярных клетках яичников цикл “пинг-понг” отсутствует, piРНК образуется в результате процессинга транскриптов локуса flamenco с помощью белкового комплекса Yb–body, затем связывается с белком piwi и выполняет функцию гидовой РНК, необходимой для поиска белком piwi активно транскрибируемых МГЭ в ядре и привлечения к этим МГЭ белков-репрессоров. В соматических клетках активность МГЭ в основном контролируется эндогенными малыми интерферирующими РНК (endo-siРНК), генерируемыми генами Dicer-2 (Dcr-2) и Argonaute 2 (AGO2) [9]. При этом endo-siРНК могут образовываться из тех же геномных локусов, из которых происходят piРНК.
Локус flamenco вовлечен в piwi-зависимый путь РНК-интерференции и контролирует транспозицию ретротранспозона-ретровируса gypsy и других МГЭ [10–13]. Он является источником piРНК в генеративных тканях и endo-siРНК в соматических тканях [8, 14]. Локус flamenco картирован генетически, но молекулярные механизмы его функционирования изучены не полностью. Методом делеционного картирования локус был отнесен к району 20А1-3 Х-хромосомы [10]. В эксперименте по инсерционному мутагенезу с использованием конструкции на основе Р-элемента была получена мутантная линия D. melanogaster с фенотипом flamenco, при этом инсерция Р[yellow] оказалась расположена на расстоянии 1.5 тпн от гена DIP1 [15]. Показано, что инсерция не влияет на транскрипцию гена DIP1, но может влиять на транскрипцию предшественника piРНК, локализующегося дистальнее гена DIP1 в районе 20А4-5 Х-хромосомы. В настоящее время считается, что локус flamencо имеет размер около 180 тпн. Последовательность локуса главным образом состоит из фрагментов транспозонов, большинство из которых относятся к группе ретротранспозонов с длинными концевыми повторами (ДКП) – ДКП-ретротранспозонов [8, 13].
Очевидно, что мутации в генах РНК-интерференции и в локусе flamencо будут приводить к повышению уровня транскрипции МГЭ и частоты их транспозиции. В частности, мутации в гене piwi приводят к стерильности и вызывают повышенную частоту транспозиции МГЭ gypsy [16] и copia [17], а также повышение уровня транскрипции других МГЭ [18]. Локус flamenco – не единственный регулятор сложного процесса транспозиции. Показано, что источником piРНК может быть не один, а несколько кластеров, локализованных на разных хромосомах, и функционирование этих кластеров до конца не изучено [19]. Очевидно, что в понимании функционирования механизма РНК-интерференции остаются пробелы, и не все участники системы РНК-интерференции и подавления активности МГЭ на сегодняшний день идентифицированы.
Проблему представляет также определение статуса flamenco у дрозофил. Фенотип flamenco не имеет внешнего проявления и может быть обнаружен только в специальных экспериментах, измеряющих транспозиционную активность МГЭ gypsy. Это делает затруднительным проведение генетического анализа при исследовании мутантов flamenco. Обнаружение надежных экспрессионных маркеров позволило бы облегчить определение фенотипа flamenco. Отметим, что экспрессия самих МГЭ не всегда является хорошим маркером из-за больших различий в копийности МГЭ у разных линий дрозофилы.
В нашей лаборатории ранее была исследована линия SS D. melanogaster, которая имеет фенотип flamenco. Она не является инсерционным мутантом, и природа мутации flamenco в ней не известна. Из этой линии путем искусственного введения активной копии МГЭ gypsy была получена линия MS, характеризующаяся повышенной частотой транспозиции gypsy [20]. Экспрессия генов системы РНК-интерференции ago3, zuc, aub и генов гетерохроматиновых белков семейства HP1 (hp1a, hp1b, hp1c, hp1e) в линии SS не изменена; при этом наблюдается пониженный уровень экспрессии гена rhino (hp1d), который кодирует белок семейства HP1, функционирующий только в тканях яичников [21].
Настоящая работа посвящена сравнительному анализу транскриптомов линий с фенотипом flamenco относительно линий дикого типа с фенотипом flamenco+, поиску экспрессионных маркеров, позволяющих определять фенотип flamenco, и поиску новых генов-кандидатов, регулирующих транспозицию gypsy.
МАТЕРИАЛЫ И МЕТОДЫ
Линии Drosophila melanogaster. В работе использовали изогенные линии с фенотипом flamenco, w1: SS, MS (содержит активный gypsy) [20] и линию Д32 с фенотипом flamenco+ [22] из коллекции кафедры генетики МГУ. Линии мух культивировали в стандартных условиях: на пищевом субстрате (агар, манная крупа, сахар, изюм, дрожжи) в стеклянных пробирках при температуре 25°С и влажности около 65%. Мух пересаживали на свежий корм в новую пробирку каждые 18 дней.
Выделение РНК и высокопроизводительное секвенирование. Выделение РНК для секвенирования транскриптома осуществляли с помощью комплекта реактивов ExtractРНК (Евроген) из взрослых самок возрастом 7 дней. Концентрацию РНК измеряли с помощью флуориметра Qubit (Thermo Scientific), оценку целостности – с помощью капиллярного электрофореза на приборе Bioanalyzer 2100 (Agilent). Подготовка библиотеки была выполнена с использованием набора TruSeq РНК sample v2 (Illumina). Перед секвенированием концентрация библиотеки была оценена с помощью флуориметра Qubit и ПЦР в реальном времени (праймеры: I-qPCR-1.1 AATGATACGGCGACCACCGAGAT и I-qPCR-2.1 CAAGCAGAAGACGGCATACGA). Библиотеки были разведены до концентрации 11 пМ и секвенированы на приборе Illumina HiSeq 2000 (длина прочтения – 50 оснований). Глубина секвенирования составила для линии MS 5 млн прочтений и для линий Д32, SS 12 млн прочтений на образец. Контроль качества проводили с помощью программы FastQC, для тримминга использовали Trimmomatic v0.32. Доля прочтений с качеством не менее 20 (по системе оценки качества PHRED) составила 98.9–99.5%. Короткие прочтения были картированы на геном D. melanogaster сборки BDGP6 с применением программы Tophat v. 2.1.0 с использованием аннотации референсного генома BDGP6.94. Доля картированных прочтений составила 94–98%. Подсчет прочтений производили с помощью программы HTSeq v. 0.6.1 с использованием референсной аннотации генома D. melanogaster BDGP6 (сборка Ensembl 94). Для оценки экспрессии конкретного гена использовали суммарное количество уникальных прочтений, локализованных на экзонах каждого гена. Дополнительные данные, используемые в качестве контрольных (транскриптомы самок возрастом 4 дня линий OregonR и w1118, глубина секвенирования – 11–17 млн прочтений на образец), получены из серии эксперимента GSE99574 базы данных GEO (https:// www.ncbi.nlm.nih.gov/geo) и процессированы согласно описанному выше протоколу.
Оценка дифференциальной экспрессии генов и поиск мутаций в секвенированных геномах in silico. Дифференциальную экспрессию генов оценивали методом DESeq. Транскриптом линии SS сравнивали попарно с транскриптомами каждой из контрольных линий (Д32, OregonR, w1118) и получили набор генов, которые имели значимые различия в каждом из трех случаев. Дифференциальную экспрессию для отдельных генов подтверждали с помощью количественной ПЦР.
Для поиска мутаций в линиях Д32, SS, MS относительно референсного генома использовали программы для анализа секвенированного транскриптома: Ensemble VEP и R VariantAnnotation.
Выделение ДНК и РНК. Выделение ДНК производили на особях одного пола согласно стандартному протоколу [23]. Выделение РНК проводили с помощью реактива ExtractRNA (Евроген) по протоколу, рекомендованному фирмой-производителем. Концентрацию РНК измеряли спектрофотометрически на приборе NanoDrop (Peqlab).
Обратная транскрипция (ОТ). Перед постановкой реакции ОТ образцы РНК обрабатывали ДНКазой I (Fermentas) согласно протоколу фирмы. Для ОТ использовали набор MMLV-RTKit (Евроген).
Полимеразная цепная реакция (ПЦР) в режиме реального времени. Амплификацию проводили в амплификаторе Mini-Opticon Real-Time-PCR System (Bio-Rad Laboratories) с использованием реакционной смеси qPCRmix-HS SYBR (Евроген), содержащей флуоресцентный краситель SYBR Green I. Параметры цикла: денатурация – 95°С, 15 с, отжиг праймеров – 55°С, 45 с, элонгация – 72°С, 60 с (40 циклов). Эффективность ПЦР оценивали с помощью серии кратных разведений ДНК или кДНК. Анализ результатов ПЦР проводили с помощью пакета программ Bio-Rad CFX Manager (версия 1.6.541.1028). Вычисление экспрессии gypsy данным программным обеспечением производили ∆∆С(t) методом. Оценивали относительный уровень экспрессии генов sosie и CR45822. Нормализацию экспрессии проводили на уровень референсных генов αTub84D, RpL40, EloB, eIF-1A. Последовательности праймеров приведены в табл. 1.
Таблица 1.
Последовательности праймеров, использованных в работе
| Ген | Прямой праймер (5'–3') | Обратный праймер (5'–3') |
|---|---|---|
| Для ОТ-ПЦР | ||
| Sosie | CCAGTACGAGAACAGCAAC | TCCTCCACATATCCGTTGTC |
| CR45822 | GTTCCGTGGTCAGTACAGC | GTCGAGCAAGTGATACAAATG |
| αTub84D | GTGCATGTTGTCCAACACCAC | AGAACTCTCCCTCCTCCATA |
| RpL40 | CTGCGTGGTGGTATCATTG | CAGGTTGTTGGTGTGTCC |
| EloB | GCACAAACATACACACTCACG | TTTCCTACTTCGCTTGCACC |
| eIF-1A | TCGTCTGGAGGCAATGTG | GAGTCCTGGTAGTCACGC |
| Для аллель-специфичной ПЦР | ||
| CG2121 | CAGATGGCCCAGGTGTG(G/А)* | CCACCCTGTTTCTCTCCAG |
| Cyp9b1 | CAGTTAGCATTAATACATCAGGTG | CCTTAAAGGTGCCAGTACT(C/Т)* |
| dgt1 | CAGTCCACCAGCTTGATTAG | CGAGGGCTTAAAAATTAGTTT(T/А)* |
| GstE5 | CGACTGCACTTCGAGACA(G/Т)* | CCAGCAATGTAATCATGTCC |
| CG31918 | TGCGACTGCTCACCTAAAG | AGCATCTGCCAGAAAGTC(G/Т)* |
| List | TTCTTCCTCATGCTATTCGTC | CCGAGAAGCCTAAAACAG(T/С)* |
| CG33346 | CAACAATGGGTGGGAAAC | ACACATTTTCGGTATTATCC(C/Т)* |
Для подтверждения однонуклеотидного полиморфизма в исследуемых линиях использовали аллель-специфичную ПЦР. Для амплификации образцов ДНК использовали праймеры к референсному геному и два праймера к полиморфному сайту гена (различаются нуклеотидом на 3'-конце). Наличие того или иного аллеля устанавливали по различию в Ct между результатами реакций с альтернативными парами праймеров для каждого образца отдельно. Для амплификации использовали праймеры, приведенные в табл. 1.
РЕЗУЛЬТАТЫ
Поиск мутаций в транскриптомах линий D. melanogaster с фенотипом flamenco
Поиск мутаций осуществляли с помощью программ Ensemble VEP и R VariantAnnotation. Алгоритмы представленных программ находят отдельные замены одного нуклеотида на другой, не анализируя соседние замены. Такой подход приводит к обнаружению большого количества нонсенс-мутаций, которые при дальнейшей проверке не подтверждаются. Проблема связана с тем, что замена одного нуклеотида может происходить вместе с соседним, тем самым не приводя к нонсенс-мутации, например замена триплета TCG на ТАТ распознается в этих программах как нонсенс-мутация, так как триплет ТАG является стоп-кодоном. Для оптимизации работы нами был разработан алгоритм для поиска аминокислотных замен и была создана его реализация на языке Scala (рис. 1). Разработанный алгоритм позволяет производить анализ замен, используя триплетный код; отбирать замены с одной группы аминокислот на другую (включая нонсенс-мутации); проводить фильтрацию результатов по представленности замены среди всех последовательностей прочтений, по количеству прочтений с заменами и общему количеству прочтений; оценивать наличие одинаковых замен в различных геномах. В качестве параметров для анализа было выбрано: минимальное число прочтений — не менее двух, минимальный процент замен относительно референсного генома — не менее 75% для каждого прочтения, пороговое значение для сравнения однонуклеотидных вариантов полиморфизма в двух линиях — не менее 30% прочтений с заменами.
Рис. 1.
Блок-схема обработки данных секвенирования транскриптома. Пункт “фильтрация по количеству прочтений” позволяет отсортировать данные с минимальным количеством прочтений, например от двух, или по вероятности данной замены среди представленных прочтений. При этом в данном блоке можно анализировать как нуклеотидные, так и аминокислотные замены. “Сопоставление двух геномов” позволяет выявить одинаковые или различные аминокислотные замены в двух и более различных геномах. “Отбор по типу замен” позволяет отобрать замены по заменяемой или заменяющей аминокислоте, включая стоп-кодон.
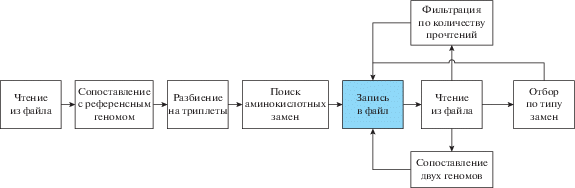
При расчетах нами учтено, что средняя частота ошибок при высокопроизводительном секвенировании составляет 0.5% [24]. Это увеличивает возможность появления ложных результатов в данных, содержащих только одно прочтение. Кроме того, необходимо учитывать возможность гетерозиготности особей по определенным генам. Очевидно, что чем больше прочтений для гена и чем больше одинаковых замен в этих прочтениях, тем более достоверным (подтверждаемым) будет наличие данной замены в исследуемой линии. Необходимый минимум – два прочтения с одинаковой нуклеотидной заменой.
С помощью описанного алгоритма было проведено сравнение экзомов линий с фенотипом flamenco (SS и MS) и линии с фенотипом flamenco+ (Д32) относительно референсного генома BDGP6 (предположительно с фенотипом flamenco+). В результате обнаружено более 15 000 замен. Меньше половины из этих замен (более 7000) значащие, т.е. приводят к замене одной аминокислоты на другую (табл. 2). Наименьшее количество замен было обнаружено в экзоме линии MS, что объясняется низким покрытием секвенирования.
При сравнении аминокислотных замен между исследуемыми линиями обнаружено, что линии MS и SS имеют более 96% одинаковых замен относительно генома линии BDGP6, что объясняется близким родством мутантных линий; линии SS и Д32 имеют около 35% одинаковых замен относительно референсного генома BDGP6 (рис. 2). Это соответствует ранее полученным данным по полиморфизму в лабораторных линиях D. melanogaster (от 33 до 63 процентов по STS-последовательностям) [25].
Рис. 2.
Диаграмма Эйлера, отражающая множества значащих замен (включая нонсенс-мутации) для линий Д32 и SS, SS и MS относительно референсного генома BDGP6.

Таким образом, для того чтобы выявить генетические различия между линиями flamenco+ и flamenco, следовало бы проверить функциональную значимость около 5000 замен, по которым различаются эти линии. Однако следует учитывать, что замена одной аминокислоты на другую может не приводить к изменению биохимических свойств белка. Определение функциональной значимости замены возможно, если доказано ее влияние на функцию белка. Для большинства белков такие данные отсутствуют, а математическое моделирование не позволяет точно предсказать влияние замены на структурные и ферментативные особенности белков. Поэтому в данной работе было принято решение провести поиск мутаций, приводящих к возникновению стоп-кодона, что однозначно должно повлиять на структуру и свойства белка. Результат поиска замен, приводящих к образованию нонсенс-мутаций, в каждой исследуемой линии мух приведен в табл. 3.
Таблица 3.
Общее число замен, приводящих к замене аминокислоты на стоп-кодон, в линиях Д32, SS и MS относительно референсного генома BDGP6
| Хромосома | Д32 | SS | MS |
|---|---|---|---|
| X | 2 | 1 | 1 |
| Y | 0 | 0 | 0 |
| 2 | 3 | 8 | 5 |
| 3 | 4 | 3 | 3 |
Обнаружено семь генов, содержащих в кодирующей последовательности стоп-кодоны: cyp9b1, List, CG2121, CG33346, CG31918, GstE5, dgt1. Три однонуклеотидные замены, приводящие к появлению нонсенс-мутации, были выявлены в линии Д32 относительно линий SS, MS и референсного генома BDGP6. Эти мутации локализованы в генах dgt1, GstE5 и CG31918. Две замены были найдены в линии SS (в генах CG2121 и Cyp9b1), но для линии MS данные замены нельзя было проверить в связи с отсутствием прочтений этих генов в экзоме линии MS. Для генов List и CG33346 нонсенс-мутации были найдены в линиях Д32, SS и MS. Соответственно, мутации в данных генах присутствуют как в линиях с фенотипом flamenco+, так и в линиях с фенотипом flamenco.
Нонсенс-мутации, обнаруженные при анализе данных, полученных при секвенировании транскриптома, в дальнейшем верифицировали с помощью аллель-специфичной ПЦР. В результате нам удалось подтвердить данные о наличии пяти из семи мутаций, взятых в анализ: три замены в линии Д32 (в генах dgt1, GstE5, CG31918), одна замена в линии SS в гене Cyp9b1 и одна замена в гене CG33346 в линиях Д32 и SS. Наличие мутации в гене CG2121 не подтвердилось. Это можно объяснить тем, что в исследуемом сиквенсе для данного гена было низкое покрытие (всего два прочтения). Наличие однонуклеотидной замены в гене List выявлено в гомозиготном состоянии для линии Д32, в линиях SS и MS мутантный аллель присутствовал в гетерозиготе наряду с аллелем дикого типа. Однонуклеотидных замен в генах cyp9b1, CG2121, CG13101, CG31918, GstE5 методом аллель-специфичной ПЦР в линии MS обнаружено не было.
Таким образом, нонсенс-мутации, которые были обнаружены в экзомах линий MS и SS, не имеют отношения к фенотипу flamenco, так как мутация в гене Cyp9b1 отсутствует у линии MS, а мутация в гене CG33346 есть также у линии дикого типа Д32.
Далее мы провели поиск мутаций в экзомах секвенированных нами линий flamenco, которые могли бы повлиять на функционирование системы РНК-интерференции. В предыдущих исследованиях было показано, что в линиях SS и MS с фенотипом flamenco транскрипция основных генов-участников системы РНК-интерференции не изменена [21]. Тем не менее это не исключало возможности нарушения экспрессии генов на уровне белка. В анализ взяли 89 генов, участвующих в РНК-интерференции [26, 27].
Наличие замен в геномах линий SS и MS сопоставляли с линиями-контролями Д32, CantonS, w1118, OregonR, имеющими фенотип flamenco+. В результате обнаружено 215 несинонимичных замен в рассматриваемых генах, из которых хотя бы в одном контроле обнаруживалось 156. Уникальные для линий MS и SS 59 замен сопоставили с двумя базами данных полиморфизма DGRP2 (~200 линий) и Ensembl. 34 замены не были обнаружены в базах данных, однако 29 из них не подходят по соотношению MM/DP (доля замен на позиции). Таким образом, только пять неизвестных вариантов полиморфизма, характерных и для линии SS, и для линии MS, обнаружены в генах системы РНК-интерференции. Это замены в следующих генах: piwi (A>V; 2L 10986085), tud (A>T, 2R 21191137), CG7504 (M>I, 3L 8135663), vret (С>Y, 3R 22744567) и Nmnat (G>V, 3R 24945787). Мы предполагаем, что аминокислотные замены, обнаруженные в генах piwi (полярная > полярная) и CG7504 (полярная > полярная), не влияют на функцию этих генов, так как являются консервативными и не затрагивают функционально значимых доменов белка. Обнаруженные замены в генах vret (неполярная > полярная) и tud (неполярная > полярная) являются радикальными, но не затрагивают функционально значимых доменов. Замена в гене Nmnat (неполярная > неполярная) является консервативной, хотя и попадает в функционально значимый домен – Cytidyltransferase-like domain (IPR004821). Выяснение роли всех этих мутаций в формировании фенотипа flamenco – задача дальнейших исследований.
Сравнительный анализ транскриптомов линий D. melanogaster с фенотипом flamenco+ и flamenco
Для выявления генов со специфической экспрессией для линий с фенотипом flamenco сравнивали транскриптомы линий SS и MS и контрольных линий Д32 и OregonR, w1118, единственно доступных в настоящее время в базе данных GEO (https:// www.ncbi.nlm.nih.gov/geo). Выделен набор из 25 генов с дифференциальной экспрессией с высоким уровнем значимости padj, среди которых 15 имеют сниженную экспрессию по сравнению с контрольными линиями, а 10 – повышенную (табл. 4).
Таблица 4.
Дифференциальная экспрессия белок-кодирующих генов в линии SS относительно линий Д32, w1118, OregonR
| Ген | SS относительно oreR | SS относительно w1118 | SS относительно Д32 | |||
|---|---|---|---|---|---|---|
| log2FoldChange | padj | log2FoldChange | padj | log2FoldChange | padj | |
| CG43348 | –7.0 | 7.05E-07 | –5.7 | 0.0150 | –7.8 | 6.29E-17 |
| CG4572 | –3.8 | 2.39E-28 | –3.5 | 7.66E-37 | –3.6 | 1.51E-13 |
| Ser8 | –3.5 | 3.10E-09 | –2.6 | 3.33E-09 | –3.6 | 5.34E-08 |
| Sosie | –3.4 | 3.40E-05 | –3.7 | 0.000471 | –3.6 | 5.03E-10 |
| Cyp4p1 | –3.1 | 5.19E-12 | –3.2 | 3.73E-12 | –3.6 | 1.18E-11 |
| CG6839 | –4.1 | 0.00588 | –2.9 | 0.0262 | –2.6 | 2.45E-07 |
| CG2233 | –1.5 | 0.000290 | –1.6 | 9.67E-15 | –2.3 | 5.27E-09 |
| CG5011 | –1.6 | 0.0262 | –2.3 | 6.18E-06 | –2.0 | 0.00144 |
| CG3819 | –3.8 | 5.77E-06 | –2.6 | 0.00313 | –1.6 | 0.00173 |
| Pst | 2.0 | 2.86E-19 | 0.9 | 3.12E-06 | 1.6 | 0.000210 |
| Moody | 1.6 | 2.15E-07 | 1.3 | 4.00E-07 | 1.6 | 0.00914 |
| CG17752 | 0.9 | 0.0184 | 1.1 | 2.85E-05 | 1.9 | 0.000873 |
| CG40472 | 6.5 | 2.27E-20 | Не определен | 6.36E-32 | 2.6 | 0.0288 |
| CG5773 | 1.5 | 0.0160 | 0.6 | 0.0435 | 3.7 | 7.45E-10 |
| CG17147 | 6.5 | 7.87E-13 | 4.6 | 2.58E-11 | 4.1 | 0.00857 |
| CG10513 | 3.3 | 1.04E-37 | 1.5 | 1.95E-08 | 6.7 | 1.72E-34 |
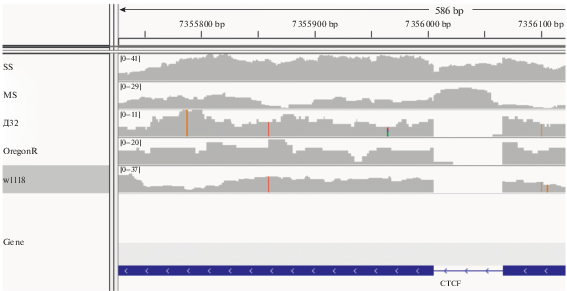
Среди оставшихся экспрессирующихся последовательностей обнаружены различия в экспрессии РНК, соответствующей 2-му интрону гена CTCF. Экспрессия значительно увеличена у линий SS и MS по сравнению с контрольными линиями (рис. 3). По всей видимости, это результат увеличения экспрессии некодирующей РНК CR45822 (antisense РНК:CR45822), которая тран-скрибируется с последовательности гена CTCF в антисмысловом направлении.
Рис. 3.
Оверэкспрессия гена CR45822 у линий MS и SS. Приведено покрытие области 2-го интрона гена CTCF у секвенированных транскриптомов самок различных линий (представление данных в программе IGV).
В качестве маркеров фенотипа flamenco нас в первую очередь заинтересовали гены sosie и CR45822. Выбор гена sosie обусловлен тем, что он демонстрирует достаточно большие различия в экспрессии относительно всех контрольных линий, при этом у него известна функция, в отличие, например, от гена CG43348. Ген CR45822, несмотря на отсутствие известной функции, интересен тем, что практически не экспрессируется у дрозофил дикого типа. Столь значительные различия в его экспрессии должны позволить надежно отличать фенотипы flamenco+ и flamenco.
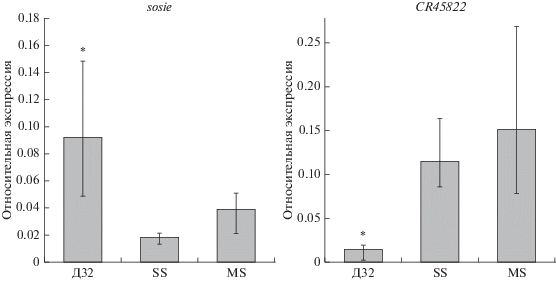
Обнаруженные различия в экспрессии генов sosie и CR45822 мы подтвердили методом количественной ПЦР (рис. 4). Следовало предположить, что повышенная транскрипция антисмысловой РНК CR45822 должна повлиять на транскрипцию гена CTCF, однако никаких различий в его экспрессии у дрозофил с разным статусом flamenco мы не выявили.
Рис. 4.
Экспрессия генов sosie и CR45822 у самок линий SS, MS и контрольной линии Д32. Планки погрешностей иллюстрируют крайние значения выборки (размах значений). * – значимые различия между выборками индивидуальных самок линии Д32 и линиями с фенотипом flamenco, тест Манна–Уитни (p-value < 0.01, N = 6).
Таким образом, экспрессия генов sosie и CR45822 может быть использована в качестве маркера фенотипа flamenco в линиях SS и MS.
ОБСУЖДЕНИЕ
Известно, что у D. melanogaster перемещения ретротранспозонов группы gypsy контролируются локусом flamenco, выступающим в качестве источника малых РНК (piРНК), необходимых для процесса РНК-интерференции [10–13]. Выделены линии, характеризующиеся повышенной активностью некоторых групп МГЭ, в частности ретротранспозонов группы gypsy; к таким линиям относятся и исследуемые в настоящей работе линии SS и MS [28]. Чаще всего повышение активности МГЭ вызвано нарушением работы генов, задействованных в системе РНК-интерференции, в том числе генов piwi и flamenco [16–18]. Однако детальные механизмы работы системы подавления активности и их взаимодействие изучены недостаточно. Так, подавление транспозиции может происходить по Dicer-зависимому и Dicer-независимому пути [9]; механизмы действия системы РНК-интерференции и набор белков, в ней участвующих, различаются в соматических и генеративных тканях [8, 14]; локус flamenco не является единственным источником piРНК [29]; строение локусов, продуцирующих piРНК, может различаться, и механизм образования таких локусов неизвестен.
В настоящее время считается, что предшественники piРНК появляются из единого транскрипта. Однако данная модель в некоторой степени спорная, так как внутри локуса flamenco картированы пики плотности piРНК, природа которых пока еще не выяснена [30]. Транскрипция локуса flamenco происходит в антисмысловом направлении по отношению к транскрипции ретротранспозонов. Этот первичный транскрипт подвергается альтернативному сплайсингу, проходит модификацию 5'-конца и процессинг 3'-конца. Однако выявлены не все участники этих процессов [31]. Кроме того, источниками piРНК у D. melanogaster являются три кластера piРНК: 42AB, trafficjam и flamenco. Flamenco является более изученным, чем остальные два. Считается, что именно этот локус является источником piРНК для препятствия транспозиции gypsy [32]. Однако выявлены только некоторые ретроэлементы, которые находятся под контролем кластера flamenco. Контроль других МГЭ этим локусом остается под вопросом.
В настоящей работе было проведено сравнительное исследование транскриптомов линий с фенотипами flamenco и flamenco+. Особое внимание мы уделяли поиску генов, экспрессия которых изменена в линиях flamenco. Поскольку мы проводили анализ полиаденилированных РНК, остальные РНК остались вне поля зрения. В частности, не были секвенированы piРНК и endo-siРНК, источником которых является локус flamencо. Однако изменение транскрипционной активности локуса flamenco и других локусов, генерирующих piРНК, свидетельствует лишь о нарушении активности белков, отвечающих за транскрипцию и процессинг РНК-предшественников. В результате проведенной работы нами не обнаружено делеций, вставок, нонсенс-кодонов и других нарушений, которые могут однозначно приводить к функциональному изменению генов в линиях с фенотипом flamenco. Кроме того, нам не удалось обнаружить функционально значимые мутации в генах, относящихся к системе РНК-интерференции. Тем не менее мы обнаружили 25 генов с дифференциальной экспрессией, среди которых отметили два перспективных маркера фенотипа flamenco – гены sosie и CR45822.
Ген sosie кодирует небольшой белок размером 186 аминокислот и две изоформы размером 74 аминокислоты. Sosie функционирует в фолликулярных клетках яичников, играет важную роль в оогенезе и участвует в формировании яйцевой камеры и миграции фолликулярных клеток яичников. Показано, что ген координирует клеточную миграцию во взаимодействии с βH-спектрином (ген kst), филамином (ген cher) и профилином (ген chik) [33]. По-видимому, ген sosie напрямую не связан с процессом регуляции транспозиции мобильных элементов в тканях яичников. Однако снижение его экспрессии в линиях flamenco коррелирует с нарушением работы системы, регулирующей транспозиции МГЭ. Ранее нами было показано, что в линиях flamenco снижен уровень транскрипции гена rhino [21], кодирующего белок, который является одним из важнейших компонентов системы подавления активности МГЭ в яичниках и направляет транскрипцию piРНК с двуцепочечного кластера 42АВ [34, 35].
Что касается гена антисмысловой РНК CR45822, про него практически ничего неизвестно. Согласно поиску BLAST, в геномах близких видов дрозофилы (D. sechellia, D. simulans, D. yakuba, D. erecta) присутствуют гомологичные нуклеотидные последовательности, идентичность доходит до 90% (данные не приведены). Это свидетельствует о стабилизирующем отборе, направленном на сохранение последовательности. Возможно, CR45822 осуществляет посттранcкрипционную регуляцию гена CTCF. Однако никаких изменений в транскрипции генов в линиях с разным статусом flamenco мы не обнаружили. Не исключено, что регуляция может осуществляться тканеспецифично, зависеть от стадии развития или возраста. Известно, что продукт гена CTCF – регулятор транскрипции, напрямую взаимодействует с продуктом гена mod(mdg4) [36]. Продукт гена mod(mdg4) вовлечен в оогенез и миграцию герминальных клеток, а также является компонентом инсуляторного комплекса, взаимодействующего с 5'-нетраслируемой областью МГЭ gypsy [37]. Не исключено, что активация транскрипции гена CR45822 в линиях SS и MS является следствием нарушения контроля активности gypsy.
Таким образом, в линиях SS и MS не обнаружены новые гены, мутации в которых могли бы привести к формированию фенотипа flamenco. Однако выявлены два гена: sosie и CR45822, уровни экспрессии которых могут использоваться в качестве фенотипических маркеров нестабильности генома, вызванной транспозицией МГЭ gypsy. Наличие таких маркеров существенно облегчит работу по картированию мутаций, приводящих к фенотипу flamenco в других линиях. Кроме того, оба гена – sosie и CR45822 прямо или косвенно вовлечены в оогенез, и изменение их экспрессии может оказывать влияние на процессы контроля транспозиции МГЭ в яичниках. Дальнейшие исследования будут направлены на выяснение причин изменения экспрессии генов sosie и CR45822 в линиях с фенотипом flamenco, что может способствовать пониманию механизмов контроля транскрипции со стороны piwi-зависимой РНК-интерференции. Не исключено, что изменение экспрессии этих генов обусловлено эпигенетическими причинами, что в настоящее время находится в стадии изучения.
Работа поддержана грантом РФФИ 17-04-01250 А.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Arkhipova I.R., Lyubomirskaya N.V., Ilyin Y.V. Drosophila retrotransposons // Molecular Biology Intelligence Unit. L.: R.G. Landes, 1995. 148 p.
Green M.M. Mobile DNA elements and spontaneous gene mutation // Eukaryotic Transposable Elements as Mutagenic Agents. Banbury Rep., 1988. P. 41–51.
Fedoroff N.V. Transposable elements, epigenetics, and genome evolution // Science. 2012. V. 338. № 6108. P. 758–767. https://doi.org/10.1126/science.338.6108.758
Warren I.A., Naville M., Chalopin D. et al. Evolutionary impact of transposable elements on genomic diversity and lineage-specific innovation in vertebrates // Chromosome Res. 2015. V. 23. № 3. P. 505–531. https://doi.org/10.1007/s10577-015-9493-5
Elbarbary R.A., Lucas B.A., Maquat L.E. Retrotransposons as regulators of gene expression // Science. 2016. V. 351. № 6274. P. aac7247. https://doi.org/10.1126/science.aac7247
Buchon N., Vaury C. RNAi: a defensive RNA-silencing against viruses and transposable elements // Heredity. 2006. V. 96. № 2. P. 195–202. https://doi.org/10.1038/sj.hdy.6800789
Lee Y.S., Nakahara K., Pham J.W. et al. Distinct roles for Drosophila Dicer-1 and Dicer-2 in the siPHK/miPHK silencing pathways // Cell. 2004. V. 117. P. 69–81. https://doi.org/10.1016/S0092-8674(04)00261-2
Brennecke J., Aravin A.A., Stark A. et al. Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila // Cell. 2007. V. 128. № 6. P. 1089–1103. https://doi.org/10.1016/j.cell.2007.01.043
Czech B., Malone C.D., Zhou R. et al. An endogenous small interfering RNA pathway in Drosophila // Nature. 2008. V. 453. P. 798–802. https://doi.org/10.1038/nature07007
Pelisson A., Song S.U., Prud’homme N. et al. Gypsy transposition correlates with the production of a retroviral envelope-like protein under the tissue-specific control of the Drosophila flamenco gene // The EMBO J. 1994. V. 13. № 18. P. 4401–4411.
Desset S., Conte C., Dimitri P. et al. Mobilization of two retroelements, ZAM and Idefix, in a novel unstable line of Drosophila melanogaster // Mol. Biol. Evol. 1999. V. 16. № 1. P. 54–66.
Desset S., Meignin C., Dastugue B., Vaury C. COM, a heterochromatic locus governing the control of independent endogenous retroviruses from Drosophila melanogaster // Genetics. 2003. V. 164. № 2. P. 501–509.
Mevel-Ninio M., Pelisson A., Kinder A.R. et al. The flamenco locus controls the gypsy and ZAM retroviruses and is required for Drosophila oogenesis // Genetics. 2007. V. 175. № 4. P. 1615–1624. https://doi.org/10.1534/genetics.106.068106
Ghildiyal M., Seitz H., Horwich M.D. et al. Endogenous siRNAs derived from transposons and mRNAs in Drosophila somatic cells // Science. 2008. V. 320. № 5879. P. 1077–1081. https://doi.org/10.1126/science.1157396
Robert V., Prud’homme N., Kim A. et al. Characterization of the flamenco region of the Drosophila melanogaster genom // Genetics. 2001. V. 158. № 2. P. 701–713.
Sarot E., Payen-Groschêne G., Bucheton A., Pélisson A. Evidence for a piwi-dependent PHK silencing of the gypsy endo-genous retrovirus by the Drosophila melanogaster flamenco gene // Genetics. 2004. V. 166. № 3. P. 1313–1321. https://doi.org/10.1534/genetics.166.3.1313
Kalmykova A.I., Klenov M.S., Gvozdev V.A. Argonaute protein PIWI controls mobilization of retrotransposons in the Drosophila male germline // Nucl. Acids Res. 2005. V. 33. № 6. P. 2052–2059. https://doi.org/10.1093/nar/gki323
Lu J., Clark A.G. Population dynamics of PIWI-interacting PHKs (piPHKs) and their targets in Drosophila // Genome Res. 2010. V. 20. № 2. P. 212–227. https://doi.org/10.1101/gr.095406.109
Yamanaka S., Siomi M.C., Siomi H. piRNA clusters and open chromatin structure // Mobile DNA. 2014. V. 5. № 1. P. 22. https://doi.org/10.1186/1759-8753-5-22
Kim A.I., Lyubomirskaya N.V., Belyaeva E.S. et al. The introduction of a transpositionally active copy of retrotransposon gypsy into the stable strain of Drosophila melanogaster causes genetic instability // Mol. Gen. Genet. 1994. V. 242. № 4. P. 472–477.
Лавренов А.Р., Нефедова Л.Н., Романова Н.И., Ким А.И. Экспрессия генов семейства hp1 и их возможная роль в формировании фенотипа flamenco у D. melanogaster // Биохимия. 2014. Т. 79. № 11. С. 1554–1560.
Разорёнова О.В., Карпова Н.Н., Смирнова Ю.Б. и др. Межлинейное распределение и особенности структуры двух подсемейств ретротранспозона Drosophila melanogaster МДГ4 (gypsy) // Генетика. 2001. Т. 37. № 2. С. 175–182.
Bender W., Spierer P., Hogness D.S., Chambon P. Chromosome walking and jumping to isolate DNA from the Ace and rosy loci and the bithorax complex in Drosophila melanogaster // J. Mol. Biol. 1983. V. 168. № 1. P. 17–33.
Mardis E.R. Next-generation sequencing platforms // Annu. Rev. Anal. Chem. 2013. V. 6. P. 287–303. https://doi.org/10.1146/annurev-anchem-062012-092628
Hoskins R.A., Phan A.C., Naeemuddin M. et al. Single nucleotide polymorphism markers for genetic mapping in Drosophila melanogaster // Genome Res. 2001. V. 11. № 6. C. 1100–1113. https://doi.org/10.1101/gr.178001
Tóth K.F., Pezic D., Stuwe E., Webster A. The piRNA pathway guards the germline genome against transposable elements // Non-Coding RNA and the Reproductive System. Dordrecht: Springer, 2016. P. 51–77. https://doi.org/10.1007/978-94-017-7417-8_4
Czech B., Preall J.B., McGinn J., Hannon G.J. A transcriptome-wide RNAi screen in the Drosophila ovary reveals factors of the germline piRNA pathway // Mol. Cell. 2013. V. 50. № 5. P. 749–761. https://doi.org/10.1016/j.molcel.2013.04.007
Урусов Ф.А., Нефедова Л.Н., Лавренов А.Р. и др. Генетический и молекулярный анализ комплементации локусов flamenco и piwi у Drosophila melanogaster // Вавиловский журн. генетики и селекции. 2014. Т. 17. № 3. С. 381–389.
Brennecke J., Aravin A.A., Stark A. et al. Discrete small RNA-generating loci as master regulators of transposon activity in Drosophila // Cell. 2007. V. 128. № 6. P. 1089–1103. https://doi.org/10.1016/j.cell.2007.01.043
Aravin A.A., Hannon G.J., Brennecke J. The Piwi-piRNA pathway provides an adaptive defense in the transposon arms race // Science. 2007. V. 318. № 5851. P. 761–764. https://doi.org/10.1126/science.1146484
Ishizu H., Siomi H., Siomi M.C. Biology of PIWI-interacting RNAs: new insights into biogenesis and function inside and outside of germlines // Genes & Development. 2012. V. 26. № 21. P. 2361–2373. https://doi.org/10.1101/gad.203786.112
Siomi M.C., Sato K., Pezic D., Aravin A.A. PIWI-interacting small RNAs: the vanguard of genome defence // Nature Rev. Mol. Cell Biol. 2011. V. 12. № 4. P. 246. https://doi.org/10.1038/nrm3089
Urwyler O., Cortinas-Elizondo F., Suter B. Drosophila sosie functions with β(H)-Spectrin and actin organizers in cell migration, epithelial morphogenesis and cortical stability // Biol. Open. 2012. V. 1. № 10. P. 994–1005. https://doi.org/10.1242/bio.20122154
Klattenhoff C., Xi H., Li C. et al. The Drosophila HP1 homolog Rhino is required for transposon silencing and piRNA production by dual-strand clusters // Cell. 2009. V. 138. № 6. P. 1137–1149. https://doi.org/10.1016/j.cell.2009.07.014
Mohn F., Sienski G., Handler D., Brennecke J. The rhino-deadlock-cutoff complex licenses noncanonical transcription of dual-strand piRNA clusters in Drosophila // Cell. 2014. V. 157. № 6. P. 1364–1379. https://doi.org/10.1016/j.cell.2014.04.031
Savitsky M., Kim M., Kravchuk O., Schwartz Y.B. Distinct roles of chromatin insulator proteins in control of the Drosophila bithorax complex // Genetics. 2016. V. 202. № 2. P. 601–617. https://doi.org/10.1534/genetics.115.179309
Gdula D.A., Gerasimova T.I., Corces V.G. Genetic and molecular analysis of the gypsy chromatin insulator of Drosophila // Proc. Natl Acad. Sci. USA. 1996. V. 93. № 18. P. 9378–9383.
Дополнительные материалы отсутствуют.


