Генетика, 2020, T. 56, № 6, стр. 726-731
Внутривидовая структура Rhododendron camtschaticum Pall. на полуострове Камчатка: генетический аспект
М. А. Полежаева 1, *, М. В. Модоров 1, А. Н. Полежаев 2, Е. А. Марчук 3
1 Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
2 Институт биологических проблем Севера Дальневосточного отделения
Российской академии наук
685000 Магадан, Россия
3 Ботанический сад-институт Дальневосточного отделения
Российской академии наук
690024 Владивосток, Россия
* E-mail: polezhaevam@mail.ru
Поступила в редакцию 10.06.2019
После доработки 04.07.2019
Принята к публикации 03.10.2019
Аннотация
Проведен анализ изменчивости трех межгенных спейсеров хлоропластной ДНК у 47 образцов Rhododendron camtschaticum на территории п-ова Камчатка и Командорских островов. Анализ молекулярной дисперсии (AMOVA) показал, что около 99% генетического разнообразия приходится на межпопуляционные различия (FST = 0.99, p < 0.001). Выявлено две генетических линии, вероятно обусловленных северным и южным миграционными потоками. Образцы из Центральной и Северной Камчатки образуют отдельный кластер и демонстрируют высокую гомогенность, что поддерживает таксономическую самостоятельность подвида R. camtschaticum ssp. glandulosum. В выборках из южной части полуострова выявлен полиморфизм, вместе с образцами с Командорских островов они соответствуют подвиду R. camtschaticum ssp. camtschaticum.
Изучение близкородственных групп растений – одна из задач систематики и эволюционной биологии. Относительно дальневосточных рододендронов существует ряд вопросов о таксономическом статусе некоторых видов. Среди них рододендрон камчатский (Rhododendron camtschaticum Pall., сем. Ericaceae). Ареал вида охватывает приокеанические области Дальнего Востока: Сахалин, Курильские, Командорские острова, Камчатку, Чукотку, побережье Охотского моря. За пределами России – Северную Японию, Алеутские острова и Аляску.
Рододендрон камчатский – листопадный кустарник 5–20 (реже 50) см высотой. Листья около 2 см длиной, обратнояйцевидные, по краю бахромчато-реснитчатые, собраны в розетки у основания плодущих побегов; на стерильных побегах расположены равномерно. Цветки ярко-розовые от 3 до 5 см в диаметре, по 1–3 на цветоносах 4–10 см высотой. Вид распространен в высокогорном альпийском и субальпийском поясах, чаще на гольцах, также спускается в верхние части лесного пояса. Типовой образец описан с п-ва Камчатка [1, 2].
Отечественные ботаники выделяют две разновидности R. camtschaticum: var. pallasianum (=ssp. camtschaticum) и var. pumilum (=ssp. glandulosum), описанные Е.А. Буш и В.Л. Комаровым [1, 3–6]. В иностранной литературе они указываются в качестве самостоятельных видов – R. camtschaticum Small и R. glandulosum Standl. ex Small [7–9]. Спорная ситуация отчасти объясняется тем, что на территории Аляски виды произрастают аллопатрично, в то время как на территории России – симпатрично (в основном на п-ве Камчатка). Считается, что на российском Дальнем Востоке географическое распространение разновидностей не имеет резкого разграничения, и R. camtschaticum ssp. glandulosum является экологической формой характерной для более сурового климата [1]. Отсутствуют упоминания областей с совместным обитанием двух подвидов и указания о возможной гибридизации.
В качестве таксономически значимых признаков в разграничении вариаций вида традиционно используют длину листа, отношение длины долей венчика к длине всего венчика; наличие/отсутствие опушения венчика, наличие простых/железистых волосков по краю листовой пластинки у ssp. camtschaticum и ssp. glandulosum соответственно [1, 7, 9]. Однако эти признаки значительно варьируют [9]. Также ssp. glandulosum характеризуется меньшими размерами растения (до 10 см) и наличием длинных столоновидных подземных побегов [3].
В последнее время для уточнения таксономии и биогеографии видов успешно используются молекулярно-генетические маркеры. Наиболее распространены маркеры ядерного и хлоропластного геномов. Однако, ставшие классическими для систематики растений маркеры ядерного ITS-региона (ITS1–5.8S рРНК–ITS2), часто малоинформативны для решения филогенетических вопросов относительно эволюционно молодых групп. Например они показали низкое разрешение в кладе близкородственных видов подсекции Rhodorastra [10], в то время как маркеры некодирующих регионов хлоропластной ДНК выявили четкую генетическую структуру [11].
В данной работе проведена оценка параметров генетической изменчивости и степени дивергенции популяций рододендрона камчатского на Камчатке с использованием локусов хлоропластной ДНК, наследуемой у покрытосеменных по материнской линии. Исследование генетической изменчивости данного вида и уточнения внутривидовой таксономии дадут новую информацию о видообразовании на Дальнем Востоке России и в целом для Берингийского региона.
Проанализировано 47 растений рододендрона камчатского из природных популяций: 30 образцов из трех популяций с южной части полуострова (окрестности г. Елизово, природный парк “Голубые озера”, окрестности вулканов Авачинский и Мутновский); десять – из центральной части (окрестности пос. Эссо), пять образцов с Командорских островов (четыре – о-в Беринга, один – о. Медный), два образца из Корякского автономного округа (Олюторский р-н) (рис. 1, табл. 1). ДНК выделяли по стандартному протоколу для растительных тканей (СТАВ-метод) [12] из свежих или высушенных в силикагеле листьев, а также из гербарных образцов. На первом этапе на панели из восьми образцов была проведена амплификация с универсальными праймерами для девяти маркерных хлоропластных фрагментов. Согласно протоколу и температурному профилю ПЦР, рекомендованными авторами, проведена амплификация части фрагментов trnK–matK (matK) [13] и psbC–trnS (CS) [14]; трех участков trnT–trnF (TabAB, TabCD, Tab EF) [15]; trnF-TrnV [16]; trnH-psbA (HA), trnS-trnG (SG) [17] и atpH-atpI [18]. Фрагменты SG и TabAB показали нестабильную аплификацию. Для остальных методом прямого секвенирования на генетическом анализаторе ABI 3130 (Applied Biosystems, USA) были определены последовательности и выявлены вариабельные сайты. Для анализа всего материала были отобраны маркерные фрагменты, в которых обнаружено больше трех вариабельных сайтов: HA, matK и FV.
Рис. 1.
а – местоположение изученных выборок, распределение обнаруженных гаплотипов хпДНК; б – дерево гаплотипов хпДНК (H1–H3). Размер окружностей отражает частоту встречаемости гаплотипов, поперечные тонкие штрихи на ветвях дерева – мутационные события.
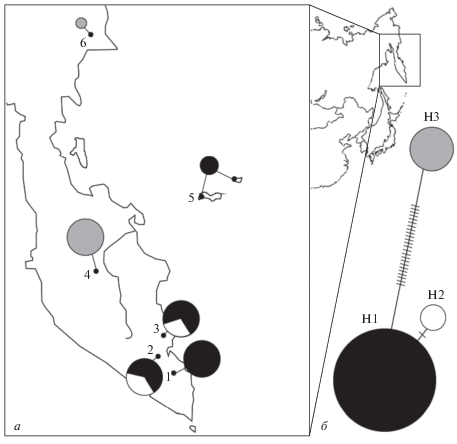
Таблица 1.
Географические координаты мест сбора R. camtschaticum и показатели генетического разнообразия хпДНК в изученных выборках
| Популяция | Координаты, с.ш./в.д. | N | Nh | Н | |
|---|---|---|---|---|---|
| 1 | Камчатский кр., Елизовский р-н, окрест. влк. Мутновской | 52°58′/158°23′ | 10 | H1:10 | 0 |
| 2 | Камчатский кр., Елизовский р-н, природный парк “Голубые озера” | 53°10′/158°08′ | 10 | H1:6 H2:4 |
0.533 |
| 3 | Камчатский кр., Елизовский р-н, окрест. влк. Авачинский | 53°25′/158°82′ | 10 | H1:7 H2:3 |
0.467 |
| 4 | Камчатский кр., Быстринский р-н, Козыревский хр., окрест. пос. Эссо | 55°94′/158°68′ | 10 | H3:10 | 0 |
| 5 | Камчатский кр., Алеутский р-н, Командорские о-ва (о. Беринга и о. Медный) | 54°51′/166°23′ | 5 | H1:5 | 0 |
| 6 | Камчатский кр., Корякский АО, Олюторский р-н, окрест. пос. Ачайваям | 60°96′/170°46′ | 2 | H3:2 | 0 |
Выравнивание последовательностей проводили вручную в программе BioEdit [19]. Расчет числа гаплотипов (Nh), показателя гаплотипического разнообразия (H), уровней дифференциации и распределения генетической изменчивости внутри и между популяциями (анализ молекулярной дисперсии, AMOVA) проводили в программе Arlequin v. 3.5.1.2 [20]. Дерево отношений гаплотипов хпДНК построено в программе Network v. 4.6.1.2 [21]. Каждая мутация (мононуклеотидная замена или индел, независимо от размера), кодировалась как единичное мутационное событие.
Длина объединенной последовательности по трем фрагментам составила 1964 п.о. (HA 1–370; matK 371–1140; FV 1141–1964). Выявлено 25 вариабельных сайтов (табл. 2), из них во фрагменте trnH-psbA – шесть мононуклеотидных замен и один микросателлит А8–А9; во фрагменте trnK-matK – восемь замен; во фрагменте trnF-TrnV – восемь замен, микросателлит А7–А8 и инсерция 98 п.о. Изменчивость сгруппировалась в три гаплотипа (Н1–Н3). Последовательности фрагментов для каждого гаплотипа размещены в GenBank под номерами доступа MN125545–MN125551. Уровень изменчивости нуклеотидных последовательностей соответствует таковому для комплекса R. dauricum s. l., для которого в четырех фрагментах хпДНК совокупно выявлено 28 вариабельных сайтов [11].
Таблица 2.
Сегрегирующие сайты для трех гаплотипов хпДНК. Референсный гаплотип H1
| Гаплотип | Позиция нуклеотида | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| trnH-psbA | matK | trnF-TrnV | |||||||||||||||||||||||
| 7 | 8 | 9 | 1 | 1 | 1 | 2 | 5 | 5 | 5 | 6 | 6 | 6 | 9 | 9 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 2 | 2 | |
| 4 | 0 | 9 | 8 | 8 | 9 | 3 | 4 | 4 | 7 | 2 | 3 | 4 | 5 | 6 | 1 | 2 | 2 | 4 | 4 | 5 | 8 | 9 | 9 | 7 | |
| 3 | 4 | 0 | 0 | 7 | 9 | 4 | 5 | 7 | 1 | 6 | 5 | 6 | 2 | 6 | 2 | 2 | 5 | 6 | 0 | 1 | 9 | ||||
| 1 | 5 | 0 | 0 | 5 | 9 | 7 | 6 | 0 | 0 | ||||||||||||||||
| H1 | A | G | А8 | C | C | A | A | G | G | G | C | A | A | A | A | C | G | A7 | A | del | C | T | T | T | T |
| H2 | . | . | . | Т | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| H3 | С | Т | А9 | . | G | G | C | T | C | T | A | T | G | C | C | G | T | A8 | T | ins | T | G | G | G | G |
Распределение гаплотипов в популяциях и их частоты отображены на рис. 1. В выборках с южной части полуострова Камчатка (выборки 2 и 3) обнаружено два гаплотипа. Минорный гаплотип Н2 отличается от гаплотипа Н1 единственной точковой заменой во фрагменте trnH-psbA (табл. 2, позиция 183). В образцах из окрестностей влк. Мутновский и с Командорских о-вов выявлен доминирующий на юге гаплотип Н1. В выборке из центральной части Камчатки, а также из Корякского автономного округа полиморфизм отсутствует, выявлен гаплотип Н3 (табл. 1), значительно отличающийся от первых двух гаплотипов (табл. 2).
Высокая дифференциация наблюдается между всеми выборками FST = 0.9982, p < 0.001, а также между регионами: из центральной и северной частей полуострова (выборки 5 и 6) с выборками с юга и с Командорских о-вов (выборки 1–4) (FST = = 0.9978, p = 0.05). Таким образом географическое распределение гаплотипов и дифференциация по данным изменчивости хпДНК соответствует двум описанным подвидам.
Ранее зарубежными авторами [22] с использованием семи ядерных локусов было показано отличие нескольких образцов из Центральной Камчатки (четыре образца из окрестностей Эссо) от южных (шесть образцов из окрестностей влк. Авачинского). Выявлены родственные отношения южнокамчатских образцов с японскими популяциями, а также с популяциями с Алеутских островов. Образцы из окрестностей Эссо в указанной публикации принимаются в качестве самостоятельного вида R. glandulosum (=Therorhodion glandulosum) и используются в качестве аутгруппы. Они значительно дивергированы, образуя отдельный генетический кластер.
Таким образом, данные по изменчивости хпДНК на большем количестве образцов с Камчатки согласуются с полученными ранее [22], поддерживая тезис о самостоятельности двух видов R. glandulosum и R. camtschaticum. Однако выявленная дифференциация по маркерам хпДНК не согласуется с разделением видов по морфологическим признакам. Так для Командорских островов и Камчатки в качестве преобладающего указывается R. camtschaticum ssp. glandulosum, типичный подвид встречается реже [4, 5]. В ходе полевых работ типичные на вид растения R. camtschaticum (15–30 см высотой), нами были собраны в популяциях из природного парка “Голубые озера” и на плато в окрестностях влк. Мутновский. Все остальные исследованные образцы, имели вид арктической расы (до 10 см высотой) и принимались нами условно за ssp. glandulosum. При рассмотрении под бинокуляром преобладали образцы с простыми волосками по краю листа. В выборке из окрестностей Эссо образцы варьировали по наличию простых или железистых волосков по краю листа как между растениями, так и в пределах одного растения. Опушение венчика присутствовало в выборках “Голубые озера” и из окрестностей вулкана Авачинский, в выборке из окрестностей Эссо – венчик голый. В выборке из окрестностей вулкана Мутновский на момент сбора не было цветущих растений, остальные образцы выделены из гербария, оценить венчик не было возможности.
Наблюдаемая дифференциация между центральными и северными выборками с одной стороны и южными совместно с Командорскими островами с другой, отражает существование на полуострове двух генетических линий вероятно обусловленных северным и южным миграционными потоками. Такой паттерн генетической структуры согласуется с историей биоты на полуострове, выявленной по ели, лиственнице и пихте [23–25]. Ограниченные центральной частью Камчатки популяции Picea jezoensis, Larix kurilensis ssp. glabra и Abies gracilis по генетическим данным представляют собой остаточные популяции видов, имевших ранее более широкий ареал на территории Дальнего Востока России. Глобальные изменения климата, повлекшие вымирание древесной растительности на территории северо-востока Азии, привели к изоляции их в благоприятных условиях на полуострове. Для лиственницы предполагается вторичный контакт с более молодым видом L. cajanderi, пришедшим с севера. Аналогичные события с повторным заселением Камчатки с севера популяциями рододендрона камчатского, находившегося в длительной изоляции на Чукотке и побережье Охотского моря и достаточно дивергировавшими от первоначального вида, можно предположить и в данном случае. Для детального восстановления истории вида необходим анализ большего количества образцов с севера и юга ареала (Чукотка, Магаданская обл., Сахалин и Курильские острова), а также использование мультилокусного анализа с привлечением ядерных маркеров.
Авторы выражают благодарность Е.А. Марчук и Е.А. Хантемировой за помощь во время полевых работ, а также А.В. Могилевой за образцы с Командорских островов.
Работа (сбор образцов) выполнена в рамках государственного задания Института экологии растений и животных УрО РАН, проведение молекулярно-генетического анализа поддержано грантом РФФИ № 18-04-00616.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Александрова М.С. Рододендроны природной флоры СССР. М.: Наука, 1975. 112 с.
Усенко Н.В. Деревья, кустарники и лианы Дальнего Востока: справочная книга. Хабаровск: Приамурские ведомости, 2010. 272 с.
Сосудистые растения советского Дальнего Востока. Т. 5 / Отв. ред. Харкевич С.С. СПб.: Наука, 1991. 390 с.
Якубов В.В., Чернягина О.А. Каталог флоры Камчатки (сосудистые растения). П-Камчатский: Камчатпресс, 2004. 165 с.
Мочалова О.А., Якубов В.В. Флора Командорских островов. Владивосток: БПИ ДВО РАН, 2004. 120 с.
Юрцев Б.А., Королева Т.М., Петровский В.В. и др. Конспект флоры Чукотской тундры. СПб.: ВВМ, 2010. 628 с.
Hulten E. Flora of Alaska and Neighboring Territories: A Manual of the Vascular Plants. Stanford: Standford University Press, 1968. 1008 p.
Viereck L.A., Little E.L., Jr. Alaska Trees and Shrubs. Fairbanks, AK: University of Alaska Press, 2007. 265 p.
Kron K.A., Judd W.S. The rorhodion // Flora of North America Editorial Committee / Eds. Flora of North America North of Mexico. 20+ vols. New York: Oxford University Press, 2009. V. 8. P. 453–543.
Куцев М.Г., Каракулов А.В. Реконструкция филогении рода Rhododendron L. (Ericaceae) флоры России на основе последовательности спейсеров ITS1_ITS2 // Turczaninowia. 2010. Т. 13. № 3. С. 59–62.
Polezhaeva M.A., Pimenova E.A., Tikhonova N.A. et al. Plastid DNA diversity and genetic divergence within Rhododendron dauricum s. l. (R. dauricum s.s., R. ledebourii, R. sichotense and R. mucronulatum; Ericaceae) // Plant Syst. Evol. 2018. V. 304. P. 763–774. https://doi.org/10.1007/s00606-018-1508-1
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinus radiata based on RFLP, RAPD and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. P. 673–679. https://doi.org/10.1007/BF00226088
Johnson L.A., Soltis D.E. Phylogenetic inference in Saxifragaceae sensu stricto and Gilia (Polemoniaceae) using matK sequences // Ann Mol. Bot. Gard. 1995. V. 82. P. 149–175. https://doi.org/10.2307/2399875
Demesure B., Sodzi N., Petit R.J. A set of universal primers for amplification of polymorphic non-coding regions of mitochondrial and chloroplast DNA in plants // Mol. Ecol. 1995. V. 4. P. 129–134. https://doi.org/10.1111/j.1365-294X.1995.tb00201.x
Taberlet P.T., Geilly L., Patou G., Bouvet J. Universal primers for amplification of three noncoding regions of chloroplast DNA // Pl. Mol. Biol. 1991. V. 17. P. 1105–1109. https://doi.org/10.1007/BF00037152
Dumolin-Lapegue S., Pemonge M.-H., Petit R.J. An enlarged set of consensus primers for the study of organelle DNA in plants // Mol. Ecol. 1997. V. 6. P. 393–397. https://doi.org/10.1046/j.1365-294x.1997.00193.x
Hamilton M.B. Four primer pairs for the amplification of chloroplast intergenic regions with intraspecific variation // Mol. Ecol. 1999. V. 8. P. 521–523.
Grivet D., Heinze B., Vendramin G.G., Petit R.J. Genome walking with consensus primers: Application to the large single copy region of chloroplast DNA // Mol. Ecol. Not. 2001. V. 1. P. 345–349. https://doi.org/10.1046/j.1471-8278.2001.00107.x
Hall T.A. Bioedit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT // Nucl. Acids Symp. Ser. 1999. V. 41. P. 95–98. https://doi.org/10.1111/jbi.12867
Excoffier L., Lischer H. Arlequin suite ver 3.5: A new series of programs to perform population genetics analyses under Linux and Windows // Mol. Ecol. Res. 2010. V. 10. P. 564–567. https://doi.org/10.1111/j.1755-0998.2010.02847.x
Bandelt H.J., Forster P., Ruhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. P. 37–48. https://doi.org/10.1093/oxfordjournals.molbev.a026036
Hata D., Higashi H., Yakubov V. et al. Phylogeographical insight into the Aleutian flora inferred from the historical range shifts of the alpine shrub Therorhodion camtschaticum (Pall.) Small (Ericaceae) // J. Biogeog. 2016. V. 44. P. 283–293. https://doi.org/10.1111/jbi.12876
Aizawa M., Yoshimaru H., Saito H. et al. Phylogeography of a northeast Asian spruce, Picea jezoensis, inferred from genetic variation observed in organelle DNA markers // Mol. Ecol. 2007. V. 16. P. 3393–3405. https://doi.org/10.1111/j.1365-294x.2007.03391.x
Polezhaeva M.A., Lascoux M., Semerikov V.L. Cytoplasmic DNA variation and biogeography of Larix Mill. in Northeast Asia // Mol. Ecol. 2010. V. 19. P. 1239–1252. https://doi.org/10.1111/j.1365-294X.2010.04552.x
Semerikova S.A., Semerikov V.L., Lascoux M. Post-glacial history and introgression in Abies (Pinaceae) species of the Russian Far East inferred from both nuclear and cytoplasmic markers // J. Biogeography. 2010. V. 38. № 2. P. 326–340. https://doi.org/0.1111/j.1365-2699.2010.02394.x
Дополнительные материалы отсутствуют.


