Генетика, 2020, T. 56, № 7, стр. 783-791
Филогеографический анализ штаммов Yersinia pestis улегейского подвида
К. А. Никифоров 1, *, **, Л. М. Куклева 1, Ж. В. Альхова 1, Е. А. Нарышкина 1, Н. П. Гусева 1, Г. А. Ерошенко 1, Е. Г. Токмакова 2, С. В. Балахонов 2, В. В. Кутырев 1
1 Российский научно-исследовательский противочумный институт “Микроб”
410005 Саратов, Россия
2 Иркутский научно-исследовательский противочумный институт
Сибири и Дальнего Востока
664047 Иркутск, Россия
* E-mail: Nikiforov6666666@mail.ru
** E-mail: rusrapi@microbe.ru
Поступила в редакцию 02.07.2019
После доработки 12.08.2019
Принята к публикации 03.10.2019
Аннотация
Исследована популяционная структура штаммов Y. pestis улегейского подвида (генетическая ветвь 0.PE5), эндемичного для природных очагов чумы Монголии и обладающего низким эпидемическим потенциалом в сравнении со штаммами основного подвида чумы. Согласно данным полногеномного SNP анализа 12 штаммов Y. pestis улегейского подвида, выделенных на территории Монголии, с учетом 1581 выявленных коровых SNPs установлено наличие двух основных генетических кластеров, соответствующих географическому распространению штаммов. Первый кластер, названный 0.PE5/1, включает штаммы из аймаков Умнеговь, Уверхангай и Говь-Алтай, расположенных на юге Монголии. Второй кластер 0.PE5/2 состоит из двух субкластеров: 0.PE5/2-1, включающий штаммы из аймаков Баян-Улгий и Ховд, и 0.PE5/2-2, представленный только штаммами из аймака Баян-Улгий. Для выяснения возможных причин избирательной вирулентности штаммов улегейского подвида проведен анализ нуклеотидных последовательностей 79 основных генов факторов патогенности, а также других участков генома. Найдены специфические мутации в генах: irp1, входящем в состав острова высокой патогенности хромосомной области пигментации, и yscQ, участвующем в регуляции синтеза белка LcrV и белков внешней мембраны (Yops), ассоциируемых с вирулентностью возбудителя чумы.
Чума – зооантропонозная особо опасная инфекция, природные очаги которой расположены в различных ландшафтно-географических зонах степей, полупустынь, пустынь и гор. На территории Российской Федерации (РФ), стран СНГ и ближнего зарубежья находятся 45 природных очагов чумы [1].
Штаммы возбудителя чумы отличаются по вирулентности, внутривидовой принадлежности и географическому распространению. Штаммы основного подвида являются высоковирулентными и обладают высоким эпидемическим потенциалом; штаммы неосновных подвидов имеют низкий эпидемический потенциал и избирательную вирулентность, вызывая эпизоотии среди мышевидных грызунов и зайцеобразных. Ранее нами предложена усовершенствованная внутривидовая классификация возбудителя чумы с выделением семи подвидов: основной, ssp. pestis; тибетский, ssp. tibetica (Китай); кавказский, ssp. caucasica (РФ, Кавказ); ангольский, ssp. angolica (Ангола); центральноазиатский, ssp. central asiatica (Россия, Китай, Кыргызстан, Таджикистан); цинхайский, ssp. qinghaica (Китай); улегейский, ssp. ulegeica (Монголия) [2, 3]. Известны случаи заболевания чумой человека, вызванные штаммами кавказского, тибетского и цинхайского подвидов [4].
Во многих пограничных с Россией государствах часто регистрируют случаи заболевания чумой человека, что в условиях активных экономических, культурных и туристических связей может привести к завозу чумы в РФ [5]. Протяженность границы трансграничных очагов чумы России и Монголии составляет 585 км. Энзоотичные по чуме территории в Монголии выявлены в 137 сомонах 17 аймаков и занимают около 30% площади страны, что составляет 443 тыс. кв. км [6, 7]. В разные годы в литературе сообщалось о выделении в Монголии от 6 до 12 природных очагов чумы различного типа и степени автономности [8–10].
В настоящее время в Монголии выделяют 12 очагов чумы, два из которых (Сайлюгемский и Хойт-Хэрленский (Северо-Керуленский)) являются трансграничными с РФ, семь (Хархира-Тургенский, Хан-Хухэйский, Сонгино-Тудэвский, Булнайский (Тэлмэн-Соготынский), Тэсийн-Шарагольский, Хурхинский, Зун-Хэнтейский) занимают приграничные территории [7]. Во многих из них протекают активные эпизоотические процессы [11]. В мае 2019 г. в Монголии зарегистрированы два случая заражения чумой человека с летальным исходом, которые произошли в аймаке Баян-Улгий, расположенном на границе с РФ (http://www.promedmail.org/index.php).
Ранее нами и зарубежными авторами проведено исследование штаммов Y. pestis из очагов Монголии по их фенотипическим свойствам с установлением внутривидовой принадлежности, а также отнесением их к основным филогенетическим ветвям [12–14]. В результате этих исследований на территории Монголии отмечена циркуляция штаммов Y. pestis основного подвида античного биовара линий 2.ANT3, 3.ANT2, 4.ANT, а также штаммов неосновных подвидов: улегейского (0.PE5) и центральноазиатского (0.PE4).
Штаммы улегейского подвида эндемичны для Монголии. Улегейский подвид наиболее эволюционно молодой среди неосновных подвидов. Однако в отличие от основного подвида штаммы улегейского подвида, как правило, не являются вирулентными для людей, вызывая лишь эпизоотии среди мышевидных грызунов и мелких зайцеообразных. Они не обладают высоким эпидемическим потенциалом.
Вирулентность Y. pestis – это полидетерминантный признак, зависящий от наличия факторов патогенности, кодируемых генами хромосомной и плазмидной локализации [15, 16]. В геноме Y. pestis находится хромосомная область пигментации, содержащая остров высокой патогенности HPI с генами сидерофор-зависимой системы потребления железа. При потере острова высокой патогенности HPI штаммы чумы становятся авирулентными [17].
Гены, кодирующие факторы патогенности возбудителя чумы, расположены на трех резидентных плазмидах: родоспецифической pCad и двух видоспецифических pFra и pPst [18, 19].
Плазмида pCad несет в своем составе гены, кодирующие систему секреции третьего типа (СС3Т). СС3Т вместе с островом высокой патогенности HPI является обязательным фактором вирулентности Y. pestis [20, 21]. Одним из основных компонентов СС3Т является эффекторный белок LcrV, кодируемый геном lcrV, принимающий участие в секреции эффекторных белков Y. pestis в клетки млекопитающих.
По данным литературы детальный молекулярно-генетический и филогенетический анализ штаммов Y. pestis улегейского подвида не проводился. Также неизвестны причины отсутствия случаев чумы у людей, вызванных штаммами улегейского подвида.
Цель настоящей работы – молекулярно-генетический и филогенетический анализ штаммов Y. pestis улегейского подвида, а также поиск возможных генетических мутаций, вызвавших отсутствие способности этих штаммов вызывать заболевание у людей.
МАТЕРИАЛЫ И МЕТОДЫ
Штаммы, изучение культурально-морфологических и биохимических свойств, выделение ДНК
Все штаммы, использованные в настоящей работе, получены из государственной коллекции патогенных бактерий при РосНИПЧИ “Микроб”. Культивирование штаммов осуществляли в соответствии с общепринятыми методами [22].
Выделение ДНК выполняли с использованием наборов “PureLink Genomic DNA Mini Kit” (Invitrogen, США).
Полногеномное секвенирование, поиск SNPs, филогенетический анализ, построение дендрограмм
Полногеномное секвенирование штаммов Y. pestis проводили в системе Ion PGM (Life technologies, США). Обработку данных секвенирования и сборку генома de novo осуществляли с использованием программ Ion Torrent Suite software 4.2 и Newbler gsAssembler 2.6. Последовательности ридов собирали в геномы, покрытие которых по геному референсного штамма СО92 (NCBI GenBank ID NC_003143.1) составляло не менее 94% с 48-кратным прочтением. Штаммы Y. pestis И-2422, И-3068, И-3193, И-3196, И-3269 депонированы в базу данных NCBI GenBank (табл. 1).
Таблица 1.
Штаммы Y. pestis, полногеномные последовательности которых использованы в работе
| Штамм Y. pestis | Филогенетическая ветвь [2] |
Номер доступа нуклеотидной последовательности в NCBI GenBank |
|---|---|---|
| 620024 | 0.PE7 | NZ_ADPM00000000.1 |
| Pestoides F | 0.PE2 | NC_009381.1 |
| Pestoides A | 0.PE4a | NZ_ACNT00000000.1 |
| 91001 | 0.PE4m | NC_005810.1 |
| KIM10 | 2.MED1 | NC_004088.1 |
| Nepal516 | 2.ANT | NC_008149.1 |
| CO92 | 1.ORI1 | NC_003143.1 |
| MGJZ3 | 3.ANT | ADSW01 |
| MGJZ12 | 4.ANT | ADSV01 |
| SCPM_O_B_6212 | 0.PE5 | PVLX01 |
| SCPM_O_B_6213 | 0.PE5 | LIZD01 |
| SCPM_O_B_6218 | 0.PE5 | PVLY01 |
| SCPM_O_B_6301 | 0.PE5 | PVLИW01 |
| SCPM_O_B_6706 | 0.PE5 | LIYO01 |
| SCPM_O_DNA_15 | 0.PE5 | PVLZ01 |
| SCPM_O_DNA_17 | 0.PE5 | PVMB01 |
| И-2422* | 0.PE5 | QANK01 |
| И-3196* | 0.PE5 | VIDY01 |
| И-3068* | 0.PE5 | VHPW01 |
| И-3193* | 0.PE5 | VIDX01 |
| И-3269* | 0.PE5 | VIDZ01 |
Филогенетический анализ и построение дендрограммы проводили на основании выявленных коровых SNPs (единичных нуклеотидных полиморфизмов) в полногеномных последовательностях исследуемых штаммов. Поиск маркерных SNPs осуществляли с использованием программы Wombac 2.0 на базе операционной системы BioLinux 8.0. Построение филогенетического дерева с применением алгоритма Maximum Likelihood выполняли в программе PhyML 3.1. Визуализацию полученных результатов проводили с помощью программы FigTree 1.4.3.
Поиск ДНК мишеней, конструирование праймеров, проведение ПЦР, фрагментное секвенирование
Поиск ДНК мишеней – SNPs, маркерных для кластеров штаммов Y. pestis улегейского подвида, осуществляли с помощью анализа матрицы коровых SNPs с применением программы Mega 6.0 (http://www.megasoftware.net/). Оценку специфичности найденных SNPs проводили с использованием алгоритма BLAST и полногеномных последовательностей штаммов Y. pestis из базы данных NCBI GenBank. Расчет праймеров выполняли с помощью программы Vector NTI 10 (http:// www.thermofisher.com). ПЦР осуществляли в амплификаторе БИС (Кольцово, Россия).
Ампликоны, полученные в ПЦР на матрице ДНК штаммов Y. pestis, секвенировали с помощью генетического анализатора Applied Biosystems 3500xL (Applied Biosystems, США). Анализ полученных последовательностей выполняли с помощью программы MEGA 6.0 и алгоритма BLAST на основе нуклеотидных последовательностей из базы данных NCBI GenBank.
РЕЗУЛЬТАТЫ
В работе проведено комплексное исследование свойств 19 штаммов Y. pestis улегейского подвида, выделенных в Монголии, и определены полногеномные нуклеотидные последовательности пяти штаммов этого подвида (табл. 1). Для анализа популяционной структуры улегейского подвида использованы также полногеномные последовательности еще семи штаммов улегейского подвида из базы данных NCBI GenBank [23], а также последовательности девяти штаммов Y. pestis, относящихся к другим филогенетическим ветвям возбудителя чумы (табл. 1). Кроме того, определение наличия в геноме SNPs, маркерных для кластеров штаммов улегейского подвида, было проведено еще у семи штаммов улегейского подвида. Все изученные штаммы улегейского подвида обладали типичными дифференциальными биохимическими свойствами: они не редуцировали нитраты, но ферментировали рамнозу, арабинозу, глицерин, что соответствовало данным литературы [24].
Ранее было показано, что штаммы улегейского подвида несут в составе своего генома специфическую мутацию – делецию 88 пн в межгенной области YPO1452–YPO1453. На эту область нами были рассчитаны праймеры и зонд и подобраны условия проведения ПЦР в режиме реального времени [25].
Для подтверждения принадлежности использованных штаммов Y. pestis к улегейскому подвиду они были изучены на наличие этой маркерной мутации методом ПЦР в режиме реального времени. У всех изученных штаммов было детектировано наличие делеции, что подтвердило их принадлежность к улегейскому подвиду.
Популяционная структура штаммов Y. pestis улегейского подвида
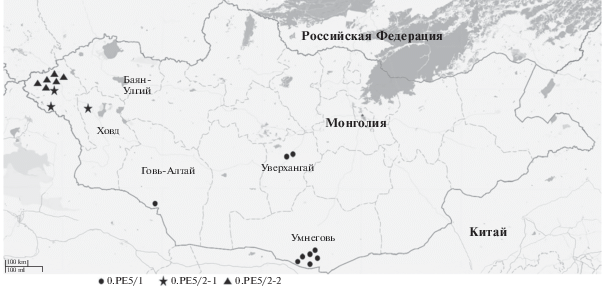
Для определения популяционной структуры штаммов улегейского подвида проведен полногеномный SNP анализ корового генома и найден 1581 полиморфный нуклеотид, на основании которого осуществлено построение дендрограммы Maximum Likelihood (рис. 1). Как видно из дендрограммы, штаммы улегейского подвида, выделенные в разные года, образовали два кластера, которые обозначены нами как 0.PE5/1 и 0.PE5/2. Кластер 0.PE5/1 представлен штаммами, выделенными на территории аймаков Умнеговь, Уверхангай и Говь-Алтай юга Монголии в период 1973–1986 гг. Кластер 0.PE5/2 в свою очередь состоит из двух субкластеров 0.PE5/2-1 и 0.PE5/2-2 (рис. 1). Первый субкластер представлен штаммами, выделенными на территории аймаков Баян-Улгий и Ховд в период 1974–1986 гг.; второй субкластер состоит из штаммов, полученных в аймаке Баян-Улгий в период 1972–1990 гг. Таким образом, кластер 0.PE5/2 включает штаммы с территории западных аймаков Монголии, при этом в аймаке Баян-Улгий циркулируют штаммы двух подкластеров 0.PE5/2-1 и 0.PE5/2-2 (рис. 2).
Рис. 1.
Дендрограмма, построенная по результатам филогенетического анализа 21 штамма разных филогенетических ветвей с использованием 1581 коровых SNPs. Алгоритм Maximum Likelihood, программа PhyML 3.1, модель HKY85.
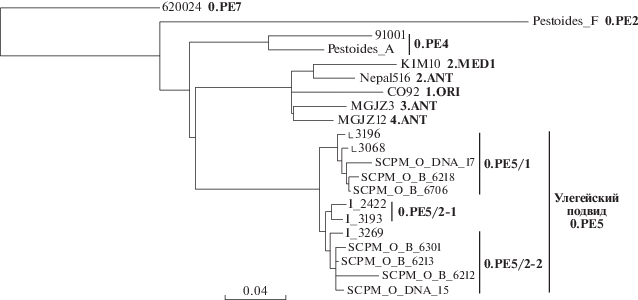
Рис. 2.
Распространение штаммов Y. pestis улегейского подвида (филогенетическая ветвь 0.PE5) на территории Монголии. Для рисунка использована карта с сайта OpenStreetMap (www.openstreetmap.org/copyright).
Был проведен поиск SNPs, специфичных для отдельных эволюционных ветвей улегейского подвида. Поиск осуществляли для выяснения филогеографических закономерностей разделения штаммов с помощью определения этих маркерных SNPs у штаммов улегейского подвида, находящихся в государственной коллекции патогенных бактерий РосНИПЧИ “Микроб”.
В результате проведенного анализа для штаммов кластера 0.PE5/1 были отобраны три маркерных SNPs. Первая мутация (замена Т→A), названная 0.PE5/11, расположена в гене ligB в позиции 54674 по геному штамма СО92 (GenBank ID NC_003143.1). Вторая маркерная замена нуклеотида (Т→G), названная 0.PE5/12, находится в гене cysG в позиции 172 494; третья специфическая мутация 0.PE5/13 (A→C) расположена в гене glgA в позиции 4 425 839 (табл. 2).
Таблица 2.
Перечень SNPs, специфичных для филогенетических ветвей штаммов Y. pestis улегейского подвида
| Позиция от начала генома* | Мутация | Ген | Функция гена | Ветви Y. pestis, у которых присутствует мутация |
|---|---|---|---|---|
| 54 674 | Т→A | ligB | НАД-зависимая ДНК-лигаза LigB | 0.PE5/1 |
| 172 494 | Т→G | cysG | Сирогемсинтаза | 0.PE5/1 |
| 4 425 839 | A→C | glgA | Гликогенсинтаза | 0.PE5/1 |
| 3 081 204 | C→A | sixA | Фосфогистидинфосфатаза | 0.PE5/2-1 |
| 4 413 306 | G→A | argE | Ацетилорнитиндеацетилаза | 0.PE5/2-1 |
| 353 703 | Т→G | YPO0344 | Форматдегидрогенеаза H | 0.PE5/2-2 |
| 2 580 558 | C→A | YPO2295 | Нуклеотидаза | 0.PE5/2-2 |
Для штаммов субкластера 0.PE5/2-1 выбраны следующие маркерные нуклеотидные замены: первая (C→A) 0.PE5/2-11 – в гене sixA (позиция 3081204), вторая (G→A) 0.PE5/2-12 – в гене argE (позиция 4413306).
Для дифференциации штаммов субкластера 0.PE5/2-2 были использованы следующие специфические замены нуклеотидов: первая (Т→G) 0.PE5/2-21 – в гене YPO0344 (позиция 353 703), вторая (C→A) 0.PE5/2-22 – в гене YPO2295 (позиция 2 580 558).
На выбранные локусы были рассчитаны праймеры (табл. 3) для установления принадлежности еще семи штаммов улегейского подвида, для которых не было проведено полногеномное секвенирование, к субкластерам 0.PE5/1, 0.PE5/2-1, 0.PE5/2-2. На матрице их ДНК получены ампликоны, которые потом были использованы для фрагментного секвенирования.
Таблица 3.
Последовательности праймеров, использованных в работе
| Название мишени | Штаммы Y. pestis, у которых присутствует мутация | Последовательность праймеров |
|---|---|---|
| 0.PE5/11 | 0.PE5/1 | 0.PE5/11-S – AAATACAGCGAGCCCGTC 0.PE5/11-AS – CCGTTCGTCCAAGGGTAAA |
| 0.PE5/12 | 0.PE5/1 | 0.PE5/12-S – TGAGTGGTCGCTGGCATA 0.PE5/12-AS – TTGCTCGGCTTACAGTGGT |
| 0.PE5/13 | 0.PE5/1 | 0.PE5/13-S – CCACAGGGTTCAAAGCGGCT 0.PE5/13-AS – AGTTGGTCGTCTTGGGGAGC |
| 0.PE5/2-11 | 0.PE5/2-1 | 0.PE5/2-11-S – AGCAGTCAGTGGATATCG 0.PE5/2-11-AS – TCTTCTTTCGCCAACGCT |
| 0.PE5/2-12 | 0.PE5/2-1 | 0.PE5/2-12-S – AGCTACGCCCTGATTTCG 0.PE5/2-12-AS – CGCTGGATTGTGATAACGCT |
| 0.PE5/2-21 | 0.PE5/2-2 | 0.PE5/2-21-S – GAAAAAGTGACCGACAGTGAGG 0.PE5/2-21-AS – TGGCTCGGTCTGGATTGGA |
| 0.PE5/2-22 | 0.PE5/2-2 | 0.PE5/2-22-S – ATAACGCCTGCAATTCGG 0.PE5/2-22-AS – GGGTGTCGCTAAGCCTGAT |
В результате показано наличие трех маркерных для ветви 0.PE5/1 нуклеотидных замен у штаммов И-3069 (Уверхангай, 1982), И-3071 (Умнеговь, 1982), И-3130 (Умнеговь, 1984), И-3131 (Умнеговь, 1984), И-2972 (Умнеговь, 1981). Это означает, что эти пять штаммов тоже входят в состав ветви 0.PE5/1. По сумме полученных данных на основе полногеномного и фрагментного секвенирования в этот кластер улегейского подвида вошли штаммы, выделенные на территории аймаков Умнеговь, Уверхангай и Говь-Алтай в период с 1973 по 1986 г.
Два SNPs, маркерных для субкластера 0.PE5/2-1 (в позициях 3081204 и 4413306), найдены у штамма Y. pestis И-2962, выделенного на территории аймака Баян-Улгий в 1977 г. Таким образом, согласно полученным данным полногеномного и фрагментного секвенирования субкластер 0.PE5/2-1 включает штаммы, выделенные на территории аймаков Баян-Улгий и Ховд в период с 1974 по 1986 г.
Маркерные для подкластера 0.PE5/2-2 SNPs – 0.PE5/2-21 и 0.PE5/2-22 с координатами 353703 и 2580558, соответственно, выявлены в геноме штамма И-3215, выделенного на территории аймака Баян-Улгий в 1987 г. В результате подкластер 0.PE5/2-2 объединяет штаммы, выделенные на территории аймака Баян-Улгий в период с 1972 по 1987 гг.
Основываясь на данных полногеномного секвенирования 12 штаммов и фрагментного секвенирования семи штаммов улегейского подвида возбудителя чумы и на установленной популяционной структуре этих штаммов можно сделать заключение о циркуляции на территории Монголии двух основных генетических ветвей улегейского подвида: первая ветвь, 0.PE5/1, состоит из штаммов, выделенных на территории южных аймаков Монголии – Умнеговь, Уверхангай и Говь-Алтай в период 1973–1986 гг.; вторая ветвь, 0.PE5/2, состоит из двух подветвей: 0.PE5/2-1 включает штаммы 1977–1990 гг. из двух расположенных рядом западных аймаков Баян-Улгий (юго-восточные сомоны аймака, граничащие с аймаком Ховд с востока и с территорией Китая с юга) и Ховд; 0.PE5/2-2 состоит только из штаммов из аймака Баян-Улгий (северные и западные сомоны аймака, расположенные вблизи границы с РФ), выделенных в 1972–1987 гг. Таким образом, штаммы улегейского подвида можно разделить по географическому принципу на “южную” ветвь 0.PE5/1 и “западную” ветвь 0.PE5/2. На территории аймака Баян-Улгий отмечена циркуляция штаммов двух подкластеров ветви 0.PE5/2: штаммы подкластера 0.PE5/2-1 занимают его юго-восточные сомоны, а штаммы подкластера 0.PE5/2-2 – северные и западные сомоны.
Анализ генов факторов патогенности у штаммов Y. pestis улегейского подвида
Нами выполнен сравнительный анализ нуклеотидных последовательностей основных генов факторов патогенности у штаммов улегейского подвида и штаммов Y. pestis других филогенетических линий. Всего исследовано 79 генов, входящих в хромосомную область пигментации, плазмиду pCad, кодирующую систему секреции третьего типа, плазмиду pFra, кодирующую, в частности, капсульный антиген и мышиный токсин, плазмиду pPst, кодирующую синтез плазминогена и пестицина [16]. В большинстве генов не было найдено мутаций, являющихся возможными причинами избирательной вирулентности улегейского подвида. Исключение составили два гена irp1 и yscQ.
При анализе нуклеотидной последовательности гена irp1, входящего в состав острова высокой патогенности хромосомной области пигментации, у всех штаммов улегейского подвида обнаружена нуклеотидная замена C→A в позиции 7869 от начала гена согласно геному штамма СО92. Однако данная мутация является синонимичной и не приводит к изменению кодируемой аминокислоты. По-видимому, эта мутация не является причиной авирулентности штаммов улегейского подвида для людей.
При анализе структуры генов СС3Т обнаружена специфическая мутация в гене yscQ, кодирующем белок YscQ (Q – белок-переносчик белков внешней мембраны). Было установлено наличие в этом гене специфической мутации – замены Т→A в позиции 296 от начала гена, приводящей к замене аминокислоты лейцин на глутамин. Продукт гена yscQ принимает участие в регуляции синтеза белка LcrV и белков внешней мембраны (Yops) СС3Т, которая является обязательным фактором патогенности возбудителя чумы [26, 27]. Возможно, что эта замена может быть одной из причин авирулентности штаммов улегейского подвида. Для подтверждения или опровержения этого требуются дальнейшие исследования.
Таким образом, при анализе 79 генов хромосомной и плазмидной локализации, ассоциированных с факторами патогенности, у штаммов улегейского подвида обнаружена мутация в гене фактора вирулентности yscQ, которая может оказывать влияние на вирулентность штаммов этого подвида.
ОБСУЖДЕНИЕ
Согласно имеющимся данным литературы исследование штаммов Y. pestis из очагов Монголии проводилось только с определением их принадлежности к основным филогенетическим ветвям. При этом филогенетического анализа штаммов улегейского подвида не проводилось.
Таким образом, нами установлена популяционная структура и изучены генетические особенности штаммов Y. pestis улегейского подвида на основании исследования 19 штаммов этого подвида.
По данным проведенного полногеномного SNP анализа 12 штаммов Y. pestis улегейского подвида с учетом 1581 выявленных коровых SNPs установлено наличие двух основных кластеров, соответствующих географическому распространению штаммов. Первый кластер, обозначенный как 0.PE5/1, включает в себя штаммы из аймаков Умнеговь, Уверхангай и Говь-Алтай. Второй кластер, 0.PE5/2, состоит из двух ветвей эволюции: 0.PE5/2-1, объединившей штаммы из аймаков Баян-Улгий (его юго-восточные сомоны), Ховд, и 0.PE5/2-2, включающей только штаммы из аймака Баян-Улгий (его северные и западные сомоны). Это означает, что улегейский подвид на территории Монголии представлен двумя основными ветвями эволюции, распространенными в определенных географических регионах. Ветвь 0.PE5/1 циркулирует в центре и на юге страны в аймаках, граничащих с Китаем, в области полупустынь, переходящих южнее в пустыню Гоби и область юго-восточных отрогов Хангайских гор, являющуюся высокогорной степью. Горные территории запада страны, относящиеся к Монгольскому Алтаю, заняты штаммами линии 0.PE5/2, внутри которой в свою очередь выделяются две субветви: 0.PE5/2-1 (южные и восточные части Монгольского Алтая), и 0.PE5/2-2 (западные и северные части Монгольского Алтая).
Штаммы улегейского подвида невирулентны для людей, не обладают высоким эпидемическим потенциалом и способны вызывать эпизоотии лишь среди мышевидных грызунов и мелких зайцеообразных. Для выяснения молекулярно-генетических причин низкой вирулентности штаммов улегейского подвида проведен анализ нуклеотидных последовательностей 79 генов, кодирующих основные факторы патогенности возбудителя чумы, а также другие участки генома. Найдены специфические мутации в генах irp1 и yscQ. Мутация в гене yscQ приводит к изменению кодируемой аминокислоты, и ее значение для проявления авирулентности штаммами улегейского подвида для человека предстоит выяснить в дальнейшем.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Онищенко Г.Г., Кутырев В.В., Федоров Ю.М. и др. Природные очаги чумы Кавказа, Прикаспия, Средней Азии и Сибири / Под ред. Онищенко Г.Г., Кутырева В.В. М.: Медицина, 2004. 192 с.
Kutyrev V.V., Eroshenko G.A., Motin V.L. et al. Phylogeny and classification of Yersinia pestis through the lens of strains from the plague foci of Commonwealth of Independent States // Front. Microbiol. 2018. V. 9. № 1106. https://doi.org/10.3389/fmicb.2018.01106
Ерошенко Г.А., Краснов Я.М., Носов Н.Ю. и др. Совершенствование подвидовой классификации Yersinia pestis на основе данных полногеномного секвенирования штаммов из России и сопредельных государств // Проблемы особо опасных инфекций. 2015. № 4. С. 58–64. https://doi.org/10.21055/0370-1069-2015-4-58-64
Cui Y., Yu C., Yan Y. et al. Historical variation in mutational rate in an epidemic pathogen Yersinia pestis // PNAS. 2013. V. 110. № 2. P. 577–582.
Попов Н.В., Карнаухов И.Г., Пакскина Н.Д. и др. Оценка современной эпидемиологической обстановки в природных очагах чумы мира. Повышение эффективности эпидемиологического надзора в природных очагах чумы Российской Федерации и прогноз их эпизоотической активности на 2019 г. // Проблемы особо опасных инфекций. 2019. № 1. С. 81–88. https://doi.org/10.21055/0370-1069-2019-1-81-88
Адъяасурэн З., Цэрэнноров Д., Отгонбаатар Д. и др. Клинико-эпидемиологические особенности чумы в Монголии // Проблемы особо опасных инфекций. 2010. № 1. С. 30–33. https://doi.org/10.21055/0370-1069-2010-1(103)-30-33
Обеспечение эпидемиологического благополучия в природных очагах чумы на территории стран СНГ и Монголии в современных условиях / Под ред. Поповой А.Ю., Кутырева В.В.: Ижевск: Принт, 2018. 336 с.
Дэмбэрэл Ж. Некоторые особенности чумных очагов Монголии // Докл. Иркут. противочум. ин-та Сибири и ДВ. Чита, 1974. № 10. С. 62–63.
Жамба Г., Галбадрах Д., Саран М. и др. К стратегии и тактике изучения природного очага чумы в МНР // Эпидемиол. и профилакт. особо опасных инф. в МНР и СССР. Улан-Батор, 1978. С. 8–11.
Некипелов Н.В. Эпизоотология чумы в Монгольской Народной Республике // Изв. Иркут. гос. науч.-исслед. противочум. ин-та Сибири и ДВ. Иркутск, 1959. № 22. С. 108–243.
Адъяасурэн З., Цэрэнноров Д., Мягмар Ж. и др. Современная ситуация в природных очагах чумы Монголии // Дальневост. журн. инфекц. патологии. 2014. № 25. С. 22–25.
Куклева Л.М., Шавина Н.Ю., Одиноков Г.Н. и др. Анализ разнообразия и определение геновариантов штаммов возбудителя чумы из очагов Монголии // Генетика. 2015. Т. 51. № 3. С. 298–305.
Kiefer D., Dalantai G., Damdindorj T. et al. Phenotypical characterization of Mongolian Yersinia pestis strains // Vector Borne Zoonotic Dis. 2012. V. 12. № 3. P. 183–188. https://doi.org/10.1089/vbz.2011.0748
Riehm J.M., Vergnaud G., Kiefer D. et al. Yersinia pestis Lineages in Mongolia // PLoS One. 2012. V. 7. № 2. e30624. https://doi.org/10.1371/journal.pone.0030624
Подладчикова О.Н. Современные представления о молекулярных механизмах патогенеза чумы // Проблемы особо опасных инфекций. 2017. № 3. С. 33–40. https://doi.org/10.21055/0370-1069-2017-3-33-40
Rasmussen S., Allentoft M.E., Nielsen K. et al. Early divergent strains of Yersinia pestis in Eurasia 5000 years ago // Cell. 2015. V. 163 № 3. P. 571–582. https://doi.org/10.1016/j.cell.2015.10.009
Kutyrev V.V., Filippov A.A., Oparina O.S. et al. Analisis of Yersinia pestis chromosomal determinants Pgm+ and Psts associated with virulence // Microb. Pathog. 1992. V. 12. P. 177–186.
Попов Ю.А., Проценко О.А., Анисимов П.И. и др. Обнаружение плазмид пестициногенности чумного микроба методом электрофореза в агарозном геле // Профилактика особо опасных инфекций. 1980. С. 20–25.
Проценко О.А., Анисимов П.И., Можаров О.Т. и др. Выявление и характеристика плазмид чумного микроба, детерминирующих синтез пестицина I, антигена фракция I и экзотоксина “мышиного” токсина // Генетика. 1983. Т. 19. № 7. С. 1081–1090.
Skurnic M., Wolf-Watz H. Analysis of the yopA gene encoding the Yop1 virulence determinants of Yersinia spp. // Mol. Microbiol. 1989. V. 3. P. 517–529.
Comelis G.R. The Yersinia Ysc-Yop “Type III” weaponry // Nat. Rev. Mol. Cell. Biol. 2002. V. 3. P. 742–752.
Лабораторная диагностика опасных инфекционных болезней. Практическое руководство // Под ред. акад. РАМН Онищенко Г.Г. и акад. РАМН Кутырева В.В.. М.: ЗАО “Шико”, 2-е изд., перераб. и доп., 2013. 560 с.
Kislichkina A.A., Bogun A.G., Kadnikova L.A. et al. Six WholeGenome Assemblies of Yersinia pestis subsp. Microtus bv. Ulegeica (phylogroup 0.PE5) strains isolated from Mongolian natural plague foci // Genome Announc. 2018. V. 6. № 25. https://doi.org/10.1128/genomeA.00536-18
Тимофеева Л.А. О таксономии чумного микроба // Проблемы особо опасных инфекций. 1972. Т. 1(23). С. 15–22.
Никифоров К.А., Оглодин Е.Г., Куклева Л.М. и др. Подвидовая дифференциация штаммов Yersinia pestis методом ПЦР с гибридизационно-флуоресцентным учетом результатов // Журн. микробиол., эпидемиол. и иммунол. 2017. № 2. С. 22–27.
Jackson M.W., Plano G.V. Interactions between type III secretion apparatus components from Yersinia pestis detected using the yeast two-hybrid system // FEMS Microbiol. Lett. 2000. V. 186. № 1. P. 85–90.
Fields K.A., Plano G.V., Straley S.C. A low-Ca2+ response (LCR) secretion (ysc) locus lies within the lcrB region of the LCR plasmid in Yersinia pestis // J. Bacteriol. 1994. V. 176. № 3. P. 569–579.
Дополнительные материалы отсутствуют.


