Генетика, 2020, T. 56, № 7, стр. 842-847
Изменчивость локуса mh44 митохондриальной ДНК в популяциях ели сибирской
А. К. Экарт 1, *, В. Л. Семериков 2, **, А. Я. Ларионова 1, А. Н. Кравченко 1
1 Институт леса им. В.Н. Сукачева Сибирского отделения Российской академии наук
660036 Красноярск, Россия
2 Институт экологии растений и животных Уральского отделения
Российской академии наук
620144 Екатеринбург, Россия
* E-mail: alya-larion@yandex.ru
** E-mail: semerikov@ipae.uran.ru
Поступила в редакцию 17.06.2019
После доработки 30.07.2019
Принята к публикации 01.08.2019
Аннотация
Изучена изменчивость локуса mh44 мтДНК в 24 природных популяциях ели сибирской (Picea obovata Ledeb.). Идентифицировано 13 аллельных вариантов, различающихся по числу повторов 32-нуклеотидного минисателлитного мотива. Определены параметры внутри- и межпопуляционного разнообразия, уровень дифференциации включенных в исследование популяций ели.
В последние два десятилетия в популяционно-генетических исследованиях хвойных активно используются ДНК-маркеры ядерного и цитоплазматических геномов. Различный тип наследования этих маркеров позволяет решать широкий круг задач популяционной генетики и ботаники. Для филогеографических исследований наиболее информативными оказались маркеры митохондриального генома [1–6 и др.]. Они позволяют не только оценить уровень генетического разнообразия, структуру и степень географической дифференциации популяций хвойных, но и могут успешно использоваться для изучения эволюции, анализа гибридизации, а также для получения информации о расселении видов в послеледниковый период.
Филогеографические исследования наиболее распространенной в России ели сибирской (Picea obovata Ledeb.) в необходимом объеме практически не проводились в связи с отсутствием пригодных для изучения этого вида полиморфных маркеров мтДНК. Широко используемый в филогенетических исследованиях других видов рода Picea минисателлитный локус второго интрона гена nad1 митохондриальной ДНК (мтДНК) оказался малоинформативным для изучения географической дифференциации популяций ели сибирской, так как был мономорфен на большей части ареала вида. Незначительная изменчивость наблюдалась лишь в нескольких популяциях из южных районов Сибири [3, 7]. Но, как показали исследования, указанный локус может успешно применяться, в том числе совместно с маркерами ядерного и хлоропластного геномов, для выявления пространственной дифференциации популяций на общем ареале видов Picea abies и Picea obovata, изучения гибридизации и интрогрессии этих видов [3, 4, 7–9]. На основании анализа полиморфизма второго интрона гена nad1 мтДНК в большой выборке популяций комплекса Picea abies–Picea obovata идентифицированы три группы высокодифференцированных друг от друга гаплотипов – южноевропейская, североевропейская и сибирская, определена граница географического разделения видов [4, 7].
Очень низкое гаплотипическое разнообразие локуса nad1 (интрон 2) мтДНК в сибирской группе популяций свидетельствует о неэффективности его использования для изучения филогеографической структуры ели сибирской. Возможно, Picea obovata имеет свою собственную историю послеледниковых миграций [8], информация о которой с помощью nad1 (интрон 2) локуса не выявляется, но может быть получена на основании анализа изменчивости каких-то других маркеров ДНК. Поэтому в последние годы большое внимание уделяется поиску и разработке на основе геномного секвенирования пригодных для анализа ели сибирской, а также других лесообразующих видов хвойных Сибири, полиморфных маркеров цитоплазматических геномов [10–12].
В задачи настоящей работы входило изучение структуры изменчивости минисателлитного локуса mh44 мтДНК ели сибирской из различных районов ее естественного распространения в азиатской части ареала с целью оценки перспективности его использования для популяционно-генетических и филогеографических исследований вида. Ранее локус mh44 был идентифицирован в митохондриальном геноме Picea abies [13] и изучен в 14 популяциях этого вида. В результате были получены данные о высоком генетическом разнообразии и значительной пространственной дифференциации популяций ели европейской по локусу mh44, свидетельствующие о том, что локус может быть полезен для выявления географической структуры популяций и исследования истории расселения P. abies в послеледниковый период. Именно это обстоятельство и определило выбор минисателлитного локуса mh44 в качестве маркера митохондриальной ДНК для изучения генетического разнообразия ели сибирской.
Материалом для исследования изменчивости локуса mh44 мтДНК ели сибирской послужили 24 выборки из популяций, расположенных в Красноярском крае (в т.ч. в Эвенкийском районе), республиках Алтай, Хакасия, Тыва, Бурятия, Якутия, а также в Новосибирской, Иркутской, Магаданской и Амурской областях и в Монголии (табл. 1). Для анализа ДНК использовали высушенную в силикагеле хвою, собранную в каждой популяции с 25–58 деревьев. Всего было проанализировано 720 индивидуумов. Выделение геномной ДНК осуществляли СТАВ-методом [14]. Для амплификации ДНК использовали наборы реагентов GenePak PCR Core (ООО “Лаборатория Изоген”, Россия) согласно рекомендациям производителя. Амплификацию проводили с парой специфических праймеров: F: GTCCCAGAACTCACAGCTTTA и R: TTCACTTGATACTCACCCCC при следующем режиме: первичная денатурация при 95°С – 5 мин, далее 35 циклов, включающих денатурацию при 94°С – 40 с, отжиг праймеров при 55°С – 40 с, элонгацию при 72°С – 45 с, и завершающая элонгация при 72°С – 8 мин. Продукты амплификации разделяли методом электрофореза в 2%-ном агарозном геле и окрашивали бромистым этидием. Для верификации структуры и размера амплифицируемого фрагмента каждый обнаруженный вариант ресеквенировали с той же парой праймеров. Варианты размера фрагментов рассматривали в качестве аллелей данного локуса.
Таблица 1.
Местоположение и параметры изменчивости популяций ели сибирской, исследованных с помощью митохондриального локуса mh44
| Популяция, регион | с.ш./в.д. | N | Nа | h | uh |
|---|---|---|---|---|---|
| Ильинка, Республика Алтай | 51°22′/85°10′ | 30 | 9 | 0.851 | 0.880 |
| Елбань, Новосибирская обл. | 54°18′/84°37′ | 28 | 3 | 0.457 | 0.474 |
| Майский, Хакасия | 53°36′/90°00′ | 29 | 11 | 0.756 | 0.783 |
| Азас, Тыва | 52°25′/96°33′ | 29 | 9 | 0.756 | 0.783 |
| Ергаки, Красноярский край | 52°46′/93°23′ | 28 | 3 | 0.255 | 0.265 |
| Туруханск, Красноярский край | 65°47′/87°58′ | 30 | 6 | 0.300 | 0.310 |
| Тура, Эвенкия | 64°18′/100°25′ | 30 | 5 | 0.742 | 0.768 |
| Тунгусский з-к, Эвенкия | 60°37′/101°39′ | 30 | 6 | 0.551 | 0.570 |
| Ванавара, Эвенкия | 60°21′/102°16′ | 30 | 9 | 0.616 | 0.637 |
| Кочечум, Эвенкия | 64°19′/100°14′ | 29 | 2 | 0.128 | 0.133 |
| Ольхон, Иркутская обл. | 53°15′/107°43′ | 27 | 5 | 0.326 | 0.339 |
| Косая Степь, Иркутская обл. | 52°51′/106°04′ | 29 | 9 | 0.842 | 0.872 |
| Тулюшка, Иркутская обл. | 54°27′/101°12′ | 25 | 3 | 0.486 | 0.507 |
| Выдрино, Бурятия | 51°26′/104°43′ | 58 | 9 | 0.530 | 0.539 |
| Малый Мамай, Бурятия | 51°27′/104°48′ | 30 | 9 | 0.787 | 0.814 |
| Убукун, Бурятия | 51°28′/106°43′ | 30 | 1 | 0.000 | 0.000 |
| Хамар-Дабан, Бурятия | 51°32′/105°52′ | 27 | 1 | 0.000 | 0.000 |
| Красноярово, Бурятия | 51°51′/107°16′ | 30 | 9 | 0.851 | 0.880 |
| Кабанск, Бурятия | 51°59′/106°32′ | 25 | 1 | 0.000 | 0.000 |
| Баргузин, Бурятия | 54°35′/110°47′ | 30 | 5 | 0.689 | 0.713 |
| Богд-Уул, Монголия | 47°48′/106°51′ | 30 | 7 | 0.747 | 0.772 |
| Якутск, Якутия | 62°02′/129°44′ | 30 | 1 | 0.000 | 0.000 |
| Талая, Амурская обл. | 49°47′/130°14′ | 30 | 9 | 0.860 | 0.890 |
| Магадан, Магаданская обл. | 59°39′/153°04′ | 26 | 1 | 0.000 | 0.000 |
| Среднее | 30 (1.26) |
5.54 (0.69) |
0.480 (0.006) |
0.497 (0.068) |
Примечание: N – размер выборки, Na – число выявленных аллелей, h – гаплотипическое разнообразие, uh – несмещенное гаплотипическое разнообразие [16]; в скобках – стандартная ошибка.
Для каждой популяционной выборки ели сибирской в пакете программ GenAlex 6.5 [15] определены основные параметры генетического разнообразия: Na – число выявленных аллелей, h – гаплотипическое разнообразие, uh – несмещенное гаплотипическое разнообразие [16]. Кроме того, был рассчитан индекс фиксации PhiPT [17], отражающий меру генетических различий между популяциями, и проведен анализ иерархической структуры изменчивости (AMOVA). Индекс фиксации PhiPT является аналогом индекса фиксации Fst для гаплоидных данных, но в отличие от Fst не зависит от величины внутрипопуляционной изменчивости. Ординация изученных популяций была произведена по данным РСА-анализа матрицы парных значений индексов фиксации PhiPT.
При исследовании локуса mh44 у 720 деревьев из 24 популяций, расположенных на территории, охватывающей значительную часть ареала этого вида (от 47°48′ с.ш. до 65°47′ с.ш. и от 84°37′ в.д. до 153°04′ в.д.), идентифицировано 13 аллельных вариантов, различающихся по числу повторов 32-нуклеотидного минисателлитного мотива. В соответствии с обозначением аллельных вариантов по Bastien et al. [13] изменчивость локуса mh44 у ели сибирской может быть представлена как (PaTR1)1–13 + (PaTR2b)1. Число аллелей, выявленных в отдельных популяциях, варьирует от 1 до 11, составляя в среднем 5.54 ± 0.69 (табл. 1). По одному аллелю обнаружено в популяциях ели из Магаданской области и Якутии (Магадан, Якутск), а также в трех популяциях из Бурятии (Хамар-Дабан, Убукун, Кабанск), в которых локус mh44 оказался мономорфным. Самое большое число аллелей наблюдалось в популяции Майский из Хакасии.
Наиболее распространенным оказался аллель, представленный одной копией мотива (табл. 2). Он обнаружен в 23 из 24 включенных в исследование популяций ели сибирской с частотой, варьирующей от 3 (Ильинка, Красноярово) до 100% (Магадан, Якутск, Хамар-Дабан, Убукун, Кабанск), со средним значением равным 53%. Частоты остальных аллелей (2–10) не превышали в среднем 10%, хотя в отдельных популяциях встречаемость некоторых из них достигала значительных величин. Так, например, в изолированной популяции ели с байкальского острова Ольхон частота аллеля 6, представленного шестью копиями мотива, составила 82%. В популяции Елбань наиболее частым среди выявленных аллелей был аллель 3 (68%), а в популяции Азас – аллель 8 (45%). Три аллеля (11, 12, 13) с наибольшим числом копий мотива являются редкими. Средние частоты этих аллелей равны соответственно 0.010, 0.004 и 0.003.
Таблица 2.
Частоты выявленных в локусе mh44 аллелей в изученных популяциях ели сибирской
| Популяция | Аллели | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | |
| Ильинка | 0.03 | 0.13 | 0.17 | 0.13 | 0.24 | – | 0.04 | 0.10 | – | 0.13 | 0.03 | – | – |
| Елбань | 0.29 | – | 0.68 | – | – | – | – | – | 0.03 | – | – | – | – |
| Майский | 0.45 | 0.04 | 0.14 | 0.04 | 0.04 | 0.04 | 0.10 | – | 0.04 | 0.04 | 0.07 | 0.03 | – |
| Азас | 0.07 | – | – | 0.07 | 0.10 | 0.07 | 0.07 | 0.45 | 0.10 | 0.03 | 0.03 | – | – |
| Ергаки | 0.86 | – | 0.07 | – | 0.07 | – | – | – | – | – | – | – | – |
| Туруханск | 0.84 | – | – | 0.04 | – | 0.03 | – | 0.03 | – | 0.03 | – | – | 0.03 |
| Тура | 0.33 | – | – | 0.03 | – | 0.30 | – | – | 0.17 | 0.17 | – | – | – |
| Тунгусский | 0.64 | 0.07 | – | – | – | – | – | 0.20 | 0.03 | 0.03 | – | 0.03 | – |
| Ванавара | 0.60 | – | 0.10 | 0.07 | 0.04 | 0.03 | 0.03 | 0.07 | 0.03 | 0.03 | – | – | – |
| Кочечум | 0.93 | – | – | – | – | – | 0.07 | – | – | – | – | – | – |
| Ольхон | 0.07 | – | – | – | – | 0.82 | 0.04 | 0.04 | – | 0.03 | – | – | – |
| Косая Степь | 0.07 | 0.04 | 0.21 | – | 0.10 | 0.21 | 0.03 | 0.21 | 0.10 | 0.03 | – | – | – |
| Тулюшка | 0.64 | 0.32 | 0.04 | – | – | – | – | – | – | – | – | – | – |
| Выдрино | 0.67 | 0.02 | 0.07 | 0.05 | 0.02 | 0.02 | 0.03 | – | 0.09 | – | 0.03 | – | – |
| М. Мамай | 0.40 | 0.03 | 0.10 | 0.13 | 0.10 | – | – | 0.07 | – | 0.07 | 0.07 | – | 0.03 |
| Убукун | 1.00 | – | – | – | – | – | – | – | – | – | – | – | – |
| Хамар-Дабан | 1.00 | – | – | – | – | – | – | – | – | – | – | – | – |
| Красноярово | 0.03 | 0.24 | – | 0.10 | 0.10 | 0.13 | 0.20 | 0.10 | 0.03 | 0.07 | – | – | – |
| Кабанск | 1.00 | – | – | – | – | – | – | – | – | – | – | – | – |
| Баргузин | 0.47 | – | 0.17 | – | – | – | – | 0.10 | 0.23 | – | 0.03 | – | – |
| Богд-Уул | – | – | 0.14 | 0.03 | 0.34 | 0.10 | 0.33 | – | 0.03 | – | – | 0.03 | – |
| Якутск | 1.00 | – | – | – | – | – | – | – | – | – | – | – | – |
| Талая | 0.20 | 0.07 | 0.17 | 0.03 | 0.13 | 0.17 | 0.10 | – | 0.10 | 0.03 | – | – | – |
| Магадан | 1.00 | – | – | – | – | – | – | – | – | – | – | – | – |
| Среднее | 0.53 | 0.04 | 0.09 | 0.03 | 0.05 | 0.08 | 0.04 | 0.05 | 0.05 | 0.03 | 0.01 | 0.004 | 0.003 |
Несмещенное гаплотипическое разнообразие uh локуса mh44 в изученных популяциях ели сибирской варьирует от 0 в популяциях, в которых локус мономорфен, до 0.890 в популяции Талая из Амурской области (табл. 1). Высокое гаплотипическое разнообразие, близкое к максимальному, наблюдается в популяциях Малый Мамай, Косая Степь, Ильинка, Красноярово (uh равно 0.814, 0.872, 0.880 и 0.880 соответственно). В нескольких популяциях, в частности Кочечум, Ергаки, Туруханск из Красноярского края, а также в популяции с о-ва Ольхон гаплотипическое разнообразие локуса mh44 было самым низким среди включенных в исследование популяций ели. Значение uh в этих популяциях составило соответственно 0.133, 0.265, 0.310 и 0.339. В среднем (uhср = 0.497) изученные популяции ели сибирской обнаруживают достаточно высокий уровень гаплотипического разнообразия по исследованному митохондриальному локусу.
Анализ иерархической структуры изменчивости AMOVA показал, что 25% гаплотипического разнообразия (PhiPT = 0.253, P = 0.01), выявленного при исследовании 24 популяций ели сибирской, распределяется между популяциями, а остальные 75% – внутри популяций. Полученные данные свидетельствуют о существенной межпопуляционной дифференциации популяций ели сибирской по локусу mh44. В то же время необходимо отметить, что установленное нами значение индекса фиксации PhiPT было существенно ниже значений, полученных для маркеров мтДНК при использовании другого индекса фиксации, Fst, у видов хвойных, имеющих ареалы, сходные с ареалом P. obovata: Larix sibirica (Fst = 64%) и Abies sibirica (Fst = 93%) [12, 18]. Это может быть вызвано гомоплазией минисателлитного локуса mh44, а также быстрым заселением всей исследованной части ареала P. obovata из одного источника, что не способствовало формированию резко выраженной географической структуры генетической изменчивости.
Наглядно иллюстрирует установленный уровень межпопуляционной дифференциации ели сибирской ординация популяций на плоскости двух координат, полученная по данным PCA анализа матрицы парных значений индексов фиксации PhiPT. На рис. 1 видно, что многие из включенных в исследование популяций ели сибирской пространственно удалены друг от друга. Наиболее обособлена от большинства популяций популяция ели сибирской с о-ва Ольхон, в которой преобладает аллель 6, имеющий в среднем достаточно низкую частоту встречаемости в остальных популяциях. Максимальный уровень дифференциации наблюдается между этой популяцией и популяциями Якутск и Убукун.
Рис. 1.
Ординация изученных популяций ели сибирской на плоскости двух координат по данным PCA-анализа матрицы парных значений индексов фиксации PhiPT.
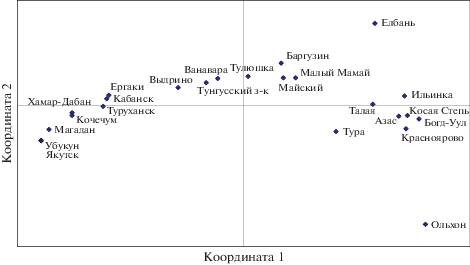
Следует, однако, отметить, что ординация популяций методом главных координат на основе парных значений PhiPT не выявила каких-либо географических закономерностей в распределении аллелей в пределах изученной части ареала ели сибирской. В связи с этим исследованный нами локус mh44 оказался непригодным для филогеографических исследований этого вида. Тем не менее благодаря своей высокой изменчивости и межпопуляционной дифференциации он может быть полезен в совокупности с другими маркерами мтДНК для исследования пространственной генетической структуры на внутри- и межпопуляционных уровнях, выявления потомства материнских деревьев и решения ряда других задач популяционной генетики, основанных на применении молекулярно-генетических маркеров.
Работа выполнена по базовому проекту № 0356-2019-0024 при частичной поддержке РФФИ (проект № 13-04-00777а).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Gugerli F., Sperisen C., Buchler U. et al. Haplotype variation in a mitochondrial tandem repeat of Norway spruce (Picea abies) populations suggests a serious founder effect during postglacial recolonization of the western Alps // Mol. Ecol. 2001. V. 10. № 5. P. 1255–1263. https://doi.org/10.1046/j.1365-294X.2001.01279.x
Ran J.-H., Wei X.-X., Wang X.-Q. Molecular phylogeny and biogeography of Picea (Pinaceae): implications for phylogeographical studies using cytoplasmic haplotypes // Mol. Phylogenet. Evol. 2006. V. 41. № 2. P. 405–419. https://doi.org/10.1016/j.ympev.2006.05.039
Tollefsrud M.M., Kissling R., Gugerli F. et al. Genetic consequences of glacial survival and postglacial colonization in Norway spruce: combined analysis of mitochondrial DNA and fossil pollen // Mol. Ecol. 2008. V. 17. № 18. P. 4134–4150. https://doi.org/10.1111/j.1365_294X.2008.03893.x
Tollefsrud M.M., Latałowa M., van der Knaap W.O. et al. Late quaternary history of North Eurasian Norway spruce (Picea abies) and Siberian spruce (Picea obovata) inferred from macrofossils, pollen and cytoplasmic DNA variation // J. Biogeogr. 2015. V. 42. № 8. P. 1431–1442.https://doi.org/10.1111/jbi.12484
Lockwood J.D., Aleksic J.M., Zou J. et al. A new phylogeny for the genus Picea from plastid, mitochondrial, and nuclear sequences // Mol. Phylogenet. Evol. 2013. V. 69. № 3. P. 717–727. https://doi.org/10.1016/j.ympev.2013.07.004
Semerikov V.L., Semerikova S.A., Putintseva Yu.A. et al. Colonization history of Scots pine in Eastern Europe and North Asia based on mitochondrial DNA variation // Tree Genetics & Genomes. 2018. V. 14. № 8. https://doi.org/10.1007/s11295-017-1222-0
Мудрик Е.А., Полякова Т.А., Шатохина А.В. и др. Пространственное распределение гаплотипов второго интрона гена nad1 в популяциях комплекса европейской и сибирской елей (Picea abies–P. obovata) // Генетика. 2015. Т. 51. № 10. С. 1117–1125. https://doi.org/10.1134/S1022795415100129
Потокина Е.К., Киселева А.А., Николаева М.А. и др. Использование маркеров органельной ДНК для анализа филогеографии восточноевропейской популяции ели европейской Picea abies (L.) H. Karst. // Вавиловский журн. генетики и селекции. 2014. Т. 18. № 4/1. С. 818–830.
Volkova P., Shipunov A., Borisova P. et al. In search of hybridity: the case of Karelian spruces // Silva Fennica. 2014. V. 48. № 2. P. 1–14. https://doi.org/10.14214/sf.1072
Семериков В.Л., Путинцева Ю.А., Орешкова Н.В. и др. Разработка новых маркеров митохондриальной ДНК сосны обыкновенной (Pinus sylvestris L.) для популяционно-генетических и филогеографических исследований // Генетика. 2015. Т. 51. № 12. С. 1386–1390. https://doi.org/10.7868/S0016675815120103
Экарт А.К., Семериков В.Л., Полежаева М.А. и др. Разработка маркеров мтДНК для популяционно-генетических исследований ели сибирской (Picea obovata Ledeb.) // Генетика популяций: прогресс и перспективы: Материалы Межд. науч. конф. (Москва, 17–21 апреля 2017 г.). М.: Ваш формат, 2017. С. 329–330.
Semerikov V.L., Semerikova S.A., Putintseva Yu.A. et al. Mitochondrial DNA in Siberian conifers indicates multiple postglacial colonization centers // Can. J. For. Res. 2019. V. 49. P. 875–883. https://doi.org/10.1139/cjfr-2018-0498
Bastien D., Favre J.M., Collignon A.M. et al. Characterization of a mosaic minisatellite locus in the mitochondrial DNA of Norway spruce [Picea abies (L.) Karst.] // Theor. Appl. Genet. 2003. V. 107. P. 574–580. https://doi.org/10.1007/s00122-003-1284-2
Devey M.E., Bell J.C., Smith D.N. et al. A genetic linkage map for Pinus radiata based on RFLP, RAPD and microsatellite markers // Theor. Appl. Genet. 1996. V. 92. P. 673–679. https://doi.org/10.1007/BF00226088
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update // Bioinformatics. 2012. № 28. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Nei M. Molecular Evolutionary Genetics. N.Y.: Columbia Univ. Press, 1987. 512 p.
Meirmans P.G. Using the AMOVA framework to estimate a standardized genetic differentiation measure // Evolution. 2006. V. 60. P. 2399–2402. https://doi.org/10.1111/j.0014-3820.2006.tb01874.x
Semerikov V.L., Semerikova S.A., Polezhaeva M.M. et al. Southern montane populations did not contribute to the recolonization of West Siberian Plain by Siberian larch (Larix sibirica): a range-wide analysis of cytoplasmic markers // Mol. Ecol. 2013. V. 22. P. 4958–4971. https://doi.org/10.1111/mec.12433
Дополнительные материалы отсутствуют.


