Генетика, 2020, T. 56, № 9, стр. 1054-1064
Анализ полиморфизма гена гефестина (HEPH) на X-хромосоме для установления принадлежности биологических образцов к диким или домашним представителям вида Sus scrofa
В. Н. Кипень 1, *, Е. В. Иванова 2, Е. В. Снытков 3, А. Н. Верчук 4
1 Институт генетики и цитологии Национальной академии наук Беларуси
220072 Минск, Республика Беларусь
2 Белорусский государственный университет
220030 Минск, Республика Беларусь
3 Международный государственный экологический институт
им. А.Д. Сахарова БГУ
220070 Минск, Республика Беларусь
4 Научно-практический центр Государственного комитета судебных экспертиз
Республики Беларусь
220114 Минск, Республика Беларусь
* E-mail: v.kipen@igc.by
Поступила в редакцию 24.09.2019
После доработки 23.10.2019
Принята к публикации 23.10.2019
Аннотация
На практическом материале (образцы дикого кабана и семь основных пород домашней свиньи, разводимых в Республике Беларусь) подтвержден высокий дифференцирующий потенциал полиморфизма H3GA0051814 (ген HEPH). Предложена тест-модель из трех SNP – H3GA0051814 (HEPH), c.367G>A (MC1R) и g.299084751C>T (NR6A1), для дифференциации дикого кабана и домашней свиньи, которая выгодно отличается от имеющихся подходов, в первую очередь своей универсальностью (подходит для дифференциации в том числе и домашних пород свиней азиатского происхождения), а также высокими точностью (99.14%) и специфичностью (100%). С использованием HRM-анализа разработан быстрый и простой подход к дифференциации на основании предложенной тест-модели, который призван сократить временны́е и финансовые затраты на молекулярно-генетическое тестирование, а также снизить риск кросс-контаминации, так как процесс является одностадийным (исключены этапы рестрикции и электрофореза).
В период 2014–2016 гг. произошла резкая депопуляция дикого кабана (Sus scrofa scrofa) на территории Республики Беларусь – с 80 тыс. особей (по состоянию на 2010 г.) до примерно 3 тыс. особей. Причина данного факта – проводимые административные меры для массового уничтожения данного вида как потенциального разносчика африканской чумы свиней. Вид Sus scrofa отличается высокой плодовитостью и через 10–15 лет будет способен восстановить свою численность до уровня, сопоставимого с показателями 2014 г., при условии отсутствия кардинальных и жестких мер административного характера.
В некоторых работах обсуждались положительные стороны и недостатки подходов, направленных на решение задачи по дифференциации дикого кабана и домашней свиньи (Sus scrofa domesticus) [1–6]. Также была показана возможность использования данных NGS-проектов для поиска “новых” генетических маркеров, в первую очередь – SNP (single nucleotide polymorphism), – обладающих высоким дифференцирующим потенциалом для различения дикого кабана и домашней свиньи, например, H3GA0051814 (rs80981028) на Х-хромосоме (Chr.X:60656889, сборка генома GCF_000000215.6) [7–9].
Также ввиду прохождения популяции дикого кабана через “бутылочное горлышко” в 2014–2016 гг., использование алгоритма для дифференциации дикого кабана и домашней свиньи, основанного на расчетах частоты встречаемости аллелей в STR-локусах, должно быть перепроверено на вновь образовавшейся популяции. Таким образом, дальнейший поиск “новых” однонуклеотидных полиморфных вариантов не потерял своего значения и является актуальной задачей. Ведь как известно при резкой значительной депопуляции вида происходит потеря в первую очередь редких аллелей, а частота самого распространенного как правило еще более возрастает. Данный факт весьма значим для SNP по причине его нахождения (более чем в 95% случаев) только в двух аллельных формах.
На данный момент основное внимание при решении задачи по дифференциации дикого кабана и домашней свиньи уделяют криминалисты при производстве экспертиз по браконьерству. В 2017 г. для реализации методики в экспертных лабораториях Государственного комитета судебных экспертиз Республики Беларусь была предложена тест-система на основе анализа двух SNP-генов MCR1 (melanocortin 1 receptor, NCBI Gene ID 494018) и NR6A1 (nuclear receptor subfamily 6 group A member 1, NCBI Gene ID 100038028) [10]. Однако при разработке данной методики ключевое внимание было уделено в первую очередь тем породам, которые наиболее распространены в свиноводческих комплексах Республики Беларусь. В то же время для пород домашних свиней азиатского происхождения, которые довольно часто встречаются на домашних подворьях, данная методика не пригодна, так как предполагается высокий уровень ложноположительных результатов (т.е. образцы домашней свиньи будут определяться как образцы дикого кабана).
Таким образом, цель данного исследования – оценить применимость полиморфизма H3GA0051814 (rs80981028) на Х-хромосоме (Chr.X:60656889), как обладающего высоким дифференцирующим потенциалом, для решения задачи по дифференциации дикого кабана и домашней свиньи в контексте имеющейся тест-системы с применением полиморфизма генов MC1R и NR6A1.
МАТЕРИАЛЫ И МЕТОДЫ
Биологические образцы. В исследование были включены следующие группы: 150 образцов биологического материала дикого кабана и 200 образцов ДНК домашней свиньи (30 – для породы белорусская крупная белая, 30 – белорусская мясная, 20 – белорусская черно-пестрая, 30 – дюрок, 30 – ландрас, 30 – йоркшир, 30 – пьетрен). Образцы диких животных были отобраны в осенне-зимний охотничий сезон 2010–2011 гг., представляли собой фрагменты мяса или ушной раковины и поступали засыпанными поваренной солью в отдельных пакетах, снабженных бирками с описанием убитого животного. Образцы ДНК домашних свиней были предоставлены Научно-практическим центром по животноводству НАН Беларуси (г. Жодино, Беларусь).
Выделение ДНК. ДНК из образцов мышечной и хрящевой ткани выделяли с использованием методики, основанной на высвобождении ДНК в ходе инкубирования образцов биологического материала в лизирующем буфере, содержащем 2% додецилсульфата натрия (SDS), 20 мM Tris-HCl, 100 мM NaCl, 20 мM EDTA (pH 8.0) и протеиназу К. При наличии фрагментов, которые подверглись лизису не полностью, осадок отделяли центрифугированием в течение 5 мин при 3000 об./мин, а супернатант переносили в новую пробирку. К супернатанту добавили 6.3 М раствор гуанидинхлорида в объеме 1/2 от объема исходной смеси, далее помещали пробирки в холодильник при 0–5°С на 10–15 мин, осадок комплекса продуктов расщепления белков с SDS удаляли центрифугированием в течение 8 мин (5000 об./мин). Далее использовали коммерческую смесь Roti®-Phenol (Германия) согласно рекомендациям производителя.
Концентрацию ДНК измеряли с использованием спектрофотометра NanoDrop-1000 (Peqlab, Германия). Среднее значение концентрации ДНК составило 89.4 ± 15.3 нг/мкл (260/280 – 1.89 ± 0.14, 260/230 – 1.82 ± 0.16).
ПЦР-ПДРФ анализ. ПЦР для генотипирования по SNP H3GA0051814 (ген HEPH) проводили в объеме 15 мкл. Количество вносимой ДНК на реакцию составляло 1.0–100.0 нг. В состав реакционной смеси входили: SynTaq-полимераза (ООО “Синтол”, РФ); 10× ПЦР-буфер (100 мМ Трис-HCl, 500 мМ KCl, 0.8% Nonidet P40, pH 8.8), 50 мМ раствора MgCl2 (финальная концентрация ионов Mg2+– 3.0 мМ); 2.0 мМ раствора смеси дНТФ; смесь прямого и обратного праймеров в концентрации 5.0 мкМ (F 5'-GGTAGATGTGGGTGGGATGGA-3'; R 5'-AACTATTGGATCTGCATCCGCT-3'). В результате ПЦР синтезировался ампликон длиной 360 пн. Полиморфизм H3GA0051814 (ген HEPH) исследовали с применением эндонуклеазы рестрикции NdeI (NEB, США). Протоколы генотипирования и результаты молекулярно-генетического анализа SNP c.367G>A (ген MC1R) и g.299084751C>T (ген NR6A1) с использованием ПЦР-ПДРФ представлены в работе В.Н. Кипень с соавт. [11].
KASP-генотипирование. Для подтверждения результатов генотипирования полиморфизма H3GA0051814 (ген HEPH), проведенных с использованием ПЦР-ПДРФ, была использована технология, основанная на конкурентной аллель-специфической ПЦР (KASP, kompetitive allele specific PCR). Генотипирование проводилось для 45 образцов с использованием KASP Assay mix (KASP by Design, KBD) и KASP Master mix (ООО “Максим Медикал”, РФ, LGC Biosearch Technologies, Великобритания) в трехкратной повторности согласно рекомендациям производителя. Концентрация ДНК для всех образцов была стандартизирована до 30 нг/мкл. Для определения нижнего предела детектируемого количества генетического материала в реакции ПЦР была использована серия разведений ДНК для девяти образцов с известным генотипом. Диапазон тестируемого количества ДНК в реакции ПЦР составил 1.0–50.0 нг/реакцию – концентрации 1.0, 2.0, 5.0, 10, 20, 50 нг/мкл.
HRM-анализ. Генотипирование SNP H3GA0051814 (ген HEPH), c.367G>A (ген MC1R) и g.299084751C>T (ген NR6A1) проводилось с использованием Precision Melt Supermix for High Resolution Melt (HRM) Analysis (Bio-Rad, США) в трехкратной повторности согласно рекомендации производителя. Результаты аллельной кластеризации были проанализированы с использованием Precision Melt Analysis™ Software v. 1.3 (Bio-Rad, США). Концентрация ДНК для всех образцов была стандартизирована до 30 нг/мкл. Протокол амплификации соответствовал рекомендуемым Bio-Rad. Для HRM-анализа были использованы оригинальные праймеры: для SNP H3GA0051814 (ген HEPH) – F 5'-TTCTGTTGTACAGCAAAGTGACCC-3' и R 5'-TATATTTGGAAGAGATAGGCAATGGG-3', для SNP c.367G>A (ген MC1R) – F 5'-GACACCATGGAGCCGCAGATG-3' и R 5'-CGTGGTGCAGCAGCTGGAC-3', для SNP g.299084751C>T (ген NR6A1) – F 5'-GGAACAGGGCTTCAGAGAGC-3' и R 5'-GCTCACCTGGAGGACAGTGTGG-3' (ЗАО “Евроген” и ООО “Биотех-Индустрия”, РФ).
Определение генотипа in silico. Генотипы in silico для животных, геномы которых были представлены в открытом доступе в NCBI (National Center for Biotechnology Information), определяли с использованием on-line алгоритма SRA-Blast (файлы в формате *.sam) и программы Unipro UGENE v. 1.31.1 [12]. Определение генотипа для трех SNP в генах HEPH, MC1R и NR6A1 с использованием алгоритма BLAST было проведено для 70 особей домашней свиньи, “сырые” данные полных геномов которых расположены в Sequence Read Archive (SRA, https://www.ncbi.nlm.nih.gov/sra). В табл. 1 суммирована информация для образцов с указанием “Sample ID” и генотипами. Используемые настройки поиска генотипа in silico отражены в работе Е.В. Ивановой [13].
Таблица 1.
Генотипы домашних свиней азиатского происхождения по трем SNP в генах HEPH, MC1R и NR6A1
| Порода (Sample ID) |
c.367G>A (MC1R) |
g.299084751 (NR6A1) |
H3GA0051814 (HEPH) |
Порода (Sample ID) |
c.367G>A (MC1R) |
g.299084751 (NR6A1) |
H3GA0051814 (HEPH) |
|---|---|---|---|---|---|---|---|
| Bamei* (SAMN04440479) |
GG | CC | CC | Meishan (SAMN06560428) |
GG | TT | CC |
| Domestic pig, Asia (SAMN02460622) |
GG | TT | CC | Meishan (SAMN06562578) |
GG | TT | CC |
| Domestic pig, Asia# (SAMN02460623) |
AG | TT | CC | Meishan (SAMN06562589) |
GG | TT | CC |
| Domestic pig, Asia# (SAMN02460625) |
AG | TT | CC | Meishan* (SAMN06562956) |
GG | CT | CC |
| Domestic pig, Asia* (SAMN02460626) |
GG | CC | CC | Meishan* (SAMN06562959) |
GG | CT | CC |
| Domestic pig, Asia* (SAMN02460627) |
AG | CC | CC | Meishan (SAMN06562971) |
GG | TT | CC |
| Ganzi Tibetan pig* (SAMN01894346) |
GG | CC | CC | Meishan* (SAMN06562977) |
GG | CT | CC |
| Ganzi Tibetan pig* (SAMN01894361) |
GG | CC | CC | Meishan* (SAMN06562978) |
GG | CT | CC |
| Jeju native# (SAMN03160627) |
AG | TT | CC | Meishan* (SAMN06562984) |
GG | CC | CC |
| Jeju native# (SAMN03160628) |
AG | TT | CC | Meishan (SAMN06563583) |
GG | TT | CC |
| Jeju native# (SAMN03160629) |
GG | TT | CC | Meishan* (SAMN06563661) |
GG | CT | CC |
| Jeju native# (SAMN03160630) |
AG | TT | CC | Meishan (SAMN06564163) |
GG | TT | CC |
| Jeju native# (SAMN03160631) |
AG | TT | CC | Meishan (SAMN06579151) |
GG | TT | CC |
| Jeju native# (SAMN03160632) |
AG | TT | CC | Meishan (SAMN06579317) |
GG | TT | CC |
| Jeju native (SAMN03160633) |
AA | TT | CC | Meishan* (SAMN06579320) |
GG | CT | CC |
| Jeju native# (SAMN03160634) |
AG | TT | CC | Meishan* (SAMN06603310) |
GG | CT | CC |
| Jiangquhai* (SAMEA3497797) |
AG | CC | CC | Meishan (SAMN06603321) |
GG | TT | CC |
| Jinhua* (SAMEA3497793) |
GG | CC | CC | Meishan* (SAMN06603369) |
GG | CT | CC |
| Jinhua* (SAMEA3497794) |
GG | CC | CC | Meishan* (SAMN06603407) |
GG | CT | CC |
| Korean native# (SAMN03160621) |
AG | TT | CC | Meishan* (SAMN06603626) |
GG | CT | CC |
| Korean native# (SAMN03160622) |
AG | TT | CC | Meishan (SAMN06603627) |
GG | TT | CC |
| Korean native# (SAMN03160623) |
AG | TT | CC | Meishan* (SAMN06606917) |
GG | CT | CC |
| Korean native (SAMN03160624) |
GG | TT | CC | Meishan* (SAMN06607518) |
GG | CT | CC |
| Korean native# (SAMN03160625) |
AG | TT | CC | Meishan* (SAMN06610205) |
GG | CT | CC |
| Korean native (SAMN03160626) |
GG | TT | CC | Meishan* (SAMN06610393) |
GG | CT | CC |
| Meishan* (SAMEA3497800) |
GG | CT | CC | Meishan# (SAMN07344467) |
AG | CT | CC |
| Meishan# (SAMEA3497801) |
AG | CT | CC | Neijiang* (SAMN01894448) |
GG | CC | CC |
| Meishan* (SAMEA3497802) |
GG | CC | CC | Rongchang* (SAMN03331745) |
GG | CC | CC |
| Meishan* (SAMEA3497803) |
GG | CC | CC | Rongchang* (SAMN04440482) |
GG | CC | CC |
| Meishan# (SAMEA3497804) |
AG | TT | CC | Tibetan pig* (SAMN01894367) |
GG | CC | CC |
| Meishan* (SAMEA3497805) |
GG | CC | CC | Tibetan pig* (SAMN01894369) |
GG | CC | CC |
| Meishan* (SAMEA3497806) |
GG | CC | CC | Tibetan pig* (SAMN01894370) |
GG | CC | CC |
| Meishan* (SAMEA3497807) |
GG | CC | CC | Wannan Spotted* (SAMEA3497811) |
GG | CC | CC |
| Meishan* (SAMEA3497808) |
AG | CC | CC | Xiang* (SAMEA3497812) |
GG | CC | CC |
| Meishan* (SAMN06556517) |
GG | CC | CC | Zang* (SAMEA3497814) |
GG | CT | CC |
п/ж * – особи, которые попадают в группу “Дикий кабан”, хотя по факту являются домашними свиньями (по результатам генотипирования только MC1R и NR6A1). курсив # – особи, отнести которые не представляется возможным ни в одну группу по существующей схеме (по результатам генотипирования только MC1R и NR6A1).
Статистический анализ данных. Оценку дифференцирующего потенциала для SNP или их совокупности проводили с использованием программы MDR v. 3.0.2 (http://www.multifactordimensionalityreduction.org/). В процессе моделирования нами были использованы высоко консервативные настройки поиска конфигурации модели, которые позволили однозначно дифференцировать наличие/отсутствие статистически значимых эффектов, а именно: количество атрибутов (attribute count range) – от 1 до n (где n – количество переменных в модели); воспроизводимость модели (cross-validation count) – 100; анализ топ-моделей (track top models) – 1000; поиск конфигурации модели (search method configuration) – всесторонний (exhaustive); метод сравнения (ambiguous cell analysis) – точный тест Фишера (Fisher’s exact test); классификация ячеек (ambiguous cell assignment) – неклассифицированные (unclassified).
Вклад конкретного генотипа определялся величиной энтропии Н (выраженной в %). При H = 100% генотип способен однозначно дифференцировать, к какой группе (дикий кабан или домашняя свинья) относится неизвестный образец. В программе MDR в результате множественных перестановок (пермутаций) первичных данных определяется наиболее оптимальная модель дифференциации. Корректность модели оценивали по значению сбалансированной точность (Balanced Accuracy), которая зависит от чувствительности и специфичности модели [14].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
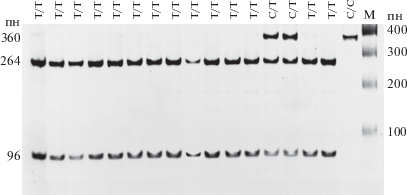
В результате генотипирования 150 образцов из выборки дикого кабана по полиморфизму H3GA0051814 гена HEPH были получены следующие результаты: генотип TT выявлен в 84.0% (126/150) случаев, генотип СТ – в 12.7% (19/150) случаев и генотип СС – в 3.3% (5/150) случаев. Таким образом, для включенных в исследование образцов дикого кабана показано наличие трех генотипов для полиморфизма H3GA0051814, при этом аллель Т распространен в данной выборке с частотой 90.3 ± 2.4%, т.е. является мажорным. Результаты электрофоретического разделения рестриктов представлены на рис. 1. Для всех животных пород домашних свиней в рамках данного исследования (n = 200) определен генотип СС, аллель С распространен в данной выборке с частотой 100%.
Рис. 1.
Результаты электрофоретического разделения рестриктов для полиморфизма H3GA0051814 гена HEPH в 8.0%-ном ПААГ. М – маркер молекулярного веса (Jena Bioscience M-214S).
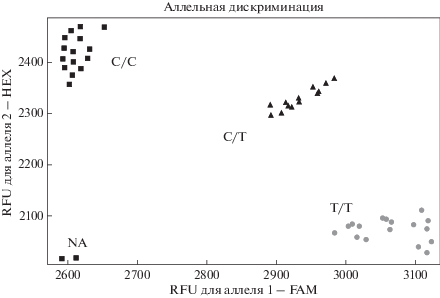
Для подтверждения результатов генотипирования полиморфизма H3GA0051814, проведенных с использованием ПЦР-ПДРФ, а также для возможности предоставить потенциальному кругу исследователей более разнообразный спектр методов, была использована технология KASP. Генотип был корректно определен после основного и дополнительного раундов амплификации для 45 образцов (15 образцов домашних свиней и 30 – диких кабанов), рис. 2.
Рис. 2.
2D-plot результатов аллельной дискриминации при генотипировании полиморфизма H3GA0051814 гена HEPH с использованием KASP-технологии (Bio-Rad CFX Manager, Bio-Rad, США). NA – отрицательный контроль.
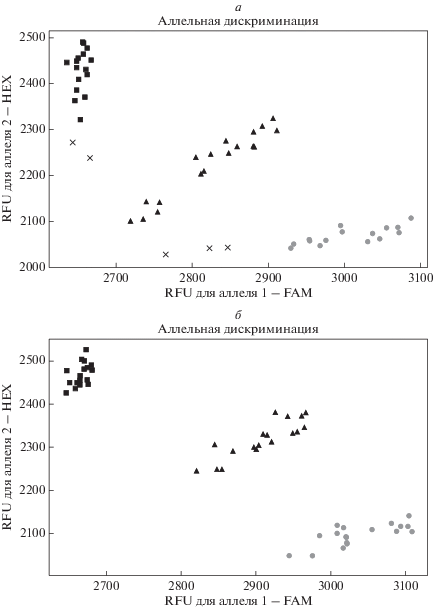
Для определения нижнего предела детектируемого количества генетического материала в реакции ПЦР была использована серия разведений ДНК для девяти образцов с известным генотипом. Диапазон тестируемого количества ДНК в реакции ПЦР составил 1.0–50.0 нг/реакцию: концентрации 1.0, 2.0, 5.0, 10, 20, 50 нг/мкл. Результат генотипирования в виде графика аллельной дискриминации после основного раунда амплификации представлен на рис. 3,а. Для тех случаев, когда после основного раунда амплификации не представилось возможным определить генотип, производитель реагентов для KASP рекомендует провести раунд дополнительной амплификации. Результат генотипирования в виде графика аллельной дискриминации после дополнительного раунда амплификации представлен на рис. 3,б. Минимальное достаточное количество ДНК для корректного определения генотипа составило не менее 1.0 нг/реакцию при 42 циклах амплификации. Чувствительность и специфичность при генотипировании полиморфизма H3GA0051814 с использованием технологии KASP составила 100%.
Рис. 3.
2D-plot результатов аллельной дискриминации при генотипировании полиморфизма H3GA0051814 гена HEPH с использованием KASP-технологии на основании серии разведения ДНК (Bio-Rad CFX Manager, Bio-Rad, США). Квадраты, треугольники, кружки, крестики – различные генотипы.
Оценку дифференцирующего потенциала для полиморфизма H3GA0051814 (ген HEPH) проводили с использованием программы MDR v. 3.0.2. Точность дифференциации дикого кабана от домашней свиньи при анализе полиморфизма H3GA0051814 гена HEPH составляет 98.33%. Ни в одном случае образец домашней свиньи не был принят за дикого кабана – специфичность модели оказалась равной 100%. Таким образом, при наличии генотипов СТ или ТТ происхождение неизвестного образца с точностью не менее 99.99% (уровень статистической значимость p < 0.01) должно быть отнесено к дикому кабану (Sus scrofa scrofa). При наличии генотипа СС происхождение неизвестного образца с точностью не менее 99.99% (уровень статистической значимость p < 0.01) должно быть отнесено к домашней свинье (Sus scrofa domesticus).
Как акцентировалось ранее, на данный момент задача по дифференциации дикого кабана и домашней свиньи решается с использованием анализа двух SNP – c.367G>A (ген MC1R) и g.299084751C>T (ген NR6A1) [10].
Однако в Республике Беларусь на частных подворьях все интенсивнее происходит разведение домашних свиней азиатского происхождения, например вьетнамской вислобрюхой свиньи или мейшан и др. По гену окраски кожи и волосяного покрова для них характерен генотип по SNP c.367G>A (ген MC1R), идентичный для дикого кабана, так как имеет место темный окрас. Также по причине большего породного разнообразия среди домашних свиней в странах Азиатского региона остается неопределенной частота распространенности аллеля С для полиморфизма g.299084751C>T (ген NR6A1), характерного для дикого кабана. В данном контексте, т.е. только при анализе полиморфных вариантов c.367G>A (ген MC1R) и g.299084751C>T (ген NR6A1), как нам представляется, имеется довольно высокая вероятность ложноположительных результатов дифференциации, т.е. мясо домашней свиньи может быть ошибочно идентифицировано как мясо дикого кабана. Негативные последствия подобной ложной дифференциации трудно переоценить, тем более в контексте криминалистики.
В NCBI имеются геномы в формате SRA для домашних свиней азиатского происхождения следующих пород: Аба (A’ba Tibetan pig), Бамей (Bamei), Дянь Сяо Эр (Diannanxiaoer), Азиатская домашняя (Domestic pig, Asia), Тибетская Ганзи (Ganzi Tibetan pig), Чеджу (Jeju native), Чжан Вэй (Jiangquhai), Цзиньхуа (Jinhua), Корейская (Korean native), Мейшан (Meishan). С использованием биоинформационной обработки данных определен генотип для трех SNP – H3GA0051814 (ген HEPH), c.367G>A (ген MC1R) и g.299084751С>T (ген NR6A1) – для 70 животных домашней свиньи, табл. 1.
Таким образом, по данным генотипов c.367G>A (ген MC1R) и g.299084751С>T (ген NR6A1), согласно методике [10], из 70 домашних свиней, имеющих азиатское происхождение, 57.14% (40/70) отнесены к группе “Дикий кабан”, т.е. имеет место неправильная дифференциация (отмечены в табл. 1 как *). Для 21.43% (15/70) случаев не представилось возможным отнести особь ни к какой группе – ни к “Домашней свинье”, ни к “Дикому кабану” (отмечены в табл. 1 как #). В то же время, для всех особей домашних свиней азиатского происхождения для полиморфизма H3GA0051814 (ген HEPH) был определен генотип CC, т.е., как и ранее было показано на образцах домашних свиней из Беларуси, для домашних свиней азиатского происхождения был показан один генотип. Таким образом, возможное решение сложившейся ситуации – введение в существующую схему (включающую два SNP – c.367G>A (ген MC1R) и g.299084751C>T (ген NR6A1)) третьего SNP – H3GA0051814 (ген HEPH), дифференцирующий потенциал которого мы оценили в рамках данной работы.
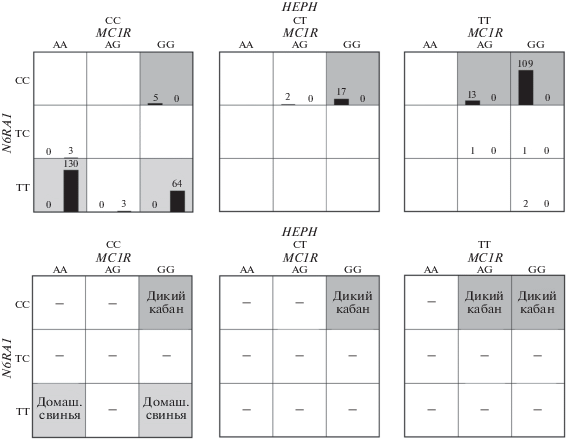
На основании результатов генотипирования по полиморфизму H3GA0051814 (HEPH) в двух выборках – “Дикий кабан” (n = 150) и “Домашняя свинья” (n = 200), а также учитывая результаты, представленные в работе В.Н. Кипень с соавт. по полиморфным вариантам c.367G>A (MC1R) и g.299084751C>T (NR6A1) [11], с использованием MDR-анализа была охарактеризована модель по дифференциации дикого кабана от домашней свиньи с использованием трех SNP, рис. 4. Точность дифференциации дикого кабана от домашней свиньи при анализе полиморфизма трех SNP – H3GA0051814 (HEPH), c.367G>A (MC1R) и g.299084751C>T (NR6A1) – составила 99.14%. Ни в одном случае образец домашней свиньи не был принят за дикого кабана – специфичность модели оказалась равной 100%.
Рис. 4.
Графическое представление модели из трех ОНП в генах HEPH, MC1R и NR6A1 для дифференциации дикого кабана и домашней свиньи, адаптировано из ПО MDR v. 3.0.2 (абсолютные значения в любой клетке для первого столбца соответствуют количеству образцов дикого кабана, для второго образца – количеству образцов домашней свиньи).
Далее мы апробировали метод HRM для генотипирования полиморфных вариантов H3GA0051814 (HEPH), c.367G>A (MC1R) и g.299084751C>T (NR6A1). Цель этапа – дать исследователю простой, точный и доступный метод генотипирования в контексте описанной выше модели. Для деградированных образцов ДНК, как нам представляется, данный способ будет весьма актуален.
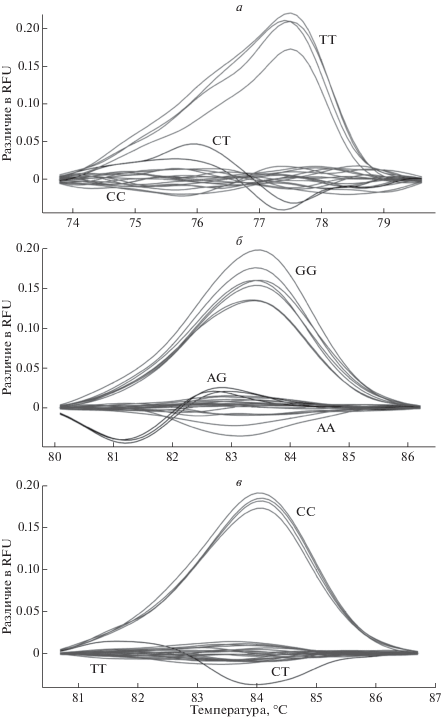
Генотипирование трех SNP проводилось с использованием Precision Melt Supermix for High Resolution Melt (HRM) Analysis (Bio-Rad, США) в трехкратной повторности. Результаты аллельной кластеризации были подтверждены с использованием ПЦР-ПДРФ и KASP. Результаты кластеризации для полиморфизма H3GA0051814 (HEPH) отражены на рис. 5,а, для SNP c.367G>A (MC1R) – рис. 5,б, для g.299084751C>T (NR6A1) – рис. 5,в.
Рис. 5.
Результат кластеризации генотипов в Precision Melt Analysis™ Software v. 1.3 (Bio-Rad, США) для полиморфизма: а – H3GA0051814 (HEPH); б – c.367G>A (MC1R); в – g.299084751C>T (NR6A1).
В рамках проведенного исследования решен ряд задач, направленных на повышение точности и специфичности имеющейся тест-системы (основанной на анализе двух SNP в генах MC1R и NR6A1) по дифференциации дикого кабана и домашней свиньи. Так, с использованием биоинформационной обработки данных определен SNP c высоким дифференцирующим потенциалом для различения дикого кабана и домашней свиньи – H3GA0051814 (HEPH), а также на практическом материале (образцы дикого кабана и семь основных пород домашней свиньи, разводимых в Республике Беларусь) подтвержден высокий дифференцирующий потенциал данного полиморфизма. Полученные с использованием метода ПЦР-ПДРФ результаты генотипирования SNP H3GA0051814 подтверждены с использованием технологии KASP. Дополнительно установлено, что полиморфизм H3GA0051814 также способен дифференцировать породы домашних свиней азиатского происхождения от особей дикого кабана. На этом основании предложена новая тест-модель из трех SNP – H3GA0051814 (HEPH), c.367G>A (MC1R) и g.299084751C>T (NR6A1), – точность которой составляет 99.14% при 100% специфичности. С использованием технологии HRM предложен легкий в исполнении, точный полуколичественный метод для генотипирования данных трех SNP.
Предложенная нами схема по дифференциации дикого кабана и домашней свиньи с использованием именно трех SNP выгодно отличается от имеющихся подходов, в первую очередь своей универсальностью (подходит для дифференциации в том числе и домашних пород свиней азиатского происхождения), а также высокими точностью и специфичностью. Анализ с использованием HRM призван сократить временные и финансовые затраты на молекулярно-генетическое тестирование, а также снизить риск кросс-контаминации, т.к. процесс является одностадийным (исключены этапы рестрикции и электрофореза).
Исследование выполнено при частичной финансовой поддержке Программы научных исследований на 2016–2020 гг., проект “Разработка методики ДНК-дифференциации биологических образцов животных вида кабан европейский (дикий и домашний)”.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Кипень В.Н., Рябцева А.О., Котова С.А. Цыбовский И.С. Современные подходы к дифференциации кабана европейского (Sus scrofa scrofa) и его домашней разновидности (Sus scrofa domesticus) с использованием молекулярно-генетических методов // Вопр. криминологии, криминалистики и суд. экспертизы. 2016. № 2(40). С. 127–134.
Brodmann P. Identifying unknown game species: Experience with nucleotide sequencing of the mitochondrial cytochrome b gene and a subsequent basic local alignment search tool search // Europ. Food Res. and Techn. 2001. V. 213(3). P. 491–496.
Maede D. A strategy for molecular species detection in meat and meat products by PCR-RFLP and DNA sequencing using mitochondrial and chromosomal genetic sequences // Europ. Food Res. and Techn. 2006. V. 224(3). P. 209–217. https://doi.org/10.1007/s00217-006-0320-2
Montiel-Sosa J. Direct and highly species-specific detection of pork meat and fat in meat products by PCR amplification of mitochondrial DNA // J. Agricultural Food Chem. 2000. V. 48(7). P. 2829–2832. https://doi.org/10.1021/jf9907438
Pascoal A. Survey of authenticity of meat species in food products subjected to different technological processes, by means of PCR-RFLP analysis // Europ. Food Res. and Techn. 2004. V. 218(1). P. 306–312.
Ramirez O. Integrating Y-chromosome, mitochondrial and autosomal data to analyse the origin of pig breeds // Mol. Biol. Evol. 2009. V. 26. P. 2061–2072. https://doi.org/10.1093/molbev/msp118
Кипень В.Н. Использование полногеномных данных проектов NGS для поиска решения криминалистической задачи по дифференциации диких кабанов и домашних свиней на основе анализа SNP // Молекулярная диагностика. 2017: IX Всероссийская научно-практическая конференция, 18–20 апреля 2017 г. Сборник материалов. Москва: 2017. С. 305–306.
Ramos A.M. Design of a high density SNP genotyping assay in the pig using SNPs identified and characterized by next generation sequencing technology // PLoS One. 2009. V. 4(8). e6524. P. 1–13. https://doi.org/10.1371/journal.pone.0006524
Ramos A.M. Identification of high utility SNPs for population assignment and traceability purposes in the pig using high-throughput sequencing // Anim. Genet. 2011. V. 42(6). P. 613–620. https://doi.org/10.1111/j.1365-2052.2011.02198.x
Котова С.А., Кипень В.Н., Рябцева А.О. и др. Методика установления принадлежности биологических образцов к диким или домашним представителям вида кабан европейский методом ПЦР-ПДРФ / Под ред. Цыбовского И.С. Минск: Право и экономика, 2017. 22 с. ISBN:978-985-552-751-1 (https://e-catalog.nlb.by/Record/BY-NLB-br0001453546).
Кипень В.Н., Рябцева А.О., Котова С.А. и др. Оценка интрогрессии генов свиньи домашней (Sus scrofa domesticus) в генофонд дикого кабана (Sus scrofa scrofa) на основе исследования полиморфизма генов MC1R и NR6A1 // Мол. и прикл. генет. 2019. Т. 26. С. 83–95.
Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: A unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. P. 1166–1167. https://doi.org/10.1093/bioinformatics/bts091
Иванова Е.В. Особенности определения генотипа для полиморфных вариантов, обладающих дифференцирующей способностью для различения Sus scrofa scrofa и Sus scrofa domesticus // Геномное секвенирование 2017: 5-я Всероссийская научно-практическая конференция по геномному секвенированию и редактированию, 18 мая 2017 г. Сборник тезисов ФГБУН Институт биоорганической химии им. академиков М.М. Шемякина и Ю.А. Овчинникова. Москва: 2017. С. 29.
Ritchie M.D., Hahn L.W., Roodi N. et al. Multifactor–dimensionality reduction reveals high–order interactions among estrogen–metabolism genes in sporadic breast cancer // Am. J. Hum. Genet. 2001. V. 69. № 1. P. 138–147. https://doi.org/10.1086/321276
Дополнительные материалы отсутствуют.


