Генетика, 2020, T. 56, № 9, стр. 1018-1033
Вариабельность генома островов патогенности нетоксигенных штаммов Vibrio cholerae O1 биовара Эль Тор
Н. И. Смирнова 1, *, А. А. Крицкий 1, Ж. В. Альхова 1, Е. Ю. Агафонова 1, Е. Ю. Щелканова 1, Д. В. Баданин 1, В. В. Кутырев 1
1 Российский научно-исследовательский противочумный институт “Микроб”
410005 Саратов, Россия
* E-mail: rusrapi@microbe.ru
Поступила в редакцию 23.09.2019
После доработки 31.10.2019
Принята к публикации 13.11.2019
Аннотация
Представлены результаты сравнительного анализа нуклеотидных последовательностей островов патогенности (ОП) VPI-1 и VPI-2 43 штаммов нетоксигенных холерных вибрионов. Установлено, что они, отличаясь друг от друга по составу и структуре генома этих ОП, образуют три генетически разные группы (ctxA−tcpA+VPI-2+, ctxA−tcpA+VPI-2del+ и ctxA−tcpA−VPI-2del+), различающиеся по адаптивному потенциалу. Обнаружено, что уровень вариабельности генома ОП, обусловленной точечными мутациями и делециями разной протяженности, был неодинаков. Нуклеотидная последовательность структурных и регуляторных генов патогенности VPI-1, присутствующего в хромосоме нетоксигенных вибрионов ctxA−tcpA+, была в основном идентична таковой референсного токсигенного штамма. Было выявлено лишь небольшое число полиморфных сайтов (5) с единичными нуклеотидными заменами в генах acfB, асfС, tcpА, tcpF, tcpH. В то же время нестабильность генома VPI-2, проявляющаяся в образовании делеций разной длины (31 131–49 986 тпн) и возникновении множественных однонуклеотидных замен во всех сохранившихся генах, была весьма выраженной. Впервые установили, что потеря ДНК VPI-2 коррелировала с появлением множественных замен в его генах утилизации аминосахаров и сиаловых кислот, локализованных в области nan-nag (57–274 замен), включая ген nanH. Обнаружено 12 ранее не описанных аллельных вариантов гена nanH. Большое генетическое разнообразие этих генов, значимых для выживания вибрионов в разных экологических нишах, является, видимо, одной из возможных причин высокого адаптивного потенциала V. choleraе О1 биовара Эль Тор сtxA−tcpA−VPI-2del+, широко распространенных в водной среде. На основе анализа нуклеотидных последовательностей гена nanH из VPI-2, а также шести генов “домашнего хозяйства” проведен кластерный анализ, определивший филогенетические связи разных групп нетоксигенных штаммов между собой и с токсигенными. Обнаружена генетическая близость нетоксигенных вибрионов ctxA−tcpA+VPI-2+ и ctxA−tcpA+VPI-2del+ с возбудителем холеры. Полученные ранее неизвестныe сведения о структуре ОП нетоксигенных вибрионов не только способствуют более глубокому пониманию молекулярных механизмов возникновения генетически разнообразных штаммов с разным адаптивным потенциалом. Нетоксигенные штаммы с установленной структурой генома, не требующие удаления при его редактировании ключевых генов вирулентности, могут быть востребованы при создании нового поколения живых холерных вакцин.
Холерные вибрионы относятся к виду Vibrio choleraе, который состоит из более 200 О-серологических групп. Среди них только V. choleraе O1-серогруппы классического и Эль Тор биоваров стали причиной семи пандемий холеры, унесших миллионы человеческих жизней [1]. Вместе с тем биовар Эль Тор генетически неоднороден и включает холерные вибрионы с разным патогенным потенциалом – как токсигенные эпидемически опасные штаммы, вызвавшие текущую, седьмую, пандемию холеры (c 1961 г. по настоящее время), так и нетоксигенные изоляты, которые могут вызывать единичные случаи, а также локальные вспышки острых кишечных инфекций (ОКИ) [2–5]. Разные патогенные свойства токсигенных и нетоксигенных штаммов V. cholerae O1 биовара Эль Тор обусловлены содержанием в их геноме различных наборов генов вирулентности. В связи с широким распространением гетерогенных популяций нетоксигенных штаммов в водных экосистемах России, несущих в геноме разные гены патогенности, существует немалый риск возникновения диарейных заболеваний различной сложности, что требует постоянного мониторинга за этим возбудителем и оценки его патогенного потенциала.
Секвенирование полного генома V. cholerae O1 биовара Эль Тор показало присутствие в клетках двух кольцевых хромосом (большой и малой), в состав которых, помимо генов “домашнего хозяйства” (housekeеping genes), входят различные мобильные генетические элементы (МГЭ) [6]. Патогенность возбудителя холеры является полидетерминантным свойством, в реализации которого принимают участие многие гены. В геноме токсигенных штаммов ключевые гены вирулентности, эпидемичности (пандемичности) и персистенции размещены на различных МГЭ. Среди них наиболее значимыми для возбудителя холеры являются профаг CTXφ, острова патогенности (ОП) VPI-1 и VPI-2 (от Vibrio pathogenicity island), острова пандемичности VSP-I и VSP-II (от Vibrio seventh pandemic island), остров персистенции в окружающей среде EPI (от environmental persistence island) [7–10]. К генам, абсолютно необходимым для развития инфекционного процесса при холере, относятся кластер генов tcpA–tcpF, входящий в состав ОП VPI-1 и определяющий биосинтез токсин-корегулируемых пилей (или TCP от toxin-coregulated pilus), а также гены ctxAB, размещенные в геноме профага CTXφ и кодирующие холерный токсин (или CT от cholera toxin) [11–14]. Продукты этих генов обеспечивают реализацию двух основных этапов инфекционного процесса – колонизацию патогеном тонкого кишечника в организме хозяина и развитие острой профузной диареи.
Принципиальное отличие нетоксигенных вибрионов V. cholerae O1 биовара Эль Тор, циркулирующих в России, от токсигенных – отсутствие в их геноме профага CTXφ с генами CT и островов пандемичности VSP-I и VSP-II [15]. Что касается островов патогенности, то геномы разных популяций нетоксигенных штаммов различались между собой по их содержанию. Нетоксигенные вибрионы с ОП VPI-1 выделяются довольно редко, тогда как большинство вибрионов лишены этого участка ДНК [3, 4, 15, 16]. Второй ОП – VPI-2, впервые обнаруженный лишь у токсигенных штаммов, впоследствии был выявлен и у нетоксигенных вибрионов [15–19].
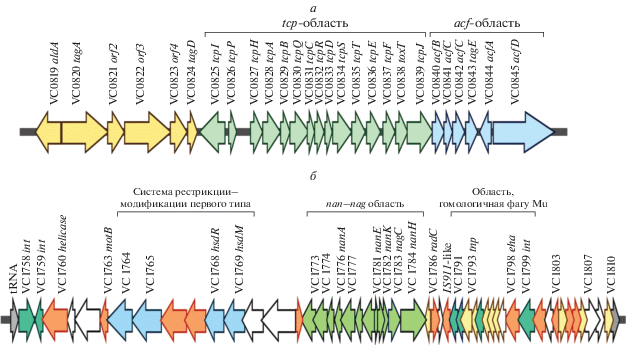
Роль ОП в патогенезе холеры, вызываемой токсигенными холерными вибрионами, хорошо изучена. Установлено, что функциональная значимость VPI-1 (41.3 тпн) состоит не только в том, что входящие в его состав гены tcpA–F кодируют TCP, необходимые для колонизации вибрионами тонкого кишечника. Эти пили служат также в качестве рецептора для фага вирулентности CTXφ, что определяет возможность формирования токсигенных клонов из нетоксигенных за счет фаговой конверсии [3, 20]. Кроме того, геном VPI-1 содержит гены, кодирующие дополнительные факторы патогенности, а также регуляторные гены, контролирующие экспрессию различных генов вирулентности на уровне транскрипции (рис. 1,а) [12, 21–23]. Геном VPI-2 (57.3 тпн) включает в себя три основные области ДНК, содержащие гены, связанные с системой рестрикции–модификации первого типа (VC1764–VC1769), с метаболизмом сиаловых кислот (VC1773–VC1784), а также гены, гомологичные генам фага Mu (VC1791–VC1799) (рис. 1,б). Среди них наиболее значимыми для выживания вибрионов в разных экологических нишах (в водной среде и организме человека) являются гены утилизации аминосахаров и сиаловых кислот, используемых в качестве альтернативного источника углерода и азота [17, 18, 24]. Нейраминидаза, кодируемая геном nanH (VC1784), наиболее важна для проявления вирулентных свойств. В кишечнике человека этот фермент, расщепляя полисиалоганглиозиды до моносиалоганглиозида GM1, являющегося рецептором для холерного токсина, усиливает его действие [18]. В то же время до сих пор отсутствует полное представление о структуре и функции островов патогенности нетоксигенных штаммов, различающихся между собой по наличию ОП VPI-1 и относящихся к разным генотипам (ctxA−tcpA+ и ctxA−tcp−). Вместе с тем в нуклеотидной последовательности их ОП могут возникать разные мутации, вызванные действием стрессовых факторов водной среды, которые могут влиять на патогенные и адаптивные свойства различных популяций. В этой связи представлял интерес сравнительный анализ структуры ОП нетоксигенных штаммов для оценки их вариабельности и поиск факторов, определяющих разный адаптивный потенциал вибрионов с различным генотипом. Ранее мы секвенировали полные геномы ряда нетоксигенных штаммов и выявили наличие делеций в VPI-2 [25]. Однако расширенный анализ этих мутаций и их распространенность среди разных популяций нетоксигенных вибрионов, циркулирующих в водоемах России, до сих пор не проведен. Кроме того, нет единого мнения о происхождении нетоксигенных вибрионов с разным составом ОП.
Настоящая работа посвящена изучению вариабельности нуклеотидной последовательности ОП VPI-1 и VPI-2 нетоксигенных штаммов V. cholerae O1 биовара Эль Тор разных генотипов и выяснению их филогенетических связей между собой.
МАТЕРИАЛЫ И МЕТОДЫ
Штаммы V. cholerae, условия культивирования. В работе использовали 53 токсигенных и нетоксигенных штамма V. cholerae О1 биовара Эль Тор, выделенных от больных и из воды открытых водоемов (табл. 1). Штаммы получали из Государственной коллекции патогенных бактерий РосНИПЧИ “Микроб”. Бактерии выращивали в бульоне и на агаре Luria–Bertani (LB) при 37°C в течение 18–20 ч. Для анализа генома ОП использовали нуклеотидные последовательности полных геномов исследуемых штаммов, определенных нами (51 штамм) или взятых из базы данных NCBI GenBank (табл. 1).
Таблица 1.
Штаммы Vibrio cholerae O1 биовара Эль Тор, полногеномные последовательности которых использованы в работе
| Штамм | Место и год выделения | Генотип | № доступа в GenBank |
|---|---|---|---|
| М988 | Туркмения, 1965 | ctxA– tcpA– | LQBX00000000* |
| 114 | Украина, 1999 | ctxA– tcpA– | NDXP00000000* |
| М1332 | Россия, Челябинск, 2000 | ctxA– tcpA– | PYCE00000000* |
| М1337 | Россия, Астрахань, 2000 | ctxA– tcpA– | NEEB00000000* |
| Р18748 | Россия, Сочи, 2004 | ctxA– tcpA– | NIFH00000000* |
| 102 | Украина, 2006 | ctxA– tcpA– | NDXO00000000* |
| М1457 | Россия, Калмыкия, 2009 | ctxA– tcpA– | VTLH00000000* |
| М1467 | Россия, Калмыкия, 2010 | ctxA– tcpA– | VTLI00000000* |
| 5U | Украина, 2011 | ctxA– tcpA– | NDXM00000000* |
| М1506 | Россия, Калмыкия, 2011 | ctxA– tcpA– | VTZX00000000* |
| М1516 | Россия, Калмыкия, 2012 | ctxA– tcpA– | VTZY00000000* |
| М1517 | Россия, Калмыкия, 2012 | ctxA– tcpA– | VTZZ00000000* |
| М1526 | Россия, Калмыкия, 2012 | ctxA– tcpA– | VUAA00000000* |
| 29 | Россия, Калмыкия, 2013 | ctxA– tcpA– | VUAB00000000* |
| 132 | Россия, Калмыкия, 2013 | ctxA– tcpA– | VUAC00000000* |
| М1522 | Россия, Татарстан, 2014 | ctxA– tcpA– | LQCA00000000* |
| 8 | Россия, Калмыкия, 2014 | ctxA– tcpA– | PYCF00000000* |
| 433 | Россия, Сочи, 2015 | ctxA– tcpA– | NEDW00000000* |
| 2843 | Россия, Калмыкия, 2016 | ctxA– tcpA– | PYCG00000000* |
| 3178 | Россия, Калмыкия, 2017 | ctxA– tcpA– | PYCH00000000* |
| 120 | Россия, Калмыкия, 2018 | ctxA– tcpA– | VTLJ00000000* |
| 136 | Россия, Калмыкия, 2018 | ctxA– tcpA– | VTLK00000000* |
| 2439 | Россия, Калмыкия, 2018 | ctxA– tcpA– | VTLL00000000* |
| 3017 | Россия, Калмыкия, 2018 | ctxA– tcpA– | VTLM00000000* |
| М139 | Туркмения, 1965 | ctxA– tcpA+ | LRMU00000000* |
| М299 | Туркмения, 1965 | ctxA– tcpA+ | LQYZ00000000* |
| М1395 | Россия, Астрахань, 1981 | ctxA– tcpA+ | LQBY00000000* |
| М1399 | Россия, Астрахань, 1982 | ctxA– tcpA+ | LQBZ00000000* |
| 85U | Украина, 1995 | ctxA– tcpA+ | NEDU00000000* |
| Р18778 | Россия, Ростов-на-Дону, 2005 | ctxA– tcpA+ | NIFI00000000* |
| М1501 | Россия, Калмыкия, 2011 | ctxA– tcpA+ | LRAE00000000* |
| М1504 | Россия, Калмыкия, 2011 | ctxA– tcpA+ | VTLN00000000* |
| М1505 | Россия, Калмыкия, 2011 | ctxA– tcpA+ | VTLO00000000* |
| М1507 | Россия, Калмыкия, 2011 | ctxA– tcpA+ | VTLP00000000* |
| М1518 | Россия, Калмыкия, 2012 | ctxA– tcpA+ | LQZR00000000* |
| 111 | Россия, Калмыкия, 2012 | ctxA– tcpA+ | VTLQ00000000* |
| М1519 | Россия, Калмыкия, 2012 | ctxA– tcpA+ | VUAD00000000* |
| М1527 | Россия, Калмыкия, 2012 | ctxA– tcpA+ | VUAE00000000* |
| М1524 | Россия, Калмыкия, 2013 | ctxA– tcpA+ | LQZS00000000* |
| 2613 | Россия, Калмыкия, 2015 | ctxA– tcpA+ | PYCA00000000* |
| 2687 | Россия, Калмыкия, 2015 | ctxA– tcpA+ | PYCB00000000* |
| 2688 | Россия, Калмыкия, 2015 | ctxA– tcpA+ | PYCC00000000* |
| 124 | Россия, Калмыкия, 2017 | ctxA– tcpA+ | PYCD00000000* |
| N16961 | Бангладеш, 1975 | ctxA+ tcpA+ | AE003852/AE003853** |
| М1293 | Россия, Дагестан, 1994 | ctxA+ tcpA+ | JFFW00000000* |
| Р17644 | Россия, Ачинск, 1997 | ctxA+ tcpA+ | JRTW00000000* |
| CIRS 101 | Бангладеш, 2002 | ctxA+ tcpA+ | CWPG00000000** |
| М1429 | Россия, Башкортостан, 2004 | ctxA+ tcpA+ | LAEM00000000* |
| Р18899 | Россия, Мурманск, 2006 | ctxA+ tcpA+ | LAKM00000000* |
| Л3226 | Россия, Москва, 2010 | ctxA+ tcpA+ | JDVX00000000* |
| КМ76 | Украина, 2011 | ctxA+ tcpA+ | MPVL00000000* |
| М1509 | Россия, Москва, 2012 | ctxA+ tcpA+ | NEDZ00000000* |
| 3265-80 | Россия, Москва, 2015 | ctxA+ tcpA+ | JRQL00000000* |
Выделение и очистку геномной ДНК проводили из бактериальной суспензии с использованием коммерческого набора QIAamp DNA mini Kit (Qiagen, Германия) в соответствии с протоколом производителя и методическими указаниями [26]. Клетки предварительно были обработаны мертиолятом натрия до конечной концентрации 1 : 10 000 (0.01%) и прогреты при 56°С в течение 30 мин.
Полногеномное секвенирование, сборка и аннотация. Полногеномное секвенирование проводили на приборе “Ion PGM” (Thermo Fisher Scientific, США) по технологии ионного полупроводникового секвенирования. Первичную обработку данных выполняли с помощью пакета программ Ion Torrent Suite Software версии 5.10.1 (Thermo Fisher Scientific). Сборку полногеномных последовательностей de novo осуществляли при помощи программы Newbler gs Assembler версии 2.6 (454 Life Sciences, США).
Идентификацию геномных островов проводили путем картирования полученных полногеномных последовательностей на референсную последовательность штамма N16961 (номер доступа в базе данных GenBank AE003852/AE003853) с помощью пакета программ DNASTAR Lasergene версии 11.2 (DNASTAR Inc., США).
Определение полиморфизма исследуемых генов, включая количество аллелей, индекс полиморфизма локусов, среднее число синонимичных/ несинонимичных замен на синонимичный/несинонимичный сайт, проводили при помощи программ MEGA версии 6.0 (Megasoftware, США) и DNASequence Polymorphism версии 5.10.01 (Университет Барселоны, Испания).
Мультилокусное сиквенс-типирование проводили на основе анализа нуклеотидных последовательностей шести генов “домашнего хозяйства” (cat, chi, dnaE, gyrB, pgm, recA), взятых из секвенированных последовательностей полных геномов исследуемых штаммов. Размеры генов, выбранных для анализа, составили: cat – 2175 пн (позиция в геноме 1671041–1673215), chi – 2550 пн (позиция в геноме 33035–35584), dnaE – 3495 пн (позиция в геноме 2397732–2401226), gyrB – 2418 пн (позиция в геноме 11103–13520), pgm – 1647 пн (позиция в геноме 2256154–2257800), recA – 1065 пн (позиция в геноме 574696–575760). Определение аллелей данных генов осуществляли при помощи онлайн сервиса PubMLST (https://pubmlst.org/).
Филогенетический анализ токсигенных и нетоксигенных штаммов осуществляли последовательно, предварительно выравнивая нуклеотидные последовательности генов в соответствии с алгоритмом MAFFT, с дальнейшим построением филогенетических деревьев по методу Байеса в программе Ugene версии 1.32 (UniPro, Россия), используя модель нуклеотидных замен GTR с гамма-коррекцией.
РЕЗУЛЬТАТЫ
Сравнение генома острова патогенности VPI-1 токсигенных и нетоксигенных штаммов V. cholerae О1 биовара Эль Тор
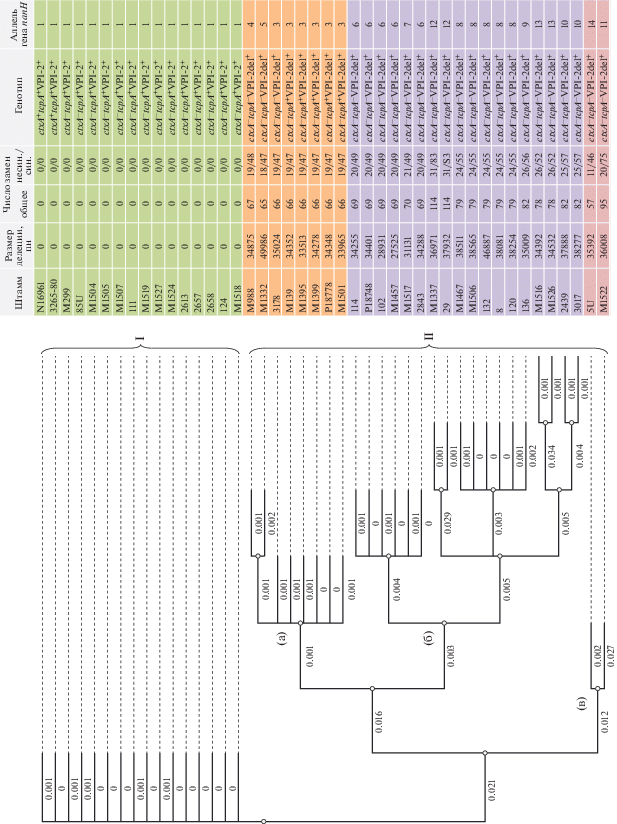
Согласно результатам сравнительного биоинформационного анализа все исследованные нетоксигенные штаммы ctxA−tcpA+ (19 изолятов) содержали в геноме весь блок генов, входящих в состав VPI-1 (VC0819–VC0845), который имел тот же самый сайт внедрения в хромосому, что и в референсном токсигенном штамме N16961. В то же время нуклеотидная последовательность отдельных генов VPI-1 нетоксигенных изолятов отличалась от таковой этих же генов референсного штамма. Так, при сопоставлении последовательностей генов, наиболее важных для проявления патогенных свойств, выявили следующее. В нуклеотидной последовательности генов acfB и асfС, входящих в кластер генов acf, кодирующих компоненты дополнительного фактора колонизации ACF (от accessory colonization factor) (рис. 1,а), присутствовали полиморфные позиции (730 и 378), в которых находились транзиции C→T и G→A соответственно (табл. 2). Вместе с тем краевой ген acfD из этого же кластера не содержал никаких полиморфных сайтов и его последовательность была идентична таковой референсного штамма. Такая же картина была характерна и для другого краевого гена aldA, кодирующего фермент альдегиддегидрогеназу (рис. 1). Что касается центрального фрагмента VPI-1, включающего кластер генов tcp, состоящий из ключевых структурных и регуляторных генов вирулентности, то среди изученных нуклеотидных последовательностей генов tcpI, tcpF, tcpP, tcpH, toxT мы выявили единичные полиморфные сайты, содержащие точечные замены, лишь в генах tcpF и tcpH (табл. 2). Следует особо отметить присутствие одной и той же замены C→G в позиции 146 в регуляторном гене tcpH у 13 штаммов из 20 изученных, выделенных в основном в одном и том же регионе – Республике Калмыкия. Полиморфные локусы в этом же гене у других штаммов, изолированных на территории Туркмении, Астрахани, Ростова-на-Дону, отсутствовали (табл. 1, 2). Это означает, что нетоксигенные штаммы из различных популяций, циркулирующих в разных регионах, могут отличаться друг от друга полиморфизмом нуклеотидных последовательностей одних и тех же генов. Отдельный интерес вызывал сравнительный анализ нуклеотидной последовательности гена tcpA, кодирующего основную субъединицу TCP – ключевого фактора колонизации, поскольку ранее среди нетоксигенных штаммов V. cholerae О1 биовара Эль Тор ctxA−tcpA+, выделенных в Китае из различных объектов окружающей среды, впервые были выявлены изоляты с разными аллелями этого гена – аллелем tcpAET, идентичным референсному токсигенному штамму N16961, и аллелем tcpAETvar, отличающимся от первого наличием 26 синономичных и несинонимичных однонуклеотидных замен [16]. Однако практически все изученные штаммы имели тот же аллель гена tcpA, что и возбудитель холеры, а именно tcpAET. Только у одного изолята (М1501) в tcpA обнаружили единичную точечную мутацию (трансверсию A→T) в положении 626. Поиски аллеля tcpAETvar среди сравниваемых штаммов были безуспешными (табл. 2). Замены оснований в генах tcpА, tcpF, tcpH и acfB сопровождались заменой соответствующей аминокислоты, однако эти замены, видимо, не влияли на функциональные свойства белков. Таким образом, уровень вариабельности генома ОП VPI-1 нетоксигенных штаммов ctxA−tcpA+ оказался невысоким. Среди десяти генов, расположенных в разных фрагментах его генома и наиболее значимых для проявления патогенных свойств, мы обнаружили лишь пять полиморфных сайтов в генах кластеров acf и tcp, содержащих единичные однонуклеотидные замены трансверсии и транзиции. При этом у всех исследуемых штаммов, независимо от года и географического региона выделения, точечные мутации в измененных генах tcpF, acfB и асfС были локализованы в одних и тех же позициях. Основные структурные и регуляторные гены вирулентности tcpА, tcpP и toxT были в целом идентичны таковым референсного штамма.
Таблица 2.
Позиции однонуклеотидных замен в генах VPI-1 нетоксигенных штаммов Vibrio cholerae О1 биовара Эль Тор ctxA–tcpA+
| Штамм | Гены VPI-1 и полиморфные позиции | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| aldA 1512 пн | tcpA* 675 пн | tcpF* 1017 пн | tcpI 1863 пн | tcpP 666 пн | tcpH* 411 пн | toxT 1512 пн | acfB* 1881 пн | acfC** 762 пн | acfD 4563 пн | |
| М139 | 0 | 0 | 842*G→A | 0 | 0 | 0 | 0 | 730 C→T | 378 G→A | 0 |
| М299 | 0 | 0 | 842G→A | 0 | 0 | 0 | 0 | 730C→T | 378G→A | 0 |
| М1395 | 0 | 0 | 842G→A | 0 | 0 | 0 | 0 | 730C→T | 378G→A | 0 |
| М1399 | 0 | 0 | 842G→A | 0 | 0 | 0 | 0 | 730C→T | 378G→A | 0 |
| 85U | 0 | 0 | 842G→A | 0 | 0 | 146 C→G | 0 | 730C→T | 378G→A | 0 |
| Р18778 | 0 | 0 | 842G→A | 0 | 0 | 0 | 0 | 730C→T | 378G→A | 0 |
| М1501 | 0 | 626А→Т | 842G→A | 0 | 0 | 0 | 0 | 730C→T | 378G→A | 0 |
| М1504 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| М1505 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| М1507 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| М1518 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| 111 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| М1519 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| М1527 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| М1524 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| 2613 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| 2687 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| 2688 | 0 | 0 | 842G→ A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
| 124 | 0 | 0 | 842G→A | 0 | 0 | 146C→G | 0 | 730C→T | 378G→A | 0 |
Нестабильность острова патогенности VPI-2 нетоксигенных штаммов
Проведенный нами анализ нуклеотидных последовательностей полных геномов 43 нетоксигенных и 10 токсигенных, включая референсный штамм (табл. 1), показал, что VPI-2, кодирующий 52 ORFs, у всех сравниваемых штаммов включен в один и тот же хромосомный сайт – в ген tRNAser. Однако нетоксигенные штаммы оказались гетерогенными по структуре генома этого мобильного элемента. Среди 19 штаммов ctxA−tcpA+, имеющих в хромосоме VPI-1, у 14 (или 73.7%) нуклеотидная последовательность этого ОП была идентична таковой токсигенных штаммов. В то же время у пяти штаммов (или 26.3%) были полностью утрачены два его фрагмента – содержащий гены системы рестрикции и модификации типа I (VC1759–VC1772) и гены, гомологичные генам фага Mu (VC1792–VC1803). Общая протяженность делеции составила 33 965–38 426 пн. Сохранились лишь краевые гены (VC1757–VC1758 и VC1805–VC1810) и гены центрального фрагмента (VC1773–VC1790), включающего ген nanH, кодирующий нейраминидазу, и прилежащую к нему область nan–nag (рис. 2). Этот ОП с редуцированным геномом обозначили как VPI-2del. Иную картину наблюдали при анализе нуклеотидной последовательности VPI-2 у нетоксигенных штаммов ctxA−tcрA−, лишенных VPI-1 (изучено 24 изолята). В отличие от штаммов с VPI-1 частота утраты фрагментов VPI-2 в этой группе составляла 100%. Это означает, что все изученные штаммы имели VPI-2del. При этом у разных штаммов размеры утраченного участка ДНК были неодинаковы. Наиболее протяженные делеции VPI-2 (47 120–49 986 тпн), захватывающие все три основные районы VPI-2, включая центральный участок ДНК с генами nan–nag, были обнаружены у трех штаммов (М1332, 132 и 433), выделенных в разные годы (2000–2015 гг.). Сохранились лишь единичные краевые гены. Однако даже при такой масштабной перестройке генома ОП у двух штаммов не был утрачен ген nanH (2423 пн). Только у штамма 433 в гене nanH была протяженная делеция размером 2311 пн, что составляло 95.4% от размера нативного гена nanH (рис. 2). Делетируемые участки VPI-2 других штаммов отличались друг от друга по длине, которая варьировала от 31 131 до 38 730 тпн вследствие полной или частичной утраты двух больших фрагментов ДНК, содержащих гены рестрикции и модификации, а также гены фагового происхождения. Вместе с тем в геноме этих штаммов делеции не затронули практически все гены, входящие в состав центральной области nan–nag, включая ген нейраминидазы (рис. 2).
Рис. 2.
Структура острова патогенности VPI-2 токсигенных и нетоксигенных штаммов Vibrio cholerae О1 биовара Эль Тор, выделенных на территории России и сопредельных стран. Схема составлена на основе анализа нуклеотидных последовательностей генов VPI-2 указанных штаммов. В графе “Штамм” зеленым цветом выделены токсигенные штаммы ctxA+tcpA+VPI-2+, включая референсный, коричневым – нетоксигенные штаммы ctxA–tcpA+VPI-2del; голубым – нетоксигенные штаммы ctxA–tcpA–VPI-2del. Под рисунком приведен процент гомологии нуклеотидной последовательности генов с таковой референсного штамма N16961.
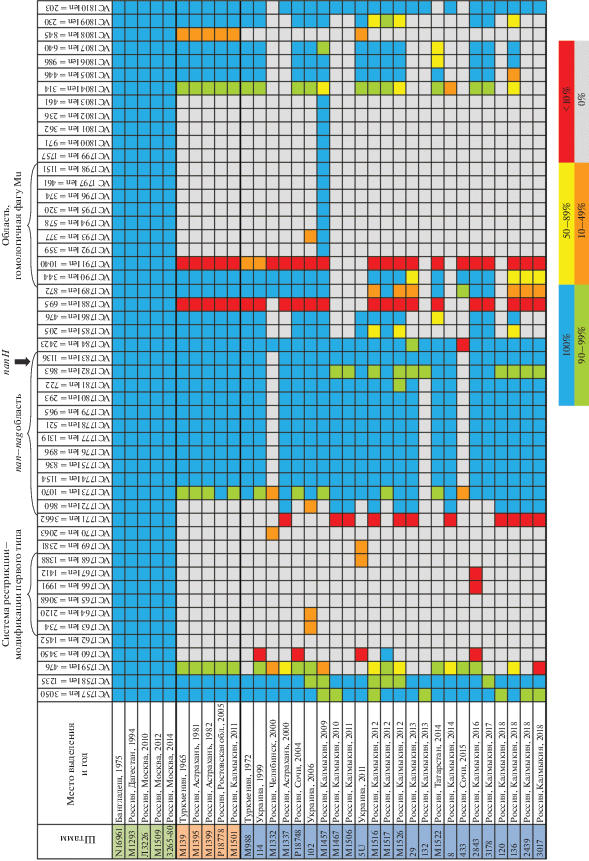
Поскольку сохранение 11 генов из области nan–nag почти во всех нетоксигенных штаммах с делетированным VPI-2, возможно, было связано с их функциональной значимостью для клетки, мы сравнили их нуклеотидную последовательность с таковой 10 токсигенных штаммов (табл. 1). Оказалось, что эти гены вибрионов c интактным геномом VPI-2 были идентичны таковым токсигенных. В отличие от них у всех нетоксигенных штаммов с делетированным геномом VPI-2 во всех генах этой области, включая nanH, были обнаружены множественные нуклеотидные замены (от 57 до 274). Детальный анализ нуклеотидной последовательности гена nanH выявил в нем 57–114 полиморфных сайтов, содержащих 47–83 синонимичных и 11–31 несинонимичных однонуклеотидных замен (табл. 3). Из них 66 полиморфных сайтов были идентифицированы среди штаммов ctxA−tcpA+, тогда как у штаммов ctxA−tcрA− их число варьировало в пределах 57–114. Этo означает, что появление точечных мутаций в гене nanH коррелировало с возникновением крупных делеционных перестроек в геноме VPI-2. Таким образом, при биоинформационном анализе полногеномных последовательностей ДНК 43 нетоксигенных штаммов впервые выявлена выраженная нестабильность генома VPI-2, проявляющаяся в образовании делеций разной длины, что сопровождалось возникновением множественных однонуклеотидных замен во всех генах области nan–nag, включая ген nanH. Сравнение последовательностей VPI-2 нетоксигенных штаммов с генотипами ctxA−tcpA+ и ctxA−tcрA− показало, что одним из их отличий является наиболее частая утрата последним участков ДНК этого ОП.
Таблица 3.
Полиморфные сайты гена nanH токсигенных и нетоксигенных штаммов V. cholerae О1 биовара Эль Тор
| Генотип штамма | Число штаммов | Число полиморфных сайтов | Нуклеотидные замены в полиморфных сайтах | Kn* | Ks** | Kn/Ks | Аллель гена nanH | |
|---|---|---|---|---|---|---|---|---|
| несинонимич-ные (n) | cинонимичные (s) | |||||||
| ctxA+tcpA+ VPI-2+ | 10 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ctxA–tcpA+ VPI-2+ | 14 | 0 | 0 | 0 | 0 | 0 | 0 | 1 |
| ctxA–tcpA+ VPI-2del+ | 5 | 66 | 19 | 47 | 0.010 | 0.092 | 0.109 | 3 |
| ctxA–tcpA– VPI-2del+ | 1 | 66 | 19 | 47 | 0.010 | 0.092 | 0.109 | 3 |
| ctxA–tcpA– VPI-2del+ | 1 | 67 | 19 | 48 | 0.010 | 0.094 | 0.106 | 4 |
| ctxA–tcpA– VPI-2del+ | 1 | 65 | 18 | 47 | 0.010 | 0.092 | 0.109 | 5 |
| ctxA–tcpA– VPI-2del+ | 5 | 69 | 20 | 49 | 0.011 | 0.096 | 0.114 | 6 |
| ctxA–tcpA– VPI-2del+ | 1 | 70 | 21 | 49 | 0.012 | 0.096 | 0.125 | 7 |
| ctxA–tcpA– VPI-2del+ | 5 | 79 | 24 | 55 | 0.013 | 0.109 | 0.119 | 8 |
| ctxA–tcpA– VPI-2del+ | 1 | 82 | 26 | 56 | 0.015 | 0.111 | 0.135 | 9 |
| ctxA–tcpA– VPI-2del+ | 2 | 82 | 25 | 57 | 0.014 | 0.113 | 0.124 | 10 |
| ctxA–tcpA– VPI-2del+ | 1 | 95 | 20 | 75 | 0.011 | 0.154 | 0.071 | 11 |
| ctxA–tcpA– VPI-2del+ | 2 | 114 | 31 | 83 | 0.017 | 0.172 | 0.099 | 12 |
| ctxA–tcpA– VPI-2del+ | 2 | 78 | 26 | 52 | 0.014 | 0.103 | 0.136 | 13 |
| ctxA–tcpA– VPI-2del+ | 1 | 57 | 11 | 46 | 0.006 | 0.090 | 0.067 | 14 |
Аллельное разнообразие гена nanH нетоксигенных штаммов
Детальный анализ нуклеотидных последовательностей гена nanH нетоксигенных штаммов указывал на присутствие у них аллельных вариантов этого гена, сходство которых с аллелем токсигенных штаммов (nanH1) составляло 96.3–97.9%. При сравнении последовательности nanH среди 42 штаммов обнаружили 13 его аллелей, из которых 12 были выявлены впервые. Частота их встречаемости была разной. Наиболее распространенным оказался аллель nanH1. Этот аллель, присущий токсигенным штаммам, был идентифицирован у всех нетоксигенных штаммов ctxA−tcpA+ (14 изолятов) с интактным VPI-2. Сравнительно высокую частоту распространения имели также аллели nanH3, nanH6 и nanH8, среди которых первый был обнаружен у всех штаммов ctxA−tcpA+VPI-2del+. Другие аллели (nanH4, nanH5, nanH7, nanH9–nanH14) были выявлены в одном или двух случаях (табл. 3). Ранее идентифицированный у нетоксигенных штаммов аллель nanH2 [27] не был найден среди изученных изолятов. Таким образом, полученные данные показали достаточно высокую консервативность гена nanH нетоксигенных штаммов ctxA−tcpA+ с интактным VPI-2 и его идентичность этому гену токсигенных штаммов. В отличие от них нуклеотидная последовательность гена nanH нетоксигенных штаммов c редуцированным геномом VPI-2 (ctxA−tcpA+VPI-2del+ и ctxA−tcpA−VPI-2del+) была весьма вариабельна.
Филогенетический анализ VPI-2 нетоксигенных штаммов
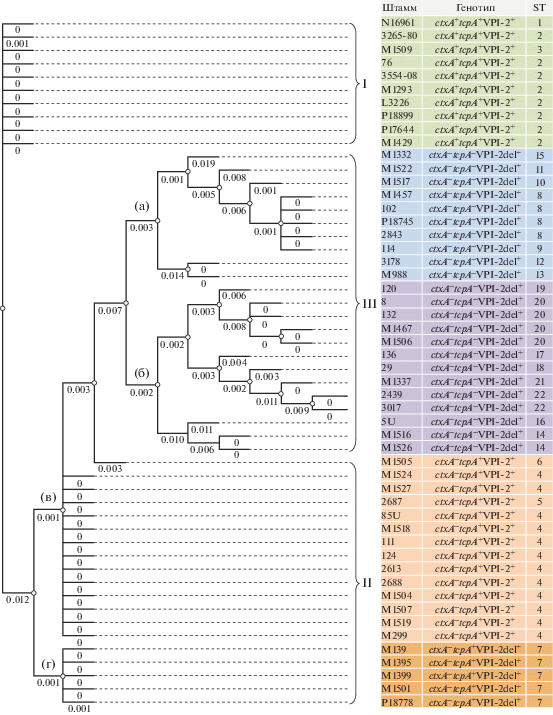
Далее, для выяснения изменений структуры VPI-2 в процессе эволюции холерных вибрионов мы построили дендрограмму по результатам сравнительного анализа нуклеотидных последовательностей его гена nanH у двух токсигенных и 42 нетоксигенных штаммов разных генотипов. В результате показали ее четкое разделение на два основных кластера (рис. 3). Первый из них содержал токсигенные штаммы ctxA+tcpA+, а также 14 нетоксигенных ctxA−tcpA+, имеющих VPI-1 и интактный VPI-2 с аллелем nanH1 в его составе. Второй кластер был представлен 28 нетоксигенными штаммами разных генотипов (ctxA−tcpA+, ctxA−tcрA−), имеющими существенные отличия от штаммов первого кластера – наличие делеции разной длины в VPI-2 и множественных мутаций в гене nanH. Этот кластер включал, по крайней мере, три подгруппы (а, б, в), штаммы которых имели 12 других аллелей nanH, отличающихся от аллеля nanH1 присутствием 57–114 полиморфных сайтов. Интерес представлял тот факт, что подгруппа “а”, содержащая пять штаммов ctxA−tcpA+, в геноме которых присутствовали VPI-1 и VPI-2del, филогенетически оказалась наиболее близкой к нетоксигенным штаммам с VPI-1 и интактным VPI-2 из кластера I. Это может означать, что указанные штаммы, выделенные в разное время (1965–2005 гг.) и имеющие аллель nanH3, были производными нетоксигенных штаммов ctxA−tcpA+ с интактным VPI-2, сформированными в результате утраты ими больших участков ДНК этого ОП. Отличия штаммов из других подгрупп кластера II от большинства изолятов подгруппы “а” определялись отсутствием VPI-1 и увеличением числа полиморфных сайтов в гене nanH (рис. 3). Несмотря на большую вариабельность гена nanH, являющегося частью VPI-2, представленные данные позволяют говорить о следующих этапах преобразования этого ОП в процессе эволюции. Выявленная филогенетическая близость нетоксигенных штаммов ctxA−tcpA+VPI-2+ с токсигенными может указывать на существование общей предковой формы, содержащей в геноме VPI-I и VPI-2. Дивергенция токсигенных штаммов от нетоксигенных ctxA−tcpA+VPI-2+ была связана с приобретением первыми профага CTXφ и островов пандемичности VSP-I и VSP-II в результате горизонтального переноса генов. Дальнейший этап – дивергенция нетоксигенных вибрионов с другими генотипами (ctxA−tcpA+VPI-2del+ и ctxA−tcрA−VPI-2del+) от нетоксигенных ctxA−tcpA+VPI-2+ коррелирует с несколькими событиями – как с сохранением VPI-1, но редукцией VPI-2, так и с последующей утратой VPI-1 и накоплением мутаций в сохранившихся генах VPI-2del.
Рис. 3.
Филогенетическое дерево Vibrio cholerae О1 биовара Эль Тор по данным байесовского анализа нуклеотидных последовательностей гена nanH из VPI-2 двух токсигенных ctxA+tcpA+VPI-2+ и 42 нетоксигенных штаммов с разным генотипом: ctxA–tcpA+VPI-2+, ctxA–tcpA+VPI-2del и ctxA–tcpA–VPI-2del. В основании ветвей указаны генетические расстояния, рассчитанные при помощи параметрической модели нуклеотидных замен GTR (General Time Reversible).
Филогенетический анализ токсигенных и нетоксигенных штаммов методом MLST
Для подтверждения представленных выше филогенетических связей различных групп нетоксигенных штаммов между собой и с токсигенными провели мультилокусное сиквенс-типирование (MLST) на основе анализа нуклеотидной последовательности шести генов (dnaE, gyrB, recA, pgm, cat, chi), локализованных в коровой части генома и кодирующих наиболее важные для обеспечения жизнедеятельности клетки белки (табл. 4). В результате анализа их аллельного профиля среди 52 изученных штаммов выявили 22 сиквенс-типа (ST) (табл. 4). Далее на основе байесовского анализа их нуклеотидных последовательностей провели реконструкцию филогенетического дерева токсигенных и нетоксигенных штаммов. Анализ дендрограммы филогенетического родства (рис. 4) показал, что исследуемые штаммы образовали три основных кластера, коррелирующих с их генотипом. Первый кластер включал все токсигенные штаммы (ctxA+tcpA+), имеющие в 80.0% случаев идентичный сиквенс-тип (ST2) и содержащие в геноме оба ОП – VPI-1 и VPI-2. Филогенетически наиболее близкими к ним были нетоксигенные штаммы ctxA−tcpA+, сохранившие VPI-1 и вошедшие в состав кластера II. Однако эти штаммы оказались генетически неоднородными и, различаясь между собой по структуре VPI-2, образовали две подгруппы – “в” и “г”. В состав подгруппы “в” вошли все штаммы ctxA−tcpA+ с интактным VPI-2, у 85.7% которых был один и тот же ST (4), в подгруппу “г” – штаммы ctxA−tcpA+ с VPI-2del (ST7). Отражением дальнейших преобразований нетоксигенных вибрионов стали штаммы ctxA−tcpA−, распределившиеся в кластер III, особенностью которых было отсутствие в их геноме VPI-1, но наличие у всех VPI-2del. В этот кластер входили, по крайней мере, две подгруппы (“а” и “б”), отличительным признаком которых были другие ST-типы – ST8–ST22 (рис. 4). Таким образом, согласно полученным данным популяционная структура нетоксигенных вибрионов включает три основные группы, относящиеся к разным филогенетическим линиям: ctxA−tcpA+VPI-2+, ctxA−tcpA+VPI-2del+ и ctxA−tcрA−VPI-2del+. Различия в структуре их генома отражают, видимо, разные этапы их эволюции. Первый – дивергенция от общей предковой формы токсигенных и нетоксигенных вибрионов. Наиболее близкими к токсигенным оказались нетоксигенные вибрионы ctxA−tcpA+VPI-2+ и ctxA−tcpA+VPI-2del+. Следующий этап эволюции нетоксигенных вибрионов, возможно, связан с утратой ими ОП VPI-1 и дальнейшей реорганизацией генома VPI-2.
Таблица 4.
Аллельный полиморфизм генов “домашнего хозяйства” токсигенных и нетоксигенных штаммов V. cholerae О1 биовара Эль Тор и их сиквенс-типы
| Штаммы | Присутствие мобильных генетических элементов вирулентности | Аллельные профили генов “домашнего хозяйства”* | Число штаммов с указанным аллельным профилем | ST-тип | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| CTXφ | VPI-1 | VPI-2 | cat | chi | dnaE | gyrB | pgm | recA | |||
| Токсигенные | + | + | + | 1; | 1; | 1; | 1; | 1; | 1; | 1 | 1 |
| + | + | + | 1; | 1; | 1; | 1; | 1; | 2; | 8 | 2 | |
| + | + | + | 1; | 1; | 1; | 1; | 24; | 2; | 1 | 3 | |
| Нетоксигенные | – | + | + | 1; | 4; | 1; | 5; | 1; | 6; | 12 | 4 |
| – | + | + | 1; | 4; | 1; | 5; | 5; | 6; | 1 | 5 | |
| – | + | + | 1; | 4; | 80; | 5; | 1; | 6; | 1 | 6 | |
| Нетоксигенные | – | + | VPI-2del | 1; | 4; | 1; | 5; | 1; | 64; | 5 | 7 |
| Нетоксигенные | – | – | VPI-2del | 6; | 5; | 5; | 6; | 6; | 7; | 4 | 8 |
| – | – | VPI-2del | 6; | 5; | 5; | 7; | 6; | 8; | 1 | 9 | |
| – | – | VPI-2del | 79; | 5; | 77; | 6; | 6; | 51; | 1 | 10 | |
| – | – | VPI-2del | 89; | 5; | 4; | 6; | 61; | 65; | 1 | 11 | |
| – | – | VPI-2del | 145; | 131; | 143; | 64; | 59; | 62; | 1 | 12 | |
| – | – | VPI-2del | 65; | 131; | 143; | 64; | 59; | 62; | 1 | 13 | |
| – | – | VPI-2del | 94; | 26; | 3; | 54; | 105; | 142; | 2 | 14 | |
| – | – | VPI-2del | 143; | 10; | 10; | 12; | 11; | 13; | 1 | 15 | |
| – | – | VPI-2del | 3; | 2; | 3; | 2; | 3; | 4; | 1 | 16 | |
| – | – | VPI-2del | 64; | 2; | 108; | 65; | 96; | 63; | 1 | 17 | |
| – | – | VPI-2del | 66; | 59; | 149; | 65; | 25; | 63; | 1 | 18 | |
| – | – | VPI-2del | 94; | 175; | 62; | 131; | 92; | 122; | 1 | 19 | |
| – | – | VPI-2del | 144; | 86; | 19; | 131; | 129; | 122; | 4 | 20 | |
| – | – | VPI-2del | 66; | 56; | 65; | 65; | 60; | 63; | 1 | 21 | |
| – | – | VPI-2del | 64; | 55; | 65; | 65; | 60; | 137; | 2 | 22 | |
Рис. 4.
Филогенетическое дерево Vibrio cholerae О1 биовара Эль Тор по данным байесовского анализа нуклеотидных последовательностей шести генов “домашнего хозяйства” (cat, chi, dnaE, gyrB, pgm, recA) 10 токсигенных штаммов ctxA+tcpA+VPI-2+ и 42 нетоксигенных штаммов с генотипами ctxA–tcpA+VPI-2+, ctxA–tcpA+VPI-2del и ctxA–tcpA–VPI-2del. В основании ветвей указаны генетические расстояния, рассчитанные при помощи параметрической модели нуклеотидных замен GTR (General Time Reversible).
ОБСУЖДЕНИЕ
При сравнении последовательностей геномов ОП VPI-1 и VPI-2 43 нетоксигенных вибрионов выявлена их вариабельность, обусловленная точечными мутациями и делециями разной протяженности. Однако уровень вариабельности этих ОП был неодинаков. Нуклеотидная последовательность VPI-1 вибрионов ctxA−tcpA+ отличалась от таковой референсного штамма в основном наличием единичных нуклеотидных замен, но их количество было невелико. Консервативность VPI-1 могла быть связана с его важной функциональной значимостью – обеспечением колонизации вибрионами кишечника человека с возможным последующим развитием ОКИ. В то же время вариабельность генома VPI-2 была существенной. Выраженная нестабильность генома VPI-2 проявлялась в образовании делеций разной длины. При этом было установлено, что одним из отличий нетоксигенных штаммов с разными генотипами оказалась различная частота утраты этих участков ДНК – в 100.0% случаев у штаммов ctxA−tcpA− и только в 26.3% случаев у штаммов ctxA−tcpA+. Появление делеций в VPI-2, возможно, обусловлено незаконной рекомбинацией. Мы впервые обнаружили, что потеря участков ДНК VPI-2 коррелировала с возникновением множественных однонуклеотидных замен во всех сохранившихся генах области nan–nag, включая ген nanH. Такая ситуация, возможно, связана с утратой такими вибрионами редактирующих компонентов репаративных систем при реорганизации их генома, что характерно и для других бактерий [28, 29]. Но эти вопросы нуждаются в дальнейшем изучении. В результате анализа последовательностей гена nanH впервые выявили 12 его аллельных вариантов среди 42 изученных нетоксигенных штаммов. Обнаруженные нуклеотидные замены в аллельных вариантах гена nanH локализуются в основном в синонимичных сайтах и не приводят, видимо, к изменению функции кодируемого белка. Обнаруженные нами новые генетические различия нетоксигенных вибрионов ctxA−tcpA+VPI-2+ и ctxA−tcpA−VPI-2del+ позволили выявить ранее неизвестные молекулярные механизмы формирования популяций с разным уровнем генетического разнообразия, что в значительной мере отражает их неодинаковый адаптивный потенциал. Так, одной из возможных причин высокого уровня адаптации к действию различных стрессовых факторов у вибрионов ctxA−tcpA−VPI-2del+ является большое генетическое разнообразие функционально значимых генов утилизации аминосахаров и сиаловых кислот, локализованных в области nan–nag VPI-2del и обеспечивающих устойчивость к неблагоприятным факторам водной среды. В то же время у вибрионов ctxA−tcpA+VPI-2+ эти гены в основном консервативны. Основным аргументом в пользу данного предположения является факт широкого распространения в водной среде холерных вибрионов ctxA−tcpA−VPI-2del+, но довольно редкая встречаемость вибрионов ctxA−tcpA+VPI-2+ [3, 4]. Таким образом, сравнительный анализ состава и структуры генома ОП показал, что изученные нетоксигенные штаммы представлены тремя генетически разными группами ctxA−tcpA+VPI-2+, ctxA−tcpA+VPI-2del+ и ctxA−tcpA−VPI-2del+ c разным адаптивным потенциалом.
Филогенетический анализ, основанный на сравнении нуклеотидных последовательностей гена nanH, а также шести генов “домашнего хозяйства”, позволил выявить новые кластеры нетоксигенных штаммов с разной структурой VPI-2 и показать возможное происхождение нетоксигенных изолятов ctxA−tcpA+VPI-2+ и ctxA−tcpA−VPI-2del+ от токсигенных, что согласуется с данными ряда исследователей.
Полученные сведения о структуре ОП нетоксигенных вибрионов не только способствуют более глубокому пониманию молекулярных механизмов возникновения генетически разнообразных штаммов с разным адаптивным потенциалом. Выявленная корреляция между появлением в VPI-2 делеций и образованием точечных мутаций в гене nanH позволяет использовать нуклеотидную последовательность этого гена в качестве нового генетического маркера для дифференциации нетоксигенных штаммов с различным адаптивным потенциалом. Кроме того, нетоксигенные штаммы с установленной структурой генома, не требующие удаления при его редактировании ключевых генов вирулентности, могут быть востребованы при создании нового поколения живых холерных вакцин.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Kaper J., Morris J., Jr., Levine M. Cholera // Clin. Microbiol. Rev. 1995. V. 8. № 1. P. 48–86.
Научная рабочая группа ВОЗ. Холера и другие диареи, вызываемые вибрионами // Бюл. ВОЗ. 1980. Т. 58. № 3. С. 277–296.
Faruque S.M., Asadulghani, Saha M.N. et al. Analysis of clinical and environmental strains of nontoxigenic Vibrio cholerae for susceptibility to CTXPhi: molecular basis for origination of new strains with epidemic potential // Infect. Immun. 1998. V. 66. № 12. P. 5819–5825.
Левченко Д.А., Кругликов В.Д., Архангельская И.В. и др. Анализ результатов мониторинга холерных вибрионов в объектах окружающей среды на административных территориях России с помощью ГИС “Холера 1989–2014” // Пробл. особо опасных инфекций. 2017. № 4. С. 99–102.
Takemura T., Murase K., Maruyama F. et al. Genetic diversity of environmental Vibrio cholerae O1 strains isolated in Northern Vietnam // Infect. Genet. Evol. 2017. V. 54. P. 146–151. https://doi.org/10.1016/j.meegid.2017.06.017
Heidelberg J.F., Elsen J.A., Nelson W.C. et al. DNA sequence of both chromosomes of the cholera pathogen Vibrio cholerae // Nature. 2000. V. 406. P. 477–483. https://doi.org/10.1038/35020000
Marsh J.W., Taylor R.K. Genetic and transcriptional analyses of the Vibrio cholerae mannose-sensitive hemagglutinin type 4 pilus gene locus // J. Bacteriol. 1999. V. 181. № 4. P. 1110–1117.
Dziejman M., Balon E., Boyd D. et al. Comparative genomic analysis of Vibrio cholerae: genes that correlate with cholera endemic and pandemic disease // Proc. Natl Acad. Sci. USA. 2002. V. 99. № 3. P. 1556–1561. https://doi.org/10.1073/pnas.042667999
Смирнова Н.И., Заднова С.П., Агафонов Д.А. и др. Сравнительный молекулярно-генетический анализ мобильных элементов природных штаммов возбудителя холеры // Генетика. 2013. Т. 49. № 9. С. 1036–1047.
Hu D., Liu B., Feng L. et al. Origins of the current seventh cholerae pandemic // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 48. P. 7730–7739. https://doi.org/10.1073/pnas.1608732113
Waldor M.R., Mekalanos J.J. Lysogenic conversion by a filamentous phage encoding cholera toxin // Science. 1996. V. 272. № 5270. P. 1910–1914. https://doi.org/10.1126/science.272.5270.1910
Karaolis D.K., Johnson J.A., Bailey C.C. et al. A Vibrio cholerae pathogenicity island associated with epidemic and pandemic strains // Proc. Natl Acad. Sci. USA. 1998. V. 95. № 6. P. 3134–3139. https://doi.org/10.1073/pnas.95.6.3134
Faruque Sh.M., Mekalanos J.J. Pathogenicity islands and phages in Vibrio cholerae evolution // Trends Microbiol. 2003. V. 11. № 11. P. 505–510. https://doi.org/10.1016/j.tim.2003.09.003
Ильина Т.С. Нитчатые бактериофаги и их роль в вирулентности и эволюции патогенных бактерий // Мол. генетика, микробиология и вирусология. 2015. Т. 30. № 1. С. 3–10.
Смирнова Н.И., Кульшань Т.А., Баранихина Е.Ю. и др. Структура генома и происхождение нетоксигенных штаммов Vibrio cholerae биовара Эль Тор с различной эпидемиологической значимостью // Генетика. 2016. Т. 52. № 9. С. 1029–1041.
Pang B., Yan M., Cui Z. et al. Genetic diversity of toxigenic and nontoxigenic Vibrio cholerae serogroups O1 and O139 revealed by array-based comparative genomic hybridization // J. Bacteriol. 2007. V. 189. P. 4837–4849. https://doi.org/10.1128/JB.01959-06
Jermyn W.S., Boyd E.F. Characterization of a novel Vibrio pathogenicity island (VPI-2) encoding neuraminidase (nanH) among toxigenic Vibrio cholerae isolates // Microbiology. 2002. V. 148. P. 3681–3693. https://doi.org/10.1099/00221287-148-11-3681
Jermyn W.S, Boyd E.F. Molecular evolution of Vibrio pathogenicity island-2 (VPI-2): mosaic structure among Vibrio cholerae and Vibrio mimicus natural isolates // Microbiology. 2005. V. 151. P. 311–322. https://doi.org/10.1099/mic.0.27621-0
Siriphap A., Leekitcharoenphon P., Kaas R.S. et al. Characterization and genetic variation of Vibrio cholerae isolated from clinical and environmental sources in Thailand // PLoS One. 2017. V. 19. № 12(1):0169324. https://doi.org/10.1371/journal.pone.0169324
Karaolis D.K., Somara S., Maneval D.R. Jr. et al. A bacteriophage encoding a pathogenicity island, a type-IV pilus and a phage receptor in cholera bacteria // Nature. 1999. V. 399. № 6734. P. 375–379. https://doi.org/10.1038/20715
Karaolis D.K.R., Lan R., Kaper J.B., Reeves P.R. Comparison of Vibrio cholerae pathogenicity islands in sixth and seventh pandemic strains // Infect. Immun. 2001. V. 69. № 3. P. 1947–1952. https://doi.org/10.1128/IAI.69.3.1947-1952.2001
Choi S.Y., Rashed S.M., Hasan N.A. et al. Phylogenetic diversity of Vibrio cholerae associated with endemic cholera in Mexico from 1991 to 2008 // mBio. 2016. V. 7. № 2.e02160–15. https://doi.org/10.1128/mBio.02160-15
Matson J.S., Withey J.H., DiRita V.J. Regulatory networks controlling Vibrio cholerae virulence gene expression // Infect. Immun. 2007. V. 75. № 12. P. 5542–5549. https://doi.org/10.1128/IAI.01094-07
Almagro-Moreno S., Boyd E.F. Sialic acid catabolism confers a competitive advantage to pathogenic Vibrio cholerae in the mouse intestine // Infect. Immun. 2009. V. 77. № 9. P. 3807–3816. https://doi.org/10.1128/IAI.00279-09
Смирнова Н.И., Агафонова Е.Ю., Щелканова Е.Ю. и др. Геномное разнообразие нетоксигенных штаммов Vibrio cholerae O1, выделенных на территории России и сопредельных стран // Мол. генетика, микробиология и вирусология. 2018. № 2. С. 76–86.
Организация работы лабораторий, использующих методы амплификации нуклеиновых кислот при работе с материалом, содержащим микроорганизмы I–IV групп патогенности: Методические указания 1.3.2569–09. М.: Федеральный центр гигиены и эпидемиологии Роспотребнадзора, 2009. 31 с.
de Abreu Figuiredo S.C., Nevers-Borges A.C., Coelho A. The neuraminidase gene is present in the non-toxigenic Vibrio cholerae Amazonia strain: a different allele in comparison to the pandemic strains // Mem. Inst. Oswaldo Cruz, Rio de Janeiro. 2005. V. 100. № 6. P. 563–569. https://doi.org/10.1590/s0074-02762005000600010
Moran N.A. Accelerated evolution and Muller’s rachet in endosymbiotic bacteria // Proc. Natl Acad. Sci. USA. 1996. V. 93. № 7. P. 2873–2878. https://doi.org/10.1073/pnas.93.7.2873
Itoh T., Martin W., Nei M. Acceleration of genomic evolution caused by enchansed mutation rate in endocellular symbionts // Proc. Natl Acad. Sci. USA. 2002. V. 99. № 20. P. 12944–12948. https://doi.org/10.1073/pnas.192449699
Дополнительные материалы отсутствуют.