Генетика, 2020, T. 56, № 9, стр. 1034-1042
Шаперон DnaK участвует в фолдинге, но не в рефолдинге термоинактивированных белков в Bacillus subtilis
Е. Ю. Гнучих 1, 3, *, И. В. Манухов 4, Г. Б. Завильгельский 2, 3, **
1 Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”,
Курчатовский геномный центр
117545 Москва, Россия
2 Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”
117545 Москва, Россия
3 НИЦ “Курчатовский институт”
123098 Москва, Россия
4 Московский физико-технический институт
141700 Московская область, Долгопрудный, Россия
* E-mail: gnuchikh_ey@genetika.ru
** E-mail: zavilgel@genetika.ru
Поступила в редакцию 07.11.2019
После доработки 12.12.2019
Принята к публикации 18.12.2019
Аннотация
Проведено исследование роли шаперонов DnaKJE и триггер фактора (ТФ) в процессах фолдинга и рефолдинга белков в Bacillus subtilis. В качестве исследуемых белков были использованы бактериальные люциферазы Photobacterium leiognathi и Photorhabdus luminescens. Показано, что в отличие от Escherichia coli в клетках B. subtilis шаперон DnaKJE участвует в основном в фолдинге белков, но не в рефолдинге термоинактивированных белков. Показано, что шаперон ТФ в B. subtilis участвует в синтезе термолабильной люциферазы P. leiognathi, но значительно снижает уровень синтеза темостабильной люциферазы P. luminescens.
В грамотрицательных бактериях Escherichia coli шаперон DnaK-DnaJ-GrpE (DnaKJE) выполняет ключевую роль в процессе контроля за качеством продукции (белки), разрушая белковые агрегаты и восстанавливая нативные структуры неправильно собранных (misfolded) и денатурированных (unfolded) белков (рефолдинг) [1–7].
Кроме того, DnaKJE участвует совместно с шапероном триггер фактором (ТФ) в формировании нативной структуры белковой макромолекулы в процессе синтеза на рибосоме (фолдинг) [8–12].
В грамположительных бактериях исследованы регуляция экспрессии белков-шаперонов [13–15], влияние шаперонов на выживаемость бактерий при повышенных (стрессовых) температурах [16]. Показано, что шаперон DnaK Bacillus subtillis комплементирует способность расти при высокой температуре (40°C) мутантный штамм E. coli ΔdnaK [17]. Показано, что у B. subtilis механизм регуляции экспрессии генов теплового шока значительно отличается от такового в клетках E. coli. Относительно же участия шаперонов в процессах фолдинга и рефолдинга белков экспериментальные данные малочисленные и носят фрагментарный характер. Например, в работе [18] показано, что шаперон DnaK B. subtilis in vitro осуществляет рефолдинг денатурированного (5 М мочевина, 25°С) белка лактат-дегидрогеназы (LDH): за 50 мин инкубации в рефолдируемой среде, содержащей ATP, фосфоэнолпируват, NADH, пируваткиназу и DnaK, процент активного фермента увеличивался с 12 (спонтанный рефолдинг) до 75.
Аминогликозиды (стрептомицин, гентамицин, канамицин и др.) значительно усиливают процесс ошибочного синтеза белков на рибосоме, что приводит к формированию в клетке misfolded полипептидов [19–21]. Было также показано, что аминогликозиды индуцируют в клетках B. subtilis синтез белков теплового шока [22]. Поэтому представляло интерес использовать стрептомицин как индуктор ошибок трансляции для получения неправильно собранных полипептидов и оценить, как шапероны DnaK и ТФ “исправляют” ошибки, возникшие в процессе трансляции белка на рибосомах.
В настоящей работе проведено исследование роли шаперонов DnaK и ТФ в процессах фолдинга и рефолдинга белков in vivo в клетках B. subtilis. В качестве исследуемых белков были использованы бактериальные люциферазы. Показано, что в отличие от E. coli в клетках B. subtilis шаперон DnaKJE участвует в основном в фолдинге белков, но не в рефолдинге термоинактивированных белков.
МАТЕРИАЛЫ И МЕТОДЫ
Бактериальные штаммы и плазмиды
Штаммы и плазмиды, использованные в работе, представлены в табл. 1.
Таблица 1.
Штаммы и плазмиды, используемые в работе
| Бактерии и плазмиды | Источник |
|---|---|
| Escherichia coli MG1655 | Получен из ВКПМ |
| Escherichia coli PK202 ∆dnaKJ14 dksA::kan | Получен от Craig E. [7] |
| Bacillus subtilis 168 | Получен из ВКПМ |
| Bacillus subtilis NBS2001 ∆dnaK-dnaJ::Spcr | Получен от Yoshikawa H. [16] |
| Bacillus subtilis NBS1001 ∆tig::Cmr | » |
| Bacillus subtilis NBS2002 ∆dnaK-dnaJ::Spcr ∆tig::Cmr | » |
| pMWAL-1TPpur_dhfr_t1t2_MCS | Настоящая работа |
| pPfbaA_MCS | » |
| pPxylA_MSC | » |
| pPfbaA_xenAB | » |
| pPfbaA_leoAB | » |
| pPxylA_xenAB | » |
Ферменты, реактивы
Эндонуклеазное расщепление, лигирование фрагментов ДНК, электрофорез в агарозном геле, выделение фрагментов ДНК из агарозного геля проводили согласно [23]. ПЦР проводили с помощью высокоточной ДНК-полимеразы Q5 (NEB, США). Реакции молекулярного клонирования проводили с использованием ферментов фирм “Promega” (США) или Gibson Assembly Master Mix (NEB, США). Субстрат бактериальной люциферазы n-деканаль получен от “Sigma” (США). Антибиотики хлорамфеникол, триметоприм, спектиномицин и стрептомицин получены от “Sigma”.
Среды и условия культивирования
Компоненты питательных сред триптон, дрожжевой экстракт, агар-агар, NaCl были приобретены в “Helicon” (Россия).
E. coli выращивали в LB-среде (1%-ный триптон, 0.5%-ный дрожжевой экстракт и 0.5% NaCl ), B. subtilis выращивали в LB или в триптоновой среде (1.5%-ный триптон, 0.5% NaCl) при 37°С, если не указано иное, и постоянной аэрации (200 об./мин). Селективные среды для мутантов B. subtilis содержат хлорамфеникол 10 мкг/мл и/или спектиномицин 150 мкг/мл в LB-среде. Для трансформации и пересева штаммов B. subtilis, содержащих плазмиды, использовалась триптоновая среда с добавлением триметоприма 8 мкг/мл; для штаммов B. subtilis 168 и NBS2001 могла использоваться также среда LB с добавлением хлорамфеникола 10 мкг/мл. Селективные среды для E. coli c плазмидами содержат ампициллин 100 мкг/мл. Твердые среды дополнительно содержат 1.5%-ный агар-агар.
Для исследования фолдинга B. subtilis выращивали в течение ночи при 30°С при постоянной аэрации (200 об./мин), в триптоновой среде с добавлением триметоприма, далее разводили в 100 раз в LB-среде (+триметоприм) и выращивали при температуре, указанной в опыте.
Для индукции промотора PxylA использовалась 1%-ная D(+)-ксилоза (AppliChem, Германия).
При исследовании роста клеток и фолдинга люцифераз в присутствии аминогликозидов использовались среда, приготовленная на деионизованной воде, и стрептомицин в концентрации 8 и 15 мкг/мл соответственно.
Трансформация
Трансформацию B. subtilis проводили по методу Спицайзена [24], E. coli трансформировали кальциевым методом.
Конструирование челночных плазмид
Праймеры, используемые при конструировании плазмид, приведены в табл. 2. Челночные плазмиды конструировали на основе pMWAL-1TPpur, содержащей два ориджина репликации – pMW118 [GenBank accession number AB005475] для E. coli и pBS72 репликон тета-типа для B. subtilis [25], а также содержащей гены bla и cat, сообщающие устойчивость к ампициллину и хлорамфениколу в E. coli и B. subtilis соответственно. Дополнительно с помощью пары праймеров P1/P2 под промотор Ppur по сайту XbaI был клонирован ген dhfr из B. cereus ATCC14579, кодирующий дигидрофолатредуктазу и сообщающий устойчивость к триметоприму при малых концентрациях фолатов в питательной среде, в результате была получена плазмида pMWAL-1TPpur_dhfr. В плазмиду pMWAL-1TPpur_dhfr с помощью пары праймеров P3/P4 по сайтам BamHI/KpnI были встроены терминатор t1t2 гена rrnB и полилинкер, в результате была получена беспромоторная плазмида pMWAL-1TPpur_dhfr_t1t2_MCS. В эту плазмиду далее были клонированы промоторы. Конститутивный промотор PfbaA [26] был клонирован с помощью пары праймеров P5/P6 по сайту рестрикции SacI, в результате была получена плазмида pPfbaA_MCS. Аналогичным образом по сайту SacI с помощью пары праймеров P7/P8 был клонирован индуцируемый промотор PxylA без CRE элемента и его репрессор xylR [27], в результате была получена плазмида pPxylA_MSC. В полученные плазмиды по сайтам рестрикции NotI/ApaI были клонированы гены luxAB с оптимизированными последовательностями Шайн-Дальгарно, кодирующие α и β субъединицы люцифераз Photorhabdus luminescens и Photobacterium leiognathi, из плазмид pXen5 [28] и pLF22ABleo [29] с помощью пар праймеров P9/P10 и P11/P12 соответственно. В результате были сконструированы плазмиды pPfbaA_xenAB, pPfbaA_leoAB и pPxylA_xenAB, используемые в настоящей работе для изучения процессов фолдинга и рефолдинга бактериальных люцифераз в клетках грамположительных бактерий B. subtilis.
Таблица 2.
Праймеры, используемые в работе
| P1 | 5-GTTTCTACCCGGCTGCCGTAATAAAGGAGGTTTACCGATGATTGTTTCATTTATGGTCGCTATG-3 |
| P2 | 5-TCGGTACCCGGGGATCCTCGATTCTCCTCCTCTTTCTATATTAGT-3 |
| P3 | 5-GTACTAATATAGAAAGAGGAGGAGAATCGAGCTGATGCAAAAACGAGGCTAGTTTAC-3 |
| P4 | 5-GAGCTCGGTACCGCGGCCGCTCGAGGGGCCCGGCGCGCCGGATCCCCATGCCGAACTCAGAAGTGAA-3 |
| P5 | 5-GCCGCGGTACCGAGCTTTTTCTCCATAACTAGGATACCAAC-3 |
| P6 | 5-AAAGAAGAGCTTTCAGGTATTCGAATCATGTCATTATGTTGCCGATTTG-3 |
| P7 | 5-AGAAGAGCTTTCAGGAATTCGTTCTATTTTAGAACTCCTTTTTCATATGAGAAGGT-3 |
| P8 | 5- GCCGCGGTACCGAGCTCGATTGAGCCAAGTTATTTCCTCCTTA-3 |
| P9 | 5-ACCGCGGCCGCTCGAGGAAGCAAGAGGAGGACTCTCTATG-3 |
| P10 | 5-GCCGGGCCCCTCGAGATTTCAACCTGGCCGTTAATAATGAATGA-3 |
| P11 | 5-ATCGCGGCCGCTCTCAGATCGGAAGGTGGAAGAA-3 |
| P12 | 5-TCGGGGCCCGTACCTCGCGAATGCATCTA-3 |
Измерение интенсивности биолюминесценции
Бактериальная люцифераза катализирует окисление длинноцепочечного альдегида (RCHO) атмосферным кислородом (O2) в присутствии восстановленного флавинмононуклеотида (FMNH2):
In vivo измерение активности люциферазы проводили с использованием люминометра Biotox 7 (OOO “Ekon”, Россия). Суспензию синтезирующей люциферазу бактериальной культуры (200 мкл) смешивали с 0.001%-ным n-деканалем (2 мкл) в этаноле и через 5–10 с проводили измерение интенсивности биолюминесценции.
Рефолдинг термоинактивированных люцифераз
В суспензию бактерий E. coli для ингибирования синтеза белка перед началом термоинактивации люцифераз вносили 167 мкг/мл хлорамфеникола. Термоинактивацию люцифераз проводили, выдерживая клетки при температуре 46°С 5 мин для люциферазы P. leiognathi и 10 мин при 48°C для P. luminescens.
В бактериях B. subtilis термоинактивацию люциферазы P. leiognathi проводили, инкубируя суспензию клеток B. subtilis с аэрацией при 46°C 30 мин, для P. luminescens – при 49°C 30 мин. Для ингибирования синтеза белка перед термоинактивацией был добавлен тетрациклин в концентрации 60 мкг/мл.
Рефолдинг люцифераз проводили при температуре, оптимальной для активности данной люциферазы: 23°C для P. leiognathi и 36°C для P. luminescens [30, 31], рефолдинг люцифераз в B. subtilis проводили с аэрацией суспензии клеток. Измерения проводили через определенные интервалы времени, отбирая пробу (200 мкл) и сразу после добавления n-деканаля (субстрат люциферазной реакции) измеряли интенсивность биолюминесценции.
Фолдинг бактериальных люцифераз в клетках B. subtilis
Для исследования фолдинга штаммы B. subtilis выращивали в течение ночи при 30°С при постоянной аэрации (200 об./мин) в триптоновой среде с добавлением триметоприма, далее разводили в 100 раз в LB-среде (+триметоприм) и инкубировали при температуре, указанной в опыте. Для индукции промотора PxylA использовалась ксилоза (1%).
При исследовании роста клеток и фолдинга люцифераз в присутствии аминогликозидов использовался стрептомицин в концентрации 8 и 15 мкг/мл соответственно.
Через определенные интервалы времени отбирали аликвоты (200 мкл) и сразу после добавления n-деканаля измеряли интенсивность люминесценции.
РЕЗУЛЬТАТЫ
Рефолдинг термоинактивированных люцифераз
На рис. 1 представлена кинетика рефолдинга термоинактивированных бактериальных люцифераз – термолабильной P. leiognathi и термостабильной P. luminescens в клетках E. coli (а) и B. subtilis (б). Оптимальные температуры для активности люцифераз в E. coli: P. leiognathi – 23°С и P. luminescens – 36°C. Активность люциферазы определяли, проводя измерение интенсивности люминесценции. Термоинактивацию люцифераз в E. coli проводили, выдерживая клетки при повышенной температуре, до снижения уровня биолюминесценции на 3–4 порядка от исходного.
Рис. 1.
Кинетика рефолдинга люциферазы P. leiognathi (1, 2) и P. luminescens (3, 4) в клетках E. coli (а): 1, 3 – MG1655 dnaKJ+, 2, 4 – PK202 ΔdnaKJ14; в клетках B. subtilis (б): 1, 3 – 168 dnaKJ+, 2, 4 – NBS2001 ΔdnaK-dnaJ.
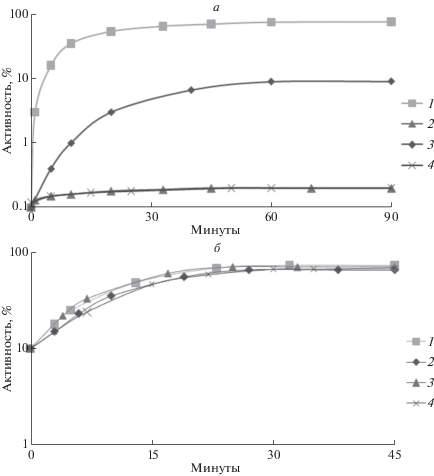
В клетках E. coli MG1655 dnaKJ+ наблюдается эффективный и быстрый рефолдинг люцифераз (для термолабильной люциферазы P. leiognathi уровень максимума рефолдинга достигает примерно 80%, а для термостабильной люциферазы P. luminescens – 8–9%), который осуществляется в основном шапероном DnaKJE, так как в мутантном штамме E. coli PK202 ΔdnaKJ14 рефолдинг термоинактивированных люцифераз практически отсутствует.
Так как оптимальные температуры для активности люцифераз в B. subtilis значительно превышают (примерно на 4–5°С) таковые в E. coli: P. leiognathi – 28°С, P. luminescens – 40°С, то для определения уровня рефолдинга люцифераз в B. subtilis предварительную термоинактивацию ферментов проводили, выдерживая клетки при 46°С для P. leiognathi и 49°С для P. luminescens в течение 30 мин с аэрацией, при этом активность люцифераз снижалась примерно в 10 раз. В клетках B. subtilis 168 dnaKJ+ наблюдается восстановление активности термоинактивированных люцифераз (как P. leiognathi, так и P. luminescens) примерно до 60–70%, однако такой же уровень рефолдинга осуществляется и в мутантном штамме B. subtilis NBS2001 ΔdnaK-dnaJ.
Фолдинг люцифераз в B. subtilis
Для оценки сборки белка в процессе синтеза (фолдинг) в B. subtilis были использованы термолабильная люцифераза P. leiognathi и термостабильная люцифераза P. luminescens. На рис. 2 и 3 представлены гистограммы синтеза активных люцифераз в клетках B. subtilis 168 и мутантных штаммах (с делециями в генах, кодирующих шапероны DnaKJ и ТФ) при различных температурах инкубации. На рис. 2 представлены гистограммы интенсивности биолюминесценции клеток B. subtilis, содержащих плазмиду pPfbaA_leoAB с генами luxAB, кодирующими термолабильную люциферазу P. leiognathi, при OD600 = 0.5 и температурах 28, 37 и 40°С.
Рис. 2.
Фолдинг люциферазы P. leiognathi при разных температурах: а – 28°С, б – 37°C, в – 40°C. Представлена интенсивность люминесценции при OD600 = 0.5 штаммов B. subtilis 168 dnaKJ+, NBS1001 Δtig, NBS2001 ΔdnaK-dnaJ, NBS2002 ΔdnaK-dnaJ; Δtig.
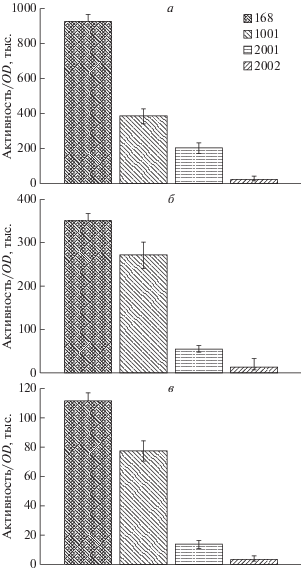
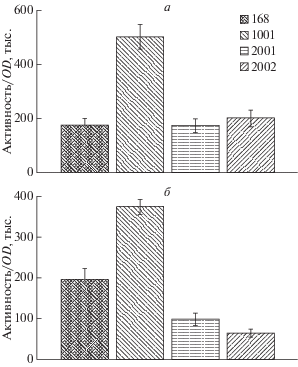
Рис. 3.
Фолдинг люциферазы из P. luminescens при разных температурах: а – 40°С, б – 46°C. Представлена интенсивность люминесценции при OD600 = 0.5 штаммов B. subtilis 168 dnaKJ+, NBS1001 Δtig, NBS2001 ΔdnaK-dnaJ, NBS2002 ΔdnaK-dnaJ; Δtig.
Как видим, уровень синтеза активной люциферазы зависит от наличия шаперонов DnaKJ и TФ, а также от температуры инкубации. При оптимальной температуре 28°С для проявления активности люциферазы, синтез нативного фермента в B. subtilis 168 при OD600 = 0.5 происходит в 40 раз более активно, чем в двойном мутанте NBS2002 Δtig ∆dnaK-dnaJ в тех же условиях. В одиночных мутантах NBS2001 ∆dnaK-dnaJ и NBS1001 Δtig уровень фолдинга активной люциферазы также снижен, но в меньшей степени (синтез активного фермента снижен в 5 раз в NBS2001 и в 2.5 раза в NBS1001). При инкубации клеток при более высоких температурах – 37°С (рис. 2,б) и 40°С (рис. 2,в) одновременно с синтезом активного фермента происходит термоинактивация белка, что видно на гистограммах по уровню биолюминесценции. Однако зависимость уровня фолдинга от активности шаперонов сохраняется, причем в одиночном мутанте NBS1001 Δtig снижение синтеза активного фермента менее значительно по сравнению с таковым при оптимальной температуре 28°С.
На рис. 3 представлены гистограммы, показывающие интенсивность биолюминесценции клеток B. subtilis, содержащих плазмиду pPfbaA-xenAB с генами luxAB, кодирующими термостабильную люциферазу P. luminescens, при OD600 = 0.5 и температурах 40 и 46°С.
При температуре 40°С (рис. 3,а) в клетках мутанта NBS1001 Δtig, содержащего только делецию гена tig, уровень синтеза активного белка примерно в 3 раза превышает таковой, характерный для бактерий дикого типа, при этом почти нет разницы между диким типом и мутантными штаммами с делецией ΔdnaK-dnaJ (NBS2001) и ΔdnaK-dnaJ; Δtig (NBS2002). Однако при повышенной температуре 46°С (рис. 3,б) делеция ΔdnaK-dnaJ снижает уровень синтеза активного фермента по сравнению с диким типом в 2 раза, при этом дополнительная делеция по гену tig еще сильнее снижает уровень фолдинга, как и в варианте с термолабильной люциферазой, но в клетках одинарного мутанта NBS1001 ∆tig сохраняется повышенный уровень синтеза активного фермента, примерно в 2 раза выше, чем в диком типе. Нужно также отметить, что уровень биолюминесценции в штамме дикого типа при температурах 40 и 46°С практически одинаков, но заметно отличается в мутантных штаммах.
Влияние антибиотиков аминогликозидов на фолдинг люцифераз в клетках B. subtilis
На рис. 4 представлены экспериментальные данные зависимости роста клеток B. subtilis (a) и синтеза люциферазы P. luminescens (б) от времени инкубации в питательной среде, содержащей стрептомицин.
Рис. 4.
Рост бактерий B. subtilis 168 dnaKJ+ и мутантных штаммов NBS1001 Δtig, NBS2001 ΔdnaK-dnaJ, NBS2002 ΔdnaK-dnaJ Δtig в присутствии стрептомицина 8 мкг/мл при 37°С за 5 ч (а). Фолдинг люциферазы P. luminescens в присутствии стрептомицина 15 мкг/мл (б). Индукцию проводили при 40°С, стрептомицин был внесен в образцы за 5 мин до индукции 1%-ной ксилозой. По оси ординат приведена активность люциферазы (за 100% принята активность в штамме B. subtilis 168 dnaKJ+), через 25 мин после начала индукции.
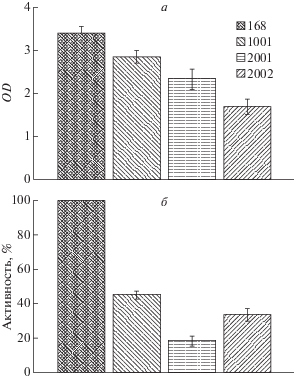
Как видим, делеции генов tig и особенно dnaK-dnaJ значительно снижают как скорость роста популяции клеток (рис. 4,а), так и синтез активных форм фермента (рис. 4,б). Что же касается двойного мутанта NBS2002 Δtig ΔdnaK-dnaJ, то при максимальном снижении скорости роста бактерий в этом штамме наблюдается несколько повышенный уровень синтеза активной люциферазы по сравнению с таковым в одиночном мутанте NBS2001 ΔdnaK-dnaJ.
ОБСУЖДЕНИЕ
В грамотрицательных бактериях основная функция АТФ-независимого шаперона ТФ – фолдинг вновь синтезируемой полипептидной цепи – тесно связана с работой рибосомы, с 50S-субъединицей, с которой ТФ формирует прочный комплекс (основной контакт с белком L23) [32]. Согласно основной схеме, ТФ, локализованный на рибосоме в области рибосомного туннеля, при высвобождении полипептидной цепи в цитоплазму осуществляет первичную сборку белка, завершение же процесса сборки белка (фолдинг) осуществляют в цитоплазме шапероны DnaKJE и GroEL/ES [10, 12, 33, 34]. ТФ в процессе первичной сборки белка, действуя совместно с рибосомой, задерживает (замедляет) окончательный фолдинг полипептида, значительно понижая вероятность формирования неправильных (misfolded) конформаций и агрегирования за счет введения в процесс дополнительных стадий, определяющих частичную раскрутку (unfolding) предварительно собранного домена, с последующим правильным процессом укладки цепи [11, 35, 36].
ATФ-зависимый шаперон DnaKJE в E. coli осуществляет в основном рефолдинг неправильно собранных и денатурированных белков, но также участвует в процессе фолдинга совместно с ТФ [1]. DnaKJE формирует комплекс с белком в результате Ван-дер-Ваальсового взаимодействия с гидрофобными участками в полипептидной цепи субстрата. С энергетической точки зрения действие шаперона DnaKJE состоит в совершении работы против барьера свободной энергии за счет гидролиза АТФ, в результате которой стабильная денатурированная форма белка-субстрата с низкой свободной энергией переходит в процессе раскручивания полипептидной цепи (активность шаперона – “unfoldase”) в “открытый” конформер с высокой свободной энергией, который затем может спонтанно перейти в состояние нативного конформера с низкой свободной энергией [37, 38].
В настоящей работе в качестве белков-субстратов использованы гомологичные ферменты – бактериальные люциферазы, характеризующиеся различной термочувствительностью. В работе [31] было показано, что в клетках E. coli шаперонная активность DnaKJE значительно понижается с ростом термостабильности белка-субстрата.
Однако согласно данным, представленным на рис. 1, в грамположительных бактериях B. subtilis рефолдинг термоинактивированных люцифераз не зависит от активности шаперона DnaKJE, а уровень рефолдинга термостабильной и термолабильной люцифераз практически одинаков.
В работе [39] было показано, что в клетках штамма E. coli Δtig ΔdnaKJ (клетки не способны расти при температуре выше 30°С) образуется примерно в 10 раз больше белков-агрегатов по сравнению с таковыми в клетках штамма с одиночной мутацией ΔdnaKJ, что прямо указывает на кооперативное участие шаперонов ТФ и DnaKJE в фолдинге синтезируемых на рибосоме полипептидов. На основании данных, представленных на рис. 2 и 3, подобный вывод справедлив и для грамположительных бактерий B. subtilis, так как в двойном мутанте NBS2002 уровень синтеза активных люцифераз значительно снижен по сравнению с таковым в клетках штаммов, содержащих одиночные мутации Δtig или ΔdnaK-dnaJ. В работе [39] также показано, что в клетках E. coli увеличение внутриклеточной концентрации ТФ примерно в 4 раза носит летальный характер, причем не только в мутантных штаммах, но также и в клетках дикого типа, причем летальность ТФ связана в основном с формированием агрегата – белка OmpF (outer membrane porin), который в процессе синтеза формирует прочный комплекс с ТФ и не способен поэтому к дальнейшему процессированию, проводимому шаперонами SecB и SecA. Можно предположить, что наблюдаемое в настоящей работе усиление фолдинга термостабильной люциферазы P. luminescens в клетках B. subtilis Δtig по сравнению с таковым в клетках дикого типа (рис. 3) также связано с формированием прочного комплекса синтезируемой на рибосоме полипептидной цепи с шапероном ТФ в результате Ван-дер-Ваальсового взаимодействия гидрофобных сайтов, замедляющего фолдинг. Необходимо отметить, что для термолабильной люциферазы P. leiognathi подобного эффекта не наблюдается (рис. 2).
Как видно из данных, представленных на рис. 4,б, в присутствии аминогликозида стрептомицина в бактериях B. subtilis уровень синтеза активной люциферазы P. luminescens значительно снижается в штамме, содержащем делецию ΔdnaK-dnaJ, и в меньшей степени в мутантном штамме Δtig. Отметим, что повышения уровня фолдинга люциферазы в одиночном мутанте Δtig по сравнению с таковым в диком штамме (рис. 3) в данном случае не наблюдается, что указывает на необходимость шаперона ТФ в проводимом в присутствии аминогликозида фолдинге белка, содержащего большое количество трансляционных ошибок.
Полученные в настоящей работе результаты, с использованием на модели бактериальных люцифераз, различающихся по термостабильности, показывают, что шаперон DnaKJE в B. subtilis принимает непосредственное участие в процессе синтеза белка на рибосоме, но в отличие от соответствующего шаперона в клетках E. coli практически не способен проводить рефолдинг термоинактивированных белков. Следовательно, система ATФ-зависимых шаперонов в грамположительных бактериях значительно отличается по механизму действия от шаперонов, работающих в грамотрицательных бактериях. Согласно нашим предварительным данным важную роль в системе ATФ-зависимых шаперонов в клетках B. subtilis выполняют белки “теплового шока” группы Hsp90.
Работа поддержана государственным заданием № 595-00003-19 ПР.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Calloni G., Chen T., Schermann S.M. et al. DnaK functions as a central hub in the E. coli chaperone network // Cell. Rep. 2012. V. 1. № 3. P. 251–264. https://doi.org/10.1016/j.celrep.2011.12.007
Hesterkamp T., Bukau B. Role of the DnaK and HscA homologs of Hsp70 chaperones in protein folding in E. coli // EMBO J. 1998. V. 17. № 16. P. 4818–4828. https://doi.org/10.1093/emboj/17.16.4818
Liu C.P., Perrett S., Zhou J.M. Dimeric trigger factor stably binds folding-competent intermediates and cooperates with the DnaK-DnaJ-GrpE chaperone system to allow refolding // J. Biol. Chem. 2005. V. 280. № 14. P. 13315–13320. https://doi.org/10.1074/jbc.M414151200
Tyedmers J., Mogk A., Bukau B. Cellular strategies for controlling protein aggregation // Nat. Rev. Mol. Cell Biol. 2010. V. 11. № 11. P. 777–788. https://doi.org/10.1038/nrm2993
Stoecklin G., Bukau B. Telling right from wrong in life — cellular quality control // Nat. Rev. Mol. Cell Biol. 2013. V. 14. № 10. P. 613–615. https://doi.org/10.1038/nrm3662
Doyle S.M., Genest O., Wickner S. Protein rescue from aggregates by powerful molecular chaperone machines // Nat. Rev. Mol. Cell Biol. 2013. V. 14. № 10. P. 617–629. https://doi.org/10.1038/nrm3660
Kang P.J., Craig E.A. Identification and characterization of a new Escherichia coli gene that is a dosage-dependent suppressor of a dnaK deletion mutation // J. Bacteriol. 1990. V. 172. № 4. P. 2055–2064. https://doi.org/10.1128/jb.172.4.2055-2064.1990
Deuerling E., Schulze-Specking A., Tomoyasu T. et al. Trigger factor and DnaK cooperate in folding of newly synthesized proteins // Nature. 1999. V. 400. № 6745. P. 693–696. https://doi.org/10.1038/23301
Frydman J. Folding of newly translated proteins in vivo: the role of molecular chaperones // Annu. Rev. Biochem. 2001. V. 70. № 1. P. 603–647. https://doi.org/10.1146/annurev.biochem.70.1.603
Agashe V.R., Guha S., Chang H.C. et al. Function of trigger factor and DnaK in multidomain protein folding: increase in yield at the expense of folding speed // Cell. 2004. V. 117. № 2. P. 199–209. https://doi.org/10.1016/s0092-8674(04)00299-5
Hoffmann A., Becker A.H., Zachmann-Brand B. et al. Concerted action of the ribosome and the associated chaperone trigger factor confines nascent polypeptide folding // Mol. Cell. 2012. V. 48. № 1. P. 63–74. https://doi.org/10.1016/j.molcel.2012.07.018
Wruck F., Avellaneda M.J., Koers E.J. et al. Protein folding mediated by trigger factor and Hsp70: new insights from single-molecule approaches // J. Mol. Biol. 2018. V. 430. № 4. P. 438–449. https://doi.org/10.1016/j.jmb.2017.09.004
Schulz A., Schumann W. hrcA, the first gene of the Bacillus subtilis dnaK operon encodes a negative regulator of class I heat shock genes // J. Bacteriol. 1996. V. 178. № 4. P. 1088–1093. https://doi.org/10.1128/jb.178.4.1088-1093.1996
Mogk A., Homuth G., Scholz C. et al. The GroE chaperonin machine is a major modulator of the CIRCE heat shock regulon of Bacillus subtilis // EMBO J. 1997. V. 16. № 15. P. 4579–4590. https://doi.org/10.1093/emboj/16.15.4579
Schumann W. Regulation of bacterial heat shock stimulons // Cell Stress Chaperones. 2016. V. 21. № 6. P. 959–968. https://doi.org/10.1007/s12192-016-0727-z
Reyes D.Y., Yoshikawa H. DnaK chaperone machine and trigger factor are only partially required for normal growth of Bacillus subtilis // Biosci. Biotechnol. Biochem. 2002. V. 66. № 7. P. 1583–1586. https://doi.org/10.1271/bbb.66.1583
Mogk A., Bukau B., Lutz R. et al. Construction and analysis of hybrid Escherichia coli–Bacillus subtilis dnaK genes // J. Bacteriol. 1999. V. 181. № 6. P. 1971–1974.
Shi L., Ravikumar V., Derouiche A. et al. Tyrosine 601 of Bacillus subtilis DnaK undergoes phosphorylation and is crucial for chaperone activity and heat shock survival // Front. Microbiol. 2016. V. 7. P. 533. https://doi.org/10.3389/fmicb.2016.00533
Kohanski M.A., Dwyer D.J., Wierzbowski J. et al. Mistranslation of membrane proteins and two-component system activation trigger antibiotic-mediated cell death // Cell. 2008. V. 135. № 4. P. 679–690. https://doi.org/10.1016/j.cell.2008.09.038
Kohanski M.A., Dwyer D.J., Collins J.J. How antibiotics kill bacteria: from targets to networks // Nat. Rev. Microbiol. 2010. V. 8. № 6. P. 423–435. https://doi.org/10.1038/nrmicro2333
Goltermann L., Good L., Bentin T. Chaperonins fight aminoglycoside-induced protein misfolding and promote short-term tolerance in Escherichia coli // J. Biol. Chem. 2013. V. 288. № 15. P. 10483–10489. https://doi.org/10.1074/jbc.M112.420380
Lin J.T., Connelly M.B., Amolo C. et al. Global transcriptional response of Bacillus subtilis to treatment with subinhibitory concentrations of antibiotics that inhibit protein synthesis // Antimicrob. Agents Chemother. 2005. V. 49. № 5. P. 1915–1926. https://doi.org/10.1128/AAC.49.5.1915-1926.2005
Sambrook J., Russell D.W. Molecular Cloning: A Laboratory Manual. 3rd ed. Cold Spring Harbor Lab. Press, 2001. 2344 p.
Spizizen J. Transformation of biochemically deficient strains of Bacillus subtilis by deoxyribonucleate // Proc. Natl Acad. Sci. USA. 1958. V. 44. № 10. P. 1072–1078. https://doi.org/10.1073/pnas.44.10.1072
Titok M.A., Chapuis J., Selezneva Y.V. et al. Bacillus subtilis soil isolates: plasmid replicon analysis and construction of a new theta-replicating vector // Plasmid. 2003. V. 49. № 1. P. 53–62. https://doi.org/10.1016/s0147-619x(02)00109-9
Guiziou S., Sauveplane V., Chang H.J. et al. A part toolbox to tune genetic expression in Bacillus subtilis // Nucl. Acids Res. 2016. V. 44. № 15. P. 7495–7508. https://doi.org/10.1093/nar/gkw624
Bhavsar A.P., Zhao X., Brown E.D. Development and characterization of a xylose-dependent system for expression of cloned genes in Bacillus subtilis: conditional complementation of a teichoic acid mutant // Appl. Environ. Microbiol. 2001. V. 67. № 1. P. 403–410. https://doi.org/10.1128/AEM.67.1.403-410.2001
Francis K.P., Yu J., Bellinger-Kawahara C. et al. Visualizing pneumococcal infections in the lungs of live mice using bioluminescent Streptococcus pneumoniae transformed with a novel gram-positive lux transposon // Infect. Immun. 2001. V. 69. № 5. P. 3350–3358. https://doi.org/10.1128/IAI.69.5.3350-3358.2001
Дерябин Д.Г., Каримов И.Ф., Манухов И.В. и др. Дифференцированная оценка бактерицидных систем сыворотки крови с использованием рекомбинантных люминесцирующих штаммов Escherichia coli и Bacillus subtilis // Бюл. эксперим. биологии и медицины. 2012. Т. 154. № 7. С. 68–73.
Тюлькова Н.А., Сандалова Т.П. Сравнительное исследование влияния температуры на бактериальные люциферазы // Биохимия. 1996. Т. 61. № 2. С. 275–287.
Manukhov I.V., Eroshnikov G.E., Vyssokikh M.Y. et al. Folding and refolding of thermolabile and thermostable bacterial luciferases: the role of DnaKJ heat-shock proteins // FEBS Lett. 1999. V. 448. № 2–3. P. 265–268. https://doi.org/10.1016/s0014-5793(99)00384-1
Kramer G., Rauch T., Rist W. et al. L23 protein functions as a chaperone docking site on the ribosome // Nature. 2002. V. 419. № 6903. P. 171–174. https://doi.org/10.1038/nature01047
Ying B.W., Taguchi H., Ueda T. Co-translational binding of GroEL to nascent polypeptides is followed by post-translational encapsulation by GroES to mediate protein folding // J. Biol. Chem. 2006. V. 281. № 31. P. 21813–21819. https://doi.org/10.1074/jbc.M603091200
Gloge F., Becker A.H., Kramer G. et al. Co-translational mechanisms of protein maturation // Curr. Opin. Struct. Biol. 2014. V. 24. P. 24–33. https://doi.org/10.1016/j.sbi.2013.11.004
Mashaghi A., Kramer G., Bechtluft P. et al. Reshaping of the conformational search of a protein by the chaperone trigger factor // Nature. 2013. V. 500. № 7460. P. 98–101. https://doi.org/10.1038/nature12293
Nilsson O.B., Müller-Lucks A., Kramer G. et al. Trigger factor reduces the force exerted on the nascent chain by a cotranslationally folding protein // J. Mol. Biol. 2016. V. 428. № 6. P. 1356–1364. https://doi.org/10.1016/j.jmb.2016.02.014
Sharma S.K., De los Rios P., Christen P. et al. The kinetic parameters and energy cost of the Hsp70 chaperone as a polypeptide unfoldase // Nat. Chem. Biol. 2010. V. 6. № 12. P. 914–920. https://doi.org/10.1038/nchembio.455
Priya S., Sharma S.K., Goloubinoff P. Molecular chaperones as enzymes that catalytically unfold misfolded polypeptides // FEBS Lett. 2013. V. 587. № 13. P. 1981–1987. https://doi.org/10.1016/j.febslet.2013.05.014
Genevaux P., Keppel F., Schwager F. et al. In vivo analysis of the overlapping functions of DnaK and trigger factor // EMBO Rep. 2004. V. 5. № 2. P. 195–200. https://doi.org/10.1038/sj.embor.7400067
Дополнительные материалы отсутствуют.


