Генетика, 2021, T. 57, № 1, стр. 103-107
Разработка системы праймеров для секвенирования митохондриального генома северного оленя Rangifer tarandus
И. В. Артюшин 1, 2, Е. А. Коноров 1, 3, *, К. А. Курбаков 1, 3, Ю. А. Столповский 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Московский государственный университет им. М.В. Ломоносова
119234 Москва, Россия
3 Федеральный научный центр пищевых систем им. В.М. Горбатова
Российской академии наук
109316 Москва, Россия
* E-mail: casqy@yandex.ru
Поступила в редакцию 24.01.2020
После доработки 05.08.2020
Принята к публикации 03.09.2020
Аннотация
Разработана система праймеров и оптимизированы условия ПЦР для амплификации митохондриального генома северного оленя Rangifer tarandus. Получены последовательности полного митохондриального генома домашнего и дикого оленя из Ненецкого автономного округа. Сравнение двух данных геномов с уже известными двумя митохондрионами домашних оленей из Китая и Японии выявило всего 160 полиморфных сайтов во всех этих выборках. Обнаружены уникальные аллели как для диких, так и домашних оленей Ненецкого автономного округа. Таким образом, разработанная методика позволяет получить новую информацию для филогеографии и изучения генетического разнообразия пород северного оленя, охватывая изменчивость по неизученным для северного оленя митохондриальным маркерам, таким как ND1–ND6 и ATP6, ATP8.
Генетике северного оленя Rangifer tarandus посвящено множество исследований, затрагивающих проблемы консервации географических популяций [1, 2], определения потока генов между дикими и домашними формами [3, 4], генетического разнообразия отдельных пород и популяций [5, 6]. В большинстве работ использовались микросателлитные маркеры, либо отдельные регионы митохондриального генома.
Участки митохондриального генома широко используются в качестве маркеров для филогенетики и баркодинга, а также для изучения внутривидовой изменчивости. Для такого рода исследований мтДНК очень удобна из-за ее высокой копийности, что улучшает успешность амплификации при небольшом количестве ДНК. Последовательность генов в митохондрионе консервативна у животных, что облегчает подбор и использование праймеров для разных групп животных. В то же время в мтДНК есть достаточное количество замен для определения различий как особей внутри вида, так и на уровне семейств. Особенно при использовании маркеров с разной скоростью накопления замен, таких как первая субъединица цитохромоксидазы (COI) и D-петля. Несмотря на обнаруженные недостатки – отклонение от нейтральности, отклонения в скоростях эволюции у отдельных таксонов, ядерные копии митохондриальных генов [7], а также на увеличение доли исследований, использующих полногеномное секвенирование – мтДНК не утратила своей роли одного из основных маркеров в популяционной генетике.
Большинство популяционно-генетических исследований северного оленя проводилось на единичном маркере, в основном D-петле контрольного региона, cytb, COI или COII, либо на комбинации из приведенных маркеров [3–9]. Использование полных митохондриальных геномов же позволяет объединить гаплотипы отдельных маркеров в единые гаплогруппы митохондрионов, сократив их количество, и дополнить информацией о полиморфизме неизученных участков мтДНК. Использование полных митохондриальных геномов позволило прояснить происхождение отдельных линий крупного рогатого скота и с большим разрешением уточнить их популяционную историю [10, 11]. В настоящее время известны митохондрионы двух северных оленей – эвенкийского оленя из Аолугуя, Китай [12, GenBank KM506758.1] и оленя из Хоронобе, Япония (Wada et al., неопубликованные данные, GenBank AB245426.1). В данных работах были использованы праймеры для Arborophila rufipectus и сычуаньской куропатки [13], что может затруднить их использование для популяционно-генетических исследований с использованием высокопроизводительного секвенирования из-за недостаточной специфичности. Поэтому нами была разработана система праймеров для амплификации мтДНК для последующего секвенирования.
Мы разработали систему из пяти пар праймеров (табл. 1) и опробовали ее на 120 образцах выщипов с относительно деградировавшей ДНК (условия хранения: температура 0–10°C, срок хранения 1–2 года), и 24 образца из свежезамороженных образцов. Для фрагментов L_2915-R_8934 и L_8328-R_13977 амплификация произошла только в половине свежих образцов и в четверти образцов с деградировавшей ДНК, скорее всего, из-за большей длины (5649 и 6019 пн соответственно). Объем смеси для ПЦР составил 25 мкл, из них: 2 мкл – 10× Encyclo буфер (Encyclo Plus PCR kit, Евроген, Россия), 0.4 мкл dNTP 50×, 0.4 мкл Hot Taq-полимераза 50×, 2× 0.04 мкл праймеры, 17.12 мкл dd H2O и 5 мкл экстракта ДНК образца.
Таблица 1.
Разработанная система праймеров для амплификации полного митохондриона R. tarandus и эффективность амплификации для хорошо сохранившейся и деградировавшей мтДНК
| Позиция начала праймера | Последовательность прямого праймера | Позиция начала праймера | Последовательность обратного праймера | % амплификации из свежих проб (n = 24) |
% амплификации из проб с деградировавшей ДНК (n = 120) |
Температура, °C |
|---|---|---|---|---|---|---|
| L_1324 | gctaagtaccccgaaaccagacgagc | R_5458 | gttggcccagttcagcacggattagt | 100 | 83.3 | 62.7 |
| L_2915 | gccattacgacctgccacatcctcaa | R_8934 | ggggcaaggcttgagtggtaaaatgc | 50 | 22.7 | 62 |
| L_8328 | acacccgctccattaattcccatgct | R_13977 | agtggtggggttggttgtggaattgt | 62.5 | 20.8 | 64 |
| L_10802 | ggccttcacctctgactacccaaagc | R_14930 | ctggggtgtagttgtctgggtctcct | 95.8 | 87.5 | 64 |
| L_15394 | tcaacacccaaagctgaagttctaatt | R_1349 | gctcgtctggtttcggggtacttagc | 100 | 100 | 55.9 |
Два образца дикого и домашнего оленя из Ненецкого автономного округа были отсеквенированы на платформе Illumina Mi Seq. Полученные чтения были выровнены на геном оленя из Хоронобе с помощью bowtie 2 [14] и собраны, используя известный геном в качестве референса. Последовательности митохондриального генома дикого и домашнего оленя из Ненецкого автономного округа депонированы в базе данных GenBank под идентификаторами MT753444 и MT726040 соответственно. Полученные нами митохондриальные геномы были выровнены с геномами северных оленей из Аолугуя и Хоронобе, а также c геномом Cervus elaphus (GenBank KP172593.1) с помощью MAFFT [15]. Также были получены из базы данных GenBank данные по отдельным митохондриальным маркерам: COI, cytb, контрольному региону [9, 16–20], и выровнены отдельно для каждого маркера. Подсчет числа полиморфных сайтов и нуклеотидного разнообразия осуществлялся с помощью DnaSP [21].
Филогенетический анализ полных митохондрионов R. tarandus с помощью Mr. Bayes выявил родство домашнего оленя из Ненецкого АО и эвенкийского оленя из Аолугуя (рис. 1,а). Геном оленя из Хоронобе находится дальше от остальных митохондрионов, однако обладает общими аллельными вариантами с диким оленем из Ненецкого АО в 11 полиморфных сайтах (рис 1,б). В 78 полиморфных сайтах из 160 эвенкийский и ненецкие олени обладают общими аллельными вариантами.
Рис. 1.
Анализ полиморфизма мтДНК для полных геномов и данных из GenBank по отдельным маркерам. а – филогения четырех особей R. tarandus с известными последовательностями митохондриальных геномов; б – число уникальных аллелей для каждого из полных митохондрионов и число общих аллелей для двух и трех особей; в – доля полиморфных сайтов на маркер для полных митохондрионов (отмечено серым) и для выборок по отдельным маркерам, взятым из GenBank.
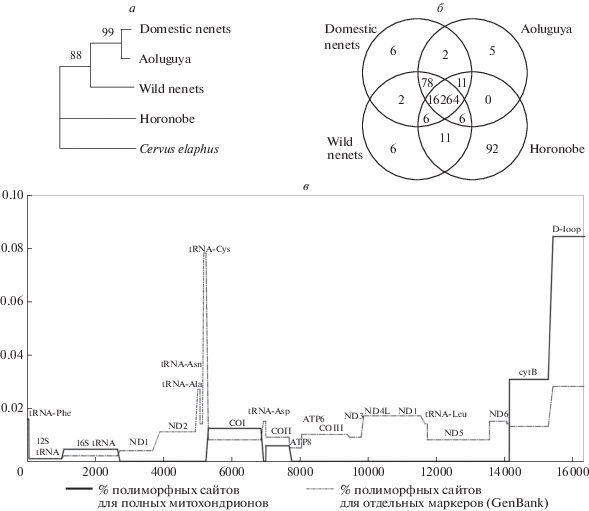
Прочитанных полных митохондриальных геномов R. tarandus на данный момент недостаточно, чтобы выявить большую долю генетической изменчивости, обнаруженную в предыдущих исследованиях с использованием последовательностей цитохрома b, контрольного региона и COI (табл. 2). Однако полученные нами данные позволяют судить о том, как распределены полиморфные сайты по митохондриальному геному R. tarandus (рис. 1,в и табл. 2). На основании данных по большему числу митохондрионов возможно объединить гаплотипы маркеров, быстро накапливающих замены (D-петля и цитохром b) в гаплогруппы, учитывая информацию по более “медленным” маркерам, в том числе не изученным для R. tarandus (таким как вторая и третья субъединицы цитохромоксидазы, NADH-дегидрогеназы и АТФазы). Наблюдаемая изменчивость по NADH-дегидрогеназам и АТФазам (рис. 1,в) может служить материалом для отбора к климатическим условиям или к высоким широтам, как это раньше наблюдалось на материале других млекопитающих [22, 23].
Таблица 2.
Нуклеотидное разнообразие (π) для некоторых митохондриальных маркеров R. tarandus
| 16S rRNA | ND2 | COI | COII | ND5 | ND6 | cytB | D-петля | |
|---|---|---|---|---|---|---|---|---|
| Полный митохондрион (n = 4) |
0.00118 | 0.00575 | 0.00475 | 0.00438 | 0.00394 | 0.00758 | 0.00715 | 0.0141 |
| Отдельные маркеры (по данным из GenBank) | 0.00272 (n = 5) | – | 0.00425 (n = 183) |
– | – | – | 0.05906 (n = 395) |
0.02795 (n = 1831) |
Разработанная система праймеров позволяет упростить задачу секвенирования митохондриальных геномов R. tarandus для изучения их популяционной истории и демографии, а также адаптационных процессов. В дальнейших исследованиях данная система будет оптимизирована для получения более устойчивой амплификации на фрагменте L_2915–R_8934 и использована для секвенирования накопленного материала по российским диким и одомашенным популяциям северного оленя.
Данная работа была поддержана грантом РФФИ № 17-29-08003.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Meng X., Aryal A., Tait A. et al. Population trends, distribution and conservation status of semi-domesticated reindeer (Rangifer tarandus) in China // J. for Nat. Conservation. 2014. V. 22. № 6. P. 539–546.https://doi.org/10.1016/j.jnc.2014.08.008
Festa-Bianchet M., Ray J.C., Boutin S. et al. Conservation of caribou (Rangifer tarandus) in Canada: An uncertain future // Canadian J. Zool. 2011. V. 89. № 5. P. 419–434. https://doi.org/10.1139/z11-025
Давыдов А.В., Холодова М.В., Мещерский И.Г. и др. Дифференциация диких и домашних форм северного оленя (Rangifer tarandus L.) по результатам анализа мтДНК // С.-х. биология. 2007. № 6. С. 1083–1093.
Cronin M.A., MacNeil M.D., Patton J.C. Mitochondrial DNA and microsatellite DNA variation in domestic reindeer (Rangifer tarandus tarandus) and relationships with wild caribou (Rangifer tarandus granti, Rangifer tarandus groenlandicus, and Rangifer tarandus caribou) // J. Heredity. 2006. V. 97. № 5. P. 525–530. https://doi.org/10.1093/jhered/esl012
Холодова М.В., Колпащиков Л.А., Кузнецова М.В., Баранова А.И. Генетическое разнообразие диких северных оленей (Rangifer tarandus) Таймыра: анализ полиморфизма контрольного региона митохондриальной ДНК // Известия РАН, Серия биологическая. 2011. № 1. С. 52–60. https://doi.org/10.7868/S0002332916060023
Кол Н.В., Королев А.Л., Захаров И.А. Полиморфизм митохондриальной ДНК в тувинской популяции северного оленя (Rangifer tarandus L.) // Генетика. 2006. Т. 42. № 1. С. 110–112.
Galtier N., Nabholz B., Glémin S., Hurst G.D.D. Mitochondrial DNA as a marker of molecular diversity: A reappraisal // Mol. Ecol. 2009. V. 18. № 22. P. 4541–4550. https://doi.org/10.1111/j.1365-294X.2009.04380.x
Cronin M.A., MacNeil M.D., Patton J.C. Variation in mitochondrial DNA and microsatellite DNA in caribou (Rangifer tarandus) in North America // J. Mammalogy. 2005. V. 86. № 3. P. 495–505. https://doi.org/10.1644/1545-1542(2005)86[495:VIMDAM]2.0.CO;2
Kvie K.S., Heggenes J., Røed K.H. Merging and comparing three mitochondrial markers for phylogenetic studies of Eurasian reindeer (Rangifer tarandus) // Ecol. and Evol. 2016. V. 6. № 13. P. 4347–4358. https://doi.org/10.1002/ece3.2199
Achilli A., Bonfiglio S., Olivieri A. et al. The multifaceted origin of taurine cattle reflected by the mitochondrial genome // PLoS One. 2009. V. 4. № 6. P. e5753. https://doi.org/10.1371/journal.pone.0005753
Achilli A., Olivieri A., Pellecchia M. et al. Mitochondrial genomes of extinct aurochs survive in domestic cattle // Curr. Biol. 2008. V. 18. № 4. P. R157–R158. https://doi.org/10.1016/j.cub.2008.01.019
Ju Y., Liu H., Rong M. et al. Complete mitochondrial genome sequence of Aoluguya reindeer (Rangifer tarandus) // Mitochondrial DNA Part A. 2016. V. 27. № 3. P. 2261–2262. https://doi.org/10.3109/19401736.2014.984171
He L., Dai B., Zeng B. et al. The complete mitochondrial genome of the Sichuan Hill Partridge (Arborophila rufipectus) and a phylogenetic analysis with related species // Gene. 2009. V. 435. № 1–2. P. 23–28. https://doi.org/10.1016/j.gene.2009.01.001
Langmead B., Salzberg S.L. Fast gapped-read alignment with Bowtie 2 // Nat. Methods. 2012. V. 9. № 4. P. 357–359. https://doi.org/10.1038/nmeth.1923
Katoh K., Asimenos G., Toh H. Multiple alignment of DNA sequences with MAFFT // Bioinf. for DNA Sequence Analysis. Humana Press, 2009. P. 39–64. https://doi.org/10.1007/978-1-59745-251-9_3
Kvie K.S., Heggenes J., Anderson D.G. et al. Colonizing the High Arctic: Mitochondrial DNA reveals common origin of Eurasian archipelagic reindeer (Rangifer tarandus) // PLoS One. 2016. V. 11. № 11. P. e0165237. https://doi.org/10.1371/journal.pone.0165237
Cronin M.A., MacNeil M.D., Patton J.C. Mitochondrial DNA and microsatellite DNA variation in domestic reindeer (Rangifer tarandus tarandus) and relationships with wild caribou (Rangifer tarandus granti, Rangifer tarandus groenlandicus, and Rangifer tarandus caribou) // J. Heredity. 2006. V. 97. № 5. P. 525–530. https://doi.org/10.1093/jhered/esl012
Wilkerson C.D., Mahoney S.P., Carr S.M. Post-glacial recolonization of insular Newfoundland across the Strait of Belle Isle gave rise to an endemic subspecies of woodland caribou, Rangifer tarandus terranovae (Bangs, 1896): Evidence from mtDNA haplotypes // Genome. 2018. V. 61. № 8. P. 575–585. https://doi.org/10.1139/gen-2017-0199
Yannic G., Pellissier L., Ortego J. et al. Genetic diversity in caribou linked to past and future climate change // Nat. Climate Change. 2014. V. 4. № 2. P. 132–137. https://doi.org/10.1038/nclimate2074
Eger J.L., Birt T.P., Gunn A., Baker A.J. Genetic diversity and history of Peary caribou (Rangifer tarandus) in North America // Proc. From the Caribou Genet. and Relationships Workshop. Edmonton, Canada. 2009. P. 73–101.
Librado P., Rozas J. DNA SP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452. https://doi.org/10.1093/bioinformatics/btg359
Yu L., Wang, X., Ting N., Zhang Y. Mitogenomic analysis of Chinese snub-nosed monkeys: Evidence of positive selection in NADH dehydrogenase genes in high-altitude adaptation // Mitochondrion. 2011. V. 11. № 3. P. 497–503. https://doi.org/10.1186/s12862-017-0896-0
Slimen H.B., Schaschl H., Knauer F., Suchentrunk F. Selection on the mitochondrial ATP synthase 6 and the NADH dehydrogenase 2 genes in hares (Lepus capensis L., 1758) from a steep ecological gradient in North Africa // BMC Evol. Biol. 2017. V. 17. № 1. P. 46.https://doi.org/10.1016/j.mito.2011.01.004
Дополнительные материалы отсутствуют.


