Генетика, 2021, T. 57, № 10, стр. 1164-1173
Гаплотипическое разнообразие монгольских и тувинских пород коз (Capra hircus) на основе полиморфизма мтДНК и Y-хромосомы
В. Н. Воронкова 1, *, А. К. Пискунов 1, Э. А. Николаева 1, М. Т. Семина 1, Е. А. Коноров 1, 2, Ю. А. Столповский 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
2 Федеральный научный центр пищевых систем им. В.М. Горбатова Российской академии наук
109316 Москва, Россия
* E-mail: valery.voronkova@gmail.com
Поступила в редакцию 14.12.2020
После доработки 03.03.2021
Принята к публикации 29.03.2021
Аннотация
Впервые на основе полиморфизма гипервариабельной области D-петли мтДНК и гена SRY проведено сравнительное исследование гаплотипического и нуклеотидного разнообразия, генетической дифференциации и филогении тувинских шерстных (двух популяций, n = 34) и монгольских пуховых коз (пяти популяций, n = 94). Проведен внутрипородный, в том числе межпопуляционный, анализ в сравнении с референсными последовательностями ДНК коз других пород. Выявлен высокий уровень генетического разнообразия исследуемых выборок. Оценка молекулярной дисперсии показала, что основной вклад в генетическое разнообразие исследуемых пород вносит внутрипопуляционное разнообразие (98.84%) и лишь 1% обусловлен межпородными отличиями. Наиболее распространена среди коз монгольской и тувинской пород гаплогруппа А мтДНК, также были обнаружены особи, относящиеся к гаплогруппам С и F; другие гаплогруппы (B, D и G) у данных выборок не обнаружены. Большинство самцов монгольской породы на основе полиморфизма гена SRY имели гаплотипы Y1A (66.7%) и Y2A (25%), в популяциях алтай улаан и три красавицы были выявлены особи с редким гаплотипом Y2B (8.3%). Все исследованные самцы тувинской породы коз относились к гаплотипу Y2A.
Козоводство – одно из наиболее древних и в то же время интенсивно развивающихся отраслей животноводства в мире. Рост интереса к козоводству обусловлен высокими вкусовыми и диетическими свойствами продуктов питания, получаемых от этих животных, их сравнительной неприхотливостью, а также рядом экономических причин, в частности высоким индексом молочности коз, качеством пуха [1]. Несмотря на важную роль и существенный потенциал козоводства в сельском хозяйстве, изучение генома козы все еще находится в зачаточном состоянии по сравнению с другими сельскохозяйственными животными: крупным рогатым скотом, овцами и свиньями и т.п. Одомашнивание безоарового козла началось около 10 тыс. лет назад [2]. Современные данные исследований биологии коз показали, что первые генетические отличия домашних коз были связаны с изменением работы иммунной системы, пищеварения (адаптацией к ксенобиотикам), а также с адаптацией к перепаду температур. Интересно, что далеко не последнюю роль в вышеуказанных процессах, судя по результатам анализа древней ДНК, мог сыграть поток или интродукция “диких” генов свободноживущих сородичей. Известным примером служит появление активного варианта гена MUC6 у ряда филогенетических ветвей домашних коз [3]. Данный ген, белковый продукт которого служит эффективной защитой от микробных и паразитических инвазий, исходно “принадлежал” не безоаровому козлу, а кавказскому туру, обитающему на влажных и теплых территориях, богатых подобными патогенами [4].
Вплоть до начала XIX в. селекция коз была слабоинтенсивной. Тем не менее козы как вид представлены большим числом пород – около 600, что составляет примерно 12% от общего числа пород всех млекопитающих [5]. По всей видимости, такое породное разнообразие возникло в результате как естественного отбора (в основном при адаптации к различным условиям существования: высокогорье, засушливые равнины, жаркие летом и холодные зимой степи, каменистые долины, практически лишенные растительности), так и искусственного, направленного на избирательное улучшение отдельных качеств животных, в основном молочной, мясной и шерстной продуктивности.
Ярким примером приспособляемости коз, а также их связи с жизнью различных народов, служат тувинские и монгольские аборигенные козы. Они прекрасно адаптированы к суровым климатическим и кормовым условиям, имеют крепкую конституцию, неприхотливы, не требуя при этом затрат на производство и хранение растительных кормов. Монгольские и тувинские козы приспособлены к круглогодичной отгонно-пастбищной системе содержания. Такой способ, в отличие от интенсивного типа разведения, положительно сказывается на экологическом состоянии агро- и фитосистем региона, которые в целом крайне чувствительны к любому антропогенному воздействию.
В настоящее время тувинские и монгольские козы слабоизучены с генетической точки зрения. Очевидно, что уникальный генофонд местных коз охарактеризован недостаточно. Широко используемые во всем мире методы оценки филогении на основе мтДНК и Y-хромосомы никогда не применялись для тувинских коз и крайне редко для монгольских. Продемонстрирован низкий уровень генетической дифференциации и географической структуры монгольских коз при высоком уровне гаплотипического разнообразия D-петли мтДНК (0.977) [6]. В отличие от других азиатских пород, таких как японские, индийские [7], казахстанские [8], у монгольской и тувинской пород коз не проводилась оценка отцовского генетического разнообразия на основе полиморфизма SRY.
Таким образом, можно выделить три основные задачи данного исследования: выявление гаплотипического разнообразия, филогенетических взаимоотношений между исследуемыми популяциями, оценка генетических рисков для тувинской и монгольской пород.
МАТЕРИАЛЫ И МЕТОДЫ
Исследуемые популяции. Сбор образцов и получение ДНК
Первичным материалом для определения гаплотипов мтДНК и гена SRY служили образцы крови домашних коз (n = 453) двух аборигенных пород: тувинской шерстной (n = 188) и монгольской пуховой (n = 255).
Образцы крови коз тувинской шерстной породы были получены в двух сельскохозяйственных производственных кооперативах (СПК) Республики Тыва: “Артыш” (n = 70, Кызылский кожуун) и “Уургай” (n = 118, Эрзинский кожуун).
Образцы крови животных монгольской пуховой породы были получены в частных хозяйствах Монголии. Эти выборки в данной работе были названы в соответствии с их географической локализацией. Всего было получено пять выборок: три красавицы (n = 48, гурван эгч, аймак Умнеговь), дархатская (n = 52, аймак Хубсугул), бурая завхан (n = 52, аймак Завхан), ульгий улаан (n = 62, аймак Увс) и алтай улаан (n = 51, аймак Ховд) (рис. 1 – карта сбора образцов).
Протокол исследования № 3 был одобрен на заседании этического комитета ИОГен РАН от 14.12.2020. Забор крови осуществлялся ветеринаром с использованием одноразовых стерильных наборов в вакуумные пробирки с К3 ЭДТА Vacuette (Greiner Bio-One, Австрия) согласно ветеринарным и этическим нормам. Для дальнейшего анализа были отобраны исключительно неродственные особи на основе микросателлитных данных оценки родства с использованием программы ML-Relate [9].
Выделение ДНК проводилось из цельной крови наборами реагентов Magna Prep 200 (ООО “Лаборатория Изоген”, Москва) позволяющими получить высокомолекулярную ДНК с концентрацией 40–60 нг/мкл, согласно инструкции изготовителя. Для секвенирования D-петли мтДНК было отобрано по 20 образцов из каждой выборки, для гена SRY – от 4 до 14 в зависимости от числа самцов в выборке (табл. 1).
Таблица 1.
Генетическое разнообразие мтДНК коз (Capra hircus) монгольской и тувинской пород
| Порода | Выборка | Код | n | H | π | n гап. | A | C | F |
|---|---|---|---|---|---|---|---|---|---|
| Тувинская | Артыш | Artysh | 15 | 0.980 ± 0.028 | 0.029 ± 0.004 | 14 | 13 | 3 | – |
| Уургай | Uurgay | 19 | 0.950 ± 0.027 | 0.032 ± 0.005 | 11 | 19 | – | – | |
| Монгольская | Три красавицы | Tri_kras | 18 | 0.990 ± 0.023 | 0.028 ± 0.007 | 15 | 14 | 3 | – |
| Дархатская | Darhat | 19 | 0.940 ± 0.03 | 0.012 ± 0.001 | 11 | 13 | 5 | – | |
| Бурая завхан | Buraya | 20 | 0.960 ± 0.033 | 0.014 ± 0.001 | 16 | 14 | 6 | – | |
| Алтай улаан | AK | 20 | 1.000 ± 0.018 | 0.022 ± 0.003 | 17 | 20 | – | – | |
| Ульгий улаан | YK | 17 | 0.990 ± 0.023 | 0.025 ± 0.004 | 15 | 13 | 3 | 1 |
Амплификация и секвенирование D-петли мтДНК
Для амплификации гипервариабельного участка D-петли мтДНК коз были выбраны праймеры, рекомендованные в публикациях [10, 11]: CAP-F (5'-CTGATTAGTCATTAGTCCATC-3'), CAP-R (5'-CTGATTAGTCATTAGTCCATC-3'). Оптимизация условий ПЦР проводилась на амплификаторе MiniAmp Plus (Thermo Fisher Scientific, США) с зоной Veriflex, позволяющей одномоментно проводить амплификацию в трех различных температурных областях для подбора наилучшей температуры отжига праймеров. В качестве реактивов для постановки ПЦР были использованы коммерческие наборы 5X MasDDTaqMIX-2025 (Диалат Лтд, Россия), содержащие термостабильную “hot-start” полимеразу SmarTaq. Результатом оптимизации стала следующая программа амплификации участка D-петли мтДНК: 94°C – 10 мин, 35 циклов: 94°C – 30 с, 61°C – 30 с, 72°C – 30 с; элонгация 72°C – 5 мин.
Очистку фрагментов, а также определение прямой и обратной нуклеотидной последовательности в исследуемом фрагменте проводили в компании Евроген (Москва, РФ) методом автоматического секвенирования на генетическом анализаторе ABI Prism 3130xl (Applied Biosystems, США) с использованием наборов Big DyeTM Terminator v3.1 Cycle Sequencing (Applied Biosystem). Для дальнейшего анализа было отобрано 128 ампликонов размером 598 пн, расположенных между позициями 15 653 и 16 250 мтДНК коз.
Амплификация и секвенирование области гена SRY
Для секвенирования фрагмента длиной 543 пн гена SRY у самцов из двух выборок из Тувинской Республики и четырех из Монголии были подобраны праймеры (SRY-F 5'-CAAAGGCACGCT- TTATCTCA-3', SRY-R 5'-GGCAGCAAAACTGGCTTTTTCAC-3') в соответствии с работами Waki и соавт. [7] и Vidal и соавт. [12].
Оптимизация условий ПЦР для амплификации фрагмента гена SRY проводилась на амплификаторе MiniAmp Plus (Thermo Fisher Scientific, США) с использованием наборов 5X MasDDTaqMIX‑2025 (Диалат Лтд, Россия), содержащих термостабильную “hot-start” полимеразу SmarTaq. В результате оптимизации температуры отжига праймеров и концентрации реактивов итоговая программа имела следующий вид: 94°C – 5 мин, 35 циклов: 94°C – 30 с, 58°C – 30 с, 72°C – 1 мин 30 с; элонгация 72°C – 5 мин.
В результате прямого и обратного секвенирования в компании Евроген (Москва, РФ) и первичной обработки данных были получены 38 последовательностей гена SRY для пяти выборок (уургай и артыш тувинской породы коз, увс, ховд, дархат и три красавицы монгольской породы коз). Для дальнейшего анализа были отобраны 30 последовательностей, не имеющих артефактов секвенирования.
Анализ данных
Редактирование нуклеотидных последовательностей осуществлялось в программном пакете R (sangeranalyzeR) [13], выравнивание – в программах UGENE Unipro v1.28.1 [14] и MEGA7 v7.0.26 [15]. С использованием программы Arlequin v3.5.2.2 [16] были посчитаны H, π и их SD, а также Fst с 1000 повторностей для получения p-значения. Гаплотипы мтДНК были определены с помощью DnaSP [17] и с добавлением последовательностей HV1 из баз данных GenBank. Байесовское дерево гаплотипов было построено в программе MrBayes3.2.7 [18], число поколений MCMC 2 000 000, модель замен определена с помощью PartitionFinder 2 [19]. Филогенетическая сеть на основе алгоритма TCS была построена и визуализирована в программе PopART v1.7 [20].
Гаплотипы SRY были определены с использованием последовательностей ДНК из GenBank в соответствии со статьями Waki и др. [7], Vidal и др. [12] и Tabata и др. [8]. Для построения сетей был использован алгоритм Median-joining с равным весом всех замен в программе NETWORK v10.1.0.0 [21].
РЕЗУЛЬТАТЫ
Вариабельность D-петли мтДНК
В результате секвенирования были определены 128 последовательностей D-петли мтДНК длиной 506 пн у коз из семи выборок монгольской и тувинской аборигенных пород. Выявлено 18 полиморфных сайтов с общим числом мутаций 31, из которых 22 транзиции и 9 трансверсий. На основе данных вариаций (мутаций) было выявлено 76 гаплотипов D-петли мтДНК. Большинство коз принадлежали гаплогруппе A (n = 107), 20 животных – гаплогруппе C и одна особь – гаплогруппе F (табл. 1).
Анализ гаплотипического и нуклеотидного разнообразия (табл. 1) выявил высокий уровень изменчивости исследуемых выборок: H от 0.940 до 1.000 для монгольской породы и 0.950–0.980 для тувинской; π от 0.012 до 0.025 для монгольской и 0.029–0.032 для тувинской. Наименьшие значения данных статистик были получены для дархатской выборки (H = 0.940 и π = 0.012), наибольшее значение гаплотипического разнообразия отмечено у выборки алтай улаан (H = 1.000), а наибольшее нуклеотидное разнообразие – у тувинской уургай (π = 0.032).
Оценка генетической дифференциации выборок монгольской и тувинской пород была проведена с помощью сравнения попарных значений индекса фиксации Fst (табл. 2) и позволила выявить незначительный уровень дифференциации исследуемых популяций (Fst = 0.000–0.080). Достоверно отличалась лишь монгольская дархатская от бурой завхан (Fst = 0.065) и от алтай улаан (Fst = 0.080).
Таблица 2.
Попарные значения индекса фиксации Fst для монгольских и тувинских коз (Capra hircus) на основе анализа последовательностей D-петли мтДНК
| Порода | Артыш | Уургай | Три красавицы | Дархат | Бурая завхан | Алтай улаан | Ульгий улаан |
|---|---|---|---|---|---|---|---|
| Артыш | 0.000 | ||||||
| Уургай | 0 | 0.000 | |||||
| Три красавицы | 0 | 0 | 0.000 | ||||
| Дархатская | 0.005 | 0 | 0 | 0.000 | |||
| Бурая завхан | 0.007 | 0.047 | 0.035 | 0.065 | 0.000 | ||
| Алтай улаан | 0 | 0.037 | 0.034 | 0.080 | 0.013 | 0.000 | |
| Ульгий улаан | 0 | 0 | 0 | 0 | 0.014 | 0.015 | 0.000 |
Анализ филогенетических связей и представленности гаплогрупп мтДНК монгольской и тувинской пород коз
Филогенетические взаимоотношения среди выборок были продемонстрированы с использованием байесовского анализа. Все исследованные нами образцы принадлежали к гаплогруппе А, С и F, другие гаплогруппы (B, D и G) у данных выборок не обнаружены. Гаплогруппа А наиболее широко представлена среди коз различных пород, например у оманских пород коз с частотой более 0.80 [11], у казахской породы – 0.97 [8], а также у различных российских пород с частотой более 0.95 [22]. Возможно, более редкие гаплотипы не попали в исследованные нами выборки или в целом не представлены у монгольской и тувинской пород.
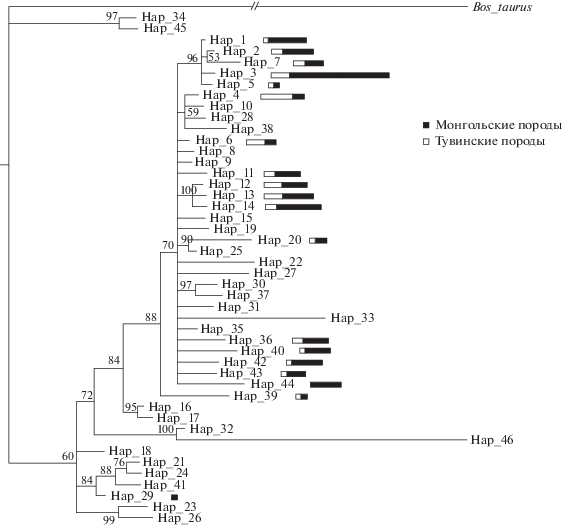
Монгольская и тувинская породы не формируют отдельные кластеры на дендрограмме (рис. 2), так как у коз нет породоспецифичных гаплотипов – отличается лишь частота их встречаемости в различных популяциях.
Рис. 2.
Байесово филогенетическое дерево на основе гаплотипов мтДНК монгольской и тувинской коз (Capra hircus), а также референсных последовательностей. В качестве внешней группы была использована последовательность мтДНК коровы (Bos taurus). Для внутренних узлов приведены апостериорные вероятности (число поколений MCMC 2 000 000).
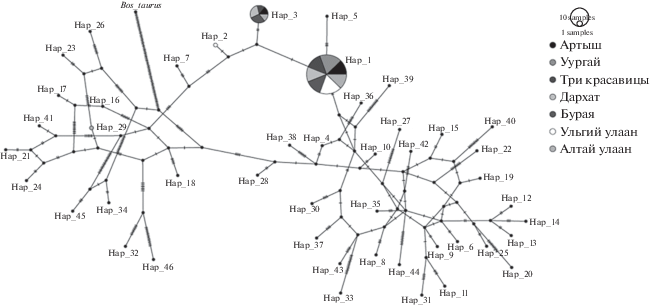
Построение TCS-сети в программе PopART позволило визуализировать филогенетические отношения среди исследуемых выборок (рис. 3). Высокий уровень разнообразия и большое число гаплотипов отражается в отсутствии сформированных породоспецифичных кластеров сети – у монгольской и тувинской пород встречаются различные гаплотипы, а популяции также отличаются лишь частотами их встречаемости.
Рис. 3.
TCS-cеть, отображающая генетические взаимоотношения между гаплогруппами мтДНК коз (Capra hircus). Размер круга соответствует числу входящих в него образцов. В качестве внешней группы использована последовательность мтДНК коровы (Bos taurus). Число прерывистых полос на линиях отражает число мутаций между двумя гаплотипами.
На основании анализа AMOVA (популяции были разделены на две группы по породам – монгольскую и тувинскую, табл. 3) было выявлено, что основной вклад в генетическое разнообразие исследуемых пород вносит внутрипопуляционное разнообразие (98.84%) и лишь 1% относится к межпородным отличиям. Для пород, обитающих в близких к естественным условиях и не находящихся под давлением селекции (искусственного отбора), типичны низкий уровень дифференциации и высокий уровень внутрипородного разнообразия.
Таблица 3.
Генетическое разнообразие тувинских и монгольских коз (Capra hircus) на основе анализа молекулярной дисперсии (AMOVA) последовательностей D-петли мтДНК
| Источник изменчивости | d.f. | SS | VC | Процент изменчивости |
|---|---|---|---|---|
| Между группами | 1 | 25.93 | 0.17 | 1.02 |
| Между популяциями внутри групп | 5 | 85.81 | 0.02 | 0.14 |
| Внутри популяций | 121 | 2025.29 | 16.74 | 98.84 |
| Всего | 127 | 2137.03 | 16.93 |
Анализ разнообразия гаплотипов Y-хромосомы на основе полиморфизма гена SRY
В данном исследовании определены гаплотипы Y-хромосомы монгольских и тувинских коз по гену SRY на основе 30 полученных нуклеотидных последовательностей длиной 548 пн. Полученные последовательности сравнивались с опубликованными ранее [7, 23, 24]. Обнаружены четыре полиморфных сайта гена SRY: g.2603A>T, g.2670A>G, g.2863T>A и g.2990A>G. Среди изученных популяций встречались три основных гаплотипа: наиболее распространенный среди азиатских пород Y1A (53.3%, n = 16), часто встречающийся у европейских пород Y2A (40%, n = 12) и редко встречающийся азиатский гаплотип Y2B (6.6%, n = 2) (табл. 4).
Таблица 4.
Число гаплотипов Y-хромосомы на основе вариабельности гена SRY у монгольских и тувинских пород коз
| Порода | Y1A | Y2A | Y2B |
|---|---|---|---|
| Дархат | 7 | 0 | 0 |
| Ульгий улаан | 6 | 2 | 0 |
| Алтай улаан | 1 | 0 | 1 |
| Три красавицы | 2 | 4 | 1 |
| Уургай | 0 | 3 | 0 |
| Артыш | 0 | 3 | 0 |
| Всего | 16 | 12 | 2 |
Большинство исследованных коз принадлежали к двум самым распространенным гаплотипам Y1A и Y2A, составив 53 и 45% соответственно. (табл. 4, рис. 4). Гаплотип Y1B не был обнаружен среди проанализированных животных: он с невысокой частотой встречается в основном в европейских популяциях коз. У монгольской породы коз встречается гаплотип Y2A, не обнаруженный в Индии, странах Юго-Восточной Азии и Японии, а также гаплотип Y2B, отсутствующий западнее Бутана, что по версии группы ученых из института Кобе [7] может быть вызвано сильным эффектом основателя на последних стадиях миграции одомашненных коз на восток. Самым распространенным гаплотипом среди монгольских популяций, исследованных нами, оказался гаплотип Y1A.
Рис. 4.
Сеть гаплогрупп Y-хромосомы на основе вариабельности гена SRY коз. Обозначения гаплотипов указаны под кругами. Размер круга отражает число входящих в него образцов. На линиях отмечены позиции мутаций.
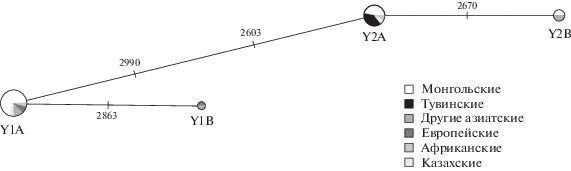
Среди тувинской породы коз был выявлен исключительно гаплотип Y2A, который наиболее полно представлен на территории Европы и Африки. Данный гаплотип также присутствует у монгольских и казахских коз (рис. 4). Вероятно, для выявления большего числа гаплотипов Y‑хромосомы требуется увеличить число выборок и исследуемых образцов тувинской породы.
ОБСУЖДЕНИЕ
Полученные в настоящем исследовании данные (в частности, высокие значения гаплотипического разнообразия от 0.940 до 1.000 и низкие значения Fst от 0 до 0.080), как и результаты подобных исследований, проводимых в Монголии в последние годы [6], свидетельствуют о слабой меж- и внутрипородной дифференциации локальных пород коз. Результаты микросателлитного анализа также свидетельствуют в пользу низкого уровня дифференциации тувинских и монгольских популяций коз [25, 26]. Дифференциация между монгольскими выборками коз составляла от 1.7 до 3.5%, что характерно для свободно скрещивающихся животных, не находящихся под давлением селекции.
Для монгольских коз характерен широкий спектр полиморфизма фенотипических признаков, начиная от разнообразия мастей (черная, бурая, белая, красная, пегая, чубарая и др.), формы рогов (винтовые или прямые) и их наличия/отсутствия (комолость), бороды и заканчивая разнообразием промеров, молочной и пуховой продуктивности [27]. Для отдельных популяций различаются частоты встречаемости данных признаков, например для коз улгий улаан (YK) характерна красная масть, однако встречаются и особи красно-коричневого и коричневого окраса. Многие породы монгольских коз в XX в. были получены путем скрещивания российских пород с аборигенными монгольскими, в том числе южно-гобийская порода три красавицы была выведена на основе местных коз и коз донской породы [27].
Дархатская (darhat) популяция монгольских коз значимо отличается от бурой (buraya) и алтай улаан (AK) (Fst 0.065 и 0.080 соответственно), что является следствием географической удаленности, а также разведения дархатской породы “в себе”, что также отражается в низком уровне гаплотипического и нуклеотидного разнообразия (H = 0.940 и π = = 0.012).
Все исследованные нами особи тувинской и монгольской породы принадлежат к гаплогруппам А, С и F, другие гаплогруппы (B, D и G) у данных выборок не обнаружены. Полученные результаты согласуются с данными других работ [6], гаплогруппа А наиболее часто встречается (90.8%) среди монгольских пород коз. Низкий уровень дифференциации тувинских и монгольских пород коз отражается в отсутствии сформированных породоспецифичных кластеров сети TCS – у монгольской и тувинской пород встречаются различные гаплотипы, а популяции незначительно отличаются лишь частотами их встречаемости. Вклад межпородного разнообразия в уровень изменчивости составил лишь 1%, в то время как различия между особями внутри популяций составили 98%.
Анализ последовательности гена SRY, локализованного на Y-хромосоме, позволил выявить три гаплотипа, присутствующих у монгольских популяций коз. Большинство самцов имели гаплотипы Y1A (66.7%) и Y2A (25%), распространенные среди азиатских и европейских пород соответственно. Также в монгольских популяциях алтай улаан (АК) и три красавицы (Tri_kras) были выявлены два самца с гаплотипом Y2B (8.3%), редко встречающимся только у азиатских пород коз [8]. Все исследованные самцы тувинской породы коз относились к гаплотипу Y2A, преобладающему в основном у европейских пород, что может косвенно свидетельствовать об интрогрессии западных пород коз и требует увеличения выборки для проверки данной гипотезы.
Видимое благополучие – высокий уровень генетического разнообразия, в том числе высокая численность животных, продукция от которых служит основным источником прибыли региона, может маскировать под собой критическую ситуацию. Суть проблемы заключается в размывании породных характеристик и возможной потери уникальных сочетаний аллелей, которые определяли ценные качества коз тувинской и монгольской аборигенных пород. Вероятно, что определенный селекционный прогресс отрасли козоводства в регионе в последние годы обусловлен эффектом гетерозиса, возникшим в результате завоза в Тыву и Монголию коз трансграничных пород в объеме нескольких миллионов особей, и последующим их скрещиванием с местными породами [28]. В результате получены синтетические популяции. Косвенным подтверждением реальности этих процессов может служить резкое снижение выживаемости коз в особенно суровые зимы (“дзуд”), в результате которых гибли миллионы животных и страна скатывалась в затяжной экономический кризис [29].
Таким образом, генетический статус изученных животных остается неопределенным, что в будущем может сыграть негативную роль для данных пород. Потенциальная утрата породных качеств, таких как крайне широкая адаптируемость к климатическим условиям и местной кормовой базе, сведет на нет пока еще экономическую целесообразность разведения данных аборигенных пород [30]. В то же время настоящее исследование показало, что наиболее изолированные популяции, в частности козы котловины Дархат, имеют наибольшую степень генетической автономности, при этом отличаясь наибольшей выносливостью и наиболее широким диапазоном физиологической адаптивности. Таким образом, не только жители Монголии и Тывы, но научное и мировое сообщество в целом должно быть заинтересовано в сохранении генофонда аборигенных коз, в изучении их физиологии и адаптивности. С социокультурной точки зрения сохранение биоразнообразия аборигенных пород коз – почти настолько же важная задача, как и сохранение находящихся под угрозой исчезновения малых народов Дархатской долины. Без пуха аратских коз в условиях типичной резко континентальной зимы с температурами до –45–50°С выживать будет значительно сложнее.
Полученные в настоящем исследовании данные о низкой дифференциации и высоком уровне генетического разнообразия на основе анализа нуклеотидных последовательностей митохондриальной ДНК и гена SRY могут привлечь внимание к проблеме генетической сохранности аборигенных пород и быть полезны при составлении программ сохранения местных пород коз Саяно-Алтайского региона.
Работа выполнена при финансовой поддержке гранта РНФ (проект № 19-76-20061).
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Zonaed Siddiki A., Miah G., Islam M.S. et al. Goat genomic resources: The search for genes associated with its economic traits // Intern. J. Genomics. 2020. 5940205. https://doi.org/10.1155/2020/5940205
Amills M., Capote J., Tosser-Klopp G. Goat domestication and breeding: A jigsaw of historical, biological and molecular data with missing pieces // Animal Genet. 2017. V. 48. № 6. P. 631–644. https://doi.org/10.1111/age.12598
Zheng Z., Wang X., Li M. et al. The origin of domestication genes in goats // Sci. Advances. 2020. V. 6. № 21. eaaz5216. https://doi.org/10.1126/sciadv.aaz5216
Simpson H.V., Umair S., Hoang V.C., Savoian M.S. Histochemical study of the effects on abomasal mucins of Haemonchus contortus or Teladorsagia circumcincta infection in lambs // Veterinary Parasitol. 2016. V. 226. P. 210–221. https://doi.org/10.1016/j.vetpar.2016.06.026
The second report on the state of the world’s animal genetic resources for food and agriculture / Eds Scherf B.D., Pilling D. FAO Commission on Genetic Resources for Food and Agriculture Assessments. Rome. 2015.
Ganbold O., Lee S.H., Paek W.K. et al. Mitochondrial DNA variation and phylogeography of native Mongolian goats // Asian-Australas J. Anim. Sci. 2020. V. 33. № 6. P. 902–912. https://doi.org/10.5713/ajas.19.0396
Waki A., Sasazaki S., Kobayashi E., Mannen H. Paternal phylogeography and genetic diversity of East Asian goats // Anim. Genet. 2015. V. 46. № 3. P. 337–339. https://doi.org/10.1111/age.12293
Tabata R., Kawaguchi F., Sasazaki S. et al. The Eurasian steppe is an important goat propagation route: A phylogeographic analysis using mitochondrial DNA and Y‑chromosome sequences of Kazakhstani goats // Anim. Sci. J. 2018. V. 90. P. 317–322. https://doi.org/10.1111/asj.13144
Kalinowski S.T., Wagner A.P., Taper M.L. ML-Relate: A computer program for maximum likelihood estimation of relatedness and relationship // Mol. Ecol. Notes. 2006. V. 6. P. 576–579. https://doi.org/10.1111/j.1471-8286.2006.01256.x
Luikart G., Gielly L., Excoffier L. et al. Multiple maternal origins and weak phylogeographic structure in domestic goats // Proc. Natl Acad. Sci. USA. 2001. V. 98. № 10. P. 5927–5932. https://doi.org/10.1073/pnas.091591198
Al-Araimi N.A., Gaafar O.M., Costa V. et al. Genetic origin of goat populations in Oman revealed by mitochondrial DNA analysis // PLoS One. 2017. V. 12. № 12. https://doi.org/10.1371/journal.pone.0190235
Vidal O., Drögemüller C., Obexer-Ruff G. et al. Differential distribution of Y-chromosome haplotypes in Swiss and Southern European goat breeds // Sci. Rep. 2017. V. 7. № 1. P. 16161. https://doi.org/10.1038/s41598-017-15593-1
Chao K.H., Barton K., Lanfear R. SangeranalyseR: simple and interactive analysis of Sanger sequencing data in R // bioRxiv. 2020.
Okonechnikov K., Golosova O., Fursov M. Unipro UGENE: A unified bioinformatics toolkit // Bioinformatics. 2012. V. 28. № 8. P. 1166–1167.
Kumar S., Tamura K., Nei M. MEGA: Molecular evolutionary genetics analysis software for microcomputers // Bioinformatics. 1994. V. 10. № 2. P. 189–191.
Excoffier L., Laval G., Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis // Evol. Bioinformatics. 2005. V. 1. P. 47–50.
Rozas J., Ferrer-Mata A., Sánchez-DelBarrio J.C. et al. DnaSP 6: DNA sequence polymorphism analysis of large data sets // Mol. Biol. Evol. 2017. V. 34. № 12. P. 3299–3302.
Huelsenbeck J.P., Ronquist F. MRBAYES: Bayesian inference of phylogenetic trees // Bioinformatics. 2001. V. 17. № 8. P. 754–755.
Lanfear R., Frandsen P.B., Wright A.M. et al. PartitionFinder 2: New methods for selecting partitioned models of evolution for molecular and morphological phylogenetic analyses // Mol. Biol. Evol. 2017. V. 34. № 3. P. 772–773.
Leigh J.W., Bryant D. POPART: full-feature software for haplotype network construction // Methods Ecol. Evol. 2015. V. 6. № 9. P. 1110–1116.
Bandelt H.J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48.
Deniskova T., Bakoev N., Dotsev A. et al. Maternal origins and haplotype diversity of seven russian goat populations based on the D-loop sequence variability // Animals (Basel). 2020. V. 10. № 9. P. 1603. https://doi.org/10.3390/ani10091603
Cañón J., García D., García-Atance M.A. et al. ECONOGENE Consortium. Geographical partitioning of goat diversity in Europe and the Middle East // Anim. Genet. 2006. V. 37. № 4. P. 327–334. https://doi.org/10.1111/j.1365-2052.2006.01461.x
Pereira F., Queirós S., Gusmão L. et al. Tracing the history of goat pastoralism: New clues from mitochondrial and Y chromosome DNA in North Africa // Mol. Biol. Evol. 2009. V. 26. № 12. P. 2765–2773. https://doi.org/10.1093/molbev/msp20
Бекетов С.В., Пискунов А.К., Воронкова В.Н. и др. Генетическое разнообразие и филогения пуховых коз Центральной и Средней Азии // Генетика. 2021. Т. 57. № 7. С. 810–819.
Takahashi H., Nyamsamba D., Mandakh B. et al. Genetic structure of Mongolian goat populations using microsatellite loci analysis // Asian-Aust. J. Anim. Sci. 2008. V. 21. № 7. P. 947–953.
Столповский Ю.А., Цендсурэн Ц., Кол Н.В. и др. Генофонды домашних животных Монголии. М.: Тов-во науч. изд. КМК, 2013. 278 с.
https://trendeconomy.com/data/h2/Mongolia/0104
Nandintsetseg B., Shinoda M., Du C., Munkhjargal E. Cold-season disasters on the Eurasian steppes: Climate-driven or man-made // Sci. Rep. 2018. V. 8. № 1. P. 14769. https://doi.org/10.1038/s41598-018-33046-1
Энхтувшин Б., Курас Л.В., Цыбенов Б.Д. Традиционное скотоводство монгольских кочевников в условиях глобализации // Вестник Бурятского науч. центра Сиб. отд. РАН. 2013. № 4. С. 208–223.
Дополнительные материалы отсутствуют.