Генетика, 2021, T. 57, № 11, стр. 1225-1240
Метилирование генов в свободной циркулирующей ДНК плазмы крови как прогностический и предиктивный факторы при раке молочной железы
С. В. Рыков 1, 2, *, Е. А. Филиппова 1, В. И. Логинов 1, Э. А. Брага 1, **
1 Научно-исследовательский институт общей патологии и патофизиологии
125315 Москва, Россия
2 Национальный исследовательский центр “Курчатовский институт” – ГосНИИгенетика,
Курчатовский геномный центр
117545 Москва, Россия
* E-mail: enterru@yandex.ru
** E-mail: eleonora10_45@mail.ru
Поступила в редакцию 13.01.2021
После доработки 18.03.2021
Принята к публикации 23.03.2021
Аннотация
Несмотря на значительный прогресс в методах ранней диагностики и лечения рака молочной железы (РМЖ), сохраняется высокий шанс рецидива. Существующие методы выявления рецидивов не пригодны для раннего обнаружения, поскольку требуют инвазивных процедур и не способны оперативно сопровождать лечение. Многообещающим методом стала так называемая жидкая биопсия, основанная на анализе генетического материала из периферической крови, в частности фракции свободной циркулирующей ДНК (сцДНК) плазмы крови. Определение мутаций, копийности и метилирования отдельных генов в сцДНК позволяет отслеживать изменения как в опухолевом очаге, так и в патологическом процессе у пациента в целом, причем мало инвазивно и с хорошей точностью и оперативностью. В представленном обзоре обобщены данные литературы по прогностической и предиктивной ценности эпигенетических маркеров, основанных на анализе метилирования генов в сцДНК плазмы или сыворотки крови у больных РМЖ. Показано, что гены, гиперметилированные в сцДНК (RASSF1A, RARB, SOX17, WNT5A и др.), могут служить эффективными маркерами общей и опухоль-специфичной выживаемости и химиорезистентности. Важным преимуществом маркеров метилирования, в отличие от маркеров на основе однонуклеотидных замен, микросателлитов и вариаций числа копий, являются универсальность, раннее проявление и ясная связь с биологией патологического процесса. Анализ метилирования наиболее эффективных маркеров может решать задачи своевременного выявления метастазирования и рецидива заболевания, оперативно наблюдать за ответом на неоадъювантную и послеоперационную химиотерапию, что является основой для персонифицированного подхода к лечению больных РМЖ.
Рак молочной железы (РМЖ) является самым распространенным злокачественным заболеванием у женщин в мире. В 2018 г. зарегистрировано более 2.1 млн новых случаев. Метастатический тип РМЖ диагностируется в зависимости от региона и этнической группы от 5–6 до 9% случаев первичного выявления [1]. РМЖ – гетерогенное заболевание, включающее различные типы опухолей, которые отличаются по морфологии, клиническому течению и чувствительности к терапии. Молекулярное типирование позволяет классифицировать опухоль, дать прогноз заболевания и персонализировать схему лечения, например определить требуемый объем хирургического вмешательства и средств терапии. В рутинной клинической практике применяется типирование по экспрессии генов рецепторов эстрогена, прогестерона и эпидермального фактора роста, которые делят РМЖ на четыре подтипа: люминальный А и Б, HER2+ позитивный и трижды негативный. Подтипы РМЖ отличаются между собой и по прогнозу течения, и ответом на медикаментозную терапию [2]. Однако детальное типирование вскрывает молекулярное разнообразие подтипов, что требует разработки и внедрения более точных и информативных групп маркеров [3].
Основной объект для молекулярного анализа – ткань опухоли, полученная с помощью биопсии. Биопсия является инвазивным методом и позволяет получить на анализ только небольшую часть опухолевого очага, который вследствие гетерогенности злокачественного процесса не всегда отражает молекулярный портрет всей опухоли. Более доступным и полноценным объектом может служить периферическая кровь, которая содержит помимо форменных элементов циркулирующие опухолевые клетки, свободную циркулирующую ДНК и экзосомы. Так называемая “жидкая биопсия” позволяет изучить генетический материал опухолевого процесса в целом. Так, можно определить генотип опухоли и его изменения с течением болезни, что делает возможным обнаружение мутаций, которые могут быть пропущены при инвазивной биопсии [4]. Жидкая биопсия может использоваться для мониторинга ответа на терапию, оценки риска возникновения устойчивости к терапии и своевременно выявлять рецидив болезни [5, 6]. Данные молекулярного профиля опухолевого процесса, полученные анализом сцДНК, дополненные данными анализа циркулирующих опухолевых клеток, РНК и белковых маркеров, позволяют дополнять и предварять классическую биопсию и в итоге найти и оптимизировать комплексный подход к персонифицированному лечению больных РМЖ [7].
Вопросам диагностики РМЖ на основе метилирования генов в сцДНК плазмы или сыворотки периферической крови посвящен ряд обзоров [8, 9]. Однако получено также много экспериментальных данных по оценке метилирования генов в сцДНК как факторов выживаемости и химиорезистентности при РМЖ, которые не получили должного обсуждения в мировой литературе. В представленном обзоре суммированы и обобщены накопленные за последнюю декаду данные о прогностическом и предиктивном значении метилирования генов и их наборов в сцДНК плазмы или сыворотки крови у пациентов с РМЖ.
СВОБОДНАЯ ЦИРКУЛИРУЮЩАЯ ДНК КРОВИ В НОРМЕ И ПАТОЛОГИИ
Свободная циркулирующая ДНК (сцДНК, cell-free circulating DNA) в норме у здорового человека имеет эндогенное происхождение и появляется в кровотоке в результате гибели ядросодержащих клеточных элементов, созревания эритроцитов и тромбоцитов и в результате активной секреции нуклеиновых кислот во внеклеточное пространство [10]. Экзогенным источником сцДНК в кровотоке могут быть вирусные и бактериальные агенты при развитии инфекционных заболеваний и ДНК плода при беременности [11, 12].
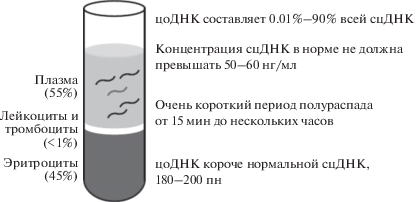
Концентрация сцДНК в крови здоровых людей, измеренная различными методами, составляет: спектрофотометрией – 15–50 нг/мл, радиоиммуноанализом – 14 нг/мл, методами с флуоресцентными красителями – 7–57 нг/мл, количественной ПЦР – 16.57 нг/мл, и в норме не должна превышать 50–60 нг/мл [10]. Концентрация сцДНК является результатом динамического равновесия между скоростью поступления ДНК в кровоток и скоростью ее гидролиза сывороточными нуклеазами и выведением олигомеров из циркуляции. СцДНК выводится из кровотока в печени и почках. Среднее время полураспада сцДНК в крови составляет 10–15 мин [13]. С помощью анализа паттерна метилирования сцДНК плазмы крови здоровых доноров определено, что сцДНК в норме происходит из белых кровяных клеток (55%), предшественников эритроцитов (30%), эндотелиальных клеток сосудов (10%) и гепатоцитов (1%) [14].
СцДНК может состоять из фракций разного размера, что отражает отличие в способе выхода из клеток. Основная фракция сцДНК в норме выходит из клеток в результате апоптоза, при котором образуются фрагменты ДНК длиной 180–200 пн и кратные этой длине мультимеры. При некрозе в кровоток попадает высокомолекулярная ДНК, размеры фрагментов которой могут быть длиной более 10 тпн [15]. В кровяном потоке сцДНК представлены в основном в комплексе с нуклеосомами [16], а также могут находиться в комплексе с сывороточными белками, в составе апоптотических телец, быть адсорбированными на поверхности форменных элементов крови [17]. Концентрация сцДНК в кровотоке повышается при травматическом поражении организма [18], радиационном поражении организма [19], аутоиммунных заболеваниях [20] и при онкологических заболеваниях [21].
Свободная циркулирующая ДНК крови при канцерогенезе происходит из различных источников, включающих первичную опухоль, циркулирующие опухолевые клетки, отдаленные метастазы, а также из здоровых гемопоэтических и стромальных клеток. Быстрое накопление в крови сцДНК происходит в основном благодаря массивному выходу ДНК из клеток, подвергшихся апоптозу и некрозу. Свой вклад в увеличение концентрации сцДНК в крови также вносит снижение скорости удаления сцДНК из кровяного потока, что обусловлено множественной органной дисфункцией на фоне системного воспалительного процесса у пациентов с онкологией [22]. Концентрация сцДНК в крови варьирует у разных пациентов и зависит от типа, местоположения и стадии опухоли, в ряде случаев не выходя за пределы нормы [23, 24]. Фракция циркулирующей опухолевой ДНК (цоДНК), которая происходит из клеток опухоли, составляет от 0.01 до 90% всей сцДНК (рис. 1).
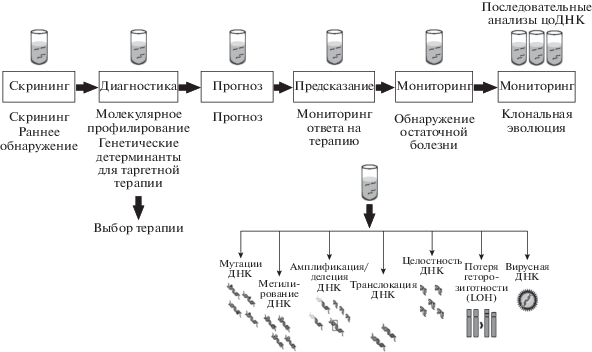
Многочисленные исследования продемонстрировали клиническую ценность анализа сцДНК на всех стадиях лечения: при диагностике, прогнозе, молекулярной классификации, при подборе стратегии лечения, определении ответа на терапию и при выявлении рецидива [25] (рис. 2).
Паттерн метилирования сцДНК при онкопатологиях отличается от такового в норме [26]. Продемонстрирован высокий потенциал метилирования сцДНК не только при обнаружении злокачественного процесса, но и при определении места локализации первичной опухоли [27].
ЦИРКУЛИРУЮЩАЯ ДНК ПРИ РМЖ
Исследования регистрируют факт повышения концентрации сцДНК у пациентов с РМЖ [21]. Оценка концентрации сцДНК в плазме/сыворотке крови проведена различными способами. Так, с помощью ПЦР в реальном времени получены следующие значения: маркер HBB – 65 нг/мл (интервал 9–566) против 13 нг/мл (интервал 3–73) у здоровых доноров [28]; маркер GAPDH – 41 600 гэ/мл (геном-эквивалентов/мл) (интервал 3644–192 482) в сыворотке против 14 764 гэ/мл (интервал 2198–87 450) у здоровых [29]; маркер GAPDH – 471 нг/мл (предел отсечения при ROC-анализе) [30]; маркер TERT – 105.2 нг/мл против 77.06 нг/мл у здоровых [31]. Концентрация сцДНК существенно снижается после хирургического удаления опухоли – с 105.5 до 59 нг/мл. В то же время увеличение концентрации сцДНК после хирургического вмешательства является предиктивным фактором рецидива [31].
Похожую тенденцию показывает исследование с применением прямого флуоресцентного измерения концентрации сцДНК: 1010 ± 642 нг/мл – перед хирургическим вмешательством и 398 ± 162 нг/мл – после, что соответствует уровню контрольной здоровой группы – 395 ± 248 нг/мл. Уровень сцДНК коррелировал с размером опухоли, метастазами в региональных лимфоузлах и со стадией [32]. Методом флуориметрии показано, что уровень сцДНК в плазме повышается в процессе прогрессирования РМЖ и снижается после хирургического лечения. Медианная концентрация сцДНК в плазме составляет 0.5, 235, 422, 1280 и 0.5 нг/мл у пациентов с РМЖ, классифицированными по стадиям I, II, III, IV и после хирургического вмешательства соответственно [33].
Важная проблема в разработке методов диагностики РМЖ – поиск предела содержания сцДНК, после которого однозначно можно говорить о начале злокачественного процесса. Выявление высоких концентраций тотальной сцДНК не является опухоль-специфичным биомаркером. Продуктивным путем применения маркера концентрации сцДНК в онкологической практике является измерение в составе тотальной сцДНК доли цоДНК. Доля цоДНК, определенная с использованием маркеров на основе химерных генов и изменения копийности, варьировала от 1.4 до 47.9%. Выявление более 0.75% цоДНК позволило диагностировать злокачественный процесс с чувствительностью 90% и специфичностью 99% [34].
СОВРЕМЕННЫЕ МЕТОДЫ ОПРЕДЕЛЕНИЯ МЕТИЛИРОВАНИЯ ЦЕЛЕВЫХ ЛОКУСОВ В сцДНК ПЛАЗМЫ И СЫВОРОТКИ ПЕРИФЕРИЧЕСКОЙ КРОВИ ПРИ РМЖ
Маркеры на основе определения метилирования целевых локусов в сцДНК обладают характеристиками, привлекательными для практического клинического применения, – стабильностью, ранним проявлением, различием паттернов при различной локализации опухолевого очага [35–37].
Получение надежных данных с помощью маркеров метилирования требует тщательной верификации и оптимизации преаналитических процедур выделения фракции сцДНК. Ограничения на использование стандартных методов возникают в силу того, что сцДНК присутствуют в ограниченном количестве, сильно фрагментированы, и часто необходимо определять статус метилирования отдельных молекул, составляющих минорную часть пула всех внеклеточных ДНК [38]. Влияние на конечный результат может оказать не столько низкая концентрация сцДНК, сколько низкая доля цоДНК, а также загрязнение фракции свободной ДНК в результате лизиса клеточных компонентов крови. Неоптимизированная процедура обращения с кровью способствует выходу геномной ДНК из лейкоцитов, что существенно снижает возможность детекции опухолевых маркеров [39, 40].
Для определения метилирования сцДНК плазмы и сыворотки крови при РМЖ применяются как методы, основанные на бисульфитной конверсии ДНК, так и методы с использованием метил-чувствительных рестриктаз. Методы, основанные на использовании метил-чувствительных рестриктаз, в современной практике применяются редко и для повышения производительности сочетаются с высокопроизводительными методами анализа. Одним из таких методов является анализ MethDet56 (methylation detection), позволяющий исследовать с помощью чипа метилирование 56 генов [38, 41].
Методы, основанные на бисульфитной конверсии, являются золотым стандартом в эпигенетических исследованиях. Метод основан на способности гидросульфита взаимодействовать с цитозином в составе одноцепочечной ДНК с конверсией последнего в урацил; 5-метилцитозин при тех же условиях модификации не подвергается [42]. Основным недостатком метода бисульфитной конверсии является существенная деградация ДНК – до 90% от поступившего в реакцию генетического материала [43]. Требуется оптимизация стадии бисульфитной конверсии ДНК при работе с таким малым количеством генетического материала. Оптимизация проводилась по параметрам времени реакции, концентрации реагентов, температуры реакции и совмещения с процедурами очистки [44]. Еще одним способом оптимизации всей процедуры анализа метилирования в сцДНК стало исключение процедуры выделения ДНК из плазмы и сыворотки крови классическими методами (метод одношаговой МС-ПЦР (one-step MSP, OS-MSP)) [45].
Анализ бисульфит-конвертированной ДНК проводят в зависимости от поставленной задачи с помощью вариантов МС-ПЦР, таргетного или полногеномного секвенирования на NGS-секвенаторах. Метил-специфическая ПЦР основана на применении двух пар праймеров – для метилированной и неметилированной бисульфит-конвертированной ДНК [46]. Оптимизированная МС-ПЦР является высокочувствительным методом, который позволяет детектировать одиночный метилированный аллель, приходящийся на тысячу неметилированных [47]. Количественный вариант МС-ПЦР улучшает чувствительность, специфичность и воспроизводимость метода и позволяет детектировать метилированный аллель в присутствии 10 000 неметилированных [48]. Разработано несколько вариантов количественного МС-ПЦР – MethyLight на основе TaqMan проб [48], метил-чувствительный анализ кривых плавления с высоким разрешением (methylation-sensitive high-resolution melting analysis, MS-HRM) [49], капельная цифровая метил-специфичная ПЦР (droplet digital methylation-specific PCR, ddMSP) [50], мультиплексная количественная МС-ПЦР cMethDNA [51]. Для масштабного анализа метилирования ДНК (как геномной, так и внеклеточной) существуют два основных подхода: технологии с использованием микрочипов (microarray) и массового параллельного секвенирования. Наиболее информативным методом с полным покрытием генома является метод полногеномного бисульфитного секвенирования (Whole-Genome Bisulfite Sequencing, WGBS) [52]. Несмотря на информативность, метод WGBS не полностью отвечает критериям для практического клинического применения в силу высокой стоимости, низкого покрытия, низкой скорости получения и обработки данных [53]. Более практично применение таргетного секвенирования бисульфит-конвертированной ДНК [54, 55].
МЕТИЛИРОВАНИЕ ЦИРКУЛИРУЮЩЕЙ ДНК ПРИ РМЖ
Процесс злокачественной трансформации характеризуется глобальным гипометилированием и гиперметилированием отдельных локусов геномной ДНК. При новообразованиях молочной железы существенно меняется не только количество сцДНК, но и ее свойства, в частности метилирование CpG-динуклеотидов. Для фракции сцДНК плазмы крови наблюдается схожий эффект, что и в тканях злокачественных новообразований молочной железы, – существенное снижение глобального геномного метилирования 5метС в сравнении как с нормой, так и с доброкачественными новообразованиями. Уровень глобального гипометилирования сцДНК при РМЖ коррелирует со стадией заболевания и метастазированием в региональные лимфоузлы [56]. Схожие результаты получены при исследовании метилирования тотальной ДНК периферической крови, которая включает также ДНК форменных элементов крови [57, 58]. Однако другие исследования показывают, что уровень метилирования тотальной ДНК периферической крови, измеренный по метилированию LINE-1 регионов, может не отличаться от нормы [59]. Кроме того, обнаружено существенное различие уровня метилирования геномной ДНК лейкоцитов крови здоровых женщин и больных РМЖ; так, гипометилирование геномной ДНК лейкоцитов крови ассоциировано с риском возникновения РМЖ [60, 61].
Гиперметилирование CpG-островков промоторных регионов генов-супрессоров опухолевого роста обнаруживается с высокой частотой при РМЖ и гиперметилированные гены используются как эффективные маркеры РМЖ [62].
ГИПЕРМЕТИЛИРОВАНИЕ ОТДЕЛЬНЫХ ЛОКУСОВ В сцДНК ПЛАЗМЫ И СЫВОРОТКИ ПЕРИФЕРИЧЕСКОЙ КРОВИ КАК ПРОГНОСТИЧЕСКИЙ ФАКТОР ПРИ РМЖ
Прогностические маркеры позволяют осуществить подразделение пациентов на группы риска и использовать дифференцированные схемы лечения, что позитивно отражается на качестве жизни пациентов и эффективности терапии. В клинической практике маркеры на основе определения гиперметилирования отдельных генов, детектируемых во фракции сцДНК, могут являться независимыми прогностическими факторами общей и опухоль-специфической выживаемости при первичном РМЖ. Маркеры гиперметилирования отдельных генов могут быть также ассоциированы с основными прогностическими факторами: размером опухоли, метастазами в региональных лимфоузлах, отдаленными метастазами, экспрессией генов рецепторов гормонов, наличием циркулирующих опухолевых клеток.
Так, обнаружение метилирования RASSF1A и/или APC в плазме крови пациентов до применения химиотерапии независимо ассоциировано с негативным прогнозом общей выживаемости (p = = 0.001) [63].
Гиперметилирование промотора гена BRCA1 в сцДНК плазмы крови ассоциировано с высокой вероятностью рецидива, негативным прогнозом как опухоль-специфической (p ≤ 0.0001; медиана 14.2 мес.), так и общей выживаемости (p = 0.0001; медиана 24.3 мес.). Гиперметилирование GSTP1 в сцДНК плазмы крови также ассоциировано с высокой вероятностью рецидива и негативным прогнозом опухоль-специфической выживаемости (p = 0.03, медиана 24.2 мес.) [64].
К маркерам худшего прогноза общей и опухоль-специфической выживаемости относятся гиперметилирование PITX2 (p = 0.001 и p = 0.023 соответственно) и гиперметилирование RASSF1A (p = 0.001 и p = 0.004). Мультивариантный анализ выживаемости показывает, что маркеры метилирования PITX2 и RASSF1A являются независимым от терапии прогностическим фактором общей выживаемости (р = 0.021, р < 0.001) [65]. Выявление метилирования минимум одного из маркеров GSTP1, RASSF1A, RARB в плазме крови пациентов с I и II клиническими стадиями указывает на худший прогноз общей выживаемости (p = 0.002).
Высокую концентрацию в плазме крови сцДНК в целом можно тоже использовать для негативного прогноза общей выживаемости (p = 0.001). Пациенты с высокой концентрацией сцДНК, а также с метилированием ряда маркеров показывали худший прогноз общей выживаемости (p = 0.001) [66].
Метилирование RARB и ESR2 наблюдали в 20 и 50% образцов сыворотки крови, что хорошо коррелировало с обнаружением в ткани опухоли. Гиперметилирование RARb2 в сыворотке крови ассоциировано с плохим прогнозом опухоль-специфической (p = 0.0001; медиана – 22.1 мес.), и общей выживаемости (p = 0.002; медиана – 23.2 мес.) [67].
Метилирование SOX17 обнаружено в 72.9% образцов опухолевой ткани и 58.1% сцДНК плазмы крови больных, а в ткани и плазме здоровых доноров метилирование SOX17 не выявляется. Важно, что наблюдается существенная корреляция между обнаружением метилирования в опухолевой ткани и плазме крови того же пациента (rs = 0.688, р < 0.001). Метилирование промотора SOX17 в ткани и плазме ассоциировано со стадией болезни (p < 0.001 и p = 0.001 соответственно) и метастазами в лимфоузлах (p < 0.001 и p = 0.001 соответственно). Мультивариантный анализ демонстрирует, что обнаружение метилирования SOX17 является независимым маркером плохого прогноза и общей (p = 0.02) и опухоль-специфической (p = 0.001) выживаемости [68].
Определены гены, метилирование которых в сцДНК может выступать эффективным прогностическим фактором общей и опухоль-специфической выживаемости при метастатическом РМЖ. Метилирование промоторной области гена SEPTIN9 обнаруживали в сцДНК плазмы крови у 11% (9/82) пациентов с первичным РМЖ, у 52% (26/50) пациентов с метастатическим РМЖ и не выявляли у здоровых доноров (0/51) [69]. Количественные показатели метилирования панели из шести генов-маркеров (AKR1B1, HOXB4, RASGRF2, RASSF1, HIT1H3C, TM6SF1), суммированные в кумулятивном индексе метилирования, позволяют дать прогноз метастатического РМЖ. Так, медианы выживаемости без прогрессирования заболевания и общей выживаемости существенно короче у пациентов с высоким кумулятивным индексом метилирования (2.1 и 12.3 мес. соответственно), чем у пациентов с низким индексом (5.8 и 21.7 мес. соответственно) [70].
Выявление метилирования трех любых генов из набора маркеров (KLK10, SOX17, WNT5A, MSH2) у пациентов с метастатическим РМЖ с ранее выявленным метилированием SOX17 или WNT5A ухудшает прогноз общей выживаемости (p = 0.042, p = 0.043, p = 0.048 соответственно). Особенно заметное снижение общей выживаемости (p = 0.011) и высокая вероятность летального исхода (p = 0.017) характерны для сочетания HER2/neu фенотипа и метилирования SOX17. Метилирование всех четырех генов указанного выше набора коррелирует с отсутствием ответа на химиотерапию (p = 0.002). В группе, получившей адъювантную терапию, обнаружение метилирования KLK10 ассоциировано с рецидивом болезни (p = 0.008) и снижением интервала до выявления рецидива (p = 0.013) [71].
Обнаружение маркеров метилирования коррелирует с присутствием в кровотоке циркулирующих опухолевых клеток. Исследована связь между клиническими маркерами метастатического процесса: циркулирующими опухолевыми клетками и метилированием сцДНК плазмы крови. Гиперметилирование одного и более генов APC, RASSF1A и ESR1 обнаружено в плазме 42 из 80 пациентов (53%), притом, что частота метилирования этих генов по отдельности составила 29, 35 и 20% соответственно, в то время как в контрольной группе метилирование этих генов найдено только у 3 из 20 (15%). Выявление маркеров метилирования в плазме коррелирует с обнаружением циркулирующих опухолевых клеток в крови (p = = 0.03) пациентов с РМЖ. Данный эффект может быть объяснен как тем, что циркулирующие опухолевые клетки выступают источником метилированной сцДНК в плазме крови, так и тем, что присутствие клеток и метилированной сцДНК является фенотипическим проявлением агрессивной опухоли [72].
Установлена корреляция между обнаружением в крови циркулирующих опухолевых клеток и определением метилированных генов APC (p = = 0.012) и GSTP1 (p = 0.001) в сцДНК плазмы пациентов с РМЖ. Обнаружение метилированных генов APC, RASSF1A и циркулирующих опухолевых клеток коррелирует с агрессивными и прогрессирующими типами РМЖ (p = 0.001, p = 0.031). Частота встречаемости метилирования APC и RASSF1A различается между метастатическим РМЖ и РМЖ без метастазов; метилированный GSTP1 чаще обнаруживается в плазме пациентов с большими опухолями (p = 0.023) и коррелирует с позитивным Her2/neu статусом (p = 0.003) [73].
Промотор гена SOX17 гиперметилирован в 86% первичных опухолей. В ДНК циркулирующих опухолевых клеток SOX17 метилирован в 34.5% ранних случаев РМЖ, 45.8% случаев метастатического РМЖ и в 1.6% здоровых доноров; в парных образцах сцДНК плазмы SOX17 метилирован в 34.5, 40.7 и 1.6% случаев соответственно. Обнаружена существенная корреляция между уровнем метилирования SOX17 в ДНК циркулирующих опухолевых клеток и сцДНК на ранних стадиях РМЖ, но не у пациентов с метастатическим РМЖ. Эти данные могут указывать на роль циркулирующих опухолевых клеток в формировании пула сцДНК плазмы периферической крови [74].
Эпигенетические механизмы вовлечены в подавление экспрессии гена эстрогенового рецептора при РМЖ, который является важным маркером прогноза выживаемости и предиктивным маркером ответа на гормональную терапию тамоксифеном и ингибиторами ароматазы. На выборке из 100 пациентов с РМЖ установлена корреляция между гиперметилированием промоторов ESR1 и PGR в плазме крови с ERα-негативным типом РМЖ (p = 0.003 и p = 0.001). Обнаружено также значительное гиперметилирование промоторов ESR1 и PGR в сцДНК плазмы при тройном негативном РМЖ (p = 0.03 и p = 0.05) [75].
Выявлена значимая ассоциация между метилированием промотора гена ESR1 в сцДНК плазмы крови и ER-статусом опухоли (p = 0.0179). Более высокие значения уровня метилирования ESR1 показывают подтипы РМЖ с плохим прогнозом – тройной негативный и Her2+ (80 и 60% соответственно), чем подтипы с лучшим прогнозом – люминальный А и Б (28 и 5.9% соответственно) [76].
Метилирование участков промотора гена ESR1 – ER3, ER4, ER5 обнаружено с частотами 65, 26.7, 61.7% в опухолевой ткани и с очень близкими частотами 57.5, 21.7, 55.8% – в сыворотке крови. Обнаружена значимая связь между метилированием ER3, ER4, ER5 в ткани и сыворотке и отрицательным статусом рецептора эстрогена ER (0.007, 0.001, 0.003 для сыворотки соответственно) [77].
Выявлена также связь между эндокринным фенотипом опухоли и метилированием других маркеров. Обнаружена корреляция между гиперметилированием BRCA1 в тканях опухоли и плазме крови с негативным статусом ER, PR и HER2, а также тройным негативным РМЖ на выборке из 100 пациентов с РМЖ [64]. Отсутствие метилирования WNT5A коррелирует с фенотипом ER+, PR+, HER2– в группе адъювантной терапии и метастатической группе (p = 0.040 и p = 0.016 соответственно). Отсутствие метилирования KLK10 статистически значимо для группы с фенотипом HER2–, PR+, получившей адъювантную терапию, и группы в устойчивой ремиссии (p = 0.027 и p = 0.004 соответственно). Присутствие метилирования KLK10 при тройном негативном РМЖ ассоциировано с высокой вероятностью рецидива (p = 0.014) [71].
Анализ метилома сцДНК при химиотерапии ER+ РМЖ эксеместаном позволяет выявить группу устойчивых к лечению опухолей. Выявлено семь регионов генома, связанных с резистентностью к эксеместану, которые характеризуются различной плотностью метилирования. ROC-анализ маркеров метилирования перед гормональной терапией выявил, что высокая плотность метилирования Chr10 (46 400 000–46 599 999, PTPN20; AUC = 0.909, p = 0.019), Chr3 (67 800 000–67 999 999; SUCLG2-AS1; AUC = 0.932, p = 0.013), Chr3 (140 200 000–140 399 999; CLSTN2; CLSTN2-AS1; TRIM42; AUC = 0.864, p = 0.037) и Chr12 (101 200 000–101 399 999; ANO4; AUC = 0.886, p = = 0.026) предсказывает устойчивую к терапии группу опухолей [78]. Данные по прогностическим факторам РМЖ сведены в табл. 1.
Таблица 1.
Прогностические маркеры на основе определения гиперметилирования генов во фракции сцДНК плазмы крови при РМЖ
| Маркеры | Тип образца | Метод | Выборка пациентов и доноров | Клиническое значение | Ссылка |
|---|---|---|---|---|---|
| APC, RASSF1A, ESR1 | Плазма и сыворотка | Количественная МС-ПЦР | 80 пациентов, 20 здоровых доноров | Корреляция с обнаружением циркулирующих опухолевых клеток и прогноз общей и опухоль-специфической выживаемости | [72] |
| APC, GSTP1, RASSF1 | Сыворотка | MethyLight | 85 пациентов, 22 здоровых донора | То же | [73] |
| SOX17 | Плазма | Количественная МС-ПЦР | 114 пациентов с первичным и метастатическим РМЖ, 60 здоровых доноров | » | [74] |
| ESR1, PGR | Сыворотка | МС-ПЦР | 100 пациентов | Корреляция с эндокринным фенотипом опухоли | [75] |
| ESR1 | Плазма | Количественная МС-ПЦР | 110 пациентов | То же | [76] |
| ESR1 | Сыворотка | МС-ПЦР | 120 пациентов | » | [77] |
| BRCA1, GSTP1 | Сыворотка | МС-ПЦР | 100 пациентов | » | [64] |
| SEPTIN9 | Плазма | Количественный МСП-ПЦР | 132 пациента, 51 здоровый донор | Прогноз общей и опухоль-специфической выживаемости | [69] |
| RASSF1A, APC | Сыворотка | MethyLight | 86 пациентов, 10 здоровых доноров | То же | [63] |
| PITX2, RASSF1A | Плазма и сыворотка | MethyLight | 428 пациентов | » | [65] |
| GSTP1, RASSF1A, RARB | Сыворотка | Одношаговая МС-ПЦР (OS-MSP) | 336 пациентов | » | [66] |
| ESR2, RARB | Сыворотка | МС-ПЦР | 100 пациентов, 30 здоровых доноров | » | [67] |
| SOX17 | Плазма | МС-ПЦР | 155 пациентов, 60 здоровых доноров | » | [68] |
| AKR1B1, HOXB4, RASGRF2, RASSF1, HIT1H3C, TM6SF1 | Сыворотка | cMethDNA | 141 пациент | » | [70] |
| KLK10, SOX17, WNT5A, MSH2 | Плазма | Количественная МС-ПЦР | 200 пациентов, 35 здоровых доноров | Прогноз общей и опухоль-специфической выживаемости, прогноз резистентности к химиотерапии | [71] |
| Chr3 (67 800 000–67 999 999), Chr3 (140 200 000–140 399 999;), Chr12 (101 200 000–101 399 999), Chr6 (32 400 000–32 599 999) | Плазма | Полногеномное секвенирование бисульфит-конвертированной ДНК | 16 пациентов | Прогноз резистентности к химиотерапии | [78] |
ГИПЕРМЕТИЛИРОВАНИЕ ОТДЕЛЬНЫХ ЛОКУСОВ В сцДНК ПЛАЗМЫ И СЫВОРОТКИ ПЕРИФЕРИЧЕСКОЙ КРОВИ КАК ПРЕДИКТИВНЫЙ ФАКТОР ПРИ РМЖ
В клинической практике маркеры на основе определения гиперметилирования отдельных генов, детектируемых во фракции сцДНК, могут применяться при контроле эффективности ответа на адъювантную и неоадъювантную химиотерапию и терапию метастатического РМЖ.
Неоадъювантная химиотерапия (НАХТ) проводится с целью уменьшения объема первичной опухоли, размеров и количества пораженных лимфатических узлов, элиминации отдаленных микрометастазов и повышения безрецидивной и общей выживаемости. Статистические оценки эффективности НАХТ разнятся в зависимости от иммуно-гистохимического типа РМЖ: для трижды негативного от 30 до 60% [79], для ER+ опухолей от 2–10% [80] до 69% [81], для HER2+ от 30–70% [82]. Выявление ответа на химиотерапию является важной клинической задачей.
Исследована динамика метилирования гена RASSF1A в плазме крови у группы из 21 пациента, прошедших НАХТ [83]. В четырех случаях, в которых достигнут полноценный ответ на терапию, метилирование RASSF1A в плазме снижается и не детектируется с 12-й нед. Напротив, у 17 пациентов с частичным или минимальным ответом на терапию метилирование RASSF1A в плазме детектируется дольше, либо вовсе сохраняется (p = 0.02).
Выявлено различие между реагирующей и резистентной группами до применения терапии по сниженной частоте метилирования маркеров BRCA1 и GSTP1 (p = 0.02 и p = 0.003 соответственно) у группы прошедших НАХТ пациентов с РМЖ [84]. Проанализирован уровень метилирования сцДНК плазмы крови пациентов в шести циклах химиотерапии по пяти маркерам: BRCA1, MGMT, GSTP1, SFN и MDR1. Выявлено, что метилирование BRCA1 существенно коррелирует с ответом на терапию и различается между реагирующей и резистентной группами. Корреляция отмечена также между метилированием BRCA1 и негативным статусом рецепторов гормонов.
Метилирование промотора RASSF1A в сцДНК плазмы крови обнаружено в 23% (20/87) образцов до НАХТ, что существенно выше, чем показатели обнаружения антигенов CEA (8.6%) и CA15-3 (7.4%) [85]. Отмечено снижение метилирования RASSF1A на выборке в целом, но степень изменения уровня метилирования зависит от ответа на терапию: наибольшее снижение наблюдается в группе пациентов с положительным откликом (р = 0.006) и в меньшей степени у резистентных пациентов. Сохранение метилирования после терапии коррелирует со степенью остаточной активности опухолевого процесса (р = 0.008). Прогностическое значение имеет сравнение уровня метилирования RASSF1A в сцДНК плазмы крови после года с момента хирургического лечения и после НАХТ. В группе пациентов, отрицательной по метилированию RASSF1A (37 пациентов), и в группе, в которой метилирования RASSF1A не обнаружено спустя год после лечения (10 пациентов), наблюдается устойчивая ремиссия. В то же время среди пациентов с метилированием RASSF1A до лечения (шесть пациентов) и с метилированием до лечения и спустя год после лечения (один пациент) – три пациента имели рецидив с метастазами.
На биопсийном материале опухолей РМЖ выявлены дифференциально метилированные регионы геномной ДНК, которые различаются между группой, отвечающей на НАХТ, и группой резистентной к терапии [86]. Проанализировано состояние метилирования промоторных областей генов SLC9A3, C1QL2, DPYS, IRF4, ADCY8, KCNQ2, TERT, SYNDIG1, SKOR2 и GRIK1 в материале опухоли и плазме крови для их использования в качестве предиктивных маркеров ответа на НАХТ у больных РМЖ. Наилучшие результаты показала диагностическая панель, состоящая всего из двух маркеров IRF4 и C1QL2, имеющая диагностическую точность 0.75 с чувствительностью 75% и специфичностью 75% [86]. В другом исследовании на выборке из 36 пациентов с РМЖ выявлено статистически значимое различие в частотах метилирования промотора гена ADCY8 в плазме крови между группами с различной степенью ответа на терапию – метилирование чаще наблюдалось в группе с плохим ответом на лечение [87].
Исследован способ определения и количественной оценки фракции сцДНК, происходящей из ткани РМЖ и нормальной ткани молочной железы, с помощью маркеров метилирования [88]. Использование маркеров на основе однонуклеотидных замен показывает низкие значения чувствительности и специфичности. Значение концентрации тотальной сцДНК плазмы крови не обладает способностью различать пациентов с РМЖ и здоровых доноров. Поэтому были использованы маркеры метилирования KRT19, LMX1B, ZNF296, специфические только для ткани молочной железы, что позволило получить высокие значения диагностического теста (чувствительность 80% и специфичность 97%). На 29 пациентах, которые получали НАХТ перед операцией, проследили динамику изменения концентрации цоДНК, специфичной для ткани молочной железы. Через 2–4 нед. после начала терапии наблюдается резкое уменьшение концентрации специфической сцДНК, которая достигает низшего значения сразу после операции, не отличаясь от здоровых показателей. Напротив, пациенты, у которых ответа на терапию не наблюдалось, перед операцией демонстрировали повышенный уровень специфической сцДНК. Уровень специфической сцДНК, измеренный в последний период неоадъювантной терапии, является предиктивным фактором остаточного заболевания. Уровень специфической сцДНК существенно ниже, по сравнению со значением перед началом терапии, у пациентов с полноценным ответом [88].
Адъювантная терапия с использованием гормональных препаратов, применяемая после хирургического удаления опухоли, способна увеличивать как безрецидивную, так и общую выживаемость больных. При гормонотерапии РМЖ применяют ингибиторы ароматазы: нестероидные (летрозол (Фемара), анастрозол (Аримидекс)) и стероидные (эксеместан), а также конкурентный ингибитор эстрогеновых рецепторов – тамоксифен.
Статус метилирования RASSF1A в сцДНК 148 пациентов был проанализирован перед хирургическим лечением и спустя один год после операции с дополнительной адъювантной терапией тамоксифеном [89]. Было обнаружено, что метилирование RASSF1A является независимым предиктивным маркером негативного прогноза (относительный риск рецидива и смерти составил 5.1 и 6.9 соответственно). Также подтверждено, что измерение уровня метилирования RASSF1A в сцДНК позволяет отслеживать эффективность терапии. Более поздние исследования подтвердили, что гиперметилирование RASSF1A снижает терапевтический эффект доцетаксела (Таутакс) при терапии РМЖ [90] и что его уровни положительно коррелируют с экспрессией рецепторов эстрогена при РМЖ [91].
Исследована динамика изменения метилирования промоторов генов RARB, MSH2, MGMT, PGR и MDGI в сцДНК плазмы периферической крови при хирургическом лечении с последующей терапией тамоксифеном на выборке из 20 пациентов ER+ РМЖ с использованием метода MethDet-56 [92]. Профиль метилирования сцДНК плазмы крови изменился: сразу после хирургического лечения выявлено снижение метилирования промоторов RARB, MSH2 и MGMT; после применения тамоксифена возросло метилирование промотора ESR1; после оперативного лечения в комбинации с тамоксифеном выявлено снижение метилирования промоторов PGR, MDGI, MGMT и RARB.
Более 30% гормон-зависимых случаев РМЖ невосприимчивы к первичной гормонотерапии и около 40% из первоначально восприимчивых случаев приобретают устойчивость. Анализ метилома сцДНК при химиотерапии экземестаном больных РМЖ с ER+ позволяет определить группу устойчивых к лечению и проконтролировать эффективность лечения на опухолях, чувствительных к терапии [78]. Выявлено семь регионов генома, связанных с резистентностью к эксеместану, которые характеризуются существенно различной плотностью метилирования. Кроме первичной устойчивости, изменения в метилировании генома обнаружены для приобретенной устойчивости. Изменение в плотности метилирования Chr6 (32 400 000–32 599 999; LA-DRA; HLADRB5; HLADRB6; HLA-DRB1) является предиктором приобретенной к эксеместану резистентности (AUC = 0.900, р = = 0.024) и ассоциировано с коротким периодом до прогрессии заболевания (3.33 против 26.31 мес., p = 0.000033).
Метилирование маркера ESR1, обнаруженное в ДНК циркулирующих опухолевых клеток пациентов после химиотерапии препаратами эверолимус (Афинитор) или эксеместан, ассоциировано с отсутствием ответа на лечение (p = 0.023) [93].
Основным методом лечения метастатической формы РМЖ является химиотерапия. Единого стандарта лечения метастатического РМЖ не существует, что отражает комплексность заболевания и насущную необходимость в определении быстрых и надежных маркеров ответа на терапию.
Исследована связь между уровнем метилирования промоторов генов ESR1 и 14-3-3-σ в сцДНК плазмы крови и клиническими проявлениями у пациентов с метастазирующим РМЖ [94]. Уровень метилирования промотора 14-3-3-σ существенно различался между группой метастазирующего РМЖ и контрольной здоровой группой (р < 0.001), а также между группой в ремиссии и группой с метастазами (р < 0.001). При проведении химиотерапии метастазирующего РМЖ уровень метилирования снижался у большинства пациентов, однако обособлялась группа, у которой уровень оставался неизменным. ROC-анализ позволяет различить группу не отвечающих на терапию пациентов с помощью величины отношения уровня метилирования перед первым циклом химиотерапии к уровню метилирования перед вторым циклом с чувствительностью 75% и специфичностью 66.7% при отсекающем значении в 2.39 [94].
Значимое снижение суммарного индекса метилирования генов-маркеров выявлялось у пациентов с ответом на терапию или без прогрессирования заболевания (p = 0.010). Выборка состояла из 29 пациентов с метастатическим РМЖ, у которых взяли образцы сыворотки перед терапией доцетакселом или капецитабином и по прошествии 18–49 дней. Данный эффект снижения индекса метилирования не наблюдался для пациентов с прогрессирующим течением заболевания. При прослеживании группы из 13 пациентов в процессе терапии у 10 наблюдали динамический характер изменения индекса метилирования в зависимости от течения болезни: снижение метилирования при первоначальном ответе на терапию и увеличение метилирования при последующем прогрессировании заболевания [51].
Методами массового параллельного секвенирования исследованы метиломы сцДНК плазмы крови пациентов с метастатическим РМЖ (отдаленные метастазы) после терапии, пациентов в устойчивой ремиссии и здоровых доноров [53]. Данные указали на различие в метилировании около 5.0 × 106 CpG-динуклеотидов между группой метастатического РМЖ и группами в ремиссии и здоровыми донорами. Между группой в ремиссии и здоровыми донорами наблюдается существенное сходство. Метилом метастатического РМЖ характеризуется глобальным гипометилированием и гиперметилированием отдельных локусов. Анализ выявил 21 новых гиперметилированных при метастатическом РМЖ CpG-островков в локусах: BEND4, CDH4, C1QL3, ERG, GP5, GSC, HTR1B, LMX1B, MCF2L2, PAX5, PCDH10, PENK, REC8, RUNX3, SP8, SP9, STAC2, ULBP1, UNC13A, VIM, VWC2. Целенаправленное секвенирование локусов GP5, PCDH10, HRR1B и UNC13A показало хорошее соответствие с данными полногеномного секвенирования и продемонстрировало статистически значимое различие в величине метилирования между метастатическим РМЖ и группой в ремиссии и здоровых доноров. Данные по предиктивным маркерам РМЖ сведены в табл. 2.
Таблица 2.
Предиктивные маркеры на основе определения гиперметилирования генов во фракции сцДНК плазмы крови при РМЖ
| Маркеры | Тип образца | Метод | Выборка пациентов и доноров | Клиническое значение | Ссылка |
|---|---|---|---|---|---|
| Chr3 (67 800 000–67 999 999), Chr3 (140 200 000–140 399 999), Chr12 (101 200 000–101 399 999), Chr6 (32 400 000–32 599 999) | Плазма | Полногеномное секвенирование бисульфит-конвертированной ДНК | 16 пациентов | Контроль ответа на адъювантную гормональную химиотерапию РМЖ | [78] |
| RASSF1A | Сыворотка | MethyLight | 148 пациентов | То же | [89] |
| RARB, MSH2, MGMT, PGR, MDGI | Плазма | MethDet-56 | 20 пациентов ER+, 20 здоровых доноров | » | [92] |
| ESR1 | Плазма | Количественная МС-ПЦР | 58 пациентов, 108 пациентов, 30 здоровых доноров |
» | [93] |
| RASSF1A | Сыворотка | МС-ПЦР с анализом по метил-чувствительным кривым плавления (MS-HRM) | 52 пациента | Контроль ответа на неоадъювантную химиотерапию РМЖ | [83] |
| BRCA1 | Сыворотка | МС-ПЦР | 30 пациентов, 30 здоровых доноров | То же | [84] |
| RASSF1A | Плазма | Одношаговая МС-ПЦР (OS-MSP) | 87 пациентов | » | [85] |
| ADCY8 | Плазма | МС-ПЦР | 36 пациентов | » | [87] |
| KRT19, LMX1B, ZNF296 | Плазма | Высокопроизводительное таргетное секвенирование ампликонов конвертированной ДНК | 29 пациентов, 64 здоровых донора | » | [88] |
| AKR1B1, HOXB4, RASGRF2, RASSF1, HIT1H3C, TM6SF1 | Сыворотка | cMethDNA | 141 пациент | Контроль ответа на химиотерапию метастатического РМЖ | [70] |
| 14-3-3-σ | Сыворотка | Количественная МС-ПЦР | 77 пациентов без обнаруженных метастазов, 34 пациента с метастазами, 34 здоровых доноров | То же | [94] |
| AKR1B1, COL6A2, GPX7, HIST1H3, HOXB4, RASGRF2, TM6SF1, ARHGEF7, TMEFF2, RASSF1 | Сыворотка | cMethDNA | 29 пациентов | » | [51] |
| GP5, PCDH10, HRR1B, UNC13A | Плазма | Полногеномное секвенирование бисульфит-конвертированной ДНК, высокопроизводительное таргетное секвенирование ампликонов конвертированной ДНК | 40 пациентов, 40 здоровых доноров, 40 пациентов в ремиссии | » | [53] |
ЗАКЛЮЧЕНИЕ
Маркеры на основе метилирования отдельных генов сцДНК показывают значительный прогностический и предиктивный потенциал при РМЖ. Анализ сцДНК плазмы крови позволяет предсказать эффективность и проследить ответ на адъювантную и неоадъювантную химиотерапию. Важным преимуществом маркеров метилирования, в отличие от маркеров на основе однонуклеотидных замен, микросателлитов и вариаций числа копий, является универсальность, раннее проявление и ясная связь с биологией патологического процесса.
Вместе с обнадеживающими результатами, продемонстрированными на клиническом материале, необходимо отметить ряд объективных технических сложностей, возникающих на практике. Результаты анализа метилирования сцДНК зависят от преаналитической стадии выделения внеклеточной ДНК, которая может осуществляться различными способами и коммерческими наборами. Устоявшихся стандартов в данной области не существует. Исследователями сообщается о различной степени корреляции данных, полученных на биопсийном опухолевом материале и материале плазмы крови. Это может быть как техническим следствием стадии анализа и интерпретирования, которая требует процедур калибровки и нормировки, так и следствием достижения предела обнаружения применяемых методов. Устоявшихся стандартов в данной области также не существует. Концептуальный характер различия данных биопсийного материала и данных сцДНК продемонстрирован для маркеров на основе определения мутаций геномной ДНК. Все это требует дальнейшей работы для внесения ясности в техническую сторону вопроса и широкого внедрения анализа метилирования сцДНК в клиническую практику.
Работа выполнена в рамках Государственного задания Минобрнауки РФ при поддержке ПНИ 0403-2020-0002 на 2020–2022 годы.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Список литературы
Harbeck N., Penault-Llorca F., Cortes J. et al. Breast cancer // Nat. Rev. Dis. Primers. 2019. V. 5. № 1. e66. https://doi.org/10.1038/s41572-019-0111-2
Sørlie T., Perou C.M., Tibshirani R. et al. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications // Proc. Natl Acad. Sci. USA. 2001. V. 98. № 19. P. 10869–10874. https://doi.org/10.1073/pnas.191367098
Lehmann B.D., Jovanović B., Chen X. et al. Refinement of triple-negative breast cancer molecular subtypes: Implications for neoadjuvant chemotherapy selection // PLoS One. 2016. V. 11. № 6. e0157368. https://doi.org/10.1371/journal.pone.0157368
Dawson S.-J., Tsui D.W.Y., Murtaza M. et al. Analysis of circulating tumor DNA to monitor metastatic breast cancer // N. Engl. J. Med. 2013. V. 368. № 13. P. 1199–1209. https://doi.org/10.1056/NEJMoa1213261
Siravegna G., Marsoni S., Siena S. et al. Integrating liquid biopsies into the management of cancer // Nat. Rev. Clin. Oncol. 2017. V. 14. № 9. P. 531–548. https://doi.org/10.1038/nrclinonc.2017.14
Nassar F.J., Chamandi G., Tfaily M.A. et al. Peripheral blood-based biopsy for breast cancer risk prediction and early detection // Front Med. (Lausanne). 2020. V. 7. e28. https://doi.org/10.3389/fmed.2020.00028
Wan J.C.M., Massie C., Garcia-Corbacho J. et al. Liquid biopsies come of age: Towards implementation of circulating tumour DNA // Nat. Rev. Cancer. 2017. V. 17. № 4. P. 223–238. https://doi.org/10.1038/nrc.2017.7
Tzanikou E., Lianidou E. The potential of ctDNA analysis in breast cancer // Crit. Rev. Clin. Lab. Sci. 2020. V. 57. № 1. P. 54–72. https://doi.org/10.1080/10408363.2019.1670615
Constâncio V., Nunes S.P., Henrique R. et al. DNA Methylation-based testing in liquid biopsies as detection and prognostic biomarkers for the four major cancer types // Cells. 2020. V. 9. № 3. https://doi.org/10.3390/cells9030624
Тамкович С.Н., Власов В.В., Лактионов П.П. Циркулирующие ДНК крови и их использование в медицинской диагностике // Мол. биология. 2008. Т. 42. № 1. С. 12–23.
Rafi I., Chitty L. Cell-free fetal DNA and non-invasive prenatal diagnosis // Br. J. Gen. Pract. 2009. V. 59. № 562. e146–e148. https://doi.org/10.3399/bjgp09X420572
Han D., Li R., Shi J. et al. Liquid biopsy for infectious diseases: A focus on microbial cell-free DNA sequencing // Theranostics. 2020. V. 10. № 12. P. 5501–5513. https://doi.org/10.7150/thno.45554
Schwarzenbach H., Pantel K. Circulating DNA as biomarker in breast cancer // Breast Cancer Res. 2015. V. 17. № 1. e136. https://doi.org/10.1186/s13058-015-0645-5
Moss J., Magenheim J., Neiman D. et al. Comprehensive human cell-type methylation atlas reveals origins of circulating cell-free DNA in health and disease // Nat. Commun. 2018. V. 9. № 1. P. 5068. https://doi.org/10.1038/s41467-018-07466-6
Jahr S., Hentze H., Englisch S. et al. DNA fragments in the blood plasma of cancer patients: quantitations and evidence for their origin from apoptotic and necrotic cells // Cancer Res. 2001. V. 61. № 4. P. 1659–1665. PMID: 11245480
Roth C., Pantel K., Müller V. et al. Apoptosis-related deregulation of proteolytic activities and high serum levels of circulating nucleosomes and DNA in blood correlate with breast cancer progression // BMC Cancer. 2011. V. 11. P. 4. https://doi.org/10.1186/1471-2407-11-4
Rykova E.Y., Morozkin E.S., Ponomaryova A.A. et al. Cell-free and cell-bound circulating nucleic acid complexes: mechanisms of generation, concentration and content // Expert Opin. Biol. Ther. 2012. V. 12. Suppl 1. P. S141–S153. https://doi.org/10.1517/14712598.2012.673577
Rodrigues Filho E.M., Ikuta N., Simon D. et al. Prognostic value of circulating DNA levels in critically ill and trauma patients // Rev. Bras. Ter. Intensiva. 2014. V. 26. № 3. P. 305–312. https://doi.org/10.5935/0103-507X.20140043
Vasilyeva I.N. Low-molecular-weight DNA in blood plasma as an index of the influence of ionizing radiation // Ann. N.Y. Acad. Sci. 2001. V. 945. P. 221–228. https://doi.org/10.1111/j.1749-6632.2001.tb03889.x
Duvvuri B., Lood C. Cell-free DNA as a biomarker in autoimmune rheumatic diseases // Front Immunol. 2019. V. 10. P. 502. https://doi.org/10.3389/fimmu.2019.00502
Rohanizadegan M. Analysis of circulating tumor DNA in breast cancer as a diagnostic and prognostic biomarker // Cancer Genet. 2018. V. 228-229. P. 159–168. https://doi.org/10.1016/j.cancergen.2018.02.002
Delmonico L., Alves G., Bines J. Cell free DNA biology and its involvement in breast carcinogenesis // Adv. Clin. Chem. 2020. V. 97. P. 171–223. https://doi.org/10.1016/bs.acc.2019.12.006
Bettegowda C., Sausen M., Leary R.J. et al. Detection of circulating tumor DNA in early- and late-stage human malignancies // Sci. Transl. Med. 2014. V. 6. № 224. e224ra24. https://doi.org/10.1126/scitranslmed.3007094
Diehl F., Schmidt K., Choti M.A. et al. Circulating mutant DNA to assess tumor dynamics // Nat. Med. 2007. V. 14. № 9. P. 985–990. https://doi.org/10.1038/nm.1789
Stewart C.M., Kothari P.D., Mouliere F. et al. The value of cell-free DNA for molecular pathology // J. Pathol. 2018. V. 244. № 5. P. 616–627. https://doi.org/10.1002/path.5048
Li L., Choi J.-Y., Lee K.-M. et al. DNA methylation in peripheral blood: A potential biomarker for cancer molecular epidemiology // J. Epidemiol. 2012. V. 22. № 5. P. 384–394. https://doi.org/10.2188/jea.JE20120003
Kang S., Li Q., Chen Q. et al. CancerLocator: Non-invasive cancer diagnosis and tissue-of-origin prediction using methylation profiles of cell-free DNA // Genome Biol. 2017. V. 18. № 1. P. 53. https://doi.org/10.1186/s13059-017-1191-5
Huang Z.H., Li L.H., Hua D. Quantitative analysis of plasma circulating DNA at diagnosis and during follow-up of breast cancer patients // Cancer Lett. 2006. V. 243. № 1. P. 64–70. https://doi.org/10.1016/j.canlet.2005.11.027
Zanetti-Dällenbach R., Wight E., Fan A.X.-C. et al. Positive correlation of cell-free DNA in plasma/serum in patients with malignant and benign breast disease // Anticancer Res. 2008. V. 28. 2A. P. 921–925.
Gong B., Xue J., Yu J. et al. Cell-free DNA in blood is a potential diagnostic biomarker of breast cancer // Oncol. Lett. 2012. V. 3. № 4. P. 897–900. https://doi.org/10.3892/ol.2012.576
Catarino R., Ferreira M.M., Rodrigues H. et al. Quantification of free circulating tumor DNA as a diagnostic marker for breast cancer // DNA Cell Biol. 2008. V. 27. № 8. P. 415–421. https://doi.org/10.1089/dna.2008.0744
Agassi R., Czeiger D., Shaked G. et al. Measurement of circulating cell-free DNA levels by a simple fluorescent test in patients with breast cancer // Am. J. Clin. Pathol. 2015. V. 143. № 1. P. 18–24. https://doi.org/10.1309/AJCPI5YHG0OGFAHM
Tangvarasittichai O., Jaiwang W., Tangvarasittichai S. The plasma DNA concentration as a potential breast cancer screening marker // Indian J. Clin. Biochem. 2015. V. 30. № 1. P. 55–58. https://doi.org/10.1007/s12291-013-0407-z
Leary R.J., Sausen M., Kinde I. et al. Detection of chromosomal alterations in the circulation of cancer patients with whole-genome sequencing // Sci. Transl. Med. 2012. V. 4. № 162. e162ra154. https://doi.org/10.1126/scitranslmed.3004742
Wang J., Han X., Sun Y. DNA methylation signatures in circulating cell-free DNA as biomarkers for the early detection of cancer // Sci. China Life Sci. 2017. V. 60. № 4. P. 356–362. https://doi.org/10.1007/s11427-016-0253-7
Warton K., Samimi G. Methylation of cell-free circulating DNA in the diagnosis of cancer // Front. Mol. Biosci. 2015. V. 2. P. 13. https://doi.org/10.3389/fmolb.2015.00013
Issa J.P. DNA methylation as a clinical marker in oncology // J. Clin. Oncol. 2012. V. 30. № 20. P. 2566–2568. https://doi.org/10.1200/jco.2012.42.1016
Брызгунова О.Е., Лактионов П.П. Современные методы исследования метилирования внеклеточных ДНК // Мол. биология. 2017. Т. 51. № 2. С. 195–214. https://doi.org/10.7868/S0026898417010074
Parpart-Li S., Bartlett B., Popoli M. et al. The effect of preservative and temperature on the analysis of circulating tumor DNA // Clin. Cancer. Res. 2017. V. 23. № 10. P. 2471–2477. https://doi.org/10.1158/1078-0432.ccr-16-1691
Sherwood J.L., Corcoran C., Brown H. et al. Optimised pre-analytical methods improve KRAS mutation detection in circulating tumour DNA (ctDNA) from patients with Non-Small Cell Lung Cancer (NSCLC) // PLoS One. 2016. V. 11. № 2. e0150197. https://doi.org/10.1371/journal.pone.0150197
Liggett T.E., Melnikov A., Yi Q. et al. Distinctive DNA methylation patterns of cell-free plasma DNA in women with malignant ovarian tumors // Gynecol. Oncol. 2011. V. 120. № 1. P. 113–120. https://doi.org/10.1016/j.ygyno.2010.09.019
Darst R.P., Pardo C.E., Ai L. et al. Bisulfite sequencing of DNA // Curr. Protoc. Mol. Biol. 2010. Chapter 7. Unit 7.9.1-17. https://doi.org/10.1002/0471142727.mb0709s91
Tanaka K., Okamoto A. Degradation of DNA by bisulfite treatment // Bioorg. Med. Chem. Lett. 2007. V. 17. № 7. P. 1912–1915. https://doi.org/10.1016/j.bmcl.2007.01.040
Yi S., Long F., Cheng J. et al. An optimized rapid bisulfite conversion method with high recovery of cell-free DNA // BMC Mol. Biol. 2017. V. 18. № 1. P. 24. https://doi.org/10.1186/s12867-017-0101-4
Yamamoto N., Nakayama T., Kajita M. et al. Detection of aberrant promoter methylation of GSTP1, RASSF1A, and RARβ2 in serum DNA of patients with breast cancer by a newly established one-step methylation-specific PCR assay // Breast Cancer Res. Treat. 2012. V. 132. № 1. P. 165–173. https://doi.org/10.1007/s10549-011-1575-2
Herman J.G., Graff J.R., Myöhänen S. et al. Methylation-specific PCR: A novel PCR assay for methylation status of CpG islands // Proc. Natl Acad. Sci. USA. 1996. V. 93. № 18. P. 9821–9826. https://doi.org/10.1073/pnas.93.18.9821
Delpu Y., Cordelier P., Cho W.C. et al. DNA methylation and cancer diagnosis // Int. J. Mol. Sci. 2013. V. 14. № 7. P. 15029–15058. https://doi.org/10.3390/ijms140715029
Eads C.A., Danenberg K.D., Kawakami K. et al. MethyLight: A high-throughput assay to measure DNA methylation // Nucl. Acids Res. 2000. V. 28. № 8. e32.https://doi.org/10.1093/nar/28.8.e32
Wojdacz T.K., Dobrovic A., Hansen L.L. Methylation-sensitive high-resolution melting // Nat. Protoc. 2008. V. 3. № 12. P. 1903–1908. https://doi.org/10.1038/nprot.2008.191
Yu M., Heinzerling T.J., Grady W.M. DNA methylation analysis using droplet digital PCR // Methods Mol. Biol. 2018. V. 1768. P. 363–383. https://doi.org/10.1007/978-1-4939-7778-9_21
Fackler M.J., Lopez Bujanda Z., Umbricht C. et al. Novel methylated biomarkers and a robust assay to detect circulating tumor DNA in metastatic breast cancer // Cancer Res. 2014. V. 74. № 8. P. 2160–2170. https://doi.org/10.1158/0008-5472.CAN-13-3392
Beck S., Rakyan V.K. The methylome: Approaches for global DNA methylation profiling // Trends Genet. 2008. V. 24. № 5. P. 231–237. https://doi.org/10.1016/j.tig.2008.01.006
Legendre C., Gooden G.C., Johnson K. et al. Whole-genome bisulfite sequencing of cell-free DNA identifies signature associated with metastatic breast cancer // Clin. Epigenetics. 2015. V. 7. P. 100. https://doi.org/10.1186/s13148-015-0135-8
Samorodnitsky E., Datta J., Jewell B.M. et al. Comparison of custom capture for targeted next-generation DNA sequencing // J. Mol. Diagn. 2015. V. 17. № 1. P. 64–75. https://doi.org/10.1016/j.jmoldx.2014.09.009
Widschwendter M., Evans I., Jones A. et al. Methylation patterns in serum DNA for early identification of disseminated breast cancer // Genome Med. 2017. V. 9. № 1. P. 115. https://doi.org/10.1186/s13073-017-0499-9
Pasha H.A., Rezk N.A., Riad M.A. Circulating cell free nuclear DNA, mitochondrial DNA and global DNA methylation: Potential noninvasive biomarkers for breast cancer diagnosis // Cancer Invest. 2019. V. 37. № 9. P. 432–439. https://doi.org/10.1080/07357907.2019.1663864
Severi G., Southey M.C., English D.R. et al. Epigenome-wide methylation in DNA from peripheral blood as a marker of risk for breast cancer // Breast Cancer Res. Treat. 2014. V. 148. № 3. P. 665–673. https://doi.org/10.1007/s10549-014-3209-y
van Veldhoven K., Polidoro S., Baglietto L. et al. Epigenome-wide association study reveals decreased average methylation levels years before breast cancer diagnosis // Clin. Epigenetics. 2015. V. 7. № 1. P. 67. https://doi.org/10.1186/s13148-015-0104-2
Wu H.-C., Delgado-Cruzata L., Flom J.D. et al. Repetitive element DNA methylation levels in white blood cell DNA from sisters discordant for breast cancer from the New York site of the breast cancer family registry // Carcinogenesis. 2012. V. 33. № 10. P. 1946–1952. https://doi.org/10.1093/carcin/bgs201
Choi J.-Y., James S.R., Link P.A. et al. Association between global DNA hypomethylation in leukocytes and risk of breast cancer // Carcinogenesis. 2009. V. 30. № 11. P. 1889–1897. https://doi.org/10.1093/carcin/bgp143
Kuchiba A., Iwasaki M., Ono H. et al. Global methylation levels in peripheral blood leukocyte DNA by LUMA and breast cancer: A case-control study in Japanese women // Br. J. Cancer. 2014. V. 110. № 11. P. 2765–2771. https://doi.org/10.1038/bjc.2014.223
Tang Q., Cheng J., Cao X. et al. Blood-based DNA methylation as biomarker for breast cancer: a systematic review // Clin. Epigenetics. 2016. V. 8. e115. https://doi.org/10.1186/s13148-016-0282-6
Müller H.M., Widschwendter A., Fiegl H. et al. DNA methylation in serum of breast cancer patients: an independent prognostic marker // Cancer Res. 2003. V. 63. № 22. P. 7641–7645.
Sharma G., Mirza S., Parshad R. et al. Clinical significance of promoter hypermethylation of DNA repair genes in tumor and serum DNA in invasive ductal breast carcinoma patients // Life Sci. 2010. V. 87. № 3–4. P. 83–91. https://doi.org/10.1016/j.lfs.2010.05.001
Göbel G., Auer D., Gaugg I. et al. Prognostic significance of methylated RASSF1A and PITX2 genes in blood- and bone marrow plasma of breast cancer patients // Breast Cancer Res. Treat. 2011. V. 130. № 1. P. 109–117. https://doi.org/10.1007/s10549-010-1335-8
Fujita N., Nakayama T., Yamamoto N. et al. Methylated DNA and total DNA in serum detected by one-step methylation-specific PCR is predictive of poor prognosis for breast cancer patients // Oncology. 2012. V. 83. № 5. P. 273–282. https://doi.org/10.1159/000342083
Mirza S., Sharma G., Parshad R. et al. Clinical significance of promoter hypermethylation of ERβ and RARβ2 in tumor and serum DNA in Indian breast cancer patients // Ann. Surg. Oncol. 2012. V. 19. № 9. P. 3107–3115. https://doi.org/10.1245/s10434-012-2323-5
Fu D., Ren C., Tan H. et al. Sox17 promoter methylation in plasma DNA is associated with poor survival and can be used as a prognostic factor in breast cancer // Medicine (Baltimore). 2015. V. 94. № 11. e637. https://doi.org/10.1097/MD.0000000000000637
Matsui S., Kagara N., Mishima C. et al. Methylation of the SEPT9_v2 promoter as a novel marker for the detection of circulating tumor DNA in breast cancer patients // Oncol. Rep. 2016. V. 36. № 4. P. 2225–2235. https://doi.org/10.3892/or.2016.5004
Visvanathan K., Fackler M.S., Zhang Z. et al. Monitoring of serum DNA methylation as an early independent marker of response and survival in metastatic breast cancer: TBCRC 005 prospective biomarker study // J. Clin. Oncol. 2017. V. 35. № 7. P. 751–758. https://doi.org/10.1200/JCO.2015.66.2080
Panagopoulou M., Karaglani M., Balgkouranidou I. et al. Circulating cell-free DNA in breast cancer: size profiling, levels, and methylation patterns lead to prognostic and predictive classifiers // Oncogene. 2019. V. 38. № 18. P. 3387–3401. https://doi.org/10.1038/s41388-018-0660-y
van der Auwera I., Elst H.J., van Laere S.J. et al. The presence of circulating total DNA and methylated genes is associated with circulating tumour cells in blood from breast cancer patients // Br. J. Cancer. 2009. V. 100. № 8. P. 1277–1286. https://doi.org/10.1038/sj.bjc.6605013
Matuschek C., Bölke E., Lammering G. et al. Methylated APC and GSTP1 genes in serum DNA correlate with the presence of circulating blood tumor cells and are associated with a more aggressive and advanced breast cancer disease // Eur. J. Med. Res. 2010. V. 15. № 7. P. 277–286. https://doi.org/10.1186/2047-783x-15-7-277
Chimonidou M., Strati A., Malamos N. et al. SOX17 promoter methylation in circulating tumor cells and matched cell-free DNA isolated from plasma of patients with breast cancer // Clin. Chem. 2013. V. 59. № 1. P. 270–279. https://doi.org/10.1373/clinchem.2012.191551
Mirza S., Sharma G., Parshad R. et al. Clinical significance of Stratifin, ERalpha and PR promoter methylation in tumor and serum DNA in Indian breast cancer patients // Clin. Biochem. 2010. V. 43. № 4–5. P. 380–386. https://doi.org/10.1016/j.clinbiochem.2009.11.016
Martínez-Galán J., Torres-Torres B., Núñez M.I. et al. ESR1 gene promoter region methylation in free circulating DNA and its correlation with estrogen receptor protein expression in tumor tissue in breast cancer patients // BMC Cancer. 2014. V. 14. P. 59. https://doi.org/10.1186/1471-2407-14-59
Hagrass H.A., Pasha H.F., Ali A.M. Estrogen receptor alpha (ERα) promoter methylation status in tumor and serum DNA in Egyptian breast cancer patients // Gene. 2014. V. 552. № 1. P. 81–86. https://doi.org/10.1016/j.gene.2014.09.016
Liu X.-R., Zhang R.-Y., Gong H. et al. Methylome variation predicts exemestane resistance in advanced ER+ breast cancer // Technol. Cancer Res. Treat. 2020. V. 19. e1533033819896331. https://doi.org/10.1177/1533033819896331
Garufi G., Palazzo A., Paris I. et al. Neoadjuvant therapy for triple-negative breast cancer: Potential predictive biomarkers of activity and efficacy of platinum chemotherapy, PARP- and immune-checkpoint-inhibitors // Expert Opin. Pharmacother. 2020. V. 21. № 6. P. 687–699. https://doi.org/10.1080/14656566.2020.1724957
Ades F., Zardavas D., Bozovic-Spasojevic I. et al. Luminal B breast cancer: Molecular characterization, clinical management, and future perspectives // J. Clin. Oncol. 2014. V. 32. № 25. P. 2794–2803. https://doi.org/10.1200/JCO.2013.54.1870
Early Breast Cancer Trialists’ Collaborative Group (EBCTCG). Long-term outcomes for neoadjuvant versus adjuvant chemotherapy in early breast cancer: meta-analysis of individual patient data from ten randomised trials // Lancet Oncol. 2018. V. 19. № 1. P. 27–39. https://doi.org/10.1016/S1470-2045(17)30777-5
Caparica R., Lambertini M., Pondé N. et al. Post-neoadjuvant treatment and the management of residual disease in breast cancer: state of the art and perspectives // Ther. Adv. Med. Oncol. 2019. V. 11. e1758835919827714. https://doi.org/10.1177/1758835919827714
Avraham A., Uhlmann R., Shperber A. et al. Serum DNA methylation for monitoring response to neoadjuvant chemotherapy in breast cancer patients // Int. J. Cancer. 2012. V. 131. № 7. e1166–e1172. https://doi.org/10.1002/ijc.27526
Sharma G., Mirza S., Parshad R. et al. DNA methylation of circulating DNA: A marker for monitoring efficacy of neoadjuvant chemotherapy in breast cancer patients // Tumour Biol. 2012. V. 33. № 6. P. 1837–1843. https://doi.org/10.1007/s13277-012-0443-y
Takahashi H., Kagara N., Tanei T. et al. Correlation of methylated circulating tumor DNA with response to neoadjuvant chemotherapy in breast cancer patients // Clin. Breast Cancer. 2017. V. 17. № 1. P. 61–69.e3. https://doi.org/10.1016/j.clbc.2016.06.006
Sigin V.O., Kalinkin A.I., Kuznetsova E.B. et al. DNA methylation markers panel can improve prediction of response to neoadjuvant chemotherapy in luminal B breast cancer // Sci. Rep. 2020. V. 10. № 1. P. 9239. https://doi.org/10.1038/s41598-020-66197-1
Кекеева Т.В., Жинжило Т.А., Ненахова Ю.Н. и др. Метилирование гена ADCY8 в плазме крови как предиктивный маркер ответа на неоадъювантную химиотерапию рака молочной железы // Мед. генетика. 2018. Т. 17. № 10. С. 38–41.
Moss J., Zick A., Grinshpun A. et al. Circulating breast-derived DNA allows universal detection and monitoring of localized breast cancer // Ann. Oncol. 2020. V. 31. № 3. P. 395–403. https://doi.org/10.1016/j.annonc.2019.11.014
Fiegl H., Millinger S., Mueller-Holzner E. et al. Circulating tumor-specific DNA: A marker for monitoring efficacy of adjuvant therapy in cancer patients // Cancer Res. 2005. V. 65. № 4. P. 1141–1145. https://doi.org/10.1158/0008-5472.CAN-04-2438
Gil E.Y., Jo U.H., Jeong H. et al. Promoter methylation of RASSF1A modulates the effect of the microtubule-targeting agent docetaxel in breast cancer // Int. J. Oncol. 2012. V. 41. P. 611–620. https://doi.org/10.3892/ijo.2012.1470
Kajabova V., Smolkova B., Zmetakova I. et al. RASSF1A promoter methylation levels positively correlate with estrogen receptor expression in breast cancer patients // Transl. Oncol. 2013. V. 6. P. 297–304. https://doi.org/10.1593/tlo.13244
Liggett T.E., Melnikov A.A., Marks J.R. et al. Methylation patterns in cell-free plasma DNA reflect removal of the primary tumor and drug treatment of breast cancer patients // Int. J. Cancer. 2011. V. 128. № 2. P. 492–499. https://doi.org/10.1002/ijc.25363
Mastoraki S., Strati A., Tzanikou E. et al. ESR1 methylation: A liquid biopsy-based epigenetic assay for the follow-up of patients with metastatic breast cancer receiving endocrine treatment // Clin. Cancer Res. 2018. V. 24. № 6. P. 1500–1510. https://doi.org/10.1158/1078-0432.CCR-17-1181
Zurita M., Lara P.C., Del Moral R. et al. Hypermethylated 14-3-3-sigma and ESR1 gene promoters in serum as candidate biomarkers for the diagnosis and treatment efficacy of breast cancer metastasis // BMC Cancer. 2010. V. 10. P. 217. https://doi.org/10.1186/1471-2407-10-217
Дополнительные материалы отсутствуют.