Генетика, 2021, T. 57, № 2, стр. 196-204
Анализ полиморфизма генома D синтетической пшеницы, полученной на основе Аe. tauschii L.
И. В. Потоцкая 1, *, В. П. Шаманин 1, **, С. С. Шепелев 1, М. Бхатта 2, А. И. Моргунов 1, 3
1 Омский государственный аграрный университет им. П.А. Столыпина
644008 Омск, Россия
2 Университет Висконсина
53705 Мэдисон, США
3 Университет штата Вашингтон
99164 Пулман, США
* E-mail: iv.pototskaya@omgau.org
** E-mail: vp.shamanin@omgau.org
Поступила в редакцию 26.03.2020
После доработки 18.05.2020
Принята к публикации 17.06.2020
Аннотация
Методом SNP-анализа с помощью панели iSelect90К изучено генетическое разнообразие линий синтетической гексаплоидной пшеницы, полученных в университете Киото (Япония) в результате гибридизации сорта твердой пшеницы Langdon (Triticum durum, 2n = 4x = 28, BBAA) с образцами Aegilops tauschii (2n = 2x = 14, DD) различного эколого-географического происхождения. Уровень геномной вариабельности оценивали по значениям генетических расстояний Emma (Efficient Mixed-Model Association) и геномной матрицы родства (G). Генетическое разнообразие генома D синтетических линий на 50% выше, чем у геномов A и В. Наибольший генетический полиморфизм отмечен у линии Langdon/Ku-2105 из провинции Пехлеви на южном побережье Каспийского моря и линии Langdon/Ig 131606 из Кыргызстана. Меньшим генетическим разнообразием по D-геному отличались линии из Индии (Langdon/Ig 48042), провинций Ирана Сари и Бальбусар (Langdon/Ku-2088, Langdon/Ku-2093, Langdon/Ku-2096) и Туркменистана (Langdon/Ig 26387). Таким образом, вовлечение в гибридизацию синтетических гексаплоидов пшеницы на основе образцов Ae. tauschii из района Каспийского бассейна позволит расширить генетическое разнообразие сортов пшеницы и повысить эффективность использования генетических ресурсов.
Использование потенциала синтетических гексаплоидов на основе эгилопса Тауша (Aegilops tausсhii Coss., геном D) доказало свою эффективность в селекции для расширения генетического разнообразия, повышения адаптивности и урожайности сортов пшеницы [1]. Более 62 сортов пшеницы, полученных на основе синтетических форм, успешно внедрены в производство 150 стран мира и их число стремительно растет [2, 3]. Основное генетическое разнообразие вида Ae. tausсhii сосредоточено на территории, простирающейся от Закавказья до восточного побережья Каспийского моря в Иране, от Центрального Ирана до Китая. В общей сложности более 600 образцов Ae. tausсhii из 20 стран мира были вовлечены в селекционные программы путем интрогрессивной гибридизации или скрещивания с синтетической пшеницей [4]. Наиболее широкое применение генетического материала синтетических гексаплоидов CIMMYT нашло в Китае, где в 2003–2005 гг. создали четыре сорта, один из которых Chuanmai 42 [5]. На основе этого высокоурожайного сорта, устойчивого к желтой ржавчине, получено 12 коммерческих сортов пшеницы [6].
Повышение эффективности и точности отбора селекционного материала с ценными интрогрессиями от синтетических гексаплоидов является важной задачей для маркер-ориентированной селекции. Первым шагом на пути решения такой задачи стало создание картирующей популяции ITMI (International Triticeae Mapping Initiative), полученной путем гибридизации сорта Opata 85 и синтетического гексаплоида W7984. С помощью картирующей популяции ITMI были выявлены QTL, в том числе D-генома, определяющие хозяйственно ценные признаки [7, 8].
SNP-локусы (однонуклеотидный полиморфизм) благодаря равномерному распределению по всему геному (одна однонуклеотидная замена на 99 пар нуклеотидов) послужили основой для разработки платформ полногеномного генотипирования [9]. Так, в китайской академии сельскохозяйственных наук (CAAS) были использованы ДНК-чипы, содержащие 600 К SNP, для идентификации QTL, контролирующих число зерен [10]. Сравнительно недавно из ДНК-чипов 35 К, 90 К и 660 К отобрано 700 и 1000 SNPs c высокой степенью детектирования для конструирования чипов 15 К и 50 К (Triticum TraitBreed array) [11]. Анализ полиморфизма ДНК с помощью SNP-маркеров успешно используется в исследованиях по изучению генетического разнообразия, по ДНК-паспортизации коммерческих сортов пшеницы, картированию QTL и методу GWAS [12].
Количественно генетическое сходство сортов можно изучить с помощью коэффициентов родства. Включение в гибридизацию родительских форм с низкими коэффициентами родства вызывает богатый формообразовательный процесс и увеличивает долю генетической дисперсии количественных признаков в гибридном потомстве [13]. В селекционных программах, наряду с анализом генеалогических профилей, прогноз геномного родства возможен за счет использования неравновесного сцепления локусов количественных признаков (QTL) и SNP-маркеров, плотно охватывающих весь геном. Данные SNP-генотипирования эффективны для выявления генетической ценности родительских форм, что позволяет повысить вероятность отбора ценных генотипов в гибридных популяциях [14].
Очевидно, что привлечение в селекцию синтетической пшеницы, имеющей большее генетическое разнообразие в сравнении с коммерческими сортами пшеницы, позволяет привнести в селекционный материал новые аллели генов [15, 16]. Однако, несмотря на проведенные исследования по генотипированию синтетической пшеницы и отбору генотипов по локусам количественных признаков [12], поиск источников для расширения генотипического потенциала сортов пшеницы и создание на их основе исходного материала, устойчивого к негативным абиотическим и биотическим стрессовым факторам в связи с проблемой потепления климата и возрастанием инфекционной нагрузки, весьма актуальны в селекции пшеницы.
Цель исследований – SNP-анализ линий синтетической пшеницы с геномом Ae. tauschii различного эколого-географического происхождения и выделение источников для расширения генотипического потенциала сортов пшеницы.
МАТЕРИАЛЫ И МЕТОДЫ
Омским ГАУ при сотрудничестве с CIMMYT в университете Nebraska-Lincoln (США) проведено генотипирование 143 образцов из коллекционного питомника ОмонГАИ, включающего 13 линий синтетической гексаплоидной пшеницы японского университета Киото [17]. Синтетические линии университета Киото получены в результате гибридизации сорта твердой пшеницы Langdon (Triticum durum, 2n = 4x = 28, BBAA) с образцами Aegilops tauschii (2n = 2x = 14, DD) различного эколого-географического происхождения (табл. 1) из коллекции университета Киото и Международного центра сельскохозяйственных исследований в засушливых регионах ICARDA (International Center for Agricultural Research in the Dry Areas) [18].
Таблица 1.
Синтетические линии университета Киото
| Синтетическая линия | Происхождение Ae. tauschii |
Подвид Ae. tauschii |
|---|---|---|
| Langdon/Ku-2105 | Пехлеви, Иран | typica |
| Langdon/Ku-2092 | Бабульсар, Иран | strangulata |
| Langdon/Ig 131606 | Талас, Кыргызстан | Не известен |
| Langdon/Ig 48042 | Джамму-Кашмир, Индия | Не известен |
| Langdon/Ku-20-9 | Бабульсар, Иран | strangulata |
| Langdon/Ku-2079 | Альябад, Иран | strangulata |
| Langdon/Ku-2100 | Рамсар, Иран | meyeri |
| Langdon/Ku-2096 | Бабульсар, Иран | strangulata |
| Langdon/Ku-2088 | Сари, Иран | strangulata |
| Langdon/Ku-2075 | Бабульсар, Иран | strangulata |
| Langdon/Ku-2093 | Бабульсар-Чалус, Иран | strangulata |
| Langdon/Ku-2097 | Бабульсар, Иран | typica |
| Langdon/Ig 126387 | Ашхабад, Туркменистан | Не известен |
SNP-анализ. В Центре генетических ресурсов университета Nebraska-Lincoln (США) проведен SNP-анализ 143 образцов питомника ОмонГАИ с помощью панели iSelect90К, содержащей 81 587 SNP, с охватом хромосом трех геномов. Выделение геномной ДНК проводили набором BioSprint 96 (QIAGEN, Valencia, Калифорния, США) в соответствии с инструкциями производителя. Метод GBS выполнен на основе протокола [19]. Создание библиотек генотипирования последовательностей ДНК проводилось c применением рестрикционных энзимов PstI и MspI. Идентификация SNP-локусов осуществлена с использованием программы TASSEL v. 5.2.40 [20], GBS v. 2 Pipeline [21] и физической карты референсного генома пшеницы от International Wheat Genome Sequencing Consortium (IWGSC, RefSeq V1.0, 2017 г.)
Статистический анализ генетического разнообразия. Уровень геномной вариабельности оценивали по значениям генетических расстояний Emma (Efficient Mixed-Model Association) [22] и геномной матрицы родства (G) [23] в оболочке R‑Studio с использованием пакета прикладных программ GAPIT 3.0 (Genomic Association and Prediction Integrated Tool) [24].
Вычисление генетических расстояний по методу Emma проводили по формуле:
где у – вектор популяционных эффектов; β – вектор фиксированных эффектов, u – случайный эффект смешанной модели; е – вектор случайных эффектов; X – матрица фиксированных эффектов; Z – матрица инцидентности случайных эффектов.Расчет геномной матрицы родства проводили по алгоритму, разработанному P.M. VanRaden [23], (метод 1) по формуле:
где: Z = M – P (матрица SNP эффектов); M – матрица размером n × m (число особей на число локусов), состоящая из элементов – оценок гомозиготных и гетерозиготных локусов, принятых за: AA = 1, AB = 0, BB = –1; P – вектор-столбец, представляющий удвоенную сумму гетерозиготных маркеров (локусов) или частоту второго SNP аллеля, где i = 2(pj – 0.5); pi – частота i-аллеля SNP (A или B) для j-го сорта.Построение дендрограммы на основании данных SNP-анализа выполнено с помощью программы SPSS версии PASW Statistics 20.0 (IBM, США).
РЕЗУЛЬТАТЫ
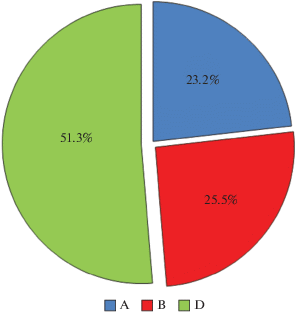
В результате SNP-анализа у синтетических линий университета Киото идентифицировано 47 525 SNP-локусов. Из данного набора линий выявлены различия по 15 553 SNP. Из 15 553 локусов – 411 с отсутствующими данными (более 2%) были исключены из анализа. Распределение 15 142 SNPs между геномами было следующим: A-, B- и D-геномы имели 3517 SNPs (23.2%), 3858 (25.5%) и 7767 (51.3%) соответственно (рис. 1, 2).
Рис. 2.
Распределение 15 142 SNPs по 21 хромосоме и SNPs с отсутствующими данными (UN) 13 синтетических линий.

Число SNPs на хромосому варьировало от 385 (4В) до 1484 (7D). Распределение SNPs между A и B геномами было практически равным, тогда как полиморфизм генома D был на 50% выше, чем у геномов A и В. Определены показатели генетических расстояний с использованием метода Emma и геномной матрицы родства (табл. 2, рис. 3).
Таблица 2.
Уровень генетического разнообразия синтетических линий пшеницы с участием образцов Ae. tauschii различного происхождения
| Синтетическая линия |
Langdon/Ku-2105 | Langdon/Ku-2092 | Langdon/Ig 131606 | Langdon/Ig 48042 | Langdon/Ku-20-9 | Langdon/Ku-2079 | Langdon/Ku-2100 | Langdon/Ku-2096 | Langdon/Ku-2088 | Langdon/Ku-2075 | Langdon/Ku-2093 | Langdon/Ku-2097 | Langdon/Ig 126387 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Ku-2105 | 1.71 | 81 | 79 | 80 | 81 | 79 | 80 | 81 | 81 | 80 | 82 | 80 | 79 |
| Ku-2092 | –0.15 | 1.42 | 79 | 80 | 83 | 80 | 81 | 81 | 84 | 82 | 84 | 81 | 79 |
| Ig 131606 | –0.12 | –0.26 | 1.65 | 84 | 79 | 77 | 78 | 78 | 79 | 78 | 80 | 78 | 83 |
| Ig 48042 | –0.15 | –0.27 | 0.69 | 1.47 | 81 | 79 | 80 | 80 | 81 | 80 | 81 | 80 | 86 |
| Ku-20-9 | –0.09 | 0.01 | –0.24 | –0.25 | 1.39 | 81 | 81 | 82 | 84 | 82 | 84 | 81 | 80 |
| Ku-2079 | –0.11 | –0.04 | –0.23 | –0.21 | –0.01 | 1.63 | 79 | 79 | 81 | 80 | 81 | 79 | 78 |
| Ku-2100 | –0.18 | –0.23 | –0.42 | –0.44 | –0.21 | –0.28 | 1.49 | 86 | 81 | 80 | 82 | 86 | 78 |
| Ku-2096 | –0.16 | –0.23 | –0.42 | –0.44 | –0.19 | –0.30 | 0.71 | 1.42 | 82 | 81 | 83 | 86 | 79 |
| Ku-2088 | –0.12 | 0.16 | –0.26 | –0.26 | 0.06 | 0.06 | –0.29 | –0.26 | 1.36 | 82 | 85 | 81 | 80 |
| Ku-2075 | –0.16 | 0.02 | –0.27 | –0.26 | 0.03 | 0.03 | –0.24 | –0.24 | 0.02 | 1.57 | 83 | 80 | 79 |
| Ku-2093 | –0.12 | 0.09 | –0.31 | –0.31 | 0.03 | –0.01 | –0.17 | –0.16 | 0.13 | 0 | 1.35 | 82 | 80 |
| Ku-2097 | –0.18 | –0.24 | –0.43 | –0.44 | –0.23 | –0.31 | 0.69 | 0.68 | –0.32 | –0.26 | –0.19 | 1.62 | 78 |
| Ig 126387 | –0.16 | –0.28 | 0.64 | 0.89 | –0.44 | –0.20 | –0.44 | –0.43 | –0.27 | –0.25 | –0.34 | –0.44 | 1.56 |
Примечание. Количество одинаковых SNPs (%) по Emma – над диагональю, коэффициент геномного родства между образцами – под диагональю, доля гомозиготных локусов каждого образца – по диагонали: варьирует от 0 (все локусы находятся в гетерозиготном состоянии) до 2 (все локусы находятся в гомозиготном состоянии).
Рис. 3.
Тепловая карта корреляционного анализа изученных 13 синтетических линий на основании данных SNP-анализа.
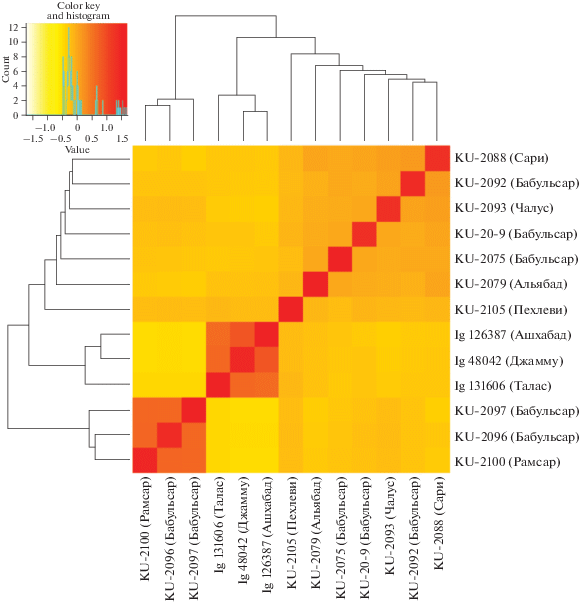
Синтетические линии, полученные с участием образцов Ae. tauschii из провинций Ирана Рамсар и Бальбусар, а также из коллекции ICARDA имеют наибольшее генетическое сходство (показатель генетического расстояния по Emma составил 0.86, или 86% общих SNP) и объединены в два кластера (рис. 3).
К первому относятся линии, полученные на основе образцов Ae. tauschii из Ирана: Langdon/Ku-2100, Langdon/Ku-2096 и Langdon/Ku-2097, ко второму – Langdon/Ig 131606 (Кыргызстан), Langdon/Ig 48042 (Индия) и Langdon/Ig 26387 (Туркменистан) (генетические дистанции по методу Emma равны 0.83, 0.84, 0.86). Высокие положительные значения коэффициента геномного родства (0.64–0.89) также свидетельствуют о близком генетическом сходстве данных линий. Остальные линии образуют отдельные субкластеры с высокой долей одинаковых SNP-локусов. Максимальные генетические различия (коэффициент геномного родства составил –0.44) отмечены между линиями Langdon/Ig 48042 (Индия) и Langdon/Ku-2100, Langdon/Ku-2096, Langdon/Ku-2097 (Иран); Langdon/Ig 126387 (Туркменистан) и Langdon/Ku-20-9, Langdon/Ku-2100, Langdon/Ku-2097 (Иран), что объясняется различным географическим происхождением образцов Ae. tauschii. Линии из разных провинций Ирана имели близкое генетическое родство, например Langdon/Ku-2088 (провинция Сари) с линиями из провинции Бальбусар – Langdon/Ku-2092 (0.16) и Langdon/Ku-2093 (0.13). Исключение составили две линии из провинции Бальбусар – Langdon/Ku-2096 и Langdon/Ku-2097, оказавшиеся в первом кластере, при этом образец Ae. tauschii Ku-2097 относится к подвиду typica. Оценка геномного родства не только подходит для анализа родственных выборок, но и учитывает степень гомозиготности образцов. Например, линии Langdon/Ku-2105 (1.71) и Langdon/Ig 131606 (1.65) имеют наибольшую долю локусов в гомозиготном состоянии, что представляет особенный интерес для селекционной работы.
Поскольку синтетические линии университета Киото получены на основе одного тетраплоидного компонента (Langdon), генетические дистанции по методу Emma у изученных линий составили 77–86%, тем не менее основной вклад в генетическую изменчивость синтетических линий вносит D-геном (51.3%). Наличие D-генома образцов Ae. tauschii разного эколого-географического происхождения позволил провести сравнительный анализ между линиями по D-геному (табл. 3).
Таблица 3.
Сравнительный анализ “уникальных” SNPs по D-геному между синтетическими линиями по методу Emma
| Синтетическая линия |
Количество SNP | Итого: | ||||||
|---|---|---|---|---|---|---|---|---|
| 1D | 2D | 3D | 4D | 5D | 6D | 7D | ||
| Langdon/Ku-2105 | 5 | 19* | 17* | 3 | 4 | 6* | 13* | 67 |
| Langdon/Ku-2092 | 2 | 9* | 12* | 0 | 0 | 3 | 9* | 35 |
| Langdon/Ig 131606 | 9* | 14* | 7* | 0 | 7* | 10* | 17* | 64 |
| Langdon/Ig 48042 | 1 | 0 | 0 | 0 | 0 | 3 | 4 | 8 |
| Langdon/Ku-20-9 | 0 | 7* | 10* | 2 | 2 | 5 | 7* | 33 |
| Langdon/Ku-2079 | 8* | 2 | 2 | 3 | 2 | 6* | 10* | 33 |
| Langdon/Ku-2100 | 5 | 8* | 2 | 1 | 4 | 4 | 1 | 25 |
| Langdon/Ku-2096 | 4 | 7 | 0 | 0 | 2 | 1 | 0 | 14 |
| Langdon/Ku-2088 | 2 | 0 | 1 | 0 | 0 | 2 | 2 | 7 |
| Langdon/Ku-2075 | 9* | 5 | 0 | 1 | 4 | 9* | 1 | 29 |
| Langdon/Ku-2093 | 4 | 2 | 1 | 1 | 4 | 2 | 4 | 18 |
| Langdon/Ku-2097 | 5 | 5 | 0 | 2 | 1 | 2 | 10* | 25 |
| Langdon/Ig 126387 | 6* | 4 | 2 | 0 | 1 | 0 | 3 | 16 |
| Итого: | 60 | 82 | 54 | 13 | 31 | 53 | 81 | |
Наибольший генетический полиморфизм также отмечен у линии Langdon/Ku-2105 (67 уникальных SNPs по геному D) из провинции Пехлеви на южном побережье Каспийского моря, а также у линии Langdon/Ig 131606 (64 SNPs) из Кыргызстана. Меньшим генетическим разнообразием по D-геному отличаются линии из Индии (Langdon/Ig 48042), провинций Ирана Сари, Бальбусар (Langdon/Ku-2088, Langdon/Ku-2093, Langdon/Ku-2096) и Туркменистана (Langdon/Ig 26387). При этом наибольшие различия отмечены между линиями по хромосомам 2D (82 SNPs) и 7D (81 SNPs), тогда как наименьшие – по хромосоме 4D (13 SNPs).
ОБСУЖДЕНИЕ
Формирование генома T. aestivum произошло вследствие многократных скрещиваний T. turgidum ssp. durum, вероятно, с ограниченным числом образцов Ae. tauschii ssp. strangulatа в местах перекрытия ареалов этих подвидов [25–27]. Современные сорта яровой пшеницы характеризуются низким генетическим разнообразием генома D из-за потери ценных генов и аллелей в результате целенаправленной селекции на повышение урожайности и многовекового возделывания этой культуры [28]. ДНК-полиморфизм геномов А и В сортов мягкой пшеницы также редуцирован в сравнении с геномами А и В твердой пшеницы. В этой связи передача генов посредством синтетической пшеницы от вида T. durum в геном T. aestivum способствует расширению генетического разнообразия исходного материала в селекции пшеницы [4, 12].
В наших предыдущих работах проведен cравнительный анализ генетического сходства линий синтетической пшеницы селекции CIMMYT и университета Киото с лучшими сортами из Северной Америки, России и Казахстана, который показал значимость синтетической пшеницы в качестве ценного генетического ресурса новых аллелей генов для селекции пшеницы. Результаты исследований выявили также генетические различия у синтетических линий не только по геному D, но также по геномам А и B [15, 29].
Для синтетических линий, полученных на основе образцов Ae. tauschii из южных районов Каспийского бассейна, характерно большее генетическое разнообразие, поскольку именно на этой территории произрастают редкие и эндемичные образцы, в том числе ssp. strangulatа – вероятный донор генома D мягкой пшеницы, продолжается активный формообразовательный процесс данного вида эгилопса [30, 31]. Полученные данные согласуются с результатами других исследований. Например, J. Wang с коллегами [32] с помощью SNP-анализа изучали генетическое разнообразие коллекции образцов Ae. tauschii и пришли к выводу, что наибольший полиморфизм D-генома отмечен у двух групп образцов, произрастающих на юге и юго-западе Каспийского бассейна.
На основе результатов SNP-aнализа построены дендрограммы для оценки степени сходства однонуклеотидного полиморфизма между изученными синтетическими линиями. На филогенетическом дереве синтетических линий выделено три кластера: синтетические линии с участием образцов Ae. tauschii из провинций Ирана Рамсар и Бальбусар, линии с участием образцов Ae. tauschii коллекции ICARDA (Туркменистан, Кыргызстан и Индия) и линии из различных провинций Ирана. Генетическая однородность линий из провинций Ирана Бабульсар, Альябад, Сари и Чалус свидетельствует о высоком филогенетическом сходстве образцов Ae. tauschii, произрастающих на территории данных провинций. В этом кластере наибольший уровень генетического разнообразия отмечен у линии Langdon/Ku-2105 из провинции Пехлеви.
Анализ полиморфизма синтетических линий с помощью генетических расстояний по методу Emma и геномной матрицы родства позволил оценить сходство синтетических линий, полученных на основе образцов Ae. tauschii разного эколого-географического происхождения. Генетическое разнообразие генома D на 50% выше, чем у геномов A и В, что свидетельствует о ценности изученных синтетических линий в качестве генетического резерва для расширения полиморфизма сортов пшеницы. Выявлены наибольшие различия между линиями по хромосомам 2D (82 SNPs) и 7D (81 SNPs).
Отмечена высокая степень генетической однородности линий по D-геному, полученных на основе образцов Ae. tauschii из провинций Ирана Сари и Бальбусар (Langdon/Ku-2088, Langdon/ Ku-2093, Langdon/Ku-2096), а также линий из Индии (Langdon/Ig 48042) и Туркменистана (Langdon/Ig 26387).
SNP-анализ у линий Langdon/Ku-20-9, Langdon/Ku-2100, Langdon/Ku-2079, Langdon/Ku-2075, Langdon/Ku-2092 и Langdon/Ku-2097 из разных провинций Ирана выявил 25–35 уникальных SNPs D-генома. Выделены две линии – Langdon/Ku-2105 из города Пехлеви на южном побережье Каспийского моря и Langdon/Ig 131606 из Кыргызстана, характеризующиеся наибольшим числом уникальных SNP-локусов D-генома – 67 и 64 и максимальными значениями генетического расстояния по VanRaden – 1.71 и 1.65 соответственно.
Проведенное исследование подтверждает особый статус образцов Ae. tauschii, произрастающих на юге Каспийского бассейна, для расширения полиморфизма генома D и поиска генов, контролирующих хозяйственно ценные признаки в селекции пшеницы. Японские синтетические линии, имеющие высокий уровень генетического разнообразия, представляют интерес для селекционных программ в качестве ценного генетического ресурса с целью расширения генотипического потенциала коммерческих сортов пшеницы и повышения эффективности использования генетических ресурсов.
Данное исследование проведено при финансовой поддержке РНФ (проект № 16-16-10005).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Rasheed A., Ogbonnaya F.C., Lagudah E. et al. The goat grass genome’s role in wheat improvement // Nat. Plants. 2018. V. 4. P. 56–58. https://doi.org/10.1038/s41477-018-0105-1
Li A., Liu D., Yang W. et al. Synthetic hexaploid wheat: yesterday, today, and tomorrow // Engineering. 2018. V. 4. P. 552–558. https://doi.org/10.1016/j.eng.2018.07.001
Singh S., Vikram P., Sehgal D. et al. Harnessing genetic potential of wheat germplasm banks through impact-oriented-prebreeding for future food and nutritional security // Sci. Rep. 2018. № 8: 12527. https://doi.org/10.1038/s41598-018-30667-4
Ogbonnaya F.C., Imtiaz M., Bariana H.S. Mining synthetic hexaploids for multiple disease resistance to improve wheat // Austr. J. Agricultural Res. 2008. V. 59(5). P. 421–431. https://doi.org/10.1071/AR07227
Li J., Wan H., Yang W.Y. Synthetic hexaploid wheat enhances variation and adaptive evolution of bread wheat in breeding processes // J. Syst. Evol. 2014. V. 52. P. 735–742. https://doi.org/10.1111/jse.12110
Hao M., Zhang L., Zhao L. et al. A breeding strategy targeting the secondary gene pool of bread wheat: introgression from a synthetic hexaploid wheat // Theor. Appl. Genet. 2019. V. 132. P. 2285–2294. https://doi.org/10.1007/s00122-019-03354-9
Баталова Г.А., Русакова И.И., Кочерина Н.В. и др. Оценка линий ITMI и картирование QTL у яровой мягкой пшеницы (Triticum aestivum L.) в условиях северо-востока Российской Федерации. Киров: Изд-во Зонального НИИСХ Северо-Востока им. Н.В. Рудницкого, 2016. 52 с.
Шаманин В.П., Шепелев С.С., Пожерукова В.Е. и др. Картирование QTL у гексаплоидной мягкой пшеницы (Triticum aestivum L.) в условиях Западно-Сибирской равнины // С-х. биология. 2018. Т. 53. № 1. С. 50–60. https://doi.org/10.15389/agrobiology.2018.1.50rus
Paux E., Sourdille P., Mackay I. et al. Sequence-based marker development in wheat: advances and applications to breeding // Biotechnol. Adv. 2012. V. 30. P. 1071–1088. https://doi.org/10.1016/j.biotechadv
Cui F., Zhang N., Fan X.L. et al. Utilization of a Wheat660 K SNP array-derived high-density genetic map for high-resolution mapping of a major QTL for kernel number // Sci. Rep-UK. 2017. V. 7. P. 3788. https://doi.org/10.1038/s41598-017-04028-6
Rasheed A., Xia X. From markers to genome-based breeding in wheat // Theor. Appl. Genet. 2019. V. 132(3). P. 767–784. https://doi.org/10.1007/s00122-019-03286-4
Rasheed A., Mujeeb-Kazi A., Ogbonnaya F.C. et al. Wheat genetic resources in the post-genomics era: promise and challenges // Ann. Bot. 2018. V. 121. P. 603–616. https://doi.org/10.1093/aob/mcx148
Мартынов С.П., Добротворская Т.В. Генеалогический и статистический анализ генетического разнообразия с помощью информационно-аналитической системы генетических ресурсов пшеницы GRIS // Тр. по прикладной ботанике, генетике и селекции. СПб: ООО “Р-КОПИ”, 2012. С. 193–210.
Crossa J., Perez P., Hickey J. et al. Genomic prediction in CIMMYT maize and wheat breeding programs // Heredity. 2014. V. 112. P. 48–60. https://doi.org/10.1038/hdy.2013.16
Bhatta M., Shamanin V., Shepelev S. et al. Marker-trait associations for enhancing agronomic performance, disease resistance, and grain quality in synthetic and bread wheat accessions in Western Siberia // G3: Genes, Genomes, Genetics. 2019. V. 19(1). P. 4209–4222. https://doi.org/10.1534/g3.119.400811
Ogbonnaya F.C., Abdalla O., Mujeeb-Kazi A. et al. Synthetic hexaploids: Harnessing species of the primary gene pool for wheat improvement // Plant Breed. Rev. 2013. V. 37. P. 35–122. https://doi.org/10.1002/9781118497869.ch2
Шаманин В.П., Потоцкая И.В., Шепелев С.С. и др. Синтетическая пшеница: монография. Омск: Изд-во ФГБОУ ВО Омский ГАУ, 2018. 172 с.
Matsuoka Y., Takumi S., Kawahara T. Natural variation for fertile triploid F1 hybrid formation in allohexaploid wheat speciation // Theor. Appl. Genet. 2007. V. 115(4). P. 509–518. https://doi.org/10.1007/s00122-007-0584-3
Poland J.A., Brown P.J., Sorrels M.E. et al. Development of high density genetic maps for barley and wheat using a novel two enzyme genotyping-by-sequencing approach // PLoS One. 2012. V. 7:e32253. https://doi.org/10.1371/journal.pone.0032253
Bradbury P.J., Zhang Z., Kroon D.E. et al. TASSEL: software for association mapping of complex traits in diverse samples // Bioinformatics. 2007. V. 23. P. 2633–2635. https://doi.org/10.1093/bioinformatics/btm308
Glaubitz J.C., Casstevens T.M., Lu F. et al. TASSEL-GBS: a high capacity genotyping by sequencing analysis pipeline // PLoS One. 2014. V. 9: e90346. https://doi.org/10.1371/journal.pone.0090346
Kang H.M., Zaitlen N.A., Wade C.M. et al. Efficient control of population structure in model organism association mapping // Genetics. 2008. V. 178. P. 1709–1723. https://doi.org/10.1534/genetics.107.080101
VanRaden P.M. Efficient methods to compute genomic predictions // J. Dairy Sci. 2008. V. 91. P. 4414–4423. https://doi.org/10.3168/jds.2007-0980
Lipka A.E., Kandianis C.B., Hudson M.E. et al. From association to prediction: Statistical methods for the dissection and selection of complex traits in plants // Curr. Opin. Plant Biol. 2015. V. 24. P. 110–118. https://doi.org/10.1016/j.pbi.2015.02.010
Кулуев А.Р., Матниязов Р.Т., Чемерис Д.А. и др. Современные представления о родственных взаимоотношениях в пшенично-эгилопсном альянсе (с краткой исторической справкой) // Биомика. 2016. Т. 8. № 4. С. 297–310.
Xu L., Tang Y., Gao S. et al. Comprehensive analyses of the annexin gene family in wheat // BMC Genomics. 2016. № 17: 415. https://doi.org/10.1186/s12864-016-2750-y
Avni R., Nave M., Barad O. et al. Wild emmer genome architecture and diversity elucidate wheat evolution and domestication // Science. V. 357(6346). P. 93–97. https://doi.org/10.1126/science.aan0032
Cox T.S., Wu J., Wang S. et al. Comparing two approaches for introgression of germaplasm from Aegilops tauschii into common wheat // Crop J. 2017. V. 5. P. 355–362.https://doi.org/10.1016/j.cj.2017.05.006
Bhatta M., Shamanin V., Shepelev S. et al. Genetic diversity and population structure analysis of synthetic and bread wheat accessions in Western Siberia // J. Appl. Genet. 2019. V. 60(3–4). P. 283–289. https://doi.org/10.1007/s13353-019-00514-x
Cox T.S. Deepening the wheat gene pool // J. Crop Prod. 1997. V. 1. P. 145–168. https://doi.org/10.1300/J144v01n01_01
Saedi H., Tabatabaei B.E.S., Rahimmalek M. et al. Genetic diversity and gene-pool subdivisions of diploid D‑genome Aegilops tauschii Coss. (Poaceae) in Iran as revealed by AFLP // Genet. Resour. Crop Evol. 2008. V. 55(8). P. 1231–1238. https://doi.org/10.1007/s10722-008-9323-0
Wang J.R., Luo M.C., Chen Z.X. et al. Aegilops tauschii single nucleotide po-lymorphisms shed light on the origins of wheat D-genome genetic diversity and pinpoint the geographic origin of hexaploid wheat // New Phytologist. 2013. V. 198. P. 925–937.
Дополнительные материалы отсутствуют.