Генетика, 2021, T. 57, № 3, стр. 280-295
Вариабельность и экспрессия паралогов фитоинсинтазы (PSY) у видов перца клады Annuum
М. А. Филюшин 1, *, Е. А. Дьяченко 1, Г. И. Ефремов 1, Е. З. Кочиева 1, А. В. Щенникова 1
1 Институт биоинженерии, Федеральный исследовательский центр
“Фундаментальные основы биотехнологии” Российской академии наук
119071 Москва, Россия
* E-mail: michel7753@mail.ru
Поступила в редакцию 08.04.2020
После доработки 04.06.2020
Принята к публикации 08.07.2020
Аннотация
В настоящем исследовании у пяти образцов трех видов перца (Capsicum annuum, C. chinense и C. frutescens), различающихся паттерном пигментации плода, идентифицированы гомологи генов PSY1 и PSY2, кодирующих ключевой фермент каротиногенеза – фитоинсинтазу. Внутри исследуемой группы перцев определена вариабельность геномных последовательностей PSY1 и PSY2 и кДНК. Последовательности белков PSY1 и PSY2 были сходны (82%) и различались N- и C-концевыми инделями NAGLRYSD и KLTSSSL, а также консервативными мотивами, характерными для групп гомологов PSY2 и PSY1. Экспрессия генов PSY1 и PSY2 проанализирована в листьях, чашелистиках, лепестках, завязи, а также в кожице и мякоти плодов в процессе созревания у анализируемых образцов перца. Показан максимальный уровень экспрессии PSY1 в лепестках, а также в перикарпе зрелых плодов образцов C. annuum и C. frutescens. У образца C. chinense Pimenta da Neyde ген PSY1 экспрессировался только в листьях. Транскрипты PSY2 обнаружены во всех анализируемых органах образцов перца, максимальный уровень – в листьях, минимальный – в перикарпе плодов. Полученные данные предполагают сохранение у видов перца гомологами PSY1 и PSY2 консервативных ключевых функций в синтезе каротиноидов в плодах (PSY1) и фотосинтезирующих тканях (PSY2).
Растения, за редким исключением, являются фотосинтезирующими организмами и для поглощения и преобразования световой энергии используют пигменты – хлорофиллы и каротиноиды, при этом каротиноиды задействованы также для защиты молекул хлорофилла от фотоокисления [1]. Более того, каротиноиды являются предшественниками фитогормонов (абсцизовой кислоты и стриголактонов), а их производные могут действовать как сигнальные молекулы в процессах развития растений и в ответ на воздействия окружающей среды [2–4]. Также растения используют каротиноиды как оптические сигналы при взаимодействии с насекомыми и другими животными, пигментируя цветковые органы и/или плоды [4].
Механизм биосинтеза каротиноидов консервативен, однако по мере эволюции растений к общей специализации данных пигментов добавлялись новые функции. Так, увеличение фотосинтезирующей поверхности при образовании плоского листа из радиально-симметричного стебля повысило необходимость фотозащиты [1]. Появление репродуктивных органов добавило необходимость окраски лепестков и тычинок для привлечения опылителей [5]. Преобразование сухого плода в сочный и окрашенный сделало его привлекательным для животных, что дало растениям большие возможности в распространении семян [6]. Такая многофункциональность продуктов каротиногенеза является следствием эволюционной дупликациии последующей диверсификации генов биосинтеза каротиноидов. Прежде всего это касается гена-предшественника фитоинсинтаз PSY (phytoene synthase), чьи продукты играют ключевую роль в инициации синтеза каротиноидов в различных органах растения [7, 8].
Фитоинсинтаза PSY локализуется в строме пластид и катализирует образование 15-цис-изомера фитоина, предшественника всех каротиноидов [7, 9]. Несколько последующих стадий приводят к синтезу ликопина, который в дальнейшем используется для синтеза α-каротина и β-каротина (оранжевый пигмент). В свою очередь, α-каротин и β-каротин могут превращаться в желтые пигменты лютеин и ксантофиллы [9, 10].
У растений описано от одного до трех паралогов PSY. Геном злаковых (Poaceae) и некоторых других видов однодольных содержит три гена PSY [11]. Двудольные виды в этом плане более разнообразны. К примеру, в геноме Arabidopsis thaliana (L.) Heynh. присутствует только один ген PSY [12]. Для других двудольных описано, как минимум, два гена фитоинсинтазы. В то же время геном люцерны и томата содержит три гена PSY [3, 13].
Первый идентифицированный ген фитоинсинтазы растений, PSY1, специфичен в основном для хромопластов и задействован в пигментации сочного плода при его созревании [14]. При отсутствии транскрипции PSY1 не происходит синтеза каротиноидов в плодах, а суперэкспрессия PSY1 значительно увеличивает каротиногенез в запасающих органах (клубнях картофеля, плодах томата и т.д.) [15, 16]. Второй ген PSY2 специфичен, прежде всего, для хлоропластов и его экспрессия максимальна в фотосинтезирующей ткани [17]. Третий паралог, PSY3, идентифицирован не у всех видов. У томата, люцерны и зерновых PSY3 активен в корнях в ответ на стресс, индуцирует синтез апокаротиноидов (окисленных производных каротиноидов), участвующих в формировании симбиотических и паразитарных отношений, а также в адаптации к дефициту некоторых пищевых элементов [3, 18].
Белки PSY1 и PSY2 высоко гомологичны, однако различаются по биохимическим свойствам [7, 19]. Тем не менее нельзя говорить об их строгой тканеспецифичности: оба гена транскрибируются в одинаковых тканях, но значительно различаются (до 100 раз) уровнями транскрипции [3]. Это обусловлено тем, что эффективность PSY1 в каротиногенезе ниже, чем PSY2, поэтому для синтеза большого количества каротиноидов в плодах необходим более высокий уровень транскрипции PSY1 [20].
Наиболее удобной и изученной моделью для исследования созревания сочных плодов, включая синтез вторичных метаболитов и вероятную эволюцию соответствующих генетических механизмов, считаются виды и сорта томата (секция Lycopersicon рода Solanum) [21]. Виды томата формируют цветки с желтыми лепестками, но различаются окраской зрелых плодов [22]. У томата овощного S. lycopersicum L. оба гена, PSY1 и PSY2, задействованы в определении окраски лепестков [7]. Это свидетельствует об изначально общей роли PSY1 и PSY2 в окраске лепестков и о более раннем происхождении PSY2 по отношению к PSY1, с приобретением PSY1 позднее новой функции – пигментация плодов [23]. Теоретически полученные знания могут быть применены к другим формирующим сочные плоды культурам.
Род Capsicum (Solanaceae) включает более 35 видов, объединенных по морфофизиологическим признакам в несколько филогенетических клад. Виды C. annuum L., C. chinense Jacq., C. frutescens L. (клада Annuum) и C. baccatum L. (клада Baccatum) были доместицированы [24, 25]. Перец формирует сочный плод, окраска которого различается у видов и сортов [26]. Как и у томата, перикарп плодов перца по мере созревания проходит стадию замещения хлоропластов хромопластами, в которых и накапливаются синтезируемые каротиноиды [27]. На сегодняшний день у C. annuum L. обнаружено два паралога гена фитоинсинтазы – PSY1 и PSY2 [28]. В отличие от томата, формирующего цветки с желтыми лепестками, цветки видов Capsicum имеют белую или антоциановую пигментацию, акаротиноидный путь в плодах Capsicum идет дальше синтеза ликопина и каротинов (обычных для плодов томата) и доходит до ксантофиллов β-каротинового пути, из которых под действием специфичного для видов Capsicum фермента капсантинка- псорубинсинтазы (CCS) образуются красные пигменты капсантин и капсорубин [26]. Состав каротиноидов в плодах перца значительно отличается от такового у томата и представлен в основном капсантином и капсорубином, а также β-каротином, β-криптоксантином, лютеином, зеаксантином, антраксантином и виолаксантином, различное сочетание и соотношение которых определяет многообразие оттенков окраски плодов [29]. Суммарное содержание каротиноидов в плодах перца значительно превышает их количество в плодах томата [26, 30]. Учитывая вышесказанное, можно предположить, что прямой перенос данных по каротиногенезу у видов томата на другие виды с сочными плодами, в частности перец, не совсем корректен и необходимы дополнительные уточняющие исследования.
Целью настоящей работы стала оценка вариабельности паралогов фитоинсинтазы у видов Capsicum клады Annuum. Для этого гены, гомологичные PSY1 и PSY2, были идентифицированы у видов перца Capsicum annuum L. (сорта Сибиряк, Сиреневый куб и Отелло), C. frutescens L. (сорт Самоцвет) и C. chinense Jacq. (сорт Pimenta da Neyde). Была охарактеризована структура и филогения идентифицированных генов и определен профиль их экспрессии в листьях, чашелистиках, лепестках, завязи, а также в кожице и мякоти плодав процессе созревания, а также возможные корреляции уровня экспрессии PSY1 и PSY2 с содержанием каротиноидов в плоде в процессе созревания.
МАТЕРИАЛЫ И МЕТОДЫ
Растительный материал. В работе были использованы образцы трех родственных видов перца, формирующих кладу Annuum: C. annuum (сорта Сибиряк, Сиреневый куб и Отелло), C. frutescens (сорт Самоцвет) и C. chinense (сорт Pimenta da Neyde) (рис. 1). Растения были выращены в пленочной теплице в Федеральном научном центре овощеводства (Московская область). Набор тканевых препаратов для каждого анализируемого образца включал молодые листья, чашелистики, лепестки, завязь, а также кожицу и мякоть плода на трех стадиях созревания: незрелый плод финального размера (MF, mature fruit); бланжевый плод (IR, intermediate ripe; соответствует переходу к зрелости с постепенной сменой окраски); плод биологической спелости (RF, ripe fruit). Выбранные для работы сорта представляют два основных паттерна изменения окраски плода в процессе созревания у видов перца комплекса Annuum. Для сорта Сибиряк (C. annuum) последовательность изменения окраски следующая: зеленая (стадия MF), мозаичная зелено-красная (стадия IR), глубокая красная (стадия RF). Для сортов Отелло и Сиреневый куб (C. annuum) – это фиолетовая (стадия MF), красная/фиолетово-красная (стадия IR), глубокая красная (стадия RF). Для вида C. frutescens (Самоцвет): фиолетовая (стадия MF), бледно-желтая (стадия IR) и красная (стадия RF). Образец C. chinense (Pimenta da Neyde) формировал плоды фиолетовые на всех стадиях развития, при этом стадия биологической спелости (RF) у данного сорта не выражена, поэтому были взяты только плоды стадий MF и IR (рис. 1). Индивидуальные ткани растений каждого образца собирали одновременно, измельчали в жидком азоте и хранили при –80°С.
Рис. 1.
Плоды анализируемых сортов перца: а – Сибиряк (C. annuum), б – Сиреневый куб (C. annuum), в – Отелло (C. annuum), г – Самоцвет (C. frutescens), д – Pimenta da Neyde (C. chinense). Цифрами обозначены стадии созревания плодов: 1 – незрелый плод финального размера (MF, mature fruit); 2 – бланжевый плод (IR, intermediate ripe; соответствует переходу к зрелости с постепенной сменой окраски); 3 – плод биологической спелости (RF, ripe fruit). Масштабная линия равна 1 см.

Таким образом, для характеристики межвидовой вариабельности генов фитоинсинтазы были взяты образцы перца с типичными для перцев паттернами пигментации плода и один сорт (Pimenta da Neyde) с аномальной каротиноидной пигментацией.
Биохимический анализ тканей анализируемых образцов перца. Содержание суммы каротиноидов в плодах исследуемых образцов перца определяли методом спектрофотометрии в хлороформ-метанольных экстрактах, согласно [31, 32], в двух биологических и трех технических повторах.
Идентификация гомологов генов PSY1 и PSY2. Из ткани листа каждого из пяти анализируемых образцов перца выделяли геномную ДНК методом СТАВ [33] и использовали (100 нг) в качестве матрицы для амплификации ДНК-последовательностей, гомологичных PSY1 и PSY2. Праймеры для амплификации и секвенирования фрагментов полноразмерных генов разрабатывали на основе доступных в базе данных NCBI полногеномных последовательностей C. annuum PSY1 (LOC107868281 bifunctional 15-cis-phytoene synthase, chromoplastic, Gene ID: 107868281) и PSY2 (LOC107859651 phytoene synthase 2, chloroplastic, Gene ID: 107859651) (табл. 1). Использовали праймеры CaPSYF/R и следующие условия ПЦР: исходная денатурация (94°C 10 мин); 36 циклов денатурации (94°C 40 с), отжига (56°C 40 с) и синтеза (65°C 4 мин); финальное достраивание фрагментов (65°C 7 мин). Гены амплифицировали с использованием полимеразы LongAmp® Hot Start Taq DNA Polymerase (New England Biolabs, Ipswich, MA, США) и термоциклера C1000 Touch Thermal Cycler (Bio-Rad Laboratories, Inc., Hercules, California, США). ПЦР-продукты ожидаемой длины очищали с помощью QIAEX® II Gel Extractionkit (QIAGEN, Hilden, Германия), клонировали в вектор pGEM®-T Easy (Promega, Madison, WI, США) и секвенировали (2–4 клона для каждого образца) на ABI Prism 3700 DNA Analyzer (Applied Biosystems, Waltham, MA, США).
Таблица 1.
Список использованных в работе праймеров
| Ген | Праймер | Последовательность (5' → 3') | Назначение |
|---|---|---|---|
| PSY1 | CaPSY1F | TCAGAATGTCTGTTGCCTTG | Амплификация, секвенирование |
| CaPSY1ex3F | ATGGTGCAGGAGAACAGAC | ||
| CaPSY1ex3R | AACTGTGTCGGACAAAGCAG | ||
| CaPSY1ex5R | CATCTACTAGCTGCGCTCAA | ||
| CaPSY1ex5F | TTAGCACAGGCAGGTCTATC | ||
| CaPSY1R | TCCTGATTTCATGTTCTTGTAGA | ||
| CaPSY1ex1R | ACATCATATACCATCYGTTC | ||
| PSY2 | CaPSY2F | AGCATGTCTGTTGCTTTGTTG | |
| CaPSY2ex3F | CTGATGAGCTTGTTGATGGC | ||
| CaPSY2ex3R | AATCTGGAAACGGTATCGG | ||
| CaPSY2ex4F | CTCTATTGTTACTATGTCGCTG | ||
| CaPSY2ex5R | AATCCTCCACTTATCAGTCAC | ||
| CaPSY2R | CTTCATTCATGTCTTTGYTAGTG | ||
| CaPSY2ex1R | CAACACCACATCATACACC | ||
| PSY1 | PSY1-F PSY1-R |
GTGAAGAGACAGCTGAGATCG TCTCCGGAGTCATTAGCATCG |
РВ-ПЦР |
| PSY2 | PSY2-F PSY2-R |
AAGGAGTCGCAGAACTGAGC GTCGTTCGCTTCAATCTCATCTAA |
|
| Actin7 | Actin7-F Actin7-R |
CATTGTGCTCAGTGGTGGTTC TCTGCTGGAAGGTGCTAAGTG |
Структурный анализ гомологов генов PSY1 и PSY2 и кодируемых ими белков. Анализ и выравнивание полученных нуклеотидных и аминокислотных последовательностей проводили с помощью программы MEGA 7.0 (https://www.megasoftware.net/) в сравнении с известными последовательностями C. annuum PSY1 (Gene ID: 107868281) и PSY2 (Gene ID: 107859651), а также с S. lycopersicum PSY1 (Gene ID: 543988) и PSY2 (Gene ID: 543964). Кроме того, был проведен биоинформационный поиск гомологичных генов в секвенированных геномах видов перца C. baccatum и C. chinense, обнаруженные последовательности были использованы в сравнительном структурном анализе. Положение нуклеотидных и аминокислотных замен определяли внутри группы Capsicum и относительно S. lycopersicum PSY1 и PSY2. Консервативные домены и мотивы в кодируемых белках определяли с помощью программ NCBI-CDD (http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi) и MEME 5.1.1 (http://meme-suite.org/tools/meme). Влияние аминокислотных замен на структуру и функции белков предсказывали с помощью программы PROVEAN (http://provean.jcvi.org/index.php). Структуру белков анализировали с использованием программы Phyre2 (http://www.sbg.bio.ic.ac.uk/~phyre2/html/page.cgi?id=index). Молекулярную массу и значение изоэлектрической точки (pI) определяли с помощью программы Isoelectric Point Calculator (http://isoelectric.org/).
Филогения генов PSY1 и PSY2. Для филогенетического анализа эволюционных отношений, как идентифицированных генов, так и видов перца, использовали известные последовательности генов, кДНК и белков PSY томата S. lycopersicum (PSY1, Gene ID: 543988; CDS ID: EU021055.1 (cv. Red Setter) и EF157835.1; PSY2, Gene ID: 107859651; CDS ID: EU021055.1 (cv. Red Setter)) и видов перца C. annuum cv. Zunla-1 (PSY1, NC_029980.1:c205334820–205328571; PSY2, NC_029978.1:142877052–142881261), C. annuum cv. Valencia (PSY1, GU085273.1), C. baccatum isolate PBC81 (PSY1, CM008446.1:19444825–19449400; PSY2, CM008444.1:141318038–141337266) и C. chinense isolate PI159236 (PSY1, CM008434.1:12099868–12105776; PSY2, CM008432.1:150147614–150156089). Анализ проводили с помощью метода ближайших соседей (NJ; модель подбирали в программе Modeltest) пакета программ MEGA 7.0.
Профиль ко-экспрессии гомологов генов PSY1 и PSY2. Препараты суммарной РНК выделяли из отобранного растительного материала, очищали от примесей ДНК и использовали для синтеза первой цепи кДНК (наборы RNeasy Plant Mini Kit и RNase free DNasy set; QIAGEN, Германия; GoScriptтм Reverse Transcription System (Promega, США).
Профиль экспрессии PSY1 и PSY2 определяли методом количественной ПЦР в реальном времени (РВ-ПЦР) c использованием набора “Реакционная смесь для проведения РВ-ПЦР в присутствии SYBR GreenI и ROX” (ООО “Синтол”, Россия) и термоциклера CFX96 Real-Time PCR Detection System (Bio-Rad Laboratories, США). Праймеры для РВ-ПЦР были разработаны ранее, на основе доступных в базе данных NCBI последовательностей мРНК C. annuum PSY1 (X68017) и PSY2 (XM_016704726.1) [34] (табл. 1). Для нормализации уровня транскрипции генов использовали экспрессию референсного гена Actin7 [35]. Реакции проводили в трех технических повторах в следующих условиях: 95°C – 5 мин; 40 циклов (95°C – 15 с, 62°C – 50 с). Статистическую обработку результатов проводили с помощью программы Graph Pad Prism v. 7.02 (https://www.graphpad.com).
РЕЗУЛЬТАТЫ
Идентификация и анализ структуры гомологов PSY1 и PSY2 у образцов трех видов перца
У образцов видов перца C. annuum (сорта Сибиряк, Сиреневый куб и Отелло), C. frutescens (сорт Самоцвет) и C. chinense (сорт Pimenta da Neyde) были определены геномные последовательности генов PSY1 и PSY2, гомологичные известным генам PSY1 и PSY2 C. annuum (NC_029980.1; NC_029978.1). Последовательности были идентифицированы, начиная с ATG-кодона, без нетранслируемых областей 5'- и 3'-UTR, где у PSY1 и PSY2 S. lycopersicum находятся нетранслируемые экзоны. Основные характеристики полученных генов приведены в табл. 2.
Таблица 2.
Характеристики гомологов генов PSY1 и PSY2 у видов и сортов Capsicum в сравнении с S. lycopersicum
| Образец перца | Ген | NCBI ID | Длина экзона/интрона, пн | Ген, пн | кДНК, пн | Белок, а.о. | MW/pI | |||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| I | II | III | IV | V | VI | |||||||
| C. annuum сорт Сибиряк | PSY1 | MT313932 | 448/96 | 51/261 | 173/328 | 236/250 | 193/649 | 159 | 2844 | 1260 | 419 | 47.140/8.25 |
| PSY2 | MT313937 | 466/101 | 51/744 | 173/193 | 236/219 | 193/429 | 180 | 2985 | 1299 | 432 | 48.535/8.53 | |
| C. annuum сорт Сиреневый куб | PSY1 | MT313933 | 448/96 | 51/261 | 173/328 | 236/291 | 193/649 | 159 | 2885 | 1260 | 419 | 47.066/8.14 |
| PSY2 | MT313938 | 466/101 | 51/744 | 173/193 | 236/219 | 193/429 | 180 | 2985 | 1299 | 432 | 48.430/8.60 | |
| C. annuum сорт Отелло | PSY1 | MT313934 | 448/96 | 51/261 | 173/328 | 236/250 | 193/649 | 159 | 2844 | 1260 | 419 | 47.094/8.25 |
| PSY2 | MT313939 | 466/101 | 51/744 | 173/193 | 236/219 | 193/429 | 180 | 2985 | 1299 | 432 | 48.405/8.43 | |
| C. frutescens сорт Самоцвет | PSY1 | MT313935 | 448/96 | 51/261 | 173/328 | 236/250 | 193/649 | 159 | 2844 | 1260 | 419 | 47.126/8.25 |
| PSY2 | MT313940 | 466/101 | 51/744 | 173/193 | 236/219 | 193/429 | 180 | 2985 | 1299 | 432 | 48.434/8.60 | |
| C. chinense сорт Pimenta da Neyde | PSY1 | MT313936 | 448/96 | 51/261 | 173/328 | 236/291 | 193/649 | 159 | 2885 | 1260 | 419 | 47.052/8.14 |
| PSY2 | MT313941 | 466/101 | 51/753 | 173/193 | 236/219 | 193/433 | 180 | 2998 | 1299 | 432 | 48.362/8.43 | |
| S. lycopersicum сорт Red Setter* | PSY1 | EF534740.1 | 412/120 | 51/423 | 173/313 | 236/518 | 193/689 | 174 | 3302 | 1239 | 413 | 46.615/8.10 |
| PSY2 | EU021055.1 | 484/107 | 51/710 | 173/267 | 242/227 | 193/398 | 180 | 3032 | 1317 | 438 | 49.326/8.05 | |
Длина генов PSY1, содержащих шесть экзонов, составила 2844 пн (C. annuum cv. Сибиряк и Отелло; C. frutescens cv. Самоцвет) и 2885 пн (C. chinense cv. Pimenta da Neyde; C. annuum cv. Сиреневый куб) (табл. 2). Разница в размере генов была обусловлена присутствием вставки 41 пн в интроне IV. Интересно, что в отличие от Pimenta da Neyde в известной последовательности PSY1 образца C. chinense (CM008434.1:12099868–12105776) такой вставки нет. Последовательность PSY1 у C. baccatum (CM008446.1:19444825–19449400) имела длину 2846 пн, а у S. lycopersicum (EF157835.1) – 3302 пн (от ATG до стоп-кодона).
Длина гомологов PSY2 у исследуемых видов/сортов перца составила 2985 пн, за исключением C. chinense cv. Pimenta da Neyde (2998 пн, за счет двух вставок – 9 и 4 пн в интронах II и V). У вида C. baccatum длина гена PSY2 составляла 2994 пн (CM008444.1:141318038–141337266), а у S. lycopersicum – 3032 пн (EU021055.1). Аналогично PSY1 ген PSY2 содержит шесть экзонов, размеры которых идентичны у анализируемых образцов перца.
В сравнении с PSY1 и PSY2 S. lycopersicum в последовательностях PSY1 и PSY2 исследуемых образцов Capsicum было обнаружено 955 и 847 мононуклеотидных вариабельных сайтов SNPs (28.24 и 27.09% от выровненных длин), из которых только 128 (13.40%) и 97 (11.45%) были локализованы в кодирующей последовательности. Между собой геномные последовательности PSY1/PSY2 образцов перца различались числом SNPs: 75 (2.60%)/51 (1.70%), больше трети которых находились в кДНК (24 (32.00%)/19 (37.25%)). При этом большая часть SNPs была локализована в экзоне I: 16 (66.70% всех экзонных замен) у PSY1 и 14 (73.70%) у PSY2.
Размер кДНК PSY1 и PSY2 у всех исследуемых образцов перца составил 1260 и 1299 пн соответственно (табл. 2). Различие в размере кДНК двух паралогов PSY обусловлено большей длиной экзонов I и VI гена PSY2.
Длина белков PSY1 и PSY2 была инвариантна у всех исследуемых образцов клады Annuum и составила 419 и 432 а.о. соответственно (табл. 2; рис. 1 и 2 в Приложении) и отличалась от последовательностей генов фитоинсинтазы C. baccatum (клада Baccatum) четырьмя (D194G, R91Q, C/Y59W, G/A40V – PSY1) и одним (V15F – PSY2) замещениями а.о. (рис. 2), которые, тем не менее, имели нейтральный характер (согласно PROVEAN). В последовательности PSY1 сорта Отелло обнаружено всего одно замещение а.о., которое внутри группы анализируемых образцов может иметь радикальный характер (согласно PROVEAN) – M207V. В последовательности PSY2 сортов Сиреневый куб и Pimenta da Neyde также было обнаружено по одному предположительно радикальному замещению а.о. – S136I и N186T соответственно.
Рис. 2.
Замещения аминокислотных остатков в последовательностях гомологов PSY1 и PSY2 у видов Capsicum в сравнении друг с другом и с референсными последовательностями PSY1 и PSY2 S. lycopersicum.

В сравнении с PSY1 томата все идентифицированные гомологи PSY1 Capsicum имели вставку R53InsQ64 (RWSFGSCLGGAQ) и делецию на С‑конце A408_R412del (ASLQR). В последовательности PSY2 Capsicum, в сравнении с гомологичным белком S. lycopersicum, выявлена делеция L32_F37del (LESSRF) (рис. 2). Анализ инделей в программе PROVEAN показал, что они носят нейтральный характер и не должны существенно изменить конформацию и функцию белка.
Несколько радикальных замещений а.о. было обнаружены при сравнении PSY1 и PSY2 перца с соответствующими белками S. lycopersicum (S. lycopersicum vs. Capsicum spp.): Q79R, P106K, G125S, M195V и W357L – для PSY1 и G71D/G, S142I, N192T и D335G – для PSY2 (рис. 2).
В целом идентифицированные последовательности перца PSY1 и PSY2 были высоко гомологичны (идентичность 82%). Главным отличием PSY2 от PSY1 стало присутствие вставок на N- и C‑конце белка: NAGLRYSD (в положении 58–65 а.о.) и KLTSSSL (в положении 423–429 а.о.).
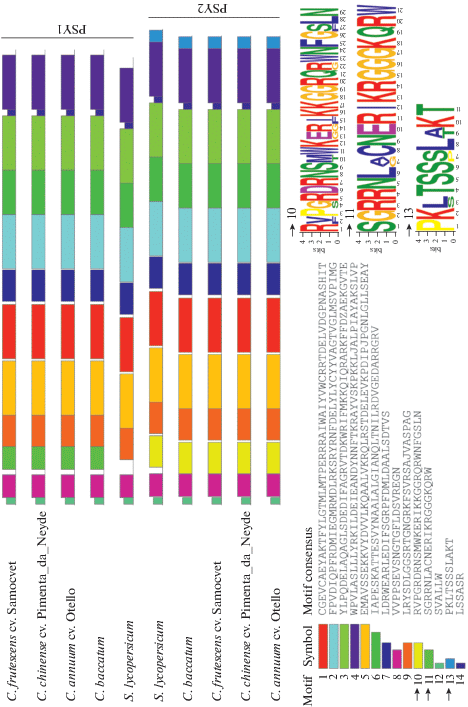
Дополнительно с использованием МЕМЕ 5.1.1 был проведен анализ возможных консервативных мотивов, специфичных для белков PSY1 и PSY2 (рис. 3). Всего было выявлено 14 достоверных мотивов, 11 из которых были общими для всех последовательностей. Были выявлены мотивы 10 и 13, характерные для клады PSY2, и мотив 11, характерный для клады PSY1. Отсутствие мотива 11 в последовательности PSY1 S. lycopersicum было обусловлено отсутствием специфичной для Capsicum вставки R53InsQ64, о которой упоминалось выше.
Рис. 3.
Схема консервативных мотивов, характерных для клад PSY1 и PSY2 Capsicum и S. lycopersicum (МЕМЕ 5.1.1). Для анализа использованы последовательности идентифицированных в данной работе фитоинсинтаз PSY1 и PSY2 сортов Самоцвет (C. frutescens), Отелло (C. annuum) и Pimenta da Neyde (C. chinense), а также видов C. baccatum (CM009446.1; CM08444.1) и S. lycopersicum (EF534740.1; EU021055.1).
Согласно результатам анализа в программе NCBI-CDD все анализируемые белки перца PSY1 (c 75 по 405 а.о.) и PSY2 (с 92 по 430 а.о.) содержат консервативный домен фитоинсинтазы. Это подтверждает принадлежность идентифицированных гомологов PSY1 и PSY2 к ферментам биосинтеза изопреноидов (Isoprenoid Biosynthesis enzymes, Class 1), входящим в суперсемейство синтаз и терпеновых циклаз (trans-isoprenyl diphosphate synthases IPPS and class I terpene cyclases).
Проведенный анализ структуры гомологов PSY1 и PSY2 в программе Phyre2 позволил предсказать возможную трехмерную структуру данных белков. С достоверностью >90% моделированы 68% (PSY1) и 67% (PSY2) аминокислотных остатков с использованием четырех структурных матриц: c4hd1A_ (129–390/135–396 а.о., 62/60%-ное покрытие; squalene synthase HpnC, Alicyclobacillus acidocaldarius); c5iysA_ (126–412/132–419 а.о., 68/66%; phytoene synthase, Enterococcus hirae dehydrosqualene synthase); c3we9A_ (126–405/131–411 а.о., 66/64%; YisP, Bacillus subtilis); c2zcpA_ (3W7F; 126–411/132–418 а.о., 68/66%; c(30) carotenoid dehydrosqualene synthase CrtM, Staphylococcus aureus) [36]. Оставшиеся 143 а.о. (в основном на N-конце) были моделированы ab initio. Было выявлено, что фолдинг PSY-белков имеет преимущественно спиральный характер (60–63%), 2% последовательности приходится на β-складки, а структура 26% неупорядочена. Гомологи обоих фитоинсинтаз имели большую центральную полость, наблюдаемую и в других изопреноидных синтазах, использующих данную полость для пристраивания туда продукта [36]. Единичные случаи обнаруженных нами радикальных миссенс-мутаций (PSY1, Отелло; PSY2, Сиреневый куб и Pimenta da Neyde) никак не влияли на фолдинг и активные центры белков.
Филогенетический анализ
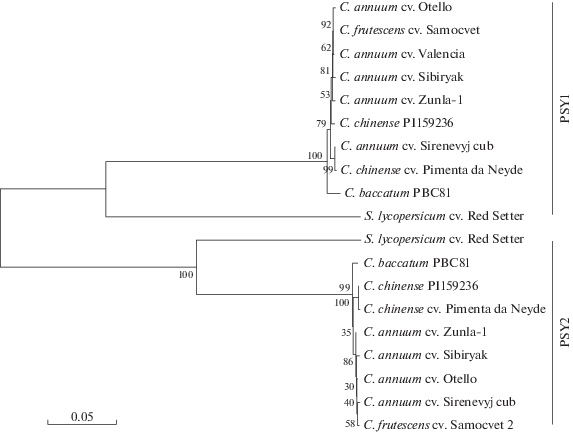
Для оценки филогении исследуемых сортов и видов перца был проведен кластерный анализ и построены дендрограммы на основе полногеномных, кодирующих и белковых последовательностей идентифицированных гомологов PSY1 и PSY2. Для сравнения в анализе были использованы известные последовательности гомологов PSY1 и PSY2 томата S. lycopersicum, а также гомологичные последовательности видов перца C. annuum, C. chinense и C. baccatum из базы данных NCBI.
Все три полученные дендрограммы были конгруэнтны и группировали образцы в два больших кластера, объединяющих последовательности гомологов PSY1 и PSY2 (рис. 4). Внутри каждого кластера последовательности S. lycopersicum формировали сестринскую ветвь кладе Capsicum. Клады PSY1 и PSY2 Capsicum организовывались сходным образом: последовательности C. annuum, C. chinense и C. frutescens формировали группу, сестринскую ветвь к которой образовывал образец C. baccatum (рис. 4).
Профиль ко-экспрессии генов PSY1 и PSY2
Экспрессия генов PSY1 и PSY2 была впервые проанализирована в листьях, чашелистиках, лепестках, завязи, а также в кожице и мякоти плодов на трех стадиях (MF, IR, RF) созревания у образцов трех видов перца (рис. 5).
Рис. 5.
Относительная экспрессия гена PSY1 (a) и PSY2 (б) в листьях (Л), чашелистиках (Ч), лепестках (ЛЕ), завязи (З), кожице (К) и мякоти (М) плодов на трех стадиях созревания (1 – незрелый плод финального размера (MF), 2 – бланжевый плод (IR), 3 – плод биологической спелости (RF)) образцов перца C. annuum (Сибиряк, Сиреневый куб, Отелло), C. frutescens (Самоцвет) и C. chinense (Pimenta da Neyde). Значения, не имеющие достоверного отличия (Pvalue > > 0.05): PSY1 – ЛЕ vs. М3 (Сибиряк, Сиреневый куб), К1 vs. М1 (Отелло); PSY2 – К2 vs. М2 и К2 vs. М3 (Сибиряк), М1 vs. К3, М1 vs. М3 и К3 vs. М3 (Сиреневый куб), М1 vs. М3 (Отелло), Л vs. З (Самоцвет).
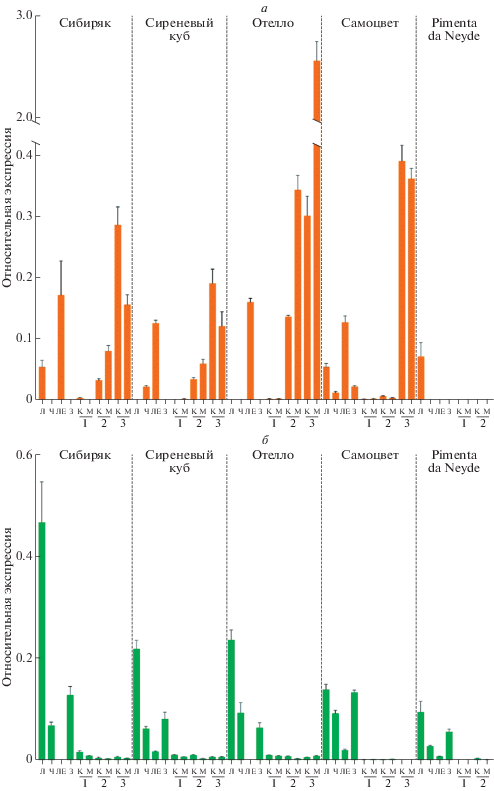
У сортов Сибиряк, Отелло, Сиреневый куб (C. annuum) и Самоцвет (C. frutescens) транскрипты PSY1 были обнаружены во всех анализируемых органах, при этом уровни транскрипции значительно варьировали между разными органами одного образца и однотипными тканями разных образцов. Максимальные уровни экспрессии PSY1 были выявлены в лепестках (на сходном уровне), а также в перикарпе зрелых плодов (стадия 3, RF). И кожица, и мякоть плода данных сортов содержали лишь следовые количества транскриптов PSY1 на стадии 1 незрелого плода (MF) и характеризовались их резким ростом в зреющих плодах (стадии 2 и 3, IR и RF). Мякоть зрелого плода сорта Отелло имела самый высокий уровень транскрипции PSY1 (в 10 раз выше, чем у остальных образцов), однако в кожице уровень экспрессии PSY1 был сопоставим с таковой у других образцов перца (рис. 5).
В листьях анализируемых образцов перца экспрессия PSY1 была на уровне либо следовых количеств (у сортов Отелло и Сиреневый куб), либо достигала 0.06–0.08 (у сортов Самоцвет, Сибиряк и Pimenta da Neyde). В отличие от остальных образцов перца у сорта Pimenta da Neyde (C. chinense) экспрессия PSY1 выявлена исключительно в листьях (рис. 5).
Экспрессия гена PSY2 была выявлена во всех анализируемых органах всех пяти сортов перца (рис. 5). Как ожидалось, максимальный уровень транскрипции был выявлен в листьях и несколько ниже – в чашелистиках и завязи, а в кожице и мякоти плодов уровень экспрессии PSY2 был минимален. У сортов Сиреневый куб, Самоцвет и Pimenta da Neyde значительные количества транскриптов PSY2 были детектированы в лепестках, при этом у первых двух сортов уровни транскрипции были сопоставимы (0.017 и 0.019), а у сорта Pimenta da Neyde – ниже в 2 раза (0.009).
Следует отметить, что анализируемые образцы значительно различались по уровню экспрессии PSY2 в листьях. Так, самый высокий уровень транскрипции PSY2 в листьях был показан для сорта Сибиряк, тогда как остальные сорта имели в 2–4 раза меньшее количество транскриптов PSY2. В целом экспрессия PSY2 в листьях сортов C. annuum была выше, чем у C. chinense и C. frutescens (рис. 5). Интересно, что в чашелистиках (другой фотосинтезирующий орган) и завязи уровни экспрессии PSY2 также различались, но при этом не коррелировали с уровнями экспрессии в листьях (рис. 5).
Содержание пигментов в перикарпе плодов анализируемых образцов видов Capsicum; взаимосвязь содержания суммы каротиноидов и уровня экспрессии PSY
Содержание суммы каротиноидов было определено в перикарпе (кожице и мякоти) плодов в процессе созревания (три стадии: незрелый плод; бланжевая спелость; биологическая спелость) пяти образцов исследуемых видов перца (рис. 6).
Рис. 6.
Содержание суммы каротиноидов (мкг/г сырой массы) в кожице (К) и мякоти (М) плодов анализируемых сортов перца на трех стадиях созревания (1 – незрелый плод финального размера (MF), 2 – бланжевый плод (IR), 3 – плод биологической спелости (RF)).
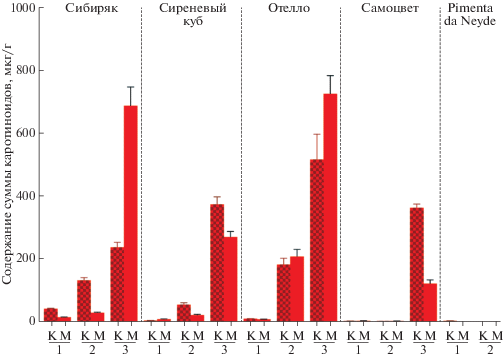
Спелый плод образцов перца, за исключением сорта Pimenta da Neyde, характеризовался высоким содержанием каротиноидов в кожице (236–514 мкг/г сырой массы) и мякоти (121–723 мкг/г) (рис. 6). В спелых плодах Pimenta da Neyde были обнаружены только следовые количества каротиноидов (менее 2 мкг/г). Можно предположить, что красный спелый плод четырех анализируемых сортов содержит типичные для перцев красные пигменты – каротиноиды капсантин и капсорубин [8, 26], при этом принадлежность к определенному виду Capsicum (C. annuum или C. frutescence) никак не связана с количеством накапливаемых каротиноидов. В зрелом плоде сорта Pimenta da Neyde (C. chinense) пигментация исключает каротиноиды и формируется за счет антоцианов [37].
Полученные результаты сравнивали с профилем экспрессии гомологов гена PSY1, экспрессия которого характерна преимущественно для созревающих плодов. Было показано, что увеличение уровней транскрипции PSY1 в перикарпе плодов перца сопровождается увеличением содержания каротиноидов.
ОБСУЖДЕНИЕ
Образцы перца, исследуемые в настоящей работе, представляют три близкородственных вида – C. annuum, C. chinense и C. frutescens, которые вместе составляют кладу Annuum [24, 25]. Самыми близкими к Annuum являются виды клады Baccatum, объединяющей C. baccatum, C. baccatum var. praetermissum (Heiser & P.G. Sm.) Hunz. и C. chacoense Hunz. [24]. Геном C. baccatum секвенирован, что дало нам возможность использовать в сравнительном структурном анализе последовательности PSY1 и PSY2 C. baccatum в качестве представителя другой клады перцев.
Для видов комплекса Annuum, кроме красной финальной окраски плода, характерны два паттерна изменения окраски плода в процессе созревания: от зеленого к красному (основной паттерн); от фиолетового к красному [24, 25]. Из взятых в анализ сортов первый паттерн характерен для сорта Сибиряк (C. annuum). Второй паттерн наблюдается у сортов Отелло и Сиреневый куб (C. annuum), Самоцвет (C. frutescens). У сорта Pimenta da Neyde окраска зрелого плода не меняется и остается фиолетовой (рис. 1), так как в кожице и мякоти не происходит накопления окрашенных каротиноидов. В результате в анализ были взяты четыре сорта с типичным для видов перца комплекса Annuum каротиноидным паттерном пигментации плода и один сорт с аномальной каротиноидной пигментацией.
Нами были идентифицированы гены PSY1 и PSY2 у пяти сортов перца видов клады Annuum. Сравнительный анализ выявил низкий уровень нуклеотидной вариабельности для гомологов каждого гена. Необычным оказалось то, что большая часть (>30%) выявленных SNPs была сконцентрирована в экзонах. Однако при этом какие-либо критичные структурные различия гомологов как между видами Annuum, так и в сравнении с C. baccatum отсутствовали. Последовательности генов не содержали никаких нонсенс-мутаций. При этом единичные случаи радикальных миссенс-мутаций (PSY1, Отелло; PSY2, Сиреневый куб и Pimente da Neyde) не влияли на фолдинг и активные центры белков (согласно Phyre2) и не входили в список мутаций, выявленных ранее при анализе 94 образцов перца и присутствие которых могло бы изменить активность фитоинсинтаз [38].
В целом идентифицированные последовательности перца PSY1 и PSY2 были высоко гомологичны (идентичность 82%), и основным отличием PSY1 от PSY2 стало отсутствие на его N- и С14-конце вставок 8 и 7 а.о. соответственно. Подобные различия характерны и для PSY-белков других видов растений. Так, ранее для фитоинсинтаз злаковых было предположено, что отсутствие/наличие таких вставок может коррелировать с локализацией белков в пластидах конкретного типа [18]. С другой стороны, данное мнение частично опровергается тем фактом, что PSY2 может компенсировать отсутствие PSY1 в плоде перца и, следовательно, PSY2 может работать не только в хлоропластах, но и в хромопластах [38, 39].
Выявленное высокое структурное сходство белков PSY1 и PSY2 у видов перца, в том числе у видов разных клад, предполагает, что и функции гомологов данных белков высоко консервативны среди сортов/видов перца. С достаточной степенью уверенности можно сказать, что по аналогии с тканеспецифичным каротиногенезом у томата S. lycopersicum идентифицированные нами гены перца могут определять биосинтез каротиноидов в созревающих плодах (гомологи PSY1), фотосинтезирующих тканях (гомологи PSY2) и лепестках (PSY1 и PSY2 совместно).
Различные сорта Capsicum классифицируются в основном по морфологическим характеристикам плода, в частности по его окраске, определяемой пигментами, главным образом присутствием и соотношением различных каротиноидов [29]. Динамика изменения окраски меняется по мере созревания плода, при этом синтез каротиноидов идет постоянно. До стадии MF окраска плода либо зеленая (хлоропласты преобладают и хлорофиллы маскируют присутствие каротиноидов), либо фиолетовая (помимо хлоропластов с хлорофиллом и каротиноидами в вакуолях накапливаются антоцианы). По мере созревания (стадии IR, RF) в образующихся хромопластах накапливается сложная смесь каротиноидов, от состава которой зависит окончательная окраска зрелого плода – зелено-коричневая, желтая, оранжевая, красная и/или темно-красная [29, 40].
Среди сортов, взятых в настоящее исследование, Сибиряк (C. annuum) формирует зеленый MF плод, а четыре остальных сорта – фиолетовый. По мере созревания плод сорта Сибиряк меняет окраску с зелено-красной (IR-плод) на темно-красную (RF-плод). Исходно фиолетовые плоды сортов Отелло, Сиреневый куб (оба – C. annuum) и Самоцвет (C. frutescens) меняют окраску с красного/оранжевого оттенка (IR) на красный (RF). Окраска плода у сорта Pimenta da Neyde (C. chinense) – фиолетовая на всех стадиях созревания.
Изменение окраски плода по мере его созревания может определяться различной активностью фитоинсинтазы PSY1. Однако у всех пяти анализируемых образцов, вне зависимости от паттерна пигментации плода и видовой принадлежности, какие-либо радикальные структурные различия между идентифицированными PSY1 отсутствуют. Это предполагает, что разный уровень биосинтеза каротиноидов в плодах данных образцов зависит не от активности фермента PSY1, а от уровня транскрипции кодирующего его гена. И действительно, уровень экспрессии гомологов гена PSY1 в плодах анализируемых сортов перца прямо коррелировал с содержанием каротиноидов и приобретаемой ими по мере созревания окраской. Так, отсутствие транскриптов PSY1 и крайне низкая экспрессия PSY2 в плодах Pimenta da Neyde (C. chinense) коррелирует с зеленой окраской мякоти плода и следовыми количествами каротиноидов. В то же время наиболее высокий уровень экспрессии PSY1 в перикарпе плода сорта Отелло (C. annuum) соответствует темно-красной окраске зрелого плода, по-видимому, за счет более активного синтеза и накопления красных каротиноидов.
Считается, что PSY1 является хромопласт-специфичной фитоинсинтазой [14]. Однако в нашем случае существенная экспрессия PSY1 наблюдалась в листьях и чашелистиках у ряда образцов (Сибиряк, Самоцвет и Pimenta da Neyde). При этом образцы различались по уровню экспрессии гена (рис. 5). Полученные результаты на перцах совпадают с данными по томату и люцерне, где также была показана транскрипция PSY1 в фотосинтезирующих органах [3]. С учетом сказанного, а также принимая во внимание способность PSY2 компенсировать PSY1 [39], можно предположить аналогичные возможности PSY1 по отношению к PSY2 и говорить о влиянии совокупного уровня экспрессии PSY2 и PSY1.
Интересно, что полученные нами паттерны экспрессии PSY1 в тканях перца в целом повторяют профиль транскрипции PSY1 S. lycopersicum [7], чего нельзя сказать о PSY2 (рис. 5). Максимальное число транскриптов PSY2 присутствует в лепестках томата, при этом в листьях, чашелистиках и завязи данный ген экспрессируется заметно слабее [7]. У перца же наибольший уровень транскрипции PSY2 был обнаружен нами в листьях (рис. 5). Мы полагаем, что подобные различия лежат в основе разной пигментации лепестков цветка томата (желтые) и перца клады Annuum (белые/фиолетовые) [23, 29]. Дополнительным тому подтверждением является то, что подавление экспрессии генов PSY1 и PSY2 приводит к почти белой окраске лепестков цветка томата [41].
Таким образом, в настоящем исследовании у пяти образцов трех видов перца клады Annuum идентифицированы и охарактеризованы гомологи генов фитоинсинтаз PSY1 и PSY2, определен профиль ко-экспрессии этих генов в вегетативных и репродуктивных органах и проведена оценка его корреляции с содержанием каротиноидов в плодах. Высокое структурное сходство гомологов между собой свидетельствует о сохранении ими консервативных ключевых функций фитоинсинтаз в синтезе каротиноидов. Отсутствие замещений в функционально значимых участках анализируемых гомологов PSY свидетельствует о корректном транспорте, фолдинге и активности этих ферментов. Показана экспрессия PSY1 не только в плодах, но также и в фотосинтезирующих органах. Предполагается, что изменения в экспрессии PSY1 и PSY2 связаны с полиморфизмом регуляторных областей. Сделано предположение о существовании взаимной функциональной компенсации ферментами PSY1 и PSY2 функций друг друга. Подтверждена прямая корреляция между уровнем экспрессии гена PSY1 и содержанием каротиноидов и, как следствие, с каротиноидной пигментацией плода в процессе созревания.
Работа выполнена при финансовой поддержке гранта Российского научного фонда № 19-16-00016, с использованием экспериментальной установки искусственного климата (ЭУИК, ФИЦ Биотехнологии РАН).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Рис. S1.
Аминокислотные последовательности PSY1 анализируемых сортов перца и сорта Zunla-1 (C. annuum; NP_013111896.1).
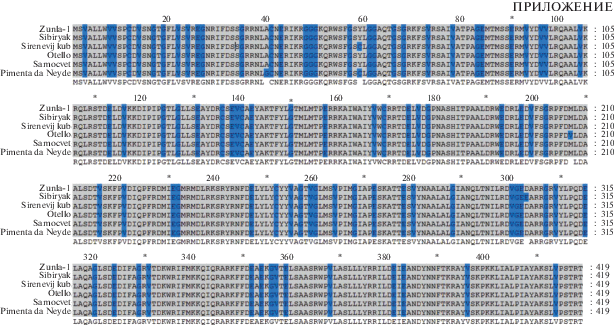
Рис. S2.
Аминокислотные последовательности PSY2 анализируемых сортов перца и сорта Zunla-1 (C. annuum; XP_016560212.1).

Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Hashimoto H., Uragami C., Cogdell R.J. Carotenoids and photosynthesis // In Carotenoids in Nature / Ed. Stange C. Basel, Switzerland: Springer, 2016. P. 111–139.
Nambara E., Marion-Poll A. Abscisic acid biosynthesis and catabolism // Annu. Rev. Plant Biol. 2005. V. 56. P. 165–185.
Stauder R., Welsch R., Camagna M. et al. Strigolactone levels in dicot roots are determined by an ancestral symbiosis-regulated clade of the PHYTOENE SYNTHASE gene family // Front Plant Sci. 2018. V. 9. Article 255. https://doi.org/10.3389/fpls.2018.00255
Sun T., Li L. Toward the ‘golden’ era: The status in uncovering the regulatory control of carotenoid accumulation in plants // Plant Sci. 2020. V. 290. Article 110331. https://doi.org/10.1016/j.plantsci.2019.110331
Rudall P.J. Colourful cones: how did flower colour first evolve? // J. Exp. Bot. 2020. V. 71. № 3. P. 759–767. https://doi.org/10.1093/jxb/erz479
Dardick C., Callahan A.M. Evolution of the fruit endocarp: Molecular mechanisms underlying adaptations in seed protection and dispersal strategies // Front Plant Sci. 2014. V. 5. Article 284. https://doi.org/10.3389/fpls.2014.00284
Giorio G., Stigliani A.L., D’Ambrosio C. Phytoene synthase genes in tomato (Solanum lycopersicum L.) – new data on the structures, the deduced amino acid sequences and the expression patterns // FEBS J. 2008. V. 275. № 3. P. 527–535. https://doi.org/10.1111/j.1742-4658.2007.06219.x
Rodriguez-Uribe L., Guzman I., Rajapakse W. et al. Carotenoid accumulation in orange-pigmented Capsicum annuum fruit, regulated at multiple levels // J. Exp. Bot. 2012. V. 63. № 1. P. 517–526. https://doi.org/10.1093/jxb/err302
Liu L., Shao Z., Zhang M., Wang Q. Regulation of carotenoid metabolism in tomato // Mol. Plant. 2015. V. 8. P. 28–39. https://doi.org/10.1016/j.molp.2014.11.006
Yoo H.J., Park W.J., Lee G.M. et al. Inferring the genetic determinants of fruit colors in tomato by carotenoid profiling // Molecules. 2017. V. 22. № 5. Article E764. https://doi.org/10.3390/molecules22050764
Ahrazem O., Diretto G., Argandoña Picazo J. et al. The specialized roles in carotenogenesis and apocarotenogenesis of the phytoene synthase gene family in saffron // Front Plant Sci. 2019. V. 10. Article 249. https://doi.org/10.3389/fpls.2019.00249
Zhou X., Welsch R., Yang Y. et al. Arabidopsis OR proteins are the major posttranscriptional regulators of phytoene synthase in controlling carotenoid biosynthesis // Proc. Natl Acad. Sci. USA. 2015. V. 112. № 11. P. 3558–3563. https://doi.org/10.1073/pnas.1420831112
Tomato Genome Consortium (Sato S., Tabata S., Hirakawa H. et al.). The tomato genome sequence provides insights into fleshy fruit evolution // Nature. 2012. V. 485. P. 635–641.
Bartley G.E., Viitanen P.V., Bacot K.O., Scolnik P.A. A tomato gene expressed during fruit ripening encodes an enzyme of the carotenoid biosynthesis pathway // J. Biol. Chem. 1992. V. 267. P. 5036–5039.
Ducreux L.J., Morris W.L., Hedley P.E. et al. Metabolic engineering of high carotenoid potato tubers containing enhanced levels of b-carotene and lutein // J. Exp. Bot. 2005. V. 56. P. 81–89.
Fraser P.D., Enfissi E.M., Halket J.M. et al. Manipulation of phytoene levels in tomato fruit: effects on isoprenoids, plastids, and intermediary metabolism // Plant Cell. 2007. V. 19. P. 3194–3211.
Bartley G.E., Scolnik P.A. cDNA cloning, expression during development, and genome mapping of PSY2, a second tomato gene encoding phytoene synthase // J. Biol. Chem. 1993. V. 268. P. 25718–25721.
Gallagher C.E., Matthews P.D., Li F., Wurtzel E.T. Gene duplication in the carotenoid biosynthetic pathway preceded evolution of the grasses // Plant Physiol. 2004. V. 135. №3. P. 1776–1783.
Fraser P.D., Schuch W., Bramley P.M. Phytoene synthase from tomato (Lycopersicon esculentum) chloroplasts – partial purification and biochemical properties // Planta. 2000. V. 211. P. 361–369.
Cao H., Luo H., Yuan H. et al. A neighboring aromatic-aromatic amino acid combination governs activity divergence between tomato phytoene synthases // Plant Physiol. 2019. V. 180. № 4. P. 1988–2003. https://doi.org/10.1104/pp.19.00384
Giovannoni J., Nguyen C., Ampofo B. et al. The epigenome and transcriptional dynamics of fruit ripening // Annu. Rev. Plant Biol. 2017. V. 68. P. 61–84. https://doi.org/10.1146/annurev-arplant-042916-040906
Peralta I.E., Spooner D.M. History, origin and early cultivation of tomato (Solanaceae) // Genetic Improvement of Solanaceous Crops. V. 2: Tomato / Eds Razdan M.K., Mattoo A.K. Science Publ. Enfield. CT, 2007. P. 1–27.
Galpaz N., Ronen G., Khalfa Z. et al. A chromoplast-specific carotenoid biosynthesis pathway is revealed by cloning of the tomato white flower locus // Plant Cell. 2006. V. 18. P. 1947–1960.
Carrizo García C., Barfuss M.H., Sehr E.M. et al. Phylogenetic relationships, diversification and expansion of chili peppers (Capsicum, Solanaceae) // Ann. Bot. 2016. V. 118. № 1. P. 35–51. https://doi.org/10.1093/aob/mcw079
Barboza G.E., Carrizo García C., Leiva González S. et al. Four new species of Capsicum (Solanaceae) from the tropical Andes and an update on the phylogeny of the genus // PLoS One. 2019. V. 14. Article e0209792. https://doi.org/10.1371/journal.pone.0209792
Berry H.M., Rickett D.V., Baxter C.J. et al. Carotenoid biosynthesis and sequestration in red chilli pepper fruit and its impact on colour intensity traits // J. Exp. Bot. 2019. V. 70. № 10. P. 2637–2650. https://doi.org/10.1093/jxb/erz086
Deruère J., Römer S., d’Harlingue A. et al. Fibril assembly and carotenoid overaccumulation in chromoplasts: a model for supramolecular lipoprotein structures // Plant Cell. 1994. V. 6. P. 119–133.
Kilcrease J., Rodriguez-Uribe L., Richins R.D. et al. Correlations of carotenoid content and transcript abundances for fibrillin and carotenogenic enzymes in Capsicum annum fruit pericarp // Plant Sci. 2015. V. 232. P. 57–66. https://doi.org/10.1016/j.plantsci.2014.12.014
Mohd Hassan N., Yusof N.A., Yahaya A.F. et al. Carotenoids of Capsicum fruits: Pigment profile and health-promoting functional attributes // Antioxidants (Basel). 2019. V. 8. № 10. Article E469. https://doi.org/10.3390/antiox8100469
Fraser P.D., Truesdale M.R., Bird C.R. et al. Carotenoid biosynthesis during tomato fruit development (evidence for tissue-specific gene expression) // Plant Physiol. 1994. V. 105. № 1. P. 405–413. https://doi.org/10.1104/pp.105.1.405
Lichtenthaler H.K. Chlorophylls and carotenoids: Pigments of photosynthetic biomembranes // Methods Enzymol. 1987. V. 148. P. 350–382. https://doi.org/10.1016/0076-6879(87)48036-1
Solovchenko A.E., Chivkunova O.B., Merzlyak M.N., Reshetnikova I.V. A spectrophotometric analysis of pigments in apples // Rus. J. Plant Phys. 2001. V. 48. № 5. P. 693–700.
Puchooa D. A simple, rapid and efficient method for the extraction of genomic DNA from lychee (Litchi chinensis Sonn.) // Afr. J. Biotech. 2004. V. 3. P. 253–255.
Филюшин М.А., Джос Е.А., Щенникова А.В., Кочиева Е.З. Зависимость окраски плодов перца от соотношения основных пигментов и профиля экспрессии генов биосинтеза каротиноидов и антоцианов // Физиол. растений. 2020. T. 67. № 6. С. 644–653.
Bemer M., Karlova R., Ballester A.R. et al. The Tomato FRUITFULL homologs TDR4/FUL1 and MBP7/FUL2 regulate ethylene-independent aspects of fruit ripening // Plant Cell. 2012. V. 24. P. 4437. https://doi.org/10.1105/tpc.112.103283
Liu C.I., Liu G.Y., Song Y. et al. A cholesterol biosynthesis inhibitor blocks Staphylococcus aureus virulence // Science. 2008. V. 319. P. 1391–1394.
Филюшин М.А., Джос Е.А., Щенникова А.В., Кочиева Е.З. Особенности экспрессии гена фактора транскрипции anthocyanin2 и его влияния на содержание антоцианов у образцов Capsicum chinense Jacq. с различной окраской плода // Генетика. 2020. Т. 56. № 10. С. 1161–1170.
Jeong H.B., Kang M.Y., Jung A. et al. Single-molecule real-time sequencing reveals diverse allelic variations in carotenoid biosynthetic genes in pepper (Capsicum spp.) // Plant Biotechnol. J. 2019. V. 17. № 6. P. 1081–1093. https://doi.org/10.1111/pbi.13039
Jang S.J., Jeong H.B., Jung A. et al. Phytoene synthase 2 can compensate for the absence of Psy1 in Capsicum fruit // J. Exp. Bot. 2020. Article eraa155. https://doi.org/10.1093/jxb/eraa155
Liu Y., Lv J., Liu Z. et al. Integrative analysis of metabolome and transcriptome reveals the mechanism of color formation in pepper fruit (Capsicum annuum L.) // Food Chem. 2020. V. 306. Article 125629. https://doi.org/10.1016/j.foodchem.2019.125629
Fray R.G., Grierson D. Identification and genetic analysis of normal and mutant phytoene synthase genes of tomato by sequencing, complementation and co-suppression // Plant Mol. Biol. 1993. V. 22. P. 589–602.
Дополнительные материалы отсутствуют.