Генетика, 2021, T. 57, № 3, стр. 345-357
Филогенетические отношения камбалообразных рыб семейства Pleuronectidae (Ostichties: Pleuronectiformes) на основе участка гена 16S рРНК
А. Д. Редин 1, *, Ю. Ф. Картавцев 1, **
1 Национальный научный центр морской биологии им. А.В. Жирмунского
Дальневосточного отделения Российской академии наук
690041 Владивосток, Россия
* E-mail: shurko92@yandex.ru
** E-mail: yuri.kartavtsev48@hotmail.com
Поступила в редакцию 13.04.2020
После доработки 11.09.2020
Принята к публикации 08.10.2020
Аннотация
На основе неполной нуклеотидной последовательности 16S рРНК, полных последовательностей Co-1 и Cyt-b исследована систематика и филогения камбал. Проанализированы 62 образца у 14 видов собственных сборов и из коллекций GenBank/BOLD. Реконструировано четыре типа генных деревьев: байесовское (BA), максимального правдоподобия (ML), минимальной эволюции (ME) и ближайшего соседства (NJ). Эти деревья показали сходную топологию. Две отдельных ветви на деревьях поддерживают выделенные ранее подсемейства Hippoglossoidinae и Pleuronectinae с монофилетическим статусом этих таксонов. Подсемейство Pleuronectinae можно считать монофилетическим, при исключении из него трибы Microstomini, с переносом рода Lepidopsetta в трибу Pleuronectini. Были сформированы и независимо исследованы три набора нуклеотидных последовательностей. Один набор включал все полученные последовательности гена 16S рРНК (291 пн), второй набор включал выборку более длинных последовательностей 16S рРНК (617 пн), третий набор состоял из последовательностей трех генов: 16S рРНК, Co-1 и Cyt-b (2926 пн). Все три набора данных дают схожий филогенетический сигнал, который согласуется с традиционными представлениями о таксономии отряда Pleuronectiformes; однако второй и третий наборы дают лучшую топологию.
Семейство настоящих камбаловых рыб Pleuronectidae, которому в статье уделяется основное внимание, является одним из крупнейших в отряде Pleuronectiformes, включая 59 номинальных видов правосторонних камбаловых рыб, распространенных в морских водах Северного полушария [1, 2]. В своем анализе Дж. Купер и Ф. Чаплау [1] рассматривали семейство Pleuronectidae как монофилетический таксон, основываясь на десяти синапоморфиях по морфологическим признакам. Важный итог, полученный вышеупомянутыми авторами, в целом согласуется с топологией ветвей семейства, установленной в нескольких исследованиях молекулярной филогенетики [3–10]. Согласно [1] это семейство включает подсемейства Hippoglossinae, Eopsettinae, Lyopsettinae, Hippoglossoidinae и Pleuronectinae, которые представлены родами, обычно состоящими из видов с высокой промысловой ценностью (например, виды рода палтусовидных камбал, Hippoglossoides). В связи с рыбохозяйственной значимостью этих и других камбал и необходимостью управлять такими ценными возобновляемыми ресурсами весьма важными являются как точная классификация образцов особей видов в пределах родов, так и вся система взаимоотношений между таксонами в этом семействе.
Таксономические исследования Pleuronectidae традиционно основывались на морфологических признаках, как следует из приведенного выше абзаца. Однако частое отсутствие четких доказательств гомологии признаков у видов даже на низких таксономических уровнях (внутри рода) делает не всегда убедительными постулируемые таксономическо-филогенетические взаимосвязи многих групп камбаловых рыб, если они обоснованы лишь с помощью морфологии. Существует несколько версий классификации камбал, которые были предложены разными авторами [1, 11–13]. Определенные разногласия также отмечаются в отношении филогенетических взаимоотношений камбал, полученных на основе морфологических и молекулярно-генетических данных [1, 4, 14, 15]. Разработка новых ядерных и митохондриальных маркеров на основе ДНК позволяет лучше идентифицировать морфологически сходные виды рыб [16], включая многие виды камбаловых. Поэтому актуальным является поиск новых или уже известных, но недостаточно разработанных молекулярных маркеров для реконструкции генных деревьев, а также комбинированных или видовых филогенетических деревьев для камбал семейства Pleuronectidae.
В настоящем исследовании, учитывая вышеизложенное, представлен сравнительный анализ неполных нуклеотидных последовательностей (далее – последовательности) гена 16S рРНК для 14 видов, относящихся к Pleuronectidae, ранее не использованных в таком объеме для камбал, с целью оценки успешности таксономической идентификации образцов и установления филогенетических и таксономических взаимосвязей в этом семействе камбал. Новизна представленного исследования заключается в том, что систематику данной группы в цитированных выше работах авторов не рассматривали на основе 16S рРНК. Соответственно, в представленной статье рассмотрели потенциал данного маркера на достаточной выборке образцов для таксономических и эволюционно-генетических исследований камбал Российской Федерации.
МАТЕРИАЛЫ И МЕТОДЫ
В общей сложности проанализировали 62 последовательности 16S рРНК и дополнительно по 24 последовательности генов 16S рРНК, Co-1 и Cyt-b для 14 видов, относящихся к семи родам семейства Pleuronectidae. Латинские имена даны в соответствии с классификацией [1]. Пробы (2–5 образцов мышечной ткани, подвергнутой фиксации этанолом, 95%) взяты из имеющейся коллекции Лаборатории молекулярной систематики, а ваучерные экземпляры самих рыб находятся на ответственном хранении в музее ННЦМБ ДВО РАН. Выделение ДНК проводили с помощью коммерческих наборов (“ДНК Экстран-2”, Синтол, Россия).
Фрагмент последовательности гена 16S рРНК амплифицировали посредством полимеразной цепной реакции (ПЦР) с помощью праймеров 16Sbr-H и 16Sar-L. Реакцию ПЦР проводили в объеме 25 мкл раствора, содержащего: дистиллированную деионизированную воду – 17.8 мкл; dNTP (ЗАО “Евроген”, Москва, Россия) – 0.5 мкл; 5× Buffer (Evrogen) – 5 мкл; праймеры в концентрации 10 мкМ/мкл – по 0.3 мкл для каждого; Taq-полимераза – 0.1 мкл. Использовали следующую тепловую программу: денатурирование при 93°С в течение 1 мин, отжиг при 55°С в течение 1 мин и элонгация при 72°С в течение 1 мин для 33 циклов. Для определения локализации и порядка расположения нуклеотидов в последовательностях продукты ПЦР (ДНК-образцы) подвергали циклическому секвенированию с помощью набора для секвенирования циклов BrightDye Terminator по следующей программе: денатурирование при 96°С – 10 с, отжиг при 45°С – 10 с, элонгация при 60°С – 2 мин.
Двунаправленные последовательности цепей ДНК генов 16S рРНК были получены для каждого ДНК-образца. Эти последовательности затем объединяли вместе для получиния консенсусных последовательностей каждого образца. Данная процедура выполнена с использованием программного пакета Geneious, Free Trial [17].
Так как длина полученных последовательностей варьировала довольно значительно, в диапазоне от 355 пар нуклеотидов (пн) до 642 пн, то для более точного дальнейшего анализа составили два набора последовательностей. Один набор представлял собой все полученные последовательности, а другой включал только наиболее длинные последовательности. Соответственно преобразованию данных набор 1 содержал 62, а набор 2 – 27 последовательностей. После проведения процедуры выравнивания и удаления гэпов (инделов) длина последовательностей двух наборов составила 291 и 617 пн соответственно.
Выравнивание последовательностей для всех таксонов было выполнено с использованием программного пакета (ПП) MEGA-X (http://megasoftware.net/) [18] на основе модуля ClustalW [19], как интегрированного продукта MEGA. Штрафы за открытие пропусков и за удлинение пропусков были установлены размером 15.0 и 5.0 соответственно (для других настроек программы выравнивания использовали параметры по умолчанию). После первого этапа выравнивания большие пробелы были удалены вручную, и окончательное выравнивание на втором этапе выполнено с уменьшенными уровнями штрафов (5.0 и 0.5 для двух опций соответственно). Все пробелы были затем снова удалены вручную.
Для увеличения информационной емкости кроме гена 16S рРНК в анализ включены последовательности генов Co-1 и Cyt-b, ранее использованные в анализе [10]; в совокупности эти данные составили третий набор последовательностей, включающий всего 24 образца длиной 2926 пн.
Для дальнейшего анализа последовательностей и построения генных деревьев подобрали оптимальную модель замены нуклеотидов для полученного набора последовательностей. Лучшая модель эволюции, которая соответствовала полученным данным, оценена посредством специального модуля программы MEGA. Для набора гена 16S рРНК с короткими последовательностями (291 пн) наилучшей моделью оказалась K2P + G (двухпараметрическая модель М. Кимуры с гамма-распределением замен) [20], для набора этого гена с длинными последовательностями (617 пн) лучшей была модель JC + G (модель Джукса–Кантора с гамма-распределением замен) [21], для набора последовательностей трех генов наилучшей моделью оказалась HKY + G (модель Хасегава–Кишино–Яно с гамма-распределением замен) [22].
Генные деревья были построены посредством четырех методов реконструкции: на основе байесовского анализа (BA), максимального правдоподобия (ML), ближайшего соседства (NJ) и минимальной эволюции (ME). Они были выполнены в MrBayes 3.2.7 (http://nbisweden.github.io/MrBayes/download.html) [23, 24] и MEGA-X [18]. Моделирование процесса реконструкции деревьев в BA проводили в течение одного миллиона поколений n (n = 106). Три другие реконструкции ML, NJ, ME проводили с повторностями равными k = 1000 копий бутстрепа (бутстреп-поддержки).
В качестве внешней группы при укоренении деревьев выбрали ветвь Platichthys stellatus, представитель которой по данным для полного митогенома (мтДНК) ранее был отнесен к внешней ветви в семействе Pleuronectidae [10]. Филогенетические деревья визуализировали и редактировали при необходимости с помощью программного обеспечения FigTree [25] и MEGA-X [18].
Все полученные последовательности по гену 16S рРНК были зарегистрированы в GenBank (https://www.ncbi.nlm.nih.gov/); часть не опубликованных ранее последовательностей Co-1 и Cyt-b также включена в статью (табл. 1).
Таблица 1.
Список видов и присвоенные им номера в генном банке
| Видовое название с лабораторным номером | Номер доступа в NCBI | ||
|---|---|---|---|
| 16S | Co-I | Cyt-b | |
| Pseudopleuronectes herzensteini 7k | MN888867 | KF386364 | KF445172 |
| Pseudopleuronectes herzensteini 05-07 | MN888868 | KF386361 | KF445169 |
| Pseudopleuronectes herzensteini 6k | MN888869 | KF386363 | KF445171 |
| Pseudopleuronectes schrenki 119-07 | MN888870 | ||
| Liopsetta pinnifasciata 90-07 | MN888871 | ||
| Liopsetta pinnifasciata 40-07 | MN888872 | ||
| Liopsetta pinnifasciata 39-07(2) | MN888873 | ||
| Liopsetta pinnifasciata 39-07 | MN888874 | ||
| Liopsetta pinnifasciata 21-07 | MN888875 | ||
| Liopsetta pinnifasciata 20-07 | MN888876 | KF386379 | KF445186 |
| Liopsetta pinnifasciata 2 | MN888877 | ||
| Liopsetta pinnifasciata 19-07 | MN888878 | KF386378 | KF445185 |
| Liopsetta pinnifasciata 13 | MN888879 | KF386377 | KF445184 |
| Pseudopleuronectes yokohamae 46 | MN888880 | KF386355 | KF445163 |
| Pseudopleuronectes yokohamae 45 | MN888881 | KF386354 | KF445162 |
| Pseudopleuronectes obscurus 42-07 | MN888882 | ||
| Pseudopleuronectes obscurus 36 | MN888883 | ||
| Pseudopleuronectes yokohamae 09-07 | MN888884 | KF386353 | KF445161 |
| Pseudopleuronectes obscurus 08-08(08-07) | MN888885 | ||
| Limanda punctatissima 89-07 | MN888886 | ||
| Limanda punctatissima 86-07 | MN888887 | KF386388 | KF445195 |
| Limanda punctatissima 58-07 | MN888888 | ||
| Limanda punctatissima 50-07 | MN888889 | ||
| Limanda punctatissima 37 | MN888890 | KF386386 | KF445193 |
| Lepidopsetta mochigarei LMO12-5 | MN888891 | ||
| Lepidopsetta mochigarei LMO12-4 | MN888892 | ||
| Lepidopsetta mochigarei LMO12-3 | MN888893 | ||
| Lepidopsetta mochigarei LMO12-2 | MN888894 | ||
| Lepidopsetta mochigarei LMO12-1 | MN888895 | ||
| Platichthys stellatus 11-07 | MN888896 | ||
| Platichthys stellatus4k | MN888897 | KF386371 | KF445178 |
| Platichthys stellatus PS6-011 | MN888898 | ||
| Platichthys stellatus PS5-011 | MN888899 | ||
| Platichthys stellatus PS4-011 | MN888900 | ||
| Platichthys stellatus Ps3-011 | MN888901 | ||
| Platichthys stellatus Ps2-011 | MN888902 | ||
| Platichthys stellatus 18-07 | MN888903 | KF386375 | KF445182 |
| Platichthys stellatus 16-07 | MN888904 | ||
| Platichthys stellatus 15-07 | MN888905 | ||
| Liopsetta pinnifasciata PG1-011 | MN888906 | ||
| Liopsetta pinnifasciata Pc2-011 | MN888907 | ||
| Liopsetta pinnifasciata Pc1-011 | MN888908 | ||
| Liopsetta pinnifasciata 2k | MN888909 | KF386376 | KF445183 |
| Limanda sakhalinensis 72(2012) | MN888910 | KF386382 | KF445189 |
| Limanda sakhalinensis 71(2012) | MN888911 | KF386381 | KF445188 |
| Limanda sakhalinensis 69(2012) | MN888912 | ||
| Limanda sakhalinensis 68(2012) | MN888913 | ||
| Hippoglossus stenolepis HST12-4 | MN888914 | ||
| Hippoglossus stenolepis HST12-3 | MN888915 | ||
| Hippoglossus stenolepis HST12-2 | MN888916 | ||
| Hippoglossus stenolepis HST12-1 | MN888917 | ||
| Hippoglossoides robustus 289 | MN888918 | KF386414 | KF445220 |
| Hippoglossoides robustus 288 | MN888919 | KF386413 | KF445219 |
| Hippoglossoides robustus 286 | MN888920 | KF386411 | KF445217 |
| Hippoglossoides elassodon 35 | MN888921 | ||
| Hippoglossoides elassodon 34 | MN888922 | KF386418 | KF445223 |
| Hippoglossoides elassodon 33 | MN888923 | KF386417 | KF445222 |
| Hippoglossoides robustus 31 | MN888924 | KF386410 | KF445216 |
| Hippoglossoides dubius 5k | MN888925 | ||
| Cleisthenes pinetorum 79-07 | MN888926 | KF386409 | KF445215 |
| Cleisthenes pinetorum 78-07 | MN888927 | KF386408 | KF445214 |
Статистический анализ нуклеотидного состава выполнен с использованием ПП MEGA-X. Дополнительно с помощью программного пакета Statistica 6 [29] провели однофакторный дисперсионный анализ (ANOVA) нуклеотидного состава отдельно по каждому гену.
РЕЗУЛЬТАТЫ
Анализ строили, базируясь на последовательностях трех представленных ранее наборов данных. Полная длина участка гена 16S рРНК (полноразмерные последовательности “от праймера до праймера”) составляет 596–631 пн. Номера полноразмерных участков гена 16S рРНК следующие: MN888911, MN888895, MN888908, MN888877, MN888901, MN888894, MN888893, MN888892, MN888917, MN888916, MN888915, MN888903, MN888924, MN888918, MN888912, MN888904, MN888905, MN888868, MN888873, MN888898, MN888899, MN888902, MN888927, MN888909, MN888907, MN888884, MN888883, MN888876.
Однако не все полученные последовательности достигли полного размера. Разница в длине последовательностей обусловлена некачественным секвенированием некоторых образцов, что привело к большему “обрезанию” участков рядом с праймерами при формировании консенсусных последовательностей. Эти погрешности секвенирования возможны в связи с тем, что часть образцов тканей хранились до анализа несколько лет. Однако короткие фрагменты не обязательно плохие для оценки изменчивости в близких таксонах и сопоставления степени их сходства-различия для реконструкции генного дерева. В связи с этим для анализа материал разбили на две группы, включающие длинные (1) и короткие (2) последовательности гена. В соответствии с изложенными подходами (в разделе “Материалы и методы”) построено четыре типа деревьев: BA, ML, NJ и ME.
Анализ всех последовательностей 16S рРНК
На рис. 1 показано укорененное ML-дерево, полученное на основе набора последовательностей гена 16S рРНК длиной 291 пн. Поддержки на деревьях указаны в следующем порядке: BA/ME/ NJ/ML.
Рис. 1.
Укорененное генное дерево, показывающее филогенетические взаимосвязи на основе 62 коротких нуклеотидных последовательностей участка 16S рРНК. Топология представлена на основе ML-реконструкции. В узлах даны значения поддержки для четырех способов реконструкции деревьев в порядке: BA/ME/NJ/ML. Для BA-дерева показаны апостериорные вероятности (%, n = 106 поколений), а для трех других реконструкций даны бутстреп-поддержки (k = 1000 реплик).
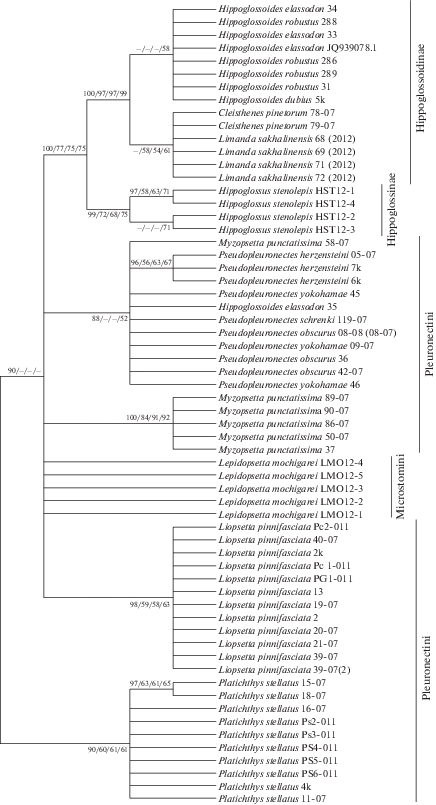
Ветвь с образцами Limanda sakhalinensis вошла в состав подсемейства Hippoglossoidinae, образуя отдельную, топологически неразрешенную ветвь (узел) вместе с Cleisthenes pinetorum подсемейства Hippoglossoidinae. Отдельная, также неразрешенная ветвь сформирована представителями трех номинальных видов палтусовидных камбал рода Hippoglossoides. Lepidopsetta mochigarei, представитель трибы Microstomini, имеет для всех пяти соответствующих ветвей на дереве неразрешенную топологию. Отдельный узел на древе формируют представители тихоокеанского белокорого палтуса Hippoglossus stenolepis из подсемейства Hippoglossinae.
Анализ более длинных последовательностей 16S рРНК
На рис. 2 представлено укорененное NJ-дерево, полученное на основе набора последовательностей гена 16S рРНК длиной 617 пн. Ветвь Limanda sakhalinensis включена в подсемейство Hippoglossoidinae, располагаясь в одном кластере с Cleisthenes pinetorum. Последовательности, представляющие подсемейство Hippoglossinae, образуют отдельный узел. Lepidopsetta mochigarei, как и ранее по короткому фрагменту, формирует неразрешенный узел, но топологически входит в трибу Microstomini.
Рис. 2.
Топология укорененного NJ-дерева, показывающая филогенетические взаимоотношения на основании данных для 27 последовательностей участка гена 16S рРНК. В узлах даны значения поддержек для BA-древа (n = 106 поколений) и трех других реконструкций в общем порядке: BA/ME/ML/NJ (k = 1000 реплик бутстрепа для трех последних методов реконструкции).
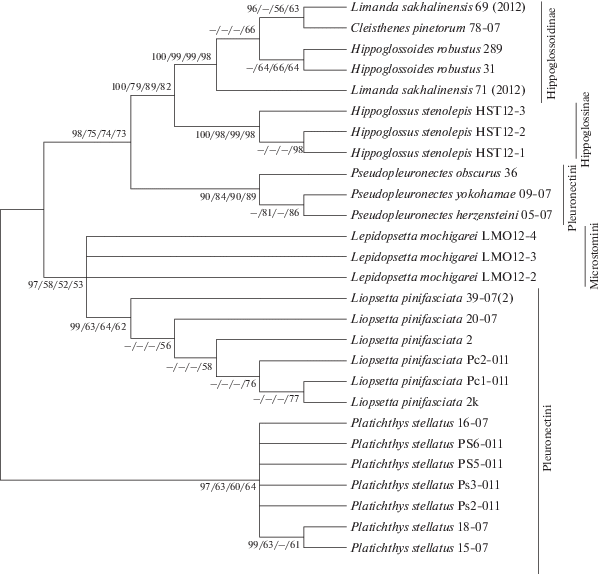
Анализ реконструкции генных деревьев по объединенным последовательностям трех генов
Для данного анализа были сопоставлены выровненные последовательности участка гена 16S рРНК, а также генов Co-1 и Cyt-b. Последовательности были конкатенированы в MEGA-X и затем подвергнуты дальнейшему анализу. Согласно полученным данным ветвь Limanda sakhalinensis включена в подсемейство Hippoglossoidinae (рис. 3). Ветвь Cleisthenes pinetorum также включена в подсемейство Hippoglossoidinae (рис. 3).
Рис. 3.
Укорененное ML-дерево, показывающее филогенетические взаимоотношения на основании данных для 24 конкатенированных последовательностей участка гена 16S рРНК, Co-1 и Cyt-b. В узлах даны значения поддержек для BA-древа (n = 106 поколений) и для трех других реконструкций в общем порядке: BA/NJ/ME/ML (k = 1000 реплик бутстрепа для трех последних методов реконструкции).
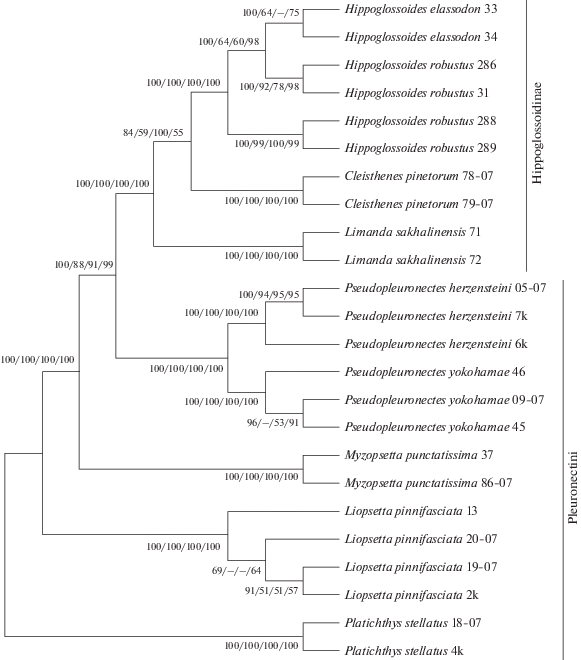
Нуклеотидный состав
Соотношение пиримидинов (T, C) и пуринов (A, G) в генах 16S рРНК, Co-1 и Cyt-b отклонялись от соотношения 50 : 50 (Приложение, рис. 4). В последовательностях 16S рРНК нет больших различий в соотношении пиримидинов (T, C) и пуринов (A, G), но можно наблюдать общую гетерогенность состава нуклеотидов с преобладанием С- и А-нуклеотидов (рис. 4,а). В случае Co-1 и Cyt-b наблюдается статистически значимое отклонение в соотношении пиримидинов к пуринам с преобладанием пиримидинов (рис. 4,б, в).
Рис. 4.
Средние значения состава (%) четырех нуклеотидов в 24 исследованных последовательностях генов 16S рРНК (а), Co-1 (б) и Cyt-b (в). По результатам однофакторного дисперсионного анализа (ANOVA). Вертикальные линии отмечают 95%-ный доверительный интервал.
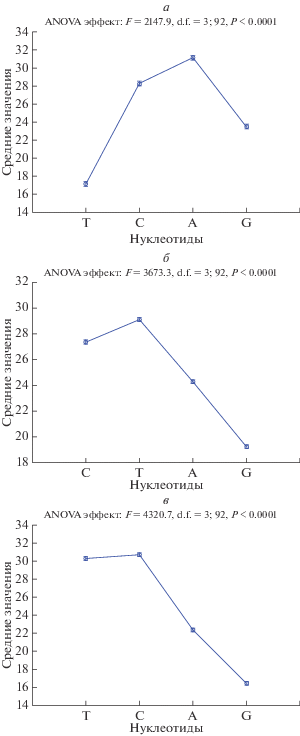
ANOVA по каждому гену обнаружил, что различия для четырех нуклеотидов статистически значимые: для 16S рРНК – F = 2147.9, d.f. = 3; 92, P < 0.0001; для Co-1 – F = 3673.3, d.f. = 3; 92, P < < 0.0001; для Cyt-b – F = 4320.7, d.f. = 3; 92, P < < 0.0001. Доля (T + C) : (A + G) для 16S рРНК, Co-1 и Cyt-b составила 45.4 : 54.6, 56.5 : 43.5 и 61.1 : : 38.9% соответственно.
ОБСУЖДЕНИЕ
Как отмечено во введении, крупнейшим подсемейством в семействе является Pleuronectinae. Это подсемейство представлено двумя трибами – Microstomini и Pleuronectini. Согласно полученным данным по 16S рРНК они не образуют монофилетические ветви (см. рис. 1, 2). Таким образом, систематика на уровне подсемейств нуждается в дальнейшем уточнении. Например для большего информационного сигнала необходимо увеличить число как ядерных, так и митохондриальных маркеров при исследовании. Это поможет уменьшить число неразрешенных топологически узлов полученных деревьев.
Как отмечалось, Дж. Купер и Ф. Чаплау [1] в своей ревизии этого семейства на основе традиционных признаков морфологии обосновали, что Pleuronectidae представляет монофилетическую группу. Монофилия камбаловых, установленная на основе классического подхода, соответствует во многих случаях молекулярно-филогенетическим реконструкциям в исследованиях этого семейства по таким маркерам как 12S рРНК, 16S рРНК, а также по генам Cо-1 и Cyt-b [4–6, 8, 9] и полному митогеному [10].
В подсемейство Hippoglossoidinae в наиболее представительном в работе материале вошли два из трех родов Cleisthenes (C. pinetorum) и Hippoglossoides (H. dubius Schmidt, 1904, H. elassodon Jordan & Gilbert, 1180, H. robustus Gill & Townsend, 1897) (рис. 3). Виды рода Hippoglossoides образуют смешанный кластер на BA-дереве (рис. 3). На основании чего можно предположить, что два таксона H. elassodon и H. robustus являются синонимами одного вида. Синонимия H. elassodon и H. robustus уже предлагалась ранее на основании морфологических и молекулярно-филогенетических данных [4, 27–31]. По принципу старшинства можно принять валидным таксон видового ранга H. elassodon Jordan & Gilbert, 1180, а H. robustus Gill & Townsend, 1897 считать младшим синонимом этого вида. Предложение о сведении в синонимию H. elassodon и H. robustus уже сделано ранее, как отмечено выше. Однако К.А. Винников с соавт. [31] в своем двустороннем анализе (морфология + + генетика) предлагают вновь ввести синонимию. Но синонимия так и не введена. В базах данных эти два таксона до сих пор фигурируют как самостоятельные виды. Одна из задач представленной статьи – заострить этот вопрос, с тем чтобы в специальной публикации наконец разрешить этот казус.
Отдельного обсуждения заслуживают данные для рода Limanda. Последовательности вида Limanda sakhalinensis оказались, как отмечено в результатах и представлено на рис. 1–3, включены в ветвь подсемейства Hippoglossoidinae. В сравнительном анатомическом исследовании Дж. Купера и Ф. Чаплау [1] монофилия этого рода не была подтверждена. В нашем же исследовании, как и в предыдущих молекулярно-филогенетических исследованиях [4, 5, 10], Limanda sakhalinensis Hubbs, 1915 оказалась включена в подсемейство Hippoglossoidinae. Учитывая все эти данные, вполне уместно рекомендовать пересмотреть позицию Limanda sakhalinensis Hubbs, 1915, переместив ее в род Hippoglossoides с именем Hippoglossoides (Limanda) sakhalinensis в составе подсемейства Hippoglossoidinae. Соответственно, необходима ревизия морфологии и видовых признаков, а также диагностических ключей, что предполагается выполнить в самостоятельной работе.
Род Lepidopsetta (L. mochigarei) оказался включенным в трибу Pleuronectini, подсемейства Pleuronectinae, тогда как в работе [1] этот род рассматривался исключительно в составе трибы Microstomini подсемейства Pleuronectinae. В молекулярно-филогенетических исследованиях на основе Co-1 и Cyt-b [4, 5, 10] род Lepidopsetta рассматривался в трибе Pleuronectini. Таким образом, род Lepidopsetta предпочтительнее рассматривать в составе трибы Pleuronectini. Однако этот вопрос, учитывая слабый топологический сигнал для данной ветви по маркеру 16S рРНК в работе, требует дальнейшего уточнения с использованием большего числа генов.
Смещение в соотношении (T + C) : (A + T) хорошо описано в литературе для многих белок-кодирующих генов [4, 32]. Представленный анализ (рис. 4, Приложение) показывает, что смещение для генов Co-1 и Cyt-b в соотношении пуринов к пиримидинам значительно отличается от смещения для гена 16S рРНК. Очевидно, что обнаруженное смещение нуклеотидного состава для изученных в работе структурных генов отражает гидрофобные свойства кодируемых ими белков [33]. Выяснение причины неоднородности нуклеотидного состава в последовательностях 16S рРНК требует дальнейшего исследования.
Исследование финансово поддержано грантом РФФИ 15-29-02456-офи по направлению исследования генетических основ биоразнообразия, а также Дальневосточным отделением РАН – грант ДВ № 18-4-040 по тематике “Комплексное исследование биоразнообразия рыб и беспозвоночных животных на основе ДНК-штрих кодирования, разработки и поддержки баз данных и биобанкинга”.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
ПРИЛОЖЕНИЕ Нуклеотидный состав для генов 16S рРНК, Co-1 и Cyt-b в 24 последовательностях камбалообразных рыб
| Вид/Средние значения | Нуклеотиды, все позиции (% от общего числа) | ||||
|---|---|---|---|---|---|
| T | C | A | G | суммарно (пн) | |
| 16S рРНК | |||||
| Cleisthenes pinetorum 78-07 | 16.8 | 28.9 | 29.9 | 24.4 | 291 |
| Cleisthenes pinetorum 79-07 | 16.8 | 28.9 | 29.9 | 24.4 | 291 |
| Hippoglossoides elassodon 33 | 16.8 | 28.9 | 30.2 | 24.1 | 291 |
| Hippoglossoides elassodon 34 | 16.8 | 28.9 | 29.9 | 24.4 | 291 |
| Hippoglossoides robustus 286 | 16.8 | 28.9 | 30.2 | 24.1 | 291 |
| Hippoglossoides robustus 288 | 17.2 | 28.5 | 30.2 | 24.1 | 291 |
| Hippoglossoides robustus 289 | 16.8 | 28.9 | 30.2 | 24.1 | 291 |
| Hippoglossoides robustus 31 | 16.8 | 28.9 | 30.2 | 24.1 | 291 |
| Limanda sakhalinensis 71 | 16.8 | 28.9 | 29.9 | 24.4 | 291 |
| Limanda sakhalinensis 72 | 16.8 | 28.9 | 29.9 | 24.4 | 291 |
| Liopsetta pinnifasciata 13 | 17.5 | 27.8 | 31.6 | 23.0 | 291 |
| Liopsetta pinnifasciata 19-07 | 17.5 | 27.8 | 31.6 | 23.0 | 291 |
| Liopsetta pinnifasciata 20-07 | 17.5 | 27.8 | 31.6 | 23.0 | 291 |
| Liopsetta pinnifasciata 2k | 17.5 | 27.8 | 31.6 | 23.0 | 291 |
| Myzopsetta punctatissima 37 | 17.2 | 27.5 | 32.3 | 23.0 | 291 |
| Myzopsetta punctatissima 86-07 | 17.2 | 27.5 | 32.3 | 23.0 | 291 |
| Platichthys stellatus 18-07 | 17.2 | 27.8 | 31.3 | 23.7 | 291 |
| Platichthys stellatus 4k | 17.2 | 27.8 | 31.6 | 23.4 | 291 |
| Pseudopleuronectes herzensteini 05-07 | 17.5 | 27.8 | 32.0 | 22.7 | 291 |
| Pseudopleuronectes herzensteini 6k | 17.5 | 27.8 | 32.0 | 22.7 | 291 |
| Pseudopleuronectes herzensteini 7k | 17.5 | 27.8 | 32.0 | 22.7 | 291 |
| Pseudopleuronectes obscurus 09-07 | 17.2 | 28.2 | 32.0 | 22.7 | 291 |
| Pseudopleuronectes obscurus 45 | 16.8 | 28.2 | 32.0 | 23.0 | 291 |
| Pseudopleuronectes obscurus 46 | 17.2 | 28.2 | 32.0 | 22.7 | 291 |
| Среднее значение | 17.14 ± 0.13 | 28.26 ± 0.13 | 31.1 ± 0.13 | 23.5 ± 0.13 | – |
| Co-1 | |||||
| Cleisthenes pinetorum 78-07 | 29.4 | 27.5 | 23.6 | 19.5 | 1540 |
| Cleisthenes pinetorum 79-07 | 29.4 | 27.3 | 23.7 | 19.5 | 1540 |
| Hippoglossoides elassodon 33 | 29.3 | 27.6 | 24.0 | 19.2 | 1540 |
| Hippoglossoides elassodon 34 | 29.1 | 27.6 | 24.0 | 19.4 | 1540 |
| Hippoglossoides robustus 286 | 29.0 | 27.8 | 24.0 | 19.2 | 1540 |
| Hippoglossoides robustus 288 | 29.2 | 27.5 | 24.0 | 19.3 | 1540 |
| Hippoglossoides robustus 289 | 29.2 | 27.4 | 24.1 | 19.3 | 1540 |
| Hippoglossoides robustus 31 | 29.0 | 27.8 | 24.0 | 19.2 | 1540 |
| Limanda sakhalinensis 71 | 28.8 | 27.1 | 24.4 | 19.7 | 1540 |
| Limanda sakhalinensis 72 | 28.6 | 27.3 | 24.4 | 19.7 | 1540 |
| Liopsetta pinnifasciata 13 | 29.4 | 27.0 | 24.8 | 18.8 | 1540 |
| Liopsetta pinnifasciata 19-07 | 29.4 | 27.0 | 24.8 | 18.8 | 1540 |
| Liopsetta pinnifasciata 20-07 | 29.4 | 27.0 | 24.8 | 18.8 | 1540 |
| Liopsetta pinnifasciata 2K | 29.4 | 27.0 | 24.8 | 18.8 | 1540 |
| Myzopsetta punctatissima 37 | 28.8 | 27.2 | 24.7 | 19.2 | 1540 |
| Myzopsetta punctatissima 86-07 | 28.8 | 27.2 | 24.7 | 19.2 | 1540 |
| Platichthys stellatus 18-07 | 29.0 | 27.2 | 24.8 | 19.0 | 1540 |
| Platichthys stellatus 4K | 28.8 | 27.3 | 24.8 | 19.0 | 1540 |
| Pseudopleuronectes herzensteini 05-07 | 28.8 | 28.0 | 23.7 | 19.5 | 1540 |
| Pseudopleuronectes herzensteini 6K | 28.7 | 28.0 | 23.7 | 19.6 | 1540 |
| Pseudopleuronectes herzensteini 7K | 28.7 | 28.0 | 23.6 | 19.7 | 1540 |
| Pseudopleuronectes obscurus 09-07 | 29.3 | 26.9 | 24.5 | 19.2 | 1540 |
| Pseudopleuronectes obscurus 45 | 29.3 | 27.0 | 24.5 | 19.2 | 1540 |
| Pseudopleuronectes obscurus 46 | 29.4 | 27.1 | 24.5 | 19.1 | 1540 |
| Среднее значение | 29.09 ± 0.07 | 27.38 ± 0.07 | 24.29 ± 0.07 | 19.25 ± 0.07 | – |
| Cyt-b | |||||
| Cleisthenes pinetorum 78-07 | 30.8 | 30.9 | 22.5 | 15.9 | 1095 |
| Cleisthenes pinetorum 79-07 | 30.8 | 30.8 | 22.6 | 15.8 | 1095 |
| Hippoglossoides elassodon 33 | 30.3 | 31.2 | 22.2 | 16.3 | 1095 |
| Hippoglossoides elassodon 34 | 30.3 | 31.2 | 22.2 | 16.3 | 1095 |
| Hippoglossoides robustus 286 | 30.3 | 31.3 | 22.2 | 16.2 | 1095 |
| Hippoglossoides robustus 288 | 30.6 | 31.1 | 22.1 | 16.3 | 1095 |
| Hippoglossoides robustus 289 | 30.6 | 31.1 | 22.2 | 16.2 | 1095 |
| Hippoglossoides robustus 31 | 30.4 | 31.3 | 22.1 | 16.2 | 1095 |
| Limanda sakhalinensis 71 | 30.8 | 30.1 | 23.4 | 15.7 | 1095 |
| Limanda sakhalinensis 72 | 30.8 | 30.2 | 23.3 | 15.7 | 1095 |
| Liopsetta pinnifasciata 13 | 29.9 | 30.9 | 22.9 | 16.3 | 1095 |
| Liopsetta pinnifasciata 19-07 | 29.7 | 31.0 | 22.9 | 16.4 | 1095 |
| Liopsetta pinnifasciata 20-07 | 29.5 | 31.3 | 23.1 | 16.1 | 1095 |
| Liopsetta pinnifasciata 2K | 29.8 | 30.9 | 23.0 | 16.3 | 1095 |
| Myzopsetta punctatissima 37 | 30.8 | 29.8 | 22.0 | 17.4 | 1095 |
| Myzopsetta punctatissima 86-07 | 30.8 | 29.9 | 22.1 | 17.3 | 1095 |
| Platichthys stellatus 18-07 | 30.0 | 30.6 | 22.6 | 16.7 | 1095 |
| Platichthys stellatus 4K | 29.8 | 30.8 | 22.9 | 16.5 | 1095 |
| Pseudopleuronectes herzensteini 05-07 | 29.9 | 31.4 | 21.4 | 17.4 | 1095 |
| Pseudopleuronectes herzensteini 6K | 30.0 | 31.0 | 21.6 | 17.4 | 1095 |
| Pseudopleuronectes herzensteini 7K | 30.0 | 31.2 | 21.4 | 17.4 | 1095 |
| Pseudopleuronectes obscurus 09-07 | 30.5 | 30.1 | 22.6 | 16.7 | 1095 |
| Pseudopleuronectes obscurus 45 | 30.4 | 30.3 | 22.2 | 17.1 | 1095 |
| Pseudopleuronectes obscurus 46 | 30.4 | 30.1 | 22.6 | 16.9 | 1095 |
| Среднее значение | 30.29 ± 0.1 | 30.77 ± 0.1 | 22.42 ± 0.1 | 16.52 ± 0.1 | – |
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Cooper J.A., Chapleau F. Monophyly and intrarelationships of the family Pleuronectidae (Pleuronectiformes), with a revised classification // Fishery Bull. 1998. V. 96. № 4. P. 686–726.
Keast A., Chapleau F. A phylogenetic reassessment of the monophyletic status of the family Soleidae, with comments on the suborder Soleoidei (Pisces; Pleuronectiformes) // Canad. J. Zool. 1988. V. 66. № 12. P. 2797–2810. https://doi.org/10.1139/z88-408
Berendzen P.B., Dimmick W.W. Phylogenetic relationships of Pleuronectiformes based on molecular evidence // Copeia. 2002. V. 3. P. 642–52. https://doi.org/10.1643/0045-8511(2002)002[0642: PROPBO]2.0.CO;2
Kartavtsev Y.P., Park T.-J., Vinnikov K.A. et al. Cytochrome b (Cyt-b) gene sequence analysis in six flatfish species (Teleostei, Pleuronectidae), with phylogenetic and taxonomic insights // Marine Biol. 2007. V. 152. № 4. P. 757–773. https://doi.org/10.1007/s00227-007-0726-9
Kartavtsev Y.P., Sharina S.N., Goto T. et al. Cytochrome oxidase 1 gene sequence analysis in six flatfish species (Teleostei, Pleuronectidae) of Far East Russia with inferences in phylogeny and taxonomy // Mitochondrial DNA. 2008. V. 19. P. 479–489. https://doi.org/10.1080/19401730802570934
Pardo B.G., Machordom A., Foresti F. et al. Phylogenetic analysis of flatfish (order Pleuronectiformes) based on mitochondrial 16S rDNA sequences // Scientia Marina. 2005. V. 69. № 4. P. 531–543.
Шарина С.Н., Картавцев Ю.Ф. Филогенетический анализ камбал (Teleostei, Pleuronectiformes), основанный на исследовании нуклеотидных последовательностей гена цитохромоксидазы 1 (Co-1) // Генетика. 2010. Т. 46. № 3. С. 401–407.
Betancur R.R., Munroe T.A., Ortí G. et al. Addressing gene tree discordance and non-stationarity to resolve a multi-locus phylogeny of the Flatfishes (Teleostei: Pleuronectiformes) // Syst. Biol. 2013. V. 62. P. 763–785. https://doi.org/10.1093/sysbio/syt039
Betancur R.R., Ortí G. Molecular evidence for the monophyly of flatfishes (Carangimorpharia, Pleuronectiformes) // Mol. Phylogenet. and Evol. 2014. V. 73. P. 18–22. https://doi.org/10.1016/j.ympev.2014.01.006
Kartavtsev Y.Ph., Sharina S.N., Saitoh K. et al. Phylogenetic relationships of Russian Far Eastern Flatfish (Pleuronectiformes, Pleuronectidae) based on two mitochondrial gene sequences, Co-1 and Cyt-b, with inferences in order phylogeny using complete mitogenome data // Mitochondrial DNA. 2014. V. 27. № 1. P. 667–678. https://doi.org/10.3109/19401736.2014.913139
Norman I.R. A systematic monograph of the flatfishes (Heterosomata). Volume I. Psettodidae, Bothidae, Pleuronectidae. London: British Museum, 1934. 459 p.
Sakamoto K. Interrelationships of the family Pleuronectidae (Pisces: Pleuronectiformes) // Memoirs of the Fac. of Fisheries, Hokkaido University. 1984. V. 31. P. 95–215.
Lindberg G.U., Fedorov V.V. Fishes of Japan Sea and nearby parts of Okhotsk and Yellow seas. Part 6. Teleostomi. Osteichthyes. Actinopterigii. XXXI. Pleuronectiformes. Sankt-Petersburg Univ. Press. Sankt-Petersburg. 1993. 272 p.
Chapleau F. Pleuronectiform relationships: A cladistic reassessment // Bull. of Marine Sci. 1993. V. 52. № 1. P. 516–540.
Vernau O., Moreau C., Catzeflis F.M., Renaud F. Phylogeny of flatfishes (Pleuronectiformes): Comparisons and contradictions of molecular and morpho-anatomical data // J. Fish Biol. 1994. V. 45. № 4. P. 685–696. https://doi.org/10.1111/j.1095-8649.1994.tb00934.x
Keith P., Lord C., Lorion J. et al. Phylogeny and biogeography of Sicydiinae (Teleostei: Gobiidae) inferred from mitochondrial and nuclear genes // Marine Biol. 2011. V. 158. P. 311–326. https://doi.org/10.1007/s00227-010-1560-z
Kearse M., Moir R., Wilson A. et al. Geneious Basic: An integrated and extendable desktop software platform for the organization and analysis of sequence data // Bioinformatics. 2012. V. 28. № 12. P. 1647–1649. https://doi.org/10.1093/bioinformatics/bts199
Tamura K., Peterson D., Peterson N. et al. MEGA5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods // Mol. Biol. and Evol. 2011. V. 28. № 10. P. 2731–2739. https://doi.org/10.1093/molbev/msr121
Thompson J.D., Higgins D.G., Gibson T.J. CLUSTAL W: Improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice // Nucl. Ac. Res. 1994. V. 22. № 22. P. 4673–4680. https://doi.org/10.1093/nar/22.22.4673
Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences // J. Mol. Evol. 1980. V. 16. P. 111–120. https://doi.org/10.1007/bf01731581
Jukes T.H., Cantor C.R. Evolution of protein molecules // Mammalian Protein Metabolism. Volume III. N. Y.: Academic Press, 1969. P. 21–132. https://doi.org/10.1016/B978-1-4832-3211-9.50009-7
Hasegawa M., Kishino H., Yano T. Dating of the human-ape splitting by a molecular clock of mitochondrial DNA // J. Mol. Evol. 1985. V. 22. P. 160–174. https://doi.org/10.1007/bf02101694
Huelsenbeck J.P., Ronquist F. MRBAYES: Bayesian inference of phylogenetic trees // Bioinformatics. 2001. V. 17. № 8. P. 754–755. https://doi.org/10.1093/bioinformatics/17.8.754
Ronquist F., Huelsenbeck J.P. MrBayes 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. 2003. V. 19. № 12. P. 1572–1574. https://doi.org/10.1093/bioinformatics/btg180
http://tree.bio.ed.ac.uk/software/figtree/
StatSoft, Inc. (1999). STATISTICA for Windows [Computer program manual]. Tulsa, OK: StatSoft, Inc. Available from: http://statsoft.ru/products/STATISTICA_Base/.
Ivankov V.N., Vinnikov K.A., Borisovets E.E., Kartavtsev Y.P. Taxonomic relations between Pseudopleuronectes yokohamae and P. schrenki // Abstract of Conference for Educated Students, Science and Education Centre. Vladivostok: Far Eastern University Press, 2002. P. 69–70.
Картавцев Ю.Ф., Свиридов В.В., Ханзава Н., Саcаки Т. Генетическая дивергенция видов дальневосточных красноперок рода Tribolodon (Pisces, Cyprinidae) и близких таксонов // Генетика. 2002. Т. 38. № 11. С. 1–14.
Винников К.А. О таксономическом статусе камбал Hippoglossoides elassodon и H. robustus (морфометрический анализ) // VI Региональная конференция по актуальным проблемам экологии, морской биологии и биотехнологии студентов, аспирантов, молодых преподавателей и сотрудников вузов и научных организаций Дальнего Востока России. Тезисы докладов. Владивосток: Изд-во Дальневосточного университета, 2003. С. 25–26.
Винников К.А., Иванков В.Н., Питрук Д.Л. Таксономический статус японской лиманды Pseudopleuronectes yokohamae и лиманды Шренка P. schrenki (Pleuronectidae, по: Cooper, Chapleau, 1998) // Вопр. ихтиологии. 2006. Т. 46. № 3. С. 316–325.
Vinnikov K.A., Thomson R.C., Munroe T.A. Revised classification of the righteye flounders (Teleostei: Pleuronectidae) based on multilocus phylogeny with complete taxon sampling // Mol. Phylogenet. and Evol. 2018. V. 125. P. 147–162. https://doi.org/10.1016/j.ympev.2018.03.014
Kim I.C., Kweon H.S., Kim Y.J. et al. The complete mitochondrial genome of the javeline goby Acanthogobius hasta (Perciformes, Gobiidae) and phylogenetic considerations // Gene. 2004 V. 336. № 2. P. 147–153. https://doi.org/10.1016/j.gene.2004.04.009
Nailor G.J., Collins T.M., Brown W.M. Hydrophobicity and phylogeny // Nature. 1995. V. 373. № 6515. P. 565–566. https://doi.org/10.1038/373565b0
Дополнительные материалы отсутствуют.


