Генетика, 2021, T. 57, № 4, стр. 384-402
Интрогрессивная гибридизация мягкой пшеницы: результаты и перспективы
И. Г. Адонина 1, *, Е. М. Тимонова 1, Е. А. Салина 1
1 Курчатовский геномный центр, Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
* E-mail: adonina@bionet.nsc.ru
Поступила в редакцию 06.05.2020
После доработки 29.07.2020
Принята к публикации 17.08.2020
Аннотация
Данный обзор посвящен интрогрессивной гибридизации мягкой пшеницы, процессу интеграции в ее геном генов других культурных или дикорастущих видов из вторичного и третичного генных пулов с целью увеличения генетического разнообразия этой важнейшей сельскохозяйственной культуры. Обобщены результаты, достигнутые в интрогрессивной гибридизации мягкой пшеницы за последние 20 лет, проанализировано распределение чужеродных транслокаций по субгеномам и по хромосомам мягкой пшеницы, обсуждаются возможности дальнейшего развития данного направления исследований и селекции, базирующейся на использовании чужеродного генетического материала.
Впервые термины “интрогрессия”, “интрогрессивная гибридизация” были использованы американским генетиком Андерсоном [1, 2]. Под чужеродной интрогрессией обычно понимают перенос в геном какого-либо биологического вида генетической информации от другого вида, а в селекции растений – это интеграция в геном культурных растений генов других культурных или дикорастущих видов. Интрогрессивная гибридизация играет значительную роль в эволюции растений. Поэтому всестороннее изучение данного явления – важное направление исследований в генетике растений. Однако прежде всего интрогрессивная гибридизация – это один из способов увеличения генетического разнообразия культурных растений и, в частности, такой важнейшей сельскохозяйственной культуры как мягкая, или хлебная пшеница (Triticum aestivum L., 2n = 6x = 42).
Дикие и культурные виды, родственные T. aestivum, условно можно отнести к нескольким генным пулам [3]. Виды, принадлежащие к первичному генному пулу, имеют геномы, гомологичные субгеномам A, B и D мягкой пшеницы. Эта группа включает, прежде всего, виды пшениц подрода Triticum [4]: T. urartu Thum. ex Gandil. (2n = 2x = 14; геном АuАu), относящийся к секции Urartu Dorof et A. Filat., чей геном наиболее близок субгеному A T. aestivum; 11 тетраплоидных видов секции Dicoccoides Flaksb. и гексаплоидные виды секции Triticum. Также к первичному генному пулу Фрибе с соавт. [3] отнесли диплоидные виды секции Monococcon Dum. подрода Boeoticum [4]: T. monococcum L., T. boeoticum Boiss., T. sinskajae A. Filat. et Kurk. (геном AbAb) и диплоидный вид рода Aegilops L. [5], Aegilops tauschii Coss. (2n = 2x = 14; геном DD), из секции Vertebrata Zhuk. Emend. Kihara. Перенос генов от этих видов может быть осуществлен путем прямой гибридизации, гомологичной рекомбинации, беккроссирования и отбора.
Вторичный генный пул включает полиплоидные виды пшениц и эгилопсы, имеющие, по крайней мере, один геном, гомеологичный субгеномам A, B, D T. aestivum. В эту группу, например, входят тетраплоидные виды секции Timopheevi Filat. et Dorof. [4]: T. timopheevii (Zhuk.) Zhuk. и T. araraticum Jakubz., диплоидные виды Aegilops L. секции Sitopsis (Jaub. & Spach.) Zhuk. [5], S-геном которых близок B-субгеному T. aestivum. Перенос генов от этих видов в мягкую пшеницу возможен за счет рекомбинации гомеологичных хромосом.
Виды, относящиеся к третичному генному пулу, считаются более отдаленными родственниками пшеницы. Хромосомы их геномов негомологичны хромосомам мягкой пшеницы. Перенос генов от этих видов затруднен и требует применения других стратегий.
В результате скрещивания мягкой пшеницы с видами вторичного и третичного генных пулов могут быть получены амфиплоиды (полный набор хромосом пшеницы и полный или частичный набор хромосом другого вида) и следующие типы линий: дополненные линии (полный набор хромосом пшеницы и дополнительная пара чужеродных хромосом), замещенные линии (пара хромосом пшеницы замещена на пару чужеродных хромосом), транслоцированные линии (одна или несколько хромосом пшеницы содержат чужеродные транслокации). Перенос генетического материала от отдаленных видов во многих случаях происходит поэтапно от дополненных и замещенных линий к линиям, несущим относительно небольшие транслокации (меньше размера плеча) с целевым геном. Стремление уменьшить размеры чужеродных транслокаций, насколько это возможно, связано с тем, что вместе с целевым геном зачастую переносится большое количество другого генетического материала, который может отрицательно влиять на хозяйственно ценные свойства пшеницы.
Первые попытки получения гибридов между пшеницей и ее дикими или культивируемыми видами-сородичами были предприняты в конце XIX в. [6]. Первоначально целью экспериментов по интрогрессивной гибридизации было изучение вопросов эволюции и происхождения видов, процесса спаривания хромосом в мейозе. Однако основной целью гибридизации являлось создание пшеницы с исключительными свойствами. В перспективе представлялось, что данный подход обеспечит гораздо более широкие возможности для обогащения генома пшеницы, чем традиционная селекция. Своего пика интрогрессивная гибридизация достигла во второй половине XX в. Этому способствовали развитие молекулярной генетики и цитогенетики, работы по секвенированию геномов злаков. Вначале проводилась только цитологическая характеристика интрогрессивных линий, которая ограничивалась описанием митотических хромосом и поведения хромосом в мейозе, а сам факт интрогрессии чужеродного генетического материала устанавливался по появлению новых признаков, например устойчивости к заболеванию.
Бурное развитие методов хромосомного анализа растений [7], таких как дифференциальное окрашивание, в частности C-бэндинг; различные модификации in situ гибридизации (GISH – геномная in situ гибридизация, FISH – флуоресцентная in situ гибридизация с использованием различных повторяющихся последовательностей ДНК в качестве зондов); микродиссекция и сортинг хромосом; геномное секвенирование; разработка разнообразных молекулярных маркеров; построение генетических карт, позволило полнее описывать геномную конституцию у межвидовых гибридов, идентифицировать чужеродные хромосомы и характеризовать транслокации. Все это привело к получению широкого разнообразия линий пшеницы с интрогрессиями различных типов от разных доноров.
Наш обзор – это обобщение данных по гибридизации мягкой пшеницы с родственными видами вторичного и третичного генных пулов за последние 20 лет. Мы проанализировали распределение чужеродных транслокаций по субгеномам и по хромосомам мягкой пшеницы. Мы также попытались оценить возможности дальнейшего развития интрогрессивной гибридизации пшеницы. Аналогичного обзора в русскоязычной литературе нами не найдено. Поэтому мы надеемся, что данный обзор будет интересен для специалистов в области генетики пшеницы и особенно для селекционеров.
ВИДЫ, ОТНЕСЕННЫЕ КО ВТОРИЧНОМУ ГЕННОМУ ПУЛУ, КАК ИСТОЧНИКИ НОВЫХ ГЕНОВ
Гибридизация мягкой пшеницы с T. timopheevii и T. araraticum
T. timopheevii и T. araraticum относятся к вторичному генному пулу. Субгеном At этих видов родственен субгеному A мягкой пшеницы, а субгеном G – субгеному B. Как следствие этого, для T. aestivum получен полный спектр замещенных линий с участием T. timopheevii: с замещением хромосом субгенома A на хромосомы субгенома AtT. timopheevii, хромосом субгенома B на хромосомы субгенома G, а также линии с замещением хромосом субгенома D на хромосомы субгеномов At или G [8]. Интрогрессии генетического материала T. timopheevii и T. araraticum в геном мягкой пшеницы в виде транслокаций представлены в табл. 1. По меньшей мере девять генов устойчивости к грибным заболеваниям перенесены от этих видов в геном T. aestivum (табл. 1): Lr18, Lr50, LrSelG12, LrTt1 – гены устойчивости к листовой ржавчине (Puccinia triticina Erikss.); Pm6, Pm27 – гены устойчивости к мучнистой росе (Blumeria graminis DC.); Sr36, Sr37, Sr40 – гены устойчивости к стеблевой ржавчине (Puccinia graminis Pers.).
Таблица 1.
Транслокации от T. timopheevii и T. araraticum в геноме мягкой пшеницы
| Хромосома | Транслокации | Хозяйственно ценные гены | Ссылки |
|---|---|---|---|
| 1A | T1AL-1AtL.1AtS | [9] | |
| 2A | 2AS-2AtS.2AtL-2AL | LrTt1 | [9] |
| 3A | T3AL.3AtL | [8] | |
| T3AL.3AtS | [8] | ||
| 7A | T7AL.7AtS | [8] | |
| T7AS.7AtL | [8] | ||
| 2B | T2BL.2GS | [8] | |
| T2B/2G | Sr36/Pm6, Lr50 | [3, 10] | |
| T2BS-2GS.2GL-1BL | [9] | ||
| T2BL/2GS (T. araraticum) | Sr40 | [3] | |
| 3B | T3BS.3GL | [8] | |
| T3BS.3BL-3GL | LrSelG12 | [11] | |
| 4B | T4BS.4GL | [8] | |
| T4B/4G | Sr37 | [3] | |
| 5B | T5BS.5BL-5GL | Lr18 | [3] |
| T5BS.5GL | [12] | ||
| T5BS.5BL-5GL | LrTt1 | [9] | |
| 6B | T6BS.6GL | [9, 12] | |
| T6B/6G | Pm27 | [13] | |
| 7D | T7DS.7AtL | [8] |
Примечание (для табл. 1–8). Lr – гены устойчивости к листовой ржавчине (Puccinia triticina Erikss.); Sr – гены устойчивости к стеблевой ржавчине (Puccinia graminis Pers.); Pm – гены устойчивости к мучнистой росе (Blumeria graminis DC.).
Пшенично-эгилопсные интрогрессии. Интрогрессии от эгилопсов секции Sitopsis
Очень интенсивно в гибридизацию с мягкой пшеницей вовлекаются виды рода Aegilops. Это объясняется, с одной стороны, тем, что в трибе Triticeae род Aegilops является наиболее близким к роду Triticum. А с другой стороны, − эгилопсы характеризуются значительным генетическим полиморфизмом. Первенство по количеству полученных с их участием различных замещенных и транслоцированных линий пшеницы принадлежит эгилопсам секции Sitopsis (табл. 2), особенно выделяется Ae. speltoides, считающийся наиболее вероятным донором B-субгенома T. aestivum [32]. Значительное число генов устойчивости к различным болезням и вредителям передано пшенице от эгилопсов этой секции (табл. 2).
Таблица 2.
Транслокации и замещения хромосом, полученные с участием видов Aegilops секции Sitopsis
| Хромосома | Транслокации, замещения | Хозяйственно ценные гены (признаки) | Ссылки |
|---|---|---|---|
| 1A | T1AS.1AL-1SL | Lr51 | [14] |
| 2A | T2AL.2SL-2SS | Sr32 | [3] |
| 3A | T3AL.3AS-3SS | [15] | |
| T3A-3S | Lr66 | [16] | |
| T3AS.3SL | [17] | ||
| T3AL.3SS | [17] | ||
| T3AL.3SsS (Ae. searsii) | Sr51 | [18] | |
| 4A | T4AS-4AL-7SS | Lr28 | [3] |
| 6A | T6AL.6SS | Pm12 | [17] |
| T6AL-6SshL.6SshS (Ae. sharonensis) | Lr56/Yr38 | [19] | |
| 7A | T7AS-7SS.7SL | Gb5, Lr47 | [3] |
| T7AS-7SS-7AS.7AL | Lr47 | [20] | |
| T7AS.7AL-7SL-7AL | Lr47 | [20] | |
| 1B | 1S(1B) | [21] | |
| T1BL.1BS-1SS | [22, 23] | ||
| T1BS.1BL-1SL | Lr51 | [14] | |
| T1BL.1SS | Pm32 | [24] | |
| T1BS.1SL | [17] | ||
| 1BL.1SlS (Ae. longissima) | [25] | ||
| 2B | T2BL/2SS | Sr32 | [3] |
| T2B/2S | Lr35/Sr39 | [3] | |
| T2BL-2SL.2SS | Sr47 | [26] | |
| T2BL.2SS | [17] | ||
| T2BS.2SL | [17] | ||
| T2BS.2BL-2S | Pm57 | [27] | |
| 3B | T3BL.3BS-3SS (Ae. longissima) | Pm13 | [3] |
| T3BL.3SsS (Ae. searsii) | Sr51 | [18] | |
| 4B | T4BL.4SS | [17] | |
| 5B | T5BS.5BL-5SL | LrAsp5 | [22] |
| T5BL.5SS | [17] | ||
| T5BS.5SL | [17] | ||
| T5BS.5BL-5SL | [21] | ||
| T5BS/5SL | Pm53 | [28] | |
| 6B | T6BS.6BL-6SL | [22] | |
| T6BS-6SS.6SL | Pm12 | [29] | |
| T6BS | Lr36 | [30] | |
| T6BS.6SL | [17] | ||
| T6BL.6BS-6SS | [21] | ||
| T6B (Ae. sharonensis) | (устойчивость к листовой и стеблевой ржавчинам) | [31] | |
| 7B | T7BL.7SS | [17] | |
| T7BS.7SL | [17] | ||
| 1D | T1DS.1DL-1SL | [21] | |
| 2D | T2DL-2SL.2SS | Sr32 | [3] |
| T2DS.2SL | LrSp2 | [12] | |
| T2DS.2DL-2SL | [21] | ||
| 3D | T3DS.3DL-3SL | [21] | |
| T3DL.3DS-3SlS (Ae. longissima) | Pm13 | [3] | |
| T3DL.3SsS (Ae. searsii) | Sr51 | [18] | |
| T3DS-3SsS.3SsL (Ae. searsii) | Sr51 | [18] | |
| 4D | 4S(4D) | [21] | |
| 5D | T5DS.5SL | [17] | |
| 7D | 7S(7D) | [15] | |
| T7SS.7SL-7DL | [23] | ||
| T7DL-7SL.7SS | [21] |
ВИДЫ, ОТНЕСЕННЫЕ К ТРЕТИЧНОМУ ГЕННОМУ ПУЛУ, КАК ИСТОЧНИКИ НОВЫХ ГЕНОВ
Интрогрессии в геном мягкой пшеницы от эгилопсов, не относящихся к секции Sitopsis
Интрогрессии от эгилопсов, не относящихся к секции Sitopsis, представлены в табл. 3.
Таблица 3.
Транслокации и замещения хромосом, полученные с участием разных видов Aegilops
| Хромосома | Транслокации, замещения | Вид | Хозяйственно ценные гены (признак) | Ссылки |
|---|---|---|---|---|
| 1A | T1AS-1AepL | Ae. peregrina | Lr59 | [33] |
| 2A | T2AS-2ML.2MS | Ae. comosa | Yr8/Sr34 | [3] |
| T2AS | Ae. ventricosa | Cre5 | [34] | |
| T2AL.2AS-2NvS | Ae. ventricosa | Rkn3, Yr17, Lr37, Sr38, Cre5; (устойчивость к пирикуляриозу) | [35, 43, 44] | |
| 5A | 5Nv(5A) | Ae. ventricosa | Cre6 | [36] |
| 6A | T6AL-6AenL.6AenS | Ae. neglecta | Lr62 | [37] |
| T6AL-6AenL.6AenS | Ae. neglecta | Yr42, Lr62 | [37] | |
| 1B | 1BS.1BL-2Sv | Ae. peregrina | CreX | [45] |
| 2B | 2U(2B) | Ae. umbellulata | PmY39 | [38] |
| T2BS.2BL-2UtL | Ae. triuncialis | Lr58 | [39] | |
| 3B | T3BS.3BL-3Sv | Ae. peregrina | CreY, Rkn2 | [45] |
| 4B | T4BL.4BS-6UL | Ae. umbellulata | Lr9 | [3] |
| 6B | T6BS.6BL-6UL | Ae. umbellulata | Lr9 | [3] |
| T6BL.6BS-6UL | Ae. umbellulata | Lr9 | [3] | |
| 7B | T7BL.7BS-6UL | Ae. umbellulata | Lr9 | [3] |
| 2D | T2DS.2DL-6UL | Ae. umbellulata | Lr9 | [3] |
| T2DS.2DL-2UL | Ae. umbellulata | [21] | ||
| T2DS-2ML.2MS | Ae. comosa | Yr8/Sr34 | [3] | |
| T2DL | Ae. kotschyi | Yr37/Lr54 | [40] | |
| 5D | T5DL.5DS-5MgS | Ae. geniculata | Lr57, Yr40 | [41] |
| T5DL-5MgL.5MgS | Ae. geniculata | Sr53 | [42] | |
| T5DL.5DS-5US | Ae. umbellulata | Lr76, Yr70 | [46] | |
| 7D | 7Nv(7D) | Ae. ventricosa | Cre2, Pch1 | [36, 43] |
| T7DL.7DS-7US | Ae. umbellulata | [21] | ||
| T7DS | Ae. geniculata | Pm29 | [47] |
Примечание. Yr – гены устойчивости к полосатой (желтой) ржавчине (P. striiformis); Cre – гены устойчивости к корневой нематоде (Heterodera avenae Woll.); Rkn – гены устойчивости к корневой нематоде (Meloidogyne spp.); Pch – гены устойчивости к церкоспореллезу (глазковая пятнистость) (Tapesia yallundae Wallwork & Spooner).
Стоит отметить, что интрогрессивные линии, полученные с участием видов Aegilops, очень разнообразны и зачастую характеризуются уникальными признаками, которых нет у пшеницы. И даже если в настоящее время эти линии не так интенсивно используются, их качества могут быть оценены в будущем. Так, например, Ae. speltiodes стал источником генов устойчивости (Sr32 и Sr39) к расе стеблевой ржавчины Ug99, появившейся в 1999 г. [48–50]. Получение транслокации T2AL.2AS-2NvS от Ae. ventricosa и внедрение ее в селекционные программы позволило остановить распространение пирикуляриоза в странах Азии, в частности в Бангладеш [44, 51]. Интересным фактом является то, что значительный процент линий мягкой пшеницы, вовлеченных в селекционные программы CIMMYT, содержит сегмент 2NvS, который помимо устойчивости к пирикуляриозу обеспечивает повышенное содержание цинка в зерне [43, 52, 53]. Возможно, транслокация T2AL.2AS-2NvS окажет значительное влияние на селекцию и производство пшеницы, как это произошло с пшенично-ржаной транслокацией 1BL.1RS.
Интрогрессии в геном мягкой пшеницы от ржи (Secale L.)
Из 14 хромосомных плеч ржи только пять (1RS, 1RL, 2RL, 3RS, и 5RS) коллинеарны пшеничным, остальные перестроены [54, 55]. Поэтому транслокации с участием других хромосом были получены в основном с использованием ионизирующего облучения (табл. 4). До настоящего времени наиболее успешными в плане использования в селекции являются транслокации 1AL.1RS и 1BL.1RS.
Таблица 4.
Транслокации от ржи в геноме мягкой пшеницы
| Хромосома | Транслокации | Хозяйственно ценные гены (признаки) | Ссылки |
|---|---|---|---|
| 1A | T1AL-1RS | Gb2/Pm17 | [3] |
| T1AL.1RS | Gb6 | [3] | |
| 2A | T2AL-2AS-1RS | Gb6 | [3] |
| T2AS-1RS-1RL | Gb6 | [3] | |
| T2AS-2RS.2RL | Lr45 | [3] | |
| 3A | T3AS.3RS | Sr27 | [3] |
| T3AL-3RS | Sr27 | [3] | |
| 4A | T4AS.4AL-6RL-4AL | H25 | [3] |
| 6A | T6AL.6RS | Pm56 | [56] |
| 7А | T7AS.4RS | [57] | |
| 1B | T1BL.1RS | Pm8/Sr31, Lr26/Y49; PmCn17; YrCN17, Sr50 (ранее SrR), Sr1RSAmigo | [3, 58–60] |
| T1BL.1RS (S. africanum) | Yr9 | [61] | |
| 2B | T2BS-2RL | H21, (устойчивость к мучнистой росе и листовой ржавчине) | [3, 62] |
| 3B | T3BL.3RS | Sr27 | [3] |
| 4B | T4BS.4BL-2RL | Lr25/Pm7 | [3] |
| T4BS.4BL-6RL | H25 | [3] | |
| T4BL.4RL | (устойчивость к мучнистой росе) | [57] | |
| 6B | T6BS-6RL | Pm20, Cre10, Yr83 | [3, 63] |
| T6BS.6BL-6RL | H25 | [3] | |
| 2D | T2DS.2RL | Sr59 | [64] |
Интрогрессии в геном мягкой пшеницы от многолетних видов Triticeae
С участием многолетних видов трибы Triticeae получены дополненные, замещенные линии мягкой пшеницы, линии с различными транслокациями (табл. 5–8). Поскольку систематика многолетних видов трибы Triticeae достаточно сложна и продолжает изменяться с получением новой информации об организации геномов этих видов, мы решили придерживаться наиболее часто используемой в настоящее время номенклатуры [97]. Интерес к этим видам был связан главным образом с надеждой на создание многолетней пшеницы. В основном для реализации этой цели использовались различные виды пырея: пырей удлиненный (Thinopyrum elongatum), пырей бессарабский (Th. bessarabicum), пырей прутьевидный (Th. junceum), пырей средний (Th. intermedium), пырей понтийский (Th. ponticum). Наиболее широкое применение в качестве доноров полезных признаков получили виды: пырей средний (Th. intermedium, синоним Agropyron glaucum) и пырей понтийский (Th. ponticum). Однако многолетний цикл жизни – сложный признак, носит количественный характер. Оказалось, что гибридные формы становятся многолетними только если доля генома, полученного от многолетних родителей, значительна [97]. В последние годы появилась тенденция создания многолетних крупнозерных сортов пырея [98–100], однако пока такой подход вызывает много вопросов. Среди диких злаков пырей средний обладает наиболее крупным зерном. В США был создан сорт Kernza, урожайность которого составляет около 30% от урожайности мягкой пшеницы [98, 101]. В Омске создан крупнозёрный сорт пырея Сова, с урожайностью 11–12 центнеров зерна с гектара (Шаманин, неопубликованная информация).
Таблица 5.
Транслокации от Thinopyrum elongatum в геноме мягкой пшеницы
| Хромосома | Транслокации | Хозяйственно ценные гены (признаки) | Ссылки |
|---|---|---|---|
| 1A | T1AS.1EL | [65] | |
| 3A | T3AS.3AL-3EL | (солеустойчивость) | [66] |
| 6A | T6AS.6AL-6EL | Sr26, SrB | [3, 67] |
| 1B | T1BL.1BS-3EL | Sr24/Lr24 | [3] |
| 5B | T5BL.6ES | Cmc2 | [3] |
| 3D | T3DS-3DL-3EL | Sr24/Lr24 | [3] |
| 4D | T4DS.4DL-1EL | (WSMR) | [3] |
| 6D | T6DS | [68] | |
| 7D | T7DS.7DL-7EL-7DL | [3] | |
| T7DL-7EL.7ES | Lr19/Sr25/Sr43 | [3] | |
| T7DS.7DL-7EL | Lr19/Sr25/Sr43 | [3] | |
| T7DS-7EL | Lr19/Sr25/Sr43 | [3] |
Таблица 6.
Транслокации и замещения хромосом, полученные с участием Thinopyrum intermedium
| Хромосома | Транслокации, замещения | Хозяйственно ценные гены (признаки) | Ссылка |
|---|---|---|---|
| 2A | T2AS.2AL-7AiL | Lr38 | [3] |
| 5A | T5AL.5AS-7AiL | Lr38 | [3] |
| 7A | T7AL.7AS-7AiS | [69] | |
| 1B | T1BS-7AiS.7AiL | (BYDR) | [3] |
| T1BL.1BS-3Ai | YrCH-1BS | [70] | |
| 4B | T4BL | Yr50 | [71] |
| 6B | T6BS.6AiL | (устойчивость к мучнистой росе и полосатой ржавчине) | [72] |
| 7B | T7BS | Pm40 | [73] |
| 1D | T1DS.1DL-7AiL | Lr38 | [3] |
| 1St(1D) | (устойчивость к полосатой ржавчине) | [74] | |
| 2D | T2DL | Pm43 | [75] |
| 3D | T3DL.3DS-7AiL | Lr38 | [3] |
| 4D | T4DL.4AiS | (WSMR) | [3] |
| 6D | T6DS.6DL-7AiL | Lr38 | [3] |
| 6Ai(6D) | Lr6Ai#2 | [76] | |
| 7D | T7DS-7AiL.7AiS | Sr, (BYDR) | [3] |
| T7DS-7AiS.7AiL | (BYDR) | [3] | |
| 7DS.7DL-7EL | Bdv3 | [77] | |
| 7E(7D) | Bdv3 | [78] | |
| T7DS-7AiL.7AiS 7AiL | Sr44 | [3] | |
| 7DL.7AiS | Sr44 | [79] |
Таблица 7.
Транслокации от Dasypyrum villosum в геноме мягкой пшеницы
| Хромосома | Транслокации | Хозяйственно ценные гены (признак) | Ссылки |
|---|---|---|---|
| 5A | T5AL.6VS | Pm55 | [80] |
| 6A | T6AL.6VS | Pm21 | [81, 82] |
| T6AS.6VL | CreV, Sr52, Pm5 | [81, 83, 84] | |
| 1B | T1BL.1VS | [80] | |
| 2B | T2BS.2VS | Pm62 | [85] |
| 6B | T6BS.6BL-2VS | [86] | |
| 1D | T1DL.1VS | [81] | |
| T1DL.1VL | [80] | ||
| 2D | T2DL.2VS | [87] | |
| T2DS.2VL | [80] | ||
| 3D | T3DS.2VL | [86] | |
| T3DL.3VS | [81] | ||
| T3DS.3VL | [81] | ||
| 4D | T4DL.4VS | (WSSMVR) | [81, 88] |
| T4DS.4VL | [81] | ||
| 5D | T5DL.5VS | Pm55 | [80, 81] |
| 6D | T6DL.6VS | Pm21 | [89] |
| 7D | T7DL.2VS | [86] | |
| T7DL.7VS | [81] | ||
| T7DS.7VL | [81] |
Таблица 8.
Транслокации и замещение хромосом, полученные с участием разных многолетних видов
| Хромосома | Транслокации | Вид | Хозяйственно ценные гены (признаки) | Ссылки |
|---|---|---|---|---|
| 2A | T2AL | Thinopyrum ponticum | [68] | |
| T2AS | Th. ponticum | Yr69 | [90] | |
| 7A | T7AL.7LrS | Leymusracemosus | Fhb3 | [91] |
| 1B | T1BL | Th. ponticum | [68] | |
| 2B | T2BL.2BS-2JS | Th. bessarabicum | [92] | |
| 3B | T3BL.3BS-3NsS | Psathyrostachys huashanica | (устойчивость к полосатой ржавчине) | [93] |
| 4B | T4BS.4BL-7LrS | L. racemosus | Fhb3 | [91] |
| T4BL-7LrS.5LrS | L. racemosus | [91] | ||
| 5B | T5BS | Th. ponticum | [68] | |
| 6B | T6BS.6BL-7LrS | L. racemosus | Fhb3 | [91] |
| T6BL.5LrS | L. racemosus | [91] | ||
| 1D | T1DL | Th. ponticum | [68] | |
| 2D | T2DL | Th. ponticum | (гены карликовости) | [68, 94] |
| 6D | 6P(6D) | A. cristatum | [95] | |
| 7D | T7DS.7DL-7AeL | Th. distichum | Lr19/Sr25 | [3] |
| T7DL | Th. ponticum | Fhb7 | [96] |
Интрогрессии в геном мягкой пшеницы от ячменя (Hordeum vulgare L.)
Ячмень имеет много характеристик, которые могут быть полезны для пшеницы, например раннеспелость, повышенное содержание β-глюканов в зерне, благоприятный аминокислотный состав, устойчивость к засолению и засухе и т.д. Однако одна из основных проблем, возникающих при попытке получения гибридов пшеницы и ячменя, – ядерно-цитоплазматическая несовместимость.
Долгое время изучение пшенично-ячменных гибридов шло по пути создания дополненных и замещенных линий [102]. Затем были получены рекомбинантные интрогрессивные линии пшеницы с транслокациями целых плеч и сегментов хромосом, а также серии линий с фрагментами разного размера от хромосом 1H, 3H, 4H, 5H, 6H, 7H [102–107]. Набор таких линий позволил провести физическое и генетическое картирование хромосом ячменя, однако в селекционных программах большая часть полученных линий не используется.
ОСОБЕННОСТИ ИНТРОГРЕССИВНОЙ ГИБРИДИЗАЦИИ МЯГКОЙ ПШЕНИЦЫ
С целью выявления возможных закономерностей в интрогрессивной гибридизации мягкой пшеницы мы проанализировали данные за период с 1996 г. по 2018 г. (табл. 1–8). Результаты проведенного анализа показывают, что больше всего чужеродных транслокаций, полученных с участием видов, относящихся к вторичному генному пулу, выявляется в хромосомах субгенома B (рис. 1,а), а с участием видов третичного генного пула – в хромосомах субгенома D (рис. 1,б). Это в первую очередь объясняется тем, что геномы соответствующих видов наиболее близки к субгеномам пшеницы B и D. Наименьшая частота чужеродных интрогрессий наблюдается по хромосомам 4А, 5А (рис. 2). Одной из причин этого может быть наличие в геноме мягкой пшеницы транслокации 4AL/5AL/7BS, а также перицентрической и парацентрической инверсий хромосомы 4A [108, 109], перестроек, произошедших в эволюционной истории пшениц на уровне тетраплоидного предшественника гексаплоидной пшеницы. Значительное число транслокаций, полученных с участием видов, относящихся к вторичному генному пулу, в хромосомах 2B и 6B (рис. 2,а) объясняется, скорее всего, тем, что виды Aegilops секции Sitopsis, принадлежащие к данному генному пулу и являющиеся донорами большего числа из этих транслокаций, несут в соответствующих гомеологичных хромосомах гаметоцидные гены, имеющие преимущество при передаче потомству [110].
Рис. 1.
Процентное распределение чужеродных транслокаций по субгеномам мягкой пшеницы: (а) транслокации от видов вторичного пула; (б) транслокации от видов третичного пула.
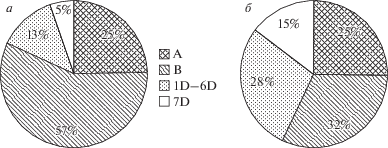
Рис. 2.
Количественное распределение чужеродных транслокаций по хромосомам мягкой пшеницы: (а) транслокации от видов вторичного пула; (б) транслокации от видов третичного пула.
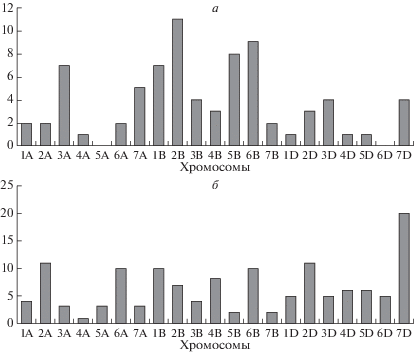
При обсуждении закономерностей интрогрессивной гибридизации мягкой пшеницы важно учитывать тот факт, что чаще всего проводится анализ гибридных линий, полученных селекционерами. Это значит, что исследуемые линии уже были отобраны по каким-то характеристикам. Наиболее часто такой характеристикой является устойчивость к биотическому стрессу. Многие чужеродные интрогрессии, представленные в табл. 1–8, выявлены именно в таких селекционных линиях. При анализе карты геномного распределения семейств генов пшеницы [111] можно заметить определенную положительную корреляцию между участками локализации генов (генных семейств), связанных с адаптацией к биотическому стрессу и распределением чужеродных интрогрессий. По-видимому, это связано с коллинеарностью хромосом пшеницы и хромосом родственных видов. Так, например, гены, связанные с адаптацией к биотическому стрессу, в хромосоме 1B сконцентрированы главным образом в ее коротком плече. Транслокации по этой хромосоме также в основном находятся в плече 1BS. Хромосома 4В обеднена генами устойчивости, почти все транслокации по этой хромосоме с генами устойчивости получены с использованием облучения и представляют собой фрагменты негомологичных хромосом других видов.
Среди всех хромосом по числу интрогрессий выделяется хромосома 7D (рис. 2). Наибольшее количество различных интрогрессивных линий получено с участием именно этой хромосомы, особенно это касается видов третичного генного пула. Возможно, одной из причин значительного числа замещений хромосомы 7D и наличия чужеродных транслокаций в хромосоме 7D является то, что хромосомы гомеологичной группы 7, кроме хромосомы 7В, играют незначительную роль в определении жизнеспособности и фертильности мягкой пшеницы по сравнению с хромосомами других групп [112]. Следует отметить, что структурная организация хромосомы 7B значительно отличается от организации хромосом 7A и 7D из-за транслокации 4AL/5AL/7BS [108, 109]. По-видимому, с этим связано и то, что хромосома 7В характеризуется небольшим числом чужеродных интрогрессий (рис. 2).
Локализация и характеристики чужеродных транслокаций в значительной степени зависят от того, насколько геном донорного вида перестроен относительно генома пшеницы. Так, например, картирование хромосом T. timopheevii показало, что хромосомы 6At и 1G и дистальный район 3AtL значительно перестроены [113], и транслокации с участием этих хромосом у мягкой пшеницы не выявлены. Геном Ae. umbellulata сильно перестроен относительно генома пшеницы [114], поэтому практически все интрогрессивные линии с участием этого вида получены при использовании радиационного облучения.
ПРОБЛЕМЫ ИТРОГРЕССИВНОЙ ГИБРИДИЗАЦИИ МЯГКОЙ ПШЕНИЦЫ
Интрогрессивная гибридизация пшеницы сталкивается со многими проблемами.
• Получение интрогрессивных линий и в особенности создание сортов на их основе является долгим и сложным процессом, даже несмотря на развитие технологий с использованием молекулярных маркеров для ускорения селекции.
• Вместе с целевым геном зачастую переносится большое количество избыточного генетического материала, который может отрицательно влиять на хозяйственно ценные свойства пшеницы.
• Чем больше эволюционное расстояние между видами, тем сложнее получить гибриды.
• Для переноса в пшеницу генов от видов, относящихся к третичному генному пулу, часто используют ионизирующее излучение, которое вызывает случайные хромосомные разрывы, из-за чего могут возникать генетически несбалансированные транслокации между негомеологичными хромосомами (участками хромосом), в результате чего потеря собственных генов не компенсируется генами донора.
• Еще одной проблемой, особенно когда в роли доноров генетического материала выступают виды рода Aegilops, являются гаметоцидные гены. Они считаются агрономически негативными, поскольку нарушают жизнеспособность гамет. Однако, с другой стороны, способность гаметоцидных генов элиминировать гаметы, несущие нежелательный аллель, может использоваться для отбора, если они связаны с генами, контролирующими полезные признаки [115]. Кроме того, гаметоцидные гены, как и ионизирующее излучение, используются для индукции разрыва донорских хромосом, для стимуляции встройки чужеродных фрагментов в хромосомы пшеницы.
Существуют и другие проблемы, с которыми можно столкнуться при попытке улучшения свойств пшеницы с помощью интрогрессивной гибридизации. Это ядерно-цитоплазматическая несовместимость, изменение экспрессии чужеродного гена в новом генетическом окружении, возможная активация мобильных элементов [116]. В некоторых случаях чужеродные гены могут инактивироваться или не функционировать должным образом. Внесение чужеродного хроматина может изменять пространственную организацию генома и, как следствие, влиять на его функционирование. Следует отметить, что эти аспекты еще изучены совершенно недостаточно.
СОВРЕМЕННЫЙ СТАТУС ИНТРОГРЕССИВНОЙ ГИБРИДИЗАЦИИ ПШЕНИЦЫ
На настоящий момент получено значительное число разнообразных интрогрессивных линий. Большинство из них несут гены устойчивости к разным факторам биотического стресса. Однако в результате интрогрессивной гибридизации было создано лишь небольшое количество коммерческих сортов. Вероятно, лучшим примером успешности чужеродной интрогрессии является транслокация 1BL.1RS. Было подсчитано, что в период с 1991 по 1995 г. 45% из 505 коммерческих сортов мягкой пшеницы в 17 странах мира имели транслокацию 1BL.1RS, которая обеспечила увеличение урожайности зерна за счет устойчивости к ржавчинным заболеваниям, повышение устойчивости к стрессу, больший вес зерна [6].
В Мексике был создан сорт Oasis 89, несущий ген Lr19 от Th. ponticum, который оказался более урожайным, чем родительский сорт Yecora 70 даже при отсутствии поражения ржавчиной [6]. В Индии сорта с сочетанием генов Lr19 и Sr25 приобрели популярность не только из-за устойчивости к ржавчине, но и благодаря повышенной продуктивности [97]. Сорта Plainsman V, Plainsman IV, Encore и Frontiersman (США), созданные с участием Ae. ovata, характеризовались увеличением содержания белка [6]. И, наконец, еще один яркий пример − китайский сорт Xiaoyan 6, у которого хромосомы 2A и 7D содержат интрогрессии от Th. ponticum [97]. Благодаря устойчивости к болезням, экологической пластичности (высокая стабильность урожая в разных условиях) и хорошим хлебопекарным качествам Xiaoyan 6 выращивался в качестве основного сорта в провинции Шэньси в течение 16 лет (1980–1995 гг.). Кроме того, на основе Xiaoyan 6 в Китае было выведено более 50 новых сортов пшеницы. Эти сорта в свою очередь занимали более 20 млн га посевных площадей и увеличили производство зерна в общей сложности на 7.5 млн т.
В нашей стране тоже получен ряд успешных сортов мягкой пшеницы с чужеродными интрогрессиями, например:
• сорта селекции Самарского НИИСХ, Тулайковская 5, Тулайковская 10 и Тулайковская 100 с замещением хромосомы 6D на соответствующую хромосому пырея, 6Ai(6D), характеризующиеся устойчивостью к листовой ржавчине и мучнистой росе [76, 117];
• сорт Фишт (Краснодарский НИИСХ им. П.П. Лукьяненко), с транслокациями 5BS.5GL, 6BS.6GL от T. timopheevii и замещением хромосом 1D, 6D на соответствующие хромосомы Ae. tauschii [12];
• сорт Челяба 75 (Челябинский НИИСХ) с транслокацией 2DS.2SL от Ae. speltoides, устойчивый к стеблевой ржавчине [115].
ПЕРСПЕКТИВЫ ИНТРОГРЕССИВНОЙ ГИБРИДИЗАЦИИ
Каковы перспективы интрогрессивной гибридизации в XXI в.?
В первую очередь необходимо развивать теоретические знания. Почти ничего не известно о том, как геном пшеницы взаимодействует с интрогрессированными генами и как он влияет на их функцию. Последние достижения в областях геномики, транскриптомики, протеомики, эпигенетики, цитогенетики обещают дать необходимую информацию.
Важно сохранение уже созданных отдельными исследователями или селекционерами коллекций интрогрессивных линий и получение новых, поскольку потенциал многих родственных видов, например видов Aegilops, еще далеко не исчерпан.
До недавнего времени использование в селекции пшеницы потенциала отдаленных видов было недостаточно эффективным. Однако современные технологии генотипирования в сочетании со специфическими стратегиями скрещиваний значительно повысили результативность интрогрессивной гибридизации с участием видов вторичного и третичного генных пулов. Применение молекулярных маркеров и современных цитогенетических методов (FISH/GISH) позволяет обнаруживать, точно характеризовать перенесенные в пшеницу чужеродные фрагменты, а также отслеживать их судьбу в последующих скрещиваниях [43]. Особенно важно то, что в настоящее время с помощью молекулярных маркеров могут быть выявлены интерстициальные интрогрессии, не имеющие значимого фенотипического проявления [26]. Многие из последних работ направлены на уменьшение размеров фрагментов генетического материала отдаленных видов, которые были ранее перенесены в геном пшеницы или получены от новых скрещиваний. Это делается для снижения отрицательного влияния сопутствующего генетического материала, перенесенного вместе с полезными целевыми генами. Так, например, успешно проведены работы по уменьшению размеров транслокаций с целевыми генами устойчивости к вирусу полосатой мозаики Wsm1 и Wsm3 у ранее отобранных интрогрессивных линий пшеницы, у которых короткое плечо хромосомы 4D и длинное плечо хромосомы 7B были полностью замещены на плечи соответствующих гомеологичных хромосом Th. intermedium [118, 119]. Ниу с соавт. [50], используя мутантный (делеционный) локус ph1b, индуцировали рекомбинацию между хромосомами 2В мягкой пшеницы и 2S Ae. speltoides. В результате после скрининга популяции из 1048 растений было отобрано 40 рекомбинантных линий. Четыре из них содержали ген Sr39 с размером прилегающего фрагмента 2.87–9.15% хромосомы эгилопса. Занг с соавт. [120, 121] также, используя локус ph1b и генотипирование с помощью SNP-маркеров, продемонстрировали алгоритм, позволяющий стимулировать и идентифицировать гомеологичную рекомбинацию между хромосомой 2В мягкой пшеницы и хромосомами 2S Ae. speltoides (генотипировано 83 рекомбинантных линии) и 2E Th. elongatum (генотипировано 67 рекомбинантных линий). И, наконец, группой ученых под руководством Джулии Кинг была выполнена масштабная работа по получению интрогрессивных популяций для ряда видов злаков. На сегодняшний день ими получено и охарактеризовано с помощью SNP-маркеров 218 интрогрессий от Ambylopyrum mutica [122], 294 интрогрессированных фрагмента от Ae. speltoides [123], 276 интрогрессированных фрагментов от T. timopheevii [124] и 12 интрогрессированных фрагментов от Th. bessarabicum [125]. Хотя влияние большей части полученных фрагментов на фенотип еще не изучено, полученный материал, скорее всего, может оказаться полезным не только для решения задач селекции, но и в фундаментальных исследованиях. Например, созданные серии интрогрессивных линий позволяют строить физические и генетические карты высокого разрешения [43, 106, 123].
Помимо совершенствования технологий традиционной интрогрессивной гибридизации мягкой пшеницы все более актуальным становится использование новых оригинальных подходов в селекции растений. Так, например, применение в селекции синтетических гексаплоидных пшениц, полученных путем скрещивания различных тетраплоидных видов пшеницы с Ae. tauschii, позволяет использовать потенциал генетического разнообразия сразу двух видов [126]. Стабильные гексаплоидные синтетики затем успешно скрещиваются с сортами мягкой пшеницы. Перспективным методом внесения изменений в геном пшеницы является цисгенез – альтернатива трансгенезу, такая технология генетической модификации, при которой манипуляции происходят с использованием ДНК близкородственного вида, с которым возможен половой процесс [127]. В отличие от трансгенных такие растения не должны содержать репортерные и селективные гены, т.е. гены неродственных организмов и гены устойчивости к антибиотикам. Следует отметить, что все большие обороты в современном мире набирают технологии редактирования генома, особенно основанные на применении системы CRISPR/Cas9 [127].
В связи с расширением круга задач, усложнением методов интрогрессивной гибридизации мягкой пшеницы и других хозяйственно важных растений было даже предложено дать новое название этому направлению исследований – интрогрессиомика (introgressiomics) [128].
Таким образом, можно сделать вывод, что интрогрессивная гибридизация мягкой пшеницы имеет значительный потенциал дальнейшего развития и в XXI в.
Работа выполнена в рамках бюджетного проекта 0324-2019-0039-C-01, при финансовой поддержке гранта РФФИ № 20-016-00122.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Anderson E., Hubricht L. Hybridization in tradescantia. III. The evidence for introgressive hybridization // Amer. J. Bot. 1938. V. 25. P. 396–402.
Anderson E. Introgressive Hybridization. N.Y., 1949. 109 p.
Friebe B., Yiang J., Raupp W.J. et al. Characterization of wheat alien translocations conferring resistance to diseases and pests: current status // Euphytica. 1996. V. 91. P. 59–87.
Дорофеев В.Ф., Филатенко А.А., Мигушова Э.Ф. и др. Пшеница // Культурная флора СССР. Л.: Колос, 1979. Т. 1. С. 7–31.
van Slageren M.W. Wild Wheats: A Monograph of Aegilips L. and Amblyopyrum (Jaub., Spach.) Eig. (Poaceae). Wageningen Agricultural Univ., Intern. Center for Agricultural Research in the Dry Areas: Veenman Drukkers, Wageningen, 1994. 512 p.
Bedő Z., Láng L. Wheat breeding: Current status and bottlenecks // Alien Introgression in Wheat Cytogenetics, Molecular Biology, and Genomics / Eds Molnár-Láng M., Ceoloni C., Doležel J. Switzerland: Springer International Publ., 2015. P. 77–101. https://doi.org/10.1007/978-3-319-23494-6
Бадаева Е.Д., Салина Е.А. Структура генома и хромосомный анализ растений // Вавиловский журн. генетики и селекции. 2013. Т. 17. № 4/2. С. 1017–1043.
Бадаева Е.Д., Будашкина Е.Б., Билинская Е.Н., Пухальский В.А. Закономерности межгеномных замещений хромосом у межвидовых гибридов пшеницы и их использование для создания генетической номенклатуры хромосом Triticum timopheevii // Генетика. 2010. Т. 46. № 7. С. 869–886.
Leonova I., Budashkina E., Flath K. et al. Microsatellite mapping of a leaf rust resistance gene transferred to common wheat from Triticum timopheevii // Cereal Res. Commun. 2010. V. 38. P. 211–219. https://doi.org/10.1556/CRC.38.2010.2.7
Brown-Guedira G., Singh S., Fritz A. Performance and mapping of leaf rust resistance transferred to wheat from Triticum timopheevii subsp. armeniacum // Phytopathology. 2003. V. 93. P. 784–789. https://doi.org/10.1094/PHYTO.2003.93.7.784
Singh A.K., Sharma J.B., Singh P.K. et al. Genetics and mapping of a new leaf rust resistance gene in Triticum aestivum L. × Triticum timopheevii Zhuk. derivative ‘Selection G12’ // J. Genet. 2017. V. 96. P. 291–297.
Адонина И.Г., Леонова И.Н., Бадаева Е.Д., Салина Е.А. Генотипирование сортов мягкой пшеницы разных регионов России // Вавиловский журн. генетики и селекции. 2016. Т. 20. № 1. С. 44–50. https://doi.org/10.18699/VJ16.107
Jarve K., Peusha H.O., Tsymbalova J. et al. Chromosomal location of a T. timopheevii-derived powdery mildew resistance gene transferred to common wheat // Genome. 2000. V. 43. P. 377–381.https://doi.org/10.1139/g99-141
Helguera M., Vanzetti L., Soria M. et al. PCR markers for Triticum speltoides leaf rust resistance gene Lr51 and their use to develop isogenic hard red spring wheat lines // Crop Sci. 2005. V. 45. P. 728–734. https://doi.org/10.2135/cropsci2005.0728
Adonina I.G., Salina E.A., Efremova T.T., Pshenichnikova T.A. The study of introgressive lines of Triticum aestivum × Aegilops speltoides by in situ and SSR analyses // Plant Breed. 2004. V. 123. P. 220–224. https://doi.org/10.1111/j.1439-0523.2004.00932.x
Marais G.F., Bekker T.A., Eksteen A. et al. Attempts to remove gametocidal genes co-transferred to wheat with rust resistance from Aegilops speltoides // Euphytica. 2009. V. 171. P. 71–85. https://doi.org/10.1007/s10681-009-9996-2
Liu W., Koo D.-H., Friebe B., Gill B. A set of Triticum aestivum–Aegilops speltoides Robertsonian translocation lines // Theor. Appl. Genet. 2016. V. 129. P. 2359–2368. https://doi.org/10.1007/s00122-016-2774-3
Liu W.X., Jin Y., Rouse M. et al. Development and characterization of wheat–Ae. searsii Robertsonian translocations and a recombinant chromosome conferring resistance to stem rust // Theor. Appl. Genet. 2011. V. 122. P. 1537–1545. https://doi.org/10.1007/s00122-011-1553-4
Marais G.F., McCallum B., Marais A.S. Leaf rust and stripe rust resistance genes derived from Triticum sharonense // Euphytica. 2006. V. 149. P. 373–380.
Dubcovsky J., Lukaszewski A.J., Echaide M. et al. Molecular characterization of two Triticum speltoides interstitial translocations carrying leaf rust and green bug resistance genes // Crop Sci. 1998. V. 38. P. 1655–1660. https://doi.org/10.2135/cropsci1998.0011183X003800-060040x
Adonina I.G., Davoyan R.O., Bebyakina I.V. et al. Chromosomal rearrangements in wheat introgression lines obtained using synthetic genome substitution and genome addition forms: IV Межд. конф. “Генофонд и селекция растений” (GPB 2018). Новосибирск, 2018. С. 13–17.
Адонина И.Г., Петраш Н.В., Тимонова Е.М. и др. Создание и изучение устойчивых к листовой ржавчине линий мягкой пшеницы с транслокациями от Aegilops speltoides Tausch // Генетика. 2012. Т. 48. № 4. С. 488–494.
Салина Е.А., Егорова Е.М., Адонина И.Г. и др. ДНК-маркеры для генотипирования линий мягкой пшеницы (Triticum aestivum L.) с генетическим материалом Aegilops speltoides Tausch и Triticum timopheevii Zhuk. // Вестник ВОГиС. 2008. Т. 12. № 4. С. 620–628.
Hsam S.L.K., Lapochkina I.F., Zeller F.J. Chromosomal location of genes for powdery mildew resistance in common wheat (Triticum aestivum L. em Thell.). 8. Gene Pm32 in a wheat–Aegilops speltoides translocation line // Euphytica. 2003. V. 133. P. 367–370.
Wang K., Lin Z., Wang L. et al. Development of a set of PCR markers specific to Aegilops longissima chromosome arms and application in breeding a translocation line // Theor. Appl. Genet. 2018. V. 131. P. 13–25. https://doi.org/10.1007/s00122-017-2982-5
Klindworth D.L., Niu Z.X., Chao S.M. et al. Introgression and characterization of a goat grass gene for a high level of resistance to Ug99 stem rust in tetraploid wheat // Genes, Genomes Genet. 2012. V. 2. P. 665–673. https://doi.org/10.1534/g3.112.002386
Liu W., Koo D.-H., Xia Q. et al. Homoeologous recombination-based transfer and molecular cytogenetic mapping of powdery mildew-resistant gene Pm57 from Aegilops searsii into wheat // Theor. Appl. Genet. 2017. V. 130. P. 841–848. https://doi.org/10.1007/s00122-017-2855-y
Petersen S., Lyerly J.H., Worthington M.L. et al. Mapping of powdery mildew resistance gene Pm53 introgressed from Aegilops speltoides into soft red winter wheat // Theor. Appl. Genet. 2015. V. 128. P. 303–312. https://doi.org/10.1007/s00122-014-2430-8
Jia J., Devos K.M., Chao S. et al. RFLP-based maps of homoeologous group-6 chromosomes of wheat and their application in the tagging of Pm12, a powdery mildew resistance gene transferred from Aegilops speltoides to wheat // Theor. Appl. Genet. 1996. V. 92. P. 559–565.
Dvorak J., Knott D.R. Location of a Triticum speltoides chromosome segment conferring resistance to leaf rust in Triticum aestivum // Genome. 1990. V. 33. P. 892–897. https://doi.org/10.1139/g90-134
Millet E., Manisterski J., Ben-Yehuda P. et al. Introgression of leaf rust and stripe rust resistance from Sharon goat grass (Aegilops sharonensis Eig) into bread wheat (Triticum aestivum L.) // Genome. 2014. V. 57. P. 1–8. https://doi.org/10.1139/gen-2014-0004
El Baidouri M., Murat F., Veyssiere M. et al. Reconciling the evolutionary origin of bread wheat (Triticum aestivum) // New Phytologist. 2017. V. 213. P. 1477–1486.
Marais G.F., McCallum B., Marais A.S. Wheat leaf rust resistance gene Lr59 derived from Aegilops peregrine // Plant Breed. 2008. V. 127. P. 340–345. https://doi.org/10.1111/j.1439-0523.2008.01513.x
Jahier J., Abelard P., Tonguy A.M. et al. The Aegilops ventricosa segment on chromosome 2AS of the wheat cultivar ‘VPM1’ carries the cereal cyst nematode gene Cre5 // Plant Breed. 2001. V. 120. P. 125–128. https://doi.org/10.1046/j.1439-0523.2001.00585.x
Williamson V.M., Thomas V., Ferris H., Dubcovsky J. An Aegilops ventricosa translocation confers resistance against root-knot nematodes to common wheat // Crop Sci. 2013. V. 53. № 4. P. 1412–1418. https://doi.org/10.2135/cropsci2012.12.0681
Ogbonnaya F.C., Seah S., Delibes A. et al. Molecular-genetic characterization of a new nematode resistance gene in wheat // Theor. Appl. Genet. 2001. V. 102. P. 623–629.
Marais F., Marais A., McCallum B., Pretorius Z. Transfer of leaf rust and stripe rust resistance genes Lr62 and and Yr42 from Aegilops neglecta Req. ex Bertol. to common wheat // Crop Sci. 2009. V. 49. P. 871–879. https://doi.org/10.2135/cropsci2008.06.0317
Zhu Z., Zhou R., Kong X. et al. Microsatellite marker identification of a Triticum aestivum–Aegilops umbellulata substitution line with powdery mildew resistance // Euphytica. 2006. V. 150. P. 149–153. https://doi.org/10.1007/s10681-006-9103-x
Kuraparthy V., Sood S., Chhuneja P. et al. A cryptic wheat–Aegilops triuncialis translocation with leaf rust resistance gene Lr58 // Crop Sci. 2007. V. 47. P. 1995–2003. https://doi.org/10.2135/cropsci2007.01.0038
Marais G.F., McCallum B., Snyman J.E. et al. Leaf rust and stripe rust resistance genes Lr54 and Yr37 transferred to wheat from Aegilops kotschyi // Plant Breed. 2005. V. 124. P. 538–541. https://doi.org/10.1111/j.1439-0523.2005.01116.x
Kuraparthy V., Chunneja P., Dhaliwal H.S. et al. Characterization and mapping of cryptic alien introgression from Aegilops geniculata with new leaf rust and stripe rust resistance genes Lr57 and Yr40 in wheat // Theor. Appl. Genet. 2007. V. 114. P. 1379–1389. https://doi.org/10.1007/s00122-007-0524-2
McIntosh R.A., Yamazaki Y., Dubcovsky J. et al. Catalogue of Gene Symbols for Wheat. 2013. Available at: http://www.shigen.nig.ac.jp/wheat/komugi/genes/.
Kishii M. An update of recent use of Aegilops species in wheat breeding // Front. Plant Sci. 2019. V. 10. Article. 585. https://doi.org/10.3389/fpls.2019.00585
Cruz C.D., Peterson G.L., Bockus W.W. et al. The 2NS translocation from Aegilops ventricosa confers resistance to the Triticum pathotype of Magna portheoryzae // Crop Sci. 2016. V. 56. P. 990–1000. https://doi.org/10.2135/cropsci2015.07.0410
Coriton O., Barloy D., Huteau V. et al. Assignment of Aegilops variabilis Eig chromosomes and translocations carrying resistance to nematodes in wheat // Genome. 2009. V. 52. № 4. P. 338–346. https://doi.org/10.1139/G09-011
Bansal M., Kaur S., Dhaliwal H.S. et al. Mapping of Aegilops umbellulata-derived leaf rust and stripe rust resistance loci in wheat // Plant Pathol. 2017. V. 66. P. 38–44. https://doi.org/10.1111/ppa.12549
Zeller F.J., Kong L., Hartl L. et al. Chromosomal location of genes for resistance to powdery mildew in common wheat (Triticum aestivum L. em Thell.). 7. Gene Pm29 in line Pova // Euphytica. 2002. V. 123. № 2. P. 187–194.
Mago R., Zhang P., Bariana H.S. et al. Development of wheat lines carrying stem rust resistance gene Sr39 with reduced Aegilops speltoides chromatin and simple PCR markers for marker assisted selection // Theor. Appl. Genet. 2009. V. 119. P. 1441–1450. https://doi.org/10.1007/s00122-009-1146-7
Mago R., Verli D., Zhan P. et al. Development of wheat–Aegilops speltoides recombinants and simple PCR-based markers for Sr32 and a new stem rust resistance gene on the 2S#1 chromosome // Theor. Appl. Genet. 2013. V. 126. P. 2943–2955. https://doi.org/10.1007/s00122-013-2184-8
Niu Z., Klindworth D.L., Friesen T.L. et al. Targeted introgression of a wheat stem rust resistance gene by DNA marker assisted chromosome engineering // Genetics. 2011. V. 187. P. 1011–1021. https://doi.org/10.1534/genetics.110.123588
Mahmud H. Wheat blast in Bangladesh threatening South Asia wheat production // Acta Sci. Microbiol. 2019. V. 2. P. 8–9. https://doi.org/10.1371/journal.pone.0197555
Juliana P., Singh R.P., Singh P.K. et al. Genomic and pedigree-based prediction for leaf, stem, and stripe rust resistance in wheat // Theor. Appl. Genet. 2017. V. 130. P. 1415–1430. https://doi.org/10.1007/s00122-017-2897-1
Velu G., Crespo-Herrera L., Guzman C. et al. Assessing genetic diversity to breed competitive biofortified wheat with enhanced grain Zn and Fe concentrations // Front. Plant Sci. 2018. V. 9. Article 1971. https://doi.org/10.3389/fpls.2018.01971
Naranjo T., Fernandez-Rueda P. Homoeology of rye chromosome arms to wheat // Theor. Appl. Genet. 1991. V. 82. P. 577–586.
Devos K.M., Atkinson M.D., Chinoy C.N. et al. Chromosomal rearrangements in the rye genome relative to wheat // Theor. Appl. Genet. 1993. V. 85. P. 673–680.
Hao M., Liu M., Luo J. et al. Introgression of powdery mildew resistance gene Pm56 on rye chromosome arm 6RS into wheat // Front. Plant Sci. 2018. V. 9. Article 1040. https://doi.org/10.3389/fpls.2018.01040
Ma P., Han G., Zheng Q. et al. Development of novel wheat-rye chromosome 4R translocations and assignment of their powdery mildew resistance // Plant Dis. 2020. V. 104. № 1. P. 260–268.https://doi.org/10.1094/PDIS-01-19-0160-RE
Ren T.H., Yang Z.J., Yan B.J. et al. Development and characterization of a new 1BL.1RS translocation line with resistance to stripe rust and powdery mildew of wheat // Euphytica. 2009. V. 169. P. 207–313. https://doi.org/10.1007/s10681-009-9924-5
Luo P.G., Zhang H.Y., Shu K. et al. Stripe rust (Puccinia striiformis f. sp. tritici) resistance in wheat with the wheat-rye 1BL/1RS chromosomal translocation // Canad. J. Plant Pathol. 2007. V. 30. P. 1–6. https://doi.org/10.1080/07060661.2008.10540540
Mago R., Zhang P., Vautrin S. et al. The wheat Sr50 gene reveals rich diversity at a cereal disease resistance locus // Nat. Plants. 2015. V. 1. Article 15186. https://doi.org/10.1038/nplants.2015.186
Yang Z.J., Li G.R., Jia J.Q. et al. Molecular cytogenetic characterization of wheat–Secale africanum amphiploids and derived introgression lines with stripe rust resistance // Euphytica. 2009. V. 167. P. 197–202. https://doi.org/10.1007/s10681-008-9861-8
Hysing S.C., Hsam S.L.K., Singh R.P. et al. Agronomic performance and multiple disease resistance in T2BS.2RL wheat-rye translocation lines // Crop Sci. 2007. V. 47. P. 254–260. https://doi.org/10.2135/ cropsci2006.04.0269
Li J., Dundas I., Dong C. et al. Identification and characterization of a new stripe rust resistance gene Yr83 on rye chromosome 6R in wheat // Theor. Appl. Genet. 2020. V. 133. P. 1095–1107. https://doi.org/10.1007/s00122-020-03534-y
Rahmatov M., Rouse M.N., Nirmala J. et al. A new 2DS.2RL Robertsonian translocation transfers stem rust resistance gene Sr59 into wheat // Theor. Appl. Genet. 2016. V. 129. P. 1383–1392. https://doi.org/10.1007/s00122-016-2710-6
Kumar A., Garg M., Kaur N. et al. Rapid development and characterization of chromosome specific translocation line of Thinopyrum elongatum with improved dough strength // Front. Plant Sci. 2017. Article 1593. https://doi.org/10.3389/fpls.2017.01593
Mullan D.J., Mirzaghaderi G., Walker E. et al. Development of wheat–Lophopyrum elongatum recombinant lines for enhanced sodium ‘exclusion’ during salinity stress // Theor. Appl. Genet. 2009. V. 119. P. 1313–1323. https://doi.org/10.1007/s00122-009-1136-9
Mago R., Zhang P., Xia X. et al. Transfer of stem rust resistance gene SrB from Thinopyrum ponticum into wheat and development of a closely linked PCR‑based marker // Theor. Appl. Genet. 2019. V. 132. P. 371–382. https://doi.org/10.1007/s00122-018-3224-1
Wang J., Xiang F., Xia G. Agropyron elongatum chromatin localization on the wheat chromosomes in an introgression line // Planta. 2005. V. 221. P. 277–286. https://doi.org/10.1007/s00425-004-1443-y
Khan I.A. Molecular and agronomic characterization of wheat–Agropyron intermedium recombinant chromosomes // Plant Breed. 2000. V. 119. P. 25–29. https://doi.org/10.1046/j.1439-0523.2000.00440.x
Zheng X., Tang C., Han R. et al. Identification, characterization, and evaluation of novel stripe rust-resistant wheat–Thinopyrum intermedium chromosome translocation lines // Plant Disease Publ. Online. 2020. https://doi.org/10.1094/PDIS-01-19-0001-RE
Liu J., Chang Z.J., Zhang X.J. et al. Putative Thinopyrum intermedium-derived stripe rust resistance gene Yr50 maps on wheat chromosome arm 4BL // Theor. Appl. Genet. 2013. V. 126. P. 265–274. https://doi.org/10.1007/s00122-012-1979-3
Zhan H., Zhang X., Li G. et al. Molecular characterization of a new wheat–Thinopyrum intermedium translocation line with resistance to powdery mildew and stripe rust // Int. J. Mol. Sci. 2015. V. 16. P. 2162–2173. https://doi.org/10.3390/ijms16012162
Luo P.G., Luo H.Y., Chang Z.J. et al. Characterization and chromosomal location of Pm40 in common wheat: a new gene for resistance to powdery mildew derived from Elytrigia intermedium // Theor. Appl. Genet. 2009. V. 118. P. 1059–1064. https://doi.org/10.1007/s00122-009-0962-0
Hu L.-J., Li G.-R., Zeng Z.-X. et al. Molecular cytogenetic identification of a new wheat–Thinopyrum substitution line with stripe rust resistance // Euphytica. 2011. V. 177. P. 169–177. https://doi.org/10.1007/s10681-010-0216-x
He R.L., Chang Z.J., Yang Z.J. et al. Inheritance and mapping of powdery mildew resistance gene Pm43 introgressed from Thinopyrum intermedium into wheat // Theor. Appl. Genet. 2009. V. 118. P. 1173–1180. https://doi.org/10.1007/s00122-009-0971-z
Salina E.A., Adonina I.G., Badaeva E.D. et al. A Thinopyrum intermedium chromosome in bread wheat cultivars as a source of genes conferring resistance to fungal diseases // Euphytica. 2015. V. 204. P. 91–101. https://doi.org/10.1007/s10681-014-1344-5
Ohm H.W., Anderson J.M., Sharma H.C. et al. Registration of yellow dwarf virus resistant wheat germplasm line P961341 // Crop Sci. 2005. V. 45. P. 805–806. https://doi.org/10.2135/cropsci2005.0805
Sharma H.C., Ohm H.W., Perry K.L. Registration of barley yellow dwarf virus resistant wheat germplasm line P29 // Crop Sci. 1997. V. 37. P. 1032–1033. https://doi.org/10.2135/cropsci1997.0011183X003700-030096x
Liu W., Danilova T., Rouse M. et al. Development and characterization of a compensating wheat–Thinopyrum intermedium Robertsonian translocation with Sr44 resistance to stem rust (Ug99) // Theor. Appl. Genet. 2013. V. 126. P. 1167–1177. https://doi.org/10.1007/s00122-013-2044-6
Zhang R., Sun B., Chen J. et al. Pm55, a developmental-stage and tissue-specific powdery mildew resistance gene introgressed from Dasypyrum villosum into common wheat // Theor. Appl. Genet. 2016. V. 129. P. 1975–1984. https://doi.org/10.1007/s00122-016-2753-8
Liu C., Qi L.L., Liu W.X. et al. Development of a set of compensating Triticum aestivum–Dasypyrum villosum Robertsonian translocation lines // Genome. 2011. V. 54. P. 836–844. https://doi.org/10.1139/g11-051
Qi L.L., Chen P.D., Liu D.J. et al. The gene Pm21 – a new source of resistance to wheat powdery mildew // Acta Agriculture Sinica. 1995. V. 21. P. 257–261.
Zhang R., Feng Y., Li H. et al. Cereal cyst nematode resistance gene CreV effective against Heterodera filipjevi transferred from chromosome 6VL of Dasypyrum villosum to bread wheat // Mol. Breed. 2016. V. 36. Article 122. https://doi.org/10.1007/s11032-016-0549-9
Li H., Dong Z., Ma C. et al. Physical mapping of stem rust resistance gene Sr52 from Dasypyrum villosum based on ph1b-induced homoeologous recombination // Int. J. Mol. Sci. 2019. V. 20. № 19. Article 4887. https://doi.org/10.3390/ijms20194887
Zhang R., Fan Y., Kong L. et al. Pm62, an adult-plant powdery mildew resistance gene introgressed from Dasypyrum villosum chromosome arm 2VL into wheat // Theor. Appl. Genet. 2018. V. 131. P. 2613–2620. https://doi.org/10.1007/s00122-018-3176-5
Chen Q.Z., Cao A.Z., Qi Z.J. et al. Structural change of 2V chromosome of Haynaldia villosa induced by gametocidal chromosome 3C of Aegilops triuncialis // Agric. Sci. China. 2008. V. 7. P. 804–811. https://doi.org/10.1016/S1671-2927(08)60117-0
Zhang R., Hou F., Feng Y. et al. Characterization of a Triticum aestivum–Dasypyrum villosum T2VS·2DL translocation line expressing a longer spike and more kernels traits // Theor. Appl. Genet. 2015. V. 128. P. 2415–2425. https://doi.org/10.1007/s00122-015-2596-8
Zheng Q.P., Li Q.A., Wang X.U. et al. Development and characterization of a Triticum aestivum–Haynaldia villosa translocation line T4VS.4DL conferring resistance to wheat spindle streak mosaic virus // Euphytica. 2005. V. 145. P. 317–320.
Li H., Chen X., Xin Z.Y. et al. Development and identification of wheat-Haynaldia villosa 6DL.6VS chromosome translocation lines conferring resistance to powdery mildew // Plant Breed. 2005. V. 124. P. 203–205. https://doi.org/10.1111/j.1439-0523.2004.01062.x
Hou L.Y., Jia J.Q., Zhang X.J. et al. Molecular mapping of the stripe rust resistance gene Yr69 on wheat chromosome 2AS // Plant Dis. 2016. V. 100. P. 1717–1724. https://doi.org/10.1094/PDIS-05-15-0555-RE
Qi L.L., Pumphrey M.O., Friebe B. et al. Molecular cytogenetic characterization of alien introgressions with gene Fhb3 for resistance to Fusarium head blight disease // Theor. Appl. Genet. 2008. V. 117. P. 1155–1166. https://doi.org/10.1007/s00122-008-0853-9
Qi Z.J., Du P., Qian B.L. et al. Characterization of a wheat–Thinopyrum bessarabicum (T2JS-2BS·2BL) translocation line // Theor. Appl. Genet. 2010. V. 121. P. 589–597. https://doi.org/10.1007/s00122-010-1332-7
Kang H., Wang Y., Fedak G. et al. Introgression of chromosome 3Ns from Psathyrostachys huashanica into wheat specifying resistance to stripe rust // PLoS One. 2011. V. 6. e21802. https://doi.org/10.1371/journal.pone.0021802
Chen G., Zheng Q., Bao Y. et al. Molecular cytogenetic identification of a novel dwarf wheat line with introgressed Thinopyrum ponticum chromatin // J. Biosci. 2012. V. 37. P. 149–155. https://doi.org/10.1007/s12038-011-9175-1
Wu J., Yang X., Li H. et al. The introgression of chromosome 6P specifying for increased numbers of florets and kernels from Agropyron cristatum into wheat // Theor. Appl. Genet. 2006. V. 114. P. 13–20. https://doi.org/10.1007/s00122-006-0405-0
Guo J., Zhang X., Hou Y. et al. High‑density mapping of the major FHB resistance gene Fhb7 derived from Thinopyrum ponticum and its pyramiding with Fhb1 by marker‑assisted selection // Theor. Appl. Genet. 2015. V. 128. P. 2301–2316. https://doi.org/10.1007/s00122-015-2586-x
Ceoloni C., Kuzmanovic L., Forte P. et al. Wheat-perennial Triticeae introgressions: major achievements and prospects // Alien Introgression in Wheat Cytogenetics, Molecular Biology, and Genomics / Eds Molnár-Láng M., Ceoloni C., Doležel J. Switzerland: Springer Intern. Publ., 2015. P. 273–313. https://doi.org/10.1007/978-3-319-23494-6
Cox T.S., Van Tassel D.L., Cox C.M., DeHaan L.R. Progress in breeding perennial grains // Crop and Pasture Sci. 2010. V. 61. P. 513–521. https://doi.org/10.1071/CP09201
Culman S.W., Snapp S.S., Ollenburger M. et al. Soil and water quality rapidly responds to the perennial grain Kernza wheatgrass // Agron. J. 2013. V. 105. P. 735–744. https://doi.org/10.2134/agronj2012.0273
Zhang X., Sallam A., Gao L. et al. Establishment and optimization of genomic selection to accelerate the domestication and improvement of intermediate wheatgrass // Plant Genome. 2016. V. 9. № 1. P. 1–18.
Айдаров А.Н., Чурсин А.С., Шаманин В.П. Оценка многолетнего зернокормового злака в условиях Западной Сибири // Научные инновации – аграрному производству: Материалы Междун. научно-практ. конф., посвященной 100-летнему юбилею Омского ГАУ. Омск, 2018. С. 582–585.
Molnár-Láng M., Linc G. Wheat–barley hybrids and introgression lines // Alien Introgression in Wheat Cytogenetics, Molecular Biology, and Genomics / Eds Molnár-Láng M., Ceoloni C., Doležel J. Switzerland: Springer Intern. Publ., 2015. P. 315–345. https://doi.org/10.1007/978-3-319-23494-6
Molnár-Láng M., Linc G., Friebe B.R., Sutka J. Detection of wheat-barley translocations by genomic in situ hybridization in derivatives of hybrids multiplied in vitro // Euphytica. 2000. V. 112. P. 117–123. https://doi.org/10.1023/A:1003840200744
Nagy E.D., Molnár-Láng M., Linc G., Láng L. Identification of wheat-barley translocations by sequential GISH and two-colour FISH in combination with the use of genetically mapped barley SSR markers // Genome. 2002. V. 45. P. 1238–1247. https://doi.org/10.1139/g02-068
Cseh A., Kruppa K., Molnár I. et al. Characterization of a new 4BS.7HL wheat-barley translocation line using GISH, FISH, and SSR markers and its effect on the β-glucan content of wheat // Genome. 2011. V. 54. P. 795–804. https://doi.org/10.1139/g11-044
Kruppa K., Sepsi A., Szakács É. et al. Characterization of a 5HS-7DS.7DL wheat-barley translocation line and physical mapping of the 7D chromosome using SSR markers // J. Appl. Genet. 2013. V. 54. P. 251–258. https://doi.org/10.1007/s13353-013-0152-2
Darkó É., Barnabás B., Molnár-Láng M. Characterization of newly developed wheat/barley introgression lines in respect of aluminium tolerance // Am. J. Plant. Sci. 2012. V. 3. P. 1462–1469. https://doi.org/10.4236/ajps.2012.310176
Liu C.J., Atkinson M.D., Chinoy C.N. et al. Nonhomoeologous translocations between group 4, 5 and 7 chromosomes within wheat and rye // Theor. Appl. Genet. 1992. V. 83. P. 305–312. https://doi.org/10.1007/BF00224276
Devos K.M., Dubcovsky J., Dvořák J. et al. Structural evolution of wheat chromosomes 4A, 5A, and 7B and its impact on recombination // Theor. Appl. Genet. 1995. V. 91. P. 282–288. https://doi.org/10.1007/bf00220890
Endo T.R. Gametocidal genes // Alien Introgression in Wheat Cytogenetics, Molecular Biology, and Genomics / Eds Molnár-Láng M., Ceoloni C., Doležel J. Switzerland: Springer Intern. Publ., 2015. P. 121–132. https://doi.org/10.1007/978-3-319-23494-6
The International Wheat Genome Sequencing Consortium (IWGSC) Shifting the limits in wheat research and breeding using a fully annotated reference genome // Science. 2018. V. 361. № 6403. eaar7191. https://doi.org/10.1126/science.aar7191
Sears E.R. The aneuploids of common wheat // Mo. Agr. Exp. Sta. Res. Bull. 1954. V. 572. P. 1–58.
Salina E.A., Leonova I.N., Efremova T.T., Roder M.S. Wheat genome structure: translocations during the course of polyploidization // Functional and Integrative Genomics. 2006. V. 1. P. 71–80. https://doi.org/10.1007/s10142-005-0001-4
Zhang H., Jia J., Gale M.D., Devos K.M. Relationships between the chromosomes of Aegilops umbellulata and wheat // Theor. Appl. Genet. 1998. V. 96. P. 69–75. https://doi.org/10.1007/s001220050710
Адонина И.Г., Букатич Е.Ю., Пискарев В.В. и др. Особенности наследования транслокации, несущей ген устойчивости к бурой ржавчине, от Aegilops speltoides Tausch на хромосому 2D мягкой пшеницы // Генетика. 2018. Т. 54. № 8. С. 969–974.
Rey E., Molnár I., Doležel J. Genomics of wild relatives and alien introgressions // Alien Introgression in Wheat Cytogenetics, Molecular Biology, and Genomics / Eds Molnár-Láng M., Ceoloni C., Doležel J. Switzerland: Springer Intern. Publ., 2015. P. 347–381. https://doi.org/10.1007/978-3-319-23494-6
Сибикеев С.Н., Крупнов В.А., Воронина С.А., Бадаева Е.Д. Идентификация чужеродной хромосомы у линии мягкой пшеницы Мульти 6R // Генетика. 2005. Т. 41. № 8. С. 1084–1088.
Qi L.L., Friebe B., Zhang P., Gill B.S. Homoeologous recombination, chromosome engineering and crop improvement // Chromosome. Res. 2007. V. 15. P. 3–19. https://doi.org/10.1007/s10577-006-1108-8
Danilova T.V., Zhang G., Liu W. et al. Homoeologous recombination‑based transfer and molecular cytogenetic mapping of a wheat streak mosaic virus and Triticum mosaic virus resistance gene Wsm3 from Thinopyrum intermedium to wheat // Theor. Appl. Genet. 2017. V. 130. P. 549–556. https://doi.org/10.1007/s00122-016-2834-8
Zhang P., Dundas I.S., Xu S.S. et al. Chromosome engineering techniques for targeted introgression of rust resistance from wild wheat relatives // Methods in Mol. Biol. 2017. V. 1659. P. 163–172. https://doi.org/10.1007/978-1-4939-7249-4_14
Zhang W., Zhu X., Zhang M. et al. Meiotic homoeologous recombination‑based mapping of wheat chromosome 2B and its homoeologues in Aegilops speltoides and Thinopyrum elongatum // Theor. Appl. Genet. 2018. V. 131. P. 2381–2395. https://doi.org/10.1007/s00122-018-3160-0
King J., Grewal S., Yang C. et al. A step change in the transfer of interspecific variation into wheat from Amblyopyrum muticum // Plant Biotech. J. 2017. V. 15. P. 217–226. https://doi.org/10.1111/pbi.12606
King J., Grewal S., Yang C. et al. Introgression of Aegilops speltoides segments in Triticum aestivum and the effect of the gametocidal genes // Ann. Bot. 2018. V. 121. P. 229–240. https://doi.org/10.1093/aob/mcx149
Devi U., Grewal S., Yang C. et al. Development and characterization of interspecific hybrid lines with genome-wide introgressions from Triticum timopheevii in a hexaploid wheat background // BMC Plant Biol. 2019. V. 19. Article 183. https://doi.org/10.1186/s12870-019-1785-z
Grewal S., Yang C., Hubbart-Edwards S. et al. Characterization of Thinopyrum bessarabicum chromosomes through genome wide introgressions into wheat // Theor. Appl. Genet. 2018. V. 131. P. 389–406. https://doi.org/10.1007/s00122-017-3009-y
Ogbonnaya F.C., Abdalla O., Mujeeb-Kazi A. et al. Synthetic hexaploids: harnessing species of the primary gene pool for wheat improvement // Plant Breed. Rev.2013. V. 37. P. 35–122. https://doi.org/10.1002/9781118497869.ch2
Hartung F., Schiemann J. Precise plant breeding using new genome editing techniques: opportunities, safety and regulation in the EU // The Plant J. 2014. V. 78. P. 742–752. https://doi.org/10.1111/tpj.12413
Prohens J., Gramazio P., Plazas M. et al. Introgressiomics: A new approach for using crop wild relatives in breeding for adaptation to climate change // Euphytica. 2017. V. 213. Article 158. https://doi.org/10.1007/s10681-017-1938-9
Дополнительные материалы отсутствуют.


