Генетика, 2021, T. 57, № 6, стр. 615-631
Генетический контроль инфицирования человека SARS-CoV-2
А. Н. Кучер 1, *, Н. П. Бабушкина 1, А. А. Слепцов 1, М. С. Назаренко 1
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
* E-mail: aksana.kucher@megenetics.ru
Поступила в редакцию 23.06.2020
После доработки 26.07.2020
Принята к публикации 25.08.2020
Аннотация
В 2019 г. впервые обнаружен бета-коронавирус SARS-CoV-2, ставший причиной пандемии тяжелой острой респираторной вирусной инфекции COVID-19 (от COronaVIrus Disease 2019). Восприимчивость к SARS-CoV-2 и характер течения клинической картины COVID-19 определяются многими факторами, в том числе и генетическими особенностями как возбудителя, так и человека. Геном SARS-CoV-2 имеет сходство с геномами других патогенных для человека коронавирусов, вызывающих тяжелое течение инфекции: 79% – с геномом SARS-CoV и 50% – с геномом MERS-CoV. Наиболее значимые различия между SARS-CoV-2 и другими коронавирусами регистрируются в структуре гена S-белка – ключевого белка, отвечающего за связывание вируса с рецептором клеток организма-хозяина. В частности, в S-белке SARS-CoV-2 выявлены замены, приводящие к формированию сайта расщепления фурином, отсутствующего у других SARS-подобных коронавирусов, что может объяснять высокую патогенность SARS-CoV-2. У человека к числу генов, значимых для начальных этапов инфицирования, можно отнести ACE2, ANPEP, DPP4 (кодируют рецепторы связывания коронавируса); TMPRSS2, FURIN, TMPRSS11D, CTSL, CTSB (кодируют протеазы, участвующие в процессах проникновения коронавируса в клетку); DDX1 (ген АТФ-зависимой РНК-хеликазы DDX1, способствующей репликации коронавирусов); IFITM1, IFITM2 и IFITM3 (кодируют интерферон-индуцируемые трансмембранные белки, обладающие противовирусным эффектом). Данные гены экспрессируются во многих тканях (в том числе восприимчивых к воздействию SARS-CoV-2), в них описаны редкие и частые варианты, влияющие на структуру кодируемого белка, его свойства и уровень экспрессии. Для ряда частых генетических вариантов с доказанной функциональной значимостью характерна вариабельность частоты аллелей в популяциях мира, что может определять межпопуляционные различия в распространенности COVID-19 и в клинических особенностях течения данной патологии. На уровень экспрессии генов, значимых для формирования восприимчивости к SARS-CoV-2, влияют эпигенетические модификации, наличие сопутствующих заболеваний на момент инфицирования, прием лекарственных препаратов, вредные привычки.
Коронавирус SARS-CoV-2, впервые выявленный в 2019 г., привел к пандемии тяжелой острой респираторной вирусной инфекции, известной как COVID-19 (от COronaVIrus Disease 2019). По данным официальной статистики на 21 июня 2020 г. пандемия охватила 215 стран (территорий), COVID-19 заразились более 8.8 млн человек, более 465 тыс. скончались [1].
SARS-CoV-2 относится к РНК-содержащим бета-коронавирусам (CoV). Для человека известны семь коронавирусов: альфа-коронавирусы HCoV-229E, HCoV-NL63 и бета-коронавирусы HCoV-OC43, HCoV-HKU1, SARS-CoV, MERS-CoV, SARS-COV-2 [2]. Коронавирусы различаются по патогенности: 229E, OC43, NL63 и HKU1 вызывают легкие формы инфекционного заболевания, а SARS-CoV, MERS-CoV и SARS-CoV-2 являются высокопатогенными (SARS-CoV явился причиной атипичной пневмонии в 2002–2003 гг., MERS-CoV – ближневосточного респираторного синдрома в 2015 г.) [2, 3]. Клиническая картина заболеваний, вызванных высокопатогенными коронавирусами, имеет как общие, так и специфические черты [4–6]. В частности, по сравнению с инфекционным заболеванием, вызываемым MERS-CoV, для COVID-19 характерны большая контагиозность, но меньшая летальность, существенная гетерогенность клинических симптомов, в том числе в разных возрастных группах и в различных странах мира [5, 7–9]. Наиболее частыми симптомами COVID-19 являются кашель, лихорадка, одышка, регистрируются также миалгия, диарея, тошнота и рвота [6, 8]. Однако индивидуальные клинические симптомы широко варьируют: от бессимптомного течения и легкой простуды до таких осложнений как бронхит, пневмония, тяжелый острый респираторный дистресс-синдром, полиорганная недостаточность [6].
Подверженность человека коронавирусной инфекции и клиническая картина заболевания зависят от многих причин, среди которых определенная роль принадлежит генетическим особенностям как вирусов, так и человека [10–13]. Начальным этапом инфицирования организма является контакт коронавируса с восприимчивыми к нему клетками. У человека известно около 100 генов, продукты которых вовлечены в процесс проникновения вирусов в клетку, отнесенных к пяти категориям “Gene Ontology”: проникновение вируса в клетку организма-хозяина (GO: 0046718); вирус-рецепторная активность (GO: 0001618); слияние вирусной оболочки с плазматической мембраной клеток организма-хозяина (GO: 0019064); клатрин-зависимый эндоцитоз вируса клеткой-хозяином (GO: 0075512); рецептор-опосредованный эндоцитоз вируса клеткой-хозяином (GO: 0019065) [14, 15]. По мере изучения взаимодействий между вирусами и клетками организма-хозяина число известных генов, от которых зависит восприимчивость к коронавирусной инфекции, будет увеличиваться. В то же время коронавирусы также различаются по структуре генома и ключевых молекул, отвечающих за их проникновение в клетки [11, 16, 17]. Поэтому не все гены человека, продукты которых вовлечены в регуляцию проникновения вирусов в клетки, оказываются значимыми в отношении формирования риска развития инфекции, вызванной разными коронавирусами.
В настоящем обзоре анализируются особенности генома SARS-CoV-2 и структурно-функциональных свойств генов и белков человека, отвечающих за восприимчивость к данному возбудителю.
ПАТОГЕНЕТИЧЕСКИ ЗНАЧИМЫЕ ОСОБЕННОСТИ ГЕНОМА SARS-COV-2
Геном SARS-CoV-2 близок по структуре к геномам двух коронавирусов летучих мышей bat-SL-CoVZC45 и bat-SL-CoVZXC21 (идентичность составляет 88–96%) и значительно меньшим сходством обладает с геномами SARS-CoV (79–80%) и MERS-CoV (около 50%) [17, 18]. Геномы отдельных образцов SARS-CoV-2, полученных от китайских пациентов, характеризовались высоким сходством, превышающим 99% [18], но оценки генетического разнообразия уточняются по мере роста количества исследований структуры геномов SARS-CoV-2019 из различных регионов мира [19, 20].
По сравнению с SARS-CoV и MERS-CoV для генома SARS-CoV-2 свойственны более низкие значения эффективного числа кодонов, что предполагает более высокую эффективность экспрессии генов, в том числе генов, кодирующих структурные белки – шипа (S), оболочки (Е), мембраны (М), нуклеокапсида (N) [2, 21]. S-белок (белок “шипа”, или spike-белок), отвечающий за прикрепление SARS-CoV-2 к мембранному рецептору, рассматривается в качестве критичного для проникновения в клетку организма-хозяина [22]. Коронавирус SARS-CoV-2 (как и HCoV-NL63, SARS-CoV, MERS-CoV) использует в качестве рецептора мембранно-связанный ангиотензин-превращающий фермент 2 (ACE2) [17, 23–25]. Соответственно SARS-COV-2 может проникать во все клетки, экспрессирующие ACE2. Однако коронавирусы для проникновения в клетки организма-хозяина могут использовать в качестве рецепторов и ряд других молекул [25–27].
По сравнению с другими коронавирусами структура S-белка SARS-CoV-2 имеет некоторые важные для проникновения в клетку организма-хозяина особенности [16, 17, 28]. Идентичность между S-белками SARS-СОV и SARS-COV-2 составляет 72–75% [16, 17]. P. Zhou с соавт. [17] к числу основных отличий в структуре последовательности гена, кодирующего S-белок SARS-CoV-2, отнесли три короткие вставки в N-концевом домене, а также изменения в четырех из пяти ключевых остатков в рецептор-связывающем мотиве. По сравнению с SARS-CoV для SARS-CoV-2 характерны: более высокая аффинность к связыванию с рецептором АСЕ2 вследствие замен в С‑терминальном домене S-белка (наблюдается больше взаимодействий с ACE2, H-связей и др.) [16, 28, 29], лучшая способность к слиянию с плазматической мембраной клетки организма-хозяина в связи с аминокислотными заменами в HR1 домене S2 субъединицы S-белка [30] и в целом более высокая активность [23].
В результате сравнительного анализа геномов изолятов SARS-CoV-2 из различных регионов мира установлены множественные мутации, в том числе и в генах, кодирующих структурные белки (включая S-белок) [20]. Например, в клинических изолятах Индии обнаружена мутация в рецептор-связывающем домене S-белка в положении 407, приводящая к изменению вторичной структуры белка, так как положительно заряженная аминокислота аргинин заменяется на гидрофобную С-бета-разветвленную аминокислоту изолейцин [19]. Обнаружены аминокислотные замены в S-белке и у SARS-CoV-2 из других географических регионов [31]. Мутации, приводящие к изменению структуры ключевых белков, ответственных за связывание коронавируса со своим рецептором, могут влиять на патогенность коронавируса.
После связывания S-белка с рецептором ACE2 происходит расщепление белка на S1 и S2 субъединицы, что инициирует слияние вирусной оболочки с клеточной мембраной и проникновение HCoV в клетку [22]. Для этих целей коронавирусы (как и другие вирусы) используют различные протеазы клеток организма-хозяина, “выбор” которых определяется особенностями структуры и доступностью сайта для протеолитического расщепления S-белка [32]. Например, было показано, что по сравнению с лабораторными штаммами 1966 г. современные клинические изоляты альфа-коронавируса HCoV-229E используют для проникновения не катепсин L, а TMPRSS2, что связано с двумя аминокислотными заменами (R642M и N714K) в S-белке [33]. SARS-CoV-2 для активации и расщепления S-белка также использует TMPRSS2 [34–36]. Кроме того, у SAR-CoV-2 в гене S-белка выявлены замены, приводящие к формированию фурин-подобного сайта расщепления, отсутствующего у других SARS-подобных коронавирусов [25, 37]. Наличие сайта расщепления фурином в SARS-CoV-2 обусловливает способность слияния “вирус–клетка” и “клетка–клетка”, что обеспечивает высокую патогенность SARS-CoV-2 по сравнению с другими бета-коронавирусами [38, 39].
Так как проникновение коронавирусов в клетку происходит при участии белковых продуктов клеток организма-хозяина, можно ожидать, что особенности в структуре генов, кодирующих данные белки, будут определять индивидуальные различия в восприимчивости к коронавирусам, в том числе и SARS-CoV-2.
ГЕНЕТИЧЕСКИЕ ФАКТОРЫ ВОСПРИИМЧИВОСТИ К SARS-COV2 У ЧЕЛОВЕКА
На основании классического близнецового исследования были рассчитаны коэффициенты наследуемости для некоторых симптомов COVID-19: для лихорадки данный показатель составил 41 (95%-ный доверительный интервал 12–70)%, для аносмии – 47 (27–67)%, для делирия – 49 (24–75)%, что позволило оценить предсказанный коэффициент наследуемости COVID-19 в 50 (29–70)% [40]. Эти оценки свидетельствуют о значимом генетическом компоненте в формировании симптомов данного заболевания. Исследования, посвященные выявлению генетических маркеров восприимчивости к SARS-CoV-2, немногочисленны [12, 41, 42].
Известен ряд генов, продукты которых могут принимать участие в процессе инфицирования человека коронавирусами, включая SARS-CoV-2 (табл. 1). Помимо генов рецепторов (ACE2, ANPEP, DPP4) и генов протеаз (TMPRSS2, FURIN, TMPRSS11D, CTSL, CTSB), способствующих проникновению коронавируса в клетку, к их числу можно отнести ген АТФ-зависимой РНК-хеликазы DDX1 (DDX1), которая способствует репликации коронавирусов (как это было показано для SARS-CoV) [56], а также гены IFITM1, IFITM2 и IFITM3, кодирующие интерферон-индуцируемые трансмембранные белки [12, 57, 58].
Таблица 1.
Характеристика генов-кандидатов восприимчивости к SARS-CoV-2 человека
| № | Ген (альтернативные названия#), хромосомная локализация | Белок | Число LoF/ миссенс-вариантов c частотой MAF ≤ 1% (по [43]) | Миссенс-варианты
c частотой MAF > 1%: rs (аллель: min–max частота) (по [14, 43, 44]) |
Число известных eQTL-вариантов (по [45, 46]) | Участие в процессах инфицирования коронавирусом | Коронавирусы, использующие продукты данных генов для проникновения в клетки организма-хозяина |
|---|---|---|---|---|---|---|---|
| 1 | ACE2 (ACEH), Xp22.2 | Ангиотензин- превращающий фермент 2 | 5/239 | Нет | 288 | Рецептор коронавируса [25] | SARS-CoV-2; SARS-CoV; HCoV-NL63 [10, 16, 23, 24, 28, 35, 39] |
| 2 | DPP4 (CD26), 2q24.2 |
Дипептидилпептидаза 4 | 60/391 | rs1129599 (A: 0–4%) | 130 | Рецептор коронавируса [47] | SARS-CoV-2; hCoV-EMC (MERS-CoV) [47–49] |
| 3 | ANPEP (APN, CD13), 15q26.1 | Аминопептидаза N | 47/547 | rs8192297 (C: 16–37%) rs25651 (T: 23–51%) rs25653 (C: 10–61%) rs17240268 (A: 0–14%)* rs41276922 (A: 0–9%)* rs17240212 (A: 0–3%) |
594 | Рецептор коронавируса [26] | HCV-229E [26, 50] |
| 4 | FURIN (FUR), 15q26.1 | Фурин | 5/339 | rs16944971 (T: 0–9%) | 295 | Расщепление S-белка; способствует проникновению вируса в клетку [25, 29, 37] | SARS-CoV-2 [29, 37, 51] |
| 5 | TMPRSS11D (HAT), 4q13.2 | Трансмембранная протеаза серина 11D | 17/210 | 0 | 191 | Расщепление S-белка и ACE2; способствует слиянию вирусной и клеточной мембран [35] | SARS-CoV; HCoV-229E [35, 52] |
| 6 | TMPRSS2, 21q22.3 | Трансмембранная протеаза серина 2 | 39/332 | rs12329760 (T: 15–36%)* rs75603675 (A: 2–40%) |
203 | Расщепление S-белка; способствует проникновению вируса в клетку [14, 34, 54] | SARS-CoV-2; SARS-CoV; MERS-CoV [34, 35, 39, 51, 53, 54] |
| 7 | CTSL, 9q21.33 |
Катепсин L | 14/192 | rs11541204 (A: 0–5%) | 258 | Активация S-белка; способствует проникновению вируса в клетку хозяина [33, 52] | SARS-CoV-2; HCoV-229E; MERS-CoV; SARS-CoV [29, 55] |
| 8 | CTSB, 8p23.1 |
Катепсин B | 41/369 | rs12338 (G: 49–67%)* rs1803250 (С: 0–15%)* rs17573 (T: 0–17%)* |
2553 | Активирует слияние мембран вируса и клетки хозяина [14, 55] | SARS-CoV-2; SARS-CoV; MERS-CoV [14, 29, 55] |
| 9 | DDX1, 2p24.3 |
АТФ-зависимая РНК-хеликаза DDX1 | 14/305 | 0 | 463 | Способствует репликации коронавируса (in vitro) [56] | SARS-CoV [56] |
| 10 | IFITM1, 11p15.5 |
Интерферон-индуцируемый трансмембранный белок 1 | 4/72 | 0 | 201 | Ограничение репликации и проникновения вирусов в клетку хозяина [14, 57] | SARS-CoV [14, 57] |
| 11 | IFITM2, 11p15.5 | Интерферон-индуцируемый трансмембранный белок 2 | 11/123 | rs1059091 (A: 31–67%)* rs1058900 (T: 0–42%) rs14408 (T: 4–62%) |
220 | Ограничение репликации и проникновения вирусов в клетку хозяина [14, 57] | SARS-CoV и другие вирусы [57, 58] |
| 12 | IFITM3, 11p15.5 | Интерферон-индуцируемый трансмембранный белок 3 | 6/92 | rs1136853 (T: 0–8%)* | 439 | Ограничение репликации и проникновения вирусов в клетку хозяина; инактивация новых вирусов, выходящих из зараженной клетки [59] | SARS-CoV-2; SARS-CoV и другие вирусы [12, 57, 58] |
Примечание. # – указаны альтернативные названия генов и белков, встречающиеся в использованных литературных источниках; * – “потенциально патогенный” эффект, по результатам использования программ предикции функционального эффекта генетических вариантов, встроенных в браузер Ensemble. LoF – вариант с потерей функции; MAF – частота редкого аллеля.
Роль АСЕ2 в качестве рецептора SARS-CoV-2 считается доказанной [16, 23, 25, 28, 39, 60]. Данные о возможности использования SARS-CoV-2 других рецепторов, известных для коронавирусов, неоднозначны. Ряд исследователей [27, 48], на основании моделирования структурных взаимодействий между S-белком и рецепторами других коронавирусов, к потенциальным рецепторам SARS-CoV-2 отнесли дипептидилпептидазу 4 (DPP4, известна также как CD26), выступающую в качестве рецептора для MERS-CoV [49]. Причем для SARS-CoV-2 и MERS-CoV характерно наличие идентичных критически значимых для связывания с S-белком участков DPP4 [47]. В то же время P. Zhou с соавт. [17] в исследовании in vitro установили, что SARS-CoV-2 не использует в качестве рецептора DPP4. Эти же авторы не подтвердили значение аминопептидазы N (ANPEP, известна также как APN и CD13) в качестве рецептора SARS-CoV-2. Интересно, что ANPEP является рецептором для HCoV-229E, но не обладает такими свойствами для HCoV-OC43 [26]. Однако для ANPEP и DPP4 характерны близкие паттерны экспрессии с ACE2 в 13 тканях, что позволило F. Qi с соавт. [61] рассматривать их в качестве потенциальных рецепторов для SARS-CoV-2. Для разрешения противоречия относительно рецепторных свойств DPP4 и уточнения возможной рецепторной роли ANPEP для SARS-CoV-2 необходимо проведение дополнительных исследований.
После прикрепления SARS-CoV-2 к рецептору ACE2 для обеспечения проникновения коронавируса в клетку необходимы протеазы, в том числе – TMPRSS2 и FURIN [29, 37, 39]. D. Bestle с соавт. [51] установили, что для проникновения SARS-CoV-2 в клетки его S-белок должен быть расщеплен в двух разных местах протеазами клеток организма-хозяина: TMPRSS2 в сайте S2 и FURIN в сайте S1/S2.
Трансмембранная протеаза TMPRSS2 и родственные протеазы (такие как TMPRSS11D) способствуют проникновению коронавируса в клетку двумя механизмами: путем расщепления рецептора ACE2 (что может стимулировать поглощение коронавируса) и путем расщепления S-белка коронавируса (что приводит к активации белка и слиянию вирусной оболочки с мембраной клетки организма-хозяина) [35, 39]. В исследовании S. Bertram с соавт. [52] показано, что TMPRSS2 активирует катепсин-независимое проникновение HCoV-229E в клетку хозяина, причем активация TMPRSS2 защищает 229E-S-зависимый вход в клетку от ингибирования интерферон-индуцируемыми трансмембранными белками. На модельных животных установлено, что дефицит данной протеазы в клетках дыхательных путей снижает тяжесть течения SARS-CoV и MERS-CoV, что свидетельствует о важности TMPRSS2 в процессах инфицирования [62].
Проникновение в клетку SARS-CoV-2 активируют также лизосомные катепсины [29]. В отличие от TMPRSS2 катепсины способствуют проникновению коронавирусов в клетку через эндоцитоз (лизосомный путь); при этом “выбор” протеазы для проникновения определяется структурными особенностями S-белка вирусов [33]. Протеаза TMPRSS2 и лизосомные катепсины оказывают кумулятивное с фурином влияние на активацию проникновения SARS-CoV-2 в клетки организма-хозяина [29].
Интерферон-индуцируемые трансмембранные белки (IFITM1, IFITM2, IFITM3) способны ограничивать репликацию коронавирусов, их проникновение в клетки и инактивировать новые коронавирусы при выходе из зараженных клеток (показано для SARS-CoV и других вирусов, таких как вирус гриппа А, вирус Денге и вирус Западного Нила) [57–59, 63]. Поскольку белки IFITM индуцируются интерферонами типа I и II, считают, что они имеют решающее значение для противовирусного действия интерферона [63]. В исследовании Y. Zhang с соавт. [12] установлено, что структурные особенности гена IFITM3 ассоциированы с тяжестью течения COVID-19.
Таким образом, большинство из включенных в табл. 1 генов относятся к категории с доказанной или потенциальной значимостью в отношении участия их белковых продуктов в процессе заражения клеток SARS-CoV-2. Пять генов (TMPRSS11D, ANPEP, DDX1, IFITM1, IFITM2) можно рассматривать как вероятно значимые, так как кодируемые ими продукты вовлечены в процессы проникновения других коронавирусов (SARS-CoV, HCoV-229E) и ряда других вирусов в клетки организма-хозяина [35, 52, 56–58] (см. табл. 1). Далее в статье все эти гены будут обозначены как гены-кандидаты восприимчивости к SARS-CoV-2.
Из числа привлеченных к рассмотрению генов только ACE2 локализован на Х-хромосоме, что может выступать в качестве возможной причины гендерных различий подверженности к SARS-CoV-2 [64–66], другие гены локализованы на восьми разных хромосомах (табл. 1).
Гены-кандидаты восприимчивости к SARS-CoV-2 экспрессируются во многих органах/тканях, в том числе и чувствительных к данному коронавирусу (по данным клинических наблюдений [7–9]). Однако отмечаются различия по уровню их экспрессии в разных органах (рис. 1). На основании уровня экспрессии генов-кандидатов восприимчивости к SARS-CoV-2 ткани группируются в несколько кластеров (рис. 1). В один кластер объединены ткани легких, аорты, коронарной артерии, щитовидной железы, висцеральной и подкожной жировой тканей; второй кластер объединяет ткани других привлеченных к сравнению органов, наибольшие отличия наблюдались по уровню экспрессии в клетках крови.
Рис. 1.
Теплокарта, отражающая уровень экспрессии генов-кандидатов восприимчивости к SARS-CoV-2 в различных органах/тканях человека (построено по медианным значениям числа транскриптов на миллион (TPM) по [45]). Уровень экспрессии нормализован по генам, где ноль соответствует среднему уровню экспрессии генов. Значения в ячейках отражают число статистически значимых eQTL, влияющих на уровень экспрессии генов в соответствующих тканях.
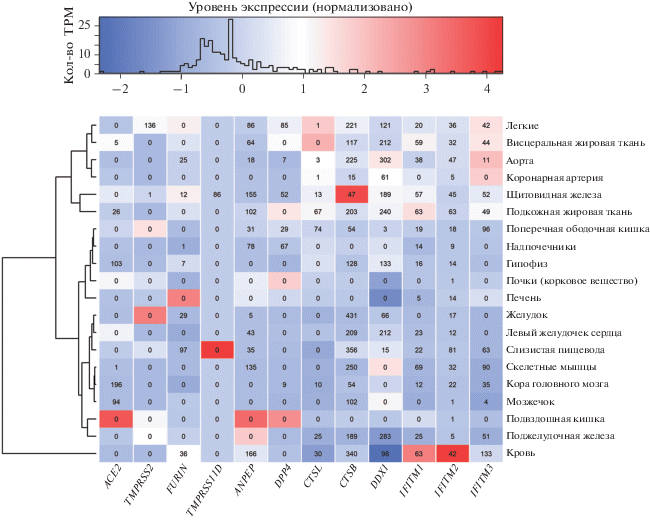
Различий по уровню экспрессии генов ACE2, TMPRSS2, CTSB и CTSL между мужчинами и женщинами не выявлено [67]. Коэкспрессия белков является важным условием проникновения в клетки и распространения вирусов в организме человека [68], а органы и ткани, где экспрессируются эти гены, могут быть потенциально восприимчивы к коронавирусной инфекции [69].
На индивидуальные различия по восприимчивости к SARS-CoV-2 могут оказывать влияние варианты в генах-кандидатах, изменяющие структуру белка или его экспрессию. Кроме того, экспрессия генов может зависеть от уровня метилирования их промоторных регионов.
Генетические варианты, влияющие на структуру белков
Несинонимичные замены регистрируются во всех генах-кандидатах восприимчивости к SARS-CoV-2, но в большинстве случаев они являются крайне редкими, низкополиморфными или регистрируются не во всех этно-территориальных группах (табл. 1; [44]). Это может быть связано с функциональной значимостью кодируемых данными генами белков и ферментов, а также их локализацией в клетке: все рассматриваемые белки, за исключением DDX1, локализуются в клеточной мембране [14]. В то же время в гене ACE2 зарегистрировано большое число редких вариантов, которые важны для проникновения SARS-CoV-2 в клетки организма-хозяина и развития инфекции, в том числе: S19P, I21T/V, E23K, A25T, K26R, T27A, E35D/K, E37K, Y50F, N51D/S, M62V, N64K, K68E, F72V, E75G, M82I, T92I, Q102P, G220S H239Q, G326E, E329G, G352V, D355N, H378R, Q388L, P389H, E467K, H505R, R514G/* и Y515C; для некоторых из них (выделены полужирным шрифтом) регистрировались межрасовые различия по частоте встречаемости вариантов [67]. Так как ген АСЕ2 локализован на Х-хромосоме, наличие таких вариантов у мужчин в гемизиготном состоянии может выступать в качестве неблагоприятного фактора инфицирования SARS-CoV-2. В генах-кандидатах восприимчивости к SARS-CoV-2 установлено большое число вариантов, приводящих к аминокислотным заменам или к потере функции белков, с частотой регистрации менее 1% (см. табл. 1 ). Не исключено, что именно такие редкие варианты как в гене ACE2 [67], так и в других рассматриваемых генах могут выступать в качестве генетических факторов, определяющих индивидуальную восприимчивость к SARS-CoV-2.
К числу высокополиморфных миссенс-вариантов в генах-кандидатах восприимчивости к SARS-CoV-2 во всех территориальных группах, охарактеризованных в рамках проекта 1000 геномов (1000 Genomes Project, Phase 3), относятся: rs12329760 (p.Val160Met) гена TMPRSS2; rs25653 (p.Arg86Gln), rs8192297 (p.Ile603Met) и rs25651 (p.Ser752Asn) гена ANPEP; rs12338 (p.Leu26Met, p.Leu26Val) гена CTSB и rs1059091 (p.Ile121Leu) гена IFITM2 (табл. 1). Причем rs12329760 гена TMPRSS2, rs12338 гена CTSB и rs1059091 гена IFITM2 отнесены к категории “потенциально патогенных”, согласно программам SIFT, PolyPhen, Mutation Assessor и MetaLR, встроенным в Ensemble [44].
Для большинства миссенс-вариантов частота регистрации аллелей различается в 2 и более раза между изученными этно-территориальными группами (табл. 1). Интерес для рассмотрения представляют также SNP, которые существенно различаются по уровню полиморфизма между регионами мира (в некоторых случаях частота редких аллелей варьирует 0 до 42%). Так, по данным проекта “1000 Геномов” [44] аллель A по rs75603675 (p.Gly8Asp) гена TMPRSS2 регистрируется с частотой 2% в популяциях Восточной Азии, 22% в популяциях Южной Азии, 0% в Африке, 27% в Америке и 40% в европейских популяциях; частота аллеля T по rs1058900 (p.Val33Ala) гена IFITM2 составляет 0, 7, 4, 20 и 42% соответственно, частота аллеля T по rs14408 (p.Met41Arg) гена IFITM2 – 4, 27, 24, 33 и 60% соответственно.
Важную роль с точки зрения формирования восприимчивости к коронавирусам могут играть миссенс-замены в функционально-значимом участке белковой молекулы (как, например, в случае rs12329760 гена TMPRSS2 замена p.Val160Met происходит в функциональном SRCR домене белка (опосредует межбелковые взаимодействия и связывание лигандов) [14]). В гене ANPEP в регионе, критичном для связывания с HCoV-229E (аминокислоты 260–353), выявлены варианты, в том числе приводящие к несинонимичным заменам [14, 50]. Такие замены могут сказаться на свойствах белков и соответственно на различиях в степени подверженности коронавирусной инфекции обладателей разных генотипов по данным вариантам.
Среди полиморфных вариантов, локализованных в экзонах привлеченных к рассмотрению генов-кандидатов восприимчивости к SARS-CoV-2, только для одного SNP ранее была установлена патогенетическая значимость в отношении риска развития инфекционных заболеваний – для rs12252 гена IFITM3 (OMIM: 605579, предрасположенность к тяжелому течению гриппа) [70, 71]. Ассоциация с более высоким риском тяжелого течения гриппа H1N1/09 была зарегистрирована для индивидов с аллелем С rs12252 [71]. Авторы процитированного исследования в условиях in vitro показали, что клеточные линии с генотипом СС характеризовались более низким уровнем экспрессии IFITM3 и более высокой восприимчивостью к инфекции, чем линии с генотипом TT по данному SNP. Несмотря на то что полиморфный вариант rs12252 приводит к синонимичной замене (p.Ser14Ser), для некоторых транскриптов эта замена локализована в регионе сплайсинга или 5'UTR [44]. Клетки, экспрессирующие укороченный на 22 аминокислоты белок, не способны ограничивать репликацию вируса по сравнению с белком IFITM3 дикого типа [71]. Частота “неблагоприятного” аллеля С варьирует от 4% в европейских популяциях до 53% в популяциях Южной Азии [44].
Показано, что носители генотипа СС rs12252 гена IFITM3 также имеют риск более тяжелого течения COVID-19 (доля носителей такого генотипа среди пациентов со средней степенью тяжести заболевания составляет 28.57%, с тяжелым течением – 50% и среди умерших – 66.7%) [12]. Несмотря на то что это исследование выполнено на небольшой выборке, с учетом ранее полученных данных об ассоциации rs12252 с тяжелым течением гриппа H1N1/09 [70, 71], о молниеносном развитии вирусной пневмонии с тяжелой клинической картиной у мышей с нокаутом гена Ifitm3 при заражении вирусом гриппа с низкой патогенностью [71], о неспособности гомозигот СС в случае синтеза укороченного белка IFITM3 ограничивать репликацию вируса гриппа H1N1/09 [71] и о критической роли 21 N-концевой аминокислоты IFITM3 (как и С-концевой трансмембранной области белка) для противовирусной активности в отношении вируса везикулярного стоматита in vitro [72], можно предположить, что данный вариант обладает высокой прогностической значимостью в отношении оценки риска развития COVID-19 и для определения характера течения инфекции, вызванной SARS-CoV-19.
Генетические варианты с регуляторным потенциалом
В генах-кандидатах восприимчивости к SARS-CoV-2 регистрируются многочисленные SNP, в том числе и варианты, которые могут оказать влияние на уровень экспрессии генов. Известны eQTL для генов-кандидатов восприимчивости к коронавирусной инфекции, число и спектр которых различны для разных органов и тканей (см. табл. 1, рис. 1; см. также [45]). Например, по сравнению с другими генотипами в легких более высокий уровень экспрессии гена TMPRSS2 характерен для обладателей генотипа АА по rs35074065, генотипа TT по rs34783969, генотипа АА по rs463727 и т.д. У индивидов с генотипом AA по rs1622599 более высокий уровень экспрессии гена CTSB наблюдался в тканях легких, клетках крови, поджелудочной железе, большеберцовой артерии, но более низкий – в слизистой оболочке пищевода и щитовидной железы [45].
К категории eQTL для гена TMPRSS2 относятся rs2070788 и rs383510, для GG и ТТ генотипов которых соответственно характерен более высокий уровень экспрессии данного гена (в том числе и в ткани легких) [45]. Эти же генотипы ассоциированы с восприимчивостью к вирусу гриппа А (H7N9) и с риском тяжелого течения гриппа A (H1N1) [73]. Между территориальными группами населения регистрируются различия по частоте “неблагоприятных” аллелей и соответственно генотипов ряда eQTL [44]. Например, “неблагоприятный” аллель G по rs2070788 гена TMPRSS2 встречался с частотой 27% в африканских популяциях, 36% в восточно-азиатских и более 46% в европейских, южно-азиатских и американских популяциях.
Интересно, что ген TMPRSS2 локализован в непосредственной близости с генами MX1 и MX2, которые кодируют две интерферон-индуцируемые динамин-подобные ГТФазы. MX1 обладает противовирусной активностью в отношении широкого спектра РНК-вирусов и некоторых ДНК-вирусов; для MX2 характерен сильный противовирусный эффект в отношении вируса иммунодефицита человека типа 1. Маркеры rs2070788 и rs383510 являются также eQTL для гена MX1 [14, 45, 74].
Ассоциированный с заболеваниями сердечно-сосудистой системы (гипертонией, ишемической болезнью сердца, атеросклерозом коронарных артерий) аллель А rs17514846, расположенного в интроне гена FURIN [75–77], приводит к более высокой, чем альтернативный аллель, транскрипционной активности данного гена в эндотелиальных клетках сосудов [45, 78]. Эти данные хорошо согласуются с клиническими наблюдениями, согласно которым большая подверженность заражению SARS-CoV-2 и более тяжелое течение COVID-19 наблюдаются у лиц с заболеваниями сердечно-сосудистой системы [79]. Частота регистрации “неблагоприятного” аллеля А rs17514846 варьирует от 16% в популяциях Восточной Азии до 88% в африканских популяциях; в европейских популяциях этот аллель встречается с частотой 46% [44].
Влияние на уровень экспрессии гена CTSL оказывают генотипы по rs3118869 и rs2378757 [45, 80]. Для первого указанного eQTL зависимость уровня экспрессии от генотипов различалась в органах/тканях (более высокий уровень экспрессии CTSL регистрировался у носителей аллеля С – в коже, большеберцовом нерве и ободочной кишке, у обладателей гомозиготных генотипов АА и СС – в подкожной жировой ткани и большеберцовой артерии, у носителей гетерозиготного генотипа – в крови). N. Mbewe-Campbell с соавт. [80] показано, что генотип СС rs3118869, который расположен в промоторе гена CTSL, ассоциирован с артериальной гипертензией. Не исключено, что пациенты с артериальной гипертензией и генотипом СС rs3118869 гена CTSL более подвержены к SARS-CoV-2 и поражению тех органов, где носительство генотипа СС приводит к более высокому уровню экспрессии данного гена. Для rs2378757 в 11 органах/тканях (в том числе в легких, аорте, подкожной жировой ткани) уровень экспрессии гена CTSL снижался в зависимости от числа аллелей А: АА > AC > CC.
Специфичность экспрессионных профилей генов-кандидатов восприимчивости к SARS-CoV-2 (в том числе и вследствие генетических особенностей индивидов) может определять межпопуляционные различия в распространении COVID-19. В частности, с особенностями уровня экспрессии гена FURIN (одного из ключевых генов, продукт которого участвует в процессе проникновения коронавируса в клетку организма-хозяина) некоторые исследователи связывают разные клинические сценарии течения COVID-19 в регионах мира (в частности, в Китае и в Италии) [81]. Для гена FURIN известно около 300 eQTL (табл. 1), но их число (рис. 1) и спектр отличаются в разных органах и тканях [45].
Приведенные примеры функционально-значимых полиморфных вариантов не отражают всего их разнообразия. A. Paniri с соавт. [82], используя in silico анализ предсказания функциональной значимости вариабельности гена TMPRSS2, выявили 21 SNPs, которые влияют на функцию и структуру кодируемого им белка – на фолдинг, посттрансляционную модификацию, сплайсинг, а также на регуляторный потенциал микроРНК. В частности, rs12329760 изменяет форму белка таким образом, что de novo формируется “карман” белка; rs875393 создает новый донорный сайт, сайты в сайленсерах и разрушает три сайта в энхансерном мотиве. Ряд SNP (rs12627374, rs456142, rs462574, rs456298) приводят к разрушению/созданию сайтов связывания, ослаблению или усилению эффектов различных микроРНК (hsa-miR-548c-3p, hsa-miR-127-3p, hsa-miR1324, hsa-miR-5089, hsa-miR-204-5p, hsa-miR-211-5p, hsa-miR-4685-3p, hsa-miR-4716-5p). Все эти структурно-функциональные особенности гена TMPRSS2, как и других генов-кандидатов восприимчивости к SARS-CoV-2, могут лежать в основе индивидуальных различий в подверженности данному инфекционному заболеванию и определять характер его течения.
Факторы, влияющие на экспрессию генов-кандидатов восприимчивости к SARS-CoV-2
Клинические наблюдения показывают, что наличие патологических состояний (заболеваний сердечно-сосудистой системы, почек, эндокринные нарушения) и пожилой возраст выступают в качестве факторов риска развития, а также тяжелого течения COVID-19 [8, 64, 83, 84]. При некоторых заболеваниях и с увеличением возраста могут регистрироваться изменения в уровне экспрессии генов-кандидатов восприимчивости к SARS-CoV-2, в том числе и вследствие эпигенетических модификаций.
С возрастом увеличивается экспрессия генов ACE2 и TMPRSS2 в сустентакулярных (вспомогательных) клетках обонятельного эпителия (показано на модельных объектах) [85]. У людей старшего возраста регистрируются более высокие активность DPP4 [86] и уровень CTSB (уровень CTSL не менялся) в различных тканях и органах [87]. Эти наблюдения могут объяснять более высокую подверженность COVID-19 лиц пожилого возраста [88].
При многих заболеваниях регистрируется изменение уровня экспрессии генов-кандидатов восприимчивости к SARS-CoV-2. У пациентов с идиопатической дилатационной кардиомиопатией и ишемической кардиомиопатией увеличивается экспрессия гена ACE2 в миокарде желудочков [66]. При этом показан более высокий риск развития COVID-19 и осложнений со стороны сердечно-сосудистой системы у лиц, страдающих заболеваниями данной системы [83, 84, 88].
Следует отметить, что уровень экспрессии ACE2 в соскобах эпителиальных клеток носа и бронхов у пациентов с бронхиальной астмой и респираторными аллергиями был снижен и показал обратно-пропорциональную зависимость от тяжести клинической картины (экспрессия ACE2 была самой низкой у пациентов с высоким уровнем аллергической сенсибилизации и с аллергической астмой), но неатопическая астма не была связана с пониженной экспрессией данного гена [89]. Авторы отмечают, что эти данные могут объяснить такое неожиданное клиническое наблюдение, как более мягкое течение COVID-19 у пациентов с респираторной аллергией, и в то же время подчеркивают, что экспрессия ACE2 – это один из факторов, который может влиять на ответ данных лиц на инфицирование SARS-CoV-2. Действительно, в бронхах при аллергической астме наблюдали увеличение экспрессии генов DPP4 и ANPEP [90], что также может влиять на восприимчивость к SARS-CoV-2. Кроме того, в легких регистрируется экспрессия всех привлеченных к рассмотрению генов-кандидатов восприимчивости к SARS-CoV-2, и для многих генов установлены eQTL, влияющие на уровень экспрессии в легких (рис. 1). Возможно, именно с генетическими особенностями территориальных групп населения связана противоречивость результатов в отношении влияния астмы на риск развития и характер течения COVID-19. Так, Z. Zhu с соавт. [91] отмечают, что взрослые с астмой имеют более высокий риск тяжелого COVID-19, причем повышенный риск наблюдался именно среди пациентов с “неаллергической” астмой.
Повышение уровня экспрессии гена DPP4 зарегистрировано в альвеолярном эпителии и альвеолярных макрофагах у пациентов с хронической обструктивной болезнью легких (ХОБЛ), с муковисцидозом [92], при инсулинорезистентности (показано на модельных животных и в культуре клеток) [93] и в условиях гипоксии [47]. Повышенный уровень белка DPP4 рассматривается в качестве предиктора возникновения метаболического синдрома [86]. У лиц с сахарным диабетом типа 2 (СД2) по сравнению со здоровыми индивидами в плазме наблюдали более высокий уровень и активность DPP4; повышенный уровень данного белка (но не активность) регистрировался также у пациентов с СД2 и ожирением по сравнению с СД2 без ожирения [94].
Модифицирующие эффекты на регуляцию работы генов-кандидатов восприимчивости к SARS-CoV-2 и синтез кодируемых ими белков может оказывать прием лекарственных препаратов. Так, уровень экспрессии гена TMPRSS11D в биоптатах легких пациентов с ХОБЛ зависел от приема лекарственных препаратов (флутиказона и сальметерола – комбинация ингаляционных стероидов и бета-агонистов длительного действия) [95]. Между пациентами с воспалительными заболеваниями кишечника и здоровыми индивидами не выявлено различий по уровню экспрессии генов ACE2 и TMPRSS2 в подвздошной кишке и толстой кишке, но медикаментозная терапия у пациентов (в частности, прием блокаторов фактора некроза опухолей) приводила к снижению уровня экспрессии ACE2 [96].
Уровень экспрессии генов-кандидатов восприимчивости к SARS-CoV-2 может модифицироваться наличием в организме других вирусов, причем направленность изменения экспрессии зависит от инфекционного агента. Несколько неожиданным является то, что некоторые вирусы (в частности, вирус птичьего гриппа H5N1) увеличивают экспрессию генов IFITM1, IFITM2 и IFITM3 [97], обладающих противовирусным эффектом [57–59, 63]. В то же время вирусы папилломы человека высокого онкогенного риска (hrHPV) подавляют экспрессию гена IFITM1 в кератиноцитах уже через 48 ч после инфицирования [98]. Возможно, изменение уровня экспрессии при воздействии вирусов зависит не только от инфекционного агента, но и от длительности инфекционного процесса, а также от генетических особенностей организма и его функционального состояния на момент инфицирования.
В качестве модифицирующих функционирование генов-кандидатов восприимчивости к SARS-CoV-2 могут выступать и другие экзогенные факторы. В легких у курильщиков выявлено повышение экспрессии генов ACE2 и FURIN, но на уровень экспрессии гена TMRPSS2 курение влияния не оказывало [99]. Курение ассоциировано с увеличением экспрессии гена ACE2 в жировой ткани [100]. Некоторые исследователи предполагают, что жировая ткань у индивидов с ожирением может выступать в качестве источника для более широкого распространения вирусов, повышенного их выделения, иммунной активации и амплификации цитокинов [64].
В ряде работ показано влияние курения на уровень метилирования сайта сg23161492, расположенного в 1-м интроне гена ANPEP (курение снижает уровень метилирования ДНК) в лейкоцитах крови [101–104]. Изменение уровня метилирования данного CpG-сайта сказывается на экспрессии гена ANPEP в ткани легкого [105]. Интересно, что для сg23161492 установлено шесть генетических вариантов (rs28565347, rs12442778, rs12440213, rs8030857, rs8031576 и rs748508), которые могут влиять на паттерн метилирования CpG-сайтов (mQTLs) в лейкоцитах крови [106]. Уровень метилирования другого cg05312779, локализованного в 3'UTR гена ANPEP, статистически значимо ассоциирован с функцией легких, в частности увеличение метилирования ДНК было связано со снижением индекса Тиффно у девушек-подростков [107]. Эти данные согласуются с клиническими наблюдениями, согласно которым курильщики имеют более высокий риск развития симптомов COVID-19 при инфицировании SARS-CoV-2 [100]. Однако оценки влияния курения на риск развития COVID-19 неоднозначны (см., например, [65]), что еще раз подчеркивает сложность регуляции ответа организма-хозяина на инфицирование SARS-CoV-2.
В качестве фактора, модулирующего ответ на инфицирование SARS-CoV-2, рассматривают также нарушение уровня метилирования ДНК и в других генах [108]. Предполагают, что гипометилирование регуляторных участков гена ACE2 в CD4+ T-клетках у пациентов с системной красной волчанкой, вследствие вызванного вирусной инфекцией окислительного стресса, может приводить к увеличению экспрессии гена АСЕ2 [108]. Гипометилирование CpG-сайтов и высокий уровень экспрессии гена ACE2 характерны для многих онкозаболеваний [109], и это может объяснять высокий риск осложнений у пациентов с COVID-19 и такими сопутствующими патологиями [83].
Таким образом, несмотря на непродолжительный период исследований, к настоящему времени накоплены данные, свидетельствующие о том, что восприимчивость и характер течения инфекции, вызванной SARS-CoV-2 могут определяться генетическими особенностями коронавируса и организма человека. Структурно-функциональные особенности S-белка SARS-CoV-2 и генов человека, кодирующих рецепторы, протеазы и другие белки и ферменты, значимые для проникновения и репликации коронавируса, а также модифицирующие функционирование данных генов факторы (в том числе – наличие сопутствующих заболеваний, вредных привычек, инфицирование другими вирусами, прием лекарственных препаратов и др.) могут определять на индивидуальном уровне восприимчивость и клиническую картину COVID-19, а на популяционном – особенности эпидемиологической ситуации.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
WHO Coronavirus Disease (COVID-19) Dashboard [Electronic resource]. URL: https://covid19.who. int/. Accessed 23.06.2020.
Malik Y.A. Properties of coronavirus and SARS-CoV-2 // Malays J. Pathol. 2020. V. 42(1). P. 3–11.
de Wit E., van Doremalen N., Falzarano D. SARS and MERS: Recent insights into emerging coronaviruses // Nat. Rev. Microbiol. 2016. V. 14. P. 523–534. https://doi.org/10.1038/nrmicro.2016.81
Liu J., Zheng X., Tong Q. et al. Overlapping and discrete aspects of the pathology and pathogenesis of the emerging human pathogenic coronaviruses SARS-CoV, MERS-CoV, and 2019-nCoV // J. Med. Virol. 2020. V. 92(5). P. 491–494. https://doi.org/10.1002/jmv.25709
Tu Y.F., Chien C.S., Yarmishyn A.A. et al. Review of SARS-CoV-2 and the ongoing clinical trials // Int. J. Mol. Sci. 2020. V. 21(7). pii: E2657. https://doi.org/10.3390/ijms21072657
Zimmermann P., Curtis N. Coronavirus infections in children including COVID-19: An overview of the epidemiology, clinical features, diagnosis, treatment and prevention options in children // Pediatr. Infect. Dis. J. 2020. V. 39(5). P. 355–368. https://doi.org/10.1097/INF.0000000000002660
Park S.E. Epidemiology, virology, and clinical features of severe acute respiratory syndrome – coronavirus-2 (SARS-CoV-2; Coronavirus Disease-19) // Clin. Exp. Pediatr. 2020. V. 63(4). P. 119–124. https://doi.org/10.3345/cep.2020.00493
Goyal P., Choi J.J., Pinheiro L.C. et al. Clinical characteristics of Covid-19 in New York City // N. Engl. J. Med. 2020. V. 382(24). P. 2372–2374. https://doi.org/10.1056/NEJMc2010419
Liu K., Chen Y., Lin R., Han K. Clinical features of COVID-19 in elderly patients: A comparison with young and middle-aged patients // J. Infect. 2020. V. 80(6). e14–e18. https://doi.org/10.1016/j.jinf.2020.03.005
Li W., Zhang C., Sui J. et al. Receptor and viral determinants of SARS-coronavirus adaptation to human ACE2 // EMBO J. 2005. V. 24(8). P. 1634–1643. https://doi.org/10.1038/sj.emboj.7600640
Li X., Geng M., Peng Y. et al. Molecular immune pathogenesis and diagnosis of COVID-19 // J. Pharm. Anal. 2020. V. 10(2). P. 102–108. https://doi.org/10.1016/j.jpha.2020.03.001
Zhang Y., Qin L., Zhao Y. et al. Interferon-induced transmembrane protein-3 genetic variant rs12252-C is associated with disease severity in COVID-19 // J. Infect. Dis. 2020. pii: jiaa224. https://doi.org/10.1093/infdis/jiaa224
Clohisey S., Baillie J.K. Host susceptibility to severe influenza A virus infection // Critical Care (London, England). 2019. V. 23(1): 303. https://doi.org/10.1186/s13054-019-2566-7
The UniProt Consortium. UniProt: the universal protein knowledgebase // Nucl. Acids Res. 2017. V. 45. D158–D169 [Electronic resource]. URL: http:// www.uniprot.org/. Accessed 10.2019.
QuickGO. [Electronic resource]. URL: https://www. ebi.ac.uk/QuickGO/. Accessed 06.2020.
Chen Y., Guo Y., Pan Y., Zhao Z.J. Structure analysis of the receptor binding of 2019-nCoV // Biochem. Biophys. Res. Commun. 2020. V. 525(1). P. 135–140. https://doi.org/10.1016/j.bbrc.2020.02.071
Zhou P., Yang X.-L., Wang X.-G. et al. Pneumonia outbreak associated with a new coronavirus of probable bat origin // Nature. 2020. V. 579(7798). P. 270–273. https://doi.org/10.1038/s41586-020-2012-7
Lu R., Zhao X., Li J. et al. Genomic characterisation and epidemiology of 2019 novel coronavirus: Implications for virus origins Wenjie and receptor binding // Lancet. 2020. V. 395(10224). P. 565–574. https://doi.org/10.1016/S0140-6736(20)30251-8
Saha P., Banerjee A.K., Tripathi P.P. et al. A virus that has gone viral: Amino-acid mutation in S protein of Indian-isolate of Coronavirus COVID-19 might impact receptor-binding, thus infectivity // Biosci. Rep. 2020. V. 40(5). pii: BSR20201312. https://doi.org/10.1042/BSR20201312
Yin C. Genotyping coronavirus SARS-CoV-2: methods and implications // Genomics. 2020. pii: S0888-7543(20)30318-9. https://doi.org/10.1016/j.ygeno.2020.04.016
Kandeel M., Ibrahim A., Fayez M., Al-Nazawi M. From SARS and MERS CoVs to SARS-CoV-2: Moving toward more biased codon usage in viral structural and nonstructural genes // J. Med. Virol. 2020. V. 92(6). P. 660–666. https://doi.org/10.1002/jmv.25754
Pillay T.S. Gene of the month: the 2019-nCoV/SARS-CoV-2 novel coronavirus spike protein // J. Clin. Pathol. 2020. V. 73(7). P. 366–369. https://doi.org/10.1136/jclinpath-2020-206658
Zhu Y., Yu D., Yan H. et al. Design of potent membrane fusion inhibitors against SARS-CoV-2, an emerging coronavirus with high fusogenic activity // J. Virol. 2020. V. 94(14):e00635-20. https://doi.org/10.1128/JVI.00635-20
Hofmann H., Pyrc K., van der Hoek L. et al. Human coronavirus NL63 employs the severe acute respiratory syndrome coronavirus receptor for cellular entry // PNAS USA. 2005. V. 102(22). P. 7988–7993. https://doi.org/10.1073/pnas.0409465102
Walls A.C., Park Y.J., Tortorici M.A. et al. Structure, function, and antigenicity of the SARS-CoV-2 spike glycoprotein // Cell. 2020. V. 181(2). P. 281–292.e6. https://doi.org/10.1016/j.cell.2020.02.058
Yeager C.L., Ashmun R.A., Williams R.K. et al. Human aminopeptidase N is a receptor for human coronavirus 229E // Nature. 1992. V. 357(6377). P. 420–422. https://doi.org/10.1038/357420a0
Li Y., Zhang Z., Yang L. et al. The MERS-CoV receptor DPP4 as a candidate binding target of the SARS-CoV-2 spike // iScience. 2020. V. 23(6): 101160. https://doi.org/10.1016/j.isci.2020.101160
Wang Q., Zhang Y., Wu L. et al. Structural and functional basis of SARS-CoV-2 entry by using human ACE2 // Cell. 2020. V. 181(4). P. 894–904.e9. https://doi.org/10.1016/j.cell.2020.03.045
Shang J., Wan Y., Luo C. et al. Cell entry mechanisms of SARS-CoV-2 // PNAS USA. 2020. V. 117(21). P. 11727–11734. https://doi.org/10.1073/pnas.2003138117
Xia S., Liu M., Wang C. et al. Inhibition of SARS-CoV-2 (previously 2019-nCoV) infection by a highly potent pan-coronavirus fusion inhibitor targeting its spike protein that harbors a high capacity to mediate membrane fusion // Cell Res. 2020. V. 30(4). P. 343–355. https://doi.org/10.1038/s41422-020-0305-x
Chang T.J., Yang D.M., Wang M.L. et al. Genomic analysis and comparative multiple sequence of SARS-CoV2 // J. Chin. Med. Assoc. 2020. V. 83(6). P. 537–543. https://doi.org/10.1097/JCMA.0000000000000335
Garten W., Braden C., Arendt A. et al. Influenza virus activating host proteases: Identification, localization and inhibitors as potential therapeutics // Eur. J. Cell. Biol. 2015. V. 94(7–9). P. 375–383. https://doi.org/10.1016/j.ejcb.2015.05.013
Shirato K., Kanou K., Kawase M., Matsuyama S. Clinical isolates of human coronavirus 229E bypass the endosome for cell entry // J. Virol. 2016. V. 91(1). pii: e01387-16. https://doi.org/10.1128/JVI.01387-16
Shulla A., Heald-Sargent T., Subramanya G. et al. A transmembrane serine protease is linked to the severe acute respiratory syndrome coronavirus receptor and activates virus entry // J. Virol. 2011. V. 85(2). P. 873–882. https://doi.org/10.1128/JVI.02062-10
Heurich A., Hofmann-Winkler H., Gierer S. et al. TMPRSS2 and ADAM17 cleave ACE2 differentially and only proteolysis by TMPRSS2 augments entry driven by the severe acute respiratory syndrome coronavirus spike protein // J. Virol. 2014. V. 88(2). P. 1293–1307. https://doi.org/10.1128/JVI.02202-13
Reinke L.M., Spiegel M., Plegge T. et al. Different residues in the SARS-CoV spike protein determine cleavage and activation by the host cell protease TMPRSS2 // PLoS One. 2017. V. 12(6): e0179177. https://doi.org/10.1371/journal.pone.0179177
Coutard B., Valle C., de Lamballerie X. et al. The spike glycoprotein of the new coronavirus 2019-nCoV contains a furin-like cleavage site absent in CoV of the same clade // Antiviral Res. 2020. V. 176: 104742. https://doi.org/10.1016/j.antiviral.2020.104742
Rabaan A.A., Al-Ahmed S.H., Haque S. et al. SARS-CoV-2, SARS-CoV, and MERS-COV: A comparative overview // Infez. Med. 2020. V. 28(2). P. 174–184.
Hoffmann M., Kleine-Weber H., Schroeder S. et al. SARS-CoV-2 Cell entry depends on ACE2 and TMPRSS2 and is blocked by a clinically proven protease inhibitor // Cell. 2020. V. 181(2). P. 271–280. https://doi.org/10.1016/j.cell.2020.02.052
Williams F.M.K., Freydin M., Mangino M. et al. Self-reported symptoms of covid-19 including symptoms most predictive of SARS-CoV-2 infection, are heritable // medRxiv. The Preprint server for Health Sciences. 2020. https://www.medrxiv.org/. https://doi.org/10.1101/2020.04.22.20072124
Zietz M., Tatonetti N.P. Testing the association between blood type and COVID-19 infection, intubation, and death // medRxiv. The Preprint server for Health Sciences 2020. V. 2020. https://doi.org/10.1101/2020.04.08.20058073
Zaidi F.Z., Zaidi A.R.Z., Abdullah S.M., Zaidi S.Z.A. COVID-19 and the ABO blood group connection // Transfus. Apher Sci. 2020. V. 59(5): 102838. https://doi.org/10.1016/j.transci.2020.102838
Genome Aggregation Database (gnomAD). [Electronic resource]. https://gnomad.broadinstitute.org/. Accessed 06.2020.
Ensembl Genome Browser 88. [Electronic resource]. https://www.ensembl.org/. Accessed 05.2020.
Genotype-Tissue Expression (GTEx) project. [Electronic resource]. https://www.gtexportal.org/home/. Accessed 05.2020.
GTEx Consortium. The Genotype-Tissue Expression (GTEx) project // Nat. Genet. 2013. V. 45(6). P. 580–585. https://doi.org/10.1038/ng.2653
Li Y., Yang L., Dong L. et al. Crosstalk between the Akt/mTORC1 and NF-κB signaling pathways promotes hypoxia-induced pulmonary hypertension by increasing DPP4 expression in PASMCs // Acta Pharmacol. Sin. 2019. V. 40(10). P. 1322–1333. https://doi.org/10.1038/s41401-019-0272-2
Vankadari N., Wilce J.A. Emerging COVID-19 coronavirus: glycan shield and structure prediction of spike glycoprotein and its interaction with human CD26 // Emerg. Microbes Infect. 2020. V. 9(1). P. 601–604. https://doi.org/10.1080/22221751.2020.1739565
Raj V.S., Mou H., Smits S.L. et al. Dipeptidyl peptidase 4 is a functional receptor for the emerging human coronavirus-EMC // Nature. 2013. V. 495(7440). P. 251–254. https://doi.org/10.1038/nature12005
Kolb A.F., Maile J., Heister A., Siddell S.G. Characterization of functional domains in the human coronavirus HCV 229E receptor // J. Gen. Virol. 1996. V. 77. Pt. 10. P. 2515–2521. https://doi.org/10.1099/0022-1317-77-10-2515
Bestle D., Heindl M.R., Limburg H. et al. TMPRSS2 and furin are both essential for proteolytic activation and spread of SARS-CoV-2 in human airway epithelial cells and provide promising drug targets (Preliminary Report) // BioRxiv. The Preprint server for Biology. 2020. https://www.biorxiv.org/. https://doi.org/10.1101/2020.04.15.042085
Bertram S., Dijkman R., Habjan M. et al. TMPRSS2 activates the human coronavirus 229E for cathepsin-independent host cell entry and is expressed in viral target cells in the respiratory epithelium // J. Virol. 2013. V. 87(11). P. 6150–6160. https://doi.org/10.1128/JVI.03372-12
Matsuyama S., Nao N., Shirato K. et al. Enhanced isolation of SARS-CoV-2 by TMPRSS2-expressing cells // PNAS USA. 2020. V. 117(13). P. 7001–7003. https://doi.org/10.1073/pnas.2002589117
Shirato K., Kawase M., Matsuyama S. Middle East respiratory syndrome coronavirus infection mediated by the transmembrane serine protease TMPRSS2 // J. Virol. 2013. V. 87(23). P. 12552–12561. https://doi.org/10.1128/JVI.01890-13
Simmons G., Gosalia D.N., Rennekamp A.J. et al. Inhibitors of cathepsin L prevent severe acute respiratory syndrome coronavirus entry // PNAS USA. 2005. V. 102(33). P. 11876–11881. https://doi.org/10.1073/pnas.0505577102
Xu L., Khadijah S., Fang S. et al. The cellular RNA helicase DDX1 interacts with coronavirus nonstructural protein 14 and enhances viral replication // J. Virol. 2010. V. 84(17). P. 8571–8583. https://doi.org/10.1128/JVI.00392-10
Huang I.C., Bailey C.C., Weyer J.L. et al. Distinct patterns of IFITM-mediated restriction of filoviruses, SARS coronavirus, and influenza A virus // PLoS Pathog. 2011. V. 7(1): e1001258. https://doi.org/10.1371/journal.ppat.1001258
Jiang D., Weidner J.M., Qing M. et al. Identification of five interferon-induced cellular proteins that inhibit west nile virus and dengue virus infections // J. Virol. 2010. V. 84(16). P. 8332–8341. https://doi.org/10.1128/JVI.02199-09
Amini-Bavil-Olyaee S., Choi Y.J., Lee J.H. et al. The antiviral effector IFITM3 disrupts intracellular cholesterol homeostasis to block viral entry // Cell Host Microbe. 2013. V. 13(4). P. 452–464. https://doi.org/10.1016/j.chom.2013.03.006
Wrapp D., Wang N., Corbett K.S. et al. Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation // Science. 2020. V. 367(6483). P. 1260–1263. https://doi.org/10.1126/science.abb2507
Qi F., Qian S., Zhang S., Zhang Z. Single cell RNA sequencing of 13 human tissues identify cell types and receptors of human coronaviruses // Biochem. Biophys. Res. Commun. 2020. V. 526(1). P. 135–140. https://doi.org/10.1016/j.bbrc.2020.03.044
Iwata-Yoshikawa N., Okamura T., Shimizu Y. et al. TMPRSS2 contributes to virus spread and immunopathology in the airways of murine models after coronavirus infection // J. Virol. 2019. V. 93(6). e01815-18. https://doi.org/10.1128/JVI.01815-18
Brass A.L., Huang I.C., Benita Y. et al. The IFITM proteins mediate cellular resistance to influenza A H1N1 virus, West Nile virus, and dengue virus // Cell. 2009. V. 139(7). P. 1243–1254. https://doi.org/10.1016/j.cell.2009.12.017
Ryan P.M., Caplice N.M. Is adipose tissue a reservoir for viral spread, immune activation and cytokine amplification in COVID-19 // Obesity (Silver Spring). 2020. V. 28(7). P. 1191–1194. https://doi.org/10.1002/oby.22843
Gaibazzi N., Tuttolomondo D., Guidorossi A. et al. Smoking prevalence is low in symptomatic patients admitted for COVID-19. // medRxiv. The Preprint server for Health Sciences. 2020. https://www.medrxiv.org//. https://doi.org/10.1101/2020.05.05.20092015
Goulter A.B., Goddard M.J., Allen J.C., Clark K.L. ACE2 gene expression is up-regulated in the human failing heart // BMC Med. 2004. V. 2: 19. https://doi.org/10.1186/1741-7015-2-19
Darbani B. The expression and polymorphism of entry machinery for COVID-19 in human: juxtaposing population groups, gender, and different tissues // Int. J. Environ. Res. Public Health. 2020. V. 17(10): 3433. https://doi.org/10.3390/ijerph17103433
Bertram S., Heurich A., Lavender H. et al. Influenza and SARS-coronavirus activating proteases TMPRSS2 and HAT are expressed at multiple sites in human respiratory and gastrointestinal tracts // PLoS One. 2012. V. 7(4). e35876. https://doi.org/10.1371/journal.pone.0035876
Fu J., Zhou B., Zhang L. et al. Expressions and significances of the angiotensin-converting enzyme 2 gene, the receptor of SARS-CoV-2 for COVID-19 // Mol. Biol. Rep. 2020. May 14. P. 1–10. https://doi.org/10.1007/s11033-020-05478-4
Online Mendelian Inheritance in Man [Electronic resource]. URL: http:www.omim.org/. Accessed 06.2020.
Everitt A.R., Clare S., Pertel T. et al. IFITM3 restricts the morbidity and mortality associated with influenza // Nature. 2012. V. 484. P. 519–523. https://doi.org/10.1038/nature10921
Weidner J.M., Jiang D., Pan X.-B. et al. Interferon-induced cell membrane proteins, IFITM3 and tetherin, inhibit vesicular stomatitis virus infection via distinct mechanisms // J. Virol. 2010. V. 84(24). P. 12646–12657. https://doi.org/10.1128/JVI.01328-10
Cheng Z., Zhou J., To K.K. et al. Identification of TMPRSS2 as a susceptibility gene for severe 2009 pandemic A(H1N1) influenza and A(H7N9) influenza // J. Infect. Dis. 2015. V. 212(8). P. 1214–1221. https://doi.org/10.1093/infdis/jiv246
EBI Search. [Electronic resource]. https://www.ebi. ac.uk/ebisearch/overview.ebi/about. Accessed 06.2020.
van der Harst P., Verweij N. Identification of 64 novel genetic loci provides an expanded view on the genetic architecture of Coronary Artery Disease // Circulation Research. 2017. V. 122(3). P. 433–443. https://doi.org/10.1161/circresaha.117.312086
Nikpay M., Goel A., Won H.H. et al. A comprehensive 1,000 Genomes-based genome-wide association meta-analysis of coronary artery disease // Nat. Genet. 2015. V. 47(10). P. 1121–1130. https://doi.org/10.1038/ng.3396
Takeuchi F., Akiyama M., Matoba N. et al. Interethnic analyses of blood pressure loci in populations of East Asian and European descent // Nat. Communications. 2018. V. 9(1): 5052. https://doi.org/10.1038/s41467-018-07345-0
Yang X., Yang W., McVey D.G. et al. FURIN expression in vascular endothelial cells is modulated by a coronary artery disease–associated genetic variant and influences monocyte transendothelial migration // J. Am. Heart Assoc. 2020. V. 9(4). e014333. https://doi.org/10.1161/JAHA.119.014333
Li B., Yang J., Zhao F. et al. Prevalence and impact of cardiovascular metabolic diseases on COVID-19 in China // Clin. Res. Cardiol. 2020. V. 109(5). P. 531–538. https://doi.org/10.1007/s00392-020-01626-9
Mbewe-Campbell N., Wei Z., Zhang K. et al. Genes and environment: novel, functional polymorphism in the human cathepsin L (CTSL1) promoter disrupts a xenobiotic response element (XRE) to alter transcription and blood pressure // J. Hypertens. 2012. V. 30(10). P. 1961–1969. https://doi.org/10.1097/HJH.0b013e328356b86a
Afsar C.U. 2019-nCoV-SARS-CoV-2 (COVID-19) infection: Crucıalıty of Furın and relevance wıth cancer // Med. Hypotheses. 2020. V. 140: 109770. https://doi.org/10.1016/j.mehy.2020.109770
Paniri A., Hosseini M.M., Akhavan-Niaki H. First comprehensive computational analysis of functional consequences of TMPRSS2 SNPs in susceptibility to SARS-CoV-2 among different populations // J. Biomol. Struct. Dyn. 2020. P. 1–18. https://doi.org/10.1080/07391102.2020.1767690
Mason K.E., McHale P., Pennington A. Age-adjusted associations between comorbidity and outcomes of COVID-19: a review of the evidence. // medRxiv. The Preprint server for Health Sciences. 2020. https://www.medrxiv.org/. https://doi.org/10.1101/2020.05.06.20093351
Lippi G., Wong J., Henry B.M. Hypertension in patients with coronavirus disease 2019 (COVID-19): A pooled analysis // Pol. Arch. Intern. Med. 2020. V. 130(4). P. 304–309. https://doi.org/10.20452/pamw.15272
Bilinska K., Jakubowska P., Von Bartheld C.S., Butowt R. Expression of the SARS-CoV-2 entry proteins, ACE2 and TMPRSS2, in cells of the olfactory epithelium: identification of cell types and trends with age // ACS Chem. Neurosci. 2020. V. 11(11). P. 1555–1562. https://doi.org/10.1021/acschemneuro.0c00210
Bassendine M.F., Bridge S.H., McCaughan G.W., Gorrell M.D. Covid-19 and co-morbidities: a role for Dipeptidyl Peptidase 4 (DPP4) in disease severity? // J. Diabetes. 2020. V. 12(9). P. 649–658. https://doi.org/10.1111/1753-0407.13052
Kakimoto Y., Sasaki A., Niioka M. et al. Myocardial cathepsin D is downregulated in sudden cardiac death // PLoS One. 2020. V. 15(3). e0230375. https://doi.org/10.1371/journal.pone.0230375
Siordia J.A., Jr. Epidemiology and clinical features of COVID-19: A review of current literature // J. Clin. Virol. 2020. V. 127. 104357. https://doi.org/10.1016/j.jcv.2020.104357
Jackson D.J., Busse W.W., Bacharier L.B. et al. Association of respiratory allergy, asthma, and expression of the SARS-CoV-2 receptor ACE2 // J. Allergy Clin. Immunol. 2020. V. 146(1). P. 203–206.е3. https://doi.org/10.1016/j.jaci.2020.04.009
van der Velden V.H., Wierenga-Wolf A.F., Adriaansen-Soeting P.W. et al. Expression of aminopeptidase N and dipeptidyl peptidase IV in the healthy and asthmatic bronchus // Clin. Exp. Allergy. 1998. V. 28(1). P. 110–120. https://doi.org/10.1046/j.1365-2222.1998.00198.x
Zhu Z., Hasegawa K., Ma B. et al. Association of asthma and its genetic predisposition with the risk of severe COVID-19 // J. Allergy Clin. Immunol. 2020. V. 146(2). P. 327–329.e4. https://doi.org/10.1016/j.jaci.2020.06.001
Meyerholz D.K., Lambertz A.M., McCray P.B., Jr. Dipeptidyl peptidase 4 distribution in the human respiratory tract: Implications for the Middle East Respiratory Syndrome // Am. J. Pathol. 2016. V. 186(1). P. 78–86. https://doi.org/10.1016/j.ajpath.2015.09.014
Cheng F., Yuan G., He J. et al. Dysregulation of DPP4 is associated with the AMPK/JAK2/STAT3 pathway in adipocytes under insulin resistance status and liraglutide intervention // Diabetes Metab. Syndr. Obes. 2019. V. 12. P. 2635–2644. https://doi.org/10.2147/DMSO.S229838
Sarkar J., Nargis T., Tantia O. et al. Increased plasma dipeptidyl peptidase-4 (DPP4) activity is an obesity-independent parameter for glycemic deregulation in Type 2 Diabetes patients // Front. Endocrinol. (Lausanne). 2019. V. 10: 505. https://doi.org/10.3389/fendo.2019.00505
van den Berge M., Steiling K., Timens W. et al. Airway gene expression in COPD is dynamic with inhaled corticosteroid treatment and reflects biological pathways associated with disease activity // Thorax. 2014. V. 69(1). P. 14–23. https://doi.org/10.1136/thoraxjnl-2012-202878
Burgueno J.F., Reich A., Hazime H. et al. Expression of SARS-CoV-2 entry molecules ACE2 and TMPRSS2 in the gut of patients with IBD // Inflamm Bowel. Dis. 2020. V. 26(6). P. 797–808. https://doi.org/10.1093/ibd/izaa085
Wang H., Chen L., Luo J., He H. NP and NS1 proteins of H5N1 virus significantly upregulated IFITM1, IFITM2, and IFITM3 in A549 cells // Afr. Health Sci. 2019. V. 19(1). P. 1402–1410. https://doi.org/10.4314/ahs.v19i1.13
Ma W., Tummers B., van Esch E.M. et al. Human papillomavirus downregulates the expression of IFITM1 and RIPK3 to escape from IFNγ- and TNFα-mediated antiproliferative effects and necroptosis // Front Immunol. 2016. V. 7: 496. https://doi.org/10.3389/fimmu.2016.00496
Cai G., Bossé Y., Xiao F. et al. Tobacco smoking increases the lung gene expression of ACE2, the receptor of SARS-CoV-2 // Am. J. Respir. Crit. Care Med. 2020. V. 201(12). P. 1557–1559. https://doi.org/10.1164/rccm.202003-0693LE
Hopkinson N.S., Rossi N., El-Sayed Moustafa J. et al. Current tobacco smoking and risk from COVID-19: results from a population symptom app in over 2.4 million people // medRxiv. The Preprint server for Health Sciences. 2020. https://www.medrxiv.org/. https://doi.org/10.1101/2020.05.18.20105288
Zhang X., Hu Y., Aouizerat B.E. et al. Machine learning selected smoking-associated DNA methylation signatures that predict HIV prognosis and mortality // Clin. Epigenetics. 2018. V. 10: 155. https://doi.org/10.1186/s13148-018-0591-z
Li S., Wong E.M., Bui M. et al. Causal effect of smoking on DNA methylation in peripheral blood: a twin and family study // Clin. Epigenetics. 2018. V. 10: 18. https://doi.org/10.1186/s13148-018-0452-9
Zeilinger S., Kühnel B., Klopp N. et al. Tobacco smoking leads to extensive genome-wide changes in DNA methylation // PLoS One. 2013. V. 8(5): e63812. https://doi.org/10.1371/journal.pone.0063812
Sayols-Baixeras S., Lluís-Ganella C., Subirana I. et al. Identification of a new locus and validation of previously reported loci showing differential methylation associated with smoking. The REGICOR study // Epigenetics. 2015. V. 10(12). P. 1156–1165. https://doi.org/10.1080/15592294.2015.1115175
Lee M.K., Hong Y., Kim S.-Y. et al. DNA methylation and smoking in Korean adults: epigenome-wide association study // Clin. Epigenetics. 2016. V. 8: 103. https://doi.org/10.1186/s13148-016-0266-6
Gao X., Thomsen H., Zhang Y. et al. The impact of methylation quantitative trait loci (mQTLs) on active smoking-related DNA methylation changes // Clin. Epigenetics. 2017. V. 9: 87. https://doi.org/10.1186/s13148-017-0387-6
Sunny S.K., Zhang H., Rezwan F.I. et al. Changes of DNA methylation are associated with changes in lung function during adolescence // Respir. Res. 2020. V. 21(1): 80. https://doi.org/10.1186/s12931-020-01342-y
Sawalha A.H., Zhao M., Coit P., Lu Q. Epigenetic dysregulation of ACE2 and interferon-regulated genes might suggest increased COVID-19 susceptibility and severity in lupus patients // Clin. Immunol. 2020. V. 215: 108410. https://doi.org/10.1016/j.clim.2020.108410
Chai P., Yu J., Ge S. et al. Genetic alteration, RNA expression, and DNA methylation profiling of coronavirus disease 2019 (COVID-19) receptor ACE2 in malignancies: a pan-cancer analysis // J. Hematol. Oncol. 2020. V. 13(1): 43. https://doi.org/10.1186/s13045-020-00883-5
Дополнительные материалы отсутствуют.


