Генетика, 2021, T. 57, № 7, стр. 737-751
Регуляторы элонгации и архитектурные белки – новые участники транскрипции генов эукариот
Н. Е. Воробьева 1, М. Ю. Мазина 1, *
1 Институт биологии гена Российской академии наук
119334 Москва, Россия
* E-mail: mazinam@genebiology.ru
Поступила в редакцию 03.08.2020
После доработки 16.10.2020
Принята к публикации 27.10.2020
Аннотация
До последнего времени основными белками-корегуляторами транскрипции принято было считать комплексы, изменяющие структуру хроматина, – ремоделеры и модификаторы гистонов. Однако новейшие исследования механизмов регуляции транскрипции позволили внести уточнения в данную классификацию. Были выделены два дополнительных класса транскрипционных корегуляторов: регуляторы элонгации и белки/белковые комплексы, изменяющие трехмерную структуру хроматина. В данном обзоре мы суммировали наиболее свежую информацию о возможностях регулирования транскрипции генов эукариот посредством влияния на элонгацию и трехмерную структуру хроматина. Мы обсуждаем такие актуальные вопросы, как регуляция “паузинга” РНК-полимеразы II и скорости элонгации транскрипции, подавление криптической транскрипции в теле гена, а также роль белков/белковых комплексов в формировании обособленных топологических доменов и обеспечении связи между энхансерами и промоторами генов.
Многие годы основными регуляторами процесса транскрипции генов эукариот, помимо общих факторов транскрипции РНК-полимеразы II, принято было считать комплексы, ремоделирующие и модифицирующие хроматин в области генов. Однако недавние успехи в развитии полногеномных технологий позволили значительно расширить данные представления. Так, в ряде работ было показано, что для большинства многоклеточных организмов стадии инициации транскрипции предшествует состояние “паузинга” РНК-полимеразы II на промоторах генов. В активации транскрипции таких генов велика роль комплексов, регулирующих элонгацию: они влияют на выход РНК-полимеразы II из состояния “паузинга” на промоторе и переход к элонгации транскрипции. К данной группе белков относят не только комплексы, взаимодействующие с РНК-полимеразой II и общими факторами транскрипции на промоторе, но и шапероны гистонов, ремоделирующие нуклеосомы перед полимеразой в кодирующих областях генов и определяющие эффективность элонгации.
Широко известно, что изменение спектра контактов промоторов с другими регуляторными элементами (энхансерами, инсуляторами) влияет на экспрессию генов, однако долгое время данная информация была ограничена лишь отдельными генетическими примерами. На сегодняшний день с развитием полногеномных методов анализа конформации хроматина (Hi-C, HiChIP, ChIA-PET) появляется все больше экспериментальных данных о роли контактов в хроматине и белков, эти контакты поддерживающих, в регуляции транскрипции. С использованием таких данных становится возможным строить емкие модели влияния архитектурных белков на транскрипцию функциональных групп генов в различные этапы онтогенеза.
В данном обзоре мы попытались объединить самую современную информацию о функциях двух новых классов транскрипционных корегуляторов, регуляторов элонгации и архитектурных белков, в транскрипции.
ТРАНСКРИПЦИОННЫЕ КОМПЛЕКСЫ, РЕГУЛИРУЮЩИЕ ЭЛОНГАЦИЮ
Единственным общим транскрипционным регулятором, присутствующим у всех живых организмов от бактерий до человека и влияющим на процессивность транскрипции, является белок NusG/Spt5 [1, 2]. У многоклеточных организмов Spt5 функционирует в виде гетеродимера с Spt4, образуя регулятор элонгации транскрипции, названный DSIF (DRB-sensitive factor) [3]. Долгое время факторами, влияющими на скорость элонгации, интересовались достаточно мало, так как считали, что основной регуляторный потенциал представлен белками, стимулирующими привлечение РНК-полимеразы II. Со временем стало понятно, что на промоторах многих генов многоклеточных РНК-полимераза II рекрутируется заранее, в неактивном состоянии, приводя их в состояние транскрипционной “паузы” [4, 5]. Регуляция транскрипции таких генов осуществляется путем стимуляции элонгации. Экспериментальные исследования, продемонстрировавшие широкое распространение “паузы” транскрипции в геноме, привлекли внимание к регуляторам элонгации транскрипции, таким как DSIF. Фактор DSIF взаимодействует с РНК-полимеразой II на промоторе генов, стимулируя транскрипционную “паузу” путем стабилизации комплекса, образуемого ферментом с негативным фактором транскрипции NELF [6]. Молекулярный механизм работы фактора DSIF хорошо иллюстрирует сложность и многоступенчатость процесса транскрипции (рис. 1,а).
Рис. 1.
а – роль фактора DSIF в начальных процессах транскрипции генов; б – транскрипционные корегуляторы, ассоциированные с РНК-полимеразой на различных этапах транскрипции. Приведены комплексы, связанные с немодифицированной РНК-полимеразой сразу после ее рекрутирования, с РНК-полимеразой, фосфорилированной по Ser5 в составе CTD, которая инициировала транскрипцию и покинула промотор, а также с РНК-полимеразой, фосфорилированной по Ser2 в составе CTD, которая находится в стадии продуктивной элонгации; в – роль ковалентных модификаций хроматина, а также корегуляторов, ассоциированных с РНК-полимеразой, в ингибировании криптической транскрипции в теле гена в ходе элонгации. Для рис. 1 и 2: более детальное описание приведено в тексте. Там же приводятся ссылки на работы, послужившие основой для формирования молекулярных моделей процессов, ассоциированных с транскрипцией. Все модели были созданы при помощи приложения BioRender.com.
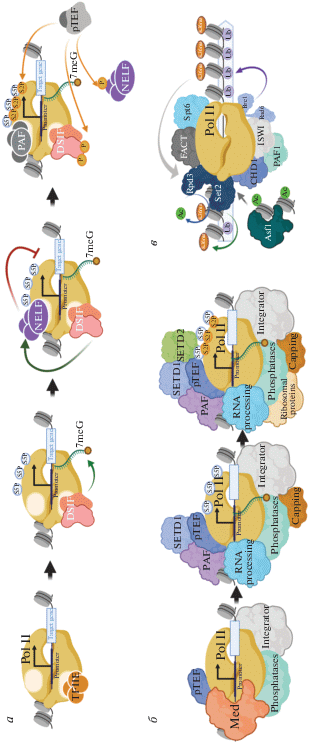
На стадии инициации транскрипции фактор DSIF не способен связать РНК-полимеразу II, так как поверхность его взаимодействия c ферментом занята общими факторами транскрипции (такими как TFIIE) [7, 8]. Связывание DSIF с РНК-полимеразой II стимулируется появлением новосинтезированной РНК, с которой взаимодействует данный фактор [9]. В свою очередь, DSIF играет важную роль в кэпировании новосинтезированной РНК для защиты ее от деградации [10]. У многоклеточных взаимодействие DSIF с РНК-полимеразой II стимулирует также связывание фактора NELF, который в свою очередь приводит к формированию репрессивной или временной транскрипционной “паузы” [6]. Выход РНК-полимеразы II из состояния “паузы” происходит путем фосфорилирования DSIF при помощи фактора p-TEFb, которое провоцирует отделение NELF и стимулирует элонгацию транскрипции [11]. Интересно, что p-TEFb фосфорилирует также NELF и саму РНК-полимеразу II, но главную роль в выходе из транскрипционной “паузы” играет именно фосфорилирование повторов в составе субъединицы Spt5 фактора DSIF. Отделение NELF от РНК-полимеразы II освобождает на ней поверхности для рекрутирования фактора TFIIS, необходимого для выхода из “паузы”, а также для связывания комплекса PAF1, позитивного регулятора элонгации транскрипции [12]. Таким образом, DSIF является активным участником ранних этапов транскрипции, начиная от синтеза первых нуклеотидов до образования продуктивного элонгирующего комплекса РНК-полимеразы II. В кодирующей области генов DSIF также играет важную роль, стимулируя процессивность РНК-полимеразы II, сплайсинг и образование мРНП [13–15].
Как видно из механизма работы DSIF, ключевым регулятором, разрушающим “паузу” транскрипции и провоцирующим переход комплекса РНК-полимеразы II в стадию продуктивной элонгации, является комплекс p-TEFb (состоящий из субъединиц CDK9 и CYCT). К настоящему моменту известно три основных способа рекрутирования p-TEFb к промоторам генов: в составе комплекса с Brd4, как часть Super Elongation Complex (SEC) или совместно c 7SK РНК. Исходно предполагалось, что именно Brd4 отвечает за привлечение комплекса p-TEFb к активно работающим промоторам путем ассоциации содержащихся в нем бромодоменов с ацетильными остатками гистонов [16]. Действительно, было показано, что активная фракция p-TEFb входит в состав комплекса с Brd4, а снижение внутриклеточного уровня белка Brd4 приводит к падению степени фосфорилирования РНК-полимеразы II по Ser2 в составе CTD (что является свидетельством нарушения работы CDK9). Однако недавно было показано, что нарушение связывания Brd4 с хроматином при помощи селективных ингибиторов не приводит к снижению уровня p-TEFb на промоторах генов [17]. Вероятно, основной функцией Brd4 во взаимодействии с p-TEFb является стимуляция функциональной активности, а не рекрутирование на промоторы. Именно способность Brd4 воздействовать на p-TEFb и, соответственно, элонгацию транскрипции сделала его перспективной фармацевтической мишенью. К настоящему моменту уже охарактеризован ряд селективных ингибиторов бромодоменов Brd4, многие из которых демонстрируют большой потенциал использования в качестве лекарственных препаратов [18, 19]. Также глубоко исследован механизм работы p-TEFb в составе комплекса SEC [20]. Дело в том, что гены, кодирующие субъединицы комплекса SEC, часто являются партнерами по транслокации с геном, кодирующим гистон-метилтрансферазу MLL [21]. Результатом такой транслокации является слитный белок, который провоцирует привлечение комплекса SEC (а вместе с ним и p-TEFb) к генам, регулируемым MLL-белком. Воздействие такого слитного регулятора приводит к высокоэффективной стимуляции элонгации транскрипции на генах-мишенях и развитию различных патологических состояний (в частности, наличие транслокаций гена MLL ассоциировано с острым лимфобластным и смешанным лейкозами) [22]. Альтернативным фактором, стимулирующим элонгацию транскрипции при помощи p-TEFb, является малая ядерная 7SK РНК. Исходно основной ролью 7SK РНК во взаимодействии с p-TEFb считалась регуляция активности p-TEFb посредством включения в неактивный киназный комплекс 7SK/HEXIM1/ p-TEFb [23]. Однако недавно было описано, что 7SK РНК участвует и в рекрутировании 7SK/ HEXIM1/p-TEFb-комплекса на хроматин: в ChIP-seq экспериментах было обнаружено, что данным комплексом обогащены промоторы более 15 000 генов человека [24]. Считается, что активация рекрутированного на хроматин комплекса 7SK/ HEXIM1/p-TEFb способствует быстрому повышению концентрации p-TEFb на промоторах за счет связывания комплекса ген-специфическими транскрипционными факторами.
Стоит отметить отдельно, что комплекс p‑TEFb является не только стимулятором выхода комплекса РНК-полимеразы II из состояния “паузы”, но и модулятором ее активности в процессе элонгации. Тот факт, что степень фосфорилирования Ser2 в составе CTD домена РНК-полимеразы II возрастает по мере продвижения ее по гену, свидетельствует об активности комплекса, ответственного за данный тип фосфорилирования на протяжении всего процесса элонгации. Вероятно, p-TEFb входит в состав элонгирующего комплекса РНК-полимеразы II. Это предположение подкрепляется и функциональными данными, свидетельствующими об активной роли p-TEFb в ранних и поздних этапах элонгации, а также его вкладе в регуляцию терминации транскрипции и полиаденилирования [25]. Регулирование степени фосфорилирования CTD РНК-полимеразы II, а также повторов в составе Spt5, используется клеткой для управления скоростью продвижения элонгирующего комплекса по гену. Отмечено, что снижение скорости транскрипции в сайте полиаденилирования связано со снижением степени фосфорилирования РНК-полимеразы II и Spt5. Замедление элонгирующего комплекса происходит под воздействием фосфатазы PP1, регулируемой CDK9. В сайте полиаденилирования PP1 дефосфорилирует Spt5 в составе DSIF, что приводит к замедлению продвижения РНК-полимеразы II по гену и провоцирует начало терминации транскрипции и полиаденилирования [26]. Отмечено, что поддержание определенной скорости продвижения РНК-полимеразы II по гену имеет существенное значение для правильного сворачивания мРНК [27] и альтернативного процессинга РНК. Действительно, в ряде работ показано, что изменение скорости элонгации РНК-полимеразы II влияет на выбор сайта сплайсинга, последующее включение экзонов в состав мРНК и на эффективность сплайсинга [28–30].
CTD-домен РНК-полимеразы II содержит в своем составе множество гептааминокислотных повторов, подвергающихся ковалентным модификациям в процессе транскрипции. Этот белковый домен, обладающий различными свойствами на различных этапах транскрипционного процесса (благодаря изменению типа и степени модификации), служит платформой для связывания комплексов, участвующих в инициации, элонгации и терминации транскрипции [31]. Недавно была проведена биохимическая очистка белковых комплексов, ассоциированных с CTD-доменами РНК-полимеразы II, которые были модифицированы различным образом (нефосфорилированных, фосфорилированных по Ser5 в CTD посредством CDK7, а также фосфорилированных по Ser2 в CTD посредством CDK9) [32]. Результаты данного эксперимента вполне соответствуют принятой модели участия комплексов в транскрипционном процессе, основанной на различных исследованиях (рис. 1,б) [33]. Было показано, что немодифицированная РНК-полимераза II ассоциирована с комплексами Mediator, Integrator и набором различных фосфатаз. Фосфорилирование РНК-полимеразы II по Ser5 в составе CTD (которое происходит одновременно с началом синтеза РНК, ее кэпированием и покиданием промотора РНК-полимеразой) приводит к нарушению контактов с Mediator, но появлению в комплексе факторов процессинга РНК, кэпирующих ферментов и комплекса PAF1. Элонгирующая РНК-полимераза, фосфорилированная по Ser2 в составе CTD, также ассоциирована с факторами процессинга РНК, комплексами Integrator и PAF1. Интересно, что CTD-домен РНК-полимеразы имеет белковые контакты с ферментативными комплексами, модифицирующими хроматин. Так, было обнаружено, что фосфорилирование CTD РНК-полимеразы II по Ser5 не только приводит к его взаимодействию с комплексом SETD1 A/B, вносящим метилирование по лизину 4 гистона Н3, но и стимулирует ферментативную активность данного комплекса [32]. Биохимические данные, полученные при исследовании комплексов, связанных с CTD РНК-полимеразы II, недавно были подтверждены микроскопическими исследованиями. Судя по всему, фосфорилирование CTD РНК-полимеразы II играет важную роль в фазовом переходе, который проходит элонгирующий фермент в процессе транскрипции между конденсатами, отвечающими за инициацию транскрипции (и обогащенными энхансер-ассоциированными белками, в частности Mediator) и процессинг РНК (включающими в себя факторы сплайсинга) [34].
Известно, что кодирующие области активно работающих генов эукариот заполнены нуклеосомами. Преодоление транскрибирующим комплексом РНК-полимеразы II этих нуклеосом является достаточно сложной задачей. Гены, транскрипция которых происходит на чрезвычайно высоком уровне (обычно в условиях стресса), теряют нуклеосомы в кодирующей области, чтобы обеспечить возможность экстремально высокой степени активации. Примером такого гена является шаперон hsp70, транскрипция которого активируется в условиях теплового шока [35]. Освобождение гена hsp70 от нуклеосом в ходе активации его транскрипции связывают с активностью белка PARP (poly(ADP-ribose) polymerase) [36]. В то же время гены, транскрибирующиеся в нормальных условиях на среднем уровне, обычно не демонстрируют сколько-нибудь значимого снижения уровня нуклеосом в кодирующей области. Полагают, что сохранение нуклеосом в генах является важным механизмом, который препятствует развитию криптической транскрипции – инициации синтеза РНК вне промоторов генов [37]. Таким образом, элонгирующий комплекс РНК-полимеразы II представляет собой эффективную систему, которая, с одной стороны, имеет возможность транскрибировать сквозь хроматин (преодолевать нуклеосомы), а с другой, – восстанавливать связь нуклеосом с ДНК после прохождения фермента для подавления криптической транскрипции. Для реализации данной программы РНК-полимераза II взаимодействует с рядом транскрипционных комплексов, многие из которых ассоциированы с ней на протяжении всего процесса элонгации [38]. Позитивную роль в преодолении РНК-полимеразой II нуклеосомного барьера играют гистоновые шапероны FACT и Spt6, а также ISWI и CHD1 комплексы, ремоделирующие хроматин [39–41]. Причем активность ремоделирующих комплексов в процессе элонгации регулируется ковалентными модификациями хроматина [37]. Поддержание определенного набора ковалентных модификаций хроматина в кодирующих областях генов играет ключевую роль и в подавлении криптической транскрипции (рис. 1,в). Так, в ходе элонгации гистоновый шаперон Asf1 восстанавливает состав нуклеосом вслед за РНК-полимеразой II [42, 43]. В процессе такого восстановления в кодирующую область гена попадают ацетилированные гистоны, присутствующие во внутриклеточном пуле. Ацетилирование гистонов, в свою очередь, провоцирует криптическую транскрипцию в кодирующей области. Для преодоления данной проблемы в состав элонгирующего комплекса РНК-полимеразы II включены компоненты деацетилазного комплекса Set2–Rpd3 [44]. Причем активность данных деацетилаз зависит от наличия ковалентной модификации H3K36me3 на нуклеосомах в кодирующей области гена [45]. Поддержание модификации H3K36me3, а также H2BUb, на нуклеосомах кодирующей области снижает уровень обмена гистонов в данных районах генома и, как следствие, препятствует криптической транскрипции [45, 46]. Как и H3K36me3, модификация H2BUb вносится на хроматин котранскрипционно. В этом процессе ведущую роль играют комплексы PAF и CHD1, ассоциированные с фосфорилированным CTD-доменом РНК-полимеразы II [47]. Недавно было обнаружено, что Rtf1-субъединица комплекса PAF напрямую стимулирует убиквитинилирование H2B, взаимодействуя непосредственно с комплексом Rad6/Bre1, ассоциированным с полимеразой [48]. Вполне вероятно, что все факторы, регулирующие скорость продвижения РНК-полимеразы II в теле гена, могут иметь непосредственное отношение к качеству синтезированной геном РНК. К сожалению, в настоящее время полногеномная оценка качества синтеза РНК является технически трудной задачей, для решения которой необходим дальнейший прогресс экспериментальных подходов [49].
ТРАНСКРИПЦИОННЫЕ КОМПЛЕКСЫ, ИЗМЕНЯЮЩИЕ 3D-СТРУКТУРУ ХРОМАТИНА
Регуляция транскрипции генов при помощи изменения трехмерной структуры ДНК – эволюционно древний процесс. Системы регуляции транскрипции, основанные на принципе приближения удаленного ДНК-связывающего активаторного белка к промотору гена, который он регулирует, встречаются даже у бактерий [50]. Более того, у бактерий имеются специализированные белки, обеспечивающие процесс образования петель ДНК [51]. Увеличение размера генома у более сложных организмов приводит к тому, что белки, изменяющие структуру ДНК, начинают играть еще более важную роль в регуляции транскрипции генов.
На протяжении долгого времени модели процессов регуляции транскрипции, основанные на изменении трехмерной структуры ДНК, были в некоторой степени гипотетическими. Они хорошо объясняли результаты многих функциональных экспериментов (например, генетических) [52]. Однако факт непосредственного взаимодействия удаленных участков ДНК долгое время было невозможно установить экспериментально (как и точно локализовать взаимодействующие участки ДНК в геноме). Ситуация существенно изменилась вместе с изобретением экспериментальных методик, позволяющих регистрировать непосредственную ассоциацию элементов ДНК в различных клетках, – методик фиксации состояния хроматина, называемых еще “С-методами” (3C, “chromatin conformation capture”, и его модификации) [53]. Применение “С-методов” продемонстрировало, что геномы многоклеточных организмов разделены на области, обогащенные хроматиновыми контактами (названные топологически ассоциированными доменами – TAD), и области, обедненные контактами и названные границами TAD [54]. Имеющиеся в геноме TADs можно разделить на активные (имеющие в своем составе активно работающие гены) и неактивные (содержащие неактивные гены, а часто и ковалентные модификации, присущие компактизованному хроматину). Тип TAD хорошо предсказывает время его репликации – активные реплицируются в ранней S-фазе клеточного цикла. Кроме того, характеристика TAD связана с плотностью контактов внутри него – неактивные TAD демонстрируют большую плотность контактов, вероятно связанную с более плотной компактизацией хроматина в данном районе. Исследования разных типов клеток, а также разных стадий развития организмов показали, что разделение генома на TAD является очень устойчивой характеристикой. Отдельные локусы демонстрируют даже эволюционную консервативность TAD-структуры [55].
Исследования структуры геномов демонстрируют нам по крайней мере два совершенно различных уровня функциональной организации трехмерной структуры ДНК: 1) устойчивый уровень, характерный для многих типов клеток (включающий в себя белки и геномные элементы, организующие TAD и их границы), а также 2) динамический уровень, отвечающий на изменение состояния клеток (включающий в себя белки и геномные элементы, организующие динамическую ассоциацию энахансеров и промоторов). Без сомнения оба уровня трехмерной организации генома играют огромную роль в регуляции транскрипции генов. Однако принципы их участия в работе генов различны.
Белки и белковые комплексы, организующие топологически ассоциированные домены
Как уже упоминалось выше, присутствие гена в TAD определенного типа предсказывает его активность. Трансген, встроившийся в неактивный TAD, с большой вероятностью не будет транскрибироваться. В соответствии с этим фактом изменение границ TADs оказывает значительное влияние на работу входящих в них генов. Данный феномен лучше всего изучен на модельных локусах, для которых хорошо исследована их структура, а также известны регуляторные элементы и их функциональная активность. Так, в раннем эмбриональном развитии Hox-гены млекопитающих, отвечающие за рост и формирование организма, формируют один общий TAD с регуляторными областями. Изменение активности генов, происходящее в ходе развития, приводит к изменению топологической структуры локуса и формированию двух раздельных TAD, различных по транскрипционному статусу [56]. Изменение границы между данными TAD приводит к нарушению транскрипционного статуса генов данного района [57]. Другая топологическая граница разделяет регуляторные элементы генов Tfap2c и Bmp7, обеспечивая независимый характер работы тканеспецифических энхансеров данного локуса [58].
Несмотря на то что функциональная значимость границ TAD продемонстрирована во многих модельных системах, молекулярный механизм формирования таких границ до сих пор недостаточно ясен. Более того, экспериментальные данные предполагают наличие различных механизмов образования границ для разных организмов [59]. Так, у млекопитающих основным механизмом образования TAD предполагается образование петель когезиновым комплексом при участии инсуляторного белка CTCF, действие которого ограничивает распространение петли [60, 61]. Данную гипотезу подтверждают данные экспериментов Hi-C высокого разрешения, которые демонстрируют наличие петли ДНК (обогащенного контактами участка) между границами TAD. В настоящее время считается, что механизм образования TAD посредством действия когезина и CTCF отсутствует у дрозофилы. Во всяком случае границы TAD у нее не образуют видимых петель. Формирование TAD в раннем эмбриогенезе дрозофилы хорошо коррелирует с активацией транскрипции генов. Высказывались предположения, что именно транскрипционная активность генов домашнего хозяйства формирует границы TAD у дрозофилы [62]. Однако ингибирование транскрипции в раннем эмбриогенезе не нарушает образование TAD полностью, демонстрируя тем самым важную, но не ведущую роль транскрипции в данном процессе [63]. Интересно, что ингибирование транскрипции у до-имплантационного эмбриона мыши также не препятствует образованию TAD [64]. Более серьезный эффект на образование структуры хроматина в раннем эмбриогенезе млекопитающих оказывает ингибирование репликации [65]. Вместе эти данные хорошо иллюстрируют отсутствие в настоящее время единого мнения о механизме образования TADs и их границ. Многие белки могут играть роль в их поддержании, но важны ли они для образования данного TAD в ходе развития – доказать достаточно сложно.
Конечно, белки, отвечающие за сохранение границ TAD, важны для поддержания адекватного уровня транскрипции входящих в них генов. Сохранение границ обеспечивает возможность своевременного образования петель от эхансеров и сайленсеров, играя существенную роль в динамической регуляции транскрипции генов в ходе развития [66, 67]. Ярким примером этого является белок CTCF млекопитающих, активность которого важна для сохранения границы в составе Hox-локуса и поддержания репрессированного состояния генов в неактивном TAD (рис. 2,а) [57]. Как упоминалось выше, роль инсуляторных белков в поддержании границ хроматиновых доменов была продемонстрирована задолго до появления “С-методов” [68]. Поэтому сразу после появления методик полногеномного исследования структуры хроматина они стали считаться наиболее вероятными кандидатами на роль белков, ответственных за поддержание 3D-архитектуры генома. Так, уже в самых первых исследованиях Hi-C геномов млекопитающих указывалось на участие инсуляторного белка CTCF в организации границ TAD [54]. Годы исследований еще более укрепили функциональную роль CTCF в качестве белка, сохраняющего границы TAD у млекопитающих [69]. Стоит отметить, что роль CTCF в организации границ у дрозофилы продемонстрирована менее явно. Предполагается, что поддержание границ TAD у этого организма организовано при участии многих инсуляторных белков, таких как BEAF-32, Chromator и CP190 [70, 71]. С другой стороны, данные типы инсуляторных белков дрозофилы колокализуются в геноме с активными регуляторными элементами (такими, как промоторы генов) [72]. Может оказаться, что за сохранение границ TAD у дрозофилы отвечают какие-то другие регуляторные белки, присутствующие в геноме на инсуляторах данного типа.
Рис. 2.
а – роль CTCF в организации топологически ассоциированных доменов в Hox-локусе млекопитающих. Нарушение границы приводит к активации генов в составе репрессированного домена; б – механизм ассоциации энхансера и промотора гена на основе взаимодействия между “жидкими” областями, образованными вокруг каждого из регуляторных элементов. Данные области образуются при участии неструктурированных областей белков, входящих в состав преинициаторного комплекса, а также комплекса Mediator.
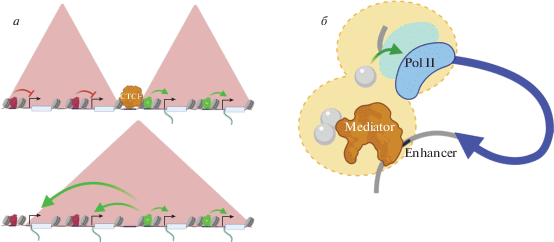
В основном границы областей TAD стабильны на протяжении развития и сохраняются в различных тканях. Изменение положения границ обычно происходит однократно, сопровождается глобальным изменением транскрипционного статуса хроматинового района и сохраняется в дальнейшем в развитии (как изменения в структуре локуса Hox-генов млекопитающих, которые происходят в ходе дифференцировки ткани). Медленный характер изменений в структуре генома хорошо иллюстрирует динамика изменения сайтов связывания инсуляторных белков под воздействием сигнала развития. На клеточной линии Кс дрозофилы было показано, что воздействие на нее гормона развития 20-гидроксиэкдизона приводит к появлению новых сайтов связывания для инсуляторных белков Su(Hw) и CP190 [73]. Однако для такого глобального изменения хроматина необходимо длительное воздействие индуцирующего сигнала (эффект наблюдается только после 48 ч воздействия гормона на клетки).
Белки и белковые комплексы, обеспечивающие взаимодействие удаленных регуляторных элементов и промоторов генов
Разделение генома клетки на топологически обособленные домены основано на частотности контактов внутри района. В отдельные домены выделяют участки хроматина, обогащенные множественными контактами. Основную массу контактов внутри TAD составляют динамические взаимодействия между элементами ДНК, регулирующими транскрипцию и репликацию. Именно изменение таких внутридоменных контактов происходит под воздействием сигналов стресса или развития, что реализуется в изменении паттерна транскрипции генов. Конечно, белки, провоцирующие или нарушающие динамическую связь между регуляторными элементами, влияют на транскрипцию генов и являются тем самым транскрипционными корегуляторами.
Лучше всего исследованы механизмы становления контактов внутри топологических районов на примере взаимодействия энхансеров и промоторов генов [74]. В некоторых случаях именно взаимодействие энхансера с промотором является триггером, достаточным для активации транскрипции гена. Так, искусственное рекрутирование локус-контролирующего района LCR к промотору β-глобинового гена приводит к активации его транскрипции [75]. В норме сближение LCR и промотора β-глобинового гена обеспечивается согласованной работой транскрипционного фактора GATA1 и белка Ldb1 [76]. Тем не менее для многих генов было показано, что взаимодействие их промоторов с энхансерами не связано с индукцией транскрипции и происходит задолго до активации гена. В частности, энхансеры экдизон-зависимых генов дрозофилы ассоциированы с промоторами генов в неактивном состоянии [77]. Показано, что важную роль в организации структуры хроматина отдельных генов экдизонового каскада играют компоненты ядерной поры. Полногеномное исследование контактов энхансеров и промоторов в клетках человека позволило обнаружить корреляцию между силой контактов и уровнем маркера H3K27Ac в хроматине энхансеров [78]. Было обнаружено, что “стабильные” энхансеры, установившие контакты с промоторами генов задолго до активации транскрипции, характеризуются высоким уровнем данного маркера хроматина. Вероятно, активность транскрипционного регулятора CBP/p300, присутствующего на энхансерах, связана с образованием контактов между энхансером и промотором.
Логично предположить, что в установлении контактов промотора с энхансерами принимают участие компоненты преинициаторного комплекса или даже сама РНК-полимераза II. На первый взгляд данному предположению противоречат экспериментальные данные, демонстрирующие наличие контактов между промоторами и энхансерами неактивных генов. Однако детальное исследование динамики контактов между регуляторными элементами в раннем эмбриональном развитии дрозофилы показало, что стабильные контакты энхансеров с промоторами неактивных генов связаны с присутствием на промоторах РНК-полимеразы II в состоянии транскрипционной “паузы” [79]. То есть сама РНК-полимераза II может быть активным участником процесса формирования и поддержания динамических контактов в геноме. В том же исследовании было продемонстрировано, что лишь в половине энхансеров, установивших контакты с регулируемыми ими промоторами, присутствуют сайты связывания для инсуляторных белков. Данный факт подтверждает ранее высказанное предположение о ведущей роли инсуляторных белков в стабильных, но не динамических трехмерных взаимодействиях в хроматине.
Среди компонентов преинициаторного комплекса ведущую роль в организации взаимодействий между энхансерами и промоторами генов играют компоненты комплекса Mediator. Данный комплекс представляет собой мультибелковую структуру, которая связывает энхансеры и управляет процессом инициации транскрипции на промоторе [80, 81]. Служа платформой для действия многих корегуляторов, Mediator координирует различные этапы транскрипции генов. Сближение Mediator с регулируемым промотором является одним из ключевых моментов активации транскрипции гена и происходит путем образования петли между энхансером и промотором. Взаимодействие субъединиц Mediator с когезиновым комплексом указывает на то, что данный комплекс может принимать непосредственное участие в формирование этой петли [82, 83]. С другой стороны, последние исследования противоречат модели физического контакта Mediator одновременно с энхансерами и промоторами генов [84]. Предполагается, что данный комплекс лишь сближается с регулируемым промотором, оказывая на него функциональное воздействие, но не устанавливая устойчивого контакта.
В последнее время большую популярность получила модель взаимодействия энхансеров и промоторов путем формирования в ядре обособленных жидких безмембранных органелл [85]. Ранее описанным примером подобных внутриядерных органелл являются тельца Кахаля, содержащие в своем составе факторы биогенеза мРНК [86]. В соответствии с данной моделью компоненты преинициаторного комплекса, а также регуляторы, ассоциированные с энхансерами, несут в составе своих белковых компонентов множество неструктурированных доменов, которые способствуют образованию в районе регуляторных элементов “жидких” областей с особыми химическими свойствами. Свойства данных областей таковы, что способствуют слиянию их в единое целое и, соответственно, сближению участков промотора и энхансера (рис. 2,б) [87]. Особые свойства данных областей позволяют попавшим в них транскрипционным регуляторам двигаться с большей скоростью и повышают вероятность функционального воздействия белкового регулятора на регуляторный элемент ДНК. В контексте данной теории особую роль приобретают пока не охарактеризованные белки, способствующие созданию “жидких” областей в районе энхансера и промотора, а также их объединению. Другими немаловажными регуляторами являются также белки, облегчающие фазовый переход на границе подобных областей для регуляторов транскрипции [34]. Поиск и характеристика свойств транскрипционных регуляторов, организующих фазовые переходы внутри ядра, – удел будущих исследований.
В настоящее время сложно выделить ведущую молекулярную модель, которая бы описывала механизм, обеспечивающий взаимодействие энхансера и промотора. До сих пор устойчивыми являются многие модели образования петель: 1) за счет элонгации РНК-полимеразы; 2) за счет олигомеризации ДНК-связывающих белков; 3) за счет образования петли при участии комплексов когезина и конденсина, а также Nipped-B; 4) за счет разделения фаз [76, 87–90]. Многие белки и белковые комплексы, опосредующие образование петель ДНК в рамках данных моделей, играют одновременно функциональные роли регуляторов транскрипции и архитектурных белков. Но для большинства из них установить ведущую и косвенную функции на основе имеющейся экспериментальной информации пока не представляется возможным.
В табл. 1 мы приводим описание основных регуляторов транскрипции, рассмотренных в данном обзоре.
Таблица 1.
Классификация транскрипционных корегуляторов
| Название белка/белкового комплекса | Функциональная (каталитическая/ ферментативная) субъединица | Молекулярные функции | Способ привлечения к регуляторному элементу |
|---|---|---|---|
| Регуляторы элонгации | |||
| y/d/hDSIF | y/d/hSpt5 | Участвует в кэпировании мРНК посредством стимуляции активности мРНК-гуанилилтрансферазы [91]. Стимулирует паузинг РНК-полимеразы II на промоторе посредством стабилизации комплекса РНК-полимеразы II с NELF [6]. Стимулирует процессивность РНК-полимеразы II, сплай- синг и образование мРНП [13–15] | РНК-опосредованное взаимодействие с РНК-полимеразой II на этапе инициации транскрипции [9]. Диссоциация путем фосфорилирования фактором p-TEFbDSIF и NELF [11] |
| d/hNELF | d/hNELFA, d/hNELFB, d/hNELFD, d/hNELFE | Экранирует поверхность РНК-полимеразы II, необходимую для рекрутированияTFIIS и связывания PAF1 [12] | Связывание с РНК-полимеразой II кооперативно с DSIF [6] |
| y/d/h p-TEFb | y/d/hCDK9 | Стимулирует элонгацию посредством фосфорилирования факторов паузинга DSIF, NELF и РНК-полимеразы II [11] | Рекрутирование к промоторам в составе комплекса с Brd4, 7SK RNA или как части Super Elongation Complex (SEC) [20] |
| y/d/hPAF1 | y/d/hPAF1 | Регулирует паузинг РНК-полимеразы II [92] и совместно с DSIF регулирует элонгацию транскрипции [93]. Cтимулирует убиквитинилирование H2B, взаимодействуя с комплексом Rad6/Bre1 [48] | Связывание с CTD-доменом РНК-полимеразы II [12] |
| d/hIntegrator | d/hIntS9, d/hIntS11 – РНК-эндонуклеазы | Участвует в терминации транскрипции паузированной РНК-полимеразы II [94]. Регулирует отрезание U1 и U2 snRNA сплайсосомы, транскрипцию энхансерной eRNA [95] | Связывание с CTD-доменом РНК-полимеразы II [96] |
| Шапероны гистонов | |||
| y/d/hFACT | d/hSSRP1, y/d/hSpt1, yPob3 | Способствует преодолению РНК-полимеразой II нуклеосомного барьера промотора, прохождению ею нуклеосом в кодирующей области генов [97] | Взаимодействиес CTD-доменом РНК-полимеразы II [98] |
| Белки, изменяющие 3D-структуру хроматина | |||
| d/hCTCF | d/hCTCF | Регулирует образование петель хроматина внутри локусов генов [57, 99], совместно с когезином формирует границы TADs [60, 61, 69, 83] и специфическую для типа клеток 3D-структуру хроматина [100] | Связывание со специфическим мотивом ДНК [101] |
| dBEAF-32 | dBEAF-32 | Участвует в поддержании границ TADs [71] | Связывание со специфическим мотивом ДНК [102] |
| dСP190 | dСP190 | Участвует в поддержании границ TADs [71], в формировании энхансер-промоторных петель [66, 73] | Связывание хроматина посредством взаимодействия с ДНК-связывающими инсуляторными белками [103, 104] и ядерными рецепторами [66] |
| y/d/hMediator | Связывает энхансеры и регулирует процесс инициации транскрипции на промоторе [80, 81] | Образование энхансер-промоторных петель происходит, возможно, посредством взаимодействия с когезином [82, 83] | |
ЗАКЛЮЧЕНИЕ
В последние годы развитие полногеномных методов исследования позволило существенно дополнить наши знания о функциях разнообразных белков и белковых комплексов-корегуляторов в процессах инициации и элонгации транскрипции. Так, если раньше считалось, что стимуляция транскрипции гена требует рекрутирования к промотору общих факторов транскрипции, РНК-полимеразы II, комплексов ремоделирования и модификации хроматина, то современные данные свидетельствуют скорее в пользу существования на большинстве генов многоклеточных инициаторного комплекса полимеразы в состоянии “паузинга”. Показано, что процесс активации и регуляции транскрипции на разных генах, в различных тканях, на отдельных этапах онтогенеза может задействовать огромное разнообразие механизмов. Для того, чтобы отследить наиболее важных участников и биологические закономерности, которым эти механизмы подчиняются, сформировать единую экспериментальную модель транскрипционного процесса, потребуется еще много работы. В данном обзоре мы постарались начать ее выполнение.
Данная работа была поддержана грантом Российского научного фонда № 18-14-00219 (рук. Н.Е. Воробьева).
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Blombach F., Smollett K.L., Grohmann D., Werner F. Molecular mechanisms of transcription initiation-structure, function, and evolution of TFE/TFIIE-Like factors and open complex formation // J. Mol. Biol. 2016. V. 428. № 12. P. 2592–2606. https://doi.org/10.1016/j.jmb.2016.04.016
Werner F. A Nexus for gene expression – molecular mechanisms of Spt5 and NusG in the three domains of life // J. Mol. Biol. 2012. V. 417. № 1. P. 13–27. https://doi.org/10.1016/j.jmb.2012.01.031
Wada T., Takagi T., Yamaguchi Y. et al. DSIF, a novel transcription elongation factor that regulates RNA polymerase II processivity, is composed of human Spt4 and Spt5 homologs // Genes Dev. 1998. V. 12. № 3. P. 343–356.
Adelman K., Lis J.T. Promoter-proximal pausing of RNA polymerase II: Emerging roles in metazoans // Nat. Rev. Genet. 2012. V. 13. № 10. P. 720–731. https://doi.org/10.1038/nrg3293
Muse G.W., Gilchrist D.A., Nechaev S. et al. RNA polymerase is poised for activation across the genome // Nat. Genet. 2007. V. 39. № 12. P. 1507–1511. https://doi.org/10.1038/ng.2007.21
Yamaguchi Y., Inukai N., Narita T. et al. Evidence that negative elongation factor represses transcription elongation through binding to a DRB sensitivity-inducing factor/RNA polymerase II complex and RNA // Mol. Cell. Biol. 2002. V. 22. № 9. P. 2918–2927. https://doi.org/10.1128/MCB.22.9.2918-2927.2002
Larochelle S., Amat R., Glover-Cutter K. et al. Cyclin-dependent kinase control of the initiation-to-elongation switch of RNA polymerase II // Nat. Struct. Mol. Biol. 2012. V. 19. № 11. P. 1108–1115. https://doi.org/10.1038/nsmb.2399
Schulz S., Gietl A., Smollett K. et al. TFE and Spt4/5 open and close the RNA polymerase clamp during the transcription cycle // Proc. Natl Acad. Sci. USA. 2016. V. 113. № 13. P. E1816–E1825. https://doi.org/10.1073/pnas.1515817113
Vos S.M., Farnung L., Urlaub H., Cramer P. Structure of paused transcription complex Pol II–DSIF–NELF // Nature. 2018. V. 560. № 7720. P. 601–606. https://doi.org/10.1038/s41586-018-0442-2
Pei Y., Shuman S. Interactions between fission yeast mRNA capping enzymes and elongation factor Spt5 // J. Biol. Chem. 2002. V. 277. № 22. P. 19639–19648. https://doi.org/10.1074/jbc.M200015200
Yamada T., Yamaguchi Y., Inukai N. et al. P-TEFb-mediated phosphorylation of hSpt5 C-terminal repeats is critical for processive transcription elongation // Mol. Cell. 2006. V. 21. № 2. P. 227–237. https://doi.org/10.1016/j.molcel.2005.11.024
Vos S.M., Farnung L., Boehning M. et al. Structure of activated transcription complex Pol II–DSIF–PAF–SPT6 // Nature. 2018. V. 560. № 7720. P. 607–612. https://doi.org/10.1038/s41586-018-0440-4
Fitz J., Neumann T., Pavri R. Regulation of RNA polymerase II processivity by Spt5 is restricted to a narrow window during elongation // EMBO J. 2018. V. 37. № 8. P. e97965. https://doi.org/10.15252/embj.201797965
Maudlin I.E., Beggs J.D. Spt5 modulates cotranscriptional spliceosome assembly in Saccharomyces cerevisiae // RNA. 2019. V. 25. № 10. P. 1298–1310. https://doi.org/10.1261/rna.070425.119
Shetty A., Kallgren S.P., Demel C. et al. Spt5 Plays vital roles in the control of sense and antisense transcription elongation // Mol. Cell. 2017. V. 66. № 1. P. 77–88.e5. https://doi.org/10.1016/j.molcel.2017.02.023
Jang M.K., Mochizuki K., Zhou M. et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription // Mol. Cell. 2005. V. 19. № 4. P. 523–534. https://doi.org/10.1016/j.molcel.2005.06.027
Winter G.E., Mayer A., Buckley D.L. et al. BET bromodomain proteins function as master transcription elongation factors independent of CDK9 recruitment// Mol. Cell. 2017. V. 67. № 1. P. 5–18.e19. https://doi.org/10.1016/j.molcel.2017.06.004
Andrews F.H., Singh A.R., Joshi S. et al. Dual-activity PI3K–BRD4 inhibitor for the orthogonal inhibition of MYC to block tumor growth and metastasis // Proc. Natl Acad. Sci. USA. 2017. V. 114. № 7. P. E1072–E1080. https://doi.org/10.1073/pnas.1613091114
Coudé M.-M., Braun T., Berrou J. et al. BET inhibitor OTX015 targets BRD2 and BRD4 and decreases c-MYC in acute leukemia cells // Oncotarget. 2015. V. 6. № 19. P. 17698–17712.
Chen F.X., Smith E.R., Shilatifard A. Born to run: Control of transcription elongation by RNA polymerase II // Nat. Rev. Mol. Cell Biol. 2018. V. 19. № 7. P. 464–478. https://doi.org/10.1038/s41580-018-0010-5
Lin C., Smith E.R., Takahashi H. et al. AFF4, A component of the ELL/P-TEFb elongation complex and a shared subunit of MLL chimeras, can link transcription elongation to leukemia // Mol. Cell. 2010. V. 37. № 3. P. 429–437. https://doi.org/10.1016/j.molcel.2010.01.026
Luo Z., Lin C., Shilatifard A. The super elongation complex (SEC) family in transcriptional control // Nat. Rev. Mol. Cell Biol. 2012. V. 13. № 9. P. 543–547. https://doi.org/10.1038/nrm3417
Nguyen V.T., Kiss T., Michels A.A., Bensaude O. 7SK small nuclear RNA binds to and inhibits the activity of CDK9/cyclin T complexes // Nature. 2001. V. 414. № 6861. P. 322–325. https://doi.org/10.1038/35104581
McNamara R.P., Reeder J.E., McMillan E.A. et al. KAP1 recruitment of the 7SK snRNP complex to promoters enables transcription elongation by RNA polymerase II // Mol Cell. 2016. V. 61. № 1. P. 39–53. https://doi.org/10.1016/j.molcel.2015.11.004
Bacon C.W., D’Orso I. CDK9: A signaling hub for transcriptional control // Transcription. 2019. V. 10. № 2. P. 57–75. https://doi.org/10.1080/21541264.2018.1523668
Parua P.K., Booth G.T., Sansó M. et al. Cdk9–PP1 switch regulates the elongation–termination transition of RNA polymerase II // Nature. 2018. V. 558. № 7710. P. 460–464. https://doi.org/10.1038/s41586-018-0214-z
Saldi T., Fong N., Bentley D.L. Transcription elongation rate affects nascent histone pre-mRNA folding and 3′ end processing // Genes Dev. 2018. V. 32. № 3–4. P. 297–308. https://doi.org/10.1101/gad.310896.117
Ip J.Y., Schmidt D., Pan Q. et al. Global impact of RNA polymerase II elongation inhibition on alternative splicing regulation // Genome Res. 2011. V. 21. № 3. P. 390–401. https://doi.org/10.1101/gr.111070.110
Fong N., Kim H., Zhou Yu. et al. Pre-mRNA splicing is facilitated by an optimal RNA polymerase II elongation rate // Genes Dev. 2014. V. 28. № 23. P. 2663–2676. https://doi.org/10.1101/gad.252106.114
Aslanzadeh V., Huang Y., Sanguinetti G., Beggs J.D. Transcription rate strongly affects splicing fidelity and cotranscriptionality in budding yeast // Genome Res. 2018. V. 28. № 2. P. 203–213. https://doi.org/10.1101/gr.225615.117
Harlen K.M., Churchman L.S. The code and beyond: Transcription regulation by the RNA polymerase II carboxy-terminal domain // Nat. Rev. Mol. Cell Biol. 2017. V. 18. № 4. P. 263–273. https://doi.org/10.1038/nrm.2017.10
Ebmeier C.C., Erickson B., Allen B.L. et al. Human TFIIH kinase CDK7 regulates transcription-associated chromatin modifications // Cell Rep. 2017. V. 20. № 5. P. 1173–1186. https://doi.org/10.1016/j.celrep.2017.07.021
Krasnov A.N., Mazina M.Y., Nikolenko J.V., Vorobyeva N.E. On the way of revealing coactivator complexes cross-talk during transcriptional activation // Cell Biosci. 2016. V. 6. P. 15. https://doi.org/10.1186/s13578-016-0081-y
Guo Y.E., Manteiga J.C., Henninger J.E. et al. Pol II phosphorylation regulates a switch between transcriptional and splicing condensates // Nature. 2019. V. 572. № 7770. P. 543–548. https://doi.org/10.1038/s41586-019-1464-0
Petesch S.J., Lis J.T. Rapid, transcription-independent loss of nucleosomes over a large chromatin domain at Hsp70 loci // Cell. 2008. V. 134. № 1. P. 74–84. https://doi.org/10.1016/j.cell.2008.05.029
Petesch S.J., Lis J.T. Activator-induced spread of Poly(ADP-Ribose) polymerase promotes nucleosome loss at Hsp70 // Mol. Cell. 2012. V. 45. № 1. P. 64–74. https://doi.org/10.1016/j.molcel.2011.11.015
Smolle M., Workman J.L. Transcription-associated histone modifications and cryptic transcription // Biochim. Biophys. Acta BBA–Gene Regul. Mech. 2013. V. 1829. № 1. P. 84–97. https://doi.org/10.1016/j.bbagrm.2012.08.008
Cheung V., Chua G., Batada N.N. et al. Chromatin- and transcription-related factors repress transcription from within coding regions throughout the Saccharomyces cerevisiae genome // PLoS Biol. 2008. V. 6. № 11. e277. https://doi.org/10.1371/journal.pbio.0060277
Chang H.-W., Nizovtseva E.V., Razin S.V. et al. Histone chaperone FACT and curaxins: Effects on genome structure and function // J. Cancer Metastasis Treat. 2019. V. 5. https://doi.org/10.20517/2394-4722.2019.31
Pathak R., Singh P., Ananthakrishnan S. et al. Acetylation-dependent recruitment of the FACT complex and Its role in regulating Pol II occupancy genome-wide in Saccharomyces cerevisiae // Genetics. 2018. V. 209. № 3. P. 743–756. https://doi.org/10.1534/genetics.118.300943
Sun M., Larivière L., Dengl S. et al. A tandem SH2 domain in transcription elongation factor Spt6 binds the phosphorylated RNA polymerase II C-terminal Repeat Domain (CTD) // J. Biol. Chem. 2010. V. 285. № 53. P. 41597–41603. https://doi.org/10.1074/jbc.M110.144568
Schwabish M.A., Struhl K. Asf1 mediates histone eviction and deposition during elongation by RNA polymerase II // Mol. Cell. 2006. V. 22. № 3. P. 415–422. https://doi.org/10.1016/j.molcel.2006.03.014
Silva A.C., Xu X., Kim H.-S. et al. The replication-independent histone H3–H4 chaperones HIR, ASF1, and RTT106 Co-operate to maintain promoter fidelity // J. Biol. Chem. 2012. V. 287. № 3. P. 1709–1718. https://doi.org/10.1074/jbc.M111.316489
Govind C.K., Qiu H., Ginsburg D.S. et al. Phosphorylated Pol II CTD recruits multiple HDACs, including Rpd3C(S), for methylation-dependent deacetylation of ORF nucleosomes // Mol. Cell. 2010. V. 39. № 2. P. 234–246. https://doi.org/10.1016/j.molcel.2010.07.003
Venkatesh S., Smolle M., Li H. et al. Set2 methylation of histone H3 lysine 36 suppresses histone exchange on transcribed genes // Nature. 2012. V. 489. № 7416. P. 452–455. https://doi.org/10.1038/nature11326
Batta K., Zhang Z., Yen K. et al. Genome-wide function of H2B ubiquitylation in promoter and genic regions // Genes Dev. 2011. V. 25. № 21. P. 2254–2265. https://doi.org/10.1101/gad.177238.111
Lee J.-S., Garrett A.S., Yen K. et al. Codependency of H2B monoubiquitination and nucleosome reassembly on Chd1 // Genes Dev. 2012. V. 26. № 9. P. 914–919. https://doi.org/10.1101/gad.186841.112
Van Oss S.B., Shirra M.K., Bataille A.R. et al. The histone modification domain of Paf1 complex subunit Rtf1 directly stimulates H2B ubiquitylation through an interaction with Rad6 // Mol. Cell. 2016. V. 64. № 4. P. 815–825. https://doi.org/10.1016/j.molcel.2016.10.008
Imashimizu M., Takahashi H., Oshima T. Visualizing translocation dynamics and nascent transcript errors in paused RNA polymerases in vivo // Genome Biol. 2015. V. 16. № 1. P. 98. https://doi.org/10.1186/s13059-015-0666-5
Bush M., Dixon R. The role of bacterial enhancer binding proteins as specialized activators of σ54-dependent transcription // Microbiol. Mol. Biol. Rev. 2012. V. 76. № 3. P. 497–529. https://doi.org/10.1128/MMBR.00006-12
Cournac A., Plumbridge J. DNA looping in prokaryotes: Experimental and theoretical approaches // J. Bacteriol. 2013. V. 195. № 6. P. 1109–1119. https://doi.org/10.1128/JB.02038-12
Kyrchanova O., Georgiev P. Chromatin insulators and long-distance interactions in Drosophila // FEBS Lett. 2014. V. 588. № 1. P. 8–14. https://doi.org/10.1016/j.febslet.2013.10.039
Schwartz Y.B., Cavalli G. Three-dimensional genome organization and function in Drosophila // Genetics. 2017. V. 205. № 1. P. 5–24. https://doi.org/10.1534/genetics.115.185132
Dixon J.R., Selvaraj S., Yue F. et al. Topological domains in mammalian genomes identified by analysis of chromatin interactions // Nature. 2012. V. 485. № 7398. P. 376–380. https://doi.org/10.1038/nature11082
Fishman V., Battulin N., Nuriddinov M. et al. 3D organization of chicken genome demonstrates evolutionary conservation of topologically associated domains and highlights unique architecture of erythrocytes’ chromatin // Nucl. Acids Res. 2019. V. 47. № 2. P. 648–665. https://doi.org/10.1093/nar/gky1103
Noordermeer D., Leleu M., Schorderet P. et al. Temporal dynamics and developmental memory of 3D chromatin architecture at Hox gene loci // eLife. 2014. V. 3. P. e02557. https://doi.org/10.7554/eLife.02557
Narendra V., Rocha P.P., An D. et al. CTCF establishes discrete functional chromatin domains at the Hox clusters during differentiation // Science. 2015. V. 347. № 6225. P. 1017–1021. https://doi.org/10.1126/science.1262088
Tsujimura T., Klein F.A., Langenfeld K. et al. A discrete transition zone organizes the topological and regulatory autonomy of the adjacent tfap2c and bmp7 genes // PLoS Genet. 2015. V. 11. № 1. P. e1004897. https://doi.org/10.1371/journal.pgen.1004897
Szabo Q., Bantignies F., Cavalli G. Principles of genome folding into topologically associating domains // Sci. Adv. 2019. V. 5. № 4. P. eaaw1668. https://doi.org/10.1126/sciadv.aaw1668
Fudenberg G., Imakaev M., Lu C. et al. Formation of chromosomal domains by loop extrusion // Cell Rep. 2016. V. 15. № 9. P. 2038–2049. https://doi.org/10.1016/j.celrep.2016.04.085
Sanborn A.L., Rao S.S.P., Huang S.-C. et al. Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes // Proc. Natl Acad. Sci. USA. 2015. V. 112. № 47. P. E6456–E6465. https://doi.org/10.1073/pnas.1518552112
Ulianov S.V., Khrameeva E.E., Gavrilov A.A. et al. Active chromatin and transcription play a key role in chromosome partitioning into topologically associating domains // Genome Res. 2016. V. 26. № 1. P. 70–84. https://doi.org/10.1101/gr.196006.115
Hug C.B., Grimaldi A.G., Kruse K., Vaquerizas J.M. Chromatin architecture emerges during zygotic genome activation independent of transcription // Cell. 2017. V. 169. № 2. P. 216–228.e19. https://doi.org/10.1016/j.cell.2017.03.024
Du Z., Zheng H., Huang B. et al. Allelic reprogramming of 3D chromatin architecture during early mammalian development // Nature. 2017. V. 547. № 7662. P. 232–235. https://doi.org/10.1038/nature23263
Ke Y., Xu Y., Chen X. et al. 3D Chromatin structures of mature gametes and structural reprogramming during mammalian embryogenesis // Cell. 2017. V. 170. № 2. P. 367–381.e20. https://doi.org/10.1016/j.cell.2017.06.029
Mazina M.Y., Ziganshin R.H., Magnitov M.D. et al. Proximity-dependent biotin labelling reveals CP190 as an EcR/Usp molecular partner // Sci. Rep. 2020. V. 10. № 1. P. 1–14. https://doi.org/10.1038/s41598-020-61514-0
Воробьева Н.Е., Мазина М.Ю. Функции инсуляторов в контексте современных полногеномных исследований // Генетика. 2019. Т. 55. №2. С. 154–162.
Maksimenko O., Georgiev P. Mechanisms and proteins involved in long-distance interactions // Front. Genet. 2014. V. 5. P. 28. https://doi.org/10.3389/fgene.2014.00028
Ghirlando R., Felsenfeld G. CTCF: Making the right connections // Genes Dev. 2016. V. 30. № 8. P. 881–891. https://doi.org/10.1101/gad.277863.116
Gambetta M.C., Furlong E.E.M. The insulator protein CTCF is required for correct hox gene expression, but not for embryonic development in Drosophila // Genetics. 2018. V. 210. № 1. P. 129–136. https://doi.org/10.1534/genetics.118.301350
Wang Q., Sun Q., Czajkowsky D.M., Shao Z. Sub-kb Hi-C in D. melanogaster reveals conserved characteristics of TADs between insect and mammalian cells // Nat. Commun. 2018. V. 9. № 1. P. 188. https://doi.org/10.1038/s41467-017-02526-9
Schwartz Y.B., Linder-Basso D., Kharchenko P.V. et al. Nature and function of insulator protein binding sites in the Drosophila genome // Genome Res. 2012. V. 22. № 11. P. 2188–2198. https://doi.org/10.1101/gr.138156.112
Wood A.M., Van Bortle K., Ramos E. et al. Regulation of chromatin organization and inducible gene expression by a Drosophila insulator // Mol. Cell. 2011. V. 44. № 1. P. 29–38. https://doi.org/10.1016/j.molcel.2011.07.035
Furlong E.E.M., Levine M. Developmental enhancers and chromosome topology // Science. 2018. V. 361. № 6409. P. 1341–1345. https://doi.org/10.1126/science.aau0320
Deng W., Lee J., Wang H. et al. Controlling long-range genomic interactions at a native locus by targeted tethering of a looping factor // Cell. 2012. V. 149. № 6. P. 1233–1244. https://doi.org/10.1016/j.cell.2012.03.051
Krivega I., Dean A. LDB1-mediated enhancer looping can be established independent of mediator and cohesin // Nucleic Acids Res. 2017. V. 45. № 14. P. 8255–8268. https://doi.org/10.1093/nar/gkx433
Pascual-Garcia P., Debo B., Aleman J.R. et al. Metazoan nuclear pores provide a scaffold for poised genes and mediate induced enhancer-promoter contacts // Mol. Cell. 2017. V. 66. № 1. P. 63–76.e6. https://doi.org/10.1016/j.molcel.2017.02.020
Rubin A.J., Barajas B.C., Furlan-Magaril M. et al. Lineage-specific dynamic and pre-established enhancer–promoter contacts cooperate in terminal differentiation // Nat. Genet. 2017. V. 49. № 10. P. 1522–1528. https://doi.org/10.1038/ng.3935
Ghavi-Helm Y., Klein F.A., Pakozdi T. et al. Enhancer loops appear stable during development and are associated with paused polymerase // Nature. 2014. V. 512. № 7512. P. 96–100. https://doi.org/10.1038/nature13417
Allen B.L., Taatjes D.J. The mediator complex: A central integrator of transcription // Nat. Rev. Mol. Cell Biol. 2015. V. 16. № 3. P. 155–166. https://doi.org/10.1038/nrm3951
Soutourina J. Transcription regulation by the Mediator complex // Nat. Rev. Mol. Cell Biol. 2018. V. 19. № 4. P. 262–274. https://doi.org/10.1038/nrm.2017.115
Kagey M.H., Newman J.J., Bilodeau S. et al. Mediator and cohesin connect gene expression and chromatin architecture // Nature. 2010. V. 467. № 7314. P. 430–435. https://doi.org/10.1038/nature09380
Phillips-Cremins J.E., Sauria M.E.G., Sanyal A. et al. Architectural protein subclasses shape 3D organization of genomes during lineage commitment // Cell. 2013. V. 153. № 6. P. 1281–1295. https://doi.org/10.1016/j.cell.2013.04.053
El Khattabi L., Zhao H., Kalchschmidt J. et al. A pliable Mediator acts as a functional rather than an architectural bridge between promoters and enhancers // Cell. 2019. V. 178. № 5. P. 1145–1158.e20. https://doi.org/10.1016/j.cell.2019.07.011
Hnisz D., Shrinivas K., Young R.A. et al. A phase separation model for transcriptional control // Cell. 2017. V. 169. № 1. P. 13–23. https://doi.org/10.1016/j.cell.2017.02.007
Meier U.T. RNA modification in Cajal bodies // RNA Biol. 2017. V. 14. № 6. P. 693–700. https://doi.org/10.1080/15476286.2016.1249091
Kantidze O.L., Razin S.V. Weak interactions in higher-order chromatin organization // Nucl. Acids Res. 2020. V. 48. № 9. P. 4614–4626. https://doi.org/10.1093/nar/gkaa261
Bonchuk A., Denisov S., Georgiev P., Maksimenko O. Drosophila BTB/POZ domains of “ttk Group” can form multimers and selectively interact with each other // J. Mol. Biol. 2011. V. 412. № 3. P. 423–436. https://doi.org/10.1016/j.jmb.2011.07.052
Dorsett D. The many roles of cohesin in Drosophila gene transcription // Trends Genet. TIG. 2019. V. 35. № 7. P. 542–551. https://doi.org/10.1016/j.tig.2019.04.002
Wang Q., Carroll J.S., Brown M. Spatial and temporal recruitment of androgen receptor and its coactivators involves chromosomal looping and polymerase tracking // Mol. Cell. 2005. V. 19. № 5. P. 631–642. https://doi.org/10.1016/j.molcel.2005.07.018
Mandal S.S., Chu C., Wada T. et al. Functional interactions of RNA-capping enzyme with factors that positively and negatively regulate promoter escape by RNA polymerase II // Proc. Natl Acad. Sci. USA. 2004. V. 101. № 20. P. 7572–7577. https://doi.org/10.1073/pnas.0401493101
Chen F.X., Woodfin A.R., Gardini A. et al. PAF1, A molecular regulator of promoter-proximal pausing by RNA polymerase II // Cell. 2015. V. 162. № 5. P. 1003–1015. https://doi.org/10.1016/j.cell.2015.07.042
Chen Y., Yamaguchi Y., Tsugeno Y. et al. DSIF, the Paf1 complex, and Tat-SF1 have nonredundant, cooperative roles in RNA polymerase II elongation // Genes Dev. 2009. V. 23. № 23. P. 2765–2777. https://doi.org/10.1101/gad.1834709
Elrod N.D., Henriques T., Huang K.-L. et al. The integrator complex attenuates promoter-proximal transcription at protein-coding genes // Mol. Cell. 2019. V. 76. № 5. P. 738–752.e7. https://doi.org/10.1016/j.molcel.2019.10.034
Rienzo M., Casamassimi A. Integrator complex and transcription regulation: Recent findings and pathophysiology // Biochim. Biophys. Acta. 2016. V. 1859. № 10. P. 1269–1280. https://doi.org/10.1016/j.bbagrm.2016.07.008
Baillat D., Hakimi M.-A., Näär A.M. et al. Integrator, a multiprotein mediator of small nuclear RNA processing, associates with the C-terminal repeat of RNA polymerase II // Cell. 2005. V. 123. № 2. P. 265–276. https://doi.org/10.1016/j.cell.2005.08.019
Pavri R., Zhu B., Li G. et al. Histone H2B monoubiquitination functions cooperatively with FACT to regulate elongation by RNA polymerase II // Cell. 2006. V. 125. № 4. P. 703–717. https://doi.org/10.1016/j.cell.2006.04.029
Kwon S.H., Florens L., Swanson S.K. et al. Heterochromatin protein 1 (HP1) connects the FACT histone chaperone complex to the phosphorylated CTD of RNA polymerase II // Genes Dev. 2010. V. 24. № 19. P. 2133–2145. https://doi.org/10.1101/gad.1959110
Splinter E., Heath H., Kooren J. et al. CTCF mediates long-range chromatin looping and local histone modification in the beta-globin locus // Genes Dev. 2006. V. 20. № 17. P. 2349–2354. https://doi.org/10.1101/gad.399506
Tang Z., Luo O.J., Li X. et al. CTCF-mediated human 3D genome architecture reveals chromatin topology for transcription // Cell. 2015. V. 163. № 7. P. 1611–1627. https://doi.org/10.1016/j.cell.2015.11.024
Kim T.H., Abdullaev Z.K., Smith A.D. et al. Analysis of the vertebrate insulator protein CTCF-binding sites in the human genome // Cell. 2007. V. 128. № 6. P. 1231–1245. https://doi.org/10.1016/j.cell.2006.12.048
Shrestha S., Oh D.-H., McKowen J.K. et al. 4C-seq characterization of Drosophila BEAF binding regions provides evidence for highly variable long-distance interactions between active chromatin // PLoS One. 2018. V. 13. № 9. P. e0203843. https://doi.org/10.1371/journal.pone.0203843
Maksimenko O., Bartkuhn M., Stakhov V. et al. Two new insulator proteins, Pita and ZIPIC, target CP190 to chromatin // Genome Res. 2015. V. 25. № 1. P. 89–99. https://doi.org/10.1101/gr.174169.114
Zolotarev N., Maksimenko O., Kyrchanova O. et al. Opbp is a new architectural/insulator protein required for ribosomal gene expression // Nucl. Acids Res. 2017. V. 45. № 21. P. 12285–12300. https://doi.org/10.1093/nar/gkx840
Дополнительные материалы отсутствуют.


