Генетика, 2021, T. 57, № 7, стр. 810-819
Генетическое разнообразие и филогения пуховых коз Центральной и Средней Азии
С. В. Бекетов 1, *, А. К. Пискунов 1, В. Н. Воронкова 1, С. Н. Петров 2, В. Р. Харзинова 2, А. В. Доцев 2, Н. А. Зиновьева 2, М. И. Селионова 3, Ю. А. Столповский 1
1 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, ГСП-1, Россия
2 Федеральный исследовательский центр – ВИЖ им. академика Л.К. Эрнста
142132 Московская область, г/о Подольск, п. Дубровицы, Россия
3 Российский государственный аграрный университет – Московская сельскохозяйственная академия
им. К.А. Тимирязева
127550 Москва, Россия
* E-mail: svbeketov@gmail.com
Поступила в редакцию 18.08.2020
После доработки 11.11.2020
Принята к публикации 17.11.2020
Аннотация
С использованием микросателлитных маркеров исследовано генетическое разнообразие, родственные связи и дрейф генов трех пород и семи популяций шерстных (пуховых) коз Центральной и Средней Азии. Получены параметры аллельного и генетического разнообразия аборигенных популяций монгольских коз (гурван эгч, дархатская, бурах завхан, ульгий уулан, алтай уулан), двух популяций местных тувинских коз, советской и таджикской шерстных пород коз и оренбургской пуховой козы. С использованием метода главных компонент выявлены две основные группы коз, в одной из которых объединены преимущественно монгольские аборигенные популяции, а в другой – среднеазиатские породы коз. Популяции тувинской местной козы разделились между соотвествующими группами. При этом монгольские козы характеризовались высоким внутрипопуляционным разнообразием и достаточно низким уровнем генетических различий между популяциями. Полученные данные свидетельствуют о существовании корреляции генетических отношений между рассматриваемыми популяциями и породами пуховых коз и их географическим распределением.
Исследование генетической изменчивости автохтонных популяций коз Евразии и пород, созданных на их основе, имеет большое значение для сохранения биоразнообразия и изучения эволюционно-филогенетических связей. В первую очередь это относится к породам и местным одомашненным популяциям шерстных коз Средней и Центральной Азии, которые изучены в гораздо меньшей степени по сравнению с пуховыми козами южной части Евразийского континента [1–3].
В последние годы для исследования генетической структуры популяций, групп или отдельных организмов практически повсеместно используют основанный на ПЦР метод ДНК-профилирования, или микросателлитного анализа. Согласно этой методике анализируют высокополиморфные короткие тандемно повторяющиеся (ди-, три- и тетрануклеотидные) последовательности ДНК, число которых может достигать нескольких тысяч. Поскольку индивидуумы имеют уникальное число повторяющихся участков ДНК, метод эффективен для установления различий и родственных связей [4].
Благодаря высокой степени полиморфизма, менделевскому типу наследования и равномерному распределению по всему геному [5] микросателлиты нашли применение для оценки уровней инбридинга, характеристики генетической структуры субпопуляций и популяций, демографической истории, определения эффективного размера популяций, величины и направления дрейфа генов между ними. Очень удобными STR-маркеры оказались для проведения филогеографических исследований, выявления биогеографической и генетической истории фауны больших регионов.
В связи с чем целью настоящей работы стало проведение сравнительного микросателлитного анализа пород и одомашненных популяций шерстных (пуховых) коз с учетом их географической принадлежности. Непосредственно в исследование были включены породы коз среднеазиатского (таджикская шерстная, советская шерстная, оренбургская) и центрально-азиатского регионов (одомашненные популяции монгольских и тувинских пуховых коз).
Характеризуя изучаемые породы и популяции, следует отметить, что современный породный тип шерстных коз среднеазиатской группы сформировался сравнительно недавно. Например, процесс выведения знаменитой оренбургской пуховой козы включал несколько этапов. Первоначально, вплоть до начала ХIX в., проводилась народная селекция среди одомашненных аборигенных пуховых коз южного склона Уральских гор. В 1937 г. для получения более высокой продуктивности по выходу пуха в Оренбургскую область завезли козлов Придонской породы [6], затем в течение 30 лет велись селекционные работы уже внутри сложившейся породы, путем отбора особей с наилучшими показателями по качеству пуха.
К созданию советской шерстной породы в Советском Союзе приступили в 1936 г. с приобретения в США 338 козлов и 400 ангорских коз, закупленных в штатах Техас и Нью-Мексико. Животные были размещены в хозяйствах Таджикистана, Узбекистана, Казахстана и Туркмении. Для повышения качества пуха местных популяций козлов ангорской породы скрещивали с грубошерстными матками аборигенных популяций. Итогом многолетней работы стало утверждение в 1962 г. породы коз – советская шерстная [7].
В 1981–1982 гг. в Республику Таджикистан в целях улучшения местных пуховых коз из Австралии было завезено 47 козлов ангорской породы, дополнительно использовали прилитие крови козлов советской шерстной породы из Казахстана и Киргизии. В результате в 2004 г. была выведена порода таджикская шерстная [8].
Если говорить о пуховом козоводстве Монголии, то здесь, несмотря на наличие двух самостоятельных пород – гоби гурван-сайхан и уулын-бор [9], в большей степени преобладают местные популяции шерстных коз.
В последние годы исследование генетического разнообразия одомашненных аборигенных коз Монголии стало предметом пристального внимания ученых разных стран мира. С использованием метода белковых маркеров крови и микросателлитного анализа была изучена генетическая структура нескольких популяций монгольских местных пуховых коз: баянделгер, улгий красная, эрчим черная, доргон, завхан буурал и сумбер [10–12]. Несмотря на это, генетическая изменчивость и межпопуляционные связи местных монгольских коз изучены еще в недостаточной степени. Так, отобранные нами аборигенные популяции коз – гурван эгч, дархатская, бурах завхан, ульгий уулан и алтай уулан ранее не были исследованы. То же самое можно сказать и о местной тувинской козе.
В отличие от Монголии в соседней с ней Республике Тыва сложилась несколько иная ситуация. Здесь с середины 50-х гг. прошлого века начали проводить качественное улучшение местных тувинских коз путем завоза племенных козлов ангорской и советской шерстной пород. И сейчас имеется только небольшое поголовье тувинских грубошерстных аборигенных коз, которых разводят в чистоте в отдаленных районах республики [13].
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследований являлись образцы биоматериала (кровь) пяти популяций аборигенных пуховых коз Монголии (n = 240): гурван эгч (аймак Умнеговь, ранее Южно-Гобийский аймак) (MGL_SGOB, n = 48), дархатская (аймак Хувсгел) (MGL_HUV, n = 48), бурах завхан (аймак Завхан) (MGL_ZAH, n = 48), ульгий уулан (аймак Увс) (MGL_UVC, n = 48), алтай уулан (аймак Ховд) (MGL_HOVD, n = 48) и двух популяций местных тувинских шерстных коз, разводимых в Республике Тыва (n = 99), включая животных СПК “Артыш” (Кызылский кожуун) (TUV_ART, n = 48) и СППК “Уургай” (Эрзинский кожуун) (TUV_UUR, n = 51).
Выделение ДНК из образцов крови осуществляли с использованием наборов ДНК–Экстран (ЗАО “Синтол”, Россия) согласно протоколу фирмы-изготовителя.
Количественную и качественную оценку полученных препаратов ДНК проводили с применением микроспектрофотометра Nano Drop 8000 (Thermo Fisher, США). Концентрацию геномной ДНК определяли на основании измерения степени поглощения при 260 нм, чистоту ДНК оценивали по соотношению степени поглощения при длинах волн 260 и 280 нм.
Исследование коз осуществляли по 14 STR-маркерам с использованием двух мультиплексных ПЦР-реакций, включающих восемь (INRA006, ILSTS087, INRA063, CSRD247, FCB20, ILSTS19, ILSTS011, MAF065) и шесть (INRA005, ILSTS008, SRCP003, SRCP008, SRCP023, MCM527) локусов микросателлитов. Для сравнительного анализа использовали генотипы оренбургской (ООО ГКХ “Губерлинское”, Оренбургская обл.) (ORB, n = 30), советской шерстной (МУП “Ангорка”, Республика Тыва) (SOV_W, n = 33), таджикской шерстной пород коз (n = 34) (ОПХ “Табошар”, Республика Таджикистан) (TAD_W, n = 33) по восьми локусам микросателлитов (INRA006, LSTS087, CSRD247, OARFCB20, ILSTS011, MAF065, ILSTS008, MCM527), предоставленные объектом инфраструктуры ФГБНУ ФИЦ ВИЖ им. Л.К. Эрнста.
Капиллярный электрофорез проводили на генетическом анализаторе ABI 3130xl (Applied Biosystems, США) с последующим определением длин аллелей микросателлитов в программном обеспечении GeneMapper v. 4.0 (Applied Biosystems).
Для оценки структуры изучаемых пород и аборигеных популяций коз использовали следующие показатели: наблюдаемая (HO) и несмещенная ожидаемая (HE(u)) гетерозиготность [14], среднее число аллелей на локус (Na), число эффективных аллелей на локус (NE), число информативных аллелей с частотой более 5% (NA ≥ 5%), аллельное разнообразие (AR) [15], коэффициент инбридинга (FIS).
Степень генетической дифференциации исследуемых пород и популяций коз оценивали на основании попарных генетических различий (индекс фиксации FST) [16], попарных генетических дистанций по Jost’s D [17]. Филогенетические отношения устанавливали путем определения построения Neighbor-net дендрограмм [18].
Результаты оценки генетической структуры популяций были получены с помощью адмикс-модели. Анализ базы данных STR-вариабельности монгольских и тувинских популяций коз проводили для k от 1 до 7 при длине burn-in периода – 100 000, модель Марковских цепей Монте-Карло (MCMC) – 1 000 000 повторов. Аналогичный анализ шерстных пород коз с использованием баз данных геномной вариабельности ОНИС БиоТехЖ выполняли для k от 1 до 12 при условии, что длина burn-in периода – 50 000 и модель Марковских цепей Монте-Карло (MCMC) – 500 000 повторов. Для каждого значения k выполняли 10 итераций.
Для формирования исходных файлов и проведения анализа использовали R версию 3.3.2 [19], программное обеспечение GenAlEx 6.5 [20] и R пакет “diveRsity”, STRUCTURE 2.3.4 [21] с последующей визуализацией в “pophelper” [22]. Анализ главных компонент (Principal Component Analysis, PCA) проводили с помощью R пакета аdegenet [23] с визуализацией в ggplot2 [24].
РЕЗУЛЬТАТЫ
Результаты оценки аллельного разнообразия в изучаемых популяциях коз представлены в табл. 1.
Таблица 1.
Параметры аллельного разнообразия монгольской и тувинской популяций коз
| Популяция | n | Na | NE | NA ≥ 5% | AR |
|---|---|---|---|---|---|
| MGL_SGOB | 48 | 8.750 ± 0.750 | 4.983 ± 0.599 | 5.750 ± 0.491 | 8.117 ± 0.656 |
| MGL_HUV | 48 | 7.500 ± 0.756 | 3.541 ± 0.548 | 4.750 ± 0.750 | 6.972 ± 0.828 |
| MGL_ZAH | 48 | 8.500 ± 0.845 | 5.102 ± 0.597 | 6.125 ± 0.639 | 8.194 ± 0.770 |
| MGL_UVC | 48 | 8.500 ± 0.627 | 4.810 ± 0.611 | 5.750 ± 0.453 | 8.013 ± 0.560 |
| MGL_HOVD | 48 | 7.625 ± 0.498 | 4.263 ± 0.479 | 5.000 ± 0.463 | 7.077 ± 0.456 |
| TUV_ART | 48 | 7.875 ± 0.639 | 3.983 ± 0.365 | 4.875 ± 0.398 | 7.104 ± 0.499 |
| TUV_UUR | 49 | 8.250 ± 0.921 | 4.699 ± 0.533 | 5.625 ± 0.532 | 7.824 ± 0.771 |
Примечание. Для табл. 1, 4: Na – среднее число аллелей на локус; NE – число эффективных аллелей на локус; NA ≥ 5% – число информативных аллелей с частотой более 5%; AR – аллельное разнообразие.
Можно видеть, что значение показателя AR в локусах микросателлитов варьировало от 6.972 ± ± 0.828 в популяции MGL_HUV до 8.194 ± 0.770 – в MGL_ZAH. При этом минимальное число эффективных аллелей было выявлено в популяциях MGL_HUV и TUV_ART – 3.541 ± 0.548 и 3.983 ± ± 0.365 соответственно. Число информативных аллелей в исследованных группах коз было относительно высоким и варьировало от 4.750 ± 0.750 в монгольской популяции коз MGL_HUV до 6.125 ± 0.639 – в MGL_ZAH. В целом исследованные географические группы коз монгольской и тувинской популяций характеризовались сопоставимым уровнем генетического разнообразия. Выявленные различия между группами были несущественны и носили характер тенденций (табл. 1).
Анализ значений коэффициента инбридинга FIS показывает наличие дефицита гетерозигот в двух популяциях монгольских коз: MGL_UVC – 0.076 [0.028; 0.124] и MGL_HOVD – 0.088 [0.022; 0.154] (табл. 2).
Таблица 2.
Параметры генетического разнообразия монгольской и тувинской популяций коз
| Популяция | n | HO | HE(u) | FIS (95%CI) |
|---|---|---|---|---|
| MGL_SGOB | 48 | 0.740 ± 0.052 | 0.779 ± 0.036 | 0.052 (–0.033; 0.137) |
| MGL_HUV | 48 | 0.690 ± 0.057 | 0.673 ± 0.051 | –0.030 (–0.129; 0.069) |
| MGL_ZAH | 48 | 0.737 ± 0.039 | 0.784 ± 0.037 | 0.050 (–0.055; 0.155) |
| MGL_UVC | 48 | 0.714 ± 0.047 | 0.769 ± 0.038 | 0.076 (0.028; 0.124) |
| MGL_HOVD | 48 | 0.685 ± 0.041 | 0.750 ± 0.031 | 0.088 (0.022; 0.154) |
| TUV_ART | 48 | 0.719 ± 0.044 | 0.734 ± 0.038 | 0.023 (–0.025; 0.071) |
| TUV_UUR | 49 | 0.735 ± 0.054 | 0.751 ± 0.059 | 0.009 (–0.060; 0.078) |
Примечание. Для табл. 2, 5: HO – наблюдаемая гетерозиготность; HE(u) – несмещенная ожидаемая гетерозиготность; FIS – коэффициент инбридинга; CI – доверительный интервал.
Генетические дистанции между исследованными группами коз монгольских и тувинских популяций, рассчитанные на основании FST и Jost’s D, обобщены в табл. 3.
Таблица 3.
Генетические дистанции между исследуемыми популяциями коз
| Популяция | MGL_ SGOB | MGL_HUV | MGL_ ZAH | MGL_UVC | MGL_HOVD | TUV_ ART | TUV_UUR |
|---|---|---|---|---|---|---|---|
| MGL_SGOB | * | 0.055 | 0.014 | 0.015 | 0.025 | 0.051 | 0.030 |
| MGL_HUV | 0.115 | * | 0.050 | 0.066 | 0.060 | 0.106 | 0.064 |
| MGL_ZAH | 0.024 | 0.106 | * | 0.017 | 0.026 | 0.056 | 0.029 |
| MGL_UVC | 0.036 | 0.146 | 0.040 | * | 0.017 | 0.050 | 0.025 |
| MGL_HOVD | 0.045 | 0.106 | 0.058 | 0.032 | * | 0.055 | 0.039 |
| TUV_ART | 0.134 | 0.252 | 0.142 | 0.130 | 0.121 | * | 0.054 |
| TUV_UUR | 0.073 | 0.145 | 0.088 | 0.049 | 0.100 | 0.138 | * |
Генетические различия между монгольскими и тувинскими группами коз были выше (FST = = 0.025–0.106, Jost’s D = 0.049–0.252), чем между популяциями коз внутри монгольской (FST = = 0.014–0.066, Jost’s D = 0.024–0.146) и тувинской групп животных (FST = 0.054, Jost’s D = 0.138).
Самостоятельный интерес представляет сравнение STR-вариабельности объединенных групп монгольских и тувинских популяций (табл. 4).
Таблица 4.
Параметры аллельного разнообразия исследуемых групп популяций и пород коз
| Группа популяций/ порода | n | Na | NE | NA >= 5% | AR |
|---|---|---|---|---|---|
| MGL | 240 | 10.750 ± 0.773 | 5.335 ± 0.827 | 5.375 ± 0.460 | 8.314 ± 0.723 |
| TUV | 97 | 9.250 ± 0.648 | 4.890 ± 0.516 | 5.500 ± 0.463 | 7.961 ± 0.586 |
| ORB | 30 | 7.500 ± 0.866 | 4.250 ± 0.445 | 5.000 ± 0.535 | 7.500 ± 0.866 |
| TAD_W | 33 | 7.875 ± 0.934 | 4.139 ± 0.422 | 4.625 ± 0.324 | 7.776 ± 0.911 |
| SOV_W | 33 | 8.375 ± 0.885 | 4.524 ± 0.455 | 4.875 ± 0.515 | 8.217 ± 0.843 |
Как показано в табл. 5, в группе монгольских популяций выявлен наиболее высокий уровень генетического разнообразия в сравнении с объединенной группой тувинских популяций и породами среднеазиатских шерстных коз, в то время как тувинская группа местных шерстных коз занимала промежуточное положение по данному показателю. Также группы монгольских и тувинских популяций характеризовались наибольшими значениями числа эффективных аллелей.
Таблица 5.
Параметры генетического разнообразия исследуемых групп популяций и пород коз
| Группа популяций/порода | n | HO | HE(u) | FIS (95%CI) |
|---|---|---|---|---|
| MGL | 240 | 0.713 ± 0.037 | 0.773 ± 0.041 | 0.074 (0.026; 0.122) |
| TUV | 97 | 0.727 ± 0.045 | 0.764 ± 0.051 | 0.044 (0.013; 0.075) |
| ORB | 30 | 0.662 ± 0.063 | 0.751 ± 0.038 | 0.127 (0.02; 0.234) |
| TAD_W | 33 | 0.769 ± 0.048 | 0.75 ± 0.029 | –0.023 (–0.112; 0.066) |
| SOV_W | 33 | 0.739 ± 0.059 | 0.771 ± 0.029 | 0.050 (–0.061; 0.161) |
Причем как в монгольской, так и в тувинской группе был выявлен дефицит гетерозигот, однако в абсолютном значении данные популяции характеризовались меньшим уровнем этого показателя по сравнению с оренбургской породой: 7.4 и 4.4% соответственно, против 12.7%.
Как видно из данных, представленных в табл. 6, в группе исследованных монгольских популяций коз наиболее генетически удалена от других популяция MGL_HUV. На дендрограммах это отражено в формировании длинной отдельной ветви (рис. 1, 2).
Таблица 6.
Генетические дистанции между исследуемыми популяциями и породами коз
| Популяция/ порода | ORB | TAD_W | SOV_W | MGL_SGOB | MGL_HUV | MGL_ZAH | MGL_UVC | MGL_HOVD | TUV_ART | TUV_UUR |
|---|---|---|---|---|---|---|---|---|---|---|
| ORB | * | 0.051 | 0.038 | 0.020 | 0.091 | 0.045 | 0.045 | 0.041 | 0.068 | 0.047 |
| TAD_W | 0.092 | * | 0.027 | 0.051 | 0.094 | 0.048 | 0.062 | 0.045 | 0.050 | 0.054 |
| SOV_W | 0.094 | 0.041 | * | 0.033 | 0.086 | 0.028 | 0.038 | 0.04 | 0.043 | 0.036 |
| MGL_SGOB | 0.066 | 0.110 | 0.061 | * | 0.055 | 0.014 | 0.015 | 0.025 | 0.051 | 0.030 |
| MGL_HUV | 0.212 | 0.202 | 0.180 | 0.115 | * | 0.050 | 0.066 | 0.060 | 0.106 | 0.064 |
| MGL_ZAH | 0.110 | 0.099 | 0.053 | 0.024 | 0.106 | * | 0.017 | 0.026 | 0.056 | 0.029 |
| MGL_UVC | 0.113 | 0.125 | 0.086 | 0.036 | 0.146 | 0.04 | * | 0.017 | 0.050 | 0.025 |
| MGL_HOVD | 0.099 | 0.082 | 0.096 | 0.045 | 0.106 | 0.058 | 0.032 | * | 0.055 | 0.039 |
| TUV_ART | 0.149 | 0.115 | 0.121 | 0.134 | 0.252 | 0.142 | 0.13 | 0.121 | * | 0.054 |
| TUV_UUR | 0.093 | 0.117 | 0.062 | 0.073 | 0.145 | 0.088 | 0.049 | 0.100 | 0.138 | * |
Рис. 1.
Neighbor-net дендрограмма взаимоотношений исследуемых популяций и пород коз, построенная на основе матрицы попарных генетических дистанций FST.
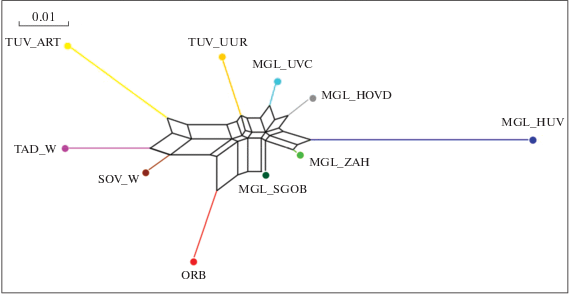
Рис. 2.
Neighbor-net дендрограмма взаимоотношений исследуемых популяций и пород коз, построенная на основе матрицы попарных генетических дистанций Jost’s D.
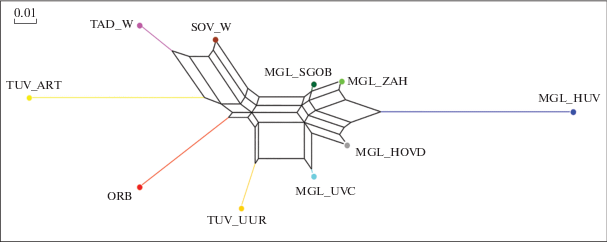
В целом же популяции аборигенных монгольских коз характеризовались, прежде всего, сродством между собой. Следует отметить также филогенетическую связь тувинской популяции коз (Эрзинскиий кожуун) TUV_UUR с монгольской аборигенной популяцией ульгий уулан (аймак Увс) MGL_UVC, что отчасти объясняется географической близостью зон их разведения.
При этом популяция местных тувинских коз (Кызылского кожууна) TUV_ART проявляет большее генетическое родство к таджикской и советской шерстным породам. На дендрограммах это проявляется в локализации данной популяции в общем кластере с этими среднеазиатскими козами (рис. 1, 2). Следует указать также на близость группы MGL_SGOB монгольских коз с оренбургской породой: генетические дистанции между MGL_SGOB и оренбургской породой были меньше, чем между MGL_SGOB и группами коз тувинской популяции (рис. 1).
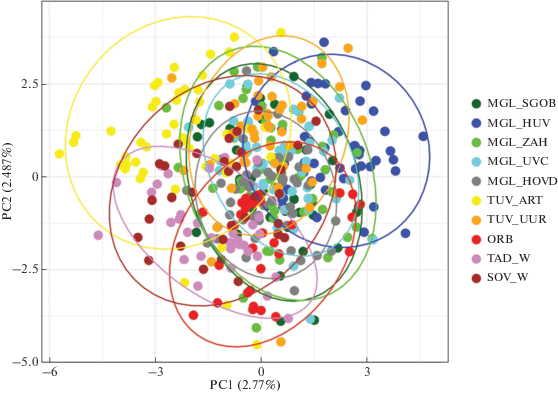
Результаты анализа главных компонент согласуются с результатами расчета генетических дистанций (рис. 3).
Рис. 3.
Проекция особей исследуемых пород и популяций коз на плоскости двух координат по данным PCA-анализа.
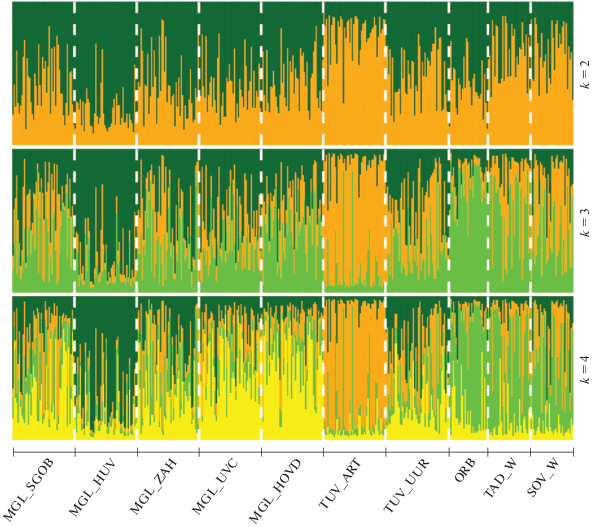
Анализ популяционно-генетической структуры изученных популяций и пород коз (рис. 4) при k = 2 показывает разделение пород на две группы, одна из которых включает все популяции местных монгольских коз, тувинскую популяцию TUV_ UUR и оренбургскую породу, а вторая – тувинскую популяцию TUV_ART, советскую шерстную и таджикскую шерстные породы, при этом наблюдается четкая адмиксия между группами. Наименьшая адмиксия отмечена для популяций пуховых коз TUV_ART и MGL_HUV.
При k = 3 популяции MGL_HUV, TUV_ART и оренбургская порода формируют собственные кластеры с наименьшей степенью адмиксии других предковых компонентов, которые сохраняются при k = 4.
ОБСУЖДЕНИЕ
Известно, что к изменчивости генетической структуры популяций приводят такие процессы как географическая изоляция, миграция и адмиксия, в ходе которых индивидуумы приобретают большее или меньшее сродство или различие, однако сделать объективные выводы о природе и времени проявления таких изменений чаще всего достаточно сложно.
С учетом того, что создание современных шерстных пород Средней Азии и попытки улучшения аборигенных популяций пуховых коз Центральной Азии проходили в новейшее время, можно относительно объективно отразить причины, повлекшие существенные изменения в их генетической структуре.
Важно отметить, что до сих пор определяющую роль в современном животноводстве Монголии продолжает играть номадизм – традиционное кочевое и полукочевое животноводство, остающееся главным видом хозяйственной деятельности сельского населения [25]. Причем неконтролируемый трансграничный перегон скота из Монголии в Туву и обратно был осуществим вплоть до введения ограничений в 1949 г. [12]. Возможно, этим обстоятельством объясняется филогенетическое родство популяции тувинской козы СППК “Уургай” (Эрзинский кожуун) с козами пограничного монгольского аймака Увс.
Как уже отмечалось выше, по своему породному составу козоводство в Монголии представлено многочисленными отродьями азиатской пуховой козы и двумя породами: гоби гурван-сайхан (Южно-Гобийский аймак) и уулын-бор (Баян-Улэгейский и Кобдоский аймаки), полученными путем скрещивания аборигенных коз с козлами придонской и горно-алтайской пород [9].
Однако, несмотря на повышение продуктивности, качество пуха у породных животных стало ниже, чем у исходных местных монгольских коз [12]. В связи с чем для улучшения общей популяции коз учеными Монголии была разработана собственная программа отбора аборигенных животных по наиболее важным хозяйствено ценным признакам.
Местные популяции монгольских пуховых коз отличаются крепкой конституцией и хорошей приспособленностью к разведению в условиях круглогодового пастбищного содержания. При этом даже в пределах одной популяции животные часто отличаются большим многообразием фенотипической изменчивости в окраске волосяного покрова [25].
Однако несмотря на повышенный интерес к монгольским популяциям коз их генетическая структура и родственные связи изучены еще в недостаточной степени, и имеются лишь единичные работы, посвященные этому вопросу. В частности, оценку генетической близости между пятью аборигенными популяциями коз Монголии определяли по 12 полимофным белковым маркерам крови: трансферрину, амилазе, щелочной фосфатазе, преальбумину-3, эстеразе D, α-глобину, β-глобину, пептидазе-B, тетразолиевой оксидазе, эстеразе-1, эстеразе-2 и каталазе. По результатам анализа было установлено, что, несмотря на морфометрические различия, выявляемые у местных коз в разных районах Монголии, генетические межпопуляционные дистанции между ними оказались очень близкими (D = 0.0002–0.0038) [10].
Похожий результат был получен в работе Tакахаши с соавт. [12] с использованием 10 микросателлитных маркеров. В частности, было установлено, что несмотря на высокое генетическое разнообразие в популяциях (оценка по ожидаемой гетерозиготности 0.719–0.746) дифференциация между ними составила всего лишь 1.7% от общей генетической изменчивости, что указывает на относительную межпопуляционную однородность и наличие интенсивного генного потока между ними [12].
Известно, что межпопуляционные генетические связи зависят, в первую очередь, от величины эффективного потока генов между субпопуляциями или подгруппами. И если интенсивность генного потока между группами высока, он направлен на гомогенизацию генетической изменчивости между ними [26].
Это подтверждается и данными, полученными в настоящей работе, согласно которым несмещенная ожидаемая гетерозиготность составляет у рассматриваемых популяций монгольских коз (0.673–0.784), а дифференциация между ними составила 3.5%.
То есть фактически монгольские популяции коз до сих пор не дифференцированы до уровня пород и имеют генетическую структуру, свойственную для неполностью одомашненных животных, что является следствием как ведения традиционного животноводства, так и очень короткой во времени истории селекции [12].
Это хорошо заметно при сравнении монгольских популяций с местными тувинскими козами, которые подверглись неоднородной метисизации, соответственно ожидаемая гетерозиготность исследованных нами тувинских популяций составила (0.734–0.751), а межпопуляционная дифференциация – 9.6%.
Если говорить в целом, то у рассматриваемых в настоящей работе средне- и центрально-азиатских пород и аборигенных популяций пуховых коз уровень гетерозиготности существенно не различался.
При этом с помощью метода главных компонент были выявлены две основные группы коз, в одной из которых были объединены преимущественно монгольские местные популяции коз, а во второй – среднеазиатские породы. Популяции тувинской местной козы разделились между соответствующими группами.
Во многом объединяющим фактором, позволившим сформировать относительно целостную группу среднеазиатских коз, является то, что в создании отдельных ее пород принимали участие ангорские козы либо напрямую, как в случае с советской и таджиской шерстными породами, либо опосредованно, как при выведении оренбургской породы (скрещивание с козами придонской породы коз). Отчасти этим же фактором можно объяснить и промежуточное положение тувинских популяций коз (прилитие крови ангорской и советской шерстной пород).
Например, ранее в работе монгольских и австралийских исследователей в ходе микросателлитного анализа по сравнению генетического сходства ангорской и кашемировой коз с монгольскими шерстными козами наименьшее генетическое расстояние отмечали у кашемировой козы – 0.096, а наибольшее – у ангорской породы – 0.276 [11]. Причем при сравнении попарных генетических дистанций ближе всего к ангорсой козе была монгольская порода коз гоби гурван-сайхан (0.192), которая, в свою очередь, была создана с участием придонской породы [12].
В нашем случае при сравнении попарных генетических дистанций по FST оренбургская порода коз ORB имеет больше сходств с MGL_SGOB (0.02), чем с советской шерстной породой SOV_W (0.03) (табл. 6), что предположительно свидетельствует о ее изначальном родстве с центрально-азиатской группой коз и прежде всего с монгольскими популяциями. В то время как тувинская популяция коз СПК “Артыш” TUV_AR оказалась генетически ближе советской шерстной породе SOV_W (0.043), а также, наряду с монгольской популяцией коз ульгий уулан, MGL_UVC (0.05) – таджикской шерстной козе TAD_W (0.05), что является результатом сравнительно недавнего прилития к тувинским козам крови советской шерстной и ангорской коз. Причем близость к монгольским популяциям коз у тувинской козы СПК “Артыш” TUV_AR оказалась больше (0.051–0.106), чем к тувинской козе СППК “Уургай” TUV_UUR (0.138).
Таким образом, в целом можно говорить о существовании корреляции генетических отношений между рассматриваемыми популяциями и породами и их географическим распределением, а полученные в исследовании результаты по генетической структуре центрально-азиатских и среднеазиатских шерстных коз и их взаимоотношениям могут иметь не только теоретическое, но и практическое значение для решения вопросов происхождения, установления родства, распространения, а также сохранения генофондов пород и популяций коз.
Исследования выполнены при финансовой поддержке РНФ в рамках проектов № 19-76-20061 и 19-76-20006.
Все применимые международные, национальные и/или институциональные принципы ухода и использования животных были соблюдены.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Mahmoudi B., Bayat M., Sadeghi R. et al. Genetic diversity among three goat populations assessed by microsatellite DNA markers in Iran // Glbl. Vet. 2010. V. 4. № 2. P. 118–124.
Nomura K., Ishii K., Dadi H. et al. Microsatellite DNA markers indicate three genetic lineages in East Asian indigenous goat populations // Anim. Genet. 2012. V. 43. P. 760–767. https://doi.org/10.1111/j.1365-2052.2012.02334.x
Elzarei M.F., Mousa E.F. Molecular and morphological characteristics of aradi goats with prediction equation of growth pattern // Intl. J. Biol. 2017. V. 9. № 2. P. 29–38. https://doi.org/10.5539/ijb.v9n2p29
Wagner R.P. Understanding inheritance: An introduction to classical and molecular genetics // Los Alamos Sci. LANL. 1992. № 20. P. 3–67.
Dakin E.E., Avise J.C. Microsatellite null alleles in parentage analysis // Heredity. 2004. V. 93. P. 504–509. https://doi.org/10.1038/sj.hdy.6800545
Зеленский Г.Г., Мишарев С.С. Оренбургская пуховая коза. Чкалов: Чкаловское кн. изд-во, 1949. 80 с.
Харзинова В.Р., Петров С.Н., Доцев А.В. и др. Популяционно-генетическая характеристика некоторых пород коз на основе анализа микросателлитов // Овцы, козы, шерстяное дело. 2019. № 3. С. 7–12.
Косимов М.А., Косимов Ф.Ф. Таджикская шерстная порода коз // Зоотехния. 2015. № 3. С. 5–7.
Альков Г.В. Пуховое козоводство в Алтайском горном регионе // Сб. науч. тр. Всерос. научно-исследовательского института овцеводства и козоводства. Ставрополь. 2007. Т. 3. С. 142–146.
Nyamsamba D., Nomura Nozawa K., Yokohama M. et al. Genetic relationship among Mongolian native goat populations estimated by blood protein polymorphism // Small Ruminant Res. 2003. V. 47. P. 3171–3181.
Bolormaa S., Ruvinsky A., Walkden-Brown S., van der Werf J. Genetic relationships among Australian and Mongolian fleece-bearing goats // Asian-Aust. J. Anim. Sci. 2008. V. 21. № 11. P. 1535–1543. https://doi.org/10.5713/ajas.2008.70635
Takahashi H., Nyamsamba D., Mandakh B. et al. Genetic structure of Mongolian goat populations using microsatellite loci analysis // Asian-Aust. J. Anim. Sci. 2008. V. 21. № 7. P. 947–953. https://doi.org/10.5713/ajas.2008.70285
Самбу-Хоо Ч.С. Продуктивные и биологические особенности коз разного происхождения в условиях Республики Тыва: Дис. … канд. с.-х. наук: ВИЖ, 2016. 145 с.
Nei M. Estimation of average heterozygosity and genetic distance from small number of individuals // Genetics. 1978. V. 89. № 3. P. 583–590.
Kalinowski S.T. Counting alleles with rarefaction: Private alleles and hierarchical sampling designs // Conserv. Genet. 2004. № 5. P. 539–543. https://doi.org/10.1023/B:COGE.0000041021.91777.1a
Weir B.S., Cockerham C. C. Estimating F-statistics for the analysis of population structure // Evolution. 1984. V. 38. № 6. P. 1358–1370. https://doi.org/10.2307/2408641
Jost L. GST and its relatives do not measure differentiation // Mol. Ecol. 2008. № 17. P. 4015–4026. https://doi.org/10.1111/j.1365-294X.2008.03887.x
Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies // Mol. Biol. Evol. 2006. V. 23. № 2. P. 254–267. https://doi.org/10.1093/molbev/msj030
R Core Team. R: A language and environment for statistical computing. R Foundation for Statistical Computing. Vienna. 2018. https://www.R-project.org.
Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research – an update // Bioinformatics. 2012. V. 28. № 19. P. 2537–2539. https://doi.org/10.1093/bioinformatics/bts460
Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data // Genetics. 2000. V. 155. № 2. P. 945–959.
Keenan K., McGinnity P., Cross T.F. et al. DiveRsity: An R package for the estimation and exploration of population genetics parameters and their associated errors // Methods Ecol. Evol. 2013. № 4. P. 782–788. https://doi.org/10.1111/2041-210X.12067
Jombart T. Adegenet: A R package for the multivariate analysis of genetic markers // Bioinformatics. 2008. V. 24. № 11. P. 1403–1405. https://doi.org/10.1093/bioinformatics/btn129
Wickham H. Ggplot2: Elegant graphics for Data Analysis. Springer-Verlag, 2009. https://doi.org/10.1007/978-3-319-24277-4
Гомбожапов А. К вопросу о проблеме номадизма в современной Монголии // Власть. 2009. № 10. С. 129–132.
Хедрик Ф. Генетика популяций. М.: Техносфера, 2003. 592 с.
Дополнительные материалы отсутствуют.