Генетика, 2021, T. 57, № 7, стр. 786-796
Соотношение летальных и мутагенных повреждений в ДНК плазмид и бактериофагов, индуцированных 8-метоксипсораленом + УФ (λ ≥ 320 нм)
В. Ю. Котова 1, 3, *, С. К. Абилев 2, Г. Б. Завильгельский 1, 3, **
1 Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”
117545 Москва, Россия
2 Институт общей генетики им. Н.И. Вавилова Российской академии наук
119991 Москва, Россия
3 Научно-исследовательский центр “Курчатовский институт”
123098 Москва, Россия
* E-mail: v-kotova@mail.ru
** E-mail: zavilgel@mail.ru
Поступила в редакцию 03.12.2020
После доработки 12.01.2021
Принята к публикации 13.01.2021
Аннотация
Проведено измерение K-соотношения количеств диаддуктов (D) и моноаддуктов (M), индуцированных 8-метоксипсораленом (8-МОП) в ДНК, упакованной в головке бактериофага λ и в ДНК плазмиды pBR322 при УФ-облучении (λ ≥ 320 нм). Определены вероятности P UVR-эксцизионной репарации моноаддуктов 8-МОП, а также S и Sm SOS-репарации диаддуктов и моноаддуктов 8‑МОП. Измерение P проводилось с использованием ангулярного производного ангелицина. Показано, что P = 0.86. Для определения S и Sm использовали бактериофаг λ11, обработанный 8-МОП + УФ (λ ≥ 320 нм) или 8-МОП + УФ (λ ≥ 380 нм), и высеянный или на предварительно облученные коротковолновым УФ (λ = 254 нм) бактерии, или на бактерии с конститутивным синтезом генов SOS-регулона, а также на бактерии, содержащие плазмиду pKM101. У бактериофага λ11 определяли степень W-реактивации (α) и частоту W-мутагенеза clear-мутаций (m). Показано, что диаддукты (“сшивки”) 8-МОП репарируются бактериальной SOS-системой с вероятностью S = 0.28–0.29, а моноаддукты 8-МОП репарируются бактериальной SOS-системой с вероятностью Sm = 0.41 лишь при участии фермента MucA’2B, гены которого расположены в конъюгативной плазмиде pKM101.
При фотосенсибилизации бактерий и бактериофагов при помощи 8-метоксипсоралена (8‑МОП) плюс УФ (λ ≥ 320 нм) в ДНК образуются два типа фотопродуктов: моноаддукты (молекула 8-МОП ковалентно связана с одним пиримидиновым основанием) и диаддукты, или межнитевые “сшивки” (молекула 8-МОП ковалентно связана с двумя пиримидиновыми основаниями из комплементарных нитей) [1–4].
Принципиальное значение при анализе действия 8-MOП на ДНК имеет величина K, характеризующая соотношение моноаддуктов и диаддуков:
где M – число моноаддуктов, D – число диаддуктов.
Величина K зависит от ряда факторов. Во-первых, K зависит от длины волны УФ, используемого при освещении раствора с 8-MOП. Например при использовании УФ с длиной волны больше 380 нм в ДНК образуются практически исключительно моноаддукты 8-МОП, т.к. для вторичной реакции, сшивающей моноаддукт с комплементарной нитью, требуется значительно более короткая длина волны УФ (320–350 нм) [5]. Во-вторых, K зависит от состояния ДНК. Например, ДНК, упакованная внутри головки бактериофагов, характеризуется меньшей величиной K по сравнению со свободной ДНК [6].
Необходимо также учитывать возможности репарации моноаддуктов и диаддуктов в бактериальной клетке. Так как моноаддукты и диаддукты 8-МОП блокируют процесс репликации, проводимый ДНК-полимеразой III, то естественно считать, что при высеве плазмид, обработанных 8-MOП, и фага на УФ-необлученных бактериях E. coli ΔuvrΔrecA в связи с отсутствием систем эксцизионной репарации (UVR) и пострепликативной репарации (RecA) каждый моноаддукт и диаддукт в ДНК носят летальный характер. В E. coli uvr+recA+ моноаддукты 8-МОП репарируются ферментами эксцизионной репарации UVR с такой же эффективностью P (P – вероятность эксцизионной репарации моноаддуктов и циклобутановых пиримидиновых димеров), как и циклобутановые димеры пиримидинов [7]. Но моноаддукты 8-МОП в ДНК фагов и плазмид не способны репарироваться пострепликативной рекомбинационной системой (RecA). Это связано с необходимостью наличия в клетке второй копии неповрежденного в этом локусе гомологичного генома, так как фаги и плазмиды инфицируют клетку, как правило, в одном экземпляре (в опыте используется низкая, много меньше единицы, множественность инфекции).
Диаддукты (“сшивки”) в ДНК фагов и плазмид при их высеве на УФ-необлученных штаммах E. coli ΔuvrΔrecA и E. coli Δuvr летальны на 100%, т.к. блокируют репликацию и не репарируются. Для репарации “сшивки” требуются выщепление “плеча” сшивки из одной из комплементарных нитей, проводимое ферментами эксцизионной репарации UVR, и последующее заполнение образуемой “бреши”. Диаддукты 8-MOП в ДНК фагов и плазмид при их высеве на необлученных бактериях uvr+ recA+, т.е. при отсутствии в клетке активной системы SOS-репарации, также 100%-но летальны, т.к. несмотря на выщепление “плеча” сшивки ферментами UVR, заполнить “брешь” система пострепликативной RecA-рекомбинационной репарации не способна из-за отсутствия в клетке второй копии генома фага или плазмиды. В работе [8] это было экспериментально подтверждено: в ДНК фага λ, высеянного на необлученных бактериях. E. coli uvr+recA+, формирование одного диаддукта в ДНК соответствует примерно одному летальному удару, в то время как моноаддукы 8-МОП c высокой эффективностью репарируются ферментами UVR.
При УФ-облучении в бактериях индуцируется SOS-система репарации ДНК, регуляция которой определяется двумя белками – репрессором LexA и белком RecA [9, 10]. В бактериях E. сoli в составе SOS-регулона расположены гены umuC и umuD [11], кодирующие субъединицы полимеразы PolV (UmuD’2C), которая преодолевает блок репликации, вызванный летальным дефектом в ДНК, вставляя напротив поврежденного нуклеотида, как правило, некомплементарный нуклеотид [12–14]. В результате процесса, который получил наименование “translesion synthesis”, одновременно увеличивается выживаемость бактерий и индуцируются мутации [15]. Показано, что в этом процессе помимо полимеразы PolV важную роль играет активированный белок RecA* (звездочка обозначает активированное состояние белка) [16, 17]. Активация RecA происходит в результате образования связи RecA с однотяжевой ДНК, которая образуется в значительном количестве в УФ-облученных бактериях в процессе репликации и ее блокирования летальными повреждениями. Белки системы SOS-репарации способны репарировать и вызывать мутации не только в хромосоме бактерии, но также в плазмидах и ДНК бактериофагов, особенно эффективно у умеренных бактериофагов (например фаг λ), в меньшей степени – у условно летальных бактериофагов, например, фаги T1, T3, T7 [18], и не способна репарировать активно летальные фаги типа T2, T4, которые убивают клетку и ингибируют основные внутриклеточные процессы уже практически в момент адсорбции [19]. Феномен возрастания выживаемости и частоты мутаций у фагов и плазмид, высеянных на бактериях с предварительно индуцируемой SOS-системой, в честь первооткрывателя эффекта J. Weigle (1953 г.) носит название W-реактивации и W-мутагенеза [9].
В настоящей работе проведено измерение K‑соотношения количеств диаддуктов (D) и моноаддуктов (M) в ДНК, упакованной в головке бактериофага λ, и в ДНК плазмиды. Определены вероятности P эксцизионной репарации моноаддуктов и S и Sm (по [20]) SOS-репарации диаддуктов и моноаддуктов 8-МОП. Измерение P проводилось с использованием ангулярного производного ангелицина, который в силу структурных особенностей, способен образовывать лишь моноаддукты и не способен формировать диаддукты [20]. Для определения S и Sm использовали бактериофаг λ11, обработанный 8-МОП + УФ (λ ≥ 320 нм) или 8‑МОП + УФ (λ ≥ 380 нм), и высеянный или на предварительно облученные коротковолновым УФ (λ = 254 нм) бактерии, или на бактерии с конститутивным синтезом генов SOS-регулона, а также на бактерии, содержащие плазмиду pKM101.
МАТЕРИАЛЫ И МЕТОДЫ
Бактериальные штаммы, бактериофаги и плазмиды
Бактериальные штаммы Escherichia coli K12: AB1157 F--- thr-1 leu-6 proA2 his-4 thi-1 argE3 lacY1 galK2 ara14 xyl-5 mtl-1 tsx-33 rpsL31 supE44; AB1886 uvrA6, AB2480 uvrA6 recA13, остальные маркеры – как у AB1157 (штаммы получены от проф. P. Howard-Flanders, CША), Escherichia coli DM1187 recA441 lexA51 sfiA11 arg+, остальные маркеры – как у AB1157 (штамм получен от проф. В.А. Ланцова, СПб государственный университет). Штаммы TK603 arg+ilvuvrA6, остальные маркеры – как у AB1157; GW514 = TK603 (pKM101 mucAB+) – получены от проф. G.C. Walker, США.
Бактериофаг λ11 (λv2v3) получен от R. Devoret (Франция). Плазмида pBR322.
Среды и условия роста бактерий
LB-среда: 1%-ный триптон, 0.5%-ный дрожжевой экстракт, 0.5%-ный NaCl; для верхнего и нижнего слоев чашек Петри использовали 0.7 и 1.8%-ный LB-агар. Бактерии растили в LB или LB плюс 100 мкг/мл ампициллина при постоянном перемешивании до средней экспоненциальной фазы при 30°С. Трансформацию бактериальных клеток, обработанных ионами Ca2+, проводили согласно [21].
Буферы и реактивы
Трис-буфер: 0.05 M трис-HCl, 0.01 M NaCl, pH 7.5; ТМ-буфер: 0.05 M трис-HCl, 0.01 M NaCl, 0.001 M MgSO4, pH 7.8. 8-Метоксипсорален (8‑МОП) – фирмы “Sigma”. Ангелицин получен от проф. G. Rodighiero (Италия).
Облучение бактерий, бактериофага и плазмид
Раствор ДНК в трис-буфере и суспензию фага в ТМ-буфере, содержащих 40 мкг/мл 8-МОП или 200 мкг/мл ангелицина, облучали при 4°С в пирексовых кюветах. Источником света λ ≥ 320 нм служила ртутная лампа высокого давления СВД-120А с фильтром УФС-6. Лампу СВД-120А с фильтром ЖС-4 использовали в качестве источника света с λ ≥ 380 нм. Расстояние лампы до образца – 20 см. Суспензию бактерий в ТМ-буфере облучали коротковолновым УФ (254 нм), источником которого служила ртутная лампа низкого давления БУВ-15. Дозу УФ-света измеряли дозиметром УФД-4 с магниевым фотоэлементом.
W-реактивация и W-мутагенез
Бактерии в экспоненциальной фазе дважды отмывали ТМ-буфером, концентрировали в пять раз в том же буфере и облучали при 20°С лампой БУВ-15 (254 нм) различными дозами. Адсорбцию фага λ на необлученных и УФ-облученных клетках проводили в течение 15 мин в ТМ-буфере при 37°С. Посев комплекса фаг-бактерия проводили двухслойным методом с добавлением индикаторной культуры AB2480. Для учета clear-мутантов использовали фаг λ11, который способен формировать две одноступенчатые мутации – clear и vir. Спонтанный фон clear-мутаций для различных препаратов фага равнялся: 8 × 10–4–1 × 10–3. У фага, обработанного 8-MOП + УФ (λ ≥ 320 нм), определяли частоту clear-мутаций среди выживших. Степень W-мутагенеза определяли как отношение частоты мутаций при высеве фага на УФ (254 нм)-облученных клетках к частоте мутаций при высеве фага на необлученных клетках. Множественность инфекции не превышала 0.01.
Для измерения W-реактивации у плазмиды pBR322 клетки обрабатывали ионами Ca2+, а затем облучали разными дозами УФ (254 нм). Степень W-реактивации (α) для фага и плазмиды определяли по формуле: α = NnnNoo/NonNno, где Nnn – титр необлученных фага и плазмиды на необлученных бактериях, Noo – титр облученных фага и плазмиды на облученных бактериях, Non – титр облученных фага и плазмиды на необлученных бактериях, Nno – титр необлученных фага и плазмиды на облученных бактериях.
РЕЗУЛЬТАТЫ
Высев фагов и плазмид на необлученных бактериях
В настоящей работе измерение P (вероятность эксцизионной репарации UVR) проводилось с использованием ангулярного производного ангелицина, который в силу структурных особенностей, способен образовывать лишь моноаддукты и не способен формировать диаддукты. Были получены кинетические кривые инактивации плазмиды pBR322, обработанной с помощью ангелицина + УФ (λ ≥ 320 нм) при трансформации необлученных штаммов E. coli AB1157 uvr+recA+ и AB2480 ΔuvrΔrecA (рис. 1), которые описываются следующими формулами:
N/N0 = e–M – для штамма AB2480, т.к. каждый моноаддукт летален,
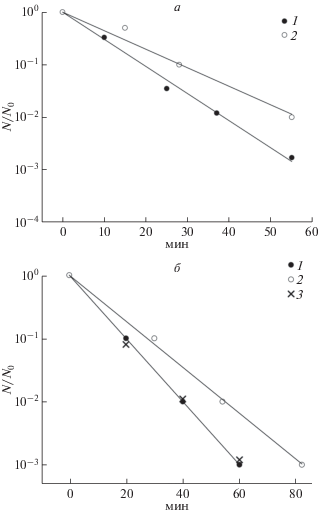
Рис. 1.
Кинетические кривые инактивации плазмиды pBR322, обработанной ангелицином + УФ (λ ≥ 320 нм) при трансформации штаммов E. coli K12. 1 – AB1157 uvr+recA+, 2 – AB2480 uvrA6 recA13. По оси абсцисс отложено время облучения плазмидной ДНК (мин), по оси ординат – степень выживаемости плазмиды N/N0.
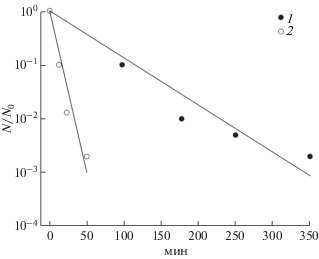
N/N0 = e–(1 –P)M – для штамма AB1157, т.к. летален только каждый нерепарированный моноаддукт.
Из полученного соотношения наклонов кинетических кривых инактивации, σΑΒ1157/σΑΒ2480 = 0.14, получаем: M × (1 – P)/M = = (1 – P) = 0.14, и вероятность эксцизионной репарации моноаддуктов P = 0.86.
Для измерения величины K (M = KD) были определены кинетические кривые 8-MOП + UV (λ ≥ 320 нм) инактивации плазмиды pBR322 и бактериофага λ при высеве на необлученных бактериях AB2480 и AB1157 (рис. 2):
Рис. 2.
Кинетические кривые инактивации обработанных 8-MOP + УФ (λ ≥ 320 нм) бактериофага λ (а) и плазмиды pBR322 (б) при инфекции или трансформации штаммов E. coli K12: 1 – AB1157 uvr+recA+; 2 – AB2480 uvrA6 recA13. По оси ординат отложена выживаемость N/N0, по оси абсцисс время облучения препарата (мин).
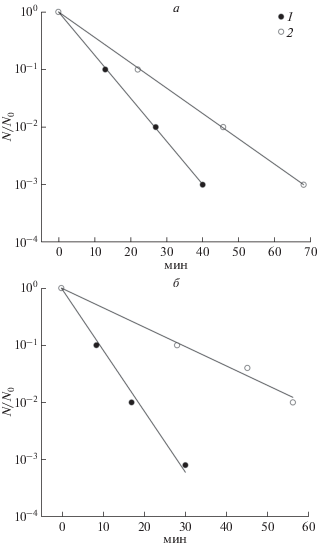
N/N0 = e–D(1 +K) – для штамма AB2480, т.к. каждые моноаддукт и диаддукт летальны;
N/N0= e–D[1 +K(1 –P)] – для штамма AB1157, т.к. каждый диаддукт летален, а моноаддукт летален с вероятностью (1 – P). Из полученного соотношения наклонов, равных σAB2480/σΑΒ1157 = 1.7 для бактериофага, и =3.3 для плазмиды и с учетом полученного ранее значения P = 0.86, определяем величины K:
σAB2480/σΑΒ1157 = (1 + K)/[1 + (1 – P)K]. K = 0.92 – для бактериофага λ, т.е. на один диаддукт образуется в среднем один моноаддукт, и K = 4.3 для плазмидной ДНК, т.е. на один диаддукт образуется в среднем примерно 4.5 моноаддуктов.
Высев фагов и плазмид на УФ-облученные бактерии AB1157 и AB1886 и на необлученные бактерии штамма DM1187
Вначале нами были определены оптимальные дозы УФ (254 нм)-облучения для бактерий AB1157 и AB1886 uvrA6, при которых в клетках максимально индуцируется система SOS-репарации. На рис. 3 и 4 представлены данные по возрастанию выживаемости фага λ11 (далее обозначен λ) (рис. 3) и плазмиды pBR322 (рис. 4), облученных или коротковолновым УФ (254 нм), индуцирующим в качестве основных, блокирующих репликацию, летальных дефектов ДНК циклобутановые пиримидиновые димеры, или УФ-светом с λ ≥ 320 нм в присутствии 8-MOП. У бактериофага λ11 определяли одновременно частоту “clear” мутаций. Доза УФ для облучения препаратов фага и плазмиды выбиралась таким образом, чтобы уровень инактивации объектов при высеве на необлученных бактериях равнялся примерно 10–3. Бактерии AB1157 и AB1886 uvrA6 облучали разными дозами коротковолнового УФ (254 нм). Степень W-реактивации α для фагов и плазмид определяется по формуле: α = NnnNoo/NonNno (см. Материалы и методы).
Рис. 3.
W-реактивация и W-мутагенез бактериофага λ11, облученного УФ (254 нм) (а) и обработанного 8-MOP + УФ (λ ≥ 320 нм) (б) при инфекции штаммов E. coli K12 AB1157 uvr+recA+ (1, 2) и AB1886 uvrA6 (3, 4). Бактерии облучались разными дозами УФ (254 нм). 1 и 3 – кривые W-реактивации, 2 и 4 – кривые W-мутагенеза. По оси абсцисс отложены дозы УФ (254 нм) на бактерии, в Дж/м2. По осям ординат отложены α-степень W-реактивации и m – частота clear-мутаций среди выживших. Доза УФ (254 нм) на фаг при высеве на AB1157 равнялась 140 Дж/м2, выживаемость фага на необлученных бактериях AB1157 равнялась 1.2 × 10–3. Доза УФ (254 нм) на фаг при высеве на AB1886 равнялась 20 Дж/м2, выживаемость фага на необлученных бактериях AB1886 равнялась 1.1 × 10–3. Время облучения фага с 8-MOП + УФ (λ ≥ 320 нм) при высеве на AB1157 равно 55 мин, выживаемость фага на необлученной культуре равно 1.7 × 10–3, а на AB1886 – 45 мин, выживаемость фага на необлученных бактериях AB1886 равна 1.4 × 10–3. Частота clear-мутаций фага на необлученных бактериях AB1886 и AB1157 равна 1.0 × 10–3.
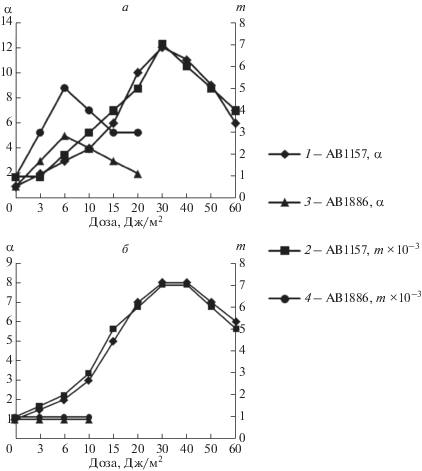
Рис. 4.
W-реактивация плазмиды pBR322, облученной УФ (254 нм) и обработанной 8-MOП + УФ (λ ≥ 320 нм) при инфекции штаммов E. coli K12 AB1157 uvr+recA+ и AB1886 uvrA6. Дозы УФ (254 нм) облучения плазмиды равны 50 Дж/м2 (AB1886) и 210 Дж/м2 (AB1157), выживаемости на необлученных клетках равны соответственно 1.5 × 10–3 и 1.3 × 10–3. Периоды времени облучения плазмиды УФ (λ ≥ 320 нм) + MOП равны: 50 мин (AB1886 uvrA6) и 90 мин (AB1157), показатели выживаемости плазмиды на необлученных бактериях равны: 1.2 × 10–3 (AB1886) и 1.4 × 10–3 (AB1157). Бактерии облучали разными дозами УФ (254 нм). По оси абсцисс отложены дозы УФ (254 нм) на бактерии в Дж/м2, по оси ординат – α-степень W-реактивации.
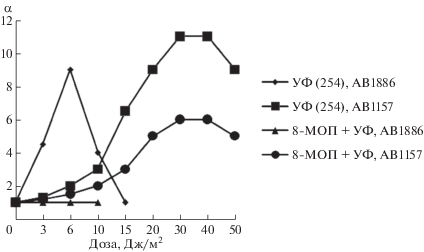
Как видно из рис. 3 и 4, выживаемость фага и плазмид увеличивается, одновременно у фага λ1l растет частота “clear” мутаций в связи с активностью системы SOS-репарации в предварительно УФ (254 нм)-облученных разными дозами бактериях. В случае УФ (254 нм)-облученного фага (рис. 3,а) и плазмиды (рис. 4), содержащих в ДНК циклобутановые димеры пиримидинов, максимальные значения α и частоты мутаций m достигаются при УФ-дозе на бактерии около 30 Дж/м2 для штамма AB1157, и около 6 Дж/м2 для штамма AB1886 uvrA6. Аналогичный результат был получен и для 8-MOП + УФ (λ ≥ 320 нм) – инактивированных фага λ11 и плазмиды, однако только при использовании в качестве хозяина бактерий AB1157, т.к. при использовании бактерий AB1886 uvrA6 SOS-репарация и индукция “clear” мутаций у фага λ11 и SOS-репарация у плазмиды не наблюдались (рис. 3,б и рис. 4).
При высеве 8-MOП + УФ (λ ≥ 320 нм)-обработанных разными дозами фагов и плазмид на предварительно УФ (254 нм)-облученных бактериях необходимо учитывать SOS-репарацию моноаддуктов и диаддуктов в ДНК. В этой серии опытов мы использовали лишь штамм AB1157 и не использовали мутантный штамм AB1886 uvrA6, так как диаддукты 8-MOП не репарируются в связи с отсутствием UVR-ферментов, выщепляющих “плечо” сшивки, а моноаддукты 8-MOП (но не моноаддукты ангелицина) не репарируются SOS-системой бактерии, т.к. фермент PolV не способен преодолевать блок репликации, вызываемый интеркалированной в ДНК молекулой 8-MOП [22].
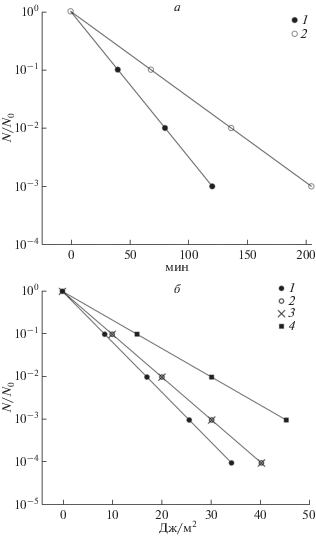
На рис. 5,а представлены кинетические кривые 8-MOП + УФ (λ ≥ 320 нм)-инактивации фага λ при высеве на необлученных и УФ (254 нм)-облученных оптимальной дозой (30 Дж/м2) бактериях AB1157. Зависимости N/N0 от количества диаддуктов Д, образующихся в ДНК, выглядят следующим образом:
Рис. 5.
Кинетические кривые инактивации обработанного 8-MOП + УФ (λ ≥ 320 нм) бактериофага λ при высеве (а) на необлученных и УФ (254 нм)-облученных оптимальной дозой (30 Дж/м2) бактериях AB1157uvr+recA+ и (б) на необлученных бактериях AB1157 uvr+recA+ (1) и необлученных бактериях DM1187 recA441 lexA51 sfiA11 arg+. Посев инфицированных фагом бактерий DM1187 на чашки Петри при 30°С (без аденина) (3) и при 42°С (+аденин) (2). По оси ординат отложена выживаемость N/N0, по оси абсцисс – время облучения препарата фага (мин).
N/N0= e–D[1 +K(1 –P)] – бактерии необлучены (каждый диаддукт летален, и летальны не репарированные UVR-ферментами моноаддукты (см. формулы выше)).
N/N0= e–D[(1 –S) +K(1 –P)] – бактерии УФ (254 нм) облучены оптимальной дозой 30 Дж/м2. В этом варианте учтено, что диаддукты репарируются ферментами SOS-системы с вероятностью S, а моноаддукты репарируются ферментами эксцизионной репарации UVR с вероятностью P, но не репарируются ферментом PolV бактериальной системы SOS (согласно [22]).
Отношение наклонов кривых инактивации σ–/σ+ на необлученных и облученных бактериях равно 1.42 (рис. 5,а). Тогда:
D[(1 – S) + K(1 – P)]/D[1 + K(1 – P)] = [(1 – S) + + K(1 – P)]/[1 + K(1 – P)] = 1/1.42, или:
S = [1 + K(1 – P)] × (1 – 1/1.42) = (1 + 0.9 × 0.14) × × (1 – 1/1.42) = 1.026 × 0.42/1.42 = 0.29.
Следовательно, вероятность репарации диаддукта 8-MOП ферментами SOS-системы и одновременного формирования мутации в ДНК бактериофага λ равняется 0.29, и, соответственно, вероятность летального действия диаддукта 8-MOП на бактериофаг λ равна 1–0.29 = 0.71.
В следующей серии опытов для высева фага λ был использован мутантный штамм E. coli DM1187 recA441 sfiA11 lexA51arg+, остальные маркеры как у AB1157. В этом штамме осуществляется конститутивный синтез всех белков SOS-системы – мутация lexA51 [23], а белок RecA (RecA441) активируется при повышенной температуре (42°С) при наличии в среде аденина (100 мкг/мл) [24]. Мутация sfiA11 обеспечивает жизнеспособность штамма при условии конститутивного синтеза SOS-регулона. Вначале была проведена проверка уровня индукции SOS-системы. Для этой цели клетки штаммов DM1187 и AB1157 (контрольный штамм с закрытой SOS-системой) были трансформированы плазмидой pColD и определены сравнительные интенсивности люминесценции клеток. Штамм DM1187 характеризуется интенсивностью люминесценции, примерно в 100 раз превышающей таковую штамма AB1157, что указывает на открытость системы SOS в штамме DM1187.
На рис. 5,б приведены кинетические кривые 8‑MOП + УФ (λ ≥ 320 нм)-инактивации фага λ при высеве на штаммах AB1157 и DM1187 без предварительного УФ (254 нм)-облучения. Чашки Петри с DM1187, содержащие в верхнем слое аденин (100 мкг/мл), инкубировали при 42°С. Зависимости N/N0 от количества диаддуктов D, образующихся в ДНК, выглядят следующим образом:
N/N0 = e–D[1 +K(1 –P)] – бактерии AB1157 (каждый диаддукт летален, и летальны нерепарированные UVR-ферментами моноаддукты),
N/N0 = e–D[(1 –S) +K(1 –P)] – бактерии DM1187 (летальны нерепарированные UVR-ферментами моноаддукты и нерепарированные SOS-системой диаддукты).
Отношение наклонов кинетических кривых σAB1157/σDM1187 = 1.38. Тогда: S = 1.026 × 0.38/1.38 = = 0.28. Cледовательно, S = 0.28, что практически совпадает с результатом для предварительно УФ (254 нм)-облученных бактерий AB1157 (рис. 5,а).
Cравнение наклонов кинетических кривых 8‑MOP + УФ (λ ≥ 320 нм)-инактивации фага λ, полученных при высевах инфицированных фагом бактерий DM1187 на чашки Петри при 30°С (без аденина) и при 42°С (+аденин) позволяет оценить вклад активированного RecA* в процесс SOS-репарации диаддуктов 8-MOП. Как видно из рис. 5,б, отношение этих наклонов, равное σ30/σ42, практически равно отношению наклонов σΑΒ1157/σ42 = 1.38. Cледовательно можно считать, что активированный белок RecA* абсолютно необходим для SOS-репарации диаддуктов 8-MOП.
W-реактивация моноаддуктов 8-МОП с помощью полимеразы MucA’2B
В следующей серии опытов для высева фага λ были использованы штаммы E. coli TK603 uvrA6 и GW514 uvrA6, содержащий плазмиду pKM101 mucAB+ [25, 26].
Препарат бактериофага был облучен УФ (254 нм) или обработан 8-MOП + УФ (λ ≥ 380 нм), и содержал в ДНК, соответственно, циклобутановые пиримидиновые димеры или в основном моноаддукты 8-MOП [5]. На рис. 6 представлены кинетические кривые 8-MOП + УФ (λ ≥ 380 нм)-инактивации (а) и УФ (254 нм)-инактивации (б) фага λ при высеве на необлученных и предварительно УФ (254 нм)-облученных оптимальной дозой (6 Дж/м2) бактериях GW514. Зависимости N/N0 от количества моноаддуктов М, образующихся в ДНК фага (рис. 6,а), выглядят следующим образом:
Рис. 6.
Кинетические кривые инактивации обработанного 8-MOП + УФ (λ ≥ 380 нм) (а) и облученного УФ (254 нм) (б) бактериофага λ при высеве на необлученных и УФ (254 нм)-облученных оптимальной дозой (6 Дж/м2) бактериях GW514 и TK603 uvrA6. а: 1 – высев на необлученных бактериях GW514, 2 – высев на УФ (254 нм)-облученных дозой 6 Дж/м2 бактериях GW514. б: 1 – высев на необлученных бактериях TK603 uvrA6, 2 – высев на УФ (254 нм)-облученных дозой 6 Дж/м2 бактериях TK603 uvrA6, 3 – высев на необлученных бактериях GW514, 4 – высев на облученных дозой 6 Дж/м2 бактериях GW514. По оси ординат отложена выживаемость N/N0, по оси абсцисс – время облучения препарата фага (мин) (а) и доза УФ (254 нм), Дж/м2 (б).
N/N0= e–М – бактерии необлучены (каждый моноаддукт летален),
N/N0 = ${{{\text{e}}}^{{--М{{{({\text{1}}--S)}}_{{\text{м}}}}}}}$ – бактерии УФ (254 нм)-облучены оптимальной дозой 6 Дж/м2. В этом варианте учтено, что моноаддукты репарируются ферментом MucA’2B с вероятностью Sм, но не репарируются ферментом PolV (UmuD’2С) бактериальной системы SOS (при высеве 8-МОП + УФ (λ ≥ 380 нм)-облученного фага на штамме E. coli TK603 uvrA6 эффекты W-реактивации и W-мутагенеза отсутствуют). Кроме того, при наличии в ДНК бактериофага моноаддуктов 8-МОП при высеве на необлученных бактериях GW514 отсутствует феномен так называемой “P-реактивации”, т.е. выживаемости фага на GW514 и TK603 равны. Отношение наклонов кривых инактивации σ−/σ+ на необлученных и УФ (254 нм)-облученных бактериях GW514 равно 1.7 (рис. 6,а). Тогда: (1 – Sm) = = 1/1.7, и, следовательно, вероятность SOS-репарации моноаддукта 8-МОП c участием полимеразы MucA’2B Sm = 1 – 1/1.7 = 0.41, и, соответственно, вероятность летального действия моноаддукта равна 1 – 0.41 = 0.59.
Несколько иначе выглядят кинетические кривые УФ (254 нм)-инактивации фага, ДНК которого содержит летальные циклобутановые пиримидиновые димеры (рис. 6,б). В этом случае имеет место феномен “P-реактивации”: выживаемость УФ (254 нм)-облученного фага на необлученных УФ (254) бактериях GW514, содержащих плазмиду pKM101 mucAB+, превышает выживаемость этого фага на необлученных бактериях TK603 uvrA6. Следовательно полимераза MucA’2B способна преодолевать блок репликации, вызываемый циклобутановыми пиримидиновыми димерами (а также моноаддуктами ангелицина – данные не представлены), даже при условии отсутствия в клетке SOS-индукции, но не способна преодолевать блок репликации, вызываемый моноаддуктами 8‑МОП без дополнительного участия ферментов бактериальной SOS-системы. Предварительное УФ (254 нм)-облучение бактерий GW514 лишь значительно усиливает W-реактивацию УФ (254 нм)-облученного фага, в частности, в связи с нарастанием экспрессии генов mucA и mucB [27]. Поэтому на рис. 6,б изображены четыре кинетические кривые инактивации УФ (254 нм)-облученного фага: на штамме GW514-необлученном и предварительно облученном дозой УФ (254 нм) 6 Дж/м2, а также на TK603 uvrA6-необлученном и предварительно облученном оптимальной дозой УФ (254 нм) 6 Дж/м2.
ОБСУЖДЕНИЕ
На основании данных, представленных в настоящей работе, при определении относительных вкладов моноаддуктов и диаддуктов 8-MOП в качестве летальных и мутагенных дефектов в ДНК бактериофагов и плазмид, необходимо учитывать, во-первых, неспособность бактериальной SOS-системы, в состав которой входит полимераза PolV, преодолевать блок репликации, вызываемый моноаддуктами 8-MOП, и, во-вторых, неспособность плазмидной полимеразы MucA’2B преодолевать блок репликации, вызываемый моноаддуктами 8-MOП (отсутствие “P-реактивации”), без дополнительной помощи клеточной SOS-системы.
Необходимо отметить, что в работе [28], в которой впервые было показано, что именно диаддукты (“сшивки”) 8-МОП определяют формирование мутаций у бактериофага λ в процессе SOS-репарации, была допущена неточность относительно предполагаемой роли моноаддуктов 8-МОП в SOS-мутагенезе. В работе [28] было показано, что ангелицин, формирующий исключительно моноаддукты, с высокой эффективностью индуцирует SOS-мутагенез при высеве фага на предварительно УФ-облученных бактериях. На этом основании было высказано предположение, что и моноаддукты 8-МОП также должны индуцировать в бактериофагах SOS-мутагенез. Однако, как нами показано в настоящей работе, моноаддукты 8-МОП индуцируют SOS-мутагенез лишь при использовании плазмидной (pKM101) полимеразы MucA’2B, но не бактериальной полимеразы PolV (UmuD’2C).
Представленные в настоящей работе данные о различной способности полимераз MucA’2B и UmuD’2C преодолевать блоки репликации, создаваемые “слабым интеркалятором” ангелицином или “сильным интеркалятором” моноаддуктом 8‑МОП, представляют интересный, но не единственный пример влияния степени перекрывания ароматического кольца плоских интеркаляторов с азотистыми основаниями в ДНК на SOS-процесс формирования мутаций в бактериях. В частности показано, что карциноген N-2-ацетиламинофлуорен, плоская молекула которого встраивается внутрь двойной спирали ДНК, при SOS-репарации индуцирует в хромосоме бактерии формирование фрейм-шифт мутаций в последовательности 5'-GGCGCC, частота которых не зависит от полимеразы UmuD’2C, но значительно возрастает при наличии полимеразы MucA’2B [29].
Остается нерешенным вопрос об относительном вкладе моноаддуктов и диаддуктов в летальное и мутагенное действия 8-MOП + УФ (λ ≥ 320 нм) на бактериальные клетки. Задача в данном случае усложнена в связи с наличием в бактериальной клетке второго генома и эффективным процессом пострепликативной рекомбинационной репарации, зависящей от значительной группы генов (RecA, RecBC, RecE и др.). В частности, можно предположить, что полимераза PolV при участии белков системы рекомбинационной репарации, возможно, в той или иной степени способна преодолевать блок репликации, вызываемый моноаддуктами 8-МОП.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Dall’Acqua F., Marciani S., Rodighiero G. Inter-strand cross-linkages occurring in the photoreactions between psoralen and DNA // FEBS Lett. 1970. V. 9. P. 121–123. https://doi.org/10.1016/0014-5793(70)80330-1
Cole R.S. Psoralen monoadducts and interstrand cross-links in DNA // Biochim. Biophys. Acta. 1971. V. 254. P. 30–39. https://doi.org/10.1016/0005-2787(71)90111-0
Dall’Acqua F., Marciani S., Ciavatta G., Rodighiero G. Formation of interstrand cross-linkings in the photoreactions berween fuurocoumarins and DNA // Z. Naturforsch. 1971. V. 26b. P. 561–565. https://doi.org/10.1515/znb-1971-0613
Rodighiero G., Dall’-Acqua F. Biochemical and medical aspectof psoralens // Photochem. Photobiol. 1976. V. 24. P. 647–653. https://doi.org/10.1111/j.1751-1097.1976.tb06887.x
Chatterjee P.K., Cantor C.R. Photochemical production of psoralen DNA monoadducts capable of subsequent photocross-linking // Nucl. Acids Res. 1978. V. 5. P. 3619–3633. https://doi.org/10.1093/nar/5.10.3619
Hradechna Z., Kittler L. Photobiology of furocoumarins.Various types of cross-linking with DNA and their interference with the development of lambda phage // ActaVirol. 1982. V. 26. P. 305–311.
Белогуров А.А., Зуев А.В., Завильгельский Г.Б. Репарация моноаддуктов и диаддуктов 8-метоксипсоралена у бактериофагов и бактерий // Мол. биология. 1976. Т. 10(4). С. 857–867.
Cassuto E., Gross N., Bardwell E., Howard-Flanders P. Genetic effects of photoadducts and photocross-links in the DNA of phage λ exposed to 360 nm light and trimethyl-psoralen or khellin // Biochim. Biophys. Acta. 1977. V. 475. P. 589–600. https://doi.org/10.1016/0005-2787(77)90319-7
Weigle J.J. Induction of mutation in bacterial virus // PNAS. 1953. V. 39. P. 628–636. https://doi.org/10.1073/pnas.39.7.628
Radman M. SOS repair hypothesis: phenomenology of an inducible DNA repair which is accompanied by mutagenesis // Mol. Mech. for Repair of DNA / Eds. Hanawalt P., Setlow R.B. N.Y.: Plenum Publ. Corp., 1975. Pt. A. P. 355–367. https://doi.org/10.1007/978-1-4684-2895-7_48
Kato T., Shinoura Y. Isolation and characterization of mutants of Escherichia coli deficient in induction of mutations by ultraviolet light // Mol. Gen. Genet. 1977. V. 156. P. 121–131. https://doi.org/10.1007/BF00283484
Tang M., Bruck L., Eritja R., Tarner J. et al. Biochemical basis of SOS-induced mutagenesis in Escherichia coli: Reconstitution of in vitro lesion bypass dependent on the UmuD’2C mutagenic complex and RecA protein // PNAS. 1998. V. 95. P. 9755–9760.https://doi.org/10.1073/pnas.95.17.9755
Tang M., Shen X., Frank E.G., Woodgate R. et al. UmuD’2C is an error-prone DNA polymerase // PNAS. 1999. V. 96. P. 8919–8924. https://doi.org/10.1073/pnas.96.16.8919
Reuven N.B., Arad G., Maor-Shoshani A., Livneh Z. The mutagenesis protein UmuC is a DNA polymerase activated by UmuD’, RecA, and SSB, and is specialized for translesion replication // J. Biol. Chem. 1999. V. 274. P. 31763–31766. https://doi.org/10.1074/jbc.274.45.31763
Defais M., Lesca C., Monsarrat B., Hanavalt P. Translesion synthesis is the main component of SOS repair in bacteriophage lambda DNA // J. Bacteriol. 1989. V. 171. P. 4938–4944. https://doi.org/10.1128/jb.171.9.4938-4944.1989
Jiang Q., Karata K., Woodgate R., Cox M.M. et al. The active form of DNA polymerase V is UmuD’2C-RecA*-ATP // Nature. 2009. V. 460. P. 359–363. https://doi.org/10.1038/nature08178
Patel M., Jiang Q., Woodgate R., Cox M.M. et al. A new model for SOS-induced mutagenesis: How RecA protein activates DNA polymerase V // Crit. Rev. Biochem. Mol. Biol. 2010. V. 45. P. 171–184. https://doi.org/10.3109/10409238.2010.480968
Завильгельский Г.Б., Белогуров А.А., Крюгер Д.Н. W‑реактивация и W-мутагенез у бактериофагов λ и Т7: сравнительный анализ действия ультрафиолетового излучения (254 нм) и фотосенсибилизаторов 8-метоксипсоралена и ангелицина // Генетика. 1982. Т. 18. № 1. С. 24–34.
Krivisky A.S., Esipova V.V., Zuev A.V. Induction of mutations and repair in bacteriophages after photosensitizing action of 8-methoxypsoralen // Biologisches Zentralblatt. 1979. V. 98. P. 175–183.
Ashwood M.J., Grant F. Conversion of psoralen DNA monoadducts in Escherichia coli to interstrand DNA cross-links by near UV light (320–360 nm): Inability of angelicin to form crosslinks in vivo // Experientia. 1977. V. 33. P. 384–386. https://doi.org/10.1007/BF02002841
Sambrook J., Fritsch E.P., Maniatis T. Molecular Cloning: A Laboratory Manual. 2nd ed. Cold Spring Harbor, New York: Cold Spring Harbor Lab. Press., 1989. 1626 pp.
Завильгельский Г.Б., Котова В.Ю. SOS-репарация моноаддуктов 8-метоксипсоралена в ДНК бактериофага λ и плазмид осуществляется полимеразой MucA’2B, но не UmuD’2C (PolV) // Генетика. 2013. Т. 49. № 12. С. 1370–1375. https://doi.org/10.7868/S001667581312014X
Pacelli L., Edmiston S., Mount D. Isolation and characterization of amber mutation in the lexA gene of Escherichia coli K12 // J. Bacteriology. 1979. V. 137. P. 568–573. https://doi.org/10.1128/JB.137.1.568-573.1979.573
George J., Buttin G. Prophage induction and cell division in Escherichia coli. I. Further characterization of the thermosensitive mutation tif-1 whose expression mimics the effectof the UV-irradiation // Mol. Gen. Genetics. 1972. V. 119. P. 139–152. https://doi.org/10.1007/BF00269133
Dobson P.P., Walker G.C. Plasmid pKM101 mediated W-reactivation in Escherichia coli K12 and Salmonella typhimurium LT: Genetic dependence, kinetics of induction and effect of chloramphenicol // Mutation Res. 1980. V. 71. P. 25–41. https://doi.org/10.1016/0027-5107(80)90004-4
Hauser J., Levine A.S., Ennis D.G., Chumakov K.M. et al. The enhanced mutagenic potential of the MucAB proteins correlates with the highly efficient processing of the MucA protein // J. Bacteriol. 1992. V. 174. P. 6844–6851. https://doi.org/10.1128/jb.174.21.6844-6851.1992
Elledge S.J., Walker G.C. The muc genes of pKM101 are induced by DNA damage // J. Bacteriol. 1983. V. 155. P. 1306–1315. https://doi.org/10.1128/JB.155.3.1306-1315.1983
Belogurov A.A., Zavilgelsky G.B. Mutagenic effect of furocoumarin monoadducts and cross-links on bacteriophage lambda // Mutation Res. 1981. V. 84. P. 11–15. https://doi.org/10.1016/0027-5107(81)90045-2
Janel-Bintz R., Maenhaut-Michel G., Fuchs R.P. MucAB but not UmuDC proteins enhance (-2) frame shift mutagenesis induced by N-2-acetylaminofluorene at alternating GC sequences // Mol. Gen. Genet. 1994. V. 245. P. 279–285. https: //doi.org/:10.1007/BF00290107
Дополнительные материалы отсутствуют.


