Генетика, 2021, T. 57, № 7, стр. 842-849
Влияние дифференциальной экспрессии гена ADAMTS1 на радиационно-индуцированный ответ клеточной линии HеLа
Р. Р. Савченко 1, *, А. А. Мурашкина 2, В. С. Фишман 3, Е. С. Сухих 4, 5, А. В. Вертинский 4, 5, Л. Г. Сухих 5, О. Л. Серов 3, И. Н. Лебедев 1, С. А. Васильев 1
1 Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
2 Национальный исследовательский Томский государственный университет
634050 Томск, Россия
3 Федеральный исследовательский центр Институт цитологии и генетики Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
4 Томский областной онкологический диспансер
634063 Томск, Россия
5 Национальный исследовательский Томский политехнический университет
634050 Томск, Россия
* E-mail: renata.savchenko@medgenetics.ru
Поступила в редакцию 02.09.2020
После доработки 20.01.2021
Принята к публикации 18.02.2021
Аннотация
Проведен анализ влияния дифференциальной экспрессии гена ADAMTS1 на радиационно-индуцированный ответ в клеточных линиях HeLa с нокаутом и сверхэкспрессией гена ADAMTS1. Клеточная линия с нокаутом гена была получена с помощью системы редактирования генома CRISPR/Cas9. Сверхэкспрессия гена ADAMTS1 обеспечивалась с помощью временной трансфекции плазмиды, содержащей кодирующую последовательность изучаемого гена. После воздействия на клеточные линии γ-излучением в диапазоне доз 2–8 Гр оценена клональная выживаемость, уровень микроядер и фокусов белков репарации ДНК γH2AX и 53BP1. Показано, что клеточная линия HeLa с нокаутом гена ADAMTS1 характеризуется снижением клональной выживаемости в 1.9 раз (p < 0.05) после облучения в дозе 2 Гр и повышенной частотой микроядер (55.3 ± 8.3‰) по сравнению с исходной клеточной линией HeLa (36.0 ± 4.2‰, p < 0.05), но не отличается по уровню фокусов белков репарации ДНК. Трансфекция клеточной линии HeLa с нокаутом ADAMTS1 плазмидой, несущей кодирующую последовательность изучаемого гена, приводит к снижению частоты радиационно-индуцированных микроядер с 55.3 ± 8.3 до 28.7 ± 10.3‰ (p < 0.05), что сопоставимо с частотой микроядер в исходной клеточной линии HeLa после облучения (36.0 ± 7.2‰). Полученные данные свидетельствуют об участии ADAMTS1 в формировании радиационно-индуцированного ответа в данном типе клеток.
Воздействие ионизирующего излучения, применяемое в разные периоды противоопухолевой терапии для более чем половины пациентов с онкологическими заболеваниями, является эффективным методом лечения, однако влечет за собой развитие ряда побочных реакций в здоровых тканях организма [1–3]. Спектр данных реакций велик и при одинаковой схеме лечения может варьировать от легких и умеренных до жизнеугрожающих и летальных [4, 5]. Известно, что данные различия обусловлены индивидуальной радиочувствительностью пациентов, в связи с чем, одним из актуальных направлений радиационной онкологии является поиск генетических маркеров, позволяющих предсказать вероятность развития осложнений в нормальных тканях [5, 6]. Выявленный на сегодняшний день полиморфизм некоторых кандидатных генов объясняет лишь небольшую часть популяционной вариабельности индивидуальной радиочувствительности, как правило, характерной для определенных видов рака или наследственных синдромов [7–10]. При этом предполагают, что оставшийся генетический вклад в формирование индивидуальной радиочувствительности обеспечивается вариациями в нуклеотидной последовательности множества других генов, распространенных в популяции, но имеющих менее выраженный эффект [5]. Интересно, что наряду с генами, продукты которых связаны с репарацией двунитевых разрывов ДНК, контролем клеточного цикла и метаболизмом ксенобиотиков, с радиочувствительностью связывают и гены, не имеющие прямого отношения к данным процессам и преимущественно обеспечивающие сигнальные функции [11]. Это обстоятельство делает актуальными полногеномные исследования для выявления белков-участников радиационно-индуцированного клеточного ответа [11, 12]. Так, проведенный нами ранее полнотранскриптомный анализ профиля экспрессии генов в лимфоцитах периферической крови здоровых людей показал, что индивиды с различным уровнем фокусов белков репарации ДНК γH2AX и частотой радиационно-индуцированных микроядер характеризуются дифференциальной экспрессией гена ADAMTS1 [13]. Данный ген кодирует белок из семейства ADAMTS и содержит несколько функциональных модулей, включая тромбоспондиновый (TS) мотив 1-го типа. Согласно данным литературы, экспрессию данного гена связывают с воспалительными процессами [14, 15], гипоксией [16–18], стимуляцией ангиогенеза [19] и ремоделированием тканей во время развития и прогрессирования рака [20–24], однако роль ADAMTS1 в онкогенезе неоднозначна, и его повышенная экспрессия ассоциирована как с про-, так и с противоопухолевыми эффектами [20, 21, 25]. Насколько известно, ген ADAMTS1 не связывают с механизмами формирования индивидуальной радиочувствительности, однако согласно полученным нами ранее данным, клетки здоровых индивидов с низким уровнем микроядер в лимфоцитах периферической крови (как до, так и после облучения in vitro) характеризуются повышенной экспрессией ADAMTS1 [13]. В связи с этим, мы предполагаем, что ADAMTS1 может быть вовлечен в процесс формирования клеточного ответа на воздействие ионизирующего излучения. Поэтому целью настоящего исследования явился анализ влияния дифференциальной экспрессии гена ADAMTS1 на формирование радиационно-индуцированного ответа в культуре клеток in vitro.
МАТЕРИАЛЫ И МЕТОДЫ
Материалом для исследования послужила радиочувствительная клеточная линия карциномы шейки матки HeLa. Интактную клеточную линию HeLa, а также полученные на ее основе модельные клеточные линии с нокаутом и сверхэкспрессией гена ADAMTS1 культивировали в среде DMEM (ПанЭко, Россия) с добавлением 10%-ной телячьей эмбриональной сыворотки (FBS; Thermo, США) и эритромицина (ПанЭко, Россия) в атмосфере с 5% CO2 при 37°C.
Создание клеточной линии с нокаутом гена ADAMTS1
Клеточная линия HeLa с нокаутом гена ADAMTS1 была получена путем внесения мутации сдвига рамки считывания с использованием системы редактирования генома CRISPR/Cas9. Химерные направляющие РНК на экзон 2 гена ADAMTS1 были получены с помощью инструмента sgRNAScorer (https://crispr.med.harvard.edu/sgRNAScorer/). Олигонуклеотиды, соответствующие смысловой и антисмысловой цепям химерных направляющих РНК (табл. 1), клонировались в плазмидный вектор с помощью рестриктазы BsmBI (NEB, Великобритания).
Таблица 1.
Олигонуклеотиды для создания химерных направляющих РНК для гена ADAMTS1 и анализа введенных мутаций с помощью секвенирования
| Олигонуклеотид | Последовательность |
|---|---|
| ADAMTS1_sgRNA1_F | 5'-CACCGGCTTGTGGCAGACCAGTCGAGTT-3' |
| ADAMTS1_sgRNA1_R | 5'-CTAAAACTCGACTGGTCTGCCACAAGCC-3' |
| ADAMTS1_sgRNA2_F | 5'-CACCGGGTTTCCACATAGCGGTGACGTT-3' |
| ADAMTS1_sgRNA2_R | 5'-CTAAAACGTCACCGCTATGTGGAAACCC-3' |
| ADAMTS1_F | 5'-GCCACCGAAAACAACGTGAG-3' |
| ADAMTS1_R | 5'-ACTGGAAGCATAAGAAAGAAGCG-3' |
Трансфекцию двух плазмид с направляющими РНК и плазмиды с генами Cas9 и GFP (Addgene plasmid, 48138) осуществляли с использованием Липофектамина 3000 (Thermo, США) в соответствии с рекомендациями производителя. Через 24 ч после трансфекции GFP-позитивные клетки отбирали на клеточном сортере FACS ARIA (BD, США) и культивировали в течение двух недель для образования колоний. Анализ присутствия мутаций в сайте редактирования осуществляли с помощью таргетного массового параллельного секвенирования. Амплификацию участков экзона 2 гена ADAMTS1 проводили с помощью ПЦР с использованием разработанных олигонуклеотидных праймеров (табл. 1). Приготовление библиотек проводили с помощью набора Nextera XT (Illumina, США) в соответствии с протоколом производителя. Секвенирование осуществляли на приборе MiSeq (Illumina, США) с использованием набора MiSeq Reagent Nano Kit v2 kit (Illumina, США) в соответствии с рекомендациями производителя.
Создание клеточной линии со сверхэкспрессией гена ADAMTS1
Создание клеточной линии HeLa со сверхэкспрессией гена ADAMTS1 осуществляли с помощью временной трансфекции плазмиды ADAMTS1_ pCSdest, содержащей кодирующую последовательность изучаемого гена, любезно предоставленной Roger Reeves (Addgene plasmid, 53807) [26]. Трансфекцию осуществляли с помощью Липофектамина 3000 (Thermo, США) по протоколу производителя.
Облучение клеточных линий
Клеточные линии подвергали воздействию ионизирующего γ-излучения 60Co на гамма-терапевтической установке “Theratron Equinox 100” (Best Theratronics, Канада) в дозе 2–8 Гр (для оценки клональной выживаемости на фоне нокаута ADAMTS1) и в дозе 2 Гр (для анализа фокусов белков репарации ДНК на фоне нокаута ADAMTS1 и проведения микроядерного теста в обеих модельных клеточных линиях). Облучение проводили на базе Томского областного онкологического диспансера. Транспортировку клеток осуществляли на льду.
Анализ экспрессии ADAMTS1
Для анализа экспрессии гена ADAMTS1 РНК из клеточных линий выделяли с использованием реагента “Лира” для выделения РНК, ДНК и белков (Биолабмикс, Россия) по протоколу производителя. Далее полученная РНК была конвертирована в кДНК с использованием набора для обратной транскрипции ОТ-M-MuLV-RH (Биолабмикс, Россия), после чего проводили ПЦР в реальном времени с помощью набора БиоМастер HS-qPCR (2×) (Биолабмикс, Россия) на амплификаторе AriaMx (Agilent Technologies, США). Для анализа экспрессии изучаемого гена после трансфекции протокол был дополнен этапом обработки РНК с использованием фермента ДНКазы и 10× буфера MgCl2 (Thermo, США) для исключения плазмидной контаминации образцов РНК. Анализ экспрессии осуществляли с использованием разработанных олигонуклеотидных праймеров (табл. 2). В качестве референсного гена для оценки экспрессии использовали ген домашнего хозяйства ACTB (табл. 2). Уровень экспрессии каждого целевого гена оценивали относительно уровня экспрессии референсного гена, рассчитывая показатель ΔΔСt [27].
Клональная выживаемость
После облучения исходной клеточной линии HeLa и клеточной линии с нокаутом ADAMTS1 определенное число клеток высевали в 6-луночные планшеты (TPP, Швейцария). Для доз 0, 2, 4, 6 и 8 Гр производился посев 1200, 3000, 7500, 30 000 и 60 000 клеток соответственно. Через 14 дней была проведена фиксация клеток с помощью инкубации в 1 мл раствора метанол-уксусная кислота (3 : 1) в течение 5 мин и окрашивание клеток с использованием красителя Гимза (ПанЭко, Россия) в течение 10 мин. Далее была проведена оценка доли выживших клеток, сформировавших колонии.
Микроядерный тест
Через 24 ч после облучения клетки фиксировали в течение 20 мин при –20°C в растворе метанол-уксусная кислота (3 : 1). После фиксации клетки наносили на предметные стекла и окрашивали красителем DAPI. Частоту микроядер оценивали в 1000 клеток на препарате с помощью микроскопа Axio Imager Z2 (Carl Zeiss, Германия) с фильтром для DAPI.
Иммуноокрашивание фокусов белков репарации ДНК
Для проведения иммуноокрашивания фокусов белков репарации двунитевых разрывов ДНК клетки HeLa выращивали на покровных стеклах (20 × 20 мм), после чего подвергали воздействию γ-излучения. Через 30 мин, 2, 4, 6, 8 и 24 ч после облучения клетки фиксировали в течение 20 мин при 4°С в растворе 3%-ного параформальдегида (Sigma, США). После фиксации покровные стекла с клетками инкубировали в 0.2%-ном Triton X-100 (Sigma, США) в течение 10 мин при 4°С, отмывали в трех сменах фосфатного буфера (PBS) по 5 мин и инкубировали в 3%-ном FBS в течение 30 мин при 4°С. Затем на покровные стекла наносили 100 мкл раствора первичных антител: моноклональных антител мыши к белку γH2AX (Novus Biologicals, США) и поликлональных антител кролика к белку RAD51 (Novus Biologicals, США), разведенных в 3%-ном FBS (1 : 400). Инкубацию с первичными антителами проводили в течение 1 ч при комнатной температуре, после чего покровные стекла отмывали в трех сменах PBS по 5 мин при комнатной температуре. Затем проводили инкубацию в течение 1 ч при комнатной температуре с раствором вторичных антител: антитела козы к антителам мыши, конъюгированные с родамином, и антитела козы к антителам кролика, конъюгированные с FITC (Novus Biologicals, США), разведенные 1 : 400 в 3%-ном FBS. После трехкратной отмывки в PBS по 5 мин клетки окрашивали DAPI и заключали в среду для микроскопирования Vectashield (Vector Labs, США) на предметных стеклах.
Микрофотографии получали на микроскопе Axio Imager Z2 (Carl Zeiss, Германия) с автоматичес-кой системой анализа препаратов Metafer (Meta-Systems, Германия) с использованием фильтров для DAPI, родамина и FITC. Изображения получали с 15 фокальных плоскостей с шагом 0.5 мкм с последующим слиянием в одно результирующее изображение. Число фокусов белков репарации двунитевых разрывов ДНК анализировали в 150–200 клетках на каждом препарате. Распределение числа фокусов в клетках аппроксимировали с помощью распределения Пуассона, как описано в [28], и в дальнейшем анализе вместо среднего числа фокусов использовали λ из распределения Пуассона.
Статистический анализ
Статистический анализ проводили с использованием программного пакета STATISTICA 8.0 (StatSoft, США). Для сравнений клеточных линий использовали t-критерий Стьюдента. Различия считали статистически значимыми при p < 0.05. Числовые данные представлены в виде среднего ± ± стандартное отклонение.
Исследование выполнено с использованием оборудования Центра коллективного пользования “Медицинская геномика” Томского НИМЦ.
РЕЗУЛЬТАТЫ
Полученная клеточная линия с нокаутом ADAMTS1 была компаундной гетерозиготой по двум делециям, которые приводили к сдвигу рамки считывания (рис. 1,а). Анализ потенциальной функциональной значимости введенных мутаций с помощью инструмента Mutation Taster [29] показал, что введенные делеции приводят к значительному укорочению белкового продукта гена ADAMTS1 в результате преждевременного появления стоп-кодона. Помимо потенциального воздействия на структуру белка, введенные делеции приводили к снижению (в 1.7 раз) экспрессии гена ADAMTS1 на уровне мРНК (рис. 1,б).
Рис. 1.
Эффекты дифференциальной экспрессии гена ADAMTS1 в клеточной линии HeLa. ADAMTS1 ko – клеточная линия HeLa с нокаутом ADAMTS1, HeLa-TF – клеточная линия HeLa после трансфекции плазмиды, кодирующей ADAMTS1, ADAMTS1 ko-TF – клеточная линия HeLa с нокаутом гена ADAMTS1 после трансфекции плазмиды, кодирующей ADAMTS1. а – делеции в экзоне 2 гена ADAMTS1, введенные с помощью системы CRISPR/Cas9, wt – референсная последовательность; полужирным начертанием выделены последовательности химерных направляющих РНК, подчеркнуты участки мотива, смежного с протоспейсером (protospacer adjacent motif, PAM); б – экспрессия гена ADAMTS1 в клеточных линиях на фоне нокаута и после трансфекции; в – клональная выживаемость после воздействия γ-излучения в дозах 2–8 Гр в клеточной линии с нокаутом гена ADAMTS1; г – частота микроядер на фоне дифференциальной экспрессии гена ADAMTS1 после воздействия γ-излучения в дозах 2 Гр; д, е – динамика уровней фокусов белков γH2AX (д) и RAD51 (е) в клеточной линии с нокаутом гена ADAMTS1 после воздействия γ-излучения в дозах 2 Гр, данные представлены в виде среднего ± стандартное отклонение (n = 4), * p < 0.05.
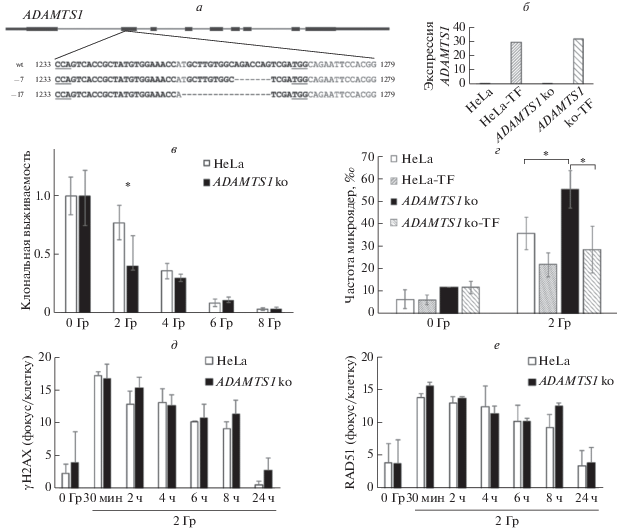
Клеточная линия с нокаутом ADAMTS1 характеризовалась снижением способности к формированию колоний в 1.9 раз (p < 0.05) после воздействия γ-излучением в дозе 2 Гр по сравнению с исходной клеточной линией HeLa (рис. 1,в). Кроме того, нокаут гена ADAMTS1 приводил к повышению частоты микроядер относительно исходной клеточной линии HeLa (55.3 ± 8.3 и 36.0 ± 4.2‰ соответственно, p < 0.05) после облучения (рис. 2 ,г). Однако уровни фокусов белков репарации двунитевых разрывов ДНК γH2AX и RAD51 после нокаута гена ADAMTS1 статистически значимо не повышались (рис. 2 ,д, е).
Анализ экспрессии гена ADAMTS1 в клеточной линии HeLa после трансфекции показал, что экспрессия данного гена возрастала в 30 раз на фоне трансфекции исходной клеточной линии HeLa и в 32 раза на фоне трансфекции клеточной линии HeLa с нокаутом ADAMTS1 (рис. 1,б).
Частота микроядер в клеточных линиях после трансфекции не отличалась от таковой в контрольных клеточных линиях до облучения (рис. 1,г), однако статистически значимые различия были выявлены после облучения в дозе 2 Гр. Примечательно, что после трансфекции клеточной линии HeLa с нокаутом ADAMTS1 частота микроядер снижалась с 55.3 ± 8.3 до 28.7 ± 10.3‰ (p < 0.05) и не отличалась статистически значимо от исходной клеточной линии HeLa (36.0 ± 7.2‰) (рис. 1,г).
ОБСУЖДЕНИЕ
Статистически значимое снижение клональной выживаемости в клеточной линии с нокаутом ADAMTS1 относительно исходной клеточной линии HeLa после облучения в дозе 2 Гр свидетельствует о том, что белок, кодируемый данным геном, по-видимому участвует в формировании радиационно-индуцированного клеточного ответа, однако его роль в данном процессе остается неясной. Возможно данный белок вовлечен в процессы контроля клеточного цикла или механизмы клеточной гибели, но не связан с процессами репарации ДНК, поскольку в клеточной линии с нокаутом ADAMTS1 наблюдается повышение частоты радиационно-индуцированных микроядер, но не фокусов белков репарации ДНК. Связь гена ADAMTS1 с механизмами контроля клеточного цикла была отмечена в исследовании M. Li с соавт. [30]. Было показано, что вызванное активацией микроРНК miR-362-3p снижение экспрессии ADAMTS1 приводит к аресту клеточного цикла на стадии G1/S и ингибированию пролиферации в культуре гладкомышечных клеток сосудов человека [30].
Согласно данным литературы, металлопротеиназы, участвующие в ремоделировании внеклеточного матрикса, играют ключевую роль в регуляции биодоступности цитокинов и факторов роста [31, 32]. В частности было показано, что металлопротеиназа ADAMTS1 может активировать трансформирующий ростовой фактор β (TGFβ) [33, 34]. В то же время известно, что экспрессия гена ADAMTS1 повышается после ингибирования TGFβ [35], что свидетельствует о возможной регуляции ADAMTS1-TGFβ по механизму отрицательной обратной связи. Это особенно важно, учитывая, что TGFβ является важным компонентом ответа клетки на повреждение ДНК [36]. Известно, что нокаут TGFβ в клетках мыши или ингибирование сигнального пути TGFβ в клетках человека приводит к нарушению активности и автофосфорилирования белка ATM, что влечет за собой снижение уровня фосфорилирования его ключевых мишеней, включая белок p53, нарушение активации контрольных точек клеточного цикла и, как следствие, повышенную радиочувствительность клеток [37–39].
Использование системы редактирования генома CRISPR/Cas9 может приводить к внесению нецелевых мутаций в геном объекта исследования, что затрудняет интерпретацию результатов экспериментов [40–44]. В данном исследовании не проводилось полногеномное секвенирование полученной клеточной линии HeLa с нокаутом ADAMTS1 с целью поиска нежелательных мутаций, внесенных системой CRISPR/Cas9, однако полученная клеточная линия была трансфецирована плазмидой, кодирующей ген ADAMTS1. Нормализация фенотипа (снижение частоты микроядер до уровня исходной клеточной линии HeLa) в нокаутной клеточной линии после трансфекции свидетельствует в пользу того, что наблюдаемые эффекты обусловлены непосредственно дифференциальной экспрессией гена ADAMTS1, а не потенциально возможными не выявленными мутациями.
Таким образом, полученные данные свидетельствуют об участии ADAMTS1 в формировании радиационно-индуцированного ответа в клеточной линии HeLa, причем повышенная экспрессия данного гена связана с уменьшением неблагоприятных радиационно-индуцированных эффектов.
В свете активного развития радиогеномики и поиска генов, ассоциированных с формированием радиочувствительности нормальных и опухолевых клеток, представляется целесообразным дальнейшее изучение гена ADAMTS1 в качестве потенциального биомаркера индивидуальной радиочувствительности.
Исследование выполнено при финансовой поддержке РФФИ в рамках научных проектов № 19-34-90143 и 16-34-50178.
Настоящая статья не содержит каких-либо исследований с использованием в качестве объекта животных.
Настоящая статья не содержит каких-либо исследований с участием в качестве объекта людей.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Foray N., Bourguignon M., Hamada N. Individual response to ionizing radiation // Mutat Res. 2016. V. 770. P. 369–386. https://doi.org/10.1016/j.mrrev.2016.09.001
Rajaraman P., Hauptmann M., Bouffler S. et al. Human individual radiation sensitivity and prospects for prediction //Annals ICRP. 2018. V. 47. № 3–4. P. 126–141. https://doi.org/10.1177/0146645318764091
Seibold P., Auvinen A., Averbeck D. et al. Clinical and epidemiological observations on individual radiation sensitivity and susceptibility // Int. J. Radiation Biol. 2020. V. 96. № 3. P. 324–339. https://doi.org/10.1080/09553002.2019.1665209
Ferlazzo M.L., Bourguignon M., Foray N. Functional assays for individual radiosensitivity: a critical review // Seminars in Radiation Oncology. WB Saunders. 2017. V. 27. № 4. P. 310–315. https://doi.org/10.1016/j.semradonc.2017.04.003
Gomolka M., Blyth B., Bourguignon M. et al. Potential screening assays for individual radiation sensitivity and susceptibility and their current validation state // Int. J. Radiation Biol. 2020. V. 96. № 3. P. 280–296. https://doi.org/10.1080/09553002.2019.1642544
Kirsch D.G., Diehn M., Kesarwala A.H. et al. The future of radiobiology // J. Natl. Cancer Inst. 2018. V. 110. № 4. P. 329–340. https://doi.org/10.1093/jnci/djx231
Barnett G.C., Thompson D., Fachal L. et al. A genome wide association study (GWAS) providing evidence of an association between common genetic variants and late radiotherapy toxicity // Radiotherapy and Oncology. 2014. V. 111. № 2. P. 178–185. https://doi.org/10.1016/j.radonc.2014.02.012
Fachal L., Gomez-Caamano A., Barnett G.C. et al. A three-stage genome-wide association study identifies a susceptibility locus for late radiotherapy toxicity at 2q24. 1 // Nat. Genetics. 2014. V. 46. № 8. P. 891–894.https://doi.org/10.1038/ng.3020
Talbot C.J., Tanteles G.A., Barnett G.C. et al. A replicated association between polymorphisms near TNFα and risk for adverse reactions to radiotherapy // Br. J. Cancer. 2012. V. 107. № 4. P. 748–753. https://doi.org/10.1038/bjc.2012.290
Seibold P., Behrens S., Schmezer P. et al. XRCC1 polymorphism associated with late toxicity after radiation therapy in breast cancer patients // Int. J. Radiat. Oncol. Biol. Phys. 2015. V. 92. № 5. P. 1084–1092. https://doi.org/10.1016/j.ijrobp.2015.04.011
Andreassen C.N., Schack L.M., Laursen L.V. et al. Radiogenomics–current status, challenges and future directions // Cancer Letters. 2016. V. 382. № 1. P. 127–136. https://doi.org/10.1016/j.canlet.2016.01.035
Guo Z., Shu Y., Zhou H. et al. Radiogenomics helps to achieve personalized therapy by evaluating patient responses to radiation treatment // Carcinogenesis. 2015. V. 36. № 3. P. 307–317. https://doi.org/10.1093/carcin/bgv007
Васильев С.А. Особенности спонтанного ииндуцированного мутагенеза в соматических клетках человека с различным эпигенетическим фоном: Дис. … д-ра биол. наук. Томск: ФГБНУ “Томский национальный исследовательский медицинский центр Российской академии наук”, 2018. 332 с.
Rodríguez-Baena F.J., Redondo-García S., Peris-Torres C. et al. ADAMTS1 protease is required for a balanced immune cell repertoire and tumour inflammatory response // Sci. Reports. 2018. V. 8. № 1. P. 1–11. https://doi.org/10.1038/s41598-018-31288-7
Karakose M., Demircan K., Tutal E. et al. Clinical significance of ADAMTS1, ADAMTS5, ADAMTS9 aggrecanases and IL-17A, IL-23, IL-33 cytokines in polycystic ovary syndrome // J. Endocrinol. Invest. 2016. V. 39. № 11. P. 1269–1275. https://doi.org/10.1007/s40618-016-0472-2
Hatipoglu O.F., Hirohata S., Cilek M.Z. et al. ADAMTS1 is a unique hypoxic early response gene expressed by endothelial cells // J. Biol. Chem. 2009. V. 284. № 24. P. 16325–16333. https://doi.org/10.1074/jbc.M109.001313
Tayman M.A., Kurgan S., Onder C. et al. A disintegrin-like and metalloproteinase with thrombospondin-1 (ADAMTS-1) levels in gingival crevicular fluid correlate with vascular endothelial growth factor-A, hypoxia-inducible factor-1α, and clinical parameters in patients with advanced periodontitis // J. Periodontol. 2019. V. 90. № 10. P. 1182–1189. https://doi.org/10.1002/JPER.18-0195
Armutcu F., Demircan K., Yildirim U. et al. Hypoxia causes important changes of extracellular matrix biomarkers and ADAMTS proteinases in the adriamycin-induced renal fibrosis model // Nephrology. 2019. V. 24. № 8. P. 863–875. https://doi.org/10.1111/nep.13572
Kelwick R., Desanlis I., Wheeler G.N. et al. The ADAMTS (A Disintegrin and Metalloproteinase with Thrombospondin motifs) family // Genome Biology. 2015. V. 16. № 1. P. 113. https://doi.org/10.1186/s13059-015-0676-3
Wen Y.C., Lin Y.W., Chu C.Y. et al. Melatonin triggered post-transcriptional and post-translational modifications of ADAMTS1 coordinately retard tumorigenesis and metastasis of renal cell carcinoma // J. Pineal Res. 2020. V. 69. № 2. P. e12668. https://doi.org/10.1111/jpi.12668
Ham S.A., Yoo T., Lee W.J. et al. ADAMTS1-mediated targeting of TSP-1 by PPARδ suppresses migration and invasion of breast cancer cells // Oncotarget. 2017. V. 8. № 55. P. 94091. https://doi.org/10.18632/oncotarget.21584
Cal S., López-Otín C. ADAMTS proteases and cancer // Matrix Biology. 2015. V. 44. P. 77–85. https://doi.org/10.1016/j.matbio.2015.01.013
Rocks N., Paulissen G., El Hour M. et al. Emerging roles of ADAM and ADAMTS metalloproteinases in cancer // Biochimie. 2008. V. 90. № 2. P. 369–379. https://doi.org/10.1016/j.biochi.2007.08.008
Lee N.V., Sato M., Annis D.S. et al. ADAMTS1 mediates the release of antiangiogenic polypeptides from TSP1 and 2 // The EMBO J. 2006. V. 25. № 22. P. 5270–5283. https://doi.org/10.1038/sj.emboj.7601400
Brancaccio M., Natale F., Falco T. et al. Cell-Free DNA Methylation: The new frontiers of pancreatic cancer biomarkers’ Discovery // Genes. 2020. V. 11. № 1. P. 14. https://doi.org/10.3390/genes11010014
Edie S., Zaghloul N.A., Leitch C.C. et al. Survey of human chromosome 21 gene expression effects on early development in Danio rerio // G3: Genes, Genomes, Genetics. 2018. V. 8. № 7. P. 2215–2223. https://doi.org/10.1534/g3.118.200144
Livak K.J., Schmittgen T.D. Analysis of relative gene expression data using real-time quantitative PCR and the 2− ΔΔCT method // Methods. 2001. V. 25. № 4. P. 402–408. https://doi.org/10.1006/meth.2001.1262
Martin O.A., Ivashkevich A., Choo S. et al. Statistical analysis of kinetics, distribution and co-localisation of DNA repair foci in irradiated cells: Cell cycle effect and implications for prediction of radiosensitivity // DNA Repair (Amst). 2013. V. 12. № 10. P. 844–855. https://doi.org/10.1016/j.dnarep.2013.07.002
Schwarz J.M., Cooper D.N., Schuelke M. et al. MutationTaster2: Mutation prediction for the deep-sequencing age // Nat. Methods. 2014. V. 11. № 4. P. 361–362. https://doi.org/10.1038/nmeth.2890
Li M., Liu Q., Lei J. et al. MiR-362-3p inhibits the proliferation and migration of vascular smooth muscle cells in atherosclerosis by targeting ADAMTS1 // Biochem. Biophys. Res. Commun. 2017. V. 493. № 1. P. 270–276. https://doi.org/10.1016/j.bbrc.2017.09.031
Apte S.S. A disintegrin-like and metalloprotease (reprolysin-type) with thrombospondin type 1 motif (ADAMTS) superfamily: Functions and mechanisms // J. Biol. Chem. 2009. V. 284. № 46. P. 31493–31497. https://doi.org/10.1074/jbc.R109.052340
Egeblad M., Werb Z. New functions for the matrix metalloproteinases in cancer progression // Nat. Rev. Cancer. 2002. V. 2. № 3. P. 161–174. https://doi.org/10.1038/nrc745
Bourd-Boittin K., Bonnier D., Leyme A. et al. Protease profiling of liver fibrosis reveals the ADAM metallopeptidase with thrombospondin type 1 motif, 1 as a central activator of transforming growth factor beta // Hepatology. 2011. V. 54. № 6. P. 2173–2184. https://doi.org/10.1002/hep.24598
Laurent M.A., Bonnier D., Théret N. et al. In silico characterization of the interaction between LSKL peptide, a LAP-TGF-beta derived peptide, and ADAMTS1 // Comput. Biol. Chem. 2016. V. 61. P. 155–161. https://doi.org/10.1016/j.compbiolchem.2016.01.012
Le Bras G.F., Taylor C., Koumangoye R.B. TGFβ loss activates ADAMTS-1-mediated EGF-dependent invasion in a model of esophageal cell invasion // Exp. Cell Res. 2015. V. 330. № 1. P. 29–42. https://doi.org/10.1016/j.yexcr.2014.07.021
Barcellos-Hoff M.H., Cucinotta F.A. New tricks for an old fox: impact of TGFβ on the DNA damage response and genomic stability // Sci. Signal. 2014. V. 7. № 341. P.:re5. https://doi.org/10.1126/scisignal.2005474
Ewan K.B., Henshall-Powell R.L., Ravani S.A. Transforming growth factor-beta1 mediates cellular response to DNA damage in situ // Cancer Res. 2002. V. 62. № 20. P. 5627–5631.
Kirshner J., Jobling M.F., Pajares M.J. et al. Inhibition of transforming growth factor-beta1 signaling attenuates ataxia telangiectasia mutated activity in response to genotoxic stress // Canser Res. 2006. V. 66. № 22. P. 10861–10869. https://doi.org/10.1158/0008-5472.CAN-06-2565
Wiegman E.M., Blaese M.A., Loeffler H. et al. TGFbeta-1 dependent fast stimulation of ATM and p53 phosphorylation following exposure to ionizing radiation does not involve TGFbeta-receptor I signalling // Radiother. Oncol. 2007. V. 83. № 3. P. 289–295. https://doi.org/10.1016/j.radonc.2007.05.013
Fu Y., Foden J.A., Khayter C. et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells // Nature Biotechnology. 2013. V. 31. № 9. P. 822–826. https://doi.org/10.1038/nbt.2623
Pattanayak V., Lin S., Guilinger J. et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity // Nature Biotechnology. 2013. V. 31. № 9. P. 839–843. https://doi.org/10.1038/nbt.2673
Lin Y., Cradick T.J., Brown M.T. et al. CRISPR/Cas9 systems have off-target activity with insertions or deletions between target DNA and guide RNA sequences // Nucl. Ac. Res. 2014. V. 42. № 11. P. 7473–7485. https://doi.org/10.1093/nar/gku402
Tsai S.Q., Zheng Z., Nguyen N.T. et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases // Nature Biotechnology. 2015. V. 33. № 2. P. 187–197. https://doi.org/10.1038/nbt.3117
Zischewski J., Fischer R., Bortesi L. Detection of on-target and off-target mutations generated by CRISPR/ Cas9 and other sequence-specific nucleases // Biotechnology Advances. 2017. V. 35. № 1. P. 95–104. https://doi.org/10.1016/j.biotechadv.2016.12.003
Дополнительные материалы отсутствуют.


