Генетика, 2021, T. 57, № 9, стр. 1062-1072
Генетическое разнообразие популяций Северной Евразии по маркерам, ассоциированным с заболеваниями, нарушающими когнитивные функции человека
А. В. Бочарова 1, *, В. А. Степанов 1
1 Научно-исследовательский институт медицинской генетики, Томский национально-исследовательский центр Российской академии наук
634050 Томск, Россия
* E-mail: anna.bocharova@medgenetics.ru
Поступила в редакцию 29.09.2020
После доработки 03.12.2020
Принята к публикации 11.02.2021
Аннотация
Впервые исследовано генетическое разнообразие 16 популяций коренного населения Северной Евразии по панели генетических маркеров генов, ассоциированных, согласно данным полногеномных ассоциативных исследований, с заболеваниями, которые ведут к нарушению когнитивных функций человека (шизофренией, болезнью Альцгеймера и их когнитивными эндофенотипами). Наблюдается снижение генетического разнообразия в географическом пространстве с запада на восток. Наибольший уровень генетического разнообразия показали европеоидные популяции. Выделяются две группы маркеров, которые преобладают у европеоидов или у монголоидов. Расположение 16 популяций в пространстве главных компонент отражает их географическую локализацию и позволяет выделить четыре кластера. В целом анализ внутрипопуляционного и межпопуляционного генетического разнообразия по 28 маркерам в 16 популяциях отражает закономерности структуры генофонда Северной Евразии, которые описаны по другим системам маркеров.
К когнитивным функциям (КФ) относятся интеллект, внимание, речь, память, разнообразные двигательные навыки, процессы восприятия информации [1]. КФ формируются в результате общей работы головного мозга в целом. При этом различные отделы его не являются равносильными. Повреждение какой-либо области в любом из отделов может привести к когнитивным нарушениям (КН). КН влияют на эффективность обучения, профессиональную, социальную и бытовую деятельность, являются одним из видов нервно-психических расстройств, включающих изменения в эмоциональной и поведенческой сфере. Все эти расстройства (эмоциональные, когнитивные и поведенческие) тесно взаимосвязаны. КН – состояния полиэтиологической природы, которые развиваются при различных психических, неврологических и соматических заболеваниях. Поскольку КФ связаны с общей деятельностью головного мозга в целом, КН закономерно развиваются при самых разных очаговых и диффузных поражениях головного мозга. Фактически любое повреждение головного мозга может вызвать КН разные по тяжести.
Во второй половине XX в. возрастной состав населения претерпел значительные изменения в сторону увеличения числа лиц пожилого возраста [2]. По данным статистики, от 5 до 25% и более (в зависимости от возраста) пожилых лиц имеют тяжелые когнитивные нарушения, в основном деменцию [3]. Частой причиной КН в молодом возрасте является шизофрения (ШЗ). Когнитивный дефицит является ключевой особенностью ШЗ. До 85% пациентов с ШЗ имеют ту или иную степень КН [4, 5].
Как ШЗ, так и болезнь Альцгеймера (БА) относятся к социально-значимым, широко распространенным многофакторным заболеваниям (МФЗ). Генетическая компонента подверженности к МФЗ, которые имеют полигенную природу, во многом зависит не только от возрастных и половых составляющих, но и от популяционных и этнических. Каждая популяция или этническая группа отличается генетическим паттерном частот аллелей в результате своего эволюционного происхождения. Частоты аллелей приобретают свою специфичность в разных популяциях в процессе этногенеза. Вероятно, это лежит в основе дифференциальной восприимчивости к распространенным МФЗ, таким как болезнь Альцгеймера и шизофрения. Характерные сочетания аллелей генов, формирующих предрасположенность к МФЗ, являются исключительными для каждой популяции. В связи с этим важно изучить вариабельность генов таких заболеваний у представителей разных этнических групп.
Цель настоящего исследования заключалась в характеристике генетического разнообразия полиморфных вариантов генов, ассоциированных, согласно данным полногеномных ассоциативных исследований (GWAS), с шизофренией, болезнью Альцгеймера и их когнитивными эндофенотипами (ЭФ), в популяциях Северной Евразии.
МАТЕРИАЛЫ И МЕТОДЫ
Популяционные выборки
В настоящей работе для выявления сходств и различий в частотах аллелей генов, ассоциированных с заболеваниями, нарушающими КФ, использованы популяционные выборки из биоколлекции НИИ медицинской генетики Томского НИМЦ РАН “Биобанк населения Северной Евразии”. Общая численность исследованных 16 популяционных выборок составила 1508 образцов, представляющих различные этно-территориальные регионы Северной Евразии: европейская часть России (русские, удмурты), Сибирь (хакасы, тувинцы, кеты, алтайцы, ханты, буряты, якуты), Средняя Азия (узбеки, казахи, киргизы северные, киргизы южные) и Дальний Восток (чукчи, коряки, нивхи).
В выборки включались неродственные неметисированные в трех поколениях индивиды, подписавшие информированное согласие на проведение исследования. Детальные характеристики выборок приведены в табл. 1. Исследование одобрено Комитетом по биомедицинской этике НИИ медицинской генетики Томского НИМЦ.
Таблица 1.
Характеристика обследуемых популяционных выборок
| № | Популяция (объем выборки) |
Локализация | Языковая принадлежность, семья/группа | Антропологический тип |
|---|---|---|---|---|
| Европеоидный расовый тип | ||||
| 1 | Русские (216) | Кемеровская обл., г. Кемерово |
Индоевропейская/славянская | Восточноевропейский |
| 2 | Удмурты (95) | Республика Удмуртия, п. Балезино | Уральско-юкагирская/ финно-угорская | Сублапоноидный |
| 3 | Узбеки (42) | Республика Узбекистан, г. Джалал-Абад |
Алтайская/тюркская | Памироиранский |
| Монголоидный расовый тип | ||||
| 4 | Алтайцы (95) | Республика Горный Алтай, с. Бешпельтир |
Алтайская/тюркская | Центральноазиатский |
| 5 | Буряты (93) | Республика Бурятия, пгт. Агинское |
Алтайская/монгольская | Центральноазиатский |
| 6 | Казахи (190) | Республика Казахстан, г. Алматы | Алтайская/тюркская | Южносибирский |
| 7 | Кеты (46) | Красноярский край, п. Келлог |
Енисейская | Енисейский |
| 8 | Киргизы северные (48) | Республика Кыргызстан, г. Бишкек | Алтайская/тюркская | Южносибирский |
| 9 | Киргизы южные (47) | Республика Кыргызстан, г. Ош |
Алтайская/тюркская | Южносибирский |
| 10 | Тувинцы (92) | Республика Тыва, г. Кызыл |
Алтайская/тюркская | Центральноазиатский |
| 11 | Хакасы (93) | Республика Хакасия, Аскизский р-н |
Алтайская/тюркская | Южносибирский |
| 12 | Ханты (94) | Ханты-мансийский автономный округ, с. Казым, д. Русскинская | Уральская/финно-угорская | Уральская |
| 13 | Якуты (93) | Республика Саха, с. Чериктей | Алтайская/тюркская | Центральноазиатский |
| 14 | Чукчи (95) | Чукотский автономный округ | Палеоазиатская | Арктический |
| 15 | Коряки (89) | Камчатский край | Чукотско-камчатская | Арктический |
| 16 | Нивхи (80) | Сахалинская обл., Охинский р-н | Палеоазиатская/нивхская | Монголоидный/ сахалиноамурский |
Выбор генетических маркеров и генотипирование
30 генетических маркеров для работы выбраны из Каталога опубликованных полногеномных ассоциативных исследований (GWAS), где эти маркеры показали статистически значимые ассоциации (уровень значимости p ≤ 5 × 10–7) с шизофренией, болезнью Альцгеймера и когнитивными эндофенотипами этих заболеваний [6–29]. ДНК выделяли методом фенол-хлороформной экстракции из цельной венозной крови [30]. Генотипирование проводили с помощью метода MALDI-TOF масс-спектрометрии как описано ранее [31]. Подробная характеристика генетических маркеров представлена в табл. 2. Подбор условий мультиплексного генотипирования для MALDI-TOF масс-спектрометрии был осуществлен с помощью разработанных подходов [32, 33].
Таблица 2.
Характеристика генетических маркеров
| № | Полное название гена | Обозначение гена | SNP | Аллели | ПА | РА | Признак | Ссылка |
|---|---|---|---|---|---|---|---|---|
| 1 | Contactin associated protein 2 | CNTNAP2 | rs10273775 | A/G | G | G | БА | [6] |
| 2 | Non-SMC condensin II complex subunit D3 | NCAPD3 | rs1031381 | C/T | C | N | Когнитивные тесты | [7] |
| 3 | Mitochondrial pyruvate carrier 2 | MPC2 | rs10489202 | G/T | G | A | ШЗ | [8] |
| 4 | Coiled-coil domain containing 60 | CCDC60 | rs11064768 | A/G | A | A | ШЗ | [9] |
| 5 | 5'-Nucleotidase, cytosolic II | NT5C2 | rs11191580 | C/T | T | T | ШЗ | [10] |
| 6 | LOC105378888/protein arginine methyltransferase 6 | LOC105378888/PRMT6 | rs12125971 | C/T | C | N | Интеллект | [11] |
| 7 | Long intergenic non-protein coding RNA 1741/SEC16 homolog B, endoplasmic reticulum export factor | LINC01741/SEC16B | rs12140439 | A/C | C | С | ШЗ | [12] |
| 8 | Transcription factor 4 | TCF4 | rs1261117 | C/T | C | T | ШЗ | [12] |
| 9 | Sorting nexin 29 | SNX29 | rs12922317 | A/G | A | G | ШЗ | [14] |
| 10 | LOC105373605 | LOC105373605 | rs12989701 | C/A | C | N | БА | [15] |
| 11 | Bromodomain containing 1 | BRD1 | rs138880 | A/C | C | C | ШЗ | [12] |
| 12 | Dachsous cadherin-related 2 | DCHS2 | rs1466662 | A/T | T | N | БА | [17] |
| 13 | Clusterin | CLU | rs1532278 | T/C | C | T | БА | [18] |
| 14 | NFKB activating protein-like | NKAPL | rs1635 | G/T | G | N | ШЗ | [19] |
| 15 | LSM1 homolog, mRNA degradation associated | LSM1 | rs16887244 | A/G | A | N | ШЗ | [8] |
| 16 | POM121 transmembrane nucleoporin-like 2 | POM121L2 | rs16897515 | C/A | C | C | ШЗ | [12] |
| 17 | Rho GTPase activating protein 31 | ARHGAP31 | rs17203055 | A/G | A | N | ШЗ | [12] |
| 18 | Transcription factor 4 | TCF4 | rs17512836 | T/C | T | C | ШЗ | [10] |
| 19 | Transcription factor 4 | TCF4 | rs17594526 | C/T | C | T | ШЗ | [21] |
| 20 | G protein-coupled receptor 89 pseudogene/tRNA-Val (anticodon AAC) 1–5 | GPR89P / TRV-AAC1-5 | rs17693963 | A/C | A | N | ШЗ | [22] |
| 21 | CD33 molecule | CD33 | rs3826656 | G/A | G | N | ШЗ | [25] |
| 22 | Acyl-CoA synthetase medium-chain family member 1 | ACSM1 | rs433598 | C/T | T | T | ШЗ | [26] |
| 23 | Apolipoprotein C1 | APOC1 | rs4420638 | A/G | G | A | БА | [17] |
| 24 | Cell adhesion associated, oncogene regulated | CDON | rs472926 | C/T | T | C | БА | [27] |
| 25 | Calcium voltage-gated channel subunit alpha1 C | CACNA1C | rs4765905 | C/G | G | N | ШЗ | [22] |
| 26 | Phosphatidylinositol binding clathrin assembly protein/ RNA, U6 small nuclear 560, pseudogene | PICALM/RNU6-560P | rs561655 | G/A | A | N | БА | [18] |
| 27 | Nectin cell adhesion molecule 2 | NECTIN2 | rs6859 | A/G | G | A | БА | [18] |
| 28 | LOC105375630 | LOC105375630 | rs7004633 | A/G | A | A | ШЗ | [22] |
| 29 | Reelin | RELN | rs7341475 | G/A | G | G | ШЗ | [28] |
| 30 | LOC105373605 | LOC105373605 | rs7561528 | G/A | A | A | БА | [18] |
Генотипирование было проведено на базе Центра коллективного пользования научно-исследовательским оборудованием и экспериментальным биологическим материалом “Медицинская геномика” НИИ медицинской генетики Томского НИМЦ.
Статистическая обработка результатов
Статистическая обработка полученных данных включала проверку распределения наблюдаемых частот генотипов на соответствие ожидаемым при равновесии Харди–Вайнберга (точный тест Фишера). Для характеристики внутри- и межпопуляционного генетического разнообразия маркеров, ассоциированных с ШЗ, БА и их когнитивными ЭФ, в популяциях Северной Евразии в качестве основной характеристики использовали ожидаемую гетерозиготность [34]. Оценку генетической дифференциации популяций проводили с помощью коэффициента фиксации Райта (Fst) методом анализа молекулярной дисперсии AMOVA [35]. Статистическая обработка полученных результатов проводилась с помощью пакетов программы “Arlequin 3.1”. Генетические взаимоотношения между популяциями анализировали с помощью метода главных компонент [36]. Анализ и построение графиков проводили с помощью пакета программ STATISTICA 7.0 (StatSoft Inc., США).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Частоты аллелей и генетическое разнообразие в популяциях коренного населения Северной Евразии
30 полиморфных вариантов генов, ассоциированных по данным GWAS с БА, ШЗ или когнитивными эндофенотипами этих заболеваний, были генотипированы с помощью MALDI-TOF масс-спектрометрии во всех 16 популяциях. Два из 30 маркеров (rs1261117 гена TCF4, rs7004633 гена LOC105375630) показали долю определяемых генотипов меньше 95% и были исключены из дальнейшего анализа для всех популяций.
Частоты предковых аллелей 28 маркеров в 16 популяциях Северной Евразии представлены в табл. 3 (данные по частотам генотипов доступны по запросу у авторов). Шесть маркеров, включенных в работу, оказались мономорфными в изученных популяциях. Так, rs17594526 гена TCF4 показал отсутствие полиморфизма в девяти популяциях: тувинцы, кеты, ханты, якуты, киргизы северные и южные, чукчи, коряки и нивхи. Другой генетический вариант этого же гена TCF4 (rs17512836) также оказался мономорфным в девяти популяциях: удмурты, хакасы, тувинцы, кеты, якуты, киргизы южные, чукчи, коряки и нивхи. Еще несколько локусов продемонстрировали отсутствие полиморфизма: rs17693963 межгенного региона GPR89P/TRV-AAC1-5 в популяциях хантов и нивхов, rs1212597 межгенного региона LOC105378888/ PRMT6 в популяциях чукчей и нивхов, rs11064768 гена CCDC60 в популяции нивхов, rs11191580 гена NT5C2 в популяции кетов (табл. 3). По опубликованным полногеномным ассоциативным исследованиям эти генетические маркеры были ассоциированы с развитием ШЗ и БА в европеоидных популяциях [9–11, 21, 22].
Таблица 3.
Частоты предковых аллелей (%) 28 SNP в 16 изученных популяциях Северной Евразии
| SNP (ген) | ПА | Европейская часть РФ | Сибирь | Средняя Азия | Дальний Восток | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| RUS | UDM | ALТ | BUR | HAK | TUV | KET | HAN | YAB | UZ | KAZ | KGN | KGS | CHU | KOR | NIV | ||
| rs10273775 (CNTNAP2) | G | 50 | 51 | 61 | 70 | 59 | 57 | 64 | 62 | 61 | 62 | 63 | 61 | 60 | 41 | 35 | 48 |
| rs1031381 (NCAPD3) | C | 63 | 44 | 44 | 52 | 60 | 51 | 46 | 55 | 66 | 54 | 53 | 56 | 50 | 28 | 51 | 22 |
| rs10489202 (MPC2) | G | 73 | 74 | 77 | 69 | 72 | 79 | 64 | 70 | 68 | 78 | 78 | 69 | 71 | 76 | 76 | 74 |
| rs11064768 (CCDC60) | A | 92 | 90 | 96 | 99 | 98 | 98 | 97 | 93 | 96 | 94 | 95 | 97 | 95 | 92 | 98 | 100 |
| rs11191580 (NT5C2) | T | 92 | 95 | 89 | 89 | 92 | 97 | 100 | 93 | 97 | 76 | 88 | 86 | 88 | 90 | 86 | 94 |
| rs12125971 (LOC105378889-PRMT6) | C | 93 | 93 | 95 | 96 | 96 | 99 | 96 | 99 | 94 | 94 | 93 | 94 | 95 | 100 | 96 | 100 |
| rs12140439 (LOC101928778-LOC105371627) | C | 68 | 79 | 74 | 74 | 87 | 80 | 85 | 79 | 70 | 74 | 80 | 80 | 82 | 86 | 94 | 94 |
| rs12922317 (SNX29) | A | 68 | 55 | 36 | 33 | 37 | 26 | 45 | 42 | 31 | 52 | 42 | 39 | 40 | 26 | 19 | 18 |
| rs12989701 (LOC105373605) | C | 87 | 87 | 98 | 96 | 91 | 96 | 97 | 95 | 96 | 93 | 94 | 96 | 95 | 95 | 98 | 95 |
| rs138880 (BRD1) | C | 19 | 17 | 21 | 18 | 19 | 16 | 18 | 15 | 15 | 21 | 19 | 11 | 19 | 21 | 24 | 16 |
| rs1466662 (DCHS2) | T | 61 | 56 | 30 | 28 | 39 | 31 | 27 | 12 | 32 | 44 | 41 | 41 | 46 | 37 | 38 | 30 |
| rs1532278 (CLU) | C | 61 | 59 | 65 | 68 | 61 | 55 | 63 | 55 | 70 | 72 | 64 | 72 | 67 | 55 | 55 | 50 |
| rs1635 (NKAPL) | G | 93 | 96 | 76 | 83 | 80 | 74 | 72 | 81 | 82 | 87 | 89 | 84 | 86 | 90 | 76 | 95 |
| rs16887244 (LSM1) | A | 74 | 67 | 82 | 75 | 69 | 83 | 74 | 87 | 77 | 77 | 78 | 77 | 80 | 76 | 82 | 49 |
| rs16897515 (POM121L2) | C | 88 | 96 | 94 | 92 | 91 | 95 | 96 | 85 | 97 | 85 | 93 | 95 | 93 | 99 | 98 | 99 |
| rs17203055 (ARHGAP31) | A | 91 | 90 | 99 | 98 | 94 | 98 | 97 | 98 | 99 | 90 | 93 | 97 | 96 | 92 | 94 | 99 |
| rs17512836 (TCF4) | T | 97 | 100 | 99 | 99 | 100 | 100 | 100 | 99 | 100 | 98 | 99 | 99 | 100 | 100 | 100 | 100 |
| rs17594526 (TCF4) | C | 97 | 95 | 99 | 99 | 97 | 100 | 100 | 100 | 100 | 99 | 90 | 100 | 100 | 100 | 100 | 100 |
| rs17693963 (GPR89P-TRV-AAC1-5) | A | 93 | 99 | 98 | 96 | 99 | 98 | 98 | 100 | 95 | 98 | 95 | 98 | 97 | 0 | 0 | 100 |
| rs3826656 (CD33) | G | 27 | 38 | 56 | 49 | 42 | 54 | 39 | 62 | 62 | 51 | 51 | 51 | 44 | 27 | 32 | 66 |
| rs433598 (ACSM1) | T | 41 | 36 | 65 | 86 | 79 | 77 | 79 | 56 | 76 | 70 | 70 | 76 | 70 | 54 | 74 | 82 |
| rs4420638 (APOC1) | G | 18 | 17 | 14 | 18 | 14 | 17 | 21 | 32 | 27 | 13 | 11 | 14 | 13 | 0 | 0 | 5 |
| rs472926 (CDON) | A | 84 | 79 | 73 | 72 | 78 | 63 | 71 | 66 | 64 | 83 | 76 | 79 | 71 | 67 | 80 | 75 |
| rs4765905 (CACNA1C) | G | 68 | 64 | 88 | 95 | 88 | 88 | 79 | 81 | 94 | 71 | 86 | 85 | 78 | 80 | 87 | 89 |
| rs561655 (PICALM-FNTAP1) | A | 67 | 66 | 64 | 57 | 35 | 43 | 42 | 37 | 48 | 62 | 57 | 57 | 56 | 46 | 50 | 35 |
| rs6859 (PVRL2) | G | 53 | 64 | 67 | 65 | 55 | 73 | 63 | 54 | 52 | 72 | 63 | 71 | 59 | 75 | 62 | 59 |
| rs7341475 (RELN) | G | 84 | 82 | 91 | 93 | 91 | 93 | 87 | 86 | 92 | 83 | 89 | 91 | 90 | 59 | 59 | 79 |
| rs7561528 (LOC105373605) | A | 28 | 29 | 15 | 8 | 18 | 11 | 12 | 9 | 6 | 23 | 11 | 16 | 15 | 9 | 4 | 9 |
Распределение генотипов в популяциях в подавляющем большинстве случаев соответствовало равновесию Харди–Вайнберга (РХВ). В проанализированном массиве данных среди 448 распределений частот генотипов было обнаружено отклонение от РХВ в 16 случаях, что не превышает ожидаемого числа случайных отклонений при 5%-ном уровне значимости. С учетом поправки Бонферрони выявлено четыре статистически достоверных отклонений от равновесия Харди–Вайнберга.
Уровень генетического разнообразия исследуемых популяций Северной Евразии оценивали на основании данных ожидаемой гетерозиготности (Не). Высокое значение средней ожидаемой гетерозиготности (>0.45) показали шесть маркеров из 28: rs10273775 гена CNTNAP2, rs1031381 гена NCAPD3, rs1532278 гена CLU, rs3826656 гена CD33, rs561655 межгенного региона PICALM/RNU6-560P, rs6859 гена NECTIN2, при этом максимальный уровень генетического разнообразия (0.5) не показал ни один из маркеров (рис. 1,а). Максимальное значение ожидаемой гетерозиготности (0.48) показали локусы rs10273775 гена CNTNAP2 и rs561655 межгенного региона PICALM/RNU6-560P. Наименьшее значение ожидаемой гетерозиготности показали два маркера гена TCF4: rs17512836 и rs17594526. В целом 79% маркеров показали уровень гетерозиготности ниже 0.45.
Рис. 1.
Средние значения ожидаемой гетерозиготности. а – отдельно по каждому генетическому маркеру; б – отдельно для каждой популяции Северной Евразии. Для рис. 1, 2: KGN – киргизы северные, KGS – киргизы южные, ALТ – алтайцы, BUR – буряты, KOR – коряки, NIVCHI – нивхи, TUV – тувинцы, HAK – хакасы, HANT – ханты, CHUKCHI – чукчи, YAB – якуты, RUS – русские, KAZAHI – казахи, UDM – удмурты, KETI – кеты, UZ – узбеки.
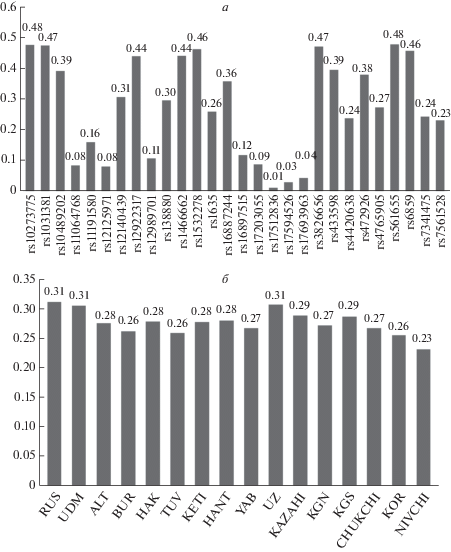
Среди исследуемых 16 популяций Северной Евразии наибольший уровень генетического разнообразия (0.31) показали европеоидные популяции: русские, удмурты и узбеки (рис. 1,б). По-видимому, формирование этих популяций происходило на основе разнородных по происхождению групп. Среди монголоидных популяций у нивхов наблюдался наименьший уровень генетического разнообразия 0.23. Нивхи представляют собой небольшой этнос, изолированный как в языковом, так и в территориальном плане. Вероятно, низкий уровень генетического разнообразия связан с длительной изоляцией этого этноса от контактов с другими популяциями. В настоящей работе наблюдается снижение генетического разнообразия с запада на восток, что также зафиксировано по другим системам маркеров. Хотя для других маркеров были описаны более высокие значения генетического разнообразия для популяций Северной Евразии, тем не менее общие закономерности в структуре генофонда сохраняются. Так, по Х-хромосомным STR и SNP маркерам наименьший уровень генетического разнообразия наблюдался в популяциях коренных этносов Сибири у бурятов (0.746 и 0.437) и тувинцев (0.715 и 0.436), в то время как наибольшим генетическим разнообразием обладала популяция русских – 0.480 по SNP маркерам и 0.779 по STR маркерам [37]. Кроме того, ранее по Y-хромосомным диаллельным маркерам было показано высокое генетическое разнообразие для европеоидных популяций русских (0.799) и удмуртов (0.778), в то время как для нивхов (0.486) было обнаружено низкое разнообразие по этим же маркерам [38].
Из 28 SNP можно выделить две группы маркеров, которые преобладают у европеоидов или у монголоидов. Так, предковые аллели превалируют в монголоидных популяциях по локусам rs433598 гена ACSM1 и rs4765905 гена CACNA1C, в то время как более высокая частота предкового аллеля в европеоидных популяциях наблюдалась для других пяти маркеров: rs12922317 гена SNX29, rs1466662 гена DCHS2, rs1635 гена NKAPL, rs561655 межгенного региона PICALM/RNU6-560P, rs7561528 гена LOC105373605.
Вероятно, все эти данные в совокупности свидетельствуют о популяционной или этнической специфичности этих маркеров. Так, предковый аллель A rs12922317 гена SNX29 преобладает у европеоидных популяций в нашем исследовании (русские – 68%, удмурты – 55%, узбеки – 52%), в то время как у монголоидов частым является альтернативный аллель G, а частота минорного аллеля А варьирует от 18% у нивхов до 45% у кетов.
Генетические взаимоотношения между популяциями и генетическая дифференциация популяций
При анализе генетических взаимоотношений между популяциями по 28 маркерам использовали метод главных компонент, который основан на многомерном анализе частот предковых аллелей. Положение 16 исследованных популяций в пространстве двух главных компонент частот 28 локусов, ассоциированных с заболеваниями ШЗ, БА и когнитивными эндофенотипами, приведено на рис. 2.
Рис. 2.
Расположение исследованных этнических групп в пространстве главных компонент аллельных частот.
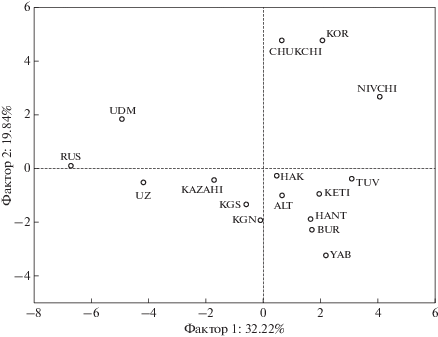
Две первые главные компоненты (ГК) в совокупности объясняют 52.06% вариабельности аллельных частот в изученных популяциях. Расположение 16 популяций в пространстве главных компонент отражает их географическую локализацию и позволяет выделить четыре кластера: популяции Дальнего Востока (чукчи, коряки, нивхи), популяции Сибири (хакасы, тувинцы, кеты, алтайцы, ханты, буряты, якуты), популяции Средней Азии (узбеки, казахи, киргизы северные, киргизы южные) и популяции европейской части России (русские, удмурты).
Генетическую дифференциацию популяций по 28 маркерам рассчитывали с использованием анализа молекулярной дисперсии (AMOVA). Оценку генетической дифференциации проводили с помощью коэффициента Fst. Необходимо отметить, что не все из 28 изученных маркеров показали статистически значимую дифференциацию, по двум локусам различия не являются статистически значимыми (rs10489202 гена MPC2 и rs138880 гена BRD1). Общий уровень генетической дифференциации изученных 16 популяций составил 3.6%. Локус rs138880 гена BRD1 (Fst = –0.00075, р = 0.603) показал более высокое внутрипопуляционное различие, чем межпопуляционное. Значения Fst в суммарной выборке по каждому локусу приведены в табл. 4.
Таблица 4.
Значения Fst в суммарной выборке по каждому локусу
| SNP | Ген | Fst |
|---|---|---|
| rs138880 | BRD1 | –0.00075 |
| rs10489202 | MPC2 | 0.00273 |
| rs17512836 | TCF4 | 0.00758 |
| rs1532278 | CLU | 0.00992 |
| rs12125971 | LOC105378888/PRMT6 | 0.01118 |
| rs11064768 | CCDC60 | 0.01322 |
| rs17693963 | GPR89P/TRV-AAC1-5 | 0.01385 |
| rs6859 | NECTIN2 | 0.01741 |
| rs12989701 | LOC105373605 | 0.01876 |
| rs17203055 | ARHGAP31 | 0.01959 |
| rs472926 | CDON | 0.02034 |
| rs4420638 | APOC1 | 0.02098 |
| rs11191580 | NT5C2 | 0.02105 |
| rs16897515 | POM121L2 | 0.02516 |
| rs10273775 | CNTNAP2 | 0.02799 |
| rs16887244 | LSM1 | 0.03112 |
| rs12140439 | LINC01741/SEC16B | 0.03307 |
| rs1635 | NKAPL | 0.04165 |
| rs561655 | PICALM/RNU6-560P | 0.04655 |
| rs17594526 | TCF4 | 0.04689 |
| rs1031381 | NCAPD3 | 0.04878 |
| rs7561528 | LOC105373605 | 0.0502 |
| rs4765905 | CACNA1C | 0.05852 |
| rs3826656 | CD33 | 0.06428 |
| rs1466662 | DCHS2 | 0.06952 |
| rs7341475 | RELN | 0.07724 |
| rs12922317 | SNX29 | 0.09171 |
| rs433598 | ACSM1 | 0.10987 |
Показано, что наибольший вклад в межпопуляционное разнообразие вносят различия в частотах аллелей локусов rs433598 гена члена семейства ацил-КоА-синтетаз-1 (ACSM1), rs12922317 гена сортирующего нексина-29 (SNX29), rs7341475 гена рилина (RELN), rs1466662 гена протокадгерина-23 (DCHS2) и rs3826656 гена молекулы CD33 (CD33). Белковые продукты этих генов участвуют в процессах развития и функционирования нервной системы. Так, белок протокадгерин-23 вовлечен в процесс аксонального поиска пути и ветвления дендритов, белок рилин контролирует межклеточные взаимодействия, важные для миграции нейронов во время развития мозга, функция белка сортирующего нексина-29 связана с регулированием мембранного транспорта при эндоцитозе в синапсах центральной нервной системы, молекула CD33 принадлежит к семейству иммуноглобулинов и экспрессируется микроглией и макрофагами в головном мозге, а белок ацил-КоА-синтетаза 1 жирных кислот является магний-зависимым ферментом, участвующим в энергетическом метаболизме митохондрий нейронов и глии.
В целом при сравнении общего уровня генетической дифференциации популяций Северной Евразии по маркерам, связанным с заболеваниями, нарушающими когнитивные функции человека, с другими системами маркеров, показано, что он чуть выше значения по Х-хромосомным STR-маркерам, для которых уровень генетической дифференциации был 2.58% [37], и значительно меньше уровня генетической дифференциации по Y-хромосомным диаллельным и STR-маркерам, где уровень генетической дифференциации был 21.61 и 22.25% соответственно [38, 39].
Таким образом, в настоящем исследовании были впервые изучены генетические различия популяций Северной Евразии по полиморфизму генов, связанных с заболеваниями, которые ведут к нарушению когнитивных функций человека. В целом анализ внутрипопуляционного и межпопуляционного генетического разнообразия по 28 маркерам в 16 популяциях отражает закономерности структуры генофонда Северной Евразии, которые описаны по другим системам маркеров: Y STR-маркеры, Х STR-маркеры, Х SNP-маркеры, аутосомные STR-маркеры и митохондриальные маркеры [37–40]. Тем не менее актуальной задачей остается накопление данных из исследований, выполненных не только в рамках GWAS в различных популяциях мира по разным системам маркеров. Но кроме этого результаты нашей работы свидетельствуют о присутствии популяционной специфики в распределении частот некоторых изученных маркеров, связанных с заболеваниями с нарушениями КФ. Данный факт является важным с точки зрения выявления у разных народов закономерностей распространения наиболее часто встречающихся МФЗ с нарушениями КФ человека, в том числе болезни Альцгеймера и шизофрении.
Исследование выполнено за счет гранта Российского фонда фундаментальных исследований (проект № 18-29-13045).
Все процедуры, выполненные в исследовании с участием людей, соответствуют этическим стандартам институционального и/или национального комитета по исследовательской этике и Хельсинкской декларации 1964 г. и ее последующим изменениям или сопоставимым нормам этики.
От каждого из включенных в исследование участников было получено информированное добровольное согласие.
Авторы заявляют, что у них нет конфликта интересов.
Список литературы
Яхно Н.Н., Захаров В.В., Локшина А.Б. и др. Деменции: руководство для врачей, 3-е изд. М.: МЕДпреcс‑информ, 2011. 272 с.
Захаров В.В., Яхно Н.Н. Когнитивные расстройства в пожилом и старческом возрасте. Методическое пособие для врачей. М., 2005. 71 с.
Alzheimer’s Association. Alzheimer’s Disease Facts and Figures // Alzheimers Dement. 2019. V. 15. № 3. P. 321–387.
Heinrichs R.W., Zakzanis K.K. Neurocognitive deficit in schizophrenia: aquantitative review of the evidence // Neuropsychology. 1998. V. 12. № 3. P. 426–445. https://doi.org/10.1037//0894-4105.12.3.426
Kontaxaki M.I., Kattoulas E., Smyrnis N., Stefanis N.C. Cognitive impairments and psychopathological parameters in patients of the schizophrenic spectrum // Psychiatriki. 2014. V. 25. № 1. P. 27–38.
Logue M.W., Schu M., Vardarajan B.N. et al. Multi-Institutional Research on Alzheimer genetic epidemiology (MIRAGE) study group. A comprehensive genetic association study of Alzheimer disease in African Americans // Arch. Neurol. 2011. V. 68. № 12. P. 1569–1579. https://doi.org/10.1001/archneurol.2011.646
Seshadri S., Fitzpatrick A.L., Ikram M.A. et al. Genome-wide analysis of genetic loci associated with Alzheimer disease // JAMA. 2010. V. 303. № 18. P. 1832–1840. https://doi.org/10.1001/jama.2010.574
Shi Y., Li Z., Xu Q. et al. Common variants on 8p12 and 1q24.2 confer risk of schizophrenia // Nat. Genet. 2011. V. 43. № 12. P. 1224–1227.
Kirov G., Zaharieva I., Georgieva L. et al. A genome-wide association study in 574 schizophrenia trios using DNA pooling // Mol. Psychiatry. 2009. V. 14. № 8. P. 796–803. https://doi.org/10.1038/mp.2008.33
Ripke S., Sanders A.R., Kendler K.S. et al. Genome-wide association study identifies five new schizophrenia loci // Nat. Genet. 2011. V. 43. № 10. P. 969–976.
Loo S.K., Shtir C., Doyle A.E. et al. Genome-wide association study of intelligence: Additive effects of novel brain expressed genes // J. Am. Acad. Child Adolesc. Psychiatry. 2012. V. 51. № 4. P. 432–440. https://doi.org/10.1016/j.jaac.2012.01.006
Aberg K.A., Liu Y., Bukszár J. et al. A comprehensive family-based replication study of schizophrenia genes // JAMA Psychiatry. 2013. V. 70. № 6. P. 573–581. https://doi.org/10.1001/jamapsychiatry.2013.288
Stefansson H., Ophoff R.A., Steinberg S. et al. Common variants conferring risk of schizophrenia // Nature. 2009. V. 6. № 460(7256). P. 744–747. https://doi.org/10.1038/nature08186
Børglum A.D., Demontis D., Grove J. et al. Genome-wide study of association and interaction with maternal cytomegalovirus infection suggests new schizophrenia loci // Mol. Psychiatry. 2014. V. 19. № 3. P. 325–333. https://doi.org/10.1038/mp.2013.2
Hu X., Pickering E., Liu Y.C. et al. Meta-analysis for genome-wide association study identifies multiple variants at the BIN1 locus associated with late-onset Alzheimer’s disease // PLoS One. 2011. V. 24. № 6(2):e16616. https://doi.org/10.1371/journal.pone.0016616
O’Donovan M.C., Craddock N., Norton N. et al. Identification of loci associated with schizophrenia by genome-wide association and follow-up // Nat. Genet. 2008. V. 40. № 9. P. 1053–1055. https://doi.org/10.1038/ng.201
Kamboh M.I., Barmada M.M., Demirci F.Y. et al. Genome-wide association analysis of age-at-onset in Alzheimer’s disease // Mol. Psychiatry. 2012. V. 17. № 12. P. 1340–1346.
Naj A.C., Jun G., Beecham G.W. et al. Common variants at MS4A4/MS4A6E, CD2AP, CD33 and EPHA1 are associated with late-onset Alzheimer’s disease // Nat. Genet. 2011. V. 43. № 5. P. 436–441. https://doi.org/10.1038/ng.801
Yue W.H., Wang H.F., Sun L.D. et al. Genome-wide association study identifies a susceptibility locus for schizophrenia in Han Chinese at 11p11.2 // Nat. Genet. 2011. V. 43. № 12. P. 1228–1231. https://doi.org/10.1038/ng.979
Sullivan P.F., Lin D., Tzeng J.Y. et al. Genome-wide association for schizophrenia in the CATIE study: results of stage 1 // Mol. Psychiatry. 2008. V. 13. № 6. P. 570–584. https://doi.org/10.1038/mp.2008.25
Purcell S.M., Wray N.R., Stone J.L. et al. Common polygenic variation contributes to risk of schizophrenia and bipolar disorder // Nature. 2009. V. 460. № 7256. P. 748–752.
Bergen S.E., O’Dushlaine C.T., Ripke S. et al. Genome-wide association study in a Swedish population yields support for greater CNV and MHC involvement in schizophrenia compared with bipolar disorder // Mol. Psychiatry. 2012. V. 17. № 9. P. 880–886. https://doi.org/10.1038/mp.2012.73
Need A.C., Attix D.K., McEvoy J.M. et al. A genome-wide study of common SNPs and CNVs in cognitive performance in the CANTAB // Hum. Mol. Genet. 2009. V. 18. № 23. P. 4650–4661. https://doi.org/10.1093/hmg/ddp413
Cirulli E.T., Kasperaviciūte D., Attix D.K. et al. Common genetic variation and performance on standardized cognitive tests // Eur. J. Hum. Genet. 2010. V. 18. № 7. P. 815–820. https://doi.org/10.1038/ejhg.2010.2
Bertram L., Lange C., Mullin K. et al. Genome-wide association analysis reveals putative Alzheimer’s disease susceptibility loci in addition to APOE // Am. J. Hum. Genet. 2008. V. 83. № 5. P. 623–632. https://doi.org/10.1016/j.ajhg.2008.10.008
Athanasiu L., Mattingsdal M., Kähler A.K. et al. Gene variants associated with schizophrenia in a Norwegian genome-wide study are replicated in a large European cohort // J. Psychiatr. Res. 2010. V. 44. № 12. P. 748–753. https://doi.org/10.1016/j.jpsychires.2010.02.002
Cummings A.C., Jiang L., Velez Edwards D.R. et al. Genome-wide association and linkage study in the Amish detects a novel candidate late-onset Alzheimer disease gene // Ann. Hum. Genet. 2012. V. 76. № 5. P. 342–351. https://doi.org/10.1111/j.1469-1809.2012.00721.x
Shifman S., Johannesson M., Bronstein M. et al. Genome-wide association identifies a common variant in the reelin gene that increases the risk of schizophrenia only in women // PLoS Genet. 2008. V. 4(2):e28. https://doi.org/10.1371/journal.pgen.0040028
Moreno-Grau S., de Rojas I., Hernández I. et al. Genome-wide association analysis of dementia and its clinical endophenotypes reveal novel loci associated with Alzheimer’s disease and three causality networks: The GR@ACE project2019 // Alzheimers Dement. 2019. V. 15. № 10. P. 1333–1347. https://doi.org/10.1016/j.jalz.2019.06.4950
Mathew C.G. The isolation of high molecular weight eukaryotic DNA // Methods Mol. Biol. 1985. V. 2. P. 31–34.
Вагайцева К.В., Бочарова А.В., Марусин А.В. и др. Разработка метода мультиплексного генотипирования полиморфных маркеров генов, ассоциированных с когнитивными способностями // Генетика. 2018. Т. 54. № 6. С. 719–726.
Степанов В.А., Трифонова Е.А. Мультиплексное генотипирование однонуклеотидных полиморфных маркеров методом MALDI-TOF масс-спектрометрии: частоты 56 SNP в генах иммунного ответа в популяциях человека // Мол. биология. 2013. Т. 47. № 6. С. 976–986.
Трифонова Е.А., Попович А.А., Вагайцева К.В. и др. Метод мультиплексного генотипирования полиморфных вариантов генов, ассоциированных с ожирением и индексом массы тела // Генетика. 2019. Т. 55. № 10. С. 1218–1230.
Nei M. Molecular Evolutionary Genetics. N.Y.: Columbia Univ. Press, 1987. 512 p.
Excoffier L., Laval G., Schneider S. Arlequin (version 3.0): An integrated software package for population genetics data analysis // Evol. Bioinform. Online. 2007. V. 1. P. 47–50.
Степанов В.А., Харьков В.Н., Трифонова Е.А., Марусин А.В. Методы статистического анализа в популяционной и эволюционной генетике человека (Наследственность и здоровье): учебно-методическое пособие / Под ред. Пузырева В.П. Томск: Печатная мануфактура, 2014. 100 с.
Вагайцева К.В. Генетическое разнообразие популяций Северной Евразии по STR иSNP маркерам Х-хромосомы и их ДНК-идентификационный потенциал: Дис. … канд. биол. наук. Томск: ФГБНУ “Научно-исследовательский институт медицинской генетики”, 2015. 224 с.
Харьков В.Н. Структура и филогеография генофонда коренного населения Сибири по маркерам Y-хромосомы: Дис. … докт. биол. наук. Томск: ФГБУ “Научно-исследовательский институт медицинской генетики” СО РАМН, 2012. 441 с.
Степанов В.А., Харьков В.Н., Пузырев В.П., Спиридонова М.Г. Генетическое разнообразие линий Y‑хромосомы у народов Сибири. Генофонд населения Сибири. Новосибирск: Изд-во Ин-та археологии и этнографии СО РАН, 2003. С. 147–152.
Голубенко М.В., Еремина Е.Р., Тадинова В.Н. и др. Территориальная дифференциация генофонда народов Сибири и Средней Азии по рестрикционному полиморфизму митохондриальной ДНК // Мед. генетика. 2002. Т. 1. № 3. С. 124–128.
Дополнительные материалы отсутствуют.


